Overview
Fonte: Alexander S. Gold1, Tonya M. Colpitts1
1 Departamento de Microbiologia, Escola de Medicina da Universidade de Boston, Laboratórios Nacionais de Doenças de Infecções Emergentes, Boston, MA
Descoberta pela primeira vez por Lederberg e Tatum em 1946, a conjugação é uma forma de transferência genética horizontal entre bactérias que depende do contato físico direto entre duas células bacterianas (1). Ao contrário de outras formas de transferência genética, como transformação ou transdução, a conjugação é um processo natural em que o DNA é secretado de uma célula doadora para uma célula receptora de forma unidirecional. Essa direcionalidade e a capacidade desse processo de aumentar a diversidade genética das bactérias deram à conjugação a reputação como uma forma de "acasalamento" bacteriano, que acredita-se ter contribuído muito para o recente aumento de bactérias resistentes a antibióticos (2, 3). Ao utilizar pressões seletivas, por exemplo, o uso de antibióticos, a conjugação tem sido manipulada para uso em ambiente de laboratório, tornando-se uma ferramenta poderosa para a transferência horizontal de genes entre bactérias e, em alguns casos, de bactérias para leveduras, plantas e células animais (4). Além das aplicações em laboratório, a transferência de genes bacterium-eucayote por conjugação é uma avenida emocionante de transferência de DNA com uma infinidade de possíveis aplicações biotécnicas e implicações naturais (5).
Acredita-se que a conjugação funcione por um "mecanismo de duas etapas" (6). Primeiro, antes que qualquer DNA possa ser transferido, a célula doadora deve fazer contato direto entre células com o receptor. Esse processo tem sido caracterizado melhor em bactérias gram-negativas, sendo a mais estudada Escherichia coli. O contato celular-célula é estabelecido pela presença de uma complexa rede de filamentos extracelulares no doador conhecido como pilus sexual, um elemento conjugado codificado pelo gene transferível conhecido como fator F (fertilidade) (7, 8). Além de estabelecer contato entre doador e receptor, diversas proteínas são transportadas através do pilus sexual para o citoplasma receptor, formando um conduíte tipo IV (T4SS) entre as duas células, estrutura necessária para a segunda etapa de conjugação, transferência de DNA (6). Combinando essa função do pilus sexual com a replicação do DNA, a célula doadora é capaz de transferir DNA na forma de um elemento transposável, como um plasmídeo ou transposon, para o receptor por um modelo de "atirar e bombear" (6). Neste caso, o "tiro" é o transporte da proteína piloto, com DNA vinculado, pelo T4SS para a célula receptora, e o "bombeamento" é o transporte ativo de DNA para o receptor, um processo dependente do T4SS e catalisado por proteínas de acoplamento (6). O maquinário utilizado neste processo é composto por uma origem da sequência de transferência(oriT),que deve ser fornecida pelo DNA em genes cis e trans, que codificam um complexo de relaxase, formação de par de mate e proteína de acoplamento tipo IV, e pode estar presente em cis ou trans (9). Esta relaxase corta o local de nic dentro da sequência oriT e se prende covalentemente ao final da cadeia transferida para produzir o relaxosome, um complexo de relaxamento de DNA de uma única cadeia com outras proteínas auxiliares (9). Uma vez formado, o relaxosome conecta-se ao complexo de formação de par de acasalamento, através da proteína de acoplamento tipo IV, que permite a transferência do complexo ssDNA-relaxase para células receptoras pelo T4SS (10). Uma vez no citoplasma do receptor, o DNA pode se integrar ao genoma receptor ou existir separadamente na forma de um plasmídeo, qualquer um dos quais permite a expressão de seus genes.
Neste experimento, a cepa de doadores de conjugação amplamente utilizada E. coli WM3064 foi usada para transferir a codificação genética para resistência à ampicilina à cepa receptora E. coli J53. Enquanto ambas as cepas das bactérias gram-negativas eram resistentes à tetraciclina, apenas a cepa doadora WM3064 tinha o gene para resistência à ampicilina, codificada no vetor pWD2-oriT, e era auxotrófica ao ácido diaminopimelic (DAP) (11-13). Este experimento consistiu em duas etapas principais, a preparação de cepas de doadores e receptores, seguidas pela transferência do gene de resistência à ampicilina de doador para receptor por conjugação (Figura 1).
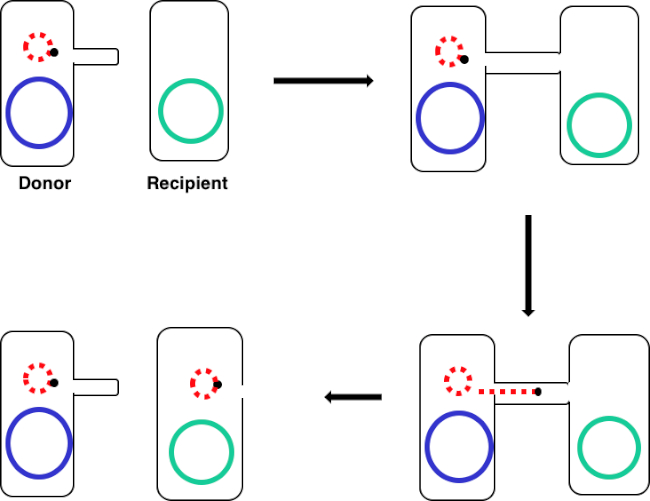
Figura 1: Esquema de conjugação. Este esquema mostra a transferência bem sucedida de um plasmídeo, apenas um exemplo de um elemento de DNA transposável, de uma célula doadora para uma célula receptora usando conjugação. Após o contato com a célula receptora pela célula doadora através do pilus sexual, o plasmid replica-se pela replicação do círculo de rolamento, move-se através do complexo multiproteína que une as duas células, e forma um novo plasmídeo de comprimento completo na célula receptora.
Ao incubar uma mistura de células doadoras e receptoras, em seguida, sucessivamente emplacando essas células na presença de tetraciclina e DAP, isso permitiu a transferência bem sucedida do gene de resistência à ampicilina. Sucessivamente, as células de revestimento cultivadas a partir dessa mistura na presença de tetraciclina e ampicilina, removeram todas as células doadoras devido à falta de DAP e de quaisquer células receptoras que possam não ter adquirido o gene de resistência à ampicilina, produzindo bactérias estritamente receptoras da cepa J53 que adquiriram resistência à ampicilina (Figura 2). Uma vez realizada, a transferência bem sucedida do gene de resistência à ampicilina foi confirmada pelo PCR. Desde que a conjugação foi bem sucedida, a cepa J53 de E. coli continha pWD2-oriT e era resistente à ampicilina, e a codificação genética para essa resistência é detectável pelo PCR. No entanto, se não tivesse havido nenhuma detecção do gene de resistência à ampicilina e a ampicilina ainda funcionaria como um antibiótico eficaz contra a cepa J53.
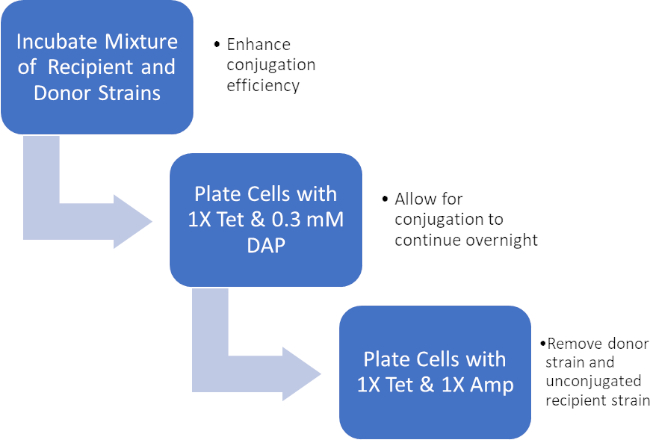
Figura 2: Esquema de protocolo. Este esquema mostra uma visão geral do protocolo apresentado.
Figura 3A: A confirmação da conjugação bem sucedida pela PCR. A) Os estoques congeladores das amostras de controle conjugadas e negativas foram listrados em placas de ágar e uma colônia foi selecionada (vermelha) para isolamento de DNA.
Procedure
1. Configuração
- Autoclave aproximadamente 1L de médium Luria-Bertani (LB). Este LB estéril será usado para fazer aproximadamente 5 mL de LB contendo ácido diaminopimelic de 0,3 mM (DAP).
- Recolher as seguintes placas: placas de ágar LB com 1X Tet e 0,3 mM DAP, placas de ágar LB apenas com 1X Tet e placas de ágar LB apenas com 1X Amp/Tet.
- Certifique-se de que um pouco de glicerol e uma caixa de pontas de pipeta de plástico pré-esterilizadas estejam próximas.
- Antes de iniciar qualquer trabalho envolvendo micróbios, esterilize o espaço de trabalho com 70% de etanol. Use sempre os equipamentos de proteção pessoal necessários, incluindo um jaleco e luvas.
- Uma vez terminado, esterilize todas as superfícies e luvas com 70% de etanol e lave as mãos.
2. Preparação da cepa de doadores e receptores
- Prepare 5 mL culturas bacterianas das cepas de doadores e receptores e cultivá-las durante a noite a 37 °C com aeração e agitação a 220 rpm. A cepa de doadores deve ser cultivada em LB com 0,3 mM DAP.
- Gire 1 mL de ambas as culturas (~3000 rpm por 5 minutos) e lave as células com PBS.
- Resuspend as células em 500 μL de salina tamponada de fosfato (PBS).
3. Conjugação
- Combine 50 μL de células receptoras com 50 μL de células doadoras em um tubo de microcentrifuus e misture por pipetação.
- Incubar a mistura celular por 1 hora a 37 °C. Isso aumentará a eficiência da conjugação antes e durante a próxima etapa de chapeamento.
- Pipet 100 μL da mistura celular em uma placa de ágar com 1X Tet e 0,3 mM DAP. Não se espalhe na placa.
- Pipet 100 μL de cultura celular receptora em uma placa de ágar com 1X Tet e 0,3 mM DAP. Não se espalhe na placa.
- Incubar ambas as placas durante a noite a 37 °C.
- Raspe a mistura de células de conjugação e a cultura celular receptora com um raspador de células estéril. Transfira as células para o tubo de microcentrifusagem estéril e resuspenque as células em 1 mL de PBS.
- Vórtice das células e gire-as suavemente para baixo (~3000 rpm por 5 minutos).
- Resuspense as células em 1 mL de PBS.
- Aplaque a mistura da célula de reação de conjugação em uma placa de ágar LB apenas com 1X Amp/Tet. Nesta placa, espera-se que apenas as bactérias receptoras que obtiveram sucesso do gene de resistência amp através da conjugação cresçam.
- Coloque as células receptoras em uma placa de ágar LB apenas com 1X Tet. Nesta placa, espera-se que apenas bactérias receptoras não julgadas, que não possuem o gene de resistência amp, cresçam.
- Incubar as placas durante a noite a 37 °C.
- Escolha colônias únicas de ambas as placas e use-as para cultivar culturas durante a noite em 5 mL de mídia (37 °C com aeração a 220 rpm).
4. Isolamento do DNA
- Isole o DNA das culturas previamente preparadas usando 4,5 mL do volume total de cultura por miniprep de DNA.
- Para isso, elute o DNA usando 35 μL de água sem nuclease.
- O DNA puro gerará uma razão de absorção (A260/280) de aproximadamente 1,8.
- Use os 0,5 mL restantes de cada cultura para preparar os estoques de glicerol, fazendo uma mistura de cultura bacteriana 1:1 e 100% glicerol.
- Armazene os estoques do congelador a -80 °C.
5. Confirmação da Conjugação de Transferência Plasmid pela PCR
- Prepare duas misturas mestres pcr, cada uma com um conjunto diferente de primers para frente e para trás, uma visando um segmento de 500 pares de base dentro do gene de resistência à ampicilina e a outra visando um segmento dentro de um gene de limpeza.
- Os primers genéticos de limpeza foram projetados para amplificar um segmento de DNA dentro da codificação genética bacteriana para o DNA gyrase B (14).
- Os seguintes volumes de reagente foram utilizados para preparar 90 μL de cada mix mestre:
7,5 μL de 10 μM primer para frente
7,5 μL de primer reverso de 10 μM
75 μL de 2X PCR Master Mix
- Prepare as seguintes seis reações de PCR usando 15 μL master mix, 10 ng de DNA de modelo e água sem nuclease até um volume final de 25 μL.
DNA de reação conjugal e primers de ampicilina
DNA de reação conjugal e primers de limpeza
Dna de controle negativo e primers de ampicillina
DNA de controle negativo e primers de limpeza
Sem primers de DNA e ampicillina
Sem dna e primers de limpeza - Transfira essas reações para uma máquina PCR com o bloco pré-aquecido para 98 C e comece a termociclamento sob as seguintes condições:
98 °C por 30 segundos
25-35 ciclos de 98 °C para 5-10 segundos, 45-72 °C por 10 a 30 segundos, e 72 °C para 15-30 segundos por kb
72 °C por 5-10 minutos
Mantenha a 4 °C - Carregue todas as seis reações do PCR em um gel de 1% de agarose e execute a ~150V por cerca de 20 minutos.
- Visualize o produto do PC usando um iluminador UV.
Células bacterianas, como e. coli,são capazes de transferir informações genéticas de célula para célula. A conjugação difere de outros mecanismos de transferência de DNA, como transdução ou transformação, na medida em que requer contato físico entre as células.
Para prosseguir, a conjugação requer uma célula doadora que expresse a fertilidade, ou F, fator e uma célula receptora sem ela, uma célula F menos. O processo requer duas etapas. O primeiro é o estabelecimento de contato direto entre células. Para isso, a célula doadora gera uma estrutura fisomada extracelular chamada pilus sexual. Chama-se isso, uma vez que a conjugação é uma forma de acasalamento para bactérias sexualmente reprodutificadas, mas deve-se notar que não é verdadeira reprodução sexual, pois nenhum gametas é trocado e nenhum descendente é formado.
O segundo passo é a entrega de DNA para a célula receptora. Após o pilus sexual estabelecer contato entre duas células, um conduíte chamado sistema de secreção tipo IV é construído permitindo a transferência de DNA. A célula doadora então começa a replicar o DNA extracromosomal que será transferido selecionado com base na presença de um elemento genético conhecido como OriT ou origem da transferência. Uma extremidade do DNA recém-replicado é enfiada no conduíte através da ligação de proteína de DNA. À medida que o DNA é ainda mais replicado, ele é bombeado através do canal, facilitado por um complexo de proteínas codificadas por genes localizados perto do OriT. Uma vez que o DNA é totalmente transferido, ele vai formar um plasmídeo cromossômico extra, ou pode se integrar ao cromossomo da célula receptora. Seja qual for o ponto final do DNA transferido, os genes que codifica serão então expressos. Esta expressão genética pode ser usada para confirmar a conjugação bem sucedida.
Por exemplo, considere um cenário em que a cepa do doador expressa resistência à ampicilina e passa isso no DNA conjugado para a bactéria receptora, mas a cepa receptora também tem um gene de resistência à tetraciclina não presente no doador. Neste caso, quando as células são banhadas em mídia LB contendo tetraciclina e ampicilina, as colônias devem crescer apenas a partir de bactérias conjugadas com sucesso, que estarão expressando ambos os fenótipos de resistência. Para confirmar ainda mais a conjugação bem sucedida, o DNA plasmídeo dessas colônias pode ser colhido e, em seguida, uma seção de DNA específica para o plasmídeo transferido pode ser amplificada usando reação em cadeia de polimerase, ou PCR. Quando o produto PCR é executado em um gel de eletroforese ao lado de uma escada de tamanhos padrão, um fragmento PCR de um tamanho conhecido deve ser visível no gel, confirmando ainda mais a conjugação bem sucedida. Neste experimento, um plasmídeo será usado para transferir o gene de resistência à ampicilina através da conjugação de uma cepa de doador para uma cepa receptora resistente à tetraciclina. Depois disso, para confirmar a conjugação, a mistura de conjugação será incubada em uma placa contendo ambos os antibióticos deixando apenas as bactérias transformadas. Finalmente, a conjugação bem sucedida será confirmada com PCR.
Antes de iniciar o procedimento, coloque os equipamentos de proteção individual adequados, incluindo um jaleco e luvas. Em seguida, esterilize o espaço de trabalho usando 70% de etanol para limpar a superfície.
Neste procedimento, o gene de resistência à ampicilina será transferido da cepa WM3064 de E. coli para a cepa J53 de E. coli via conjugação. A cepa de doadores WM3064 é resistente à tetraciclina e ampicillina e requer ácido diaminopimelic, ou DAP, para crescer. A cepa receptora J53 é resistente apenas à tetraciclina e não requer o crescimento do DAP. Isso significa que as células conjugadas com sucesso devem ser resistentes à tetraciclina e à ampicilina e podem crescer sem DAP.
Prepare a cultura da cepa do doador inoculando cinco mililitros de LB contendo 0,3 milias de DAP com um pedaço do estoque de glicerol de doação congelado. Em seguida, prepare a cepa do receptor inoculando cinco mililitros de caldo LB sem DAP com um pedaço do estoque de glicerol de cepa do receptor congelado. Cresça essas culturas durante a noite a 37 graus Celsius com aeração e tremendo a 220 RPM em uma incubadora tremendo. Uma vez que as culturas tenham crescido para um OD 600 de dois, remova um mililitro de cultura de cada um e coloque isso em dois novos tubos de microcentrifuuge separados de 1,5 mililitro. Em seguida, centrifugar essas alíquotas a 3000 RPM por cinco minutos para pelotar as células bacterianas. Descarte o supernatante e lave cada pelota com 250 microlitres de 1X PBS. Centrifugar as amostras novamente e, após descartar o supernascer, resuspend cada pelota em 500 microliters de PBS.
Para iniciar o procedimento de conjugação, primeiro combine 50 microliters de células receptoras com 50 microliters de células doadoras em um tubo de microcentrifuuge de 1,5 mililitro e misture por tubos para cima e para baixo suavemente. Em seguida, pipeta 100 microliters da cultura celular receptora em outra placa de tetraciclina 1X contendo DAP. Em seguida, prepare seu controle negativo pipetando 100 microliters da cultura celular receptora apenas em uma placa de ágar não-seletiva contendo DAP. Em seguida, incubar as placas de conjugação e controle negativo durante a noite a 37 graus Celsius.
No dia seguinte, pegue um raspador de células estéril e colhia células da placa de conjugação coletando colônias. Em seguida, transfira as colônias para um tubo de microcentrífuga de 1,5 mililitro estéril contendo um mililitro de 1X PBS. Repita este processo para coletar as células receptoras da outra placa.
Depois disso, o vórtice as amostras para misturar. Depois de misturar, transfira os tubos para uma centrífuga para de pelotas suavemente as células. Descarte o supernasce, depois lave as pelotas celulares em um mililitro de PBS e vórtice dos tubos para resuspensar as células. Pellet as células novamente por centrifugação. Descarte o supernatante novamente e resuspenque ambas as pelotas celulares em um mililitro de PBS. Agora, usando uma ponta de pipeta estéril, placa 100 microliters da mistura de célula de reação de conjugação em uma placa de ágar LB sem DAP contendo 1X tetraciclina e 1X ampicillina. Repita o método de chapeamento usando 100 microliters de uma diluição dez vezes da mesma mistura celular em PBS em outra placa de ágar LB sem DAP contendo tetraciclina 1X e 1X ampicillina.
Finalmente, pipeta 100 microliters da mistura de célula de controle negativo em uma única placa de ágar LB apenas com 1X tetraciclina. Após a incubação durante a noite a 37 graus Celsius, as colônias devem ser visíveis. Usando uma ponta de pipeta estéril, escolha uma única colônia da placa de reação conjugal e adicione-a a um tubo contendo cinco mililitros de mídia LB seletiva contendo ambos os antibióticos. Em seguida, repita o isolamento da colônia selecionando uma única colônia da placa celular receptora. Cresça essas culturas durante a noite a 37 graus Celsius com aeração a 220 RPM.
No dia seguinte, limpe a bancada com 70% de etanol e retire as placas da incubadora. Use um mini kit de preparação de DNA para isolar DNA de 4. 5 mililitros de cada cultura de acordo com as instruções do fabricante. Depois de completar a mini preparação de DNA, elute o DNA usando 35 microliters de água sem nuclease. Finalmente, use o 0 restante. 5 mililitros de cada cultura para preparar um mililitro de glicerol estoques adicionando 0,5 mililitros de 100% glicerol para uma diluição um-para-um. Coloque essas alíquotas a menos 80 graus Celsius para armazenamento até que seja necessário.
Para confirmar a conjugação bem sucedida por PCR, primeiro prepare um mix mestre de PCR adicionando 75 microliters de mistura master 2X PCR a um tubo de microcentrifuuge. Em seguida, adicione 7,5 microliters cada um de um primer dianteiro de 10 micromolar e um primer reverso de 10 micromolar projetado para amplificar o gene de resistência à ampicilina do plasmídeo. Em seguida, prepare uma segunda mistura mestre pcr adicionando 75 microliters de 2X PCR master mix a um tubo de microcentrifuuge e, em seguida, adicionando 7,5 microliters cada um de um primer dianteiro de 10 micromolar e 10 primer reverso micromolar projetado para amplificar um gene de limpeza, neste caso DNA gyrase B.
Agora, adicione 15 microliters da primeira mistura mestre a um tubo PCR e, em seguida, adicione 10 nanogramas, aproximadamente dois microliters do modelo DNA experimental ao mesmo tubo. Leve a reação até um volume final de 25 microliters com água sem nuclease. Repita estas etapas para produzir as cinco reações restantes, de modo que os tubos contenham os componentes aqui mostrados. Agora, transfira essas reações para um termociclador com o bloco pré-aquecido a 98 graus Celsius e, em seguida, inicie o programa. Após a conclusão do PCR, remova os tubos da máquina. Em seguida, carregue dois microliters de cada reação misturados com dois microliters de corante de carga e quatro microliters de um marcador de peso molecular em poços consecutivos de um gel de 1% de agarose. Coloque o gel para rodar a 150 volts por 20 minutos. Por fim, visualize o gel usando um iluminador UV.
Neste experimento, a transferência bem sucedida do gene de resistência à ampicilina via conjugação foi confirmada via PCR. Aqui, uma banda de aproximadamente 500 pares de base deve ser observada no poço contendo o DNA conjugado e primers de ampicilina, bem dois neste exemplo. Um gene de limpeza, o DNA gyrase B, foi carregado em poços três e cinco com DNA conjugado e DNA celular receptor, respectivamente. Bandas observadas nesses poços atuam como um controle positivo para garantir que o modelo de DNA estivesse presente e que o PCR tenha sido bem sucedido. As bandas não devem ser observadas no poço que contém a reação para o DNA celular receptor e o par de primer ampicillin, bem quatro neste exemplo, porque as células receptoras não são resistentes à ampicilina. Além disso, nenhuma banda deve ser observada nas reações sem DNA de modelo, poços seis e sete aqui. Se essas condições forem atendidas, isso confirmará a transferência bem sucedida do gene de resistência à ampicilina, conferindo resistência à ampicilina da cepa WM3064 de E. coli à cepa J53 de E. coli.
Subscription Required. Please recommend JoVE to your librarian.
Results
Se a conjugação for bem sucedida, um produto PCR de banda de 500 pares base será observado no poço em que a reação pcr 1 foi carregada (Bem #2 na Figura 3B), enquanto nenhuma banda será observada no poço em que a reação pcr 3 foi carregada (Bem #4 na Figura 3B). A presença desta banda confirma a transferência bem sucedida do gene de resistência à ampicilina, conferindo assim resistência à ampicilina à cepa J53 de E. coli.

Figura 3B: A confirmação da conjugação bem sucedida por PCR. B) A análise da PCR foi feita utilizando-se DNA isolado da colônia selecionada. O conteúdo de cada poço é o seguinte: 1) escada de DNA, 2) DNA de conjugação e primers de ampicilina, 3) DNA de conjugação e primers de limpeza, 4) Dna de controle negativo e primers de ampicillina, 5) DNA de controle negativo e primers de limpeza, 6) Sem primers de DNA e ampicilina, e 7) Sem primers de DNA e controle negativos. A presença de um produto PCR de banda base de ~ 500 pares base da reação PCR 1 (bem 2), e a falta deste produto da reação PCR 3 (bem 4), confirma a conjugação bem sucedida.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
A conjugação é um processo natural de transferência genética horizontal que depende do contato direto celular-célula de uma célula doadora e de uma célula receptora. Esse processo é compartilhado entre todos os tipos de bactérias e tem sido fundamental na evolução bacteriana, principalmente a resistência a antibióticos. Em laboratório, a conjugação pode ser usada como um método eficaz de transferência genética que é muito menos disruptivo quando comparado a outras técnicas. Fora do laboratório, a capacidade de transferir DNA de bactérias para eucariotes via conjugação oferece uma nova e excitante via de terapia genética e entender as implicações dessas transferências genéticas que ocorrem naturalmente, por exemplo, a relação entre infecção bacteriana e câncer, é uma área de pesquisa rapidamente emergente.
Subscription Required. Please recommend JoVE to your librarian.
References
- Lederberg J, Tatum, E.L. Gene recombination in Escherichia coli Nature. 1946;158:558.
- Holmes R.K. J, M.G. Genetics: Exchange of Genetic Information. 4th Edition ed. Baron S, editor. Galveston, TX: University of Texas Medical Branch at Galveston; 1996.
- Cruz F, Davies, J. Horizontal gene transfer and the origin of species: lessons from bacteria. Trends in Microbiology. 2000;8:128-33.
- Llosa M, Cruz, F. Bacterial conjugation: a potential tool for genomic engineering. Ressearch in Microbiology. 2005;156:1-6.
- Lacroix B, Citovsky, V. Transfer of DNA from Bacteria to Eukaryotes. mBio. 2016;7(4):1-9.
- Llosa M, et al. Bacterial conjugation: a two-step mechanism for
- DNA transport. Molecular Microbiology. 2002;45:1-8.
- Grohmann E, Muth, G., Espinosa, M. Conjugative Plasmid Transfer in Gram-Positive Bacteria. Microbiology and Molecular Biology Reviews. 2003;67:277-301.
- Firth N, Ippen-Ihler, K, Skurray, RA. Structure and function of the F factor and mechanism of conjugation. Escherichia coli and salmonella: cellular and molecular biology. 1996;2:2377-401.
- Smillie C, Garcillan-Barcia MP, Francia MV, Rocha EPC, De La Cruz F. Mobility of Plasmids. Microbiology and Molecular Biology Reviews. 2010;74(3):434-52.
- Cascales E. Definition of a Bacterial Type IV Secretion Pathway for a DNA Substrate. 2004;304(5674):1170-3.
- Wang P, Yu Z, Li B, Cai X, Zeng Z, Chen X, et al. Development of an efficient conjugation-based genetic manipulation system for Pseudoalteromonas. Microbial Cell Factories. 2015;14(1):11.
- Yi H, Cho YJ, Yong D, Chun J. Genome Sequence of Escherichia coli J53, a Reference Strain for Genetic Studies. Journal of Bacteriology. 2012;194(14):3742-3.
- Baumann RLB, E. H.; Wiseman, J. S.; Vaal, M.; Nichols, J. S. Inhibition of Escherichia coli Growth and Diaminopimelic Acid Epimerase by 3-Chlorodiaminopimelic Acid. Antimicrobial Agents and Chemotherapy 1988;32:1119-23.
- Rocha D, Santos, CS, Pacheco LG. Bacterial reference genes for gene expression studies by RT-qPCR: survey and analysis. Antonie Van Leeuwenhoek. 2015;108:685-93.




