Introduction
Fosfatidyletanolamin metyltransferas (PEMT) enzymer katalyserar kovalent bindning av en eller flera metylgrupper som använder S -adenosylmethionine (SAM) som metylgruppsdonator på PE, monometyl-PE eller dimetyl-PE till bildning av monometyl-PE, dimetyl-PE och / eller fosfatidylkolin (PC). Dessa enzymer är nästan allmänt förekommande i djurceller och svampar. De kan också hittas i vissa växter 1 och cirka 10% av bakterier, särskilt de som interagerar med eukaryoter 2.
PEMTs är relevanta för biologin av cellen, inte bara genom att bidra till produktionen av PC, som är den viktigaste lipidklassen i djurceller, utan även genom att uppfylla andra viktiga cellulära funktioner. Hos däggdjur är PEMTs uttrycks huvudsakligen i levern där de behövs för normal utsöndring av mycket low-density lipoprotein och de bidrar också till dietinducerad övervikt 3, åderförkalkning 4, och insulin motståning 5. Dessutom är däggdjurs PEMT också uttryckt i adipocyter, om än i lägre nivåer och medverka i fett nedfall 6, 7. PEMT roll i utvecklingen av cancer 8, apoptos 9, och celltillväxt 10 har också påvisats. I bakterier har PEMT enzymer visats vara viktiga för normal celltillväxt 2, virulens 2, och symbios med värdväxten 11.
Målet och syftet med det nuvarande protokollet är att mäta PEMT aktivitet från helcellextrakt utan att behöva rena enzymet. Två distinkta protokoll har utvecklats för att mäta PEMT aktivitet. Den första och vanligaste mäter överföringen av tritierad metylgruppen från radioaktivt SAM till PE, som är ämnet för denna artikel. Detta protokoll har ursprungligen utvecklats för att mäta PEMT aktivitet från jäst 12 och däggdjursceller (lever) 13 för att få en understAnding PC biosyntesen i dessa celler samt att bestämma specificiteten av dessa enzymer. Senare har denna teknik tillämpats på andra celltyper såsom bakterier 2 (med användning av ett basiskt pH-värde för analysen fastän 15) och protozoparasiter 14. Denna teknik kan användas med helcellextrakt liksom renat enzym, och kan potentiellt tillämpas på alla cellextraktet systemet. En icke radioaktiv analys har också utformats som förlitar sig på den enzymatiska kvantifiering av S -adenosylhomocysteine, den transmetyleringsaktivitet produkten av SAM 16. Den senare analysen kan vara mer praktiskt eftersom det inte innebär radioaktivitet men det är endast lämplig för renade enzymer.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Cellextrakt Framställning
- Odla Leishmania-celler i en steril plastflaska försluten med lufttät lock vid 26 ° C i ett medium tillverkat av 1x M199 supplementerat med 20 mM HEPES pH 7,4, 100 U / ml penicillin, 100 | ig / ml streptomycin, 5 | ig / ml heme , 0,35 g / I NaCOs 2 H, 0,1 mM adenin, och 2 | ig / ml biopterin utan skakning. Skörda cellerna genom centrifugering vid 1500 g under 5 min vid 4 ° C när de når en celltäthet av 1-2 x 10 7 / ml.
- Kassera supernatanten med en pipett och tvätta cellerna genom att återsuspendera cellpelleten med en serologisk pipett i halv av kulturens volym kall fosfatbuffrad koksaltlösning (137 mM NaCI, 2,7 mM KCI, 10 mM Na 2 HPO 4, 1,8 mM KH 2 PO 4, pH 7,4). Avyttra cellsupematanter enligt BL2 säkerhetsanvisningar.
- Centrifugera cellerna igen vid 1500 g under 5 min vid 4 ° C. Kassera supernatanten med en pipett. Fortsätt to nästa steg eller knäppa frysa cellpelleten i flytande kväve för långtidslagring vid -80 ° C (upp till tre månader).
- Förbered 2x lysbuffert (0,5 M sackaros, 0,1 M TrisHCl, pH 7,5, 2 mM EDTA, och 2x proteasinhibitorcocktail) och hålla den vid 4 ºC på is.
- Resuspendera cellpelleten (färsk eller frusen) i lika stor volym av 2x lyseringsbuffert. Lägg 1x volym av glaspärlor. Skaka kraftigt vid 4 ° C i 10 min.
- Tillsätt 2 volymer av 1 x lyseringsbuffert och blanda. Centrifugera cellextrakt vid 1500 xg vid 4 ° C i 10 min för att pelletera obrutna celler och kärnor.
- Överför supernatanten med en pipett till en frisk sval centrifugrör och hålla cellextrakt på is till dess att experimentet.
2. Bestäm proteinkoncentration av cellextraktet Använda Protein Uppskattning Kit Såsom bicinkoninsyra analys
- Förbered bicinkoninsyra (BCA) lösning (1 ml / rör) genom blandning BCA och koppar (II) sulfate i ett förhållande av 49: 1 (volym / volym).
- Förbered proteinstandarder av 0, 10, 20, 30, 40, 50, och 60 | ig / ml genom spädning av ett 10 mg / ml bovint serumalbumin (BSA) stamlösning i 1 ml alikvoter av BCA lösningen.
- Tillsätt 2 | j, l av celler extrakt i 1 ml BCA lösning i dubbletter. Inkubera standarder och proteinprover för 10 min i en förvärmd 60 ° C vattenbad.
- Överför proverna till is under 3 min. Mät absorbansen hos de standarder och proteinprover med en spektrofotometer vid en våglängd av 562 nm.
- Beräkna proteinkoncentrationen av cellextrakten med användning av BSA-standarden som referens, såsom beskrivs i tillverkarens protokoll. Späd cellextrakt till en proteinkoncentration av 10 mg / ml med 1 x lyseringsbuffert.
3. enzymatisk analys i 200 pl per Tube
OBS: Bär följande steg i en kemisk huva.
- Testa varje prov i två exemplar på en 15 mlkoniskt rör. Förbered 20 l 1 M TrisHCl pH 7,5 per rör och hålla den på is. Bered 2 ml kloroform / metanol (1: 1 (vol / vol); stopplösning) vid RT för varje rör.
- Pipett 20 pl 1 M TrisHCl pH 7,5 i varje 15 ml koniska rör på is.
- Följ riktlinjerna strålsäkerhet härifrån. Lägg motsvarande 0,06 ^ M (0,2 | iCi) S - [metyl-3H] adenosyl-L-metionin och 50 | iM kall SAM per rör för totalt 50,06 iM av SAM. Lägg till x il kallt vatten där x = 200- (20 (för buffert) + 20 (för cellextrakt) + volym kallt och radioaktivt SAM) per rör.
- Överför varje koniskt rör till en förvärmd 30 ° C vattenbad. Lägg till varje rör 20 pl cellextrakt (ekvivalent av 200 | j, g protein) för att starta reaktionen. Inkubera under den önskade tiden (0 till 45 min).
- Stoppa reaktionen genom att tillsätta 2 ml kloroform / metanol (1: 1; volym / volym; stopplösning) till varje rör. Överför det koniska röret till RT (20-256, C).
4. lipidextraktion
OBS: Bär följande steg i en kemisk huva.
- Lägg 700 | il vatten till varje rör innehållande den enzymatiska reaktionsprov. Skaka kraftigt under 30 sekunder. Centrifugera vid 1500 xg under 5 min vid RT för att separera den organiska från vattenfasen.
- Överför den undre organiska fasen in i en ny 15 ml koniska rör med en pipett. Tillsätt 1 ml vatten till varje "lägre fas" innehållande röret och virvla kraftigt under 30 sek. Centrifugera åter vid 1500 xg under 5 minuter för att separera den organiska från vattenfasen.
- Överför den undre organiska fasen i en scintillations-rör med en pipett. Torra proverna under en ström av N2. Avyttra vattenfaserna innehåller den icke-införlivade radioaktiva SAM och radioaktiva koniska rör som per riktlinjer strålnings.
- Tillsätt 2 ml / rör av scintillationsvätska. Mät den inkorporerade radioaktiviteten med en scintillationsräknare counter enligt tillverkarens protokoll och instrumentets användning.
- Beräkna den enzymatiska aktiviteten i nmol / mg protein med användning av följande allmänna ekvation:
cpm värde x 10 3 x [totalt (radioaktivt och kall) koncentrationen av SAM (mM)] x 5 specifik aktivitet av radioaktivt SAM (Ci / mmol) x [koncentration av radioaktivt SAM (mM)]
OBS: Ovanstående protokoll kan ändras för SAM koncentrationsberoende eller ett protein beroende PEMT analys. För SAM koncentrationsberoende enzymatisk analys, är tid hålls konstant (15 min, vilket är i det linjära intervallet) och olika mängder kallt SAM sätts till analysen samtidigt med avseende på protein beroende PEMT analys, SAM koncentration (vi valde 0,05 mM) och tid hålls konstanta (15 min). Även pH hos bufferten kan bytas ut vid behov, om vissa PEMT enzymer har olika optimala pH-värde.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Figur 1 visar en tidsberoende PEMT analys, som genomfördes med Leishmania helcellextrakt som en enzymkälla använder endogen PE som ett substrat. Mängden radioaktivitet i den organiska fasen kvantifierades genom scintillationsräkning. De resulterande numren användes för att beräkna mängden tritierad metylgrupper överförs till PE. Den PEMT aktiviteten var linjär för ca 20 min. Den nådde sedan en platå vid omkring 30 minuter, varefter den stannade konstant under ytterligare 15 minuter. Som väntat var PEMT aktivitet detekterades inte när inga cellextrakt sattes till analysen (figur 2). Vidare var denna aktivitet upphävdes i närvaro av 100 | iM octadecyltrimethylammonium bromid, som är en hämmare av L. större PE metyltransferaser LMJ PEM1 och LMJ PEM2 14. PEMT aktivitet var också proteinkoncentration dependent, och denna aktivitet var linjärt proportionell mot mängden protein som används för enzymatisk analys (fig 3). Slutligen har ett SAM-koncentrationsberoende PEMT analysen utförs, i vilken ökande koncentrationer av SAM testades (Figur 4). PEMT aktivitet nådde en platå hos SAM koncentration av ca 0,5 mM. Sammantaget dessa fyra analyser visar att PEMT verksamhet är specifik och kan mätas från helcellextrakt utan att behöva rena enzymet (s).
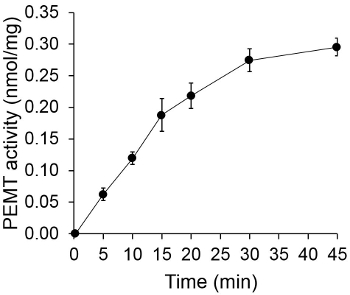
Figur 1. Tidsberoende PEMT analys. Den enzymatiska analysen utfördes två gånger i duplikat med 0,2 mg av hela Leishmania cellextrakt som en funktion av tiden. PEMT aktivitet representeras som nmol metylgrupper överförs på PE per mg protein och per h. För tiden "O", var cellextrakten först blandas med 2 ml stopplösning gjord av kloroform / metanol (1: 1, räknat på volym) innan de tillsätts till analyslösningen innehållande SAM. Standardavvikelser visas.
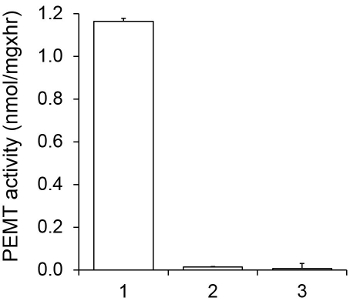
Figur 2. Specificitet av PEMT aktivitet. PEMT analysen utfördes två gånger i duplikat för 15 min i närvaro av 0,05 mM SAM. 1, 0,2 mg protein-extrakt; 2, ingen cellextrakt; 3, 0,2 mg protein och 0,1 mM octadecyltrimethylammonium bromid. Standardavvikelser visas.
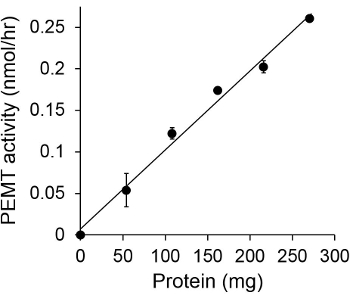
Figur 3. Protein beroende PEMT analys. Den enzymatiska analysen genomfördes två gånger iduplicera i frånvaro (punkt "O") eller närvaro av olika mängder av Leishmania proteiner med 0,05 mM SAM under 15 minuter. Standardavvikelser visas.
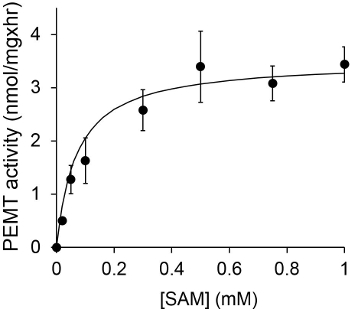
Figur 4. PEMT analysen utfördes två gånger i duplikat med 0,2 mg helcellextrakt Leishmania i närvaro av olika koncentrationer av SAM för 15 min. Standardavvikelser visas.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Denna enkla, snabba PEMT analysen möjliggör kvantifiering av denaturerad former av PE som härrör från överföringen av radioaktiva metylgrupper från SAM på PE använder helcellextrakt som proteinkälla. Den är snabb, känslig, reproducerbar, och även lämplig för renade enzymer 17. Monometyl- eller dimetyl-PE kan sättas till analys om metyltransferas av intresse är specifik för dessa substrat snarare än mot PE 12,13,18,19. Om renat PEMT enzym används, kan PE sättas till analysen. En begränsning av detta protokoll är att analysen inte identifiera de reaktioner produkter (monometyl-PE, dimetyl-PE eller PC). Emellertid kan identiteten av reaktionsprodukterna (monometyl-PE, dimetyl-PE, PC) ytterligare analyseras genom en endimensionell tunnskiktskromatografi såsom beskrivs i 20,21. Vidare kan några av de SAM nedbrytningsprodukter såsom S -adenosylhomocysteine och 5'-metyltioadenosin inhibera PEMT verksamhet By feedback inhibition. Emellertid Leishmania besitter en S -adenosylhomocysteine hydrolas 22, som klyver S -adenosylhomocysteine in adenin och S -ribosylhomocysteine, och en metyltioadenosin fosforylas, som producerar adenin och methylthioribose-1-fosfat 23. Det är dock inte känt om S -adenosylhomocysteine hydrolas och metyltioadenosin fosforylas aktiviteter är tillräckligt hög för att effektivt metabolisera S -adenosylhomocysteine och metyltioadenosin, respektive, så att ingen inhibition av PEMT aktivitet sker. I fallet S -adenosylhomocysteine hydrolas och / eller 5'-metyltioadenosin metaboliska enzym är frånvarande i cellen av intresse, tillsats av respektive renade, rekombinanta enzymerna kan tillsättas till analysen för att lindra feedback-inhibering av SAM nedbrytningsprodukter 24,25, 26.
Det finns fyra viktiga steg i detta protokoll: i)den proteasinhibitorcocktail pulver måste tillsättas till lysbufferten omedelbart före användning (steg 1,1); ii) Helcellextrakt ska kunna utnyttjas inom följande timmar efter beredning (efter steg 1,4); iii) under lipid-extraktionssteg (steg 4,2 och 4,4), måste försiktighet som skall tillämpas på inte överföra någon av interfasen eller vattenfasen, som innehåller överskottet av tritierad SAM, och iv) för att alikvot det radioaktiva S - [Metyl- 3 H] adenosyl-L-metionin och kallt reagens vid mottagandet som upprepade cykler av frysning och upptining försämra den i 5'-metyltioadenosin och homoserinlakton följt av hydrolys till adenin och S -pentosylmethionine 26,27,28, vilket kan förklara bristen på mätbar PEMT aktivitet. Bad helcellextrakt kan också vara ansvarig för någon enzymatisk aktivitet. I detta fall kan bedömas kvaliteten av cellextraktet genom att mäta en annan känd enzymatisk aktivitet. Slutligen brist på PEMT aktivitet kan lita på PEMT enZyme är specifik för monometyl-PE eller dimetyl-PE i stället för PE. Tillsats av monometyl-PE och / eller dimetyl-PE substrat till analysen kan återställa PEMT aktivitet.
En alternativ, har icke-radioaktiv SAM beroende metyltransferas analys utvecklats, som förlitar sig på den enzymatiska kvantifieringen av homocystein, de transmetyleringsreaktioner produkter av SAM 29. Detta protokoll har tillämpats med framgång för att mäta PE specifika metyltransferas aktivitet 16. Tyvärr är denna analys endast lämplig för renat enzym och inte för hela cellextraktet, och inte avslöja identiteten av reaktionsprodukterna heller.
Föreliggande protokoll kan möjligen appliceras på alla celltyper. Dessutom kan denna analys användas för att testa potentiella läkemedel som är specifika för en PEMT av intresse i samband med att testa nya antimikrobiella föreningar för att bekämpa bakterieinfektioner eller nya behandlingar mot dietinducerad ateroskleros, fetma, Och insulinresistens.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Ingen intressekonflikt deklarerade
Materials
| Name | Company | Catalog Number | Comments |
| S-[Methyl-3H]adenosyl-L-methionine (specific activity of 5-15 Ci/mMole) | Perkin Elmer | NET155050UC | Aliquot the reagent and freeze at -20 °C; follow radiation safety guidelines while using this reagent |
| Protease inhibitor cocktail | Roche Life Sciences | 11836170001 | dilute it fresh |
| Glass beads, acid washed, 425-600 mm | Sigma Aldrich | G8772 | |
| Bicinchoninic acid solution | Sigma Aldrich | B9643 | |
| Copper (II) sulfate | Sigma Aldrich | C2284 | |
| Scintillation counter MicroBeta2 with 1-detector | Perkin Elmer | 2450-0010 | |
| Spectrophotometer Biomate 3 | Thermo Scientific | 840208300 | |
| BSA stock solution (10 mg/ml) | New England Biolabs | B9001S | |
| Scintillation liquid | Research Product International Corp | 111198 | |
| S-(5'-Adenosyl)-L-methionine chloride (hydrochloride) | Cayman Chemicals | 13956 | dilute the reagent in 20 mM HCl and freeze aliquots at -80 °C |
References
- Keogh, M. R., Courtney, P. D., Kinney, A. J., Dewey, R. E. Functional characterization of phospholipid N-.methyltransferases from Arabidopsis and soybean. J Biol Chem. 284 (23), 15439-15447 (2009).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim Biophys Acta. 1831 (3), 503-513 (2013).
- Gao, X., et al. Decreased lipogenesis in white adipose tissue contributes to the resistance to high fat diet-induced obesity in phosphatidylethanolamine N-.methyltransferase-deficient mice. Biochim Biophys Acta. 1851 (2), 152-162 (2015).
- Zhao, Y., et al. Lack of phosphatidylethanolamine N-.methyltransferase alters plasma VLDL phospholipids and attenuates atherosclerosis in mice. Arterioscler Thromb Vasc Biol. 29 (9), 1349-1355 (2009).
- Vance, D. E. Phospholipid methylation in mammals: from biochemistry to physiological function. Biochim Biophys Acta. 1838 (6), 1477-1487 (2014).
- Nishimaki-Mogami, T., Suzuki, K., Takahashi, A. The role of phosphatidylethanolamine methylation in the secretion of very low density lipoproteins by cultured rat hepatocytes: rapid inhibition of phosphatidylethanolamine methylation by bezafibrate increases the density of apolipoprotein B48-containing lipoproteins. Biochim Biophys Acta. 1304 (1), 21-31 (1996).
- Noga, A. A., Zhao, Y., Vance, D. E. An unexpected requirement for phosphatidylethanolamine N-.methyltransferase in the secretion of very low density lipoproteins. J Biol Chem. 277 (44), 42358-42365 (2002).
- Li, D., et al. Epigenetic repression of phosphatidylethanolamine N-.methyltransferase (PEMT) in BRCA1-mutated breast cancer. Oncotarget. 5 (5), 1315-1325 (2014).
- Cui, Z., Houweling, M., Vance, D. E. Suppression of rat hepatoma cell growth by expression of phosphatidylethanolamine N-.methyltransferase-2. J Biol Chem. 269 (40), 24531-24533 (1994).
- Cui, Z., Shen, Y. J., Vance, D. E. Inverse correlation between expression of phosphatidylethanolamine.N-.methyltransferase-2 and growth rate of perinatal rat livers. Biochim Biophys Acta. 1346 (1), 10-16 (1997).
- Minder, A. C., de Rudder, K. E., Narberhaus, F., Fischer, H. M., Hennecke, H., Geiger, O. Phosphatidylcholine levels in.Bradyrhizobium japonicum. membranes are critical for an efficient symbiosis with the soybean host plant. Mol Microbiol. 39 (5), 1186-1198 (2001).
- Kodaki, T., Yamashita, S. Yeast phosphatidylethanolamine methylation pathway. Cloning and characterization of two distinct methyltransferase genes. J Biol Chem. 262 (32), 15428-15435 (1987).
- Tanaka, Y., Amano, F., Maeda, M., Nishijima, M., Akamatsu, Y. Purification and properties of phosphatidyl-N-.monomethylethanolamine N-.methyltransferase, the enzyme catalyzing the second and the third steps in the phosphatidylethanolamine N-.methyltransferase system, from mouse liver microsomes. Jpn J Med Sci Biol. 43 (3), 59-73 (1990).
- Bibis, S. S., Dahlstrom, K., Zhu, T., Zufferey, R. Characterization of Leishmania major phosphatidylethanolamine methyltransferases LmjPEM1 and LmjPEM2 and their inhibition by choline analogs. Mol Biochem Parasitol. 196 (2), 90-99 (2014).
- deRudder, K. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti. mutants deficient in phospholipid N-.methyltransferase still contain phosphatidylcholine. J Bacteriol. 179 (22), 6921-6928 (1997).
- Aktas, M., Narberhaus, F. In vitro characterization of the enzyme properties of the phospholipid N-.methyltransferase PmtA from Agrobacterium tumefaciens. J Bacteriol. 191 (7), 2033-2041 (2009).
- Ridgway, N. D., Vance, D. E. Phosphatidylethanolamine N-.methyltransferase from rat liver. Methods Enzymol. 209, 366-374 (1992).
- Gaynor, P. M., Carman, G. M. Phosphatidylethanolamine methyltransferase and phospholipid methyltransferase activities from Saccharomyces cerevisiae. Enzymological and kinetic properties. Biochim Biophys Acta. 1045 (2), 156-163 (1990).
- Arondel, V., Benning, C., Somerville, C. R. Isolation and functional expression in Escherichia coli. of a gene encoding phosphatidylethanolamine methyltransferase (EC 2.1.1.17) from Rhodobacter sphaeroides. J Biol Chem. 268 (21), 16002-16008 (1993).
- Wessel, M., Klusener, S., Godeke, J., Fritz, C., Hacker, S., Narberhaus, F. Virulence of Agrobacterium tumefaciens. requires phosphatidylcholine in the bacterial membrane. Mol Microbiol. 62 (3), 906-915 (2006).
- Klusener, S., Aktas, M., Thormann, K. M., Wessel, M., Narberhaus, F. Expression and physiological relevance of Agrobacterium tumefaciens. phosphatidylcholine biosynthesis genes. J Bacteriol. 191 (1), 365-374 (2009).
- Henderson, D. M., et al. Cloning of the gene encoding Leishmania donovani.S.-adenosylhomocysteine hydrolase, a potential target for antiparasitic chemotherapy. Mol Biochem Parasitol. 53 (1-2), 169-183 (1992).
- Koszalka, G. W., Krenitsky, T. A. 5'-Methylthioadenosine (MTA) phosphorylase from promastigote of Leishmania donovani. Purine and Pyrimidine Metabolism in Man V, Adv Exp Med Biol. Nyhan, W. L., Thompson, L. F., Watts, R. W. E. 131, Springer US. 559-563 (1986).
- Biastoff, S., Teuber, M., Zhou, Z. S., Dräger, B. Colorimetric activity measurement of a recombinant putrescine N.-methyltransferase from Datura stramonium. Planta Med. 72 (12), 1136-1141 (2006).
- Hendricks, C. L., Ross, J. R., Pichersky, E., Noel, J. P., Zhou, Z. S. An enzyme-coupled colorimetric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 326 (1), 100-105 (2004).
- Cannon, L. M., Butler, F. N., Wan, W., Zhou, Z. S. A stereospecific colorimetric assay for (S.,S.)-adenosylmethionine quantification based on thiopurine methyltransferase-catalyzed thiol methylation. Anal Biochem. 308 (2), 358-363 (2002).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S.-adenosyl-L-methionine. Biochemistry. 25 (15), 4444-4449 (1986).
- Wu, S. E., Huskey, W. P., Borchardt, R. T., Schowen, R. L. Chiral instability at sulfur of S.-adenosylmethionine. Biochemistry. 22 (12), 2828-2832 (1983).
- Dorgan, K. M., et al. An enzyme-coupled continuous spectrophotometric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 350 (2), 249-255 (2006).



