ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
클러스터는 정기적으로 짧은 구조 반복/CRISPR 관련 된 단백질 9 (CRISPR/Cas9) interspaced 시스템 유전 공학에 대 한 유망한 도구를 제공 하 고 transgenes 대상된 통합의 가능성을 엽니다. (HMEJ)에 가입 하는 homology 중재 끝 설명-효율적인 DNA 비보에 통합 대상 및 CRISPR/Cas9를 사용 하 여 유전자 치료를 대상으로 전략을 기반으로.
Abstract
유망한 게놈 편집 플랫폼으로 서 CRISPR/Cas9 시스템 특히 transgenes 대상된 통합에 대 한 효율적인 유전자 조작에 대 한 큰 잠재력이 있다. 그러나, 동종 재결합 (HR)의 낮은 효율과 비 동종 끝 결합 (NHEJ)의 다양 한 indel 돌연변이 때문-기반 전략 비 분할 셀, vivo에서 게놈 남아 편집에 큰 도전. 여기, 우리가 설명 (HMEJ)에 가입 하는 homology 중재 끝-정확한 타겟 통합 효율적인 vivo에서 CRISPR/Cas9 시스템을 기반으로. 이 시스템에는 기증자와 타겟된 게놈 벡터 포함 하 상 동 팔 (~ 800 bp) 단일 가이드 RNA (sgRNA) 대상 시퀀스는 CRISPR/Cas9에 의해 죽 습에 의해 형벌. 이 HMEJ 기반 전략 마우스 zygotes, 뿐만 아니라 vivo에서hepatocytes에 효율적인 transgene 통합을 달성 한다. 또한, HMEJ 기반 전략은 hepatocytes에서 fumarylacetoacetate 가수분해 효소 (파) 돌연변이의 수정에 대 한 효율적인 접근 방식을 제공 하 고 파를 구출-결핍 유발 간부 쥐. 함께, 통합 대상에 초점을 맞추고,이 HMEJ 기반 전략 유전자 변형된 동물 모델 및 타겟된 유전자 치료의 세대를 포함 하는 응용 프로그램의 다양 한 유망 도구를 제공 합니다.
Introduction
정확 하 고 타겟 게놈 편집 하는 것은 종종 생산 하는 유전자 변형 동물 모델과 임상 치료 필요 합니다. 효율적인 타겟된 게놈 아연 손가락 nuclease (ZFN), 같은 녹음 방송 활성 제 같은 이펙터 nucleases (TALENs), 편집에 대 한 다양 한 전략을 개발 하기 위해 많은 노력 했다 및 CRISPR/Cas9 시스템. 이러한 전략 타겟된 DNA 두 배 물가 틈 (DSB) 게놈에서 만들고 동종 재결합 (HR)1,2, microhomology-중재 끝 결합 (MMEJ)3 등 본질적인 DNA 복구 시스템 , 4 , 5, 그리고 비 동종 끝 결합 (NHEJ)6,,78 transgenes1,9의 타겟된 통합을 유도 하 라. 시간 기반 전략은 현재 가장 일반적으로 사용 되는 셀 라인에 매우 효율적 이지만 비 분할 셀 후반 S/g 2 단계에서 제한 된 그것의 발생 때문에 쉽게 액세스할 수 없는 접근을 편집 하는 게놈. 따라서, 시간 기반 전략 vivo에서 게놈 편집에 대 한 적용 되지 않습니다. 최근, NHEJ 기반 전략에 대 한 효율적인 유전자 노크 마우스 조직8에서 개발 되었다. 그럼에도 불구 하 고, NHEJ 기반 방법 일반적으로 접합, 어렵게 정확한 게놈 편집, 프레임에 융합 유전자8를 구성 하려고 할 때 특히 생성에서 indels를 소개 합니다. MMEJ 기반 대상된 통합은 정확한 게놈 편집 가능 합니다. 그러나, 그것은 단지 겸손 하 게 이전 보고서5대상된 통합 효율성 증가. 따라서 vivo에서 정확한 타겟된 통합의 효율성 개선 시급히 필요 광범위 한 치료 응용 프로그램3합니다.
최근에 출판 된 작품에서 우리 시연 (HMEJ)에 가입 하는 homology 중재 끝-모든 보고 전략 둘 다 생체 외에서 그리고 vivo에서10최고의 타겟된 통합 효율을 보여준 전략을 기반으로. 여기, 우리는 HMEJ 시스템의 설립을 위한 프로토콜을 설명 하 고 또한 관심과 기증자의 유전자를 대상으로 단일 가이드 RNA (sgRNA) 벡터의 벡터 은닉 sgRNA 대상 사이트와 ~ 800 bp 상 동 팔 (그림 1)의 . 이 프로토콜에서 우리 또한 vivo에서조직 대상된 통합에 대 한 DNA 노크에서 마우스와 간단한 단계의 세대에 대 한 자세한 내용은 설명합니다. 또한, HMEJ 기반 전략의 개념 증명 연구 Fah 돌연변이 수정 하 고 구조 파-/- 간 실패 마우스, 더 치료 잠재력을 공개 하는 기능을 설명 했다.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
동물 주제를 포함 한 모든 절차 생물 과학 (CAS) 상하이 연구소에서 생물 의학 연구 윤리 위원회에 의해 승인 되었습니다가지고.
1입니다. 기증자 플라스 미드의 디자인
-
SgRNA의 선택
- 온라인 CRISPR 디자인 도구를 사용 하 여 대상 지역11,12,13,,1415에 sgRNAs를 예측. Cdx2 로커 스에 대 한 6 개의 서로 다른 sgRNAs 디자인 (Cdx2-sgRNA1 ~Cdx2-sgRNA6) 주위에 정지 codon 높은 순위 및 더 낮은 잠재력으로 오프-대상 (그림 1A)16.
- BbsI 소화 (1 µ L BbsI의 20 µ L의 볼륨에서 1 U / µ L의 최종 농도에서 37 ° C에서 2 시간에 대 한)에 의해 Cas9-CMV-EGFP 식 벡터와 sgRNA의 2 µ g를 선형화. 다음 1 × 태 버퍼에 1 %agarose 젤 젤 정화 키트에 의해 제품을 정화.
- 믹스 한 쌍 sgRNA oligonucleotides 50 µ M. 최종 농도를 1 × T4 DNA 리가 버퍼의 10 µ L에서의 온도 변화 속도 5 ° C/5 분 (5 분 동안 95 ° C 95 ° C에서 25 ° C의 온도 기울기를 사용 하 여 올리고 솔루션을 품 어 5 분, 5 분, 등등을 위한 85 ° C에 대 한 다음 90 ° C.),이 oligos anneal 것입니다.
- 혼합 4 µ L의 제품, 1 × T4 DNA 리가 버퍼의 10 µ L에 T4 DNA 리가의 1 µ L로 선형화 벡터의 2 µ L을 단련 하 고 1-2 h (그림 1B) 22 ° C에서 선.
-
SgRNA의 측량 nuclease 분석 결과
참고: 노크에서 실험을 위해 사용 하는 sgRNA의 대상 효율 측량 nuclease 분석 결과 평가 (일컬어 T7 endonuclease 나 (T7EI) 분석 결과)17. 높은 DNA 분열 효율과 sgRNA 절단 사이트와 정지 codon 사이의 낮은 거리는 sgRNA를 선택 합니다.- Cas9-sgRNA-EGFP 식 벡터 N2a 셀 라인에 transfect DMEM 보충 10% 태아 둔감 한 혈 청에서에서 교양, 1 %PSG, 그리고 1% 비 필수 아미노산은 transfection에 의해 키트 ( 재료의 표참조). 5% CO2에서 37 ° C에서 transfected 세포를 품 어.
- 외피의 48 h 후 수집 5000 transfected 셀 (GFP+) 형광 활성화 (FACS) 정렬 제어로 비 페 셀을 사용 하 여.
- 2-5 µ L oflysis 버퍼 (0.1% 트라이 톤 X-100, 0.1% 트윈 20, 및 100 µ g/mL 가수분해 K) 30 분 동안 56 ° C에서 수집 된 세포를 소화 하 고 십 분 동안 95 ° C에 K 성분을 비활성화.
- 중첩 된 PCR (표 1) 제조업체의 프로토콜을 사용 하 여 샘플을 증폭. PCR 제품의 크기는 300-500 bp로 설정 됩니다.
- DNA 중 합 효소와 sgRNA 대상 사이트 (0.1 µ M, 최종 농도) 시퀀스를 인식 하는 외부 뇌관의 쌍 세포 제품의 믹스 1 µ L (표 1), 20 µ L의 볼륨에서 기본 PCR을 수행 하 고.
- 5 분 동안 95 ° C에서 DNA 중 합 효소를 활성화 하 고 30 95 ° C에서 30 사이클에 대 한 기본 PCR을 수행 s, 60 ° C 30에 대 한 s, 그리고 24 72 ° C s (1 분/1 킬로바이트), 5 분 동안 72 ° C에서 최종 확장.
- 1 µ L 기본 PCR 제품의 중첩 된 내부 뇌관의 쌍을 사용 하 여 2 차 PCR을 수행 합니다.
- 변성 고 다시 1 × T7EI 반응 버퍼 (50 m m NaCl, 10 mM Tris HCl, 10 m m MgCl2, 1mm DTT pH 7.9)의 20 µ L에서 순화 된 PCR 제품의 300-600 기 anneal 속도/5 분의 5 ° C 95 ° C에서 25 ° C의 온도의 그라디언트를 사용 하 여.
- 단련 된 PCR 제품 및 다이제스트 2 h 37 ° C에서 T7EI 효소의 1 µ L를 추가 합니다. 파편 ( 재료의 표참조) 분리 될 때까지 다음 1 × 태 buffer 120 V 40 분 2 %agarose 젤에 소화 제품을 실행 합니다.
- ImageJ를 사용 하 여 잘라내기 및 포경된 DNA의 밴드 농도 결정. 메서드를 사용 하 여 이전 indel 빈도 보고9 (그림 1C)를 계산 합니다.
-
기증자 벡터의 건설
참고: Cdx2 유전자에 대 한 HMEJ 기증자 벡터를 생성 하려면 구성 DNA 기증자 (800 bp HAL-p2A-mCherry-800 bp 하겠다) 23 형벌 nt Cdx2 sgRNAs 타겟팅 (D 그림 1및 그림 1E) 양쪽에서 시퀀스. 대상 시퀀스의 PAM 동종 팔의 끝에 인접 했다. 깁슨 어셈블리 HMEJ 기증자 복제에 대 한 것이 좋습니다.- 앞으로 뇌관-1 층으로 800 bp 왼쪽된 상 동 팔 (HAL)을 증폭 (벡터, 23에서에서 15-20 nt 중첩 시퀀스가 포함 된 nt Cdx2 sgRNA 대상 시퀀스, 그리고 HAL에서 약 20 nt 시퀀스) 및 뇌관-1 라운드 (15-20 bp 겹침 순서에서 포함 된 역 p2A mCherry와 HAL에서 약 20 nt 시퀀스) 0.1 µ M 최종 농도 200 ng / µ L (D그림 1, 표 1)에서 마우스 게놈 DNA를 사용 하 여에서.
- 앞으로 뇌관-2 층 (HAL에서 15-20 nt 겹침 순서와 삽입 부분에서 약 20 nt 시퀀스를 포함 하는)와 p2A mCherry 삽입 단편 증폭 하 고 뇌관-2R (포함 된 무기에서 15-20 nt 중첩 시퀀스에서 약 20 nt 순서 반전 삽입 부분)에서 0.1 µ M 최종 농도가 100 ng / µ L 또는 30 ng / µ L (D그림 1, 표 1)에서 기자 시퀀스 게놈 DNA 또는 플라스 미드를 사용 하 여.
- 앞으로 뇌관-3 층으로 800 bp 오른쪽 상 동 팔 (무기)를 증폭 (벡터, 23에서에서 15-20 nt 중첩 시퀀스가 포함 된 nt Cdx2 sgRNA 대상 시퀀스, 그리고 무기에서 약 20 nt 시퀀스) 반전 뇌관-3R (15-20에서 nt 중복 시퀀스를 포함 하 고 p2A mCherry 및 무기에서 약 20 nt 시퀀스) 0.1 µ M 최종 농도 200 ng / µ L (D그림 1, 표 1)에서 마우스 게놈 DNA를 사용 하 여에서.
- 1 × 태 버퍼에 1 %agarose 젤에 PCR 제품을 실행 하 고 제조업체의 지침 (표 1)에 따라 젤 추출 키트에 의해 예상 되는 크기의 PCR 제품을 정화.
- KpnI 및 XbaI 구조 벡터의 50-100 ng을 소화. 믹스 2 µ L 3 PCR와 30-40 ng / µ L에서 선형화 벡터의 깁슨 믹스 x 2에서 조각 (각, 100-200 ng / µ L에 대 한 1 µ L)를 증폭. H2O 10 µL.Incubate 60 분 50 ° C에서 믹스를 최종 볼륨 조절 추가.
- 유능한 대장균 을 변형 모든 조립된 제품 및 플라스 미드 DNA 추출 키트 제조업체의 지침에 따라 하 여 구성 하는 추출 셀. DNA 시퀀싱에 의해 HMEJ 기증자를 확인 합니다.
2. 게놈 HMEJ 기반 메서드를 사용 하 여 마우스 배아에서 편집
-
Cas9 mRNA의 생산
- Cas9 mRNA 준비, T7 발기인 순서는 Cas9 표 1에 나열 된 적절 한 뇌관 쌍을 사용 하 여 PCR 증폭에 의해 지역 코딩을 추가 합니다. 0.1 µ M의 최종 농도 20에서 뇌관 Cas9 F/R 추가 1 × 고 충실도 DNA 중 합 효소 혼합에 벡터를 표현 하는 Cas9의 ng. H2o. 50 µ L 최종 볼륨을 조정
- 5 분 동안 95 ° C에서 DNA 중 합 효소를 활성화 하 고 30 95 ° C에서 36 사이클에 대 한 PCR을 수행 s, 60 ° C 30에 대 한 s, 그리고 4 분 (1 분/1 킬로바이트), 10 분 동안 68 ° C에서 최종 확장명이 68 ° C.
- 생체 외에서 전송 (IVT), T7 Cas9 PCR 제품을 정화 하 고 다음 녹음 0.5-1 µ g의 DNA mRNA 전사 키트 제조업체의 지침에 따라 20 µ L의 전체 볼륨에 8 h 37 ° C에 의해 ( 재료의 표참조).
- 15 분 추가 폴 리 A 꼬리 37 ° C에서 45 분 동안 37 ° C에서 DNA 템플렛을 제거 하 고 제조업체의 지침에 따라 RNA 정화 키트, 여 Cas9 mRNA를 복구 하는 혼합물에 DNase의 1 µ L를 추가 ( 재료의 표참조).
-
SgRNA의 생산
- 고 충실도 DNA 중 합 효소로 위에 T7 발기인에 의해 구동 sgRNA 서식 파일을 생성 합니다. 템플릿으로 벡터를 포함 하는 sgRNA 비 계를 선택 합니다. 사용 하는 뇌관은 표 1에 나열 됩니다.
- T7 sgRNA PCR 제품을 정화 하 고 0.5-1을 사용 하 여 µ g sgRNA 짧은 RNA 전사 키트를 사용 하 여 제조업체의 지침에 따라 20 µ L의 전체 볼륨에 6 h를 37 ° C에서의 녹음 방송 생체 외에서 에 대 한 템플릿으로 DNA ( 테이블의 자료를 참조 하십시오 < / c11 >).
- 1 µ L DNase의 혼합물에 추가 하 고 DNA 템플렛을 제거 하려면 15 분 동안 37 ° C에는 부 화를 계속. sgRNAs RNA 정화 키트에 의해 정화 ( 재료의 표참조) 이상으로.
- RNase 무료 물에 500 ng / µ L을 sgRNA를 희석 하 고 최대 3 개월까지 −80 ° C에서 샘플을 저장 합니다.
참고: CRISPR ribonucleoproteins (RNPs)는 더 나은 절단 효율성18,,1920대체 대체 됩니다.
-
배아 컬렉션, microinjection 및 생체 외에서 문화
- 여성 B6D2F1 (C57BL/6 × DBA2J) 쥐 (7-8 주 이전) 임신 암 말 혈 청 생식 샘 자극 호르몬 (PMSG)에 의해 뒤에 인간의 융 생식 샘 자극 호르몬 (hCG) 48 h 후 superovulate HCG 주사 후 집 여성 B6D2F1 남성과 하룻밤.
- CO2 마 취, hCG 주사 후 24 h에 의해 여성을 희생. M2 매체에서 (30-50 배아 각 여성에 대 한)와 그들의 oviducts에서 수정 된 배아를 수집 합니다.
- 장소 KSOM 중간에 수정 된 배아 (약 300 달걀 1 일 주입에 대 한) (5.55 g/L NaCl, KCl, 0.05 g/L KH2포4, 0.05 g/L MgSO4•7H2O, 0.04 g/L 포도 당, 1.12 g/L 나트륨 0.19 g/L 젖이 나올, 2.1 g/L NaHCO3 , 0.02 g/L 나트륨 pyruvate, 0.25 g/L CaCl2•2H2O, 0.004 g/L EDTA, 0.146 g/L L-글루타민, 및 1 g/L 소 혈 청 알 부 민) 5% CO2배양 기에서 37 ° C에서.
- 혼합 Cas9 mRNA (100 ng / µ L), sgRNA (50 ng / µ L), 그리고 HMEJ 기증자 벡터 (100 ng / µ L), H2O 10 µ L. 최종 볼륨을 조정 하는 얼음에 혼합물을 넣어 추가.
- 모 세관 바늘 (외부 직경 1.0 m m, 내경 필 라 멘 트와 0.78 m m)를 당겨 Micropipette 끌어당기는 사람을 사용 하 여 (매개 변수: 열, 74, 풀, 60, 속도, 80, 200 시간/지연, 압력, 300. 재료의 표참조). 상업적인 바늘은 microinjection에 대 한 대체 대체 될 것 이다.
- HEPES CZB 매체 포함 하는 5 µ g/mL cytochalasin 상수는 microinjector를 사용 하 여 B의 물방울에 잘 정의 된 pronuclei와 혼합물의 가능성이 볼륨 zygotes의 세포질으로 주입 흐름 설정 (그림 2A) (참조 재료의 표)21.
참고: 각 그룹 zygotes의 주입 한다 20-30 분 내 Cytochalasin B는 주사 후 마우스 zygote의 생존 능력을 증대 시킬 수. 또는, microinjection22위에서 설명한 대로 피에 조 시스템으로 동작할 수 있습니다. - 37 ° C 미만 5% CO2 는 blastocyst까지 형광 관찰 (그림 2B , 2C)에 대 한 일 후 무대에서 KSOM 중간에 삽입 된 zygotes 문화.
-
배아 전송 및 쥐의 생성
- 주입으로 같은 날에 vasectomized ICR 남성 마우스 estrous ICR 여성 쥐를 친구.
- 5% CO2, 그리고 0.5 하루 게시물 coitum (dpc)에서 pseudopregnant ICR 여성의 oviducts로 전송 25-30 2 셀 배아에서 37 ° C에서 2 셀 단계에 주입된 zygotes 문화. 받는 사람 어머니 19.5 dpc에서 새끼를 제공합니다.
-
마우스 유전형
- 제조업체의 지침에 따라 DNA 추출 키트를 사용 하 여 발가락 또는 꼬리 샘플에서 마우스 게놈 DNA를 추출 ( 재료의 표참조).
- 5'과 3' 접합 PCR amplification를 수행 하기 위해 템플릿으로 UV/vis 분 광 분석에 의해 측정 하는 genomic DNA의 200-400 ng를 사용 하 여 노크에서 이벤트를 식별 합니다.
- 5 분 동안 95 ° C에서 DNA 중 합 효소를 활성화 하 고 30 95 ° C에서 38 사이클에 대 한 PCR을 수행 s, 60 ° C 30에 대 한 s, 그리고 10 분 동안 72 ° C에서 최종 확장으로 1 분 (1 분/1 킬로바이트), 72 ° C. 5' 접합, 노크-조각 (p2A-mCherry)에 반대 한 HAL의 업스트림에서 앞으로 뇌관을 사용 합니다. 3' 접합로 노크-조각 (p2A-mCherry)에 앞으로 뇌관을 사용 하 여 무기 (표 1)의 하류에 반대 하나.
- 1 × 태 버퍼 및 예상된 조각 크기에 대 한 확인에 1 %agarose 젤에 PCR 제품의 6 µ L를 실행 합니다. 다음 DNA 시퀀싱 (그림 2D)으로 그들을 확인 합니다.
3. HMEJ-기반 Vivo에서 게놈 Hepatocytes에서 편집
- 금지 장치에 받는 사람 C57BL/6J 마우스 (8 주)를 배치 하 고 슬릿을 통해 꼬리를 넣어.
- HMEJ 기증자 벡터 (30 µ g)과 spCas9 식 벡터 (30 µ g) 염의 2 개 mL에 혼합. 제어 실험에 대 한 염 (그림 3A)의 2 ml HMEJ 기증자 벡터 (30 µ g)을 일시 중단 합니다.
- 70% 에탄올과 마우스 꼬리를 청소. 삽입 꼬리에 바늘 정 맥 및 플라스 미드 DNA 솔루션 5-7 미에서 주사 바늘을 제거 하 고 억제 장치에서 마우스를 놓습니다.
- 주입 후 5-9 일 후 CO2 마 취에 의해 쥐를 희생. 0.9% 식 염 수와 쥐 transcardially perfuse, 4 %paraformaldehyde 연동 펌프를 사용 하 여 및 4 ° c.에 하룻밤 간 수정
- 하룻밤, 튜브의 바닥에 싱크 될 때까지 30% 자당을 사용 하 여 조직을 탈수.
- 섹션 간 샘플 10 µ m의 두께에 냉동된 조직.
- 0.1 M 인산 버퍼 (PB)에 세 번 섹션 린스와 1 차 항 체로 그들을 품 어: 안티-mCherry 토끼 (5%에 희석 NGS) 4 ° c.에서 하룻밤
- 세 번에 PB, 섹션을 세척 하 고 다음 2 차 항 체로 그들을 품 어: Cy3-AffiniPure 염소-토끼 IgG 궤도 셰이 커에 실 온에서 2 h에 대 한.
- 20 분 동안 DAPI와 섹션 및 형광 관찰 (그림 3B)에 대 한 유리 슬라이드에 글 리세 린과 산 counterstain.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
HMEJ 기반 게놈 마우스 배아에서 편집: 마우스 zygotes에서 노크에 HMEJ 기반 방법의 효율성을 정의 우리 Cas9 mRNA, Cdx2 의 마지막 codon에 p2A mCherry 취재 원 유전자를 융합 설계 되었습니다 sgRNA 마우스 zygotes에 HMEJ 기증자와 Cdx2 유전자를 대상으로 전달 유전자 (그림 2A). 주입 된 zygotes blastocysts 문화에로 발전. 노크에 효율성을 평가 하 우리는 형광 현미경으로 mCherry 형광을 분석 하 고 우리는 HMEJ 기증자를 받는 blastocysts의 12.9%는 trophectoderm에서 엄격 하 게 표현 되었다 mCherry에 대 한 긍정적인 발견 (그림 2B 2 C). PCR 긍정적인 쥐, 시퀀싱에 의해 우리는 또한 모든 통합 이벤트 5'과 3' 접합 (그림 2D)에서 정확한 프레임에 통합 했다 검사 발견.
성인 조직 및 HMEJ 중재 하는 유전자 치료에 HMEJ 기반 게놈 편집: HMEJ 기반 게놈 편집 수 적용 여부 성인 조직에 조사, 우리는 꼬리 정 맥에 의해 C57/B6J 마우스 간은 하 Actb-HMEJ 구조를 시험 하 여 Actb 유전자의 정지 codon 앞 mCherry 카세트를 삽입 유체역학 주입 (그림 3A). 주사의 7 일 후 우리는 transfected hepatocytes의 거의 절반 표현 mCherry (그림 3B) 간 섹션에 얼룩이 발견.
HMEJ 기반 전략을 사용 하 여 유전자 치료에 대 한 가능성을 탐험, 우리 fumarylacetoacetate 가수분해 효소 (파) 고용-결핍 쥐. 파-/- 마우스 기초가 튼튼한 유전 tyrosinemia 유형 I 다음 시퀀스23frameshift 돌연변이 일으키는 Fah 유전자의 exon 5에서에서 삽입 조각 항구 (HTI) 마우스 모델 이다. 파-/- 생쥐를 유지 하려면 우리는 티로신 catabolic 통로, 2-(2-nitro-4-trifluoromethylbenzoyl)-1,3-cyclohexanedione (NTBC)24의 업스트림의 억제제와 파-/- 생쥐 취급. 여기 우리는 MMEJ 및 HMEJ 중재 유전자 수정 파-/- 마우스에서 Fah 돌연변이 구출 수 여부 보고 밖으로 설정 합니다. 우리는 hydrodynamically Cas9 구문 주입 파 -/- 파-MMEJ 또는 파-HMEJ 구문, exon 5 ~ 14 intron Fah 유전자의 4에의 파 cDNA를 삽입 하도록 간 (를 마우스 그림 3 C). 주입 후 1 주일 NTBC 유발 간 손상 (그림 3C)를 철회 했다. NTBC, 파의 철수 후- 파-HMEJ 및 Cas9 구문 파-/- 생쥐의 수정된 hepatocytes 보였다 MMEJ 기반 방법 (그림 3D 보다 더 효과적인 확산 ).

그림 1 : 생체 외에서통합 대상 HMEJ 중재.
(A) sgRNAs의 선택에 대 한 실험 계획: 6 다른 sgRNAs (Cdx2-sgRNA1 ~Cdx2-sgRNA6) 더 높은 순위 및 오프 대상 잠재력 Cdx2 로커 스의 정지 codon 주위 했다 선택한 디자인에 따라 온라인 CRISPR 도구입니다. Protospacer 인접 한 모티브 (PAM) 시퀀스는 빨간색에서입니다. (B) 실험 설계: The Cas9-CMV-GFP 식 플라스 미드 표현 하는 sgRNA, Cas9, 그리고 GFP N2a 세포에 도입 되었다. GFP+ 세포 주 3 측량 분석 결과에 정렬 했다. (C) 측량 Cdx2 대상에 대 한 분석 결과: 6 다른 sgRNAs 측량 분석 결과 대 한 설계 되었습니다. 보통 N2a 셀 genomic DNA 컨트롤 역할을 합니다. *, Cdx2사용 sgRNA-2A-mCherry 노크에서 실험. (D) HMEJ 기증자 깁슨 어셈블리를 사용 하 여의 건설의 도식 개요. (E) Cdx2 로커 스에서 전략 대상으로 HMEJ 중재 하는 유전자의 도식 개요. HAL/무기, 왼쪽/오른쪽 상 동 팔; 삼각형, sgRNA 대상 사이트; / 또는, 외부 앞으로 또는 반전 뇌관; 경우 / IR, 내부/역회전 뇌관. 그림10이전 보고서 수정입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 2 : 통합 대상 게놈 HMEJ 중재를 통해 마우스 배아에서 편집
Microinjection의 (A) 실험 계획: Cas9 mRNA의 혼합물 (100 ng / µ L), sgRNA (50 ng / µ L), 및 기증자 플라스 미드 (100 ng / µ L) 마우스 zygotes에 주입 했다. (B) 마우스 배아 HMEJ 전략에 의해 편집의 대표적인 형광 이미지. 바, 20 µ m. (C) 노크에서 효율 mCherry+ 의 비율 blastocysts 표시. 각 바, 총 blastocysts 계산 위에 번호입니다. (D) 시퀀스 Cdx2 로커 스에서 마우스 유전자 편집의 분석. 5'과 3' 접합 사이트에서 증폭 PCR 제품 시퀀싱 했다. 상단, 상 동 팔; 보라색, p2A; 빨간색, mCherry; 무기 또는 HAL, 오른쪽 또는 왼쪽 동종 팔. 파선 영역 편의상 생략을 표시 합니다. 그림10이전 보고서 수정입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 3 : Vivo에서통합 대상 HMEJ 중재.
(A) 유체 꼬리 정 맥 주입의 도식 개요. 플라스 미드 기증자 시퀀스와 sgRNA, 그리고 spCas9을 표현 하는 플라스 미드의 혼합 유체 꼬리 정 맥 주입을 통해 간 전달 되었다. (B) hepatocytes의 대표적인 면역 형광 이미지. 간 섹션 수집된 7 일 게시물 주입 했다. 스케일 바, 50 µ m. GFP, transfected 세포. (C) 플라스 미드 중 MMEJ 또는 HMEJ 중재 유전자 대체 전략의 intron 4 Fah 유전자의 exon 5 ~ 14의 파 cDNA 삽입 하도록 설계 된 유체를 주입 하 여 파-/- 마우스 간으로 전달 되었다. 에 NTBC: 파-/- 생쥐 NTBC 물;에 유지 되었다 떨어져 NTBC: NTBC 물 철수 (NTBC 철수의 첫날 하루 0, 주입 후 7 일으로 정의 된). (D) 파 immunohistochemistry 파-/- 생쥐 MMEJ 또는 HMEJ 플라스 미드 주입에서 간 섹션의 얼룩. 스케일 바, 100 µ m. 그림 수정 이전 보고서5,10. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
HMEJ 기증자 플라스 미드의 건축에서 가장 중요 한 단계는: (1) 높은 DNA 분열 효율과 낮은 거리 sgRNA 절단 사이트와 정지 codon, 및 (2) 적절 한 건설 HMEJ 기증자의 간의 sgRNA의 선택. CRISPR/Cas9-중재 분열 (sgRNA 대상 사이트와 ~ 800 bp 상 동 팔 포함) 모두 transgene 기증자 벡터에 타겟된 게놈은 효율적이 고 정확한 타겟된 통합에 필요한 vivo에서. HMEJ 기반 방법을 사용 하 여 노크에서 쥐의 세대의 가장 중요 한 단계는: Cas9 mRNA와 sgRNA (Cas9 mRNA와 sgRNA에 존재 하는 아무 변성), 높은 품질의 준비 (1) 및 (2) 높은 품질 HMEJ 기증자 플라스 미드의 준비. 플라스 미드는 배아 발달에 아무 독성 효과 보여줍니다.
최근, NHEJ 기반 방법 또한 효율적인 vivo에서 게놈8편집에 대 한 보고 왔다. 그럼에도 불구 하 고, 이전 보고서8, 정확한 통합을 달성 하기 어려운 만들기에 설명 된 대로 다양 한 유형의 indel 돌연변이 접합에서 유도 일반적으로 했다. 여기, 우리가 위에서 설명한 HMEJ 기반 전략 거의 모든 indel 돌연변이와 정확한 타겟된 통합을 했다. 따라서, HMEJ 기반 전략 NHEJ 기반 방법에 적용 되지 않습니다 올바른 것으로 돌연변이 시퀀스 (예: 점 돌연변이)를 대체 하기 위한 이상적인 플랫폼을 수 있습니다.
Mosaicism은 유전자 배아에서 편집에 대 한 주요 문제입니다. 이전 배아 단계에서 mRNA 대신 Cas9 단백질의 주입 transgene 노크-에서 mosaicism 없이 한 셀 단계에서 얻을 수 있습니다. 임상 응용 프로그램에 대 한 성인 조직에 CRISPR/Cas9 시스템의 납품은 여전히 도전적 이다.
HMEJ 기반 게놈 편집의 많은 미래의 잠재적인 사용을 확인 하 고 있습니다. 그것은 유전자 변형된 동물 모델 생성에 사용할 수 있습니다. 그것의 노크에 고효율 배아에서 고려이 유전자 변형된 동물 모델 생성에 필요한 동물 수를 크게 줄일 수 있는 메서드와 특히 비 인간 영장류 유전자 모델 생성의 가능성을 열어. HMEJ 기반 게놈 편집 수 있습니다 성인 조직, 계보 추적 셀 종류를 특히 유용 동물 모델에 대 한 사용 가능한 동물 모델, 비 인간 영장류의 부족 때문. 그것은 타겟된 유전자 요법을 사용할 수 있습니다: HMEJ 기반 전략의 가장 매력적인 응용은 유전자 치료 클리닉 사용. 이 연구에서 우리가 수정 유전 tyrosinemia 형식의 Fah 돌연변이 나 표시 된 벡터의 유체를 주입 하 여 마우스. 그러나, 성인 조직으로 CRISPR/Cas9 시스템의 배달은 여전히 임상 사용을 위한 주요 기술 도전 유체 주입 환자에 수행 될 것 이다. 현재, 추가 배달 전략의 개선 시급히 필요 하다 병원에이 HMEJ 기반 메서드를 변환 하기 전에.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
저자는 공개 없다.
Acknowledgments
이 작품은 CAS 전략적 우선 순위 연구 프로그램 (XDB02050007, XDA01010409), 국가 하이테크 R & D 프로그램 (863 프로그램; 2015AA020307), 중국의 국가 자연과학 기초에 의해 지원 되었다 (NSFC 부여 31522037, 31500825, 31571509, 31522038), 중국 청소년 천 재능 프로그램 (HY), 휴식 프로젝트를 통해 중국 과학 아카데미의, 상하이 시 위원회의 과학 기술 프로젝트 (HY에 16JC1420202), 과학과 기술의 중국 (대부분; 2016YFA0100500).
Materials
| Name | Company | Catalog Number | Comments |
| pX330 | Addgene | 42230 | |
| pAAV vector | Addgene | 37083 | |
| pX260 | Addgene | 42229 | |
| AAV_Efs_hSpCas9_NLS_FLAG-SV40 | Addgene | 97307 | AAV vector for encoding a human codon-optimized SpCas9 driven by EFs promoter |
| AAV_Actb HMEJ donor_U6_sgRNA_EF1a_GFP_polyA | Addgene | 97308 | HMEJ donor for fusing a p2A-mCherry reporter to mouse Actb. EGFP driven by EF1a promoter and U6-driven sgRNAs targeting Actb. AAV backbone. |
| AAV_Cdx2 HMEJ donor | Addgene | 97319 | HMEJ donor for fusing a p2A-mCherry reporter to mouse Cdx2. |
| Lipofectamine 3000 Transfection Reagent | Life Technology | L3000015 | |
| Nuclease-Free Water | Life Technologies | AM9930 | |
| Bbs I | New England Biolabs | R0539S | |
| NEB Buffer 2 | New England Biolabs | B7002S | |
| T7 endonuclease I | New England Biolabs | M0302L | |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621L | |
| Plasmid EndoFree-Midi Kit | Qiagen | 12143 | |
| MMESSAGE MMACHINE T7 ULTRA | Life Technologies | AM1345 | |
| MEGACLEAR KIT 20 RXNS | Life Technologies | AM1908 | |
| MEGASHORTSCRIPT T7 KIT 25 RXNS | Life Technologies | AM1354 | |
| Flaming/Brown Micropipette Puller | Sutter Instrument | P-97 | Micropipette Puller (parameters: heat, 74; pull, 60; velocity, 80; time/delay, 200; pressure, 300) |
| Borosilicate glass | Sutter Instrument | B100-78-10 | type of capillaries (outer diameter 1.0 mm, inner diameter 0.78 mm with filament) |
| FemtoJet microinjector | Eppendorf | ||
| Freezing microtome | Leica | CM1950-Cryostat | thickness of 40 μm for brain, 10 μm for liver |
| Rabbit anti-mCherry | GeneTex | ||
| Cy3-AffiniPure Goat Anti-Rabbit IgG | Jackson Immunoresearch | ||
| DMEM | Gibco | 11965092 | |
| FBS | Gibco | 10099141 | |
| NEAA | Gibco | 11140050 | |
| Pen,Strep,Glutamine | Gibco | 10378016 | |
| Gel Extraction Kit | Omega | D2500-02 | |
| FACS | BD AriaII | ||
| PMSG | Ningbo Sansheng Medicine | S141004 | |
| HCG | Ningbo Sansheng Medicine | B141002 | |
| Cytochalasin B | Sigma | CAT#C6762 | |
| KSOM+AA with D-Glucose and Phenol Red | Millipore | CAT#MR-106-D | |
| M2 Medium with Phenol Red | Millipore | CAT#MR-015-D | |
| Mineral oil | Sigma |
References
- Yang, H., et al. Generation of Mice Carrying Reporter and Conditional Alleles by CRISPR/Cas-Mediated Genome Engineering. Cell. 154 (6), 1370-1379 (2013).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nature Biotechnology. 29 (8), 731-734 (2011).
- Nakade, S., et al. Microhomology-mediated end-joining-dependent integration of donor DNA in cells and animals using TALENs and CRISPR/Cas9. Nature Communications. 5, 5560 (2014).
- Hisano, Y., et al. Precise in-frame integration of exogenous DNA mediated by CRISPR/Cas9 system in zebrafish. Scientific reports. 5, 8841 (2015).
- Yao, X., et al. Cas9 - Mediated Precise Targeted Integration In Vivo Using a Double Cut Donor with Short Homology Arms. EBioMedicine. , (2017).
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome research. 24 (1), 142-153 (2014).
- Maresca, M., Lin, V. G., Guo, N., Yang, Y. Obligate ligation-gated recombination (ObLiGaRe): custom-designed nuclease-mediated targeted integration through nonhomologous end joining. Genome Research. 23 (3), 539-546 (2013).
- Suzuki, K., et al. In vivo genome editing via CRISPR/Cas9 mediated homology-independent targeted integration. Nature. 540 (7631), 144-149 (2016).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Yao, X., et al. Homology-mediated end joining-based targeted integration using CRISPR/Cas9. Cell Research. 27 (6), 801-814 (2017).
- Han, D. W., et al. Direct reprogramming of fibroblasts into epiblast stem cells. Nature Cell Biology. 13 (1), 66-71 (2011).
- Han, D. W., et al. Direct Reprogramming of Fibroblasts into Neural Stem Cells by Defined Factors. Cell Stem Cell. , (2012).
- Ambasudhan, R., et al. Direct reprogramming of adult human fibroblasts to functional neurons under defined conditions. Cell Stem Cell. 9 (2), 113-118 (2011).
- Sparman, M., et al. Epigenetic reprogramming by somatic cell nuclear transfer in primates. Stem Cells. 27 (6), 1255-1264 (2009).
- Schatten, G., Mitalipov, S. Developmental biology: Transgenic primate offspring. Nature. 459 (7246), 515-516 (2009).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Quadros, R. M., et al. Easi-CRISPR: a robust method for one-step generation of mice carrying conditional and insertion alleles using long ssDNA donors and CRISPR ribonucleoproteins. Genome Biology. 18 (1), 92 (2017).
- Park, K. E., et al. Targeted Gene Knockin in Porcine Somatic Cells Using CRISPR/Cas Ribonucleoproteins. International journal of molecular sciences. 217 (6), (2016).
- Woo, J. W., et al. DNA-free genome editing in plants with preassembled CRISPR-Cas9 ribonucleoproteins. Nature biotechnology. 33 (11), 1162-1164 (2015).
- Harms, D. W., et al. Mouse Genome Editing Using the CRISPR/Cas System. Current protocols in human genetics. 83, 11-27 (2014).
- Yang, H., Wang, H., Jaenisch, R. Generating genetically modified mice using CRISPR/Cas-mediated genome engineering. Nature protocols. 9 (8), 1956-1968 (2014).
- Grompe, M., et al. Loss of Fumarylacetoacetate Hydrolase Is Responsible for the Neonatal Hepatic-Dysfunction Phenotype of Lethal Albino Mice. Genes & development. 7 (12), 2298-2307 (1993).
- Paulk, N. K., et al. Adeno-associated virus gene repair corrects a mouse model of hereditary tyrosinemia in vivo. Hepatology. 51 (4), 1200-1208 (2010).
Tags
유전학 문제 133 CRISPR/Cas9 대상된 통합 호몰로지 중재 끝에 합류 Vivo에서 배아 유전자 변형 마우스 유체 주입Erratum
Formal Correction: Erratum: CRISPR/Cas9-mediated Targeted Integration In Vivo Using a Homology-mediated End Joining-based Strategy
Posted by JoVE Editors on 03/10/2021.
Citeable Link.
An erratum was issued for: Studying TGF-β Signaling and TGF-β-induced Epithelial-to-mesenchymal Transition in Breast Cancer and Normal Cells. The phrases "surveyor assay" and "Surveyor Nuclease" have been updated to "T7E1 assay" to " T7 endonuclease I" respectively.
Step 1.2 in the Protocol has been updated from:
- Surveyor nuclease assay of sgRNA
NOTE: The targeting efficiency of the sgRNA used for the knock-in experiment is evaluated by surveyor nuclease assay (also known as T7 endonuclease I (T7EI) assay)17. Select the sgRNA with high DNA cleavage efficiency and a low distance between the sgRNA cutting site and the stop codon.
to:
- T7 endonuclease assay of sgRNA
NOTE: The targeting efficiency of the sgRNA used for the knock-in experiment is evaluated by T7 endonuclease (T7EI) assay17. Select the sgRNA with high DNA cleavage efficiency and a low distance between the sgRNA cutting site and the stop codon.
Figure 1 in the Representative Results has been updated from:
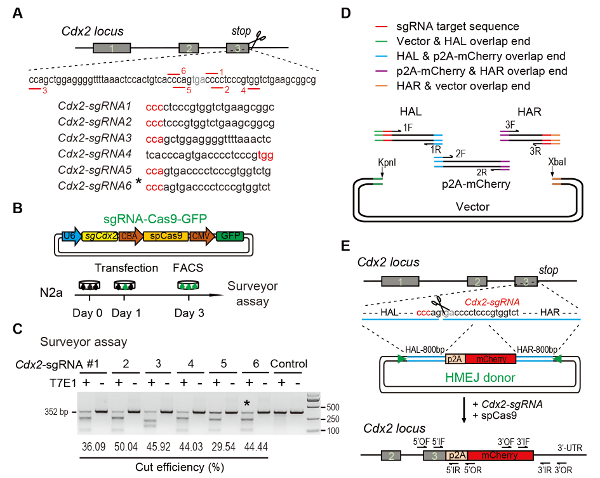
Figure 1: HMEJ-mediated targeted integration in vitro.
(A) Experimental scheme for selection of sgRNAs: Six different sgRNAs (Cdx2-sgRNA1~Cdx2-sgRNA6) around the stop codon of the Cdx2 locus with a higher rank and off-target potential were chosen based on online CRISPR design tool. The protospacer adjacent motif (PAM) sequence is in red. (B) Experimental design: The Cas9-CMV-GFP expression plasmids expressing sgRNA, Cas9, and GFP were introduced into N2a cells. GFP+ cells were sorted at day 3 for surveyor assay. (C) Surveyor assay for Cdx2 targeting: 6 different sgRNAs were designed for surveyor assay. Normal N2a cell genomic DNA serves as control. *, the sgRNA used for Cdx2-2A-mCherry knock-in experiment. (D) Schematic overview of construction of HMEJ donors using Gibson assembly. (E) Schematic overview of HMEJ-mediated gene targeting strategy at Cdx2 locus. HAL/HAR, left/right homology arm; triangles, sgRNA target sites; OF/OR, outer forward/reverse primer; IF/IR, inner forward/reverse primer. Figure modified from previous report10. Please click here to view a larger version of this figure.
to:
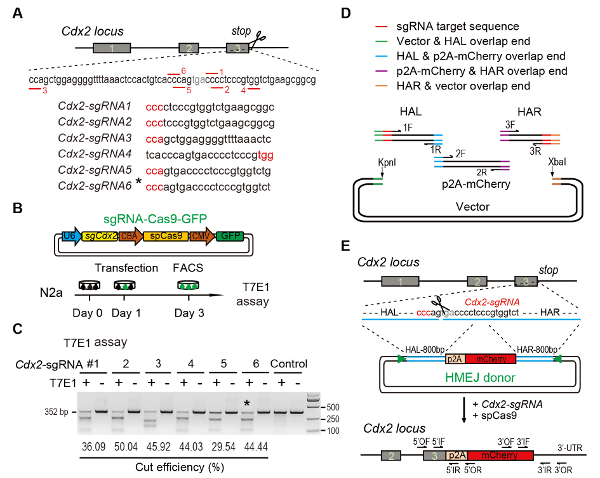
Figure 1: HMEJ-mediated targeted integration in vitro.
(A) Experimental scheme for selection of sgRNAs: Six different sgRNAs (Cdx2-sgRNA1~Cdx2-sgRNA6) around the stop codon of the Cdx2 locus with a higher rank and off-target potential were chosen based on online CRISPR design tool. The protospacer adjacent motif (PAM) sequence is in red. (B) Experimental design: The Cas9-CMV-GFP expression plasmids expressing sgRNA, Cas9, and GFP were introduced into N2a cells. GFP+ cells were sorted at day 3 for T7EI assay. (C) T7EI assay for Cdx2 targeting: 6 different sgRNAs were designed for T7EI assay. Normal N2a cell genomic DNA serves as control. *, the sgRNA used for Cdx2-2A-mCherry knock-in experiment. (D) Schematic overview of construction of HMEJ donors using Gibson assembly. (E) Schematic overview of HMEJ-mediated gene targeting strategy at Cdx2 locus. HAL/HAR, left/right homology arm; triangles, sgRNA target sites; OF/OR, outer forward/reverse primer; IF/IR, inner forward/reverse primer. Figure modified from previous report10. Please click here to view a larger version of this figure.



