Summary
이 프로토콜은 레이저 미세 조사에 의한 DNA 수리 단백질 모집 측정과 같은 하류 현미경 연구를 위한 S상 세포를 효율적으로 식별하는 비침습적 방법을 설명합니다.
Abstract
DNA 손상 복구는 반응성이 높은 환경에서 세포의 유전 적 무결성을 유지합니다. 세포는 대사 활동 또는 UV 방사선과 같은 내인성 및 외인성 소스로 인해 다양한 유형의 DNA 손상을 축적할 수 있다. DNA 복구없이, 세포의 유전 코드는 단백질의 구조와 기능을 훼손하고 잠재적으로 질병을 일으키는 손상된다.
다양한 세포 주기 단계에서 다른 DNA 수리 경로의 현면역학을 이해하는 것은 DNA 손상 복구 분야에서 매우 중요합니다. 현재 형광 현미경 검사는 DNA 손상 유도 후 다른 수리 단백질의 모집 역학을 측정하는 훌륭한 도구를 제공합니다. 세포 주기의 S 단계 도중 DNA 합성은 DNA 복구에 관하여 세포 운명에 있는 특이한 점입니다. 그것은 실수에 대한 전체 게놈을 선별하는 독특한 창을 제공합니다. 동시에, DNA 합성 오류는 또한 비 분할 세포에서 발생하지 않는 DNA 무결성에 위협을 제기합니다. 따라서, DNA 복구 과정은 세포 주기의 그밖 단계에 비교된 S 상에서 현저하게 다르고, 그 다름은 제대로 이해되지 않습니다.
다음 프로토콜은 405 nm 레이저 라인을 갖춘 레이저 스캐닝 공초점 현미경을 사용하여 로컬로 유도된 DNA 손상 부위에서 S 상에서 DNA 수리 단백질의 역학의 제조 및 측정을 설명합니다. 태그된 PCNA(mPlum와 함께)는 AcGFP-표지된 수리 단백질(즉, EXO1b)과 결합된 세포 주기 마커로서 S 상에서 DNA 손상 모집을 측정합니다.
Introduction
몇몇 DNA 복구 통로는 세포에서 생길 수 있는 DNA 병변의 다른 모형을 해결하기 위하여 발전했습니다, 모두는 공간과 시간 둘 다에서 높게 통제됩니다. 세포 주기의 가장 취약한 기간 중 하나는 DNA 합성이 발생할 때 S 단계입니다. 확산은 삶의 기본이지만, 또한 중요한 도전을 제공합니다. 세포는 돌연변이가 미래 세대에게 전해지는 것을 피하기 위하여 그들의 게놈의 충실한 복제를 보장할 필요가 있습니다. 따라서, 증식은 종양학 분야의 치료 접근법의 발달을 위해 사용된 내정간섭의 치료 포인트를 제공합니다.
DNA 병변에서 단백질 모집을 연구하는 데 사용되는 모든 주요 기술은 강점과 한계를 가지고 있습니다. 미세 조사는 이온화 방사선 유도 된 foci (IRIF), 크로마틴 -면역 침전 (ChIP) 또는 생화학 분획의 면역 형광 화상 진찰과 같은 대체 방법의 대부분보다 더 나은 공간 및 측두성 해상도1을 갖는다. 그러나, 마이크로 조사는 다수의 세포를 동시에 샘플링할 수 있는 전술한 기술의 견고성을 결속시한다.
S 단계에서 DNA 수리를 조사하기 위해 비동기 세포 배양 집단에서 S 상 세포를 구별할 수 있어야 합니다. 세포의 동기화 또는 다른 세포 주기 단계의 시각화를 포함하는 이 문제를 해결하기 위하여 많은 잘 알려진 방법이 있습니다. 그러나 두 가지 접근 법은 중요한 도전과 가능한 유물을 소개합니다. 초기 S 상에서 세포를 농축하는 데 널리 사용되는 화학 적 동기화 방법 (예를 들어, 이중 티미딘 블록, 아피디콜린 및 하이드록수유레아 처리)는 복제 스트레스의 유도를 통해 동기화를 달성하고 결국 DNA 손상 자체. 이것은 S 상2에서DNA 복구 프로세스를 연구하기 위하여 이 방법의 사용을 제한합니다. 혈청 기아 및 방출을 통한 동기화는 비변형 세포주에 비해 세포 주기 진행을 위한 성장 인자에 덜 의존하는 암 세포주를 주로 제외하는 제한된 수의 세포주에만 적용됩니다. 형광 유비퀴틴 세포 주기 표시기 (FUCCI) 시스템은 세포 주기를 연구하는 데 특히 유용한 도구이지만 S와 G2 세포 주기 단계3을 분화 할 때 근본적인 한계를 가지고 있습니다.
여기서 는 형광 태그PCNA를 S 상에 대한 비침습적 마커로서 사용함으로써 화학 세포 주기 동기화 방법의 단점을 제한하면서 FUCCI 시스템보다 더 특이성과 유연성을 허용하는 것으로 나타났다. 단일 마커로서 PCNA는 비동기 집단에서 S-위상 세포를 강조할 수 없을 뿐만 아니라 S 상(즉, 조기, 중, 또는 후기 S-phase)4내의 세포의 정확한 진행을 보여줄 수도 있다. 태그가 붙은 PCNA의 낮은 발현 수준은 세포 주기 진행 및 DNA 복구 과정 모두에 대한 최소한의 간섭을 보장합니다. 중요한 것은, PCNA는 또한 여러 DNA 병변의 수리에 관여하고 국소 유도된 DNA 손상 부위1,4로모집되기 때문에 적절한 DNA 손상 유도에 대한 내부 통제 역할을 한다.
여기에 제시된 실험은 S 단계에서 EXO1b의 채용 역학을 측정하는 방법과 이것이 잘 확립 된 PARP 억제제인 olaparib에 의해 어떻게 영향을받는지 보여줍니다. EXO1b 뉴클레아제 활성은 불일치 수리(MMR), 뉴클레오티드 절제 수리(NER), 이중 좌초 브레이크(DSB) 수리를 포함한 광범위한 DNA 수리 경로와 관련이 있다. S 단계에서 EXO1b는 DNA 절제술5동안 3'sSDNA 오버행의 형성을 통해 상동성 재조합(HR)에서 중요한 역할을 한다. EXO1b는 검사점 활성화의 역할과 함께 DNA 복제에 더욱 연루되어 정체된 DNA 포크뿐만 아니라복제5에서가닥 변위 중 지연가 닥치는 가닥에서 프라이머 제거 및 오카자키 단편 성숙을 다시 시동하고 있다. 손상된 DNA 부위에 대한 EXO1b 모집은 폴리(ADP-ribose)(PAR)6,7과의직접적인 상호 작용에 의해 조절된다. EXO1b의 수많은 세포 주기 특이적 의미로 인해 PCNA를 이용한 S-Phase 특정 채용 연구에 탁월한 선택입니다.
Protocol
1. 인간 골육종 유래 세포의 재배 (U-2 OS)
참고: U-2 OS 세포는 평평한 형태, 큰 핵을 가지고 있으며 유리를 포함한 여러 표면에 강하게 부착되기 때문에 이러한 연구에 이상적입니다. 유사한 특성을 가진 그밖 세포주도 이용될 수 있었습니다.
- U-2 OS 세포주를 재배하려면 10% 태아 소 혈청(FBS) 및 항생제(100 U/mL 페니실린 및 100μg/mL 연쇄절제술)로 보충된 McCoy의 5A 배지를 사용하십시오. 5%CO2를함유하는 가습된 대기에서 37°C에서 세포를 배양한다. 현미경 검사법의 경우, 충분한 세포 수를 제공하기 위해 10cm 접시에 세포 배양을 유지합니다.
- 세포가 90%의 합류(~7 x 106 세포/10cm 접시)에 접근할 때, 세포를 분할합니다.
- PBS로 세포를 헹구고 혈청 내에 함유 된 트립신 억제제를 씻어 낸다.
- 트립신-EDTA의 1mL을 추가하고 세포 층이 동일하게 덮여 있는지 확인합니다.
- 세포 층이 플레이트에서 들어올릴 때까지 37°C에서 배양(약 6분).
- 트립신을 비활성화하는 매체를 포함하는 혈청에서 트립시네이징 된 세포를 다시 중단하고 부피의 1/10th (~0.7 x 106 셀)를 보충 된 성장 배지 10 mL을 포함하는 새로운 10cm 플레이트에 추가합니다.
- 실험 전에 제조업체의 권고에 따라 범용 마이코플라즈마 검출 키트를 사용하여 마이코플라즈마 오염에 대한 세포를 일상적으로 테스트합니다.
2. 레트로바이러스 감염
참고: BSL-2 안전 대책의 경우 재조합 바이러스와 함께 작업하는 동안 NIH 지침, 섹션 III-D-3: 조직 배양에 있는 재조합 바이러스를 참조하십시오.
- 종자 4 x 106 HEK293T 셀은 10cm 배양 접시로 도금 한 후 24 시간 이내에 ~60 % 인플루엔성을 달성한다.
- HEK293T 재배를 위해 이 프로토콜의 1.1-1.3에 설명된 U-2 OS의 재배 단계를 따르십시오. HEK293T의 경우 McCoy의 5A 매체를 DMEM용으로 대체합니다. HEK293T 세포를 조직 배양 판에 약하게 부착할 때 항상 부드럽게 씻어야 합니다.
- 플라스미드의 바이러스 성 포장을 위한 지질 계 형 형질 전환 시약을 사용하는 Transfect HEK293T 세포.
- 레트로바이러스 벡터의 경우, VSV-G(Addgene #8454)의 1.5 μg와 pUMVC(Addgene #8449) 패키징 벡터의 1.5 μg와 관심 유전자를 포함하는 벡터의 3 μg(백본에서 푸르오마이신 저항성)을 옵티 메미센트 마이크로리의 250L에 결합한다. Opti-MEM/DNA 혼합물(이 경우 6 μL)에 추가된 각 DNA μg에 대해 P3000 시약 1μL을 추가하고 탭하여 부드럽게 섞습니다. 소용돌이 나 파이펫을 위아래로하지 마십시오.
- 또 다른 미세센심분리기 튜브에서는 옵티-MEM 감소혈청 의 250 μL과 형질 검사 시약의 μg DNA 당 2 μL(이 경우 12 μL)을 결합합니다.
- 두 혼합물 (500 μL 결합, 소용돌이하지 말고 부드러운 도청으로만 섞음)을 결합하고 실온에서 15 분 동안 배양하십시오.
- 조심스럽게, 세포를 분리하지 않고 시드 HEK293T 세포에 혼합물을 드롭 와이즈로 추가합니다. 접시를 부드럽게 소용돌이시다.
- 안정적인 세포주를 생성하는 바이러스 감염.
- FEFe후 72h의 HEK293T 세포로부터 상류체를 함유하는 바이러스를 제거한다. 0.45 μm 필터로 용액을 조심스럽게 필터링하여 세포 이물질과 분리된 세포를 제거합니다. 선택적으로, 바이러스 성 감염을 용이하게하기 위해 바이러스 성 상수에 8 μg / mL 폴리브레인을 추가합니다.
- 10cm 접시 (~3 x 106 셀)에서 ~50 % 합류에서 U-2 OS 세포에 수퍼를 포함하는 바이러스를 추가합니다. 전날 U-2 OS 셀을 시드합니다.
- 바이러스 함유 상체를 제거하고 폐기하기 전에 6-16h에 감염하십시오.
참고: 관심 있는 유전자에 대해 원하는 양의 과발현을 달성하기 위해 고정된 시간 동안 일련의 바이러스 희석을 배양합니다. 내인 성 수준과 비교하는 서양 얼룩을 가진 각각새로 확립된 세포주에서 전환유전자의 발현 수준을 확인하십시오. - 세포가 적절한 항생제의 존재하에서 선택할 수 있도록 (2 μg /mL 최종 농도에서 푸오마이신의 경우 3-4 일 동안) 현미경하에서 관심있는 형광 단백질 태그 유전자의 발현을 확인하십시오.
- 이중 표지된 셀주를 생성하려면 이러한 단계를 반복합니다. 여기에 제시된 실험에서 mPlum-PCNA는 EXO1B-AcGFP와 결합된 레트로바이러스 벡터(pBABE)로부터 발현되었으며, 또한 레트로바이러스 벡터로부터 발현되었다(pRetroQ-AcGFP1-N1).
3. 미세 조사를 위한 세포 준비
- 도금 세포: 실험 전 24시간, 총 8.0 x 104 세포를 500 μL-1 mL 사이의 부피로 플레이트하여 4번의 잘 챔버커버글래스에 1.5호 보로실리케이트 유리 바닥에 미세한 미생물및 미세레이저 에 대한 이상적인 결과를 제공한다. 세포 결도가 높을수록 단일 시야(FOV)에서 측정되는 더 많은 세포가 허용됩니다. 그러나 완전히 컨할 수 있는 슬라이드는 세포 주기 요철을 소개합니다.
- 이미징 미디어: 마이크로 조사 한 시간 전에, 플루오로브리테 DMEM에 대한 정기적 인 성장 매체를 교환 10% FBS, 100 U / mL 페니실린과 100 μg / mL 연쇄 절제술, 15 mM HEPES (pH = 7.4) 및 1mM 나트륨 피바테. 이 이미징 미디어는 신호 대 잡음 비율을 최대화하여 매우 희미한 형광을 감지할 수 있도록 도와줍니다. HEPES를 함유하고 있기 때문에 5% CO2 대기가 없는 경우 pH도 안정화됩니다.
- 이 단계에서 이미징 전에 추가 치료를 적용하십시오. 여기에 제시된 실험에서, 세포는 olaparib (PARP 억제제, 1 μM 최종 농도에서) 또는 차량 제어 (DMSO)1,8,9로이미징하기 전에 1 시간 전에 미리 처리되었다.
4. 현미경을 준비하고 이미징을 위한 S 상 세포를 선택합니다.
- 최상의 결과를 위해 여기에 설명된 시스템과 유사한 특성을 가지고 있는 공초점 시스템을 사용합니다. 여기에 제시된 실험은 반전된 현미경 스탠드에 장착된 공초점 현미경을 사용하여 수행되었습니다(재료표참조).
참고 : 여기에 사용되는 현미경은 50mW 405 nm FRAP 레이저 모듈과 60x 1.4 NA 오일 계획 - 아포크로마트 목표를 장착했다. 공초점 스캔헤드에는 갈바노 스캐너(고해상도용) 및 공진 스캐너(고속 이미징용)의 두 가지 스캐너 옵션이 있습니다.- 소프트웨어 제어 XY 갈바노 장치를 통해 샘플에 광표백(FRAP) 레이저(FRAP) 레이저후 형광 복구를 소개합니다. 488 nm 레이저 라인을 사용하여 AcGFP와 561 nm 또는 594 nm 레이저 라인을 자극하여 mPlum을 흥분시킵니다.
참고: 다음 필터 조합은 최적의 결과를 제공합니다: 560 nm 길이의 패스 필터를 사용하여, 560 nm보다 낮은 파장을 가진 방출 광은 AcGFP를 위한 525/50 nm 방출 필터를 통과하고, 560nm 이상의 파장을 가진 방출 광은 595/50 nm 방출 필터를 통과시켰다. 최소한의 형광 출혈을 보장하는 모든 적절한 필터 세트(예: FITC/TRITC, GFP/mCherry, FITC/TxRed)를 사용할 수 있습니다.
- 소프트웨어 제어 XY 갈바노 장치를 통해 샘플에 광표백(FRAP) 레이저(FRAP) 레이저후 형광 복구를 소개합니다. 488 nm 레이저 라인을 사용하여 AcGFP와 561 nm 또는 594 nm 레이저 라인을 자극하여 mPlum을 흥분시킵니다.
- 환경 챔버와 현미경 구성 요소를 켭니다.
- 가열(스테이지, 목표 및 환경 챔버 가능시),CO2 공급 및 습도 조절기를 실험 시작 4시간 이상 켜면 안정적인 이미지 수집을 위한 열 평형을 보장한다.
- 현미경으로 세포의 전송 하기 전에 적어도 1 h 레이저 라인과 함께 광 원을 초기화.
- 형광 태그PCNA를 마커로 사용하여 비동기 집단에서 S-phase 세포를 선택합니다. 아래 단계에 따라 이 작업을 수행합니다.
- S 상에서 mPlum 태그PCNA의 고유한 국소화 패턴을 찾아이 세포 주기 단계를 식별할 수 있습니다. PCNA는 세포 주기의 G1 및 G2 상에서 핵에 완전히 균일한 분포를 가지며, 핵에서 제외된다. S-phase에서 PCNA는 핵에서 replisomes의 위치에 foci를 형성한다. 도 1은 S-phase 전반에 걸쳐 PCNA 포시의 다양한 패턴을 나타내며, 이를 통해 초기, 중, 후반 S-phase를 구별할 수 있습니다.
- 현미경을 통해 현미경 조사를 위한 충분한 S-위상 세포가 있는 FOV를 선택하십시오. 비동기 U-2 OS 세포는 일반적으로 S 단계에서 그들의 인구의 30-40%를 가지고 있습니다.
- PCNA와 관심 있는 단백질(POI)에 대한 발현 수준(밝고 희미한 세포)에서 극한을 피하십시오.이 경우 EXO1b-AcGFP는 실험적인 유물로 이어질 수 있습니다.
- 적합한 FOV를 찾을 때 광표백 및 원치 않는 DNA 손상을 최소화하기 위해 장시간 필드를 스캔하지 마십시오.
- 마이크로 조사를 위해 원하는 관심 영역(ROI)을 설정합니다. 관련 소프트웨어(재료 표 참조)를사용하여 먼저 바이너리 라인을 삽입하여 원하는 ROI를 설정합니다(원하는 라인 수 및 간격 설정). 바이너리를클릭한 다음 삽입 선을 클릭| 원 | 타원은 원하는 줄 수를 그립니다.
- 이러한 이진 선을 ROI로 변환하고 마지막으로 이러한 ROI를 자극 ROI로 변환합니다. 이렇게 하려면 먼저 ROI를클릭한 다음 이진 이동을 ROI로클릭한 다음 ROI를 마우스 오른쪽 단추로 클릭하고 자극 ROI: S1로 사용을선택합니다. 이러한 선을 FOV에 배치하여 세포의 핵을 통과한다. 전체 FOV에 걸쳐 1024 픽셀의 길이를 가진 ROI는 프로토콜 전반에 걸쳐 사용되었습니다.
5. 면역 형광 염색 또는 시간 경과 이미징에 대한 미세 조사.
- 최적의 미세 조사 설정을 결정합니다.
- 세포의 미세 조사 전에 FOV의 고해상도 이미지를 사용하여 나중에 분석을 위해 PCNA 포시를 식별하십시오. 순차적 스캐닝 대신 두 파장에서 스캔하는 사이의 세포 이동을 피하기 위해 사용되는 광학 채널(녹색과 빨간색)을 동시에 기록합니다. foci의 적절한 해상도를 위해 1x 줌(여기서 사용되는 이미징 시스템의 0.29 μm 픽셀 크기)이 있는 최소 1024 x 1024 픽셀/필드 해상도를 사용하며, 평균 2배의 1/8 프레임/s 스캐닝 속도(4.85 μs/pixel)를 사용합니다. 이러한 매개 변수가 A1 LFOV 컴팩트 GUI및 A1 LFOV 스캔 영역 창에서 설정되면 캡처 버튼을 눌러 FOV를 기록합니다.
참고: 유사한 결과를 보장하기 위해 실험 전반에 걸쳐 동일한 픽셀 크기를 유지하는 것이 중요합니다. - 마이크로 조사를 설정하려면 이미징 소프트웨어의 ND 자극 탭을 열어 시간 일정(A1 LFOV/Galvano Device) 창에 액세스합니다. 이것은 갈바노 스캐너를 사용하여 일련의 사전 자극 이미지를 획득하고 자극 (LUN-F 50 mW 405 nm FRAP 레이저사용)한 다음 갈바노 스캐너를 사용하여 일련의 자극 후 이미지를 다시 획득합니다. 먼저 시간 일정 창에 세 단계를 설정합니다. Acq/Stim 열에서 인수 | 선택 표백 | 각각 3단계에 대한 인수. 표백 단계의 경우 S1을 ROI로 설정합니다.
참고: 여기에 제시된 실험에서는 자극 단계에서 이미지가 수집되지 않았습니다. - Galvano XY 창에서마이크로 조사를 위한 주요 요소를 설정: 405 nm 레이저 출력, 거주 시간 (반복은 이 시스템에 기본적으로 1). 여기에 제시된 실험에서, 세포는 1000-3000 μs 가성 시간으로 100% 출력에서 405 nm FRAP 레이저(섬유 팁에서 50mW)로 조사되었다.
참고: 레이저 거주 시간은 픽셀 단위로 유지되므로 픽셀 크기가 동일하게 유지되는 한 거주 시간과 전력 밀도 간의 관계는 다른 FOV 간에 유사합니다. 도 2A는 특정 손상 유도에 대한 레이저 전력 설정을 최적화하기 위해 DNA 손상 반응(DDR) 경로 특이적 단백질(DSBs및 NTHL1용 DXL10)의 사용을 나타낸다. 이러한 안정적인 세포주 프로토콜의 섹션 2 다음 바이러스 감염으로 생성되었다.
- 세포의 미세 조사 전에 FOV의 고해상도 이미지를 사용하여 나중에 분석을 위해 PCNA 포시를 식별하십시오. 순차적 스캐닝 대신 두 파장에서 스캔하는 사이의 세포 이동을 피하기 위해 사용되는 광학 채널(녹색과 빨간색)을 동시에 기록합니다. foci의 적절한 해상도를 위해 1x 줌(여기서 사용되는 이미징 시스템의 0.29 μm 픽셀 크기)이 있는 최소 1024 x 1024 픽셀/필드 해상도를 사용하며, 평균 2배의 1/8 프레임/s 스캐닝 속도(4.85 μs/pixel)를 사용합니다. 이러한 매개 변수가 A1 LFOV 컴팩트 GUI및 A1 LFOV 스캔 영역 창에서 설정되면 캡처 버튼을 눌러 FOV를 기록합니다.
- 시간 경과 이미징.
- 시간 일정, A1 LFOV 컴팩트 GUI 및 A1 LFOV 스캔 영역 창을 사용하여 원하는 시간 창 및 간격에 대한 시간 경과 이미징을 설정합니다. 여기에 제시된 실험에서 EXO1b와 PCNA의 모집은 12분 동안 이미지화되어 5초마다 FOV를 스캔하여 1024 x 1024 픽셀/필드에서 1x 줌(여기서 사용되는 이미징 시스템에서 0.29 μm 픽셀 크기)을 사용하여 평균 0.35 프레임/s 스캐닝 속도(1.45 μs/픽셀)를 표백하지 않고 평균 적으로 감소시켰습니다.
- A1 LFOV 컴팩트 GUI 창에서 이미징 중에 사진 표백을 줄이기 위해 레이저 전력%%, 게인 및 오프셋 설정을 최적화합니다. POI와 PCNA를 모두 측정하는 것을 목표로 하는 경우, 두 개의 별도 형광에 대한 필드를 스캔하는 사이의 세포 이동을 피하기 위해 순차적 스캐닝 대신 동시 스캐닝을 사용합니다.
- 이미징 시스템은 다음 설정과 함께 사용되었습니다. 488 nm 레이저 라인 (20 mW)의 경우 : 7 % 레이저 전력, 게인 : 45 (GaAsP 검출기) 와 2, 561 nm 레이저 라인 (20 mW)에 대한 오프셋 : 4 % 레이저 전력, 40 (GaAsP 검출기)와 2의 오프셋.
- 단백질의 운동학에 따라 이미지 간의 간격또는 총 시간 경과의 간격을 연장하거나 단축합니다. 시간 일정 창에서 세 번째 단계 수집 행에 대해 원하는 간격 과 기간을 설정합니다.
- 지금 실행을 눌러 미세 조사 및 후속 시간 경과 이미징을 실행합니다.
- 시간 경과 이미징의 끝에서 자극 ROI를 별도의 이미지로 저장하여 분석에 사용되는 다운스트림 소프트웨어에서 미세 조사 좌표를 식별하는 데 유용합니다.
- 면역 형광 염색.
참고: 단계 5.1.3 및 도 2A는 마이크로 조사에 의해 도입된 DNA 병변의 종류를 평가하기 위하여 알려진 DNA 복구 단백질의 사용을 보여줍니다. 특정 DNA 병변은 또한 세포를 고정한 후에 특정 항체를 사용하여 검출될 수 있습니다. 또한 내인성 단백질의 항체 검출에 의한 POI의 모집을 검출하는 것도 가능하다. DSB를 확인하는 γH2A.X의 시각화는아래(그림 2B)에서입증된다. 도 3은 내인성 및 외인성 태그 PCNA 모두에 대한 세포 주기 전반에 걸쳐 PCNA 국소화 및 모집의 일관성을 나타낸다.- 5.1.3 단계 후, mPlum-PCNA의 모집에 따라 적절한 FRAP 이벤트를 보장하기 위해 미세 조사 후 하나의 이미지를 가져 가라. 면역 형광 라벨 링 후 나중에 필드를 찾기 위해 FOV의 정확한 좌표를 기록하십시오.
- 5-10 분 동안 5 %CO2를 포함하는 가습 된 분위기에서 현미경및 37 °C에서 세포 배양 챔버를 꺼내서 세포를 배양하십시오.
참고: 파라포름알데히드(PFA)는 독성이 있으며 통풍이 잘 되는 부위 나 연기 후드에서 작업을 수행해야합니다. 모든 후속 세척 및 배양은 4 웰 챔버 슬라이드에서 0.5 mL 볼륨으로 수행됩니다. 인큐베이션 시간 후, PBS(137m NaCl, 2.7m MM KCl, 8m MM Na2HPO4,2mM KH2PO4)로세포를 세척하고 실온(RT)에서 10분 동안 PBS에서 4%의 PFA0.5mL로 수정한다. - PBS로 세포를 한 번 씻은 다음 50m NH4Cl로 씻어 잔류 PFA를 담금질하십시오.
- PBS에서 0.1% 트리톤 X-100으로 RT에서 15분 동안 세포를 퍼메아빌화합니다.
- 블로킹 버퍼(5% FBS, 3% BSA, PBS에서 0.05% 트리톤 X-100)로 1h의 샘플을 차단합니다.
- 차단 용액을 제거하고 RT에서 1h에 대한 차단 버퍼에 희석 된 1 차 항체 (안티 γH2A.X, 1:2000)를 추가합니다.
- 블로킹 버퍼 3 x 10 분으로 우물을 씻으시면됩니다.
- 희석 된 이차 항체 (안티 마우스 알렉사 488 플러스 공주, 1:2000)를 RT에서 1 h에 대한 버퍼를 차단하는 데 추가하십시오.
- 블로킹 버퍼 3 x 10 분으로 우물을 씻으시면됩니다.
- 15 분 동안 PBS에서 1 μg / mL DAPI 용액으로 핵을 카운터 스테인하십시오.
- PBS로 한 번 셀을 씻으시다. 이미징은 PBS 또는 안티페이드 시약(예를 들어, AFR3)을 사용하여 PBS 용액에서 직접 수행하여 광표백을 줄일 수 있습니다.
6. 채용 분석
참고: 그림 4A는 DMSO 또는 olaparib의 존재에 엑소1b및 PCNA 모집의 대표적인 이미지를 보여줍니다. 도 4B는 데이터 분석을 위한 대표적인 이미지를 나타낸다. 평균 형광 값은 피지를 사용하여 다른 시점에서 mPlum-PCNA (A, 노란색 사각형)에 의해 강조 된 레이저 트랙을 따라 사각형을 사용하여 평균 AcGFP 강도를 측정하여 계산되었다. PCNA는 ROI 좌표를 따라 성공적인 조사를 강조하는 내부 제어 역할을 할 수 있습니다. 유사하게, 평균 AcGFP 형광 값은 또한 핵의 손상되지 않은 영역에 대해 계산되었다 (B, 파란색 사각형). 배경 신호 강도는 채밀되지 않은 영역(C, 적색 사각형)에서 측정되었으며 평균 형광값(그림 A 및 B)에서빼었다. 따라서, 각 데이터 수집 점에 대한 상대평균 형광 단위(RFU)는 방정식 RFU =(A-C)/(B−C)8,9에의해 계산되었다. 마이크로 조사 영역의 결과 RFU 값은 미세 조사 전에 RFU 값으로 정규화됩니다.
- 미세 조사 부위의 영역 A를 정의하기 위해, 세포의 뉴클레올라 영역, 복제 포시 및 불규칙한 핵 영역을 측정에서 배제한다. 피지에서 두 개의 ROI를 그리는 사이에 시프트 키를 두 개의 별도 영역을 하나로 그룹화합니다.
참고: 단백질 모집은 다른 유전자 와 조사 조건 사이에서 변화할 것입니다; 따라서 지역 A의 크기는 개별적으로 결정되어야 합니다. 지역 A의 픽셀 너비가 결정되면 비교 채용에 대해 일정하게 유지되어야 합니다. 여기에 제시된 실험에서 7픽셀 너비 사각형이 사용되었습니다. - 기록된 비디오의 기간 동안 이동한 셀을 분석에서 제외합니다. 고도로 이동성 셀을 포함하려면, 기술된 분석은 프레임별 수행되어야 한다.
- 채용 프로필을 시각화하려면 통계 소프트웨어를 사용하여 정규화된 RFU 값을 시간에 대해 플롯합니다.
- Mann-Whitney 테스트를 사용하여 DMSO와 olaparib(n=31) 치료 사이의 표시된 시간 지점에서 차이를 계산합니다.
Representative Results
세포는 또한 어떤 세포 주기 단계에 달려 있는 특정 방식으로 DNA 병변의 각 모형을 다룹니다. 예를 들어, 마이크로 조사 후, 이중 좌초 휴식(DSB)은 세포 주기 단계에 따라 비동형 종결 결합(NHEJ) 또는 HR에 의해 처리됩니다. 세포 주기의 S 및 G2 단계 도중 가장 광범위하게 작용하는 핵은 적당한 HR을 위해 결정적인 DNA 오버행을 만듭니다. S 상에서 세포의 평가를 촉진하기 위해 PCNA는 단일 색 세포 주기 마커로 사용되었습니다. 도 1A는 세포 주기 진행 중 mPlum-PCNA의 국소화 프로파일을 나타낸다. PCNA는 G1 및 G2 상에서 핵에 완전히 균일한 분포를 갖는다(또한 핵에서 주로 제외되는 동안). S 단계에서 PCNA는 핵에서 밝은 반점으로 시각화할 수 있는 DNA 복제 부위를 국소화합니다. 초기 S 상 세포에서, 반점은 세포의 핵을 통해 상대적으로 작고 동등하게 분포된다. S 단계 중반으로 진행하면 반점이 흐려지고 핵과 핵의 둘레를 향해 더 많이 국한됩니다. 후반 S 단계에서는 PCNA가 후기 복제사이트(그림 1B)에집중됨에 따라 반점이 감소하지만 점점 더 커집니다. 중요한 것은, pBABE 벡터 백본으로부터의 외인성 PCNA 발현은 내인성 수준보다 적었지만 세포 주기 진행 및 DDR의 잠재적 인 유물을 최소화하는 현미경 검사법에 의한 검출에 충분했다. 도 1C는 내인성 수준에 비해 PCNA 과발현의 정도를 나타낸다. mPlum-PCNA에 해당하는 밴드는 크기가 커서 느리게 마이그레이션됩니다.
우리는 S 단계에서 이러한 병변에 EXO1b의 PARP1/2 의존적 채용을 조사하기 위해 미세 조사 중에 DSB를 도입하는 것을 목표로했습니다. 도 2A는 저용량 에너지(1000 μs 거주 시간)가 EGFP-FBXL10, DSB 응답자(FRUCC 복합체 8의성분)의 모집을 유도하지 않는 반면, NTHL1-mCherry의 모집을 유도하기에 충분했지만, 염기 절제 수리(BER) 경로 단백질, 산화 DNA 손상1,12, 121의부위에 모집하는 것을 나타낸다. 3000 μs 거주 시간, EGFP-FBXL10 및 NTHL1-mCherry 모집, 산화 병변과 DSB를 모두 생성 하는 레이저 출력을 시연. 이러한 결과 강화, 도 2B는 γH2A.X에 대 한 염색 면역 fluorescence 보여줍니다 (DSB 마커), 이는 분명히 더 높은 에너지 용량을 사용 하는 경우. PCNA는 레이저 거주 시간 설정으로 적절하게 모집하기 때문에 성공적인 미세 조사를위한 세포 주기 마커와 마커 역할을합니다. 중요하게도, 외인성 및/또는 내인성 형광 단백질 태그 PCNA 모두 유사하게 행동함에 따라 이 리포터 기능에 사용될 수있다(도 3). 내인성 태그 PCNA는 PCNA 궤적13의 하나의 대립구체에 첫 번째 엑시온과 함께 프레임에 mRuby를 삽입하여 설계되었다 (세포선은 외르그 맨스펠트의 종류 선물이었다).
도 4A 및 도 4C는 S상 세포에서 AcGFP 태그EXO1b의 모집을 나타낸다. EXO1b는 약 1 분 정도 미세 조사 사이트에 축적의 최대 수준에 도달하고 천천히 나중에 DNA 병변에서 분리하기 시작합니다. 미세 조사 부위의 농축은 그래프의 > 1 상대 형광 장치에 의해 표시됩니다. olaparib의 존재에서, 1 분에 레이저 스트라이프에 EXO1b의 축적은 차량 제어에 비해 상당히 적습니다. 이러한 결과는 문헌6,7과일치한다. 도 4B는 프로토콜의 점 6에 설명된 바와 같이 정량화(영역 A, B 및 C)에 대한 대표 영역을 보여 줍니다. 도 4D는 마이크로 조사에 사용되는 세포에서 내인성 EXO1b 및 외인성 EXO1b-AcGFP의 유사한 발현 수준을 나타낸다.
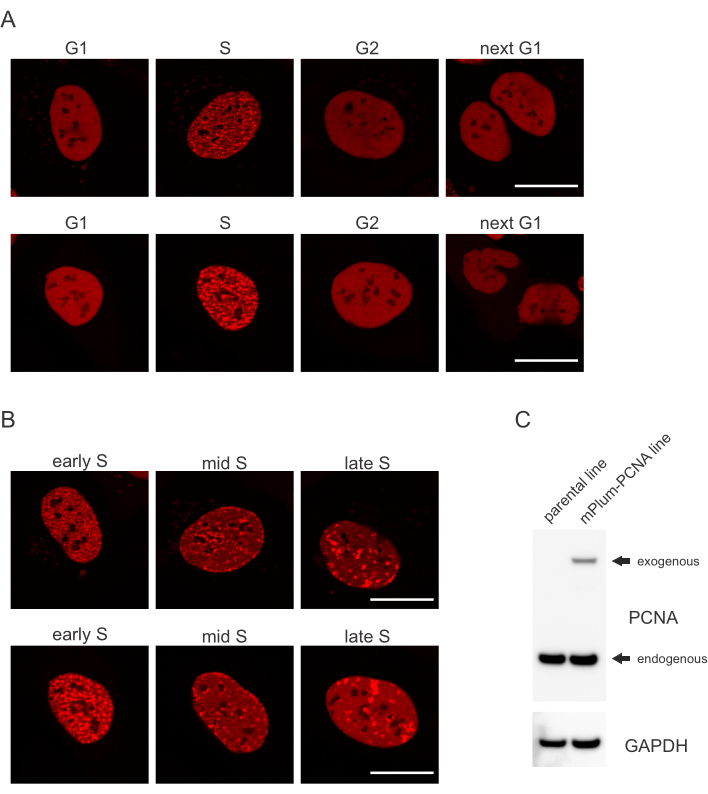
그림 1: PCNA의 국소화 패턴. (A)이미지는 U-2 OS 세포에서 세포 주기 전반에 걸쳐 안정적으로 통합된 외인성 PCNA의 국소화 패턴을 보여준다. (B)이미지는 U-2 OS 셀에서 S 상(초기, 중, 후기)의 상이한 단계에서 PCNA 포시 패턴을 보여준다. (C)영상에 사용되는 U-2 OS 세포에서 내인성 및 외인성 수준의 PCNA를 나타내는 서양 블롯. 배율 막대는 20 μm을 나타냅니다.

도 2: 최적화된 레이저 출력을 통해 DSB의 유도. (A)레이저 설정은 다양한 형태의 DNA 손상을 유도하도록 최적화할 수 있다. EFP-FBXL10 및 NTHL1-mCherry를 안정적으로 발현하는 U-2 OS 세포는 각각 DSB및 산화 병변 부위를 식별하는 데 사용되었다. 405 nm 레이저 라인을 가진 마이크로 조사는 1000 μs 또는 3000 μs 의 거주 시간을 가진 비동기 U-2 OS 세포에서 수행되었다. 스케일 바는 20 μm을 나타낸다.(B)γH2A.X에 대한 면역형광 염색은 mRuby 태그 내생 PCNA를 갖는 인간 망막 색소 상피 세포(hTERT RPE-1)에서 수행되었다. 세포는 1000 μs 또는 3000 μs 가거주 시간으로 미세 조사 후 5분 후에 고정 및 처리되었습니다. 배율 막대는 20 μm을 나타냅니다.

그림 3: 내인성 mRuby-PCNA 및 외인성 mPlum-PCNA를 1000 μs 또는 3000 μs 레이저 거주 시간에 미세 조사 부위에 비교 모집합니다. 내인성 및 외인성 태그PCNA 는 S 단계 동안 복제 foci를 형성한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: S 단계에서 EXO1b의 PARP1/2 의존 모집. EXO1b-AcGFP 및 mPlum-PCNA를 안정적으로 발현하는 U-2 OS 세포는 3000 μs 거주 시간을 사용하여 405nm FRAP 레이저 라인으로 마이크로 조사되었다. (A)차량 제어(DMSO) 또는 olaparib(1 μM)를 통해 사전 처리 후 표시된 시점에서 마이크로 조사 된 세포의 대표적인 이미지. 스케일 바는 채용 분석을 위해 A, B 및 C 영역의 정의된 영역의 대표적인 이미지인 20 μm.(B)를나타낸다. 스케일 바는 20 μm을 나타낸다.(C)DNA 손상 모집 역학은 살아있는 세포 이미징에 의해 포착되었다. 상대적 평균 형광 값과 이미지는 12 분 동안 5 초마다 획득되었습니다. 각 조건에 대해, ≥30세포를 평가하였다. 평균 상대형광값(솔리드 블랙라인)과 표준 오차(그늘진 영역에 의해 시각화된 범위)가 시간에 대해 플롯되었다. 대시 라인은 마이크로 조사 후 1 분에서 채용 값을 보여줍니다. DMSO(n=32)와 올라파리브(n=31) 치료의 차이는 Mann-Whitney 테스트를 사용하여 계산하였다. 아스테릭스는 p<0.0001을 나타냅니다. (D)서양 블롯은 마이크로 조사에 사용되는 세포에서 내인성 EXO1b 및 외인성 EXO1b-AcGFP의 발현 수준을 비교한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Discussion
중요한 단계 및 잠재적 프로토콜 문제 해결/수정
미세 조사를 위한 적당한 조직 배양 용기는 성공을 위해 중요합니다. 대부분의 고해상도 이미징 시스템은 0.17mm 커버 유리 두께에 최적화되어 있습니다. 더 높거나 낮은 두께의 이미징 챔버 또는 플라스틱 폴리머로 만든 이미징 챔버(405nm 이미징에 최적화되지 않음)를 사용하면 이미지 품질을 크게 줄일 수 있습니다. 유리 표면을 사용할 때 세포 접착력을 향상시키기 위해 치료된 조직 배양인지 확인하십시오. 그(것)들이 처리되지 않는 경우에, 이 챔버는 세포를 종자하기 전에 폴리 D-lysine와 예를 들면, 코팅될 필요가 있을 것입니다. 챔버 커버 글래스로 세포를 도금 할 때, 이상적인 세포 밀도는 세포 주기 요철과 세포에 추가 스트레스를 피하기 위해 가장 중요합니다. 실험 전에 현미경 성분의 적절한 열 평형은 안정적인 온도를 유지하기 위해 시간 경과 이미징 전반에 걸쳐 초점을 유지하는 데 매우 중요하며 시간과 샘플에 걸쳐 균일한 DDR을 보장해야합니다.
세포가 미세 조사 전에 건강한 상태에 있어 관절 데이터를 줄이는 것이 중요합니다. 세포가 감염 후/선택 후 불규칙한 형태학이 있는 경우, 형태학이 정상으로 돌아올 때까지 세포가 여러 구절을 통해 진행되도록 합니다. 항상 사용되는 세포 선이 마이코플라즈마 오염이 없는지 확인하십시오. 마이코플라즈마 감염의 많은 부작용 중에서도 숙주 세포에 DNA 손상을 일으키고 DDR 경로14,15에영향을 줄 수 있다. 세포 배양에서 마이코플라즈마를 검출하는 가장 민감한 방법은 PCR(DAPI 또는 Hoechst로 의 검출)을 통해서입니다.
관심있는 수리 단백질의 최적의 과발현은 내인 성 수준과 유사해야하지만 검출을 위해 충분히 높어야합니다. 바이러스 벡터, 감염 시 바이러스 성 티터 및 감염 시간의 길이에 사용되는 프로모터는 모두 이상적인 발현 수준에 맞게 조정할 수 있습니다. 일관된 결과를 위해 개별 세포 클론을 분리하여 균일한 발현 수준과 정상 세포 형태를 보장합니다. 적절한 세포 주기 및 DNA 복구 마커 기능에 대한 내인 성 수준보다 높은 태그PCNA를 과발현하지 않는 벡터 구조를 사용하는 것이 좋습니다. PCNA 과발현의 낮은 수준조차도 S-위상 세포를 차별하기에 충분합니다. 레트로 바이러스 pBABE 벡터는 성공적으로이 목적을 위해 사용 되었습니다 (Addgene #1764, #1765, #1766, #1767). PCNA는 임의의 단황레드(예: mPlum, mCherry, mRuby 등) 또는 단황 녹색 형광 단백질(예: mEGFP, AcGFP, mWasabi, mNeonGreen, mEmerald 등)으로 태그할 수 있으며, 이는 대체 태그 POI와 결합될 수 있습니다. 형광 태그 POI를 과도하게 표현하는 것은 몇 가지 제한사항과 고려 사항이 있습니다. 형광 태그는 정상적인 단백질 기능 및 국소화를 방해 할 수 있습니다. 따라서 태그(N 또는 C-단자)의 위치를 고려해야 합니다. 비 단황 변이체의 올리고머화가 POI의 기능에 영향을 줄 수 있기 때문에 항상 단황 형광 단백질을 사용합니다.
레이저 설정은 광학 경로의 많은 구성 요소가 세포로 전달되는 실제 전력에 영향을 미치기 때문에 각 이미징 시스템에 대해 결정되어야 합니다. 레이저 미세 조사는 흥분 파장에 따라 여러 종류의 DNA 병변을 유발할 수 있으며, FRAP 레이저의 전력 출력 및 사전 감광제(Bromodeoxyuridine 또는 Hoechst)가 사용된 경우. 405 nm 레이저산화 DNA 손상을 일으킬 수 있습니다., 단일 및 이중 좌초 휴식16,17. 더 높은 레이저 출력 설정을 사용하면 DSB의 양이 증가합니다. 이 프로토콜에서 사전 감세화 방법은 활용되지 않았지만 이러한 기술은 문헌에서 크게 다루어지고 아래 토론에서 다시 제한됩니다. 우리의 의견으로는, 원하는 병변이 생성되는 경우에 시험하는 가장 좋은 쪽은 알려진 DNA 손상 통로 특정 유전자의 모집을 위한 시험에 의하여입니다. NTHL1 또는 OGG1의 모집, BER 통로의 성분은 산화 된 DNA 기지10,11,17,18,19,FBXL10 또는 XRCC5의 존재를 나타내는 동안8,20,21의존재를 나타낸다. XRCC1의 모집은 산화된 DNA 염기와 단일 좌초 휴식(SSB)22,23의존재를 나타낼 수 있다. XPC(즉, RAD4)는 자외선(UV)17,24에의해 생성된 부피가 큰 DNA 교반을 제거하는 NER의 좋은 지표이다. 외인성 단백질을 모집하면 특정 불규칙성을 유발할 수 있기 때문에 내인성 DNA 수리 단백질 또는 마커(예: 이중 좌초 휴식의 경우 γH2A.X)의 면역형염색은 특정 DNA 병변의 존재를 확인할 수 있다. 대안적으로, DNA 병변의 특정 모형에 대하여 제기된 항체는 또한 이용될 수 있었습니다. 전달된 레이저 전력을 조정하기 위해, 거주 시간과 레이저 전력을 모두 변경할 수 있습니다.
수학적 모델링의 도움으로 POI의 채용 특성에 대한 귀중한 통찰력을 제공할 수 있는 상세한 운동 분석(예: 여러 DNA 바인딩 도메인의 기여, 다른 신호 이벤트에 대한 민감성 등)을 수행할 수 있습니다. 자동화된 채용 평가 및 셀 트래킹을 결합하여 강력한 워크플로우 1,25를만들 수 있습니다.
DNA 사전 감도의 장점과 한계
미세 조사 전에 DNA의 사전 민감화는 DNA 복구 단백질 모집을 위해 일반적으로 사용되는도구입니다(16,17). 미세 조사 전에 DNA를 민감하게 하는 것은 DSB에 더 취약하게 만듭니다. DNA 사전 감작을 위한 2개의 일반적인 방법은 Bromodeoxyuridine (BrdU) 또는 Hoechst 염료를 가진 세포의 전처리입니다. 높은 레이저 전력에서 미세 조사를 할 수 없는 시스템의 경우, 이러한 방법은 DSB와 같은 DNA 병변을 유도하는 데 필요할 수 있습니다. 그러나, DNA 사전 감감각은 중요한 합병증을 소개할 수 있습니다. BrdU(10μM의 최종 농도에서 사용)는 DNA에 적절하게 통합하기 위해 24시간(또는 사용된 세포주에서 전체 세포 주기와 동등한 시간)에 첨가되어야 하며 세포 주기간섭(26)을유발할 수 있다. Hoechst 33342 (1 μg/mL의 최종 농도에서 사용)는 긴 잠복 기간 후에 세포 독성이지만 염료로 핵을 포화시키는 충분한 시간이 필요합니다. 따라서, 그것은 단지 적용 해야 15-20 마이크로 조사 전에 분; 그렇지 않으면 채용 데이터가 일치하지 않습니다. 이러한 방식으로 염색된 세포는27,28시간이상 문화권에 보관할 수 없다. Hoechst 33342 염료와 같이 세포 투과성이 없는 Hoechst 33358을 사용하지 않도록 하십시오. 사전 감광은 또한 실험 중 불필요한 분산을 소개하고 세포 밀도의 차이에 더욱 민감하게 실험하게 할 수 있습니다 (이것은 통합 된 염료 / 세포의 양에 영향을 미치기 때문에).
공초점 현미경 검사법의 장점과 한계
공초점 현미경 검사법의 화상 진찰 속도는 와이드 필드 현미경 검사법과 비교할 때 제한될 수 있습니다. 그러나 공진 스캐너가 장착된 공초점 현미경은 회전 디스크 현미경 검사법의 속도에 가까워지는 이미징 속도(해상도 비용)를 크게 향상시킬 수 있습니다. 3가지 기능을 통해 A1R HD25 공초점 시스템은 여기에 제시된 프로토콜에 대한 탁월한 선택입니다. 첫째, 시스템의 25mm FOV는 단일 스캔 필드(일반 설정에서 5-10셀 대)에서 15-20셀 사이를 이미지할 수 있게 해 주며, 통계 분석을 위해 충분한 세포를 얻는 데 필요한 수집 횟수를 제한합니다. 둘째, FRAP 모듈과 2개의 스캔헤드를 사용하면 셀을 순차적으로 이미지화하고 미세하게 조사할 수 있습니다. 마지막으로 공진스캐너와 갈바노 스캐너를 모두 사용하는 유연성은 형광의 담금질을 최소화하는 뛰어난 속도로 고시간 해상도 이미징과 느린 스캐닝 속도를 이용하여 더 높은 신호 대 노이즈 비율로 이미지를 생성하는 고공간 해상도 이미징 사이에서 쉽게 전환할 수 있는 기능을 제공합니다. 전술한 유연성을 위해 사용된 시스템은 보다 널리 이용 가능한 공초점 현미경 구성과 유사하게 허용되었지만, 제시된 실험(마이크로 조사 및 후속 이미징 모두)에 갈바노 스캐너만 사용되었습니다.
미세 조사의 장점과 한계
미세 조사는 타의 추종을 불허하는 공간 및 시간적 해상도를 제공하지만 제한없이는 아닙니다. 레이저 미세 조사에 의한 DNA 손상은 자연적으로 발생하는 손상 제에 비해 핵의 특정 부분에 고도로 클러스터된다. 따라서, 미세 조사로 인한 크로마틴 반응은 균질적으로 분산된 손상에 비해 다를 수 있다. 추가적으로, 미세 조사는 시간이 많이 걸리고 단지 수십 개의 세포에서 만 수행 될 수있다, 큰 인구 기반 생화학 적 방법 (크로마틴 분획, 면역 침전, ChIP) 한 번에 세포의 수천을 공부하여 증가 견고성을 제공 할 수 있지만. 전통적인 생화학 기술로 미세 조사로 관찰을 확인하는 것은 신뢰할 수있는 결론을위한 효과적인 전략입니다. 특정 FOV에 있는 많은 세포의 동시 미세 조사가 가능하더라도, 화상 진찰 시스템은 작업을 수행하는 데 더 많은 시간이 필요합니다. 따라서 DNA 병변에 매우 빠르게 채용되는 단백질의 역학을 측정하면 동시에 사용되는 미세 조사에 가능한 ROI의 수가 제한됩니다. 이 프로토콜에 사용되는 이미징 시스템에서 단일 1024 픽셀 긴 ROI의 마이크로 조사는 1000 μs 거주 시간 및 3000 μs 를 사용하여 3088 ms를 사용하여 1032 ms를 소요하여 완료하는 데 걸리는 시간을 제공합니다. 여러 회 선을 사용하는 ROI는 미세 조사를 완료하는 데 필요한 시간을 크게 증가시킬 것입니다 (예를 들어, 7 x 1024 픽셀 긴 ROI는 1000 μs 거주 시간 및 3000 μs 거주 시간을 사용하여 21598 ms를 사용하여 14402 ms를 사용합니다). 이번에는 이미지 수집에서 손실되며 고려해야 합니다. 빠른 모집 이벤트를 이미징할 때 가능한 가장 짧은 ROI를 사용하고 한 번에 하나의 셀만 마이크로 조사합니다.
동기화 방법에 대한 장점 과 제한
세포 주기 특정 연구 결과에 대 한, 기존 방법은 특정 세포 주기 단계로 세포의 동기화 또는 형광 기자를 사용 하 여 세포의 특정 세포 주기 단계를 식별 하는 것을 포함. 그러나 이러한 각 메서드는 고유한 도전과 한계를 제공합니다.
FUCCI 시스템3 (CDT1과 Geminin의 형광 단백질 태그 잘린 형태에 의존)는 세포 주기 연구에 특히 유용한 도구이지만 세포 주기의 S와 G2 단계 사이의 분화에 관해서는 한계가 있습니다. Geminin 수준은 이미 중반 S 단계에서 높고 M 단계까지 높은 상태를 유지하므로 이러한 단계를 분리하기가 어렵습니다. FUCCI 시스템을 사용하면 현미경의 두 개의 광학 채널이 POI 이미징에 사용할 수 없음을 의미합니다.
비암 세포주는 혈청 (혈청 기아)에서 발견되는 성장 인자의 제거에 의해 G0으로 동기화될 수 있어 세포에 DNA 손상을 거의 또는 전혀 유발하지 않을 수 있습니다. 그러나, 대부분의 암 세포 주 부분적으로 그들의 미디어에 혈 청의 적절 한 금액 없이 세포 주기를 통해 진행 을 계속 합니다. 추가적으로, 세포는 부분적으로 늦은 G1, 초기 S 단계에 의해 동기화를 잃기 시작합니다. 혈청 기아 외에도 세포 주기 동기화를 달성하기 위한 수많은 화학 적 방법이 있습니다. 하이드록수에레아, 아피디콜린 및 티미딘 블록은 세포를 초기 S 단계로 동기화하기 위해 DNA 복제를 중지하는 방법입니다. 이러한 방법은 저렴 하 고 간단 하는 동안, 그들은 DNA 손상 결과 복제 스트레스를 소개 합니다. 이러한 DNA 복제 억제제는 H2A의 인산화를 유도하는 것으로 나타났다. X, DSBs2,29의잘 알려진 마커 . 태그-PCNA를 S-phase 세포에 대한 마커로 사용하는 방법은 화학적 동기화로 인한 동맥의 가능성을 감소시키고 혈청 기아에 비해 광범위한 세포주에 적용될 수 있다.
결론
DNA 손상은 돌연변이 병변이 세포의 악성 변환으로 이어질 수있는 유전 질환의 원동력입니다. DNA 합성 기계를 표적으로 하는 것은 암 같이 과증증성 질병의 처리에 있는 근본적인 치료 전략입니다. 이러한 질병을 보다 표적으로 한 방식으로 치료하기 위해서는 DNA 병변을 복구하는 단백질에 대한 더 나은 이해가 필요합니다. 여기에 설명된 프로토콜은 기존의 동기화 방법으로 제시된 과제를 최소화하여 가능한 유물을 줄이고 실험의 재현성을 증가시킴으로써 S 단계의 미세 조사 기반 연구를 지원합니다.
Disclosures
저자는 제시 된 작품의 출판이 니콘 공사에 의해 후원되었다고 말합니다. 저자는 경쟁적인 이해관계가 존재하지 않는다고 선언합니다.
Acknowledgments
저자들은 M. 파가노뿐만 아니라 D. 시모네스키, A. 마르지오, G. 탕의 원고에 대한 비판적 검토에 감사드립니다. B. 미와타니-Minter는 R. 미와타니와 비 민터의 지속적인 지원에 감사를 표했습니다. G. 로나는 K. 로나네 쥬라스와 G. 로나에게 지속적인 지원을 해준 것에 대해 감사를 표했습니다.
Materials
| Name | Company | Catalog Number | Comments |
| Ammonium chloride | Sigma-Aldrich | A9434-500G | For quenching formaldehyde |
| Anti-EXO1 Rabbit Polyclonal Antibody | Proteintech | 16253-1-AP | primary antibody |
| Anti-phospho-Histone H2A.X (Ser139) Antibody, clone JBW301 | Millipore | 05-636 | primary antibody |
| Bovine Serum Albumin | Sigma-Aldrich | 3117332001 | BSA for blocking |
| BrdU (5-Bromo-2'-deoxyuridine) | Merck | 19-160 | pre-sensitizing agent |
| Citifluor™ Mountant Solution AFR3 | Electron Microscopy Sciences | 17973-10 | antifade containing PBS solution for imaging |
| DAPI | Sigma-Aldrich | D9542-1MG | nucleic acid stain |
| DMEM Medium | Thermo Fisher Scientific | 10569010 | Cell culture medium for HEK293T cells |
| DMSO | Sigma-Aldrich | D2650-100ML | Vehichle control and dissolution solvent |
| EGFP-FBXL10 | Addgene | #126542 | viral expression vector for EGFP-FBXL10 |
| EXO1b-AcGFP (in pRetroQ) | custom cloning | na | EXO1b cDNA was cloned in the NheI, BamHI sites of pRetroQ-AcGFP1-N1 vector. |
| Fetal Bovine Serum | Gibco | 16140071 | Media supplement |
| FluoroBrite DMEM | Thermo Fisher Scientific | A1896701 | Phenol red free medium for microscopy |
| Goat anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 488 | Thermo Fisher Scientific | A32723 | secondary antibody |
| HEK293T cells | ATCC | ATCC CRL-3216 | Cell line for viral packaging |
| HEPES | Sigma-Aldrich | H0887-100ML | Buffering agent to supplement live cell imaging medium |
| Hoechst 33342 | Thermo Fisher Scientific | H3570 | pre-sensitizing agent |
| Lipofectamine 3000 | Thermo Fisher Scientific | L3000015 | Transfection reagent |
| McCoy’s 5A (Modified) Medium | Life Technologies | 16600-108 | Cell culture medium for U-2 OS cells |
| mCherry-PCNA | Addgene | #55117 | non-viral PCNA construct suitable for cell cycle marker |
| mPlum-PCNA | Addgene | #55994 | non-viral PCNA construct suitable for cell cycle marker |
| mPlum-PCNA (in pBABE) | custom cloning | na | mPlum-PCNA cDNA was cloned from Addgene #55994 in the BamHI, SalI sites of pBABE (puro) |
| Nikon A1R-HD25 Confocal Scanhead and Controller | Nikon | na | confocal imaging system |
| Nikon LUN4 laser unit | Nikon | na | excitation system |
| Nikon LUN-F 50 mW 405 nm FRAP laser unit | Nikon | na | FRAP laser unit |
| Nikon NIS Elements Confocal Controller Software | Nikon | na | Confocal controlling software |
| Nikon Ti2-E Inverted Microscope | Nikon | na | inverted epifluorescent microscope base |
| Nikon Ti2-LAPP Modular Illumination System | Nikon | na | illumination system |
| NTHL1-mCherry (in pRetroQ) | custom cloning | na | NTHL1 cDNA was cloned in the NheI, SalI sites of pRetroQ-mCherry-N1 vector. |
| Nunc Lab-Tek II Chambered Coverglass (4 well) | Thermo Fisher Scientific | 155382PK | Live cell microscopy cell culture chamber |
| Olaparib | Selleck Chemicals | S1060 | PARP inhibitor |
| Opti-MEM reduced serum media | Thermo Fisher Scientific | 31985062 | Dilution medium for transient transfection |
| Paraformaldehyde aqueous solution (32%) | Thermo Fisher Scientific | 50-980-494 | Fixative |
| pBABE (hygro) | Addgene | #1765 | retroviral expression vector (for low expression levels) |
| pBABE (neo) | Addgene | #1767 | retroviral expression vector (for low expression levels) |
| pBABE (puro) | Addgene | #1764 | retroviral expression vector (for low expression levels) |
| pBABE (zeo) | Addgene | #1766 | retroviral expression vector (for low expression levels) |
| PCNA Antibody (PC10) | Santa Cruz | sc-56 | primary antibody |
| Penicillin-Streptomycin-Glutamine (100x) | Gibco | 10378016 | Media supplement |
| polybrene | Sigma-Aldrich | TR-1003 | Increase viral infection efficiency |
| pRetroQ-AcGFP-C1 | Takara | 632506 | retroviral expression vector |
| pRetroQ-AcGFP-N1 | Takara | 632505 | retroviral expression vector |
| pRetroQ-mCherry-C1 | Takara | 632567 | retroviral expression vector |
| pRetroQ-mCherry-N1 | Takara | 632568 | retroviral expression vector |
| pUMVC | Addgene | #8449 | Viral packaging vector |
| Sodium-pyruvate | Thermo Fisher Scientific | 11360070 | Supplement for live cell imaging medium |
| Triton X-100 aqueous solution (10%) | Sigma-Aldrich | 11332481001 | Dilute in PBS for cell permeabilization buffer |
| Trypsin-EDTA Solution 10X | Sigma-Aldrich | 59418C-100ML | Dilute in PBS to split cells |
| U-2 OS Cells | ATCC | HTB-96 | Optimal cell line for microscopy experiments |
| Universal Mycoplasma Detection Kit | ATCC | 30-1012K | PCR based Mycoplasma detection kit |
| VSV-G | Addgene | #8454 | Viral protein envelope vector |
References
- Aleksandrov, R., et al. Protein dynamics in complex DNA lesions. Molecular Cell. 69 (6), 1046-1061 (2018).
- Darzynkiewicz, Z., Halicka, H. D., Zhao, H., Podhorecka, M. Cell synchronization by inhibitors of DNA replication induces replication stress and DNA damage response: Analysis by flow cytometry. Methods in Molecular Biology. 761, 85-96 (2011).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Herce, H. D., Rajan, M., Lattig-Tunnemann, G., Fillies, M., Cardoso, M. C. A novel cell permeable DNA replication and repair marker. Nucleus. 5 (6), 590-600 (2014).
- Keijzers, G., et al. Human exonuclease 1 (EXO1) regulatory functions in dna replication with putative roles in cancer. International Journal of Molecular Sciences. 20 (1), (2018).
- Cheruiyot, A., et al. Poly(ADP-ribose)-binding promotes Exo1 damage recruitment and suppresses its nuclease activities. DNA Repair (Amsterdam). 35, 106-115 (2015).
- Zhang, F., Shi, J., Chen, S. H., Bian, C., Yu, X. The PIN domain of EXO1 recognizes poly(ADP-ribose) in DNA damage response. Nucleic Acids Research. 43 (22), 10782-10794 (2015).
- Rona, G., et al. PARP1-dependent recruitment of the FBXL10-RNF68-RNF2 ubiquitin ligase to sites of DNA damage controls H2A.Z loading. elife. 7, (2018).
- Young, L. M., et al. TIMELESS forms a complex with PARP1 distinct from its complex with TIPIN and plays a role in the dna damage response. Cell Reports. 13 (3), 451-459 (2015).
- Kong, X., et al. Laser microirradiation to study in vivo cellular responses to simple and complex dna damage. Journal of Visualized Experiments. (131), e56213 (2018).
- Kong, X., et al. Condensin I recruitment to base damage-enriched DNA lesions is modulated by PARP1. PLoS One. 6 (8), 23548 (2011).
- Lan, L., et al. Novel method for site-specific induction of oxidative DNA damage reveals differences in recruitment of repair proteins to heterochromatin and euchromatin. Nucleic Acids Research. 42 (4), 2330-2345 (2014).
- Zerjatke, T., et al. Quantitative cell cycle analysis based on an endogenous all-in-one reporter for cell tracking and classification. Cell Reports. 19 (9), 1953-1966 (2017).
- Ji, Y., Karbaschi, M., Cooke, M. S. Mycoplasma infection of cultured cells induces oxidative stress and attenuates cellular base excision repair activity. Mutation Research. 845, 403054 (2019).
- Sun, G., et al. Mycoplasma pneumoniae infection induces reactive oxygen species and DNA damage in A549 human lung carcinoma cells. Infection and Immunity. 76 (10), 4405-4413 (2008).
- Gassman, N. R., Wilson, S. H. Micro-irradiation tools to visualize base excision repair and single-strand break repair. DNA Repair (Amsterdam). 31, 52-63 (2015).
- Muster, B., Rapp, A., Cardoso, M. C. Systematic analysis of DNA damage induction and DNA repair pathway activation by continuous wave visible light laser micro-irradiation. AIMS Genetics. 4 (1), 47-68 (2017).
- Ikeda, S., et al. Purification and characterization of human NTH1, a homolog of Escherichia coli endonuclease III. Direct identification of Lys-212 as the active nucleophilic residue. Journal of Biological Chemistry. 273 (34), 21585-21593 (1998).
- Rosenquist, T. A., Zharkov, D. O., Grollman, A. P. Cloning and characterization of a mammalian 8-oxoguanine DNA glycosylase. Proceedings of the National Academy of Science U. S. A. 94 (14), 7429-7434 (1997).
- Reid, D. A., et al. Organization and dynamics of the nonhomologous end-joining machinery during DNA double-strand break repair. Proceedings of the National Academy of Science U. S. A. 112 (20), 2575-2584 (2015).
- Taccioli, G. E., et al. Ku80: product of the XRCC5 gene and its role in DNA repair and V(D)J recombination. Science. 265 (5177), 1442-1445 (1994).
- Marsin, S., et al. Role of XRCC1 in the coordination and stimulation of oxidative DNA damage repair initiated by the DNA glycosylase hOGG1. Journal of Biological Chemistry. 278 (45), 44068-44074 (2003).
- Thompson, L. H., Brookman, K. W., Jones, N. J., Allen, S. A., Carrano, A. V. Molecular cloning of the human XRCC1 gene, which corrects defective DNA strand break repair and sister chromatid exchange. Molecular and Cell Biology. 10 (12), 6160-6171 (1990).
- Scharer, O. D. Nucleotide excision repair in eukaryotes. Cold Spring Harbor Perspective Biology. 5 (10), 012609 (2013).
- Oeck, S., et al. High-throughput evaluation of protein migration and localization after laser micro-irradiation. Science Reports. 9 (1), 3148 (2019).
- Mistrik, M., et al. Cells and stripes: A novel quantitative photo-manipulation technique. Science Reports. 6, 19567 (2016).
- Durand, R. E., Olive, P. L. Cytotoxicity, mutagenicity and dna damage by hoechst 33342. Journal of Histochemistry and Cytochemistry. 30 (2), 111-116 (1982).
- Tobey, R. A., Oishi, N., Crissman, H. A. Cell cycle synchronization: reversible induction of G2 synchrony in cultured rodent and human diploid fibroblasts. Proceedings of the National Academy of Science U. S. A. 87 (13), 5104-5108 (1990).
- Podhorecka, M., Skladanowski, A., Bozko, P. H2AX phosphorylation: Its role in DNA damage response and cancer therapy. Journal of Nucleic Acids. 2010, (2010).



