Summary
合成生物学は、非正規のアミノ酸の co-translational の挿入を使用して前例のないプロパティとタンパク質工学を可能です。ここでは、我々 は選択圧定款 (SPI) 経由のエシェリヒア属大腸菌の「ゴールド」蛍光タンパク質 (GdFP) と呼ばれる、蛍光分光特性を持つ型 GFP 蛍光のスペクトル赤シフト変形を生成する方法を提案しました。
Abstract
蛍光タンパク質は、生命科学のための基本的なツール特に生きている細胞の蛍光顕微鏡像。近赤外領域のスペクトル ギャップのまま (avGFP)オワンクラゲから緑色蛍光タンパク質だけでなく、他の種から同族体の野生型とエンジニア リングのバリエーションは既に光学スペクトルの大きい部分をカバーする、どの avGFP ベース fluorophores が付いては、ご利用いただけません。赤いシフト蛍光タンパク質 (FP) の亜種が複数の分子種のスペクトル分離のためのツールキットを大幅に拡大、サンゴ由来の天然赤シフト FPs またはイソギンチャク低い蛍光量子収率と下写真安定 avGFP 亜種に比べて。さらに操作し、発色団の共役系赤外分光地域への可能な拡張も遺伝コードで定める 20 標準的なアミノ酸のレパートリーによって制限されます。これらの制限を克服するために、合成生物学は、さらにスペクトル赤 - を介してシフト発色団トライアドに非正規のアミノ酸の挿入を実現できます。SPI の新規分光特性を備えたエンジニア avGFP バリアントへの応用について述べる。トリプトファン栄養要求性大腸菌タンパク質発現を行うひずみと適切なインドール前駆体で成長媒体を補足。細胞内これらの前駆物質は対応するトリプトファン誘導体に変換、UGG コドンへの応答におけるリボソームの機械によってタンパク質に組み込みます。電子寄付 4 aminotryptophan によって avGFP (生さ) の強化された「シアン」バリアントで Trp 66 の置換結果 108 nm ストークス シフトと強く赤いシフト排出最大 GdFP (574 nm) よりも熱力学的に安定でありながらその前身は生さ。非正規のアミノ酸残基特異定款は、質量分析法による分析されます。GdFP の分光学的性質は、生命科学の遺伝子にエンコードされた FPs の貴重なアプリケーションの一つとして時間分解蛍光分光法によって特徴付けられます。
Introduction
クラゲオワンクラゲ1962年1 (avGFP) で緑色蛍光蛋白質及び 1994年2他の真核生物の細胞内で最初の異種発現の発見以来蛍光タンパク質 GFP 家族となっています。非常に貴重なツールおよび生命科学のターゲット。種特異的なコドン使用の調整、加速度の折りたたみ、改良された成熟、輝度が向上、重合防止及びスペクトルと光化学物性仕立てに広範な遺伝学的および分子工学が含まれて可逆 photoswitch3,4,の5,6能力を含みます。GFP を負ってその 4-(p稿) から蛍光発色団 imidazolidin-5-1 (HBDI)。後者は、アミノ酸 (Ser-65/Tyr-66/Gly-67 で avGFP) のいわゆる発色団トライアドから autocatalytically 分子酸素7の影響の下でペプチッド バックボーン内の追加の共有結合の形成の後に形成します。共鳴安定共役系は、可視範囲およびこれらの蛋白質の特徴的な緑の蛍光性の吸収を可能にする分子環境と動的に対話します。
発色団トライアド内芳香族アミノ酸の存在は必須です。ただし、標準的なアミノ酸のレパートリーには、わずか 4 芳香族残基 (彼、Phe、Trp と Tyr) が装備されています。これは最も赤いシフト自然 FPs Discosoma 症coralimorphs から下流8または9 mKate/mNeptune などを基準にして大幅に以上の赤いシフト avGFP 変種を達成するために従来の突然変異誘発方法を制限します。イソギンチャクEntacmaea quadricolor。したがって、600 上の光学スペクトルの遠と近赤外線の部分 nm まばら GFP の亜種で覆われています。これは、もちろん、同時に数種の fluorophore のスペクトル分離を必要とする蛍光顕微鏡的アプローチの深刻な制限です。たとえば、長波長マーカーも作るに必要です皮膚組織深部組織イメージング10の設定で 700-1,000 nm 間の低吸収の政体の使用します。
AvGFP から派生した蛍光タンパク質は、分光学的性質とその発色団11の化学的性質に基づくいくつかのクラスに分かれています。中性フェノール フォーム間の平衡混合物として存在する野生型発色団のトライアドは Ser-65/Tyr-66/Gly-67 と (λmax = 395 nm、ε = 21,000 M-1cm-1) と陰イオン phenolate フォーム (λmax = 475 nm、ε = 7,100 M-1cm-1)、発光スペクトルが 508 で単一ピークを有し nm。Ser 65 のヒドロキシル グループは重要、それは発色団周辺の Glu 222 に水素結合を寄付 (距離: 3.7 Å)、このカルボン酸のイオン化を促進します。私は EGFP のようにの陰イオン phenolate 発色団によって特徴付けられるクラス (Phe-64-ルー/Ser-65-スレオニン; λmax = 488 nm、ε = 35,600 M-1cm-1、λem = 509 nm)。Ser-65-Thr(Ala,Gly) 代替による中性フェノール フォームの 395 nm 励起ピークが抑制され、470-475 nm のピーク アニオン性 phenolate は 5 〜 six 倍強化し、490 にシフト nm。クラス II には、中立的なフェノールの発色団、サファイア GFP のようにが付いている蛋白質が装備されています。木-203-イル置換がほぼ完全に 399 ピークのみを残して、475 nm 励起を抑制するここでは、nm。陰イオンの発色団溶媒和は正しくすることができないので、中立的な形式が好まれています。クラス III 装備「黄色」蛍光亜種 (EYFP;Ser-65-Gly/Val-68-Leu/Ser-72-Ala/Thr-203-Tyr;Λmax ε = 514 nm、ε = 84、600 M-1cm-1、λem = 527 nm) まで 20 につながる π スタッキング相互作用、芳香族の側鎖の phenolate、Thr-203-His(Trp,Phe,Tyr) の置換によってもたらされると、nm赤いシフト排出マキシマ (木-203-Tyr)。さらに置換 (Gln-69-Lys) の結果が別の 1-2 nm 赤シフト 529 nm、最も赤いシフト avGFP のバリアントが11を知られています。インドール (Tyr-66-Trp) のフェノールの交換作成シアン蛍光生さように、クラス IV (Ser-65-木/Tyr-66-Trp; λmax1 = 434 nm、ε = 24,800 M-1cm-1; λmax2 = 452 nm、ε = 23,600 M-1cm-1;Λem1 = 477 nm、λem2 = 504 nm)。かさばるインドールの宿泊施設はおそらく他の代償的な突然変異によって有効です。生さ励起と放射マキシマはニュートラルまたは陰イオンの発色団が付いている蛋白質のわたってるしきを落ちる。クラス V タンパク質、フェノールの代わりにイミダゾールを抱く (Tyr-66-彼)、 e.g。、ブルー蛍光タンパク質 EBFP のような。クラス VI は最も青シフトの励起・発光ピーク位置に排他的に、ニュートラルな発色団のフォームを好むフェノール-フェニル exchange によって生成される (360 nm と 442 nm、それぞれ)。
クラシック サイト指示された突然変異誘発は、特に 65 67 トリペプチドと 20 標準的なアミノ酸のフレームで相互作用する残基の置換による、小説 avGFP 発色団の変形の生産に適しています。リボソームタンパク質合成12中に芳香族アミノ酸の非標準的なバリアントを導入している場合、これらの可能性をさらに拡張することができます。原則として、これを達成するために 2 つの方法があります。最初の戦略は、アミノアシル tRNA シンテターゼ (Aars) 関連アミノ酸類似への特に、タンパク質翻訳機械の基板耐性に依存します。高効率、栄養要求性でこれを達成するために大腸菌式が採用されている対応する天然のアミノ酸を合成することができます。これにより培養液に、その適切な非正規アミノ酸 (ncAAs) または前駆物質を追加することによって、後者の交換。この戦略は、として知られている選択的な圧力定款 (SPI)13,14, 有効 ncAA のグローバル設立、残基特異の代替品にします。2 番目の方法を使用して、終止コドンによって ncAA の満たされるサプレッサー Trna 設計 aaRS 酵素。これはフレームの終止コドンのリードスルーの結果、サイト固有の ncAA 定款をできます。その結果、停止コドン抑制 (SCS) のこのメソッドは、遺伝コード15の拡大に します。突然変異の誘発による停止コドンは目的のサイトでの標的遺伝子に配置されます。原則として、SPI もメットや Trp など珍しい標準アミノ酸が置換のために選ばれる与えられた遺伝子組換えペプチドおよびタンパク質のユニークな ncAA インストールを軸受を作成する使用できます。4 F、5 - F と 6 F Trp 7 字 Trp、4-オハイオ州-、5-オハイオ州-、Trp と同様、4-、5 NH2など類縁体の大規模な様々 な仕事に、Trp と SPI のアプローチが示されているトリプトファンやも (thienopyrrolyl) β アラニン誘導体16 、17,18,19,20。したがって、SPI はスペクトルとこれらの FPs のストークス シフトをさらに調整する可能性を探索する非標準的なバリアントで GFP の発色団の芳香族アミノ酸を置換するため非常に有利な可能性があります。すべての蛋白質シーケンス変更については FP の折りたたみと発色団の成熟との互換性は実験的テストされなければなりません。
この作品は、クラス IV 生さ21野生型 avGFP Tyr、その発色団トライアド内 Trp 残基ではなく運ぶを駆使します。4-アミノ-トロピカルでこの Trp 66 (とのみ他 Trp 残基生さで Trp-57) SPI を使用すると、置換は発色団内 4-アミノ-Trp の電子寄付のアミノ グループの存在を支持 108 nm ストークス シフトに恵まれている励起状態のところ赤シフト プロトン移動 (ESPT) の共鳴安定化します。この「ゴールド」蛍光蛋白質 (GdFP) を構成する最大の蛍光の最大の赤い転位と変形 (574 nm) すべての avGFP 由来のタンパク質の間で。我々 SPI による GdFP タンパク質生産法および質量分析法による結果変更された蛋白質の必須の分析のためプロトコルを説明します。さらに、GdFP の活用および時間分解蛍光分光法によるアプローチで分析できる方法を紹介します。
Protocol
1. Trp 栄養要求性大腸菌の変換
- 化学的に変換または electrocompetent Trp 栄養要求性大腸菌株、例えば(50 μ L) の細胞します。ATTC 49980 (WP2、エシェリヒア属大腸菌の緊張 B/R22から派生した突然変異体)、熱ショックや穿孔をそれぞれ使用中等-80 L His6 生さプラスミドの 1 ng/μ L 水溶液の 1 μ L で。詳細についてはゼウス科学教育データベース23,24を参照してください。
注: 表現のベクトルを N 末端 6 彼の付けられた生さ21 x ラック オペレーターと細菌 T5 プロモーターによって駆動される中等-80 L His6 生さをエンコードします。さらに、アンプR選択マーカーと複製の colE1 起源を運ぶ (pQE-80 L ベクター バックボーンが見つけることができます: https://www.qiagen.com/mx/resources/resourcedetail?id=c3b71572-4d82-4671-a79b-96357fe926d1&lang=en自己暗示 = true)。(後に発色団成熟25) His6 ECFG 野生型蛋白質の理論分子量は 28303.92 ダ。変換後のターゲット蛋白質シーケンスです (タグ下線、太字でベクトルの派生シーケンス) を次のように: MRGSHHHHHHGSMVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTWGVQCFSRYPDHMK
QHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYISHNVYITADKQKNGIKANFKIRHNIEDGS
VQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYK。 - ブドウ糖 10 g/L、100 μ g/mL アンピシリンを添加した LB 寒天培地プレート (表 1) に変換された細胞をプレートし、37 ° C でプレートを一晩インキュベートします。
2. 組換え蛋白質の表現
-
大腸菌ATCC 49980 pQE-80 L His6 生さの一晩の文化
- LB 培地 5 mL を準備 (表 1; 100 μ g/mL アンピシリン ブドウ糖 10 g/L を添加した) 14 mL の滅菌にポリスチレン文化が好気性の成長のためチューブと予防接種、滅菌ピペット チップまたは接種ループを用いた寒天平板からの単独のコロニー。
注: 新しく変換されたセルからコロニーを使用して勧めします。(ステップ 1.2) から細菌のコロニーでプレートは、数日間 4 ° C で保存できます。 - 一晩で 200-250 rpm で軌道シェーカー 37 ° C 細胞を孵化させなさい。
- LB 培地 5 mL を準備 (表 1; 100 μ g/mL アンピシリン ブドウ糖 10 g/L を添加した) 14 mL の滅菌にポリスチレン文化が好気性の成長のためチューブと予防接種、滅菌ピペット チップまたは接種ループを用いた寒天平板からの単独のコロニー。
-
野生型生さの式
- 10 mL の新鮮な LB 培地に接種 (表 1; 100 μ g/mL アンピシリン ブドウ糖 10 g/L を添加した) 100 mL 三角フラスコに一晩かけて培養の 100 μ L で。37 ° c 200 rpm で軌道シェーカーでフラスコを孵化させなさい。
注: 必要に応じて、この手順 100 μ g/L アンピシリンと 0.5 mM L トリプトファン 10 mL NMM19 培 (表 1) で実行することができます (また、インドールを使用できます)。 - 600 の光学濃度を測定 nm (外径600) 20 分毎を優先的に計測細胞の密度 600 の絶滅を決定することによって常にパスの長さ 1 cm. のキュベットを用いた分光光度計で nm (外径600) 参照を実行対応する培養培地を用いた測定。サンプルおよびサンプルは 0.1 から 0.8 の測定値を取得するそれでは計算外径600希釈ファクターを使用してミックスを希釈します。詳細については、前の文書26を参照してください。
- (接種後約 2-3 時間) の 0.5 から 0.8 の OD600値に達したら、SDS ページ (ナトリウム dodecyl 硫酸塩のポリアクリルアミドゲル電気泳動、手順 4) の"誘導"する前にサンプルを取る。
- 0.5 mm IPTG (イソプロピル β-D-1-thiogalactopyranoside、1 M 原液から) 液体培養を調整することによってターゲット蛋白質の表現を誘発して 30 ° c 4 8 時間 200 rpm で軌道シェーカーでインキュベートします。
注: シアン蛍光蛋白質は通常2737 ° C 以下の温度で表されます。 - SDS ページ (手順 4) の"式"の後のサンプルを取る。
- 5,000 × g と 4 ° C で 10 分間の遠心分離によって細菌細胞を収穫します。
- デカントで上澄みを廃棄し、-20 ° C または-80 ° C で細胞ペレットを凍結ターゲット蛋白質の浄化まで。
- 10 mL の新鮮な LB 培地に接種 (表 1; 100 μ g/mL アンピシリン ブドウ糖 10 g/L を添加した) 100 mL 三角フラスコに一晩かけて培養の 100 μ L で。37 ° c 200 rpm で軌道シェーカーでフラスコを孵化させなさい。
-
GdFP を生産するための SPI
- 100 μ g/mL アンピシリン、15 μ M のトリプトファン、100 mL 三角フラスコに一晩かけて培養の 10 μ L を添加した NMM19 培地 (表 1) の 10 mL を接種して、200 rpm で軌道シェーカーで 30 ° C で一晩培養用フラスコを孵化させなさい。
注:エシェリヒア属大腸菌および SPI の耕作のための化学的に定義されているメディアの様々 な利用可能です。NMM はここで使用、に加えてモップ中28ブドウ糖・無機塩中29, デイビス最小限中30, M9 最小媒体31、または GMML32を使用することができます。 - 値が 0.05 以上 30 分未満でのみ変更するまで、次の日は外径600 30 分毎を測定します。高原の値は、約 1 をする必要があります。
注: 偏差 ± 0.3 単位によっては許容されます。菌株、使用媒体によって初期トリプトファン濃度 (ステップ. 2.3.1) は、調整を必要があります。 - SDS ページ (手順 4) の"誘導"する前にサンプルを取る。
- 5,000 × g と 4 ° C で 10 分間の遠心分離によって細菌細胞を収穫します。デカントで上澄みを廃棄します。
- 100 mL 三角フラスコに 10 mL の 100 μ g/mL アンピシリンと NMM19 培地に細胞を再懸濁します、4-アミノ-インドールを最終濃度 1 mM 50 mM 原液を使用してに追加します。200 rpm で軌道シェーカーで 30 ° C で 30 分間インキュベーションを続行します。
注: この手順はアンピシリンの低の化学安定性のためお勧めします、4-アミノ-インドールの細胞内取り込みを保証します。 - 最終濃度 1 M 在庫を使用して 0.5 mM の IPTG を追加することによってターゲット蛋白質の表現を誘発して一晩で 200 rpm で軌道シェーカー 30 ° C サンプルをインキュベートします。
注: シアン蛍光蛋白質は通常2737 ° C 以下の温度で表されます。 - 次の日は、外径600を測定します。
- SDS ページ (手順 4) の"式"の後のサンプルを取る。
- 5,000 × g と 4 ° C で 10 分間の遠心分離によって細菌細胞を収穫し、上清をデカントによって破棄します。
- 遠心分離のような容器が使用できない場合に、へらを使用して 50 mL のコニカル ポリスチレン チューブに細胞ペレットを転送します。-20 ° C または-80 ° C で細胞ペレットを凍結ターゲット蛋白質の浄化まで。
- 100 μ g/mL アンピシリン、15 μ M のトリプトファン、100 mL 三角フラスコに一晩かけて培養の 10 μ L を添加した NMM19 培地 (表 1) の 10 mL を接種して、200 rpm で軌道シェーカーで 30 ° C で一晩培養用フラスコを孵化させなさい。
3 固定化金属イオンアフィニ ティー ・ クロマトグラフィー (IMAC) を介してターゲット蛋白質の浄化
-
細菌の細胞の換散
- 10-20 分間氷の上細胞ペレットを解凍します。
- 5 mL の冷たい結合バッファー (表 1) を使用して、氷の上 50 mL コニカル ポリスチレン管細胞ペレットを再懸濁します。
- 50 mg/mL リゾチームの 20 μ L、1 Mg/ml の DNase ・ 20 μ L 1 mg/mL RNase a. のチューブを閉じます、5 回の反転で穏やかに混合保管して 30 分間氷の上の 20 μ L を追加します。
注: リゾチーム触媒としてに、部分的なセル中断が発生します。 - 2 スラッシュ アイスで冷却 15 mL のポリスチレン管内 3 分の 3 つのサイクルを用いた超音波ホモジナイザー探針を用いた音声化によって細胞を溶解 4 パルスの一時停止および 45% の振幅の s。
注: 別の方法として、高圧均質化使用できますが、例えば。、14,000 psi に 20 サイクル。必要に応じて、希釈する結合バッファーを使用して最小限の楽器のボリュームに到達します。また、タンパク質抽出試薬はセル中断に使用できます。例の材料表を参照。 - 15,000-18,000 × g、4 ° C で 30 分間遠心するサンプル
- 液量下で注意し新鮮なチューブに上清を転送します。
- ソリューションを 5 mL プラスチック ルアーロック注射器を用いた 0.45 μ m シリンジ フィルターを介してフィルターし、ポリフッ化ビニリデン (PVDF) シリンジ フィルター。
- SDS ページ (手順 4) の「溶解」サンプルを取る。
- DdH2O (旧ライセートと等しいボリューム) 細胞残骸ペレットを再懸濁します。
- SDS ページ (手順 4) のサンプル「ペレット」を取る。
-
IMAC の浄化
- 遮へいまたは製造元の指示に従って IMAC FPLC (タンパク質の高速液体クロマトグラフィー) をパックドカラム自己 1 mL を使用します。カラムの平衡化だけでなく細胞ライセートが列に適用されて後に続く洗浄ステップの結合バッファー (表 1) を使用します。
- 収集し、可視光黄金色によって識別することができます GdFP と溶出画分をプールします。
注: 必要に応じて、ターゲット蛋白質が溶出イミダゾールの線形グラデーションを使用して (0-250 mM) 自動 FPLC システムを使用します。 - 466 で消散係数の文献値を用いたタンパク質濃度を決定する nm (ɛ466 nm = 23,700 M-1 cm-1)33参照として溶出バッファー。手順の詳細については、前の文書26を参照してください。
- SDS ページのサンプル「溶」とクマシー染色の場合レーンあたり蛋白質の 1-10 μ g を使用。
注: SDS サンプル量、染色法や染料の感度によって異なります。 - 透析バッファーまたは分子量カットオフ (MWCO) 5,000-10,000 の膜を使用して MS のバッファーに対して溶出画分の因数を dialyze します。製造元の指示に従って透析膜を準備します。3 回少なくとも 2 時間 100 mL バッファーに対して 1 mL サンプルを dialyze します。この手順の詳細については、前の出版物34を参照してください。
- ストレージ、-80 ° C で透析バッファーの蛋白質のサンプルを凍結します。
注: 因数は、少なくとも 6 ヶ月間安定する必要があります。
4 エシェリヒア属大腸菌の全細胞抽出液の SDS-PAGE のサンプル準備
- 転送細胞懸濁液外径600の 1 mL に相当 = 1 懸濁液 (e.g。 OD600500 μ l = 2) 1.5 mL 遠心チューブに。
- 5000 × g、室温で 10 分間遠心分離によって細胞を収穫します。ピペットで上澄みを廃棄します。
- DdH2O の 80 μ L と 20 を追加ピペッティングにより細胞ペレットとミックスする色素バッファー (表 1) を読み込み 5 x SDS の μ L。
- 水お風呂や熱ブロックで 5 分 95 ° C まで加熱することにより細胞を変化させなさい。その後、部屋の温度に試料を冷却します。
- 以前の文書35に従って Coomassie ステンド SDS ページの 10 μ L を使用します。
注: SDS サンプル量、染色法や染料の感度によって異なります。
5 エレクトロ スプレー イオン化飛行時間質量分析 (LC ESI TOF MS) と結合した高速液体クロマトグラフィー (HPLC) による無傷タンパク質質量分析
注: 高速液体クロマトグラフィーのグラデーション、設定およびバッファー分離カラムと使用楽器によって異なる場合があります。模範的な装置のための材料表を参照。
- (ステップ 3.2.3。) を使用して上記のようにサンプルの蛋白質の集中が MS のバッファーに対して透析を決定する MS のバッファー (参照材料表) 参照として。
- 0.1 mg/ml 80 μ L の最終巻の MS のバッファーを使用して蛋白質のサンプルを希釈、慎重なピペッティングで混ぜる、ガラスを挿入すると MS オートサンプラー バイアルにソリューションを転送、キャップを閉じます。バイアルをフリックすることで、空気の泡を削除します。
- 1 mL の MS のバッファーでガラスを挿入 (バッファー空白) せず第 2 オートサンプラー バイアルを埋めます。
- ウォーム アップに楽器を許可します。測定器の校正を実行します。液体クロマトグラフィー用溶媒の十分な量が利用できることを確認してください (> 100 mL)。
- 5% から 80% のバッファー バッファー B と組み合わせて (0.1% ギ酸 ddH2O) への線形 20 分高速液体クロマトグラフィー グラデーションをプログラム (0.1% ギ酸アセトニ トリル中で)。
- 0.3 mL/min の流量で高速液体クロマトグラフィーを起動し、カラムの圧力が安定するまで待ちます。
- LC ESI-TOF MS 法の 5 μ L のオートサンプラー注入量を設定、サンプルの実行に続いて実行空白のワークリストを作成し、対応するオートサンプラー バイアル位置を割り当てます。ワークリストを実行します。
- ワークリストの完了後、生成されたサンプル データ ファイルを開きます。デコンボリューションの総イオン現在 (TIC) の印刷範囲を選択し、MS スペクトルの最大エントロピー デコンボリューション アルゴリズムを使用してようになりました。
注: 実験条件によって非成熟 FP から発生することができます種の追加またはバッファー イオン付加します。
6. 蛍光寿命測定と GdFP の崩壊によるスペクトル (DAS)
注: 時間分解蛍光分光計測、材料表を参照してください模範的な機器のしてください。吸光度、蛍光励起、蛍光タンパク質の発光スペクトルは、研究室の紫外/可視、蛍光分光光度計を使用しても記録できます。
-
GdFP の波長分解蛍光寿命測定
- PH 7 で PBS バッファー (表 1) に希釈による GdFP の 1 μ M 溶液 2 mL を準備します。1 cm 石英キュベットにソリューションを入力します。
- Ps パルス 470 nm レーザー励起のサンプルと 488 nm ロングパス ・排出フィルターをインストールし、600 L/mm 波長時間相関単一光子計数36 (TWCSPC) 検出器の波長政権獲得のための格子を調整500-700 nm。
- 単一光子計数ソフトウェアと蛍光減衰曲線の買収の最大の約 10 の3カウントが蓄積されるまでは、約 200 × 103光子/秒のカウント レートでの蛍光性の放出を取得します。
-
楽器の応答関数37 (IRF) の測定
- PBS バッファー ph 7 で 1 g/L コロイド状シリカ (~ 220 m2/g) を充填した 1 cm 石英キュベットとサンプル キュベットを交換してください。
注: シリカ懸濁液は、400 グラム/L の水性懸濁液を使用して準備されます。 - 488 nm ロングパス ・排出フィルターと挿入灰色 100 × 103カウント/秒以下に TWCSPC 検出器でカウント レートを調整するフィルターを削除します。
- 16 ch TWCSPC 検出器のチャネル 8 の 470 nm 光子の獲得のための格子を調整します。
- 約 10 x 103カウントが最大の排出に蓄積されるまでは、IRF を取得します。
- 変換蛍光減衰曲線とグローバルな ASCII データ ファイルに IRF38プログラムの継手です。
- 行為グローバル有効期間のリンクのパラメーターとして 3 つの指数要素の合計のモデルによると合います。
- プロット崩壊に関連付けられているスペクトル (DAS) 個々 の腐敗成分の振幅分布として波長の依存性のデータ解析ソフトウェア。
- PBS バッファー ph 7 で 1 g/L コロイド状シリカ (~ 220 m2/g) を充填した 1 cm 石英キュベットとサンプル キュベットを交換してください。
Representative Results
選択圧定款の手法を用いた、生さ (とのみ他 Trp 残基生さで Trp-57) の発色団のトライアドの Trp 66 取替えることができる Trp、4-アミノ - 異なる波長特性を持つ赤シフト GdFP を生成するため。質量分析法は、図 1に示す結果とタンパク質に非正規のアミノ酸の目的の化学量論的統合を示すため使用する必要があります。その後、我々 は顕微鏡、紫外可視吸収分光法としての pH 依存性にフォーカスを持つ GdFP 蛍光体の特性の特性評価に定常状態および時間と波長分解蛍光分光法からデータを提供する、スペクトル。
4 アミノ Trp によって生さで 2 つの Trp 残基の交換を確認するため質量分析を実施します。GdFP の代表的な deconvoluted の MS スペクトルを図 1に示します。野生型生さ発色団成熟後 28,283.9 Da の計算されたタンパク質の質量を持っているが、GdFP の対応する質量は 28,313.9 Da です。GdFP の deconvoluted の MS スペクトルを表わす 28,314.1 ± 0.1 質量主峰 Da 10 ppm 未満で理論値から逸脱します。解析25のこのタイプの典型的な精度の範囲内であること、これは SPI 経由で ncAA の定款を確認 (野生型生さの実験値: 28,283.7 Da)。
共焦点レーザー蛍光顕微鏡 (CFIM) 細菌のセル PBS バッファー内の細菌の巻き上げに生さ、EGFP と EYFP GdFP を表現するイメージを図 2に示します。すべての画像は、サンプルごとに同じエネルギーについてで、UV 目的とレーザーの励起を搭載した顕微鏡に買収されました。
図 3 aは、常に非常によく似た励起エネルギー (波長の図 2のように) を使用して監視、GdFP を含む様々 な FPs を表現するエシェリヒア属大腸菌細菌の CFIM 画像のオーバーレイを示しています。図 3 bは、示されている FP の亜種の発色団の構造を示しています。生さに比べて GdFP の明るさについて (蛍光量子収率 φ = 0.4)、EGFP (φ = 0.6) と EYFP (φ = 0.6) GdFP、蛍光ライトのより広範な取得範囲に注意することが重要だ (30 nm) 20 と対照をなして使用された他の spe 用 nmcies は、近い値に画像の明るさを調整するために。若干低い消散係数と特異な光物性の結果として減らされた量子収率、GdFP の明るさは示す他の fps に比べて低い。
生さ (図 3) の吸収スペクトルに 2 つの特徴的なマキシマは、434 nm、452 nm。対照的に、GdFP は 466 で最大の広い赤いシフト吸収バンド 1 つによって特徴付けられる nm。EGFP の吸収はさらに 488 に赤シフト nm。しかしながら、GdFP の多くの大きいストークス シフト (108 nm) 生さに比べて (41 nm)、EGFP (20 nm)、GdFP の発光スペクトルは、最も赤いシフトすべて 3 GFP 誘導体のここを検討した (図 3 D)。生さの蛍光性の放出は 475 で 2 つの特性のマキシマを示しています間 nm と 505 nm、EGFP が 508 で 1 つ広いメイン発光バンドのピーク 540 で若干肩 (λmax) nm nm。GdFP の蛍光は、約 565 nm (λ以内)(図 3 D)。その発光スペクトルも 475 で小さな肩として表示される野生型生さの小さな貢献が含まれている nm。このほんの生さ説明33として SPI プロシージャ中に誘導する前に合成されています。
図 3E GdFP の吸収スペクトルの pH 依存性変化を示しています。5 8 からの pH 変化、最大排出量を赤に若干シフトし、吸収帯のわずかな拡大が観察されます。ただし、吸収の振幅の減少は 8 の pH と pH 5、GdFP 色素の基底状態の性質が非常に弱く pH によって変更されたことを示すと約 10%。
時間解決単一光子計測法によって監視されている蛍光性の放出は図 4に示します。550 を中心としたスペクトル チャネルの監視減衰曲線 nm と 600 nm (図 4 a) 展示 600 でわずかに高速蛍光減衰 550 で減衰と比較して nm nm。蛍光性のグローバル フィットの結果崩壊曲線 1.0 の時定数を持つ 2 つのスペクトル区別蛍光減衰コンポーネントの 2 つの指数コンポーネント結果 ns と 3.3 ns (図 4 とD)。
GFP 家族の多くの蛍光蛋白質の変形のために典型的である、GdFP の蛍光性の放出は強く、pH に依存します。図 4 bは、5 の pH と pH 8、分光特性を一定にしたまま、低い pH での蛍光強度の減少を明確に示すと GdFP の蛍光性の放出を比較します。
GdFP の崩壊によるスペクトル (DAS)39 (図 4 とD)、2 つの異なる発光バンドによって特徴付けられます。遅い 3.3 貢献 ns 成分は顕著に短波長域で約 550 nm (60%) より高速なコンポーネント (40%) のマイナーな貢献。600 nm、両方のコンポーネントが同じ振幅について。PH からシフトに DAS の ph 6 (図 4)、分光特性 7 (図 4) をほとんど変更し、グローバル フィット ルーチンから時定数も同じ (DAS 時定数の精度は ± 0.15 ns) であります。ただし、完全に同じの pH 変化を図 4 bに減らされた蛍光発光振幅のアカウント 2 つの DAS コンポーネントの絶対振幅の違いを明確に感じ取ることがします。
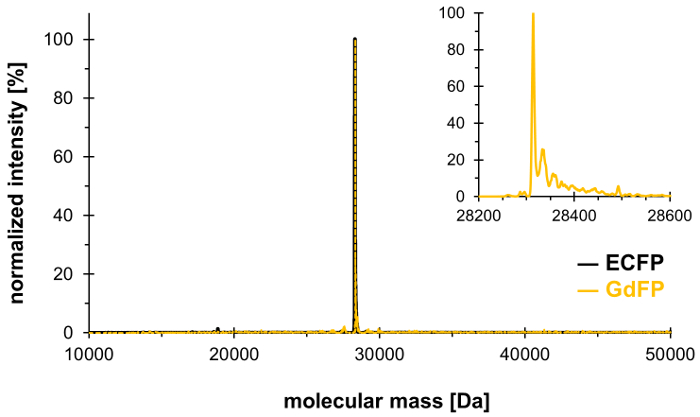
図 1: GdFP の代表的な deconvoluted ESI MS スペクトル。GdFP (ゴールド カラー、拡大のプロットを挿入として示す) の MS スペクトル 28314.1 Da で主なピークを示しています (計算値 28313.9 Da)。野生型生さのスペクトルは、黒で表示されます。この図の拡大版を表示するのにはここをクリックしてください。

図 2: 共焦点レーザー蛍光顕微鏡画像様々 な FPs を表現する細菌の集団から。次の波長設定がイメージ獲得のため使用された: 生さ (λex = 457 nm、検出: 461-480 nm)、EGFP (ex λ = 488 nm、検出: 495 515 nm)、GDFP (λex = 476 nm、検出: 560 590 nm)、EYFP (ex λ = 514 nm、検出: 520-530 nm)。この図の拡大版を表示するのにはここをクリックしてください。

図 3: GdFP のスペクトル特性。(A) CFIM PBS バッファーにおける細菌の再懸濁後生さ、EGFP と GdFP を発現する細菌細胞の混合物のイメージ。(B) (4-アミノ-Trp 残基 66 の代わりに) GdFP、(位置 66 Trp) を持つ親生さ (位置 66 Tyr) と EFGP の発色団の構造。(C) GdFP 生さ、EGFP、正規化された吸収スペクトルの比較に対し (D) 生さの正規化された蛍光発光スペクトル (430 で励起 nm) EGFP と GdFP (両方の蛍光発光スペクトルと比較して450 に興奮した nm)。(E) 吸収スペクトルの pH 依存性 (280 で吸収を正規化 nm)。この図の拡大版を表示するのにはここをクリックしてください。

図 4: GdFP の時間分解蛍光します。(A) 蛍光 GdFP の崩壊監視時間と波長分解による単一光子計数 550 を中心とした分光チャンネルで nm と 600 nm (± 12.5 nm) 470 nm レーザー パルス励起後。インストゥルメンタル応答関数 (IRF) は、使用されるセットアップの時間分解能に関する情報を提供します。(B) GdFP pH に依存しての発光スペクトルの変化 (振動を 460 nm)。(C, D)崩壊関連 (DAS) のスペクトル GdFP pH 7 (C) pH 6 (D) 時間と波長分解蛍光のデコンボリューション崩壊後に決定と 2 つの指数関数のグローバル設定ですべてのチャンネルで崩壊のグローバル フィットリンク時間の定数です。この図の拡大版を表示するのにはここをクリックしてください。

図 5: 生さ (黒) と GdFP (ゴールド) 発色団の分子内電荷移動の構造します。NcAA の一部としてアミノ グループの良い電子ドナーによる発色団システムのサイズの増加により、励起状態の共鳴安定化を達成するためによりアザ構造の形成です。半円として FP 足場へのコネクション ポイントが表示されます。この図の拡大版を表示するのにはここをクリックしてください。
| 原液 | 濃度、溶媒 | メモ | |
| 20% グルコース | 200 g/L D グルコース ddH2O | 0.45 μ m 孔サイズのシリンジ フィルターを通して濾過滅菌します。 | |
| インドール | イソプロパノール 50 mM | ||
| 4-アミノ-インドール | 20% エタノール (100 mL の ddH2O でいっぱいの最終巻でエタノール 20 mL) 50 mM | ||
| IPTG | DdH2O で 1 M | ||
| L トリプトファン | 15 mM は ddH21 M HCl を使用して O に溶解 (粉が dissoved になるまで攪拌下で塩酸を滴下追加) | ||
| リゾチーム | 50 Mg/ml と ddH2O | ||
| DNase 私 | 1 Mg/ml と ddH2O | ||
| RNase A | 1 Mg/ml と ddH2O | ||
| Amp100 | ddH2O で 100 mg/mL アンピシリン | ||
| ドデシル硫酸ナトリウム (SDS) | 200 g/L ddH2O | ||
| アンモニウムの硫酸塩 ((NH4)2SO4) | DdH2O で 1 M | オートクレーブに入れることによって殺菌しなさい | |
| リン酸二水素カリウム (KH2PO4) | DdH2O で 1 M | オートクレーブに入れることによって殺菌しなさい | |
| リン酸水素二カリウム (K2HPO4) | DdH2O で 1 M | オートクレーブに入れることによって殺菌しなさい | |
| 硫酸マグネシウム (MgSO4) | DdH2O で 1 M | オートクレーブに入れることによって殺菌しなさい | |
| D-グルコース | DdH2O で 1 M | 0.45 μ m 孔サイズのシリンジ フィルターを通して濾過滅菌します。 | |
| 塩化ナトリウム (NaCl) | DdH2O 5 M | オートクレーブに入れることによって殺菌しなさい | |
| 塩化カルシウム (CaCl2) | 1 グラム/L | 0.45 μ m 孔サイズのシリンジ フィルターを通して濾過滅菌します。 | |
| 塩化鉄 (ii) (した FeCl2) | 1 グラム/L | 0.45 μ m 孔サイズのシリンジ フィルターを通して濾過滅菌します。 | |
| チアミン | 10 グラム/L | 0.45 μ m 孔サイズのシリンジ フィルターを通して濾過滅菌します。 | |
| ビオチン | 10 グラム/L | 0.45 μ m 孔サイズのシリンジ フィルターを通して濾過滅菌します。 | |
| 微量元素ミックス | 硫酸銅 (CuSO4)、亜鉛塩化物 (ZnCl2), 塩化マンガン (MnCl2), モリブデン酸アンモニウム (NH4)2MoO4);ddH2O の各 1 mg/L | 0.45 μ m 孔サイズのシリンジ フィルターを通して濾過滅菌します。 | |
| 19 アミノ酸ミックス | 1。) 0.5 g L-フェニルアラニン及び粉末を溶解するまで攪拌下 1 M 塩酸 100 ml ddH2O を滴下することで 0.5 g L-チロシンを溶解します。 | ||
| 0.5 g (L-トリプトファン) を除く残りの L-アミノ酸酸のそれぞれのうち 2。) 重量を量る。22 mL fo とミックス 1 M KH2PO4と 1 M K2HPO448 mL。約 800 mL に ddH2O を追加します。ソリューションが明確になるまで攪拌します。 | |||
| 3.) 追加溶存 L-フェニルアラニンとステップ 1 から L チロシン)ddH2o. で 1 L にボリュームを調整し、 | |||
| 4.) 定置滅菌ボトル上部フィルター付け真空濾過によりアミノ酸混合物。 | |||
| バッファーとメディア | 組成/準備 | ||
| SDS 色素バッファーの読み込み、5 倍濃縮 | 0.25 M トリス pH 6.8, 50 %v/v のグリセロール、0.25% の w/v bromphenol ブルー 0.5 M didhiothreitol (DTT; また 5% β-メルカプトエタノール)、硫酸ドデシル ナトリウム 10 %w/v (SDS) | ||
| 結合バッファー | 50 mM りん酸二水素ナトリウム (NaH2PO4)、500 mM の NaCl、10 mM のイミダゾール pH 8 | ||
| 溶出バッファー | 50 mM りん酸二水素ナトリウム (NaH2PO4)、500 mM の NaCl、250 mM のイミダゾール pH 8 | ||
| 透析バッファー | 50 mM りん酸二水素ナトリウム (NaH2PO4)、150 mM の NaCl、100 mL/L グリセリン pH 8 | ||
| MS のバッファー | 10 mM トリス-HCl、pH 8 | ||
| L-トリプトファン (NMM19) を除く 19 L アミノ酸を含む新しい最小媒体 | 次の最終濃度を取得するすべての貯蔵液をミックス: 7.5 mM (NH4)2SO4、1.7 mM, 22 mM KH2PO4、50 ミリメートル K2HPO4、1 mM MgSO4、20 mM D-グルコース、塩化ナトリウム 50 mg/L の19 アミノ酸ミックス、1 μ g/L CaCl2、1 μ g/L した FeCl2、10 μ g/L のチアミン、ビオチンは 10 mg/L、0.01 mg/L の微量元素ミックスします。 | ||
| LB 培地 | 組成: 10 g/L トリプトン 5 g/L 酵母エキス、10 g/L 塩化ナトリウム、pH 7.0 ddH2O | ||
| 準備: | |||
| 50 g トリプトンうち 1.) 重量を量る、25 グラム酵母エキス、1 L のガラス瓶に 5 g 塩化ナトリウム。 | |||
| 2。) 追加 ddH2O 〜 800 mL とかき混ぜて溶解コンポーネント。 | |||
| 3.) pH を測定し、必要に応じて 1 M HCl または 1 M NaOH 滴下することで 7 の pH を調整します。最大 1 l. ddH2O を追加します。 | |||
| 4.) 定置滅菌、オートクレーブに入れることによっては、ボリュームの損失の後でチェックし、滅菌 ddH2O 必要に応じて補正するを追加します。使用するまで 4 ° C で保存します。 | |||
| LB 寒天培地プレート | 組成: 10 g/L トリプトン 5 g/L 酵母エキス、10 グラム/L の NaCl、寒天 15 g/L、ddH2O で pH 7.0 | ||
| 準備: | |||
| 50 g トリプトンうち 1.) 重量を量る、25 g 酵母エキス、塩化ナトリウム、1 L のガラス瓶に 7.5 g 寒天 5 g。 | |||
| 2。) 追加 ddH2O 攪拌下で 500 mL とディゾルブ コンポーネントまで。 | |||
| 3.) pH を測定し、必要に応じて 1 M HCl または 1 M NaOH 滴下することで 7 の pH を調整します。最大 1 l. ddH2O を追加します。 | |||
| 4.) 定置滅菌、オートクレーブに入れることによっては、ボリュームの損失の後でチェックし、必要に応じて滅菌 ddH2O を補うためを追加します。(注: LB 寒天培地は、LB 寒天培地プレートの準備のために使用されるまで 4 ° C で保存ことができます。慎重に電子レンジを使用して固化した寒天を溶かす) | |||
| 5.) ソリューションがまだ暖かい (30-40 ° C)、最終濃度 100 μ G/ml にアンピシリンを追加 | |||
| 6。) 注ぐ手順 5 からの液体の約 15 mL。)滅菌 10 cm シャーレ無菌条件の下で。寒天が固化したプレートは使用するまで 4 ° C で 1 週間保存できます。 | |||
| リン酸緩衝生理食塩水 (PBS) | 組成: 137 mM NaCl、KCl、2.7 mM 10 mM ナ2HPO4、1.8 mM KH2PO4、1 mM CaCl2、0.5 mM MgCl2pH 7。オートクレーブ滅菌、ろ過滅菌します。 | ||
表 1: 在庫ソリューションとバッファー。
Discussion
非常に高い ncAA の取り込み効率を達成するために SPI の栄養要求株ベース メソッドは、ncAA の対応する自然な対応を合成することができない代謝改変させた宿主細胞の使用に依存します。エシェリヒア属大腸菌のような株が容易に利用できます。同じタンパク質にも同時複数 ncAAs 定款は、multiauxotrophic 系統を用いた実現可能です。交換と同様の化学類縁体に制限される化学のレパートリーの残基に固有モードは、欠点として見なすことができます。それにもかかわらず、多数の蛋白質の変形は自然細菌の翻訳装置許容多数のアミノ酸の類縁体としても製作できます。たとえば、50 以上の ncAAs タンパク質の in vitro翻訳、再割り当て40できる遺伝コードのすべてのコドンの約 73% を使用して組み込むことができます。さらに、SPI はターゲット蛋白質の41の効率的なマルチサイト ラベルをできます。原則として、SPI 方法論エシェリヒア属大腸菌、制限されていない、栄養要求性株および定義された耕作メディアが利用他のホスト、正規 20 アミノ酸のそれぞれを働くことができます。たとえば、メチオニン類似を 2 つ、azidohomoalanine (Aha) と homopropargylglycine (Hpg) は商業的に分類する蛋白質および多様な生物のプロテオームの分析のために使用されます。さらに、Aha は細胞内で生産し、その後組み込まれて蛋白質42できます。この ncAA の bioorthogonal 動詞クリックケミストリー Tirrel および協力者によって開発されたなどに特に最適です: たとえば、植物カイコ幼虫43、ショウジョウバエシロイヌナズナの組織細胞44ゼブラフィッシュ稚魚45と同様、ニューロン46を含む哺乳類の細胞、タンパク質に Aha47,48が付けすることができます。同様に、Trp 類縁体は正常に Trp 栄養ラクトコッカス乳酸菌株49抗菌ペプチドに取り込まれました。SPI も Xenobiology50,51生活の基本的な化学メイクアップする選択肢を探るに便利です。たとえば、52 大腸菌と枯草53以前の作品を基に、大腸菌の菌株はによって最近開発された選択的な圧力の代わりに thienopyrrole を利用する進化的戦略インドール、遺伝コード54thienopyrrole アラニン、トリプトファンのプロテオーム的置換の結果します。一般的に、標準的なアミノ酸が 1 つの三重項 (UGG) でエンコードされている、Trp は多数の化学変化を提供するインドール化学の豊富なファセットによるタンパク質工学の有望なターゲットを示します。最近と SPI に基づく定款の代替として、SCS プラットフォーム細菌と真核生物の両方のホストで site-specifically Trp 類縁体を組み込むことができるがされている小説は55を報告しました。これはさらにスペクトル特性の変更を含む ncAA ベースのタンパク質工学生体内でのツールボックスを広げます。
栄養式のホストの使用は、以外にも SPI プロトコルは両方ターゲット式タイミングと ncAA 混入効率およびターゲット蛋白質収量に達するために培地の組成の面で厳格な発酵条件を必要とします。56. 主要な塩のほか窒素 (アンモニウム塩) と炭素 (グルコース)、ビタミン ・微量元素のソースで本質的に含まれている化学的に定義された最小媒体を用いて栽培。さらに auxotrophies、残りのアミノ酸の不在で必須ではありません (20-n, nアミノ酸が置き換えられる場合) 通常57細菌の増殖を促進するために追加されます。ターゲット蛋白質の表現の誘導前に、の初期成長段階中、濃度を制限するnに交換する標準的なアミノ酸が追加されました。細胞の増殖は、対象となる必須アミノ酸が枯渇している、実験として静止した OD600によって示されるまでを処理します。その後、培養培地は、枯渇したアミノ酸が欠けている、豊富な濃度で ncAA を含む新鮮な媒体に置き換えられます。このプロトコルで示すように、トリプトファンの類縁体のリボソーム定款、あるなるトリプトファン合成酵素58対応するトリプトファン誘導体に変換細胞内アナログ インドールは供給されます。次に、ターゲット蛋白の発現が誘導されます。この段階で、細胞は細胞数とフィットネスのバランスとして、対数の成長の終わりの近くにあります。存在と定款、標準的なアミノ酸は、野生型タンパク質の生産につながる、必須アミノ酸の誘導前に枯渇は完全にするために重要です。同様に、質量分析法による一般的 ncAA 混入ターゲット蛋白質の効率性を検討するために必須です。場合に標準的なアミノ酸調整する栽培条件の必要性、例えば、初期成長期に不可欠な amino acid(s) の濃度または後者の期間を変更することにより実質的な存在。NcAA に向かって低 aaRS 活動、場合内因性酵素の過剰発現または ncAA の方よりアクティブな別の aaRS の共発現が実施59をすることができます。
標準的なアミノ酸の Trp は 3 つの顕著な機能に恵まれている: (i) 蛋白質の自然な豊かさが低いです。(ii) その生物物理・化学的性質が一意 (e.g、タンパク質・ ペプチドの本質的な蛍光性の支配的な起源では通常)、および (iii) それは様々 な生化学的相互作用に貢献し、関数を含む。Π スタッキング、水素結合とカチオン-π 相互作用。これらすべての機能は、GdFP を超えて疑いで Trp → 4 アミノ酸トリプトファン置換時に根本的に変更、avGFPs の「ゴールド」のクラスのデザインはオーダーメイドされる蛋白質工学の顕著な例。異なる波長特性を持つ突然変異誘発と ncAA の設立を介して特定のスペクトルの windows に向けて FPs を調整できます。GdFP の場合にこれは、単純な化学交換 H → NH2フレーム内生さ発色団トライアドに含まれるインドール環ので。図 5には、発色団内 ncAA の影響が表示されます。4-アミノ-インドール (細胞内 4 アミノ酸トリプトファンに変換) から発信された電子寄付グループの紹介により様々 なアザの構造安定励起状態を説明することができます。分光学的、その拡大のストークス シフトと赤いシフトの蛍光性の放出結果拡張共役系のこれらの特性から。以前、GdFP 発色団内で強化された分子電荷は ph (図 4 b) 本質的に機密の報告し、S0地盤と S1の双極子モーメントの大きい変化を伴うとして励起状態生さ33に相対します。代替の電子寄付のグループとしてトリプトファン類縁体のヒドロキシ基と置換インドール環を使用モデル蛋白質バルスター41との比較研究で報告されました。
GdFP の吸収及び蛍光スペクトルが生さや EGFP と比較して広がる (図 3 とD)。吸収と蛍光バンドの均質な広がりと、発色団の振動モードと、さらに、さらに振動モード60タンパク質中に存在する色素の結合によって、通常発生します。局所的なタンパク質の環境への結合度は、発色団にローカライズされた料金によってサポートされます。発色団の振動スペクトルと蛋白質の残りの部分との間のような結合が電荷の非局在化とアザの状態のとおりでサポートされる蛋白質の構造の不均一性は、振動スペクトルのローカル変化につながると図 5。このカップリングはまた大きなストークス シフトをサポートし、必ずしも蛍光量子収率が減少します。他の赤いシフトの FPs と比較して GdFP も改良されたタンパク質の安定性と凝集33,,6162低傾向を展示します。他の FP の亜種から色が異なるだけでなく、大幅に高められた熱安定性と強化された協同組合33を折りたたみも展示します。その蛍光強度は少なくとも 90% 生さ蛍光使用量を約 30% 中 60 ° C に加熱保持されます。タンパク質、芳香族アミノ酸は、しばしば一般タンパク質の立体構造の安定化効果がある相互作用の側鎖のネットワークに貢献します。avGFP は、このような側鎖ネットワーク自体も、Phe 165, と彼-148、Tyr 145 発色団から成るを隠し持っています。これらの側鎖はのみ GdFP 構造33にかなり剛性ではありませんが、重要なは、彼らは発色団と疎水性の接触を形成します。GdFP で識別される最も顕著な特徴は、アミノ化色素は Phe 165 により近位です。この相互作用は、他の知られている avGFPs で見られない機能です。2 つの残基は 3.2 から 4.5 Å 離れて、現在のも芳香族アミノ酸相互作用かもしれません。発色団のアミノ化反応による共鳴安定化とともにこれらのほとんどは、この協調的アミノ酸の疎水性ネットワークを安定させます。色素の基底状態と比較すると励起状態でのこれらの相互作用によってより効果的な分子内電荷移動をサポート可能性があり、少なくとも部分的に占めて 108 nm ストークス シフト33,62.
蛍光特性の合理的な設計は、非局在 π システムのサイズの増加を予測して、赤いシフト励起波長が発生します。ニュートラルな発色団に位置 66 つながる中アミノ酸のシリーズによって親指のこのルールに従ったは: Phe (λmax = 355 nm) < 彼 (λma x= 386 nm) < Tyr (λmax = 395 nm) < Trp (λmax = 436 nm)63。自然の中でさまざまな戦略でこの発色団の π 結合の共役系の拡張を実現しています。Discosoma 症から下流のそれは従って λmax 573 nm64へのシフト、その他アミノ酸の統合によって拡張されます。AsFP595 の発色団 (λmax = 595 nm)作用 sulcataから65π システムを拡大、イミノ グループによって拡張されました。GdFP、その他 avFPs の発色団は同じサイズのため、異なる原理は拡大した DsRed や asFP595 の発色団の範囲で波長を伴う必要があります。108 の深遠なストークス シフト nm、蛍光タンパク質の設計の新しい光物性原理を明らかにする GdFP の発色団の明瞭な構造に起因します。( 62で報告された) として予備的な計算は、GdFP の励起状態の発色団の双極子モーメントが生さのそれぞれの値とは異なり、基底状態でより大幅に大きいことを示しています。生さ発色の変化が緩やかではなく GdFP の双極子モーメントは S1〜 15 に 〜 3 D (デバイ) S0の状態から増加、一方 (~ 4 D から 〜 6 D)。したがって、GdFP のユニークな黄金の蛍光は、増加する共鳴安定化を可能にする (図 5参照) 可能なアザ構造の様々 な発色団内で実質的な分子内電荷移動によるものです。これは排出量が発生したエネルギー レベルを減らします。励起双極子モーメントの深遠な変更の結果として分子内電荷分離は発色団環境の静電ポテンシャルの変化の主な理由です。周辺蛋白質のマトリックスを順番に、発色団励起後の電荷分布の変化を調整します。それ以降の構造緩和は、その電荷転送文字のため赤に蛍光スペクトルをシフトする興奮の発色団のエネルギー レベルを下げます。同じ理由で、結果として大きなストークス シフトと無輻射過程、GdFP の蛍光量子収率の向上率が減少生さ33と比較。
高い量子収率と生さ、EGFP の小型のストークス シフトの自由度を減らす発色団の剛体タンパク質環境に起因する通常、その結果、支持する励起状態の発光緩和過程への内部変換66です。 したがって、残りの蛋白質のマトリックスに減らされた結合をもっと厳密に埋め込まれた発色団の分子設計は、遠く赤いシフト GFP 誘導体と高い蛍光量子収率を生成するためのガイドとして役立つかもしれない。したがって、赤いシフトされる蛋白質を作り出す方法をさらに工学の π 電子系と弱結合タンパク質環境へと発色団剛構造の拡大は非常に望ましい。GFP の発色団に直接または発色団周辺で希望の ncAAs の配置によって、そのような変更を導入もできます。
Disclosures
著者は、彼らは競合する金銭的な利益があることを宣言します。
Acknowledgments
この作品は f. j. s. に、ドイツ研究振興協会 (「統一概念で触媒の卓越性のクラスター) タスクフォースオニオンと注意して連邦教育省と科学 (BMBF プログラム「HSP 2020」、TU WIMIplus プロジェクト SynTUBio) によって支えられた
Materials
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| 4-aminoindole | Sigma-Aldrich | 525022 | |
| acetonitrile | VWR | HiPerSolv CHROMANORM ULTRA for LC-MS, 83642 | LC-MS grade required |
| agar-agar | Carl Roth | 5210 | |
| ammonium molybdate ((NH4)2MoO4) | Sigma-Aldrich | 277908 | |
| ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| ampicillin sodium salt | Carl Roth | K029 | |
| biotin | Sigma-Aldrich | B4501 | |
| bromophenol blue | Sigma-Aldrich | B0126 | |
| calcium chloride (CaCl2) | Sigma-Aldrich | C5670 | |
| colloidal silica | Sigma-Aldrich | Ludox HS-40, 420816 | |
| Coomassie Brillant Blue R 250 | Carl Roth | 3862 | |
| copper sulfate (CuSO4) | Carl Roth | CP86.1 | |
| D-glucose | Carl Roth | 6780 | |
| di-sodium hydrogen phosphate (Na2HPO4) | Carl Roth | X987 | |
| di-potassium hydrogen phosphate (K2HPO4) | Carl Roth | P749.1 | |
| 1,4-dithiothreitol (DTT) | Carl Roth | 6908 | |
| DNase I | Sigma-Aldrich | D5025 | |
| ethanol | Carl Roth | 9065.1 | |
| formic acid | VWR | HiPerSolv CHROMANORM for LC-MS, 84865 | LC-MS grade required |
| glycerol | Carl Roth | 3783 | |
| imidazole | Carl Roth | X998 | |
| indole | Sigma-Aldrich | I3408 | |
| iron(II) chloride (FeCl2) | Sigma-Aldrich | 380024 | |
| isopropanol | Carl Roth | AE73.1 | |
| isopropyl β-D-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I6758 | |
| lysozyme | Sigma-Aldrich | L6876 | |
| magnesium chloride (MgCl2) | Carl Roth | KK36.1 | |
| magnesium sulfate (MgSO4) | Carl Roth | 8283.2 | |
| manganese chloride (MnCl2) | Sigma-Aldrich | 63535 | |
| β-mercaptoethanol | Carl Roth | 4227.3 | |
| potassium chloride (KCl) | Carl Roth | 6781.3 | |
| potassium dihydrogen phosphate (KH2PO4) | Sigma-Aldrich | P5655 | |
| RNase A | Carl Roth | 7156 | |
| sodium chloride (NaCl) | Carl Roth | P029 | |
| sodium dihydrogen phosphate (NaH2PO4) | Carl Roth | T879 | |
| sodium dodecyl sulphate (NaC12H25SO4) | Carl Roth | 0183 | |
| thiamine | Sigma-Aldrich | T4625 | |
| Tris(hydroxymethyl)-aminomethane (Tris) | Carl Roth | 5429 | |
| Tris hydrochloride (Tris-HCl) | Sigma-Aldrich | 857645 | |
| tryptone | Carl Roth | 8952 | |
| yeast extract | Carl Roth | 2363 | |
| zinc chloride (ZnCl2) | Sigma-Aldrich | 229997 | |
| Name | Company | Catalog Number | Comments |
| amino acids | |||
| L-alanine | Sigma-Aldrich | A7627 | |
| L-arginine | Sigma-Aldrich | A5006 | |
| L-asparagine | Sigma-Aldrich | A8381 | |
| L-aspartic acid | Sigma-Aldrich | A0884 | |
| L-cysteine | Sigma-Aldrich | C7352 | |
| L-glutamic acid | Sigma-Aldrich | G2128 | |
| L-glutamine | Sigma-Aldrich | G3126 | |
| L-glycine | Sigma-Aldrich | G7126 | |
| L-histidine | Sigma-Aldrich | H8000 | |
| L-isoleucine | Sigma-Aldrich | I2752 | |
| L-leucine | Sigma-Aldrich | L8000 | |
| L-lysine | Sigma-Aldrich | L5501 | |
| L-methionine | Sigma-Aldrich | M9625 | |
| L-proline | Sigma-Aldrich | P0380 | |
| L-phenylalanine | Sigma-Aldrich | P2126 | |
| L-serine | Sigma-Aldrich | S4500 | |
| L-threonine | Sigma-Aldrich | T8625 | |
| L-tryptophan | Sigma-Aldrich | T0254 | |
| L-tyrosine | Sigma-Aldrich | T3754 | |
| L-valine | Sigma-Aldrich | V0500 | |
| Name | Company | Catalog Number | Comments |
| Lab materials | |||
| 0.45 µm syringe filter with PVDF membrane | Carl Roth | CCY1.1 | |
| 1.5 mL microcentrifuge tubes | Eppendorf | 30120086 | |
| conical polystyrene (Falcon) tubes, 50 mL | Fisher Scientific | 14-432-22 | |
| Luer-Lock syringe 5 mL | Carl Roth | EP96.1 | |
| dialysis membrane, Molecular Weight Cut-Off (MWCO) 5,000 | Spectrum Medical Industries | Spectra/Por MWCO 5000 dialysis membrane, 133198 | |
| Immobilized Metal ion Affinity Chromatography (IMAC) column 1 mL, Ni-NTA | Macherey Nagel | Protino series, 745410.5 | |
| petri dishes (polystyrene, sterile) | Carl Roth | TA19 | |
| pQE-80L plasmid vector | Qiagen | no longer available | replaced by N-terminus pQE Vector set Cat No./ID: 32915 |
| protein extraction reagent BugBuster | EMB Millipore | 70921-4 | |
| round-bottom polystyrene tubes, 14 mL | Fisher Scientific | Corning Falcon, 14-959-1B | |
| Trp-auxotrophic E. coli strain | ATCC | ATCC 49980 | Bridges BA et al., Chem Biol Interact., 1972, 5(2):77-84; see main text for alternatives |
| Name | Company | Catalog Number | Comments |
| Mass Spectrometry equipment | |||
| mass spectrometer for LC-ESI-TOF-MS | Agilent | Agilent 6530 Accurate-Mass QTOF | coupled with Infinity LC system |
| mass spectrometry data analysis software | Agilent | MassHunter Qualitative Analysis software v. B.06.00 | |
| High-Performance Liquid Chromatography (HPLC) column for LC-ESI-TOF-MS | Sigma-Aldrich | Supelco Discovery BIO Wide Pore C5 HPLC column, 3 µm particle size, 10 cm x 2.1 mm | |
| HPLC autosampler vials 1.5 mL | Sigma-Aldrich | Supelco 854165 | with conical 0.1 mL glass inserts, screw caps and septa |
| Name | Company | Catalog Number | Comments |
| General equipment | |||
| benchtop centrifuge for 1.5 mL Eppendorf tubes | Eppendorf | 5427 R | |
| cooling centrifuge for 50 mL Falcon tubes | Eppendorf | 5810 R | |
| high pressure microfluidizer for bacterial cell disruption | Microfluidics | LM series with “Z” type chamber | |
| peristaltic pump for LC | GE Healthcare | P-1 | |
| Fast Protein Liquid Chromatography (FPLC) system | GE Healthcare | ÄKTA pure 25 L | |
| orbital shaker for bacterial cultivation | Infors HT | Minitron | |
| UV/Vis spectrophotometer | Biochrom | ULTROSPEC 2100 | |
| ultrasonic homogenizer for bacterial cell disruption | Omnilab | Bandelin SONOPULS HD 3200, 5650182 | with MS72 sonifier tip |
| Name | Company | Catalog Number | Comments |
| Fluorescence spectroscopy equipment | |||
| ps-pulsed laser 470 nm | Picoquant GmbH | PDL-470 | |
| time- and wavelength-correlated single photon counting (TWSPC) acquisition software | Picoquant GmbH | SymPhoTime 64 | |
| time- and wavelength-correlated single photon counting (TWSPC) detector | Picoquant GmbH | PML-16C | 16 spectral channels, to be selected by grating settings |
| single photon counting software | Picoquant GmbH | SPCM 9.75 | |
| global fitting software | Picoquant GmbH | SPC2Glo(R) | |
| fluorescence decay data analysis software | Picoquant GmbH | FluoFit program | |
| data analysis software | OriginLab Inc. | Origin 9.2 | |
| neutral density filter set | Schott | NG1 to NG11 | (400 - 650 nm, transmission 50 %, 20%, 10 %, 5 %) |
| 488 nm long-pass emission filter | AHF Analysentechnik | AHF-488 | |
| quartz cuvette | Thorlabs GmbH | CV10Q1400 | 1 cm pathlength |
References
- Shimomura, O., Johnson, F. H., Saiga, Y. Extraction, Purification and Properties of Aequorin, a Bioluminescent Protein from the Luminous Hydromedusan, Aequorea. J Cell Compar Physl. 59 (3), 223-239 (1962).
- Chalfie, M., Tu, Y., Euskirchen, G., Ward, W. W., Prasher, D. C. Green fluorescent protein as a marker for gene expression. Science. 263 (5148), 802-805 (1994).
- Andresen, M., et al. Structure and mechanism of the reversible photoswitch of a fluorescent protein. P Natl Acad Sci USA. 102 (37), 13070-13074 (2005).
- Andresen, M., et al. Structural basis for reversible photoswitching in Dronpa. P Natl Acad Sci USA. 104 (32), 13005-13009 (2007).
- Brakemann, T., et al. A reversibly photoswitchable GFP-like protein with fluorescence excitation decoupled from switching. Nat Biotechnol. 29 (10), 942-947 (2011).
- Kremers, G. -J., Gilbert, S. G., Cranfill, P. J., Davidson, M. W., Piston, D. W. Fluorescent proteins at a glance. J Cell Sci. 124 (Pt 2), 157-160 (2011).
- Shimomura, O. Structure of the chromophore of aequorea 0. shimomura green fluorescent protein. FEBS Lett. 104 (2), 220-222 (1979).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N. G., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22 (12), 1567-1572 (2004).
- Shcherbo, D., et al. Bright far-red fluorescent protein for whole-body imaging. Nat Methods. 4 (9), 741-746 (2007).
- Shcherbakova, D. M., Subach, O. M., Verkhusha, V. V. Red fluorescent proteins: advanced imaging applications and future design. Angew Chem Int Edit. 51 (43), 10724-10738 (2012).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Curr Protein Pept Sc. 9 (4), 338-369 (2008).
- Wang, L., Xie, J., Deniz, A. A., Schultz, P. G. Unnatural amino acid mutagenesis of green fluorescent protein. J Org Chem. 68 (1), 174-176 (2003).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Sharma, N., Furter, R., Kast, P., Tirrell, D. A. Efficient introduction of aryl bromide functionality into proteins in vivo. FEBS Lett. 467 (1), 37-40 (2000).
- Liu, C. C., Schultz, P. G. Adding new chemistries to the genetic code. Annu Rev Biochem. 79, 413-444 (2010).
- Twine, S. M., Murphy, L., Phillips, R. S., Callis, P., Cash, M. T., Szabo, A. G. The Photophysical Properties of 6-Azaindole. J Phys Chem B. 107 (2), 637-645 (2003).
- Lepthien, S., Hoesl, M. G., Merkel, L., Budisa, N. Azatryptophans endow proteins with intrinsic blue fluorescence. P Natl Acad Sci USA. 105 (42), 16095-16100 (2008).
- Budisa, N., et al. Probing the role of tryptophans in Aequorea victoria green fluorescent proteins with an expanded genetic code. Biol Chem. 385 (2), 191-202 (2004).
- Ross, J. B., et al. Spectral enhancement of proteins: biological incorporation and fluorescence characterization of 5-hydroxytryptophan in bacteriophage lambda cI repressor. P Natl Acad Sci USA. 89 (24), 12023-12027 (1992).
- Soumillion, P., Jespers, L., Vervoort, J., Fastrez, J. Biosynthetic incorporation of 7-azatryptophan into the phage lambda lysozyme: Estimation of tryptophan accessibility, effect on enzymatic activity and protein stability. Protein Eng Des Sel. 8 (5), 451-456 (1995).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Bridges, B. A., Mottershead, R. P., Rothwell, M. A., Green, M. H. L. Repair-deficient bacterial strains suitable for mutagenicity screening: tests with the fungicide captain. Chem Biol Interact. 5 (2), 77-84 (1972).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: The Heat Shock Method. J Vis Exp. , (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: Electroporation. J Vis Exp. , (2017).
- Grigorenko, B. L., Krylov, A. I., Nemukhin, A. V. Molecular modeling clarifies the mechanism of chromophore maturation in the green fluorescent protein. J Am Chem Soc. , (2017).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to the Spectrophotometer. J Vis Exp. , (2017).
- Goedhart, J., et al. Structure-guided evolution of cyan fluorescent proteins towards a quantum yield of 93%. Nat Commun. 3, 751 (2012).
- Neidhardt, F. C., Bloch, P. L., Smith, D. F. Culture medium for enterobacteria. J Bacteriol. 119 (3), 736-747 (1974).
- Hörnsten, E. G. On culturing Escherichia coli on a mineral salts medium during anaerobic conditions. Bioprocess Eng. 12 (3), 157-162 (1995).
- Davis, B. D. The Isolation of Biochemically Deficient Mutants of Bacteria by Means of Penicillin. P Natl Acad Sci USA. 35 (1), 1-10 (1949).
- Sambrook, J., Russell, D. W. Molecular Cloning: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY, USA. (2001).
- Wang, Y. -S., et al. The de novo engineering of pyrrolysyl-tRNA synthetase for genetic incorporation of L-phenylalanine and its derivatives. Mol Biosyst. 7 (3), 714-717 (2011).
- Bae, J. H., et al. Expansion of the genetic code enables design of a novel "gold" class of green fluorescent proteins. J Mol Biol. 328 (5), 1071-1081 (2003).
- JoVE Science Education Database. Dialysis: Diffusion Based Separation. J Vis Exp. , Cambridge, MA. (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Separating Protein with SDS-PAGE. J Vis Exp. , (2017).
- Petrásek, Z., et al. Excitation energy transfer from phycobiliprotein to chlorophyll d in intact cells of Acaryochloris marina studied by time- and wavelength-resolved fluorescence spectroscopy. Photoch Photobio Sci. 4 (12), 1016-1022 (2005).
- Kolber, Z. S., Barkley, M. D. Comparison of approaches to the instrumental response function in fluorescence decay measurements. Anal Biochem. 152 (1), 6-21 (1986).
- Pelet, S., Previte, M. J. R., Laiho, L. H., So, P. T. C. A fast global fitting algorithm for fluorescence lifetime imaging microscopy based on image segmentation. Biophys J. 87 (4), 2807-2817 (2004).
- Loefroth, J. E. Time-resolved emission spectra, decay-associated spectra, and species-associated spectra. J Phys Chem. 90 (6), 1160-1168 (1986).
- Hartman, M. C. T., Josephson, K., Lin, C. -W., Szostak, J. W. An expanded set of amino acid analogs for the ribosomal translation of unnatural peptides. PLoS One. 2 (10), e972 (2007).
- Budisa, N., et al. Global replacement of tryptophan with aminotryptophans generates non-invasive protein-based optical pH sensors. Angew Chem Int Edit. 41 (21), 4066-4069 (2002).
- Ma, Y., Biava, H., Contestabile, R., Budisa, N., di Salvo, M. L. Coupling bioorthogonal chemistries with artificial metabolism: intracellular biosynthesis of azidohomoalanine and its incorporation into recombinant proteins. Molecules. 19 (1), 1004-1022 (2014).
- Teramoto, H., Kojima, K. Incorporation of Methionine Analogues Into Bombyx mori Silk Fibroin for Click Modifications. Macromol Biosci. 15 (5), 719-727 (2015).
- Deal, R. B., Henikoff, J. G., Henikoff, S. Genome-wide kinetics of nucleosome turnover determined by metabolic labeling of histones. Science. 328 (5982), 1161-1164 (2010).
- Hinz, F. I., Dieterich, D. C., Tirrell, D. A., Schuman, E. M. Non-canonical amino acid labeling in vivo to visualize and affinity purify newly synthesized proteins in larval zebrafish. ACS Chem Neurosci. 3 (1), 40-49 (2012).
- Dieterich, D. C., et al. In situ visualization and dynamics of newly synthesized proteins in rat hippocampal neurons. Nat Neurosci. 13 (7), 897-905 (2010).
- Dieterich, D. C., Link, A. J., Graumann, J., Tirrell, D. A., Schuman, E. M. Selective identification of newly synthesized proteins in mammalian cells using bioorthogonal noncanonical amino acid tagging (BONCAT). P Natl Acad Sci USA. 103 (25), 9482-9487 (2006).
- Glenn, W. S., et al. Bioorthogonal Noncanonical Amino Acid Tagging (BONCAT) Enables Time-Resolved Analysis of Protein Synthesis in Native Plant Tissue. Plant Physiol. 173 (3), 1543-1553 (2017).
- Zhou, L., et al. Incorporation of tryptophan analogues into the lantibiotic nisin. Amino Acids. 48 (5), 1309-1318 (2016).
- Acevedo-Rocha, C. G., Budisa, N. Xenomicrobiology: a roadmap for genetic code engineering. Microb Biotechnol. 9 (5), 666-676 (2016).
- Agostini, F., Völler, J. -S., Koksch, B., Acevedo-Rocha, C. G., Kubyshkin, V., Budisa, N. Biocatalysis with Unnatural Amino Acids: Enzymology Meets Xenobiology. Angew Chem Int Edit. 56 (33), 9680-9703 (2017).
- Bacher, J. M., Ellington, A. D. Selection and characterization of Escherichia coli variants capable of growth on an otherwise toxic tryptophan analogue. J Bacteriol. 183 (18), 5414-5425 (2001).
- Wong, J. T. Membership mutation of the genetic code: loss of fitness by tryptophan. Pc Natl Acad Sci USA. 80 (20), 6303-6306 (1983).
- Hoesl, M. G., et al. Chemical Evolution of a Bacterial Proteome. Angew Chem Int Edit. 54 (34), 10030-10034 (2015).
- Italia, J. S., et al. An orthogonalized platform for genetic code expansion in both bacteria and eukaryotes. Nat Chem Biol. 13 (4), 446-450 (2017).
- Völler, J. -S., Thi To, T. M., Biava, H., Koksch, B., Budisa, N. Global substitution of hemeproteins with noncanonical amino acids in Escherichia coli with intact cofactor maturation machinery. Enzyme Microb Tech. 106, 55-59 (2017).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Völler, J. -S., Budisa, N. Coupling genetic code expansion and metabolic engineering for synthetic cells. Curr Opin Biotech. 48, 1-7 (2017).
- Johnson, J. A., Lu, Y. Y., Van Deventer, J. A., Tirrell, D. A. Residue-specific incorporation of non-canonical amino acids into proteins: recent developments and applications. Curr Opin Chem Biol. 14 (6), 774-780 (2010).
- Somsen, O. J., van Grondelle, R., van Amerongen, H. Spectral broadening of interacting pigments: polarized absorption by photosynthetic proteins. Biophys J. 71 (4), 1934-1951 (1996).
- Kurschus, F. C., Pal, P. P., Bäumler, P., Jenne, D. E., Wiltschi, B., Budisa, N. Gold fluorescent annexin A5 as a novel apoptosis detection tool. Cytom Part A. 75 (7), 626-633 (2009).
- Lepthien, S., Wiltschi, B., Bolic, B., Budisa, N. In vivo engineering of proteins with nitrogen-containing tryptophan analogs. Appl Microbiol Biot. 73 (4), 740-754 (2006).
- Wachter, R. M., Elsliger, M. -A., Kallio, K., Hanson, G. T., Remington, S. J. Structural basis of spectral shifts in the yellow-emission variants of green fluorescent protein. Structure. 6 (10), 1267-1277 (1998).
- Verkhusha, V. V., Lukyanov, K. A. The molecular properties and applications of Anthozoa fluorescent proteins and chromoproteins. Nat Biotechnol. 22 (3), 289-296 (2004).
- Martynov, V. I., Savitsky, A. P., Martynova, N. Y., Savitsky, P. A., Lukyanov, K. A., Lukyanov, S. A. Alternative cyclization in GFP-like proteins family. The formation and structure of the chromophore of a purple chromoprotein from Anemonia sulcata. J Biol Chem. 276 (24), 21012-21016 (2001).
- Piatkevich, K. D., Malashkevich, V. N., Morozova, K. S., Nemkovich, N. A., Almo, S. C., Verkhusha, V. V. Extended Stokes shift in fluorescent proteins: chromophore-protein interactions in a near-infrared TagRFP675 variant. Sci Rep. 3 (1), 1847 (2013).



