ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Detta dokument beskriver protokollen för provberedning, datareduktion och dataanalys i neutronspinn eko (NSE) studier av lipidmembran. Omdömesgill deuteriummärkning av lipider ger tillgång till olika membrandynamik på mesoskopiska längd- och tidsskalor, över vilka viktiga biologiska processer uppstår.
Abstract
Lipid bilayers utgör huvudmatrisen av cellmembran och är den primära plattformen för näringsutbyte, proteinmembraninteraktioner och viral spirande, bland andra viktiga cellulära processer. För effektiv biologisk aktivitet bör cellmembranen vara tillräckligt styva för att upprätthålla cellens och dess facks integritet men ändå tillräckligt flytande för att membrankomponenter, såsom proteiner och funktionella domäner, ska kunna spridas och interagera. Denna känsliga balans mellan elastiska och flytande membranegenskaper och deras inverkan på biologisk funktion kräver en bättre förståelse av kollektiv membrandynamik över mesoskopisk längd och tidsskalor av viktiga biologiska processer, t.ex. membrandeformationer och proteinbindningshändelser. Bland de tekniker som effektivt kan undersöka detta dynamiska område är neutronspinneko (NSE) spektroskopi. I kombination med deuteriummärkning kan NSE användas för att direkt komma åt böjnings- och tjockleksfluktuationer samt mesoskopisk dynamik hos utvalda membranfunktioner. Detta dokument ger en kort beskrivning av NSE-tekniken och beskriver procedurerna för att utföra NSE-experiment på liposommembran, inklusive detaljer om provberednings- och deuteringssystem, tillsammans med instruktioner för datainsamling och minskning. I dokumentet införs också dataanalysmetoder som används för att extrahera viktiga membranparametrar, såsom böjningsstyvhetsmodulus, områdeskomprimerbarhetsmodulus och viskositet i plan. För att illustrera den biologiska betydelsen av NSE-studier diskuteras utvalda exempel på membranfenomen som undersökts av NSE, nämligen effekten av tillsatser på membranböjningsstelhet, domänbildningens inverkan på membranfluktuationer och den dynamiska signaturen av membran-proteininteraktioner.
Introduction
Förståelsen för cellmembran och deras funktion har utvecklats anmärkningsvärt under de senaste decennierna. Den tidigare synen på cellmembran som passiva lipid bilayers som definierar cellgränser och husmembranproteiner 1 har gradvis förvandlats till endynamisk modell där lipid bilayers spelar en viktig roll för att reglera vitala biologiska processer, inklusive cellulär signalering, molekylärt utbyte och proteinfunktion - för att nämnanågra 2,3,4,5,6. Denna insikt om att cellmembran är mycket dynamiska, ständigt genomgår ombyggnad och molekylär omfördelning, har uppmanat vetenskapliga utforskningar bortom jämviktsstrukturer imembran 7,8,9. Följaktligen har flera metoder utvecklats för att studera de olika dynamiska lägena i biologiska och bioinspirerade lipidmembran. Hittills har majoriteten av dessa studier främst fokuserat på diffusiva molekylära rörelser10,11,12,13 och makroskopiska formfluktuationer14,15,16, vilket lämnar ett betydande gap i förståelsen av mellanliggande membrandynamik, dvs kollektiva fluktuationer i lipidenheter som består av få 10-100-tal lipidmolekyler. Denna dynamik förekommer över längdskalor på få tiotals till få 100 Å och med tiden skalar av sub-ns till några hundra ns (se figur 1),här kallas mesoskopiska skalor. Det är verkligen på dessa skalor som viktig biologisk aktivitet äger rum på membrannivå17. Detta inkluderar viral spirande18,kanalgating19, och membran-protein interaktioner20. Det är också viktigt att påpeka att energilandskapet hos membranproteiner21,22 visar att konformationsförändringar i proteiner – som är nödvändiga för deras regleringsroll – sker över ns tidsskalor23 av kollektiva membranfluktuationer, vilket ytterligare betonar vikten av mesoskopisk dynamik i cellmembranens biologiska funktion och deras bioinspirerade analoger20. Detta dokument fokuserar på de två primära mesoskopiska dynamiska lägena i lipidmembran, nämligen böjningsfluktuationer och tjockleksfluktuationer.
Den största utmaningen med att direkt sondera dessa fluktuationslägen är svårigheten att samtidigt komma åt deras rumsliga och temporala skalor med hjälp av standardspektroskopimetoder. Den andra utmaningen är att direktkontakttekniker kan påverka samma fluktuationer som de är avsedda att mäta16. Detta förvärras ytterligare av sammansättningen och strukturell komplexiteten hos biologiskamembran 24,25, vilket resulterar i icke-homogena membranfunktioner, inklusive lipiddomänbildning26,27,28,29,30 ochmembranasymmetri31,32,33- kräver selektiva sonder för att förstå dynamiken i olika membranfunktioner. Lyckligtvis kan dessa utmaningar övervinnas med icke-invasiva neutronspektroskopimetoder, såsom neutronspinneko (NSE), som i sig får tillgång till de önskade längd- och tidsskalor, och ytterligare möjliggör studier av selektiva membranfunktioner utan att ändra deras fysikokemiska miljö34. Under de senaste åren har NSE-spektroskopi utvecklats till en unik och kraftfull sond av kollektiv membrandynamik35. Resultat från NSE-studier på lipidmembran har gett nya insikter imekaniska 36,37 och viskoelastiska38,39 egenskaper hos lipidmembran och har kastat nytt ljus över deras potentiella roll i biologisk funktion40,41.
NSE-spektroskopitekniken är baserad på en interferometrisk instrumentdesign, först föreslagen av Mezei42, med hjälp av en serie spin-flippers och magnetiska spolar för att kontrollera precessionen av neutronspinn när neutroner korsar instrumentet. Konstruktionen vilar på magnetisk spegling av magnetfältselementen med avseende på provpositionen (figur 1A). Detta innebär att neutronen i avsaknad av energiutbyte mellan neutronen och provet utför samma antal spinnföreskrifter, i motsatt riktning, under instrumentets första och andra halva (lägg märke till π-flippern mellan de två precessionsspolarna). Som ett resultat förblir neutronens slutliga spinntillstånd oförändrat i förhållande till det ursprungliga tillståndet - ett fenomen som kallas spinn-eko (se transparent neutron i figur 1A). Men när neutronen energiskt interagerar med provet ändrar energiutbytet antalet spinnföreskrifter under instrumentets andra halva, vilket leder till ett annat slutligt spinntillstånd (se figur 1A). Detta upptäcks experimentellt som en förlust i polarisering, vilket kommer att visas senare i detta dokument. För mer information om NSE-tekniken hänvisas läsaren till dedikerade tekniska papper42,43,44,45.
Här presenteras en förenklad beskrivning för att ge en grov uppskattning av längd- och tidsskalor som är tillgängliga med NSE. Längdskalorna bestäms av intervallet av uppnåeliga vågvektoröverföringar, Q = 4π sin θ/λ, där 2θ är spridningsvinkeln och λ är neutronvåglängden. Man kan se att Q ställs in av våglängdsområdet och rotationsriktningen på spektrometerns andra arm (se figur 1A). Ett typiskt Q-intervallpå NSE spektrometrar är ~0,02-2 Å-146,47, och upp till 0,01-4 Å-1 med de senasteuppgraderingarna 48,49, motsvarande rumsliga skalor på ~1-600 Å. Å andra sidan beräknas den tillgängliga tidsskalan från den totala precessionsvinkeln (eller fasen) som neutronen förvärvat inom de magnetiska precessionsspolarna och befinns vara50:  . I detta uttryck är t fouriertiden definierad som , var
. I detta uttryck är t fouriertiden definierad som , var  är
är  neutron gyromagnetic förhållandet,
neutron gyromagnetic förhållandet,  är spolelängden och
är spolelängden och  är styrkan i spolens magnetfält. Det är värt att påpeka att Fourier-tiden är en mängd som är strikt beroende av instrumentgeometrin, magnetfältstyrkan och neutronvåglängden. Till exempel, med neutroner av våglängd
är styrkan i spolens magnetfält. Det är värt att påpeka att Fourier-tiden är en mängd som är strikt beroende av instrumentgeometrin, magnetfältstyrkan och neutronvåglängden. Till exempel, med neutroner av våglängd  = 8 Å och instrumentinställningar
= 8 Å och instrumentinställningar  på = 1,2 m
på = 1,2 m  och = 0,4 T, beräknas Fourier-tiden vara t ~ 50 ns. Experimentellt justeras Fourier-tiden genom att ändra strömmen i precessionsspolarna (dvs. magnetisk fältstyrka) eller använda olika neutronvåglängder, vilket resulterar i typiska NSE-tidsskalor på ~ 1 ps till 100 ns. De senaste uppgraderingarna i NSE-spektrometrar har dock gjort det möjligt att få tillgång till längre Fourier-tider, upp till ~400 ns på J-NSE-Phoenix spektrometer vid Heinz Maier-Leibnitz Zentrum51 och SNS-NSE spektrometer vid Oak Ridge National Lab48, och upp till ~1 000 ns vid IN15 NSE spektrometer vid Institut Laue-Langevin (ILL)49.
och = 0,4 T, beräknas Fourier-tiden vara t ~ 50 ns. Experimentellt justeras Fourier-tiden genom att ändra strömmen i precessionsspolarna (dvs. magnetisk fältstyrka) eller använda olika neutronvåglängder, vilket resulterar i typiska NSE-tidsskalor på ~ 1 ps till 100 ns. De senaste uppgraderingarna i NSE-spektrometrar har dock gjort det möjligt att få tillgång till längre Fourier-tider, upp till ~400 ns på J-NSE-Phoenix spektrometer vid Heinz Maier-Leibnitz Zentrum51 och SNS-NSE spektrometer vid Oak Ridge National Lab48, och upp till ~1 000 ns vid IN15 NSE spektrometer vid Institut Laue-Langevin (ILL)49.
Förutom direkt tillgång till membrandynamikens längd och tidsskala har NSE de inneboende förmågorna hos neutronisotopkänslighet52. Specifikt resulterar neutronernas förmåga att interagera annorlunda med vätets isotoper, det vanligaste elementet i biologiska system, i en annan neutronspridningslängdtäthet,34 eller NSLD (motsvarigheten till det optiska indexet för refraktion50), när protium ersätts med deuterium. Detta möjliggör en metod som kallas kontrast variation, som ofta används för att markera specifika membran funktioner eller dölja andra – det senare scenariot kallas kontrast matchning. En frekvent tillämpning av kontrastvariation/matchning är ersättning av vatten (NSLD = -0,56 × 10-6 Å-2)med tungt vatten eller D2O (NSLD = 6.4 × 10-6 Å-2) för att förstärka neutronsignalen från protierade lipidmembran (NSLD ~ 0 × 10-6 Å-2). Detta tillvägagångssätt är mycket effektivt i studier av membranstruktur eftersom penetrationen av D2O i membranets huvudgruppsregion möjliggör noggrann bestämning av membrantjocklekarna (se figur 2A, vänster panel) och placeringen av olika lipidundergrupper när mer sofistikerade modeller appliceras53,54. Detta dokument belyser några exempel på användning av kontrastvariation för studier av kollektiv dynamik i biomimetiska membran och utvalda membranfunktioner.
Här illustreras NSE:s effektivitet när det gäller att ge unika insikter om dynamiska och funktionella membranegenskaper genom konkreta exempel på NSE-studier på modell- och biologiskt relevanta lipidmembransystem med tonvikt på mesoskaladynamik i fristående membran, i form av liposomala suspensioner. För NSE-mätningar av membrandynamiken ombord hänvisas läsaren till dedikerade publikationer om neutronspin-ekospektroskopi (GINSES)55,56 och andra studier av justerade multilamellära membranstaplar57,58,59,60.
För enkelhetens skull belyser detta dokument tre olika system för membrandeuteration illustrerad på en välstuderad domänbildande, eller fasavskiljande, lipid bilayer-system på 1,2-dimyristoyl-sn-glycero-3-fosfokolin (DMPC) och 1,2-distearoyl-sn-glycero-3-phosphocholine (DSPC)blandningar 61,62. De två lipiderna kännetecknas av en missmatchning i kolvätekedjans längd (14 kol/svans i DMPC vs 18 kol/svans i DSPC) och deras övergångstemperatur för gelvätska (Tm, DMPC = 23 °C vs Tm, DSPC = 55 °C). Detta resulterar i lateral fasseparation i DMPC: DSPC-membran vid temperaturer mellan blandningens övre och nedreövergångstemperaturer 63. De utsutringningssystem som beaktas här väljs för att demonstrera de olika dynamiska lägen som är tillgängliga i NSE-mätningar på liposomala membran, nämligen böjningsfluktuationer, tjockleksfluktuationer och selektiva böjnings-/tjockleksfluktuationer i laterala domäner. Alla lipidkompositioner rapporteras för DMPC: DSPC bilayers beredda vid en molfraktion på 70:30, med hjälp av kommersiellt tillgängliga protiated och perdeuterated varianter av DMPC och DSPC. Alla provberedningssteg är baserade på 4 ml liposomal suspension i D2O, med en lipidkoncentration på 50 mg/ml, för en total lipidmassa på Mtot = 200 mg per prov.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Deuterationssystem som krävs för försöket
- För böjningsfluktuationsmätningar, gör fullt protierade liposomer i D2O (D 99,9%) eller D2O-buffert (t.ex. fosfatbuffert beredd med D2O istället för H2O). Använd helt protiated DMPC (C36H72NO8P) och DSPC (C44H88NO8P) med
 133,4 mg, Där XDMPC och XDSPC är molfraktionerna DMPC och DSPC, här inställda på 0,7 respektive 0,3, och MwDMPC respektive MwDSPC är molvikterna som ges av 677,9 g/mol respektive 790,1 g/mol. På samma sätt mDSPC = 66,6 mg. Detta utlösningsschema ökar spridningskontrasten mellan membranet (NSLD ~ 0 × 10-6 Å-2) och den avutererade bufferten (NSLD ~ 6,4 × 10-6 Å-2) och förstärker signalen från membranutsudlingar (se figur 2A vänster panel).
133,4 mg, Där XDMPC och XDSPC är molfraktionerna DMPC och DSPC, här inställda på 0,7 respektive 0,3, och MwDMPC respektive MwDSPC är molvikterna som ges av 677,9 g/mol respektive 790,1 g/mol. På samma sätt mDSPC = 66,6 mg. Detta utlösningsschema ökar spridningskontrasten mellan membranet (NSLD ~ 0 × 10-6 Å-2) och den avutererade bufferten (NSLD ~ 6,4 × 10-6 Å-2) och förstärker signalen från membranutsudlingar (se figur 2A vänster panel). - För att mäta böjningsdynamiken hos utvalda laterala membranfunktioner, t.ex. matrisdynamik i fasavskiljande DMPC:DSPC-membran, använd protiated DMPC (C36H72NO8P) och avuterated, DSPC-d83 (C44H5NO8PD83,Mw 873,7 g/mol), så att mDMPC = 128,8 mg och mDSPC-d83 = 71,2 mg. Det här utmatningssystemet minimerar spridningen från de oönskade DSPC-rika domänerna, vilket möjliggör selektiva mätningar av böjningsfluktuationer från dmpc-omfattande matrisen (se figur 2B i mitten).
OBS: För att hitta den optimala lipiddeuteration som krävs för ett specifikt kontrastmatchningsschema använder du tillgängliga webbaserade spridda längdtäthetskalkylatorer (SLD), till exempel den som utvecklats av NIST Center for Neutron Research64. Dessa webbaserade gränssnitt är utrustade med användarvänliga verktyg för enkel beräkning av lipids SLD med olika grader av utmatning, liksom lipidblandningar. - För NSE-mätningar av genomsnittliga membrantjockleksfluktuationer (utan sidokontrast) ska du använda svansdeuterated varianter av de ingående lipiderna, dvs. DMPC-d54 (C36H18NO8PD54,732,3 g/mol) och DSPC-d70 (C44H18NO8PD70, 860,1 g/mol)35,38, så att mDMPC-d54 = 133,0 mg och mDSPC-d70 = 67,0 mg. Detta kontrastschema (Figur 2A, höger panel) förstärker spridningssignalen från lipidhuvudgrupperna (NSLD ~ 4,5 × 10-6 Å-2) genom att kontrastmatcha svansgruppen (NSLD ~ 6,4 × 10-6 Å-2) till den avutererade bufferten som möjliggör detektering av fluktuationer i membrantjockleken.
- För tjockleksfluktuationsstudier av utvalda membranfack, T.ex. DMPC-rik matris, använd samma strategi som beskrivs i steg 1.2 genom att ersätta protierade DMPC-lipider med deras svansdeuterated analoger, dvs DMPC-d54, så att de DSPC-rika domänerna kontrastmatchas med den avuterated bufferten och den primära spridningssignalen kommer från huvudgruppsregionen i den svansdeuterade DMPC-rika matrisen.
2. Beredning av lipidupphängning för extrudering
- Beräkna massan för varje beståndsdel i provet, beroende på provsammansättning. Som en tumregel, för prover med flera molekylära komponenter, ges massan av en komponent av dess molmassa, Mw i , viktadavdess molfraktion, Xi, och normaliserad över alla komponenter så att: där M
 tot är den totalamassan, inställd här till 200 mg. Se exemplet ovan för DMPC-DSPC lipid bilayers med olika utsuttningssystem.
tot är den totalamassan, inställd här till 200 mg. Se exemplet ovan för DMPC-DSPC lipid bilayers med olika utsuttningssystem. - Väg de beräknade massorna av lipider (och andra provbeståndsdelar, t.ex. proteiner, nanopartiklar osv.) med hjälp av en digital halvmikrobalans och tillsätt dem i en flaska eller kolv med rund botten – kom ihåg att väga injektionsflaskan eller kolven i förväg. Tillsätt 1 ml lösningsmedel för att lösa upp de vägda komponenterna genom att blanda manuellt inuti en huva. För rena lipidprover, använd kloroform eller etanol. För prover med ytterligare icke-lipidkomponenter (t.ex. nanopartiklar) väljer du ett gemensamt lösningsmedel som sprider alla komponenter.
- För små lipidmängder (<10 mg), förbered en stamlösning och pipett den önskade volymen i blandningen.
OBS: Tillsätt inte alltför stora mängder lösningsmedel eftersom det avsevärt saktar ner det torksteg för lösningsmedel som beskrivs nedan.
- För små lipidmängder (<10 mg), förbered en stamlösning och pipett den önskade volymen i blandningen.
- Torka lipidlösningen inuti en huva genom att försiktigt strömma en inert gas (t.ex. kväve, argon) i injektionsflaskan samtidigt som injektionsflaskan långsamt roteras i en vinkel. Håll injektionsflaskan i lutat läge för att skapa en tunn film av torkad lipid på injektionsflaskans väggar, vilket möjliggör jämn torkning. Placera flaskan i ett vattenbad vid 35 °C för att kringgå avdunstningsmedierad kylning, vilket kommer att bromsa avdunstningen av lösningsmedel.
- Placera injektionsflaskan över natten i en vakuumugn vid ~35 °C för att helt avlägsna restlösningsmedlet. För omättade lipider, rensa vakuum med en inert gas för att minimera oxidation.
- För att säkerställa fullständig borttagning av lösningsmedel, väg injektionsflaskan efter lipidtorkning och bekräfta att det inte finns någon överskottsmassa utöver de uppmätta mängderna material. Gör detta genom att subtrahera injektionsflaskan från den uppmätta massan efter torkning. Om det finns överskottsmassa, torka provet under vakuum i ytterligare 6 timmar. Upprepa den här processen efter behov.
- Hydrera lipidfilmen med 4 ml D2O eller D2O-buffert för att erhålla en lipidkoncentration på 50 mg/ml. För lipider med höga övergångstemperaturer, såsom DMPC-DSPC-blandningar, värm bufferten till över övergångstemperaturen (60 °C) för att säkerställa jämn blandning.
OBS: Eftersom NSE-experiment kräver relativt stora provvolymer (~4 ml), överväg att hydrera provet med hälften av den nödvändiga bufferten, dvs. I det här fallet lägger du till den återstående halvan av buffertpostens extrudering. Observera att kapaciteten hos sprutor som används vid extrudering är begränsad till 1 ml. Således skulle återfuktning med 4 ml buffert kräva fyra uppsättningar extrudering. - Vortex-blanda den hydratiserade lipidlösningen tills lipidfilmen är helt upplöst och inte längre syns på injektionsflaskans väggar. I detta skede bildar de hydratiserade lipiderna multilamellar vesiklar och mikronstora multilamellarstaplar och suspensionen verkar mjölkvit.
- För att underlätta brytningen av lipidstaplarna och för att minska multilamelliteten, utför fem frys-/töväderscykler genom att placera injektionsflaskan med hydratiserad lipidlösning i en frys av labbkvalitet (helst -80 °C frys) tills den är helt frusen och sedan överföra injektionsflaskan till ett 35 °C vattenbad tills lipidlösningen är helt tinad. Virvel den tinade lösningen tills den är homogen. Upprepa fyra gånger till.
OBS: Alternativt kan ett torrt isbad förberedas för snabb frysning genom att kombinera aceton och torr is.
3. Extrudering av den hydratiserade lipidlösningen
- Montera extruderinställningen med hjälp av ett polykarbonatmembran mellan två membranstöd och tillsätt två pappersfilter på varje sida för att ge ytterligare stöd. Använd ett polykarbonatmembran med en porstorlek som matchar mål liposomstorleken (vanliga porstorlekar för NSE-experiment är 50 nm och 100 nm – vanligtvis möjliggör liposomer med 100 nm diameter mindre begränsade membranfluktuationer, men mindre 50 nm liposomer kan användas för krökningsstudier). Se till att polykarbonatmembranet är helt sträckt innan du slutför monteringen och drar åt det yttre extruderhöljet.
- Hydrera polykarbonatmembranet genom att passera ~0,3 ml D2O eller D2O-buffert några gånger genom membranenheten med lufttäta glassprutor. Använd samma buffert som används vid provberedning. Låt den vara i minst 10 minuter och sug sedan ut bufferten helt innan du introducerar provet.
- Fyll en 1 ml gastät spruta med den beredda lipidlösningen och sätt in i ena änden av extruderapparaten. Sätt sedan in en tom spruta i motsatt ände. När sprutorna är anslutna till extruderenheten placerar du den i extruderblocket.
- Om förhöjda temperaturer behövs för extrudering, som vid mättade lipider med höga övergångstemperaturer (t.ex. DSPC, Tm = 55 °C), förvärm extruderuppvärmningsblocket över lipidövergångstemperaturen (t.ex. 60 °C), genom att placera värmeblocket på en värmeplatta eller genom att använda ett cirkulationsbad enligt figur 3A.
OBS: Detta steg är avgörande för att säkerställa homogen blandning av lipider och för att undvika att utöva extremt tryck under extrudering, vilket kan bryta polykarbonatmembranet. För lipidprover med låga övergångstemperaturer (<25 °C) utför du extruderingen vid rumstemperatur. - För att extrudera lipidlösningen, fäst extruderuppsättningen på en programmerbar sprutpump med en aluminium/stålram enligt figur 3A. För temperaturkontrollerade profiler, tillsätt en specialbyggd extruderbas med en vätskekanal och fäst på ett cirkulerande vattenbad.
- Programmera sprutpumpen att utföra 15-20 extruderingscykler enligt tillverkarens handbok. När extruderad, färgen på lipidlösningen ändras från mjölkvita till transparent opal blå (Figur 3B, C), vilket indikerar en slutlig liposomal storlek som är mindre än våglängden av synligt ljus, som förväntat. För den typ av sprutpump som visas i figur 3A, följ stegen nedan.
- Börja med att justera pumpinställningarna. Håll ned hastighetsknappen och ange extruderingshastigheten (50,99 ml/h) och tryck sedan på diameterknappen och ange sprutdiametern (4,606 mm). Använd uppilerna under varje siffra på skärmen för att ändra det siffran.
- Placera extruderuppsättningen med provsprutan åt höger (se figur 3A). Tryck på knappen Dra tillbaka tills utryckningslampan tänds. Tryck på Start och vänta tills provet doseras i den vänstra (tomma) sprutan.
- Tryck på stoppknappen precis innan provsprutan (höger) är helt tom. Registrera den dispenserade volymen och använd den för att programmera extruderingscykeln. Håll ned hastighetsknappen tills fas 1 (PH:01) visas på skärmen. Tryck på volymknappen för att ange den dispenserade volymen som spelats in tidigare. I denna fas, se till att Withdraw-lampan är avstängd – detta dispenserar provet i rätt riktning.
- Tryck på hastighetsknappen igen och använd den högra uppåtpilen för att komma åt fas 2 (PH:02). Tryck på Volym för att ange samma värde för den dispenserade volymen som registrerats tidigare. I den här fasen trycker du på knappen Dra tillbaka tills uttagslampan är tänd – detta delar ut provet till vänster.
- Upprepa den här cykeln genom att trycka på hastighetsknappen igen och använda pilen längst upp till höger för att komma åt fas 3 (PH:03). Tryck på volymknappen tills LP:SE visas på skärmen och ställ in den på 20. Detta är antalet slingor eller upprepningar som pumpen kommer att utföra. Tryck slutligen på hastighetsknappen, åtkomstfas 4 (PH:04) och tryck på volymknappen för att komma till stoppfunktionen. Pumpen är nu inställd för automatiserad extrudering.
- Tryck på Start för att starta extruderingscykeln.
- Töm sprutan som innehåller den extruderade lipidupphängningen i en ren injektionsflaska och förbered för förvaring eller mätningar. För lipidprover med hög smälttemperatur, förvara provet ovanför vätskefasövergången tills det mäts. Annars, håll prover i rumstemperatur.
- Frys inte extruderade prover eftersom frysning kommer att orsaka att blåsorna spricker (suspensionen blir mjölkvit igen).
4. NSE-mätningar för proverna och minskning av insamlade data
- Före NSE-experimentet karakterisera det extruderade liposomprovet från steg 3.7 med hjälp av tillgängliga metoder för att säkerställa tillräcklig provkvalitet. En lista över möjliga charkateriseringsmetoder som kan användas för att bedöma kvaliteten på liposomala suspensioner för NSE-experiment, t.ex. storleksfördelning, multilamellaritet, lateral membranstruktur, ingår i diskussionsavsnittet.
- Bestäm Q-området och motsvarande instrumentinställningar som krävs för experimentet. För böjning styvhetsmätningar av lipid bilayers, använd ett Q-intervall på ~(0,04 - 0,2) Å-1. För studier av membrantjockleksfluktuationer, använd ett Q-intervall på ~(0,04 - 0,2) Å-1 motsvarandemembrantjockleken 35,66,67.
OBS: Diskutera den experimentella installationen med instrumentforskaren innan experimentet påbörjas. Som nämnts tidigare är SANS-karakterisering av provet nödvändigt, särskilt om tidigare information om spridningssignalen inte är tillgänglig, som i selektivt avuterated membran. Alternativt kan du köra statiska (även kallade diffraktionsmätningar) över ett begränsat Q-intervall på NSE-instrumentet, med förbehållet att sådana mätningar tar mycket längre tid jämfört med SANS. - Använd en spruta eller en överföringspipett och ladda den extruderade liposomupphängningen i de angivna provcellerna som finns tillgängliga vid NSE-strållinjer. Observera att vanliga NSE-provceller finns i tjocklekar på 1, 2, 3 och 4 mm. Välj celltjockleken på ett sådant sätt att spridarsignalen optimeras samtidigt som den inkonsekventa bakgrundssignalen behållits till en rimlig intensitet.
OBS: Använd som tumregel provceller med 1 eller 2 mm banlängd för protierade liposomer i avuterated buffert – tjockare celler kan resultera i flera spridningseffekter som är svåra att korrigera för. För liposomer med högre nivåer av deuterationer (t.ex. svanskontrastmatchade liposomer eller asymetrisk liposomer med enstaka protierade broschyrer), överväg att använda en tjockare provcell (t.ex. 3 eller 4 mm pathlength) för att förbättra räknestatistiken om provet finns tillgängligt i större mängder – ibland kan detta vara kostnadsöverkomligt. - Förbered en identisk provcell för bufferten. Använd samma buffert som i liposomupphängningen. Mätningar på bufferten är nödvändiga för intensitetsnormalisering och bakgrundskorrigeringar (BKG).
- Placera provcellerna i provhållaren på NSE-spektrometern, programmera mätkörningarna och samla in ekodata. Rådgör med instrumentforskaren om programmering av mätningarna om en första gången NSE-användare.
- Utför ytterligare två uppsättningar mätningar som behövs för dataminskningen: Upplösning (R) och transmissionsmätningar(T).
- Utför upplösning (R) mätning på en elastisk spridningsreferens (t.ex. kol) – som ska köras under samma inställningar; dvs. samma våghals och Fourier gånger som prov- och buffertmätningarna.
- Utförtransmissionsmätningar (T)på provet och bufferten för att beräkna intensiteten hos den överförda neutronstrålen (se steg 4.9 nedan). Överföringen beräknas som förhållandet mellan neutronantalet från provet eller bufferten dividerat med neutronräkningarna för en öppen stråle (dvs. med en tom provposition).
- Använd den dedikerade dataminskningsprogramvaran för NSE-spektrometern som mätningarna utförs på för att minska de insamlade data.
Obs: Olika spektrometrar kan använda olika program eller användargränssnitt. Nedan är ett exempel på NSE-dataminskning med hjälp av DAVE (Data Analysis and Visualization Environment)68 programvara som är särskilt skriven för NSE-spektrometern vid NIST Center for Neutron Research.- Öppna DAVE-programvaran och välj Minska NSE-data från datareduceringsmenyn. Flera popup-fönster visas.
- Ladda upp datafilerna över olika Q-värden med hjälp av Öppna .echo-filer från arkivmenyn. Dessa filer motsvarar rådatafilerna med spinn eko-signalerna och har tillägget .echo i filnamnet. När filöverföringen är klar visas filerna under tillgängliga datauppsättningar.
- Högerklicka på den valda filen och märk den enligt den mätning den motsvarar. dvs exempel, cell (för tom cell eller buffert) eller upplösning.
- Gruppera detektorpixlarna i 2 x 2 för att förbättra signal-till-brus-förhållandet med hjälp av fliken Datamängd. Använd samma binning på alla filer. dvs. upplösning, cell och prov.
- Kontrollera data över alla pixelgrupper och maskera dem med dåliga signaler (se figur 4B)genom att trycka på m-tangenten på tangentbordet. Tryck på Retur för att komma åt ett popup-fönster för att använda samma mask på alla Fourier-tider eller efterföljande Fourier-tider. Detta kan också tillämpas på enskilda pixlar när som helst under dataminskningen. Maskerade pixlar blir gröna.
- Se till att de insamlade uppgifterna är i form av en ekosignal, dvs.
OBS: Fasströmmen står i proportion till neutronspinns precessionsvinkel. Därför är det vanligt att fasströmmen representeras som en fasvinkel enligt figur 4A. För mätningar på pulsade källor tillämpas ytterligare tid för flygberäkningar på data för att erhålla ekosignalerna som en funktion av incident neutronvåglängd inom en neutronpuls. - Börja med att montera lösningsfilen. Välj en lösningsfil i den uppladdade fillistan och högerklicka på filen. Välj Anpassa åtgärder: Anpassa ekon (upplösning) på popup-menyn.
- Se till att ekosignalernas passform ger ett antal monteringsparametrar, inklusive parametern A, som krävs i steg 4.8. Anfallen utförs automatiskt med följande uttryck.

Här är ζ ekosignalens period (dvs. cosinusfunktion i figur 4A),σ är bredden på det gaussiska kuvertet som bestäms av den genomsnittliga våglängden och våglängdsspridningen av den tillbuds neutronstrålen, Φc är fasströmmen och Φ0 är ekopunkten som beror på den fältväg som neutronerna50 upplev. Fysisk information om provet kodas i amplituden A, för cosininfunktionen i ekvationen (1).
OBS: Bredden på det gaussiska kuvertet baseras på värden som instrumentforskaren förutbestämt och bör inte ändras. De andra paramtersna är variabler som är monterade på den specifika ekosignalen över varje pixel. - Inspektera passformsresultaten genom att klicka på varje pixel för att visa de resulterande monteringsparametrarna, passformens kvalitet och passformens genomsnittliga kvadratavvikelse. Om du vill kontrollera felet som är associerat med varje monteringsparameter över hela detektorn väljer du Bildalternativ och väljer sedan den anpassningsparameter som är av intresse. Detta genererar en karta med värdet av den passande paramtern över varje pixel. Högerklicka på detektorbilden. Ett popup-fönster visas som visar en felfältskarta över den valda anpassningsparametern.
- Om passformen över en viss pixel är otillfredsställande (t.ex. montera parameeters med stora felstaplar) ska du montera om signalen över den specifika pixeln. Markera pixeln, tryck på fliken Anpassa och tryck sedan på Anpassa pixel. Mata in nya startparametrar för fasen (Φ0) och period (ζ) på fliken Montering för att få en mer tillfredsställande passform.
OBS: Det är användbart att plotta den monterade fasen som en funktion av Fourier Time. Det gör du genom att gå till huvuddiagramfönstret och välja Anpassa fas v. Fourier-tid. Denna tomt ska vara jämn och kontinuerlig. Inspektera avbrott i detta diagram och montera om pixlarna som de motsvarar.
- Minska exempel- eller cellfilen genom att välja motsvarande fil i den uppladdade och märkta fillistan.
- Inspektera alla pixlar och maskera pixlarna med dålig statistik enligt beskrivningen i steg 4.7.5.
- Högerklicka på filen och välj Anpassa åtgärder: Importera faser (exempel, cell). Detta importerar faserna och den tillämpade masken från resolutionsfilen.
- Montera ekosignalerna med samma procedur som beskrivits tidigare för resolutionsfilen (steg 4.7.8-4.7.10). När du monterar exempel- och cellfilerna ska du inte ändra värdena för perioden och ekofaspunkten som importeras från upplösningen passar. Dessa parametrar beror på de instrumentella inställningarna och bör inte variera med prover.
- Innan du fortsätter till dataminskningen anger du strålcentret för alla datafiler. Markera datafilen, gå till fliken Allmänt och ange X- och Y-strålcentervärden. Dessa värden registreras under experimentet.
- När filerna Exempel, Cell och Upplösning är klara beräknar du den normaliserade mellanliggande spridningsfunktionen som ska användas senare i dataanalys och tolkning. För att göra det högerklickar du på exempelfilen för att minskas från listan över monterade filer och väljer Beräkna I(Q) på popup-menyn. Ett fönster visas med postval för filerna Upplösning och cell (dvs. buffert) och antalet Q-bågar (se steg 4.9). När du har gått in i all nödvändig informaiton trycker du på OK-knappen. Resultaten visas i ett nytt fönster.
OBS: Dataminskningen utförs enligt följande ekvation för att erhålla den normaliserade mellanliggande spridningsfunktionen69.
Där t är Fourier tid, Nupp och Nner är neutronräkningarna i icke-spin-flip- och spin-flip-konfigurationerna (mätt med π/2-flippers av respektive π-flipper off respektive on) och de upphöjda, BKG och R, motsvarar bakgrunds- respektive upplösningsmätningarna enligt definitionen i steg 4.4 respektive 4.6. Observera att strålpolariseringen , således förändras i spinntillståndet på grund av energiutbyte mellan neutronen och provet detekteras som en minskning av polariseringen (från enhet).
, således förändras i spinntillståndet på grund av energiutbyte mellan neutronen och provet detekteras som en minskning av polariseringen (från enhet).
- Gruppera slutligen detektorpixlar i Q-bågar som visas i figur 4B för att få Q-beroendet av den normaliserade mellanliggande spridningsfunktionen, S(Q,t) / S(Q, 0). Detta kallas tekniskt datalagring och bör göras på ett omdömesgillt sätt, dvs.
- För starkt spridande prover, dela detektorn i fler Q-bågar samtidigt som rimliga felstaplar bibehålls på den resulterande mellanliggande spridningsfunktionen, S(Q,t) / S(Q, 0). Detta ger fler Q-datapunkter och är viktigt för dataanalysförfarandet som beskrivs nedan. Var medveten om att för svagt spridande prover resulterar överdriven binning i dåliga förfallssignaler, dvs. stora felstaplar på S(Q,t) / S(Q, 0), vilket kan leda till stora osäkerheter.
5. Dataanalys och tolkning
- Montera de normaliserade mellanliggande spridningsfunktionerna, S(Q,t) / S(Q, 0), som erhålls från dataminskningen ovan till en sträckt exponentiell funktion med en sträckningsförmedlade exponent på 2/370.

OBS: Ett exempel på dessa anfall finns i figur 5B. Passar S(Q,t) / S(Q, 0) till ekvation (3) ger de Q-beroende avslappningshastigheterna Γ(Q). - Plott Γ( Q )somen funktion av Q och passar till en lämplig modell för att extrahera relevanta membranparametrar.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
NSE-studier som får tillgång till böjningsfluktuationer utförs vanligtvis över ett Q-intervall på ~ (0,04 - 0,2) Å-1. Detta Q-intervall motsvarar mellanliggande längdskalor mellan membrantjockleken och liposomradien, där böjningsdynamiken dominerar. Mätning över ett utökat Q-intervall kan ge tillgång till ytterligare dynamiska lägen, inklusive liposomal diffusion och intramembrandynamik. För mer information om cross-over i membrandynamik som nås av NSE, kontrollera dessa relevanta publikationer25,71. Det är viktigt att betona att NSE-signaler står i proportion till:  , där jagcoh respektive Iinc är den sammanhängande och inkonsekventa spridningsintensiteten från provet. Därför är det lämpligt att förbereda NSE liposomalprover i avfettade buffertar (dvs. buffertar som framställs med D2O i stället för H2O) för att minimera den osammanhängande spridningssignalen, främst till hjälp av provets väteinnehåll. I vissa fall kan dock mellanliggande utmatningssystem (dvs. användning av blandningar av D2O och H2O) vara nödvändiga för att uppnå optimala kontrastförhållanden. Vanligtvis utförs NSE-mätningar av membranbockningsfluktuationer på fullständigt protierade liposomer i avuterated buffert, så kallade helt kontrasterade liposomer i figur 5. Detta utsuttringsschema resulterar i en stor NSLD-skillnad mellan membrankärnan (~0 × 10-6 Å-2)och dess avutetrerade vätskemiljö (~6,4 × 10-6 Å-2), vilket avsevärt förbättrar spridningssignalen från liposomala membran och förbättrar mätstatistiken för böjningsdynamik. Detta kontrastschema(figur 2A vänster panel) används ofta i studier av böjande styvhet i lipidmembran medenstaka 38,72 och flera39,66 lipidkomponenter och i studier av membranmjukhet/förstyvning genom biologiska inneslutningar (t.ex. kolesterol, läkemedelsmolekyler, peptider/proteiner)36,37,73,74,75, och syntetiska tillsatser (t.ex. nanopartiklar)76,77.
, där jagcoh respektive Iinc är den sammanhängande och inkonsekventa spridningsintensiteten från provet. Därför är det lämpligt att förbereda NSE liposomalprover i avfettade buffertar (dvs. buffertar som framställs med D2O i stället för H2O) för att minimera den osammanhängande spridningssignalen, främst till hjälp av provets väteinnehåll. I vissa fall kan dock mellanliggande utmatningssystem (dvs. användning av blandningar av D2O och H2O) vara nödvändiga för att uppnå optimala kontrastförhållanden. Vanligtvis utförs NSE-mätningar av membranbockningsfluktuationer på fullständigt protierade liposomer i avuterated buffert, så kallade helt kontrasterade liposomer i figur 5. Detta utsuttringsschema resulterar i en stor NSLD-skillnad mellan membrankärnan (~0 × 10-6 Å-2)och dess avutetrerade vätskemiljö (~6,4 × 10-6 Å-2), vilket avsevärt förbättrar spridningssignalen från liposomala membran och förbättrar mätstatistiken för böjningsdynamik. Detta kontrastschema(figur 2A vänster panel) används ofta i studier av böjande styvhet i lipidmembran medenstaka 38,72 och flera39,66 lipidkomponenter och i studier av membranmjukhet/förstyvning genom biologiska inneslutningar (t.ex. kolesterol, läkemedelsmolekyler, peptider/proteiner)36,37,73,74,75, och syntetiska tillsatser (t.ex. nanopartiklar)76,77.
Mätningar av böjningsfluktuationer resulterar i avslappningshastigheter som följer ett Q3-beroende, som förutspåtts av Zilman och Granek för termiskt böljande elastiska tunnaark 70. En raffinerad form av detta Q-beroendeerhålls från teoretiska korrigeringar av Watson och Brown78, som tar hänsyn till effekterna av intermonolayer friktion som föreslås av Seifert och Langer79. Genom att dessutom definiera det neutrala planet som ska vara vid gränssnittet mellan de hydrofila huvudgrupperna och membranets hydrofoba svansar kan de böjande avslappningshastigheterna sedan monteras på följandeuttryck 38.

Där ηbuff är buffertviskositeten, kBT är termisk energi, κ och är det uppmätta membranets böjande styvhet (eller den kontrasterade delen av membranet i selektivt avuterated system). Denna typ av mätning möjliggör direkt beräkning av membranelastiska egenskaper i form av böjstyvhetsmodulen. Observera att κ extraheras från lutningen på den linjära passformen på Γ jämfört med Q3, som visas i figur 5C.
Å andra sidan visar NSE-mätningar av membrantjockleksfluktuationer avvikelser från Q3-beroendeti Γ(Q)runt Q-värden som motsvarar membrantjockleken (se figur 2 i ref.66). För att isolera tjockleksfluktuationssignalen kan man dividera ΓQmed Q3, som visas i figur 5D. De resulterande data visar att överskottsdynamiken på grund av tjockleksfluktuationer följer en Lorentzian-funktion i Q, som nyligen bekräftades i grovkorniga molekylära dynamiksimuleringar (MD)67. För att passa den observerade överskottsdynamiken utvecklade Nagao et al.38 ett uttryck baserat på den teoretiska ramen för membranfluktuationer av Bingham et al.80 enligt följande.

I detta uttryck är Q0 det högsta Q-värdetsom motsvarar membrantjockleken (som kan erhållas oberoende av SANS-mätningar), μ är membranviskositeten i planet, AL är området per lipid (mätt med SANS/SAXS), och KA är områdets komprimerbarhetsmodul. Förutsatt att KA kan beräknas från κ med polymerborstmodellen, reducerar detta uttryck till en passformsparameter, nämligen membranviskositeten μ , vilket presenterar ett nytt tillvägagångssättför att mäta membranviskositet utan behov av fluorescensmärkning eller partikel tjudring /spårning 13. Förutsättningen är att enligt deformationsmodeller av elastiska tunnaark 81, κ och KA är beroende av varandra så att: ,  där tm är den mekaniska (eller deformerbara) membrantjockleken och β är en konstant som beskriver interleafletkoppling. Antagandet är att β = 12 för helt kopplade broschyrer, β = 48 för helt frikopplade broschyrer, och β = 24 för mellanliggande kopplade broschyrer. Den senare kallas polymerborste modell81 och har visat sig gälla i enkomponents och binära flytande lipidmembran39. Detta måste dock hanteras med försiktighet. Till exempel, senaste simuleringar av Doktorova et al. 82 visade att för att polymerborstmodellen ska hålla i omättade lipidmembran som innehåller kolesterol måste ett modifierat uttryck för den mekaniska membrantjockleken användas. Helst, om en oberoende mätning av KA är möjlig, t.ex. med hjälp av mikropipette strävan83, då kombinera KA resultat med NSE böjning styvhet mätningar skulle utgöra en unik möjlighet att undersöka interleaflet koppling i modell och biologiska membran – en långvarig fråga i membran biofysik och strukturell biologi. När värdena för KA har validerats kan de användas i ekvation 5 för att erhålla den mesoskopiska membranviskositeten.
där tm är den mekaniska (eller deformerbara) membrantjockleken och β är en konstant som beskriver interleafletkoppling. Antagandet är att β = 12 för helt kopplade broschyrer, β = 48 för helt frikopplade broschyrer, och β = 24 för mellanliggande kopplade broschyrer. Den senare kallas polymerborste modell81 och har visat sig gälla i enkomponents och binära flytande lipidmembran39. Detta måste dock hanteras med försiktighet. Till exempel, senaste simuleringar av Doktorova et al. 82 visade att för att polymerborstmodellen ska hålla i omättade lipidmembran som innehåller kolesterol måste ett modifierat uttryck för den mekaniska membrantjockleken användas. Helst, om en oberoende mätning av KA är möjlig, t.ex. med hjälp av mikropipette strävan83, då kombinera KA resultat med NSE böjning styvhet mätningar skulle utgöra en unik möjlighet att undersöka interleaflet koppling i modell och biologiska membran – en långvarig fråga i membran biofysik och strukturell biologi. När värdena för KA har validerats kan de användas i ekvation 5 för att erhålla den mesoskopiska membranviskositeten.

Figur 1:NSE-instrumentets design och synergistiska överlappning med längd/tidsskalor av mesoskopisk membrandynamik. Den markerade neutronen indikerar förändring i spinnorientering (eller polariseringsförlust) på grund av energiutbyte mellan neutronen och provet, medan den genomskinliga neutronen representerar spinn-eko, dvs ingen förändring i neutronspinn på grund av nollenergiutbyte. Den grå pilen anger möjligheten att rotera spektrometerns andra arm för att komma åt större spridningsvinklar. (B) Bildrepresentation av hierarkisk dynamik i lipidmembran, som visar olika dynamiska lägen som sträcker sig över flera längd- och tidsskalor. Det skuggade området representerar de längd- och tidsskalor som NSE har tillgång till, som överlappar mesoskalan för kollektiva membranfluktuationer, nämligen böjnings- och tjockleksfluktuationer. Klicka här om du vill visa en större version av den här figuren.

Figur 2: Exempel på möjliga utsutringningssystem i NSE-experiment på lipidmembran. (A) Vänster: Helt kontrasterade membran, t.ex. protierade membran i avuterated buffert, som visar NSLD-profilen längs det normala till membranytan. Skillnaden i NSLD mellan svansregionen (~0 × 10-2 Å-2)och huvudgruppsregionen (~4,5 × 10-6 Å-2)i membranet beror på huvudgruppens hydrering med avuterated buffert. Höger: Svanskontrastmatchade membran så att kolvätesvansregionen i membranet har samma NSLD som bufferten, vilket visas i motsvarande NSLD-profil längs membranet normalt. (B) Domänbildande membran med två neutronkontrastscheman där domänerna (mitten) eller matrisen (vänster) kontrastmatchas med bufferten, vilket möjliggör selektiva studier av matris- respektive domändynamik. Denna siffra har ändrats från Nickels et al., JACS 201541. C)Asymmetriska membran som framställs genom cyklodextrinutbyte mellan protierade och avuterated lipid vesiklar, vilket resulterar i utmattning av en membran broschyr samtidigt som den andra bipacksedeln behålls protiated. Detta möjliggör studier av den protierade broschyrens böjningsdynamik och ger insikter om den mekaniska kopplingen mellan motsatta broschyrer i asymmetriska membran. Denna siffra har ändrats från Rickeard et al., Nanoscale 202040. Klicka här om du vill visa en större version av den här figuren.

Bild 3:Illustration av installationen förautomatiserad extrudering av liposomer. (A) Specialbyggd automatiserad extruder med hjälp av en sprutpump, en mini extruderuppsättning och en aluminium / stålram för att möjliggöra cykliska profiler. BochC visarskillnadeni visuellt utseende av lipid suspensioner före (mjölkvita) och efter (transparent opal blå) extrudering. Detta beror på den första bildandet av lipidstaplar i mikronstorlek eller gigantiska blåsor som är i storleksordningen, eller större än, våglängden av synligt ljus. Efter extrudering kommer suspensionen att omfatta nanoskopiska blåsor (~ 100 nm), som är mindre än våglängden av synligt ljus, vilket ger en transparent suspension. Klicka här om du vill visa en större version av den här figuren.

Figur 4: Representativa data från NSE-experiment på liposomala suspensioner. (A) Exempel på en ekosignal över en enda detektorpixel (markerad pixel i panel B), som visar ekosignalens anfall med hjälp av ekvation (1), med en illustration av de olika parametrar som krävs i ekopassningen. Observera att ekosignalen ritas som en funktion av fasvinkeln i stället för fasströmmen som beskrivs i steg 4.7 i protokollet. (B) NSE-detektorbild som visar variationen i neutronantal per pixel. Bilden visar också eliminerade detektorpixlar (gröna) på grund av dåliga ekosignaler. Binning av detektorpixlar i Q-bågar (även känd som Debye-Scherrer-ringar) ger Q-beroendet av den mellanliggande spridningsfunktionen, som behövs för att analysera och tolka NSE-data. Denna siffra ändrades från Ashkar, J. Appl. Phys. 202050. Klicka här om du vill visa en större version av den här figuren.

Figur 5: Representativa resultat från NSE-experiment på liposomala suspensioner med olika utsuttringssystem. A)Spridningsgeometri för en neutron som interagerar med en liposom, som visar spridningsvinkeln,2 θ,och vågvektoröverföringen.  (B) Mellanliggande spridningsfunktioner, S(Q,t) / S(Q,0), uppvisar förfall som en funktion av Fourier-tiden. Passformen av de uppmätta förfallen till en sträckt exponentiell funktion som ges av ekvation 3 ger avslappningshastigheterna, Γ(Q). C)För fullt protierade liposomer i avuterated buffert följer Γ(Q) ett Q3-beroende, typiskt för böjningsdynamik. Den linjära passformen hos de erhållna data till en Zilman-Granek-modell ger membranets böjande styvhetsmodul. (D) För svansdeuterated liposomer observeras överskottsdynamik förutom böjningsfluktuationer och är mest uttalad vid Q-värden som motsvarar membrantjockleken. Genom att anpassa överskottsdynamiken till en Lorentzian-funktion (ekvation 5) kan extraktion av membranets viskositet. Datamängder samlades in på NSE-spektrometern vid NIST. Klicka här om du vill visa en större version av den här figuren.
(B) Mellanliggande spridningsfunktioner, S(Q,t) / S(Q,0), uppvisar förfall som en funktion av Fourier-tiden. Passformen av de uppmätta förfallen till en sträckt exponentiell funktion som ges av ekvation 3 ger avslappningshastigheterna, Γ(Q). C)För fullt protierade liposomer i avuterated buffert följer Γ(Q) ett Q3-beroende, typiskt för böjningsdynamik. Den linjära passformen hos de erhållna data till en Zilman-Granek-modell ger membranets böjande styvhetsmodul. (D) För svansdeuterated liposomer observeras överskottsdynamik förutom böjningsfluktuationer och är mest uttalad vid Q-värden som motsvarar membrantjockleken. Genom att anpassa överskottsdynamiken till en Lorentzian-funktion (ekvation 5) kan extraktion av membranets viskositet. Datamängder samlades in på NSE-spektrometern vid NIST. Klicka här om du vill visa en större version av den här figuren.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
NSE är en kraftfull och unik teknik för att mäta mesoskopisk dynamik i lipidmembran under olika förhållanden. Det effektiva utnyttjandet av NSE beror på provkvalitet, neutronkontrast och det intervall av tillgänglig dynamik som kan undersökas för ett visst prov. Således krävs flera kritiska steg för att utföra framgångsrika NSE-experiment och samla in högkvalitativa data. Ett viktigt steg för att säkerställa effektiv användning av neutronstråletid under ett NSE-experiment är att karakterisera liposomala suspensioner med labbbaserade metoder före NSE-experimentet. För exmapel kan storleksfördelningen (eller diffusionskonstanten) av extruderade liposomer bestämmas av dynamisk ljusspridning (DLS), lätt tillgänglig i enskilda laboratorier eller i delade anläggningar84. Cryo-elektronmikroskopi är en annan charcaterization metod som nyligen validerats på liposomal prover, där högupplösta mikroskopi bilder på cryomicrotomed delar av liposomal suspension kan effektivt användas för att undersöka liposomal unilamellarity65,domänbildning 85,86, eller införlivande av tillsatser såsom nanopartiklar76 och proteiner87. Alternativt kan småvinkelröntgenspridning (SAXS) användas för att karakteriseramembranstrukturen 88, bedöma liposomal multilamellaritet65, eller utvärdera effekterna av tillsatser på membranets strukturella egenskaper89. Förutom dessa labbbaserade tekniker är det mycket tillrådligt att NSE-mätningar på liposomala prover paras ihop med strukturella studier med hjälp av liten vinkel neutronspridning (SANS)54,90. SANS är ett utmärkt komplement till NSE, inte bara för att förvärva strukturell membraninformation utan också för att undersöka intensiteten hos neutronspridningssignalen från provet, bekräfta kontrastschemat och göra ett välgrundat val om det Q-intervall över vilket NSE-mätningarna bör utföras. Därför rekommenderas att NSE-användare begär SANS-stråltid när de ansöker om NSE-experiment.
NSE lider dock av provbegränsningar i studier av biologiska membran. En av de viktigaste begränsande faktorerna för sådana experiment är den standardmängd provmängd som krävs för NSE-mätningar (2-4 ml) och de höga provkoncentrationerna på 100–200 mg membranmaterial (lipider och proteiner) för att erhålla data av hög kvalitet. I många fall är det inte genomförbart att producera sådana mängder biologiskt material eller är kostnadsöverkomligt. I sådana scenarier är det möjligt att minska koncentrationen till 20–25 mg/ml, men detta skulle kräva minst en 4-faldig ökning av förvärvstiden för att få jämförbar statistik med prover med 50 mg/ml koncentrationer. Dessa stränga krav på provvolym och koncentration skulle kunna lindras med nästa generations NSE-spektrometrar på neutronkällor med högre flöde, såsom den andra målstationen vid Oak Ridge National Lab och European Spallation Source. En annan kritisk begränsning vid utförandet av NSE-experiment på lipidmembran som kräver selektiva utmattningssystem är bristen på kommersiell tillgänglighet hos vissa avuterated varianter av lipidmolekyler eller deras orimliga priser, om sådana finns tillgängliga. I vissa fall kan dessa begränsningar kringgås genom att begära syntes av avutererade lipider (eller kolesterol, proteiner) genom användardeuterationsanläggningar, såsom biodeuterationslabbet vid Oak Ridge National Lab, den nationella utmatningsanläggningen vid ANSTO eller utmattningsanläggningen vid ISIS Neutron och Muon Source. Tillgång till dessa anläggningar och deras synteskapacitet är tillgänglig genom inlämnade användarförslag som är peer-reviewed baserat på vetenskapliga meriter av den föreslagna materialsyntesen och dess avsedda användning i isotopkänsliga studier.
Trots dessa begränsningar har tillämpningen av NSE-spektroskopi i studier av membranmekanik lett till bestämning av böjningsstelhetsmoduli av membran med olika grad av komplexitet, från enkomponent lipidmembran35,38 till multikomponent biomimetiskamembran 41,66,91, som alla har främjat vår förståelse av lipidmembranens dynamiska natur. Till exempel har NSE-böjningsstyvhetsmätningar av lipidmembran med olika molekylära enheter, t.ex. lipider av olika aylkedjelängder och kedjemättnad38,72,92, gett viktig information om molekylär kemins roll i membranmekaniken. När de paras ihop med strukturell information, såsom membrantjocklek ellermolekylär packning 93, börjar dessa mätningar ge nya perspektiv på det ömsesidiga beroendet mellan membranstruktur och dynamik och hur de påverkar membranets funktion. De mesoskopiska skalor av NSE placerar det unikt för sådana grundläggande undersökningar av struktur-egendomsrelationer, mest relevanta på längden på molekylära enheter. Detta ämne undersöktes nyligen i två NSE-studier på kolesterolrika lipidmembran36 och i binära lipidmembran med hydrofobisk missmatchning mellan de två lipidkomponenterna39. Båda studierna fann starka bevis för att membranmekanik skalar med området per lipid, vilket bekräftar slutsatserna från en nyligen all-atom MD simuleringar av Doktorova et al.82. Dessa resultat betonar den självmonterade karaktären av lipidmembran och ger en enande bild av molekylär förpackning som en nyckelparameter för att definiera membran dynamiska och funktionella egenskaper.
Andra tillämpningar av NSE involverar studier av membranens mekaniska svar på små tillsatser, inklusive biologiska molekyler som kolesterol36,37,trehalos92, och melittin73,94, eller oorganiska tillsatser som nanopartiklar för läkemedelsleveransapplikationer76. NSE har också använts för att förstå hur membranmekanik svarar på förändringar i deras miljö, inklusive temperatur92, pH74, och närvaron av trängsel makromolekyler96. Sådana studier bidrar till en bättre förståelse av de faktorer som påverkar mjukning eller förstyvning av lipidmembran, under biologiska förhållanden relaterade till hälsa och sjukdom, och i kontrollerade miljöer för terapeutiska tillämpningar. Noterbart är att NSE-mätningar också har använts för att undersöka effekten av antimikrobiella peptider påmembrandynamiken 73,94,95. Ytterligare exempel på NSE-tillämpningar på biomembran inkluderar studier av dynamiken hos platta membranstrukturer, kallade tylakoider, som inrymer den fotosyntetiska maskinen i cyanobakteriellaceller 97,98.
Man kan också använda selektiv lipiddeuteration i NSE-studier för att undersöka dynamiken hos specifika membranfunktioner som är relevanta för biologisk funktion. Nickels et al. använde till exempel selektiv lipiddeuteration i domänbildande lipidmembran för att generera lateral kontrast i membranet, vilket tidigare illustrerats av Heberle et al.28. Detta utmatningssystem möjliggjorde oberoende mätningar av lipiddomänernas böjningsstyvhet och värdfettmatrisen41 (se figur 2B). Resultaten bekräftade att de två membran facken har distinkt böjning styvhet moduli, som kan vara en körmekanism för domänbildning i cellulära membran. I en nyare studie använde Rickeard et al. cyklodextrinutbyte mellan protierade och avuterated liposomer för att få asymmetriska liposomer med isotopiskt märktabroschyrer 40 (Figur 2C). Deras slutliposomer hade en protiated broschyr och en utstött broschyr som är kontrast matchad med bufferten, möjliggör studier av enskilda broschyrdynamik och ger en första direkt experimentell redogörelse för effekten av asymmetri och broschyr koppling på membran böjning fluktuationer.
Selektiv membrandeuteration har också använts i NSE-studier av membrantjockleksfluktuationer, ett länge förutspått dynamiskt läge i lipidmembran99 som först nyligen observerades med tillkomsten av NSE-spektroskopi35,100. Dessa mätningar använder svansdeutade membran för att förstärka signalen från membranhuvudgruppens regioner och lösa tjockleksfluktuationssignalen. Denna typ av NSE-experiment är relativt nya, men det har effektivt använts för att förstå det ömsesidiga beroendet av membranelastiska och trögflytandeegenskaper 38, för att utforska skalningen av böjande styvhet och viskositet med molekylär förpackning i blandade lipidmembran39, och för att undersöka de lokala effekterna av kolesterol på membranviskositet36. Ett annat område av biologisk betydelse där detta dynamiska läge kan få långtgående konsekvenser är mesoskopiska membranproteininteraktioner95. Det är känt att funktionen hos membranproteiner är tätt kopplad till hydrofobisk matchning mellan proteinet och värdmembranet. Variationer i membrantjocklek, på grund av tjockleksfluktuationer, kan således fungera som en regleringsmekanism för funktionen av membranproteiner. NSE är extremt väl lämpat för sådana studier eftersom det direkt kan undersöka effekterna av proteinbindning och införande på membrantjockleksfluktuationer. Nyligen genomförda NSE mätningar från vår grupp (opublicerade) tyder på att transmembran protein insättningspunkten kan avsevärt undertrycka membran tjocklek fluktuationer och kan presentera en potentiell mekanism för att reglera signalering händelser. Detta är ett pressande, men underutvecklat, forskningsområde där NSE kan ha betydande inverkan på förståelsen av membranens dynamiska svar på proteinbindning och införande på längd- och tidsskalor av viktiga biologiska funktioner som förmedlas av interaktioner mellan proteiner och cellmembran.
Sammanfattningsvis har NSE utvecklats under de senaste åren som ett kraftfullt verktyg för att förhöra membrandynamik över rumsliga och temporala skalor av vitala biologiska funktioner. Tekniken får snabbt ett brett intresse och dess potential att svara på viktiga frågor i membranfunktionen blir välkänd. Kontrastvariationskapaciteten inom NSE har positionerat den som ett unikt tillvägagångssätt för att mäta mesoskopiska membranegenskaper som annars skulle vara utmanande att få. En annan betydande fördel med NSE jämfört med traditionella spektroskopimetoder i studier av membrandynamik är dess överlappning med de längd- och tidsskalor som är tillgängliga med MD-simuleringar, vilket möjliggör synergistiska experimentella / beräkningsstudier för att få en molekylär nivåförståelse för de olika molekylära komponenterna som utgör membran. Trots löftet finns det fortfarande vissa begränsningar i användningen av NSE i biologiska membranstudier, inklusive kravet på stora provvolymer, svårigheten att selektiv uttlåning i biologiska system och det relativt låga neutronflödet på NSE-spektrometrar, vilket resulterar i längre mättider och begränsad stråltidstillgänglighet. Dessa brister skulle dock kunna övervinnas inom en snar framtid med en ständig utveckling av neutronkällor och instrumentering tillsammans med framsteg inom utsuttringsanläggningar.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Författarna förklarar inga intressekonflikter och har inget att avslöja.
Acknowledgments
R. Ashkar tackar M. Nagao, L.-R. Stingaciu och P. Zolnierczuk för många användbara diskussioner och för deras frekventa hjälp med NSE-experiment på sina respektive strållinjer. Författarna erkänner användningen av neutron spin echo spektrometrar vid NIST och ORNL. NSE-spektrometern vid NIST stöds av Center for High Resolution Neutron Scattering, ett partnerskap mellan National Institute of Standards and Technology och National Science Foundation enligt avtal nr. DMR-1508249. NSE-spektrometern vid ORNL:s Spallation Neutron Source stöds av Scientific User Facilities Division, Office of Basic Energy Sciences, US Department of Energy. Oak Ridge National Laboratory förvaltas av UT-Battelle, LLC under US DOE Contract No. DE-AC05-00OR22725.
Materials
| Name | Company | Catalog Number | Comments |
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. Ø, Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes--a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich's model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a "modeling manifesto". Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D. Neutrons in Soft Matter. Imae, T., Kanaya, T., Furusaka, M., Torikai, N. , Wiley. ch6 147-182 (2011).
- Pynn, R. Neutron Spin Echo. Mezei, F., Pappas, C., Gutberlet, T. , Springer. Berlin Heidelberg. 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , Oxford University Press. (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- Neutron activation and scattering calculator. , Available from: https://www.ncnr.nist.gov/resources/activation/ (2021).
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , Walter de Gruyter, Inc. 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles - as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O'Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -R., O'Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).
Tags
Biologi Nummer 171 kollektiv lipiddynamik böjmodulus områdeskomprimerbarhet tjockleksfluktuationer membranviskositet selektiv avutluftning liposompreparatErratum
Formal Correction: Erratum: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions
Posted by JoVE Editors on 08/06/2021.
Citeable Link.
An erratum was issued for: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions. The Introduction, Protocol, and Representative Results sections have been updated.
In the Introduction, the fith pargraph was updated from:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 2 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
to:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 0 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
In the Protocol, step 1.1 was updated from:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 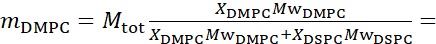 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
to:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 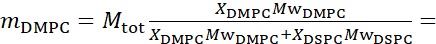 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
In the Representative Results, the fist pagargaph was updted from:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 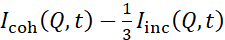 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
to:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 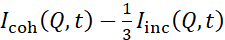 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
In the Representative Reults, Figure 2 was updated from:
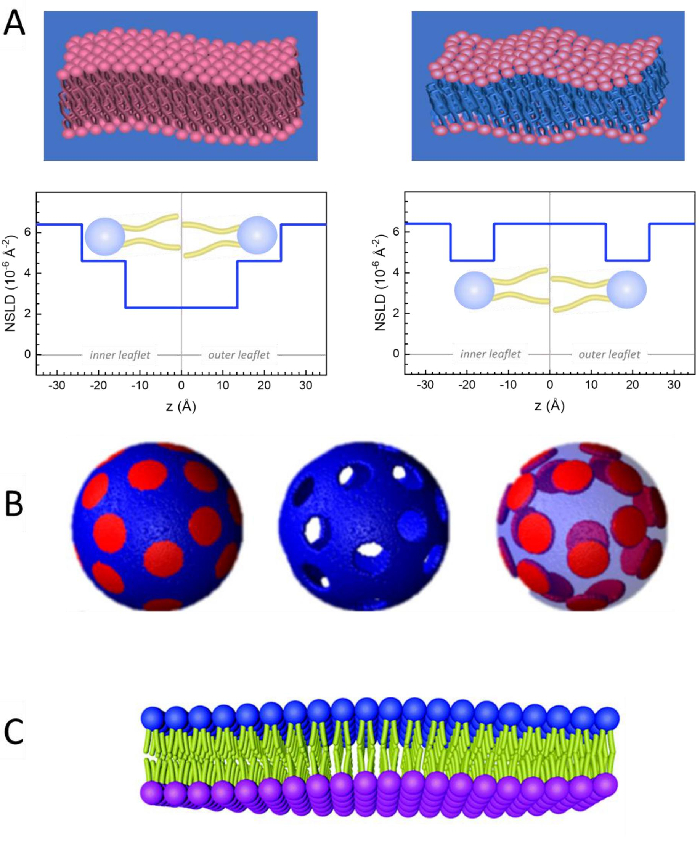
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the headgroup (~2 × 10-2 Å-2) and tail region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.
to:
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the tail region (~0 × 10-2 Å-2) and headgroup region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.



 133,4 mg, Där XDMPC och XDSPC är molfraktionerna DMPC och DSPC, här inställda på 0,7 respektive 0,3, och MwDMPC respektive MwDSPC är molvikterna som ges av 677,9 g/mol respektive 790,1 g/mol. På samma sätt mDSPC = 66,6 mg. Detta utlösningsschema ökar spridningskontrasten mellan membranet (NSLD ~ 0 × 10-6 Å-2) och den avutererade bufferten (NSLD ~ 6,4 × 10-6 Å-2) och förstärker signalen från membranutsudlingar (se figur 2A vänster panel).
133,4 mg, Där XDMPC och XDSPC är molfraktionerna DMPC och DSPC, här inställda på 0,7 respektive 0,3, och MwDMPC respektive MwDSPC är molvikterna som ges av 677,9 g/mol respektive 790,1 g/mol. På samma sätt mDSPC = 66,6 mg. Detta utlösningsschema ökar spridningskontrasten mellan membranet (NSLD ~ 0 × 10-6 Å-2) och den avutererade bufferten (NSLD ~ 6,4 × 10-6 Å-2) och förstärker signalen från membranutsudlingar (se figur 2A vänster panel). tot är den totalamassan, inställd här till 200 mg. Se exemplet ovan för DMPC-DSPC lipid bilayers med olika utsuttningssystem.
tot är den totalamassan, inställd här till 200 mg. Se exemplet ovan för DMPC-DSPC lipid bilayers med olika utsuttningssystem.

 , således förändras i spinntillståndet på grund av energiutbyte mellan neutronen och provet detekteras som en minskning av polariseringen (från enhet).
, således förändras i spinntillståndet på grund av energiutbyte mellan neutronen och provet detekteras som en minskning av polariseringen (från enhet).
