ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
De huidige studie rapporteert een protocol voor chromosoomscreening van menselijke embryo's waarbij gebruik wordt gemaakt van gebruikt kweekmedium, waardoor embryobiopsie wordt vermeden en chromosoomploïdie-identificatie met behulp van NGS mogelijk wordt. Het huidige artikel presenteert de gedetailleerde procedure, inclusief de voorbereiding van kweekmedium, whole genome amplification (WGA), next-generation sequencing (NGS) bibliotheekvoorbereiding en data-analyse.
Abstract
Bij klinische in-vitrofertilisatie (IVF) vereist de heersende methode voor PGT-A een biopsie van enkele cellen uit het trophectoderm (TE). Dit is de afstammingslijn die de placenta vormt. Deze methode vereist echter gespecialiseerde vaardigheden, is invasief en lijdt aan valse positieven en negatieven omdat het aantal chromosomen in de TE en de binnenste celmassa (ICM), die zich ontwikkelt tot de foetus, niet altijd hetzelfde zijn. NICS, een technologie die sequencing vereist van DNA dat vrijkomt in het kweekmedium van zowel TE als ICM, kan een uitweg bieden voor deze problemen, maar het is eerder aangetoond dat het een beperkte werkzaamheid heeft. De huidige studie rapporteert het volledige protocol van NICS, dat bemonsteringsmethoden voor kweekmediums, amplificatie van het hele genoom (WGA) en bibliotheekvoorbereiding omvat, en NGS-gegevensanalyse door analysesoftware. Gezien de verschillende cryopreservatietijden in verschillende embryolaboratoria, hebben embryologen twee methoden voor het verzamelen van embryokweekmedium die kunnen worden geselecteerd op basis van de werkelijke omstandigheden van het IVF-laboratorium.
Introduction
Geassisteerde voortplantingstechnologieën (ART's) worden steeds vaker gebruikt voor de behandeling van onvruchtbaarheid. Het slagingspercentage van ART, zoals IVF, is echter beperkt en het zwangerschapsverliespercentage is aanzienlijk hoger dan dat van de normale populatie1. De belangrijkste oorzaak van deze problemen zijn chromosomale afwijkingen, die vaak voorkomen in pre-implantatie menselijke embryo's2. PGT-A is een effectieve methode om embryo's te screenen op chromosomale balans vóór implantatie 3,4. Sommige onderzoeken hebben aangetoond dat PGT-A het aantal abortussen kan verminderen en het aantal zwangerschappen kan verbeteren 5,6,7,8. PGT-A vereist echter complexe technische expertise die specifieke training en ervaring vereist. De invasieve embryobiopsieprocedure kan mogelijk ook schade aan de embryo's veroorzaken9. Studies hebben aangetoond dat blastomeerbiopsie de latere ontwikkeling kan belemmeren en dat het aantal biopsieën van TE's de implantatiesnelheden kan beïnvloeden10. Hoewel het bioveiligheidsprobleem op lange termijn van embryobiopsie nog niet grondig is geëvalueerd bij mensen, hebben dierstudies aangetoond dat het een negatieve invloed heeft op de ontwikkeling van embryo's11,12,13.
Eerdere rapporten gaven aan dat sporen van DNA-materiaal werden uitgescheiden in het kweekmedium tijdens de ontwikkeling van het embryo, en er zijn inspanningen geleverd om uitgebreide chromosoomscreening (CCS) uit te voeren met behulp van gebruikt embryocultuurmedium 14,15,16,17,18. De detectiepercentages en de nauwkeurigheid van de tests voldeden echter niet aan de vereisten voor uitgebreid klinisch gebruik. De huidige studie rapporteerde een verbetering in de NICS-test voor het verhogen van de detectiepercentages en de nauwkeurigheid van de NICS-test19. In de afgelopen jaren is blastocoelevloeistof (BF) bestudeerd als een analytisch monster van minimaal invasieve PGT-A. Het aandeel van succesvolle genoombrede amplificatie en detecteerbaar DNA in blastocystvloeistofmonsters varieert echter van 34,8% tot 82%20,21,22. Het volume BF dat in verschillende onderzoeken wordt gerapporteerd, varieert van 0,3 nL tot 1 μL. Gezien de lage hoeveelheid DNA in BF is het mogelijk om de hoeveelheid celvrij DNA te verhogen door blastocystvloeistof en kweekmedium te mengen om het slagingspercentage en de consistentie van de detectie te verbeteren. Kuznyetsov et al.23 en Li et al.24 behandelden de zona pellucida met een laser en gaven blastocystvloeistof af in het kweekmedium om de totale hoeveelheid embryonaal DNA te verbeteren, en de amplificatiesnelheid van het gecombineerde medium/BF-monsters na WGA was respectievelijk 100% en 97,5%. Jiao et al.25 verkregen ook een slagingspercentage van 100% door dezelfde methode te gebruiken.
De huidige studie rapporteert een gedetailleerd protocol dat de voorbereiding van gebruikte mediamonsters, NGS-voorbereiding en gegevensanalyse omvat. Door zorgvuldig cumuluscellen uit eicellen te verwijderen, voerde de huidige studie intracytoplasmatische enkelvoudige sperma-injectie (ICSI) en blastocystkweek uit. Het medium van dag 4 - dag 5 / dag 6 werd verzameld voor de voorbereiding van de WGA- en NGS-bibliotheek. Door gebruik te maken van NICS-technologie stroomlijnde de huidige studie de voorbereidingsstappen van de WGA- en NGS-bibliotheek in ongeveer 3 uur en verkreeg het niet-invasieve CCS-resultaten in ongeveer 9 uur.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Ethische toestemming werd verkregen van de ethische commissie van het derde ziekenhuis van de Universiteit van Peking.
1. Voorbereiding
NOTITIE: De benodigde materialen en apparatuur staan vermeld in de materiaaltabel.
- Reagentia
- Voorverwarmen en equilibraat (gebalanceerd) 20-30 μL gametenmedium/bevruchtingsmedium en splijtings-/blastocyststadiumkweekmedium (bedekt met minerale olie) en hyaluronidase (in een goed afgesloten buisje) bij 37 °C, 5% CO 2 en 5% O2 in een Tri-gas incubator 's nachts voor gebruik.
- Verwarm hyaluronidase voor tot 37 °C op een werkblad in een zuurkast.
- Bereid de verglazingsbuffer en de monsterafnamereagentia voor volgens de instructies van de fabrikant.
- Gereedschap
- Bereid monsterafname- en transferpipetten (binnendiameter van ~200 tot 250 μm), denudatie-/stripperpipetten (binnendiameter van ≥150 μm, ~130-140 μm en ~120 μm) en pipetten voor het wassen (binnendiameter van ~150 μm) voor door glazen pasteurpipetten te trekken om vuurgepolijste open fijne punten te genereren.
OPMERKING: De pipetten die worden gebruikt voor het verzamelen/overbrengen van monsters, denudatie en wassen, kunnen direct worden gekocht. De houdnaalden en injectienaalden kunnen ook direct worden gekocht.
- Bereid monsterafname- en transferpipetten (binnendiameter van ~200 tot 250 μm), denudatie-/stripperpipetten (binnendiameter van ≥150 μm, ~130-140 μm en ~120 μm) en pipetten voor het wassen (binnendiameter van ~150 μm) voor door glazen pasteurpipetten te trekken om vuurgepolijste open fijne punten te genereren.
2. Protocol nr. 1: Monsterneming
- Voorbehandeling van oöcyt-corona-cumuluscomplex (OCCC's) vóór vertering met hyaluronidase
- Bereik ovariële stimulatie met zowel follikelstimulerend hormoon (FSH) als humane menopauzale gonadotrofine (hMG) preparaten. Wanneer de loodfollikel >18 mm is, gebruik dan 10.000 IE choriongonadotrofine (hCG) voor de uiteindelijke rijping van de eicel.
- Voer 36 uur na het triggershot het ophalen van de eicel uit. Pak de eicellen op en breng ze over in weefselkweekschaaltjes met 2,5 ml voorverwarmde m-HTF bedekt met minerale olie.
- Breng de OCCC's snel over naar het centrale putje van een orgaankweekschaaltje met 1 ml bevruchtingsmedium met behulp van een transferpipet en incubeer vervolgens met de eicellen bij 37 °C in een incubator van 5% CO 2 en 5 % O 2 gedurende2-4 uur.
- Verbeter OCCC's met hyaluronidase door 1 ml voorverwarmde hyaluronidase van 37 °C (80 IE/ml) toe te voegen aan het centrale putje van een orgaankweekschaaltje met OCCC's (stap 2.1.3). Houd de eindconcentratie hyaluronidase op 40 IE/ml en meng grondig.
- Incubeer de OCCC's gedurende 2 minuten op een thermisch platform van 37 °C. Observeer de veranderingen elke 30 seconden onder een microscoop totdat er nog maar 1-2 lagen granulosacellen overblijven.
- Denudatie van granulosacellen
- Breng de verteerde OCCC's snel over in de kweekschaal voor eicelbehandeling en bedek ze in elk putje met minerale olie.
- Observeer de gescheiden granulosacellen onder een microscoop. Zuig de eicellen voorzichtig op en laat ze 5 keer los om resterende granulosacellen rond de eicellen te verwijderen.
- Herhaal de vorige stap in de resterende 3 putjes om de granulosacellen volledig te verwijderen.
OPMERKING: De bovenstaande stappen (2.1-2.3) kunnen worden uitgevoerd volgens de routinematige werking van elk laboratorium.
- Evaluatie van de eicel
- Evalueer de volledigheid van granulosacelverwijdering met behulp van een microscoop. Als de cellen niet volledig kunnen worden verwijderd, is het behoud van 5 of minder granulosacellen op dit moment acceptabel.
OPMERKING: Als er nog steeds cumuluscellen aan de eicel vastzitten, kan het restant later op dag 3 worden verwijderd voordat het embryo wordt overgebracht van kweekmedium in het splitsingsstadium naar kweekmedium in het blastocyststadium.
- Evalueer de volledigheid van granulosacelverwijdering met behulp van een microscoop. Als de cellen niet volledig kunnen worden verwijderd, is het behoud van 5 of minder granulosacellen op dit moment acceptabel.
- Breng na het uitvoeren van intracytoplasmatische sperma-injectie (ICSI)26 de eicellen over in microdruppels van 20-30 μl splijtingsembryokweekmedium (één eicel komt overeen met één microdruppel) met behulp van transferpipetten en incubeer ze in een incubator van 37 °C, 5% CO 2 en 5 % O2.
- Noteer de dag van ICSI als dag 0. Controleer de embryo's en scoor volgens de Istanbul consensus workshop over embryobeoordeling van dag 1 voor bevruchting (ongeveer 18 uur), dag 2 (ongeveer 45 uur) en dag 3 (ongeveer 68 uur) voor embryosplitsing27.
- Embryo wassen
- Bereid 20-30 μl microdruppels blastocystkweekmedium voor elk embryo bedekt met minerale olie in weefselkweekschaaltjes op dag 2 in een incubator van 37 °C, 5% CO 2 en 5% O2.
- Bereid nog eens drie microdruppels bedekt met minerale olie en label de nieuwe weefselkweekschalen voor het wassen nr. 1-3.
- Breng de embryo's van dag 3 over in de wasmicrodruppels. Zuig de embryo's voorzichtig op en laat ze 3 keer in elke druppel los met behulp van denudatiepipetten.
OPMERKING: Deze procedure kan ook helpen bij het verwijderen van de resterende korrelige cellen die aan het embryo zijn bevestigd. - Observeer en evalueer de embryo's onder een microscoop op dag 3 voordat het medium werd veranderd van een kweekmedium in het splitsingsstadium naar een blastocystkweekmedium voor morfologische scores. Als er nog steeds cumuluscellen aan het embryo vastzaten, pipetteer dan op de juiste manier op en neer in een andere voorverwarmde en gebalanceerde blastocystkweekmediumdruppel bedekt met minerale olie met een stripperpipet totdat de cumuluscellen volledig waren verwijderd.
OPMERKING: Alle aangehechte cumuluscellen moesten op dag 3 volledig worden verwijderd voordat het embryo werd overgebracht van de plaat van het kweekmedium in het splitsingsstadium naar de plaat van het kweekmedium in het blastocyststadium. Eventuele resterende cumuluscellen zullen interfereren met de uiteindelijke analyse en vals-negatieve resultaten geven.
- Twee opties voor het verzamelen van kweekmedia
OPMERKING: Het IVF-centrum kan kiezen uit een van de twee methoden voor het verzamelen van kweekmedia op basis van de middelen, eisen en voorkeuren van het centrum.- Optie 1: Embryowassen en kweken
OPMERKING: Deze optie is voor IVF-laboratoria die vitrificatie uitvoeren op de ochtend van dag 5.- Breng het embryo over in voorverwarmde (37 °C) microdruppels kweekmedium en was elk embryo voorzichtig serieel in 3 microdruppels door te pipetteren op dag 4 's middags.
- Breng elk embryo over in een unieke voorverwarmde (37 °C) enkele microdruppel kweekmedium voor monsterafname. Het volume van een enkele druppel kweekmedium mag niet groter zijn dan 25 μL.
- Voer blastocyst-embryokweek uit op dag 5/dag 6 bij 37 °C, 5% CO 2 en 5% O2.
- Optie 2: Embryo wassen en kweken
OPMERKING: Deze optie is voor IVF-laboratoria die vitrificatie uitvoeren op dag 5 middag of dag 6.- Breng het embryo over in voorverwarmde (37 °C) microdruppels van 10-15 μL kweekmedium en was elk embryo voorzichtig serieel in 3 microdruppels door op dag 5 te pipetteren.
- Breng elk embryo over in een unieke voorverwarmde (37 °C) enkele microdruppel kweekmedium voor monsterafname. Het volume van een enkele druppel kweekmedium mag niet groter zijn dan 15 μL.
- Voer blastocyst-embryokweek uit op dag 5/dag 6 bij 37 °C en 5% CO2.
- Optie 1: Embryowassen en kweken
- Sample Collectie
- Pas de ICM voorzichtig aan op een aanzienlijke afstand van het beoogde punt van de laserstraal, die zich richt op de celovergang van het trophectoderm om een klein gaatje in het trophectoderm te genereren om de vloeistof uit de blastocoelholte vrij te maken. Vervolgens worden de embryo's verplaatst naar een invriesoplossing voor cryopreservatie volgens het conventionele proces.
- Breng kweekmedium van elk gekweekt embryo over in een RNase/DNase-vrije PCR-buis met 5 μL cellysebuffer.
- Verzamel dezelfde hoeveelheid kweekmedium zonder te worden gebruikt voor embryokweek als negatieve controle. Vries alle verzamelde monsters onmiddellijk in vloeibare stikstof in en bewaar ze vervolgens bij −80 °C nadat ze zijn verzameld totdat ze aan de NICS-test worden onderworpen.
- Voer vitrificatie uit zoals beschreven in het protocol.
3. Protocol 2: Bouw van bibliotheken
- Kweek medium lysis
- Verdun 1 μL positieve controle (10 ng humaan gDNA) met 199 μL vers kweekmedium. Meng grondig en centrifugeer de buis kort (200 x g gedurende 5 s).
- Breng 10 μL van dag 5-dag 6 blastocystkweekmedium, verdunde positieve controle en vers kweekmedium over naar nieuwe PCR-buisjes van 0,2 ml.
- Voeg 1 μL MT Enzyme Mix toe aan elke PCR-buis en meng grondig door te pipetteren en centrifugeer onmiddellijk gedurende 2-3 s bij 200 x g.
- Plaats de PCR-buis(jes) uit stap 3.1.3 in een voorverwarmd NICS-monstervoorbereidingsstation en voer het lysisprogramma als volgt uit: 10 min bij 75 °C; 4 min bij 95 °C; houden bij 22 °C.
OPMERKING: Het monstervoorbereidingsstation is vergelijkbaar met een standaard PCR-machine.- Klik op het Lysis-pictogram om het installatiescherm te openen.
- Selecteer Tube voor Control-modus ; ingang 10 μL voor monstervolume; selecteer Aan voor Hotlid-regeling en voer 105 °C in voor de temperatuur. Selecteer Nee voor Pauzeren bij de eerste seg. Klik op OK om verder te gaan.
- Wacht tot Remain-tijd --:-- :-- aangeeft, wat het einde van het programma aangeeft, en klik vervolgens op Stop om het programma te beëindigen.
- Stop het programma nadat het proces is voltooid. Ga direct door naar de volgende stap.
- Pre-bibliotheek voorbereiding
- Ontdooi de Pre-Lib Buffer tot RT. Meng grondig door te pipetteren en centrifugeer onmiddellijk gedurende 2-3 s op 200 x g.
- Bereid als volgt een mastermix voor prelibrary-reactie: voeg 2 μL Pre-Lib Enzyme Mix toe aan 60 μL Pre-Lib Buffer, meng de reactie grondig en centrifugeer kort.
- Voeg 60 μL prelibrary-reactiemengsel toe aan elk voorbehandeld mediummonster uit de vorige stap. Meng grondig door te pipetteren en centrifugeer onmiddellijk gedurende 2-3 s op 200 x g.
- Plaats de PCR-buis(jes) uit stap 3.2.3 in het Sample Prep Station en voer het prelibrary-programma als volgt uit: 95 °C gedurende 2 min; 12 cycli van 15 °C gedurende 40 s, 22 °C gedurende 40 s, 33 °C gedurende 30 s, 65 °C gedurende 30 s, 72 °C gedurende 40 s, 95 °C gedurende 10 s en 63 °C gedurende 10 s; en houd op 4 °C.
- Klik op het pictogram Pre_Lib om het installatiescherm te openen.
- Selecteer Tube voor Control-modus ; ingang 70 μL voor monstervolume; selecteer Aan voor Heet deksel en voer 105 °C in voor de temperatuur. Selecteer Nee voor Pauzeren bij de eerste seg. Klik op OK om verder te gaan.
- Wacht tot Remain-tijd --:-- :-- aangeeft, wat het einde van het programma aangeeft, en klik op Stop om het programma te beëindigen.
- Stop het programma wanneer het proces is voltooid. Ga direct door naar de volgende stap.
- Voorbereiding van de bibliotheek
- Ontdooi de Library Buffer tot RT. Meng grondig door te pipetteren en centrifugeer onmiddellijk gedurende 2-3 s op 200 x g.
- Bereid als volgt een mastermix voor bibliotheekreactie: voeg 1,6 μL Library Enzyme Mix toe aan 60 μL Library Buffer, meng de reactie grondig en centrifugeer kort.
- Voeg 60 μL bibliotheekreactiemix en 2 μL Barcode Primer toe aan elk prelibrary-product uit stap 3.2.3. Meng de reactie grondig en centrifugeer kort.
- Plaats de PCR-buis(jes) uit stap 3.2.3 in de thermische cycler en voer het bibliotheekvoorbereidingsprogramma als volgt uit: 94 °C gedurende 30 s; 17 cycli van 94 °C gedurende 25 s, 62 °C gedurende 30 s en 72 °C gedurende 45 s); en vervolgens vasthouden bij 4 °C.
- Klik op het pictogram Lib_Prep om het installatiescherm te openen.
- Selecteer Tube voor Control-modus ; ingang 130 μL voor monstervolume; selecteer Aan voor Hotlid-regeling en voer 105 °C in voor de bijbehorende temperatuur. Selecteer Nee voor Pauzeren bij de eerste seg. Klik op OK om verder te gaan.
- Wacht tot Remain-tijd --:-- :-- aangeeft, wat het einde van het programma aangeeft, en klik op Stop om het programma te beëindigen.
- Bibliotheek zuivering
- Haal Magbeads uit de opslag bij 2-8 °C gedurende ten minste 20 minuten vóór de zuiveringsstap. Draai en meng de Magbeads gedurende 20 s. Doseer voldoende korrels voor de zuiveringsstap in een nieuwe microcentrifugebuis van 1,5 ml en verwarm de korrels tot RT.
- Voeg 1x Magbeads toe aan elke bibliotheek. Mix door ≥10 keer op en neer te pipetteren en 5 minuten op RT te incuberen.
OPMERKING: Voeg bijvoorbeeld 100 μL Magbeads toe aan 100 μL bibliotheekmonster. - Centrifugeer de buis na incubatie kort en plaats deze op een magnetische standaard.
- Wacht ongeveer 5 minuten tot de oplossing helder wordt. Terwijl u de buis op de magnetische standaard houdt, zuigt u de oplossing voorzichtig op en gooit u deze weg.
- Voeg 200 μL vers bereide 80% ethanol toe aan de tube. Incubeer gedurende 30 s bij RT en verwijder voorzichtig het supernatant. Herhaal dit nog een keer.
- Verwijder de ethanol zo volledig mogelijk. Laat de kralen ongeveer 5-10 minuten aan de lucht drogen op de magnetische standaard bij RT.
- Verwijder de buis van de magnetische standaard, voeg 17.5 μL elutiebuffer toe en draai de buis om de kralen opnieuw te suspenderen. Centrifugeer de buis kort en incubeer gedurende 5 minuten bij RT.
- Plaats de buis op de magnetische standaard en wacht tot de oplossing helder wordt. Breng voorzichtig 15 μL supernatans over in een nieuw buisje.
- Kwantificering van de bibliotheek
- Kwantificeer gezuiverde bibliotheken met behulp van de fluorometer volgens de gebruikershandleiding van de qubit dsDNA HS Assay kits28. De opbrengst van de bibliotheken varieert van ~15 tot 300 ng.
- Bundeling van bibliotheken
- Gebruik 10 nanogram van elk bibliotheekmonster voor pooling.
- Sequencing
- Raadpleeg de gebruikershandleiding voor sequencing (15027617 v01)29.
- Gezuiverde bibliotheeksequenties van 50 bp aan een enkel uiteinde op het platform leverden ongeveer 2 miljoen lezingen op voor elk monster, en een sequentiediepte van 0,03 × werd aanbevolen.
- Data-analyse
- Voer de naam en het wachtwoord van de gebruiker in op de inlogpagina
- Nadat u bent ingelogd op het systeem, klikt u op Analyse en er verschijnt een nieuwe pagina. Klik op Inzending maken onder het tabblad NICS-A. Kies vervolgens NGS voor het platform, selecteer corporatie, kies NICSInst voor het reagens, voer de projectinformatie in het vak onder Project-ID in, stel de analysevoorkeuren in en upload de bestanden. Zodra alle sequentiebestanden zijn geüpload, klikt u op Verzenden om de analyse te starten (Afbeelding 3A).
- Klik op Inzendingen bekijken om de lijst met ingediende projecten weer te geven. Zodra de analyse is voltooid, wordt de status van een project Voltooid en verschijnt er een knop Weergeven in het rapportveld. Klik op de knop Weergeven om de overzichtstabel van de NICS-analyse te bekijken (Figuur 3B).
- Klik op de knop Rapport exporteren om de rapporten op te slaan (Figuur 3C).
OPMERKING: Voor elke analyse worden drie soorten bestanden geëxporteerd. Een grafisch bestand met alle copy number variation (CNV)-plots voor elk chromosoom en het hele genoom, dat wordt opgeslagen in de map "grafiek"; een spreadsheet met de voorbeeld-QC-details van deze analyserun; een documentbestand dat de door de gebruiker aangepaste NICS-rapporten bevat; en een spreadsheet met de overzichtsgegevens van deze analyse.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
De huidige studie paste de voorgestelde methode toe op een patiënt. IRB-goedkeuring en geïnformeerde toestemming werden verkregen vóór de toepassing van NICS-analyse. De huidige studie verkreeg 6 blastocysten van patiënten en voerde NICS uit op alle 6 embryo's op dag 4 tot dag 5 medium. Chromosoomafwijkingen veroorzaakt door de gebalanceerde translocatie van de ouders werden gedetecteerd in vijf chromosomen met de NICS-test; daarom konden ze niet worden gebruikt voor overdracht (figuur 4A-E). De NICS-resultaten van de twee embryo's toonden hetzelfde karyotype 45, en XN en -18 (×1) waren beide chromosoom 18-deleties (Figuur 4A, B). Het karyotype 46, XN, -1p (pter→p21.1, ×1) is slechts de korte arm van de chromosoom 1 pter→p21.1 regio deletie (Figuur 4D).
De NICS-resultaten toonden karyotype 46, XN, +1p (pter→p21.2, ×3) en -18 (q21.32→qter, ×1), wat aangaf dat zowel de deletie van de lange arm van chromosoom 18 q21.32→qter als de korte arm van chromosoom 1 pter→p21.2-regio werden gedupliceerd (Figuur 4E). Hoewel karyotypen 46, XN, +5q (×4) en -8 (×1, mos) chromosoom 5-duplicaties zijn en 8 mozaïekverschillen vertonen, kan de NICS-test alle 24 chromosomen screenen op aneuploïdie. Dit proces biedt een nieuwe methode voor het overbrengen van enkelvoudige normale karyotype blastocysten.

Figuur 1. De volledigheid van de verwijdering van cumuluscellen. (A) De eicellen met cumuluscellen. (B) De eicellen zonder aangehechte cumuluscellen. Klik hier om een grotere versie van deze figuur te bekijken.

Figuur 2. Cumuluscellen worden op D3 uit een embryo verwijderd voordat ze worden overgebracht naar de BM. Alle aangehechte cumuluscellen moeten worden verwijderd voordat het medium verandert van de initiële plaat van het splitsingsembryokweekmedium naar de blastocystkweekmediumplaat, die zich op dag 3 bevindt nadat de embryo's het 8-cellige stadium hebben bereikt. Cumuluscellen die niet worden verwijderd, zullen interfereren met de uiteindelijke analyse en vals-negatieve resultaten opleveren. Klik hier om een grotere versie van deze figuur te bekijken.

Figuur 3. Data-analyse. (A) Er zijn verschillende opties voor de gebruikerstoepassing. Voor sequencing platform corporation kunnen gebruikers kiezen voor Illumina, Ion Torrent of MGI. De gebruikers kunnen kiezen of de geslachtsinformatie wordt gerapporteerd. Als u klaar bent met de bovenstaande parameterinstelling, klikt u op het vakje onder Bestand uploaden en kiest u de juiste volgorde van de bestanden die u wilt uploaden. Kies voor Illumina de bestanden met de extensie fastq.gz. Klik op Verzenden om de analyse te starten nadat deze is geüpload. (B) De weergave van de overzichtstabel. De overzichtstabel bestaat uit de volgende informatie: Voorbeeldnaam: De naam van elk NICS-monster wordt vermeld; Gegevens QC: Geeft aan of het sequentiebestand de QC voor NICS-analyse doorstaat; AI-classificatie: de classificatie (A, B of C) voor elk NICS-monster; AI_Rating Interpretatie: Evaluatie van het implantatiepotentieel van embryo's; AI-beoordeling: de score voor elke NICS-steekproef; CNV-plot (Whole Genome): Bekijk de CNV-profielen van alle chromosomen; (c) De pagina Rapport opslaan. Klik op de knop Rapport exporteren naast de samenvatting van de resultaten. Selecteer de informatie die u in het eindrapport wilt weergeven en klik op Exporteren. De rapporten worden opgeslagen in de downloadmap van uw computer. Klik hier om een grotere versie van deze figuur te bekijken.

Figuur 4. Embryoscreening en -selectie met behulp van NICS van een patiënt. In totaal ontwikkelden zes embryo's zich met succes tot het blastocyststadium, en van elk embryo werd Day4-Day5-kweekmedium verzameld voor de NICS-test. (A) en (B) zijn de NICS-resultaten van de twee blastocystembryo's die hetzelfde karyotype 45, XN, -18(×1) beide chromosoom 18-deletie lieten zien. (C) toonde karyotype 46, XN, +5q (×4), -8(×1, mos) is chromosoom 5 duplicatie en 8 mozaïek. (D) toonde karyotype 46, XN, -1p (pter→p21.1, ×1) is alleen de korte arm van chromosoom 1 pter→p21.1 regiodeletie, terwijl (E) karyotype 46, XN, +1p (pter→p21.2, ×3), -18(q21.32→qter, ×1) is de korte arm van chromosoom 1 pter→p21.2-regioduplicatie en de lange arm van chromosoom 18 q21.32 → qter-regio (F) vertoonde een evenwichtige chromosomale samenstelling. De x-as betekent 22 autosomen in rood en blauw, de y-as geeft het kopienummer van elk autosoom aan. De grijze stippen zijn de liniaalschaal van het kopienummer, de respons van elk magazijnvenster en het normale karyotype van het kopienummer moet 2 zijn. Klik hier om een grotere versie van deze figuur te bekijken.
Tabel S1. De slagingspercentages van DNA-detectie Optie 1 en Optie 2. Klik hier om deze tabel te downloaden.
Tabel S2. De concordantie tussen NICS en PGT-A in verschillende opties. Klik hier om deze tabel te downloaden.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Wijzigingen en probleemoplossing
Als de NICS-resultaten besmet zijn met ouderlijk genetisch materiaal, zorg er dan voor dat alle cumulus-corona radiata-cellen worden verwijderd en zorg ervoor dat ICSI wordt uitgevoerd voor bevruchting. Onjuiste mediumopslag of sjabloonvoorbereidingsprocessen worden vermeden, waardoor DNA kan worden afgebroken. De werkruimte werd grondig gezuiverd met DNase- en RNase-decontaminatiereagentia. Om besmetting door andere embryo's te voorkomen, werd vanaf dag 4 altijd één embryo gekweekt in een enkele druppel medium om kruisbesmetting te voorkomen. Het fenomeen van besmetting wordt geminimaliseerd bij het uitstellen van de plaatsing van embryo's in de uiteindelijke kweekdruppel30,31,32,33. Om besmetting van de moeder tot een minimum te beperken, wijzigde Kuznyetsov34 de embryokweekprocedures van dag 0 tot dag 4, inclusief het zorgvuldig verwijderen van resterende coronacellen door pipetteren en spoelen.
Lane et al.30 laat zien dat bij het nemen van het embryokweekmedium van dag 4 tot dag 5, de nauwkeurigheid van euploïdiedetectie wordt verbeterd, de consistentie van de embryoploïdie meer dan 95% is en de consistentie van het geslachtschromosoom 100% bereikt. Lledo et al.33 ontdekten dat het coïncidentiepercentage tussen het kweekmedium van dag 3 en dag 5 en TE-monsters 74,6% en 92,0% was wanneer embryo's werden gekweekt van dag 4 tot dag 6.
Ook onze interne gegevens ondersteunen deze conclusie, zoals blijkt uit tabel S1. Vergeleken met de conventionele kweekmethode op dag 3 en dag 5, werden de granulosacellen verder verwijderd als gevolg van nog een verandering in kweekmedium op dag 4 of dag 5. We verstrekken interne gegevens (tabel S1) waaruit blijkt dat onze twee methoden (optie 1 en optie 2) een goede consistentie hebben in vergelijking met PGT-A, wat beter is dan de bemonsteringsmethode zonder de grondige verwijdering van CC.
IF-geamplificeerde producten verschenen in de negatieve controle, en externe DNA-materialen kunnen het reagens of de werkruimte hebben besmet. De werkruimte moet worden schoongemaakt door DNA/RNA-verwijderende reagentia, nucleasevrije materialen moeten worden gebruikt en de reagentia moeten na het eerste gebruik worden gealiquoteerd.
Verschillen in de slagingspercentages tussen optie 1 en optie 2 worden besproken in tabel S1 en tabel S2.
Beperkingen van de NICS-test
Er zijn twee belangrijke beperkingen van NICS. 1) Vóór ICSI moeten alle cumuluscellen (meestal van maternale oorsprong, meestal normale chromosoomsamenstelling) worden verwijderd. Als de verwijdering onvolledig is, kunnen de cumuluscellen DNA afgeven tijdens de ontwikkeling van het embryo en wordt het externe DNA geamplificeerd, wat de oorzaak kan zijn van vals-negatieve detectie. 2) Het is moeilijk om het sperma dat aan de zona pellucida is bevestigd te verwijderen, en de NICS-procedure wordt ten zeerste aanbevolen om met ICSI te worden uitgevoerd. Hoewel de regelmatige vervanging van splijtingsmedia op dag 3 de kans op besmetting door cumuluscellen en overtollig sperma kan verminderen, moet deze besmetting tot een minimum worden beperkt als NICS wordt gebruikt bij klinische IVF. Er is echter een methode ontwikkeld voor het detecteren van NICS in IVF-embryo's, inclusief de functie van het herkennen van exogeen DNA, die in de nabije toekomst zal worden gedemonstreerd.
Deze studie vergeleek de verschillen tussen verschillende media niet, aangezien grootschalige klinische onderzoeken kweekmedia hebben vergeleken. Acht centra gebruikten 4 verschillende kweekmedia, sequentieel en continu, en 2 verschillende percentages albuminesuppletie (5% en 10%), en deze verschillen hadden geen significante effecten op de nauwkeurigheid van embryonale cfDNA-resultaten31. Deze bevindingen ondersteunen de mogelijke toepasbaarheid van embryonale cfDNA-analyse op elk IVF-laboratorium bij het werken volgens het specifieke protocol.
Betekenis ten opzichte van bestaande methoden
De NICS-methode vermijdt embryobiopsie en verbetert zo de gebruiksveiligheid aanzienlijk. Vergeleken met blastocysten is NICS een eenvoudige, tijdbesparende, gevoelige en reproduceerbare pre-implantatiescreeningstechniek die geschikt is voor geassisteerde voortplantingspopulaties met een hoge kans op aneuploïdie. In tegenstelling tot invasieve biopsie, die aanzienlijke en professionele kennis vereist voor de blastocystbiopsieprocedure, kan NICS op grote schaal worden toegepast omdat de eenvoudige verzameling van verbruikt medium alleen de reguliere werking van IVF19 volgt en in sommige landen geen PGS/PGD-kwalificatie vereist.
Toekomstige toepassingen
NICS heeft het potentieel voor brede toepasbaarheid voor chromosoomscreening bij klinische IVF, niet alleen voor ICSI maar ook voor IVF-embryo's. Hoewel ICSI ten zeerste wordt aanbevolen, zijn methoden voor het verwijderen van het sperma dat aan de zona pellucida is gehecht vereist om de invloed van sperma te voorkomen.
Morfologische beoordeling is een traditionele methode voor embryo-evaluatie, maar in de meeste gevallen kunnen chromosomaal abnormale embryo's morfologisch vergelijkbaar lijken met chromosomaal normale (euploïde) embryo's. Het combineren van morfologische beoordeling met de NICS-test bij het overbrengen van ploïde embryo's met een goede morfologie in de baarmoeder zou het aantal doorgaande zwangerschappen en het aantal levendgeborenen kunnen verbeteren. Er zal een gerandomiseerde klinische studie worden uitgevoerd om de klinische werkzaamheid van de overdracht van één embryo met behulp van NICS-technologie te evalueren.
Kritieke stappen in het protocol
Alle cumulus-corona radiata-cellen moeten vóór de bevruchting uit de eicellen worden verwijderd. Eicellen werden bevrucht door intracytoplasmatische sperma-injectie (ICSI). Het toevoegen van van mensen afkomstige eiwitten/supplementen aan het kweekmedium werd vermeden. Het kweekmedium werd op dag 4 veranderd en verzameld op dag 5-dag 6 toen de blastocysten volledig uitzetten. Embryo's werden gekweekt in individuele druppeltjes kweekmedium vanaf dag 4. Bij het verzamelen van het kweekmedium werden de transferpipetten tussen de monsters verwisseld om besmetting te voorkomen.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yaxin Yao, Jieliang Ma, Jing Wang en Sijia Lu zijn werknemers van Yikon Genomics Co., Ltd.
Acknowledgments
De auteurs willen Shiping Bo en Shujie Ma bedanken voor hun hulp bij de analyse van NGS-gegevens. Financiering: dit werk werd ondersteund door het National Key Research and Development Program (subsidienr. 2018YFC1003100).
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
References
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Jr Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T. Jr, Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid? Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- Thermo Fisher Scientific. Qubit dsDNA HS Assay Kit. , Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015).
- Miseq system use guide. , Available from: https://support.illumina.com/downloads/miseq_system user _ guide 15027617.html (2016).
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Tags
Chromosoomscreening Menselijke pre-implantatie-embryo's Gebruikt kweekmedium Monsterafname Chromosomale ploïdieanalyse In-vitrofertilisatie (IVF) Pre-implantatie Genetische Testen voor Aneuploïdie (PGT-A) Trophectoderm (TE) Binnencelmassa (ICM) Valse positieven en negatieven NICS-technologie DNA-sequencing Kweekmediumbemonstering Whole Genome Amplification (WGA) Bibliotheekvoorbereiding NGS-gegevensanalyse Cryopreservatie IVF-laboratoriumErratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021.
Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
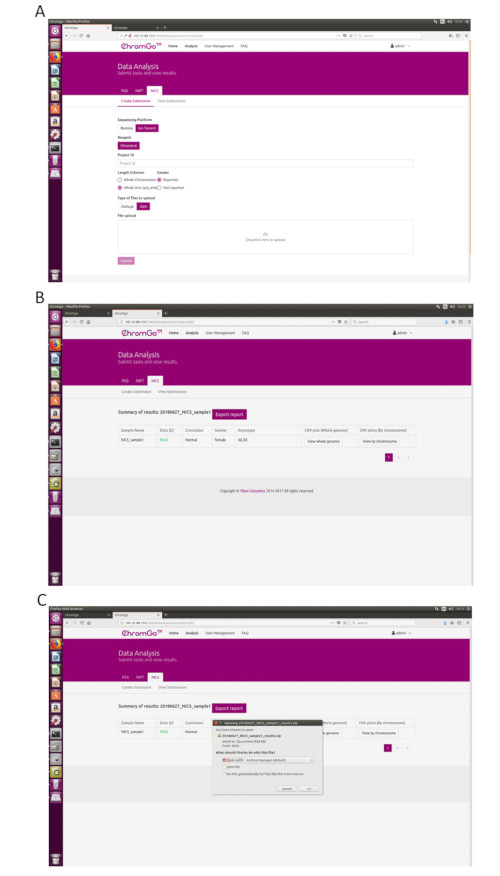
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
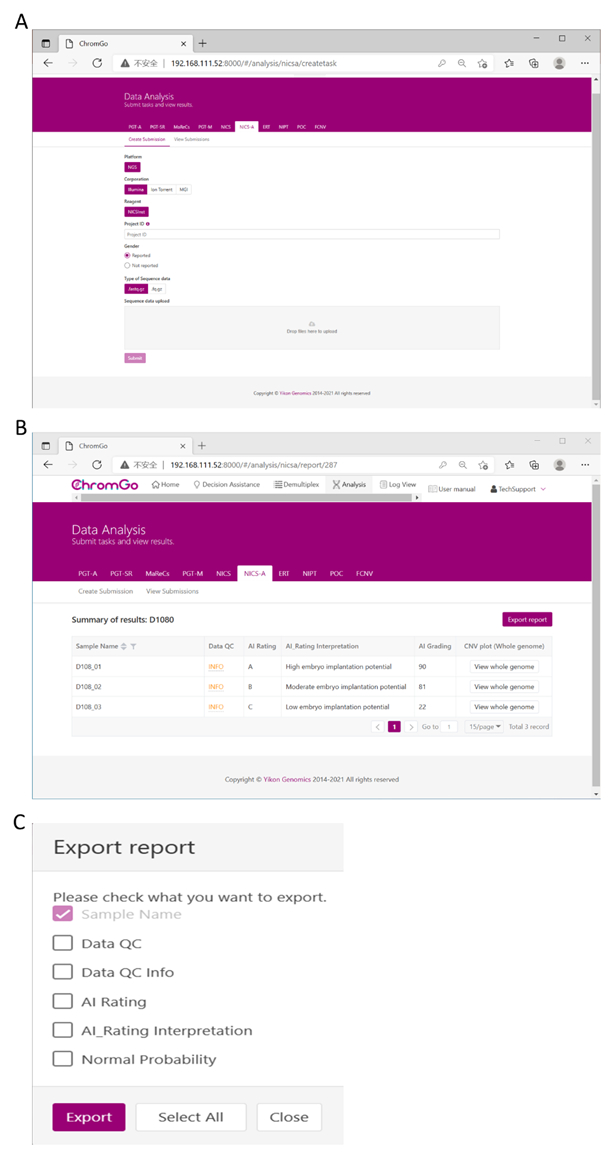
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.



