ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Denne studien rapporterer en protokoll for kromosomscreening av humane embryoer som bruker brukt kulturmedium, som unngår embryobiopsi og muliggjør kromosomploidiidentifikasjon ved hjelp av NGS. Denne artikkelen presenterer den detaljerte prosedyren, inkludert utarbeidelse av kulturmedium, hele genomamplifikasjon (WGA), neste generasjons sekvensering (NGS) bibliotekforberedelse og dataanalyse.
Abstract
Ved klinisk in vitro-fertilisering (IVF) krever den rådende metoden for PGT-A biopsi av noen få celler fra trophektoderm (TE). Dette er linjen som danner morkaken. Denne metoden krever imidlertid spesialiserte ferdigheter, er invasiv og lider av falske positive og negative fordi kromosomtallene i TE og den indre cellemassen (ICM), som utvikler seg til fosteret, ikke alltid er de samme. NICS, en teknologi som krever sekvensering av DNA som slippes ut i kulturmediet fra både TE og ICM, kan tilby en vei ut til disse problemene, men har tidligere vist seg å ha begrenset effekt. Den nåværende studien rapporterer hele protokollen til NICS, som inkluderer prøvetakingsmetoder for kulturmedium, helgenomamplifisering (WGA) og bibliotekforberedelse, og NGS-dataanalyse ved analyseprogramvare. Tatt i betraktning de forskjellige kryokonserveringstidene i forskjellige embryolaboratorier, har embryologer to metoder for å samle embryokulturmedium som kan velges i henhold til de faktiske forholdene i IVF-laboratoriet.
Introduction
Assistert befruktning (ART) har i økende grad blitt brukt til behandling av infertilitet. Imidlertid har suksessraten for ART, som IVF, vært begrenset, og graviditetstapsraten er betydelig høyere enn for den normale befolkningen1. Hovedårsaken til disse problemene er kromosomale abnormiteter, som vanligvis eksisterer i preimplantasjon menneskelige embryoer2. PGT-A er en effektiv metode for screening av embryoer for kromosombalanse før implantasjon 3,4. Noen studier har vist at PGT-A kan redusere abortfrekvensen og forbedre graviditetsraten 5,6,7,8. PGT-A krever imidlertid kompleks teknisk ekspertise som krever spesifikk trening og erfaring. Den invasive embryobiopsiprosedyren kan også potensielt forårsake skade på embryoene9. Studier har vist at blastomerbiopsi kan hindre senere utvikling, og antall biopsierte TEs kan påvirke implantasjonsratene10. Selv om det langsiktige biosikkerhetsproblemet med embryobiopsi ennå ikke har blitt evaluert grundig hos mennesker, har dyreforsøk vist sin negative innflytelse på embryoutvikling11,12,13.
Tidligere rapporter indikerte at spormengder av DNA-materialer ble utskilt i kulturmediet under embryoutvikling, og det har blitt gjort forsøk på å utføre omfattende kromosomscreening (CCS) ved bruk av brukt embryokulturmedium 14,15,16,17,18. Deteksjonsratene og nøyaktigheten av testene har imidlertid ikke oppfylt kravene til omfattende klinisk bruk. Denne studien rapporterte en forbedring i NICS-analysen for å øke deteksjonsraten samt nøyaktigheten av NICS-testen19. De siste årene har blastocoelevæske (BF) blitt studert som en analytisk prøve av minimalt invasiv PGT-A. Imidlertid varierer andelen vellykket genom-bred amplifisering og detekterbart DNA i blastocystvæskeprøver fra 34,8% til 82%20,21,22. Volumet av BF rapportert i ulike studier varierer fra 0,3 nL til 1 μL. I lys av den lave mengden DNA i BF, er det mulig å øke mengden cellefritt DNA ved å blande blastocystvæske og kulturmedium for å forbedre suksessraten og konsistensen av deteksjon. Kuznyetsov et al.23 og Li et al.24 behandlet zona pellucida med en laser og frigjorde blastocystvæske i kulturmediet for å forbedre den totale mengden embryonalt DNA, og amplifikasjonshastigheten til de kombinerte medium / BF-prøvene etter WGA var henholdsvis 100% og 97,5%. Jiao et al.25 oppnådde også en suksessrate på 100% forsterkning ved å bruke samme metode.
Den foreliggende studien rapporterer en detaljert protokoll som inkluderer forberedelse av brukte medieprøver, NGS-forberedelse og dataanalyse. Ved forsiktig å fjerne cumulusceller fra oocytter, utførte denne studien intracytoplasmatisk enkeltspermieinjeksjon (ICSI) og blastocystkultur. Dag 4-dagers 5/dag 6 brukt medium ble samlet inn for WGA og NGS bibliotek forberedelse. Ved å bruke NICS-teknologi strømlinjeformet denne studien WGA- og NGS-bibliotekforberedelsestrinnene på omtrent 3 timer og oppnådde CCS-resultater ikke-invasivt på omtrent 9 timer.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Etisk tillatelse ble innhentet fra etikkomiteen ved Peking University Third Hospital.
1. Forberedelse
MERK: De nødvendige materialene og utstyret er oppført i Materialfortegnelse.
- Reagenser
- Forvarm og bespar (balansert) 20-30 μL kjønnscellemedium/befruktningsmedium og spaltning/blastocyst-stadium kulturmedium (dekket med mineralolje) og hyaluronidase (i tett lukket rør) ved 37 °C, 5% CO 2 og 5% O2 i en Tri-gass inkubator over natten før bruk.
- Forvarm hyaluronidase til 37 °C på en arbeidsflate i en avtrekkshette.
- Forbered vitrifikasjonsbuffer og prøveinnsamlingsreagenser i henhold til produsentens instruksjoner.
- Redskapene
- Forbered prøveinnsamling og overføringspipetter (indre diameter på ~200 til 250 μm), denudasjons-/stripperpipetter (indre diameter på ≥150 μm, ~130-140 μm og ~120 μm) og pipetter for vask (indre diameter på ~150 μm) ved å trekke Pasteur-pipetter i glass for å generere brannpolerte åpne fine spisser.
MERK: Pipetter som brukes til prøveinnsamling/overføring, denudasjon og vasking kan kjøpes direkte. Holdekanylene og kanylene kan også kjøpes direkte.
- Forbered prøveinnsamling og overføringspipetter (indre diameter på ~200 til 250 μm), denudasjons-/stripperpipetter (indre diameter på ≥150 μm, ~130-140 μm og ~120 μm) og pipetter for vask (indre diameter på ~150 μm) ved å trekke Pasteur-pipetter i glass for å generere brannpolerte åpne fine spisser.
2. Protokoll 1: Prøveinnsamling
- Forbehandling av oocytt-corona-cumuluskompleks (OCCC) før fordøyelse med hyaluronidase
- Oppnå ovarial stimulering med både follikkelstimulerende hormon (FSH) og humant menopausalt gonadotropin (hMG) preparater. Når blyfollikkelen er >18 mm, bruk 10.000 IE koriongonadotropin (hCG) for endelig oocyttmodning.
- Utfør oocythenting 36 timer etter utløserskudd. Plukk opp og overfør oocytter til vevskulturretter med 2,5 ml forvarmet m-HTF dekket av mineralolje.
- Overfør raskt OCCCene til den sentrale brønnen i en organkulturskål som inneholder 1 ml befruktningsmedium ved hjelp av en overføringspipette og inkuber deretter med oocyttene ved 37 ° C i en 5% CO 2 og 5% O 2 inkubator i2-4 timer.
- Fordøy OCCC med hyaluronidase ved å tilsette 1 ml 37 °C forvarmet hyaluronidase (80 IE/ml) til den sentrale brønnen i en organkulturskål som inneholder OCCC (trinn 2.1.3). Hold den endelige konsentrasjonen av hyaluronidase på 40 IE/ml og bland godt.
- Inkuber OCCCene på en 37 °C termisk plattform i 2 minutter. Observer endringene under et mikroskop hver 30. s til bare 1-2 lag granuloseceller gjensto.
- Denudasjon av granulosa celler
- Overfør raskt de fordøyde OCCCene i dyrkningsskålen for oocytthåndtering og dekk med mineralolje i hver brønn.
- Observer de separerte granulosacellene under et mikroskop. Aspirer forsiktig og slipp oocyttene 5 ganger for å fjerne gjenværende granulosa-celler rundt oocytter.
- Gjenta forrige trinn i de resterende 3 brønnene for å fjerne granulosacellene helt.
MERK: Trinnene ovenfor (2.1-2.3) kan utføres i henhold til rutineoperasjonen til hvert laboratorium.
- Evaluering av oocytten
- Evaluer fullstendigheten av granulosacellefjerning ved hjelp av et mikroskop. Hvis cellene ikke kunne fjernes helt, er retensjonen av 5 eller færre granulosa-celler akseptabel på dette tidspunktet.
MERK: Hvis cumulusceller fortsatt er festet til oocytten, kan resten fjernes senere på dag 3 før embryoet overføres fra spaltningsstadiumkulturmedium til blastocyst-stadium kulturmedium.
- Evaluer fullstendigheten av granulosacellefjerning ved hjelp av et mikroskop. Hvis cellene ikke kunne fjernes helt, er retensjonen av 5 eller færre granulosa-celler akseptabel på dette tidspunktet.
- Etter å ha utført intracytoplasmatisk spermieinjeksjon (ICSI)26, overfør oocyttene til 20-30 μL spaltning av embryokulturmedium mikrodråper (en oocytt tilsvarer en mikrodråpe) ved hjelp av overføringspipetter og inkubere i en 37 ° C, 5% CO 2 og 5% O2 inkubator.
- Registrer dagen for ICSI som dag 0. Sjekk embryoene og score i henhold til Istanbul-konsensusverkstedet om embryovurdering på dag 1 for befruktning (ca. 18 timer), dag 2 (ca. 45 timer) og dag 3 (ca. 68 timer) for embryospaltning27.
- Embryo vask
- Forbered 20-30 μL blastocystkulturmediummikrodråper for hvert embryo dekket med mineralolje i vevskulturretter på dag 2 i en 37 ° C, 5% CO 2 og 5% O2 inkubator.
- Forbered ytterligere tre mikrodråper dekket med mineralolje, og merk de nye vevskulturfatene for vasking nr. 1-3.
- Overfør dag 3 embryoer til vaskemikrodråpene. Aspirer forsiktig og frigjør embryoene 3 ganger i hver dråpe ved hjelp av denudasjonspipetter.
MERK: Denne prosedyren kan også bidra til å fjerne de gjenværende granulære cellene som er festet til embryoet. - Observer og evaluer embryoene under et mikroskop på dag 3 før mediet ble endret fra spaltningsstadiumkulturmedium til blastocystkulturmedium for morfologisk skåring. Hvis cumulusceller fortsatt var festet til embryoet, pipetterte du riktig opp og ned i en annen forvarmet og likevektet blastocystkulturmediumdråpe dekket med mineralolje med en stripperpipette til cumuluscellene ble fullstendig fjernet.
MERK: Alle tilknyttede cumulusceller måtte fjernes helt på dag 3 før embryoet ble overført fra spaltningsstadiet kulturmediumplate til blastocyst-stadium kultur mediumplate. Eventuelle gjenværende cumulusceller vil forstyrre den endelige analysen og gi falske negative resultater.
- To alternativer for innsamling av kulturmedium
MERK: IVF-senteret kan velge mellom en av to metoder for innsamling av kulturmedium basert på senterets ressurser, krav og preferanser.- Alternativ 1: Embryovask og dyrkning
MERK: Dette alternativet er for IVF-laboratorier som utfører vitrifisering om morgenen på dag 5.- Overfør embryoet til forvarmede (37 °C) mikrodråper av dyrkningsmedium, og vask hvert embryo forsiktig serielt i 3 mikrodråper ved pipettering på dag 4 ettermiddag.
- Overfør hvert embryo til en unik forvarmet (37 ° C) enkelt mikrodråpe av kulturmedium for prøvetaking. Volumet av en enkelt dråpe kulturmedium kan ikke overstige 25 μL.
- Utfør blastocystembryokultur på dag 5/dag 6 ved 37 °C, 5 % CO2 og 5 %O2.
- Alternativ 2: Embryovask og dyrkning
MERK: Dette alternativet er for IVF-laboratorier som utfører vitrifikasjon på dag 5 ettermiddag eller dag 6.- Overfør embryoet til forvarmede (37 °C) mikrodråper med 10-15 μL kulturmedium, og vask hvert embryo forsiktig serielt i 3 mikrodråper ved pipettering på dag 5.
- Overfør hvert embryo til en unik forvarmet (37 ° C) enkelt mikrodråpe av kulturmedium for prøvetaking. Volumet av en enkelt dråpe kulturmedium kan ikke overstige 15 μL.
- Utfør blastocystembryokultur på dag 5/dag 6 ved 37 °C og 5 % CO2.
- Alternativ 1: Embryovask og dyrkning
- Eksempel på innsamling
- Juster ICM forsiktig i betydelig avstand fra laserstrålens målrettede punkt, som fokuserer på cellekrysset i trofhektodermen for å generere et lite hull i trofhektodermen for å frigjøre væsken fra blastokoelhulen. Deretter flyttes embryoer til fryseløsning for kryopreservering i henhold til konvensjonell prosess.
- Overfør dyrkningsmedium fra hvert dyrket embryo til et RNase/DNase-fritt PCR-rør inneholdende 5 μL cellelysebuffer.
- Samle samme mengde kulturmedium uten å bli brukt til embryokultur som en negativ kontroll. Frys alle oppsamlede prøver umiddelbart i flytende nitrogen og oppbevar deretter ved -80 °C etter å ha blitt samlet inn til de utsettes for NICS-analysen.
- Utfør vitrifikasjon som beskrevet i protokollen.
3. Protokoll 2: Bibliotekkonstruksjon
- Kultur medium lysis
- Fortynn 1 μL positiv kontroll (10 ng humant gDNA) med 199 mikrol friskt kulturmedium. Bland godt og sentrifuge røret kort (200 x g i 5 s).
- Overfør 10 μL dag 5-dag 6 blastocystdyrkningsmedium, fortynnet positiv kontroll og friskdyrkningsmedium til nye 0,2 ml PCR-rør.
- Tilsett 1 μL MT Enzyme Mix til hvert PCR-rør og bland grundig ved pipettering og sentrifuge umiddelbart i 2-3 s ved 200 x g.
- Sett PCR-røret/-rørene fra trinn 3.1.3 i en forvarmet NICS prøveklargjøringsstasjon og kjør lysisprogrammet som følger: 10 min ved 75 °C; 4 min ved 95 °C; hold ved 22 °C.
MERK: Prøveklargjøringsstasjonen kan sammenlignes med en standard PCR-maskin.- Klikk på Lysis-ikonet for å gå til oppsettskjermen.
- Velg Tube for kontrollmodus; inngang 10 μL for prøvevolum; velg På for Hotlid-kontroll og angi 105 °C for temperaturen. Velg Nei for pause ved første seg. Klikk OK for å fortsette.
- Vent til oppholdstiden vises --:- -:-, som angir slutten på programmet, og klikk deretter Stopp for å avslutte programmet.
- Stopp programmet etter at prosessen er fullført. Fortsett til neste trinn umiddelbart.
- Forberedelse før biblioteket
- Tine Pre-Lib buffer til RT. Bland grundig ved pipettering og sentrifuge umiddelbart i 2-3 s ved 200 x g.
- Forbered en masterblanding for prelibrary-reaksjon som følger: tilsett 2 μL Pre-Lib Enzyme Mix til 60 μL Pre-Lib Buffer, bland reaksjonen grundig og sentrifuge kort.
- Tilsett 60 μL prelibrary reaksjonsblanding i hver forbehandlet mediumprøve fra forrige trinn. Bland grundig ved pipettering og sentrifuge umiddelbart i 2-3 s ved 200 x g.
- Plasser PCR-røret/-rørene fra trinn 3.2.3 i prøveforberedelsesstasjonen og kjør forbibliotekprogrammet som følger: 95 °C i 2 minutter; 12 sykluser på 15 °C i 40 s, 22 °C i 40 s, 33 °C i 30 s, 65 °C i 30 s, 72 °C i 40 s, 95 °C i 10 s og 63 °C i 10 s; og hold ved 4 °C.
- Klikk på Pre_Lib-ikonet for å gå inn på oppsettskjermen.
- Velg Tube for kontrollmodus; inngang 70 μL for prøvevolum; velg På for Varmlokkkontroll og angi 105 °C for temperaturen. Velg Nei for pause ved første seg. Klikk OK for å fortsette.
- Vent til oppholdstiden vises --:- -:-, som indikerer slutten av programmet, og klikk Stopp for å avslutte programmet.
- Stopp programmet når prosessen er fullført. Fortsett til neste trinn umiddelbart.
- Forberedelse av bibliotek
- Tine bibliotekbufferen til RT. Bland grundig ved pipettering og sentrifuge umiddelbart i 2-3 s ved 200 x g.
- Forbered en masterblanding for bibliotekreaksjon som følger: tilsett 1,6 μL Library Enzyme Mix til 60 μL bibliotekbuffer, bland reaksjonen grundig og sentrifuge kort.
- Tilsett 60 μL bibliotekreaksjonsmiks og 2 μL strekkodeprimer til hvert forhåndsbibliotekprodukt fra trinn 3.2.3. Bland reaksjonen grundig og sentrifuge kort.
- Plasser PCR-røret/-rørene fra trinn 3.2.3 i termisk syklist og kjør bibliotekforberedelsesprogrammet som følger: 94 °C i 30 s; 17 sykluser på 94 °C i 25 s, 62 °C i 30 s og 72 °C i 45 s); og hold deretter ved 4 °C.
- Klikk på Lib_Prep-ikonet for å gå inn på oppsettskjermen.
- Velg Tube for kontrollmodus; inngang 130 μL for prøvevolum; velg På for Hotlid-styring og angi 105 °C for tilsvarende temperatur. Velg Nei for pause ved første seg. Klikk OK for å fortsette.
- Vent til oppholdstiden vises --:- -:-, som indikerer slutten av programmet, og klikk Stopp for å avslutte programmet.
- Bibliotek Rensing
- Fjern Magbeads fra lagring ved 2-8 °C i minst 20 minutter før rensetrinnet. Vortex og bland Magbeads i 20 s. Dispenser nok perler til rensetrinnet i et nytt 1,5 ml mikrosentrifugerør og varme perler til RT.
- Legg til 1x Magbeads i hvert bibliotek. Bland ved å pipettere opp og ned ≥10 ganger og ruge på RT i 5 minutter.
MERK: Legg for eksempel til 100 μL magbeads til 100 μL bibliotekprøve. - Etter inkubering, sentrifuge røret kort og sett på et magnetisk stativ.
- Vent i ca. 5 minutter til oppløsningen er klar. Mens du holder røret på magnetstativet, må du suge oppløsningen forsiktig og kaste.
- Tilsett 200 μL ferskt tilberedt 80% etanol til røret. Inkuber ved RT i 30 s og fjern forsiktig supernatanten. Gjenta en gang til.
- Fjern etanolen så fullstendig som mulig. Lufttørk perlene på magnetstativet i ca. 5-10 min ved RT.
- Fjern røret fra magnetstativet, tilsett 17,5 μL elueringsbuffer og virvel røret for å resuspendere perlene. Sentrifuger røret kort og rug på RT i 5 min.
- Plasser røret på magnetstativet og vent til løsningen blir klar. Overfør forsiktig 15 μL supernatant til en ny tube.
- Kvantifisering av bibliotek
- Kvantifiser rensede biblioteker ved å bruke fluorometeret i henhold til brukerhåndboken til qubit dsDNA HS Assay kits28. Utbyttet av bibliotekene varierer fra ~ 15 til 300 ng.
- Sammenslåing av bibliotek
- Bruk 10 nanogram av hver bibliotekprøve for sammenslåing.
- Sekvensering
- Se brukerhåndboken for sekvensering (15027617 v01)29.
- Rensede biblioteksekvenser på 50 bp i en enkelt ende på plattformen ga omtrent 2 millioner avlesninger for hver prøve, og en sekvenseringsdybde på 0,03 × ble anbefalt.
- Analyse av data
- Skriv inn brukernes navn og passord på påloggingssiden
- Etter å ha logget inn på systemet, klikk på Analyse og en ny side vises. Klikk på Opprett innsending under kategorien NICS-A. Velg deretter NGS for plattformen, velg selskap, velg NICSInst for reagenset, skriv inn prosjektinformasjonen i boksen under Prosjekt-ID, angi analyseinnstillingene og last opp filene. Når alle sekvensfilene er lastet opp, klikker du på Send for å starte analysen (figur 3A).
- Klikk Vis innsendinger for å vise listen over innsendte prosjekter. Når analysen er fullført, endres statusen for et prosjekt til Fullført, og en Vis-knapp vises i rapportfeltet. Klikk på Vis-knappen for å vise sammendragstabellen for NICS-analyse (figur 3B).
- Klikk på Eksporter rapport-knappen for å lagre rapportene (figur 3C).
MERK: Tre typer filer eksporteres for hver analyse. En grafisk fil som inneholder alle kopinummervariasjonsplott (CNV) for hvert kromosom og hele genom, som vil bli lagret under "graf" -mappen; et regneark som inneholder eksempel-QC-detaljene for denne analysekjøringen; en dokumentfil som inneholder NICS-rapportene tilpasset av brukeren; og et regneark som inneholder eksempelsammendragsinformasjonen for denne analysekjøringen.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Den foreliggende studien brukte den foreslåtte metoden på en pasient. IRB-godkjenning og informert samtykke ble innhentet før anvendelse av NICS-analyse. Den foreliggende studien innhentet 6 blastocyster fra pasienter og utførte NICS på alle 6 embryoene på dag 4 til dag 5 medium. Kromosomavvik forårsaket av foreldrenes balanserte translokasjon ble påvist i fem av kromosomene med NICS-analysen; derfor kunne de ikke brukes til overføring (figur 4A-E). NICS-resultatene fra de to embryoene viste samme karyotype 45, og XN og -18 (×1) var begge kromosom 18-delesjoner (figur 4A, B). Karyotypen 46, XN, -1p (pter→p21.1, ×1) er bare den korte armen til kromosomet 1 pter→p21.1 regiondelesjon (figur 4D).
NICS-resultatene viste karyotype 46, XN, +1p (pter→p21.2, ×3) og -18(q21.32→qter, ×1), noe som indikerte at både den lange armen av kromosom 18 q21.32→qter-regionen og den korte armen av kromosom 1 pter→p21.2-regionen ble duplisert (figur 4E). Selv om karyotypene 46, XN, +5q (×4) og -8 (×1, mos) er kromosom 5-duplikasjoner og viser 8 mosaikkforskjeller, kan NICS-analysen skjerme alle 24 kromosomer for aneuploidi. Denne prosessen gir en ny metode for overføring av enkeltnormale blastocyster av normal karyotype.

Figur 1. Fjerningen fullstendighet av cumulus celler. (A) Oocyttene med cumulusceller. (B) Oocyttene uten cumulusceller festet. Klikk her for å se en større versjon av denne figuren.

Figur 2. Kumulusceller fjernes fra et embryo ved D3 før de overføres til BM. Alle tilknyttede cumulusceller må fjernes før mediet endres fra den første spaltningsembryokulturmedieplaten til blastocystkulturmedieplaten, som er på dag 3 etter at embryoene når 8-cellestadiet. Eventuelle cumulusceller som ikke fjernes, vil forstyrre den endelige analysen og gi falske negative resultater. Klikk her for å se en større versjon av denne figuren.

Figur 3. Dataanalyse. (A) Det finnes forskjellige alternativer for brukerapplikasjonen. For sekvenseringsplattformselskap kan brukere velge Illumina, Ion Torrent eller MGI. Brukerne kan velge om kjønnsinformasjonen skal rapporteres. Ferdig med parameterinnstillingen ovenfor, klikk på boksen under Filopplasting og velg passende sekvenseringsfiler du vil laste opp. For Illumina velger du filene med filtypen fastq.gz. Klikk på Send for å starte analysen etter at opplastingen er fullført. (B) Visningen av sammendragstabellen. Sammendragstabellen består av følgende informasjon: Eksempelnavn: Navnet på hver NICS-prøve er oppført; Data-QC: Angir om sekvenseringsfilen passerer QC for NICS-analyse; AI-vurdering: Vurderingen (A, B eller C) for hvert NICS-utvalg; AI_Rating Tolkning: Evaluering av embryoimplantasjonspotensial; AI-gradering: Poengsummen for hvert NICS-utvalg; CNV-plott (Whole Genome): Se CNV-profilene til alle kromosomer; (c) Lagre rapport-siden. Klikk på Eksporter rapport-knappen ved siden av Sammendrag av resultater. Velg informasjonen du vil vise i den endelige rapporten, og klikk på Eksporter. Rapportene lagres i nedlastingsmappen på datamaskinen. Klikk her for å se en større versjon av denne figuren.

Figur 4. Embryoscreening og seleksjon ved hjelp av NICS fra en pasient. Totalt seks embryoer vellykket utviklet til blastocyststadiet, og Day4-Day5 kulturmedium fra hvert embryo ble samlet inn for NICS-analysen. (A) og (B) er NICS-resultatene av de to blastocystembryoene viste at samme karyotype 45, XN, -18 (×1) begge er kromosom 18-delesjon. (C) viste karyotype 46, XN, +5q (×4), -8 (×1, mos) er kromosom 5 duplisering og 8 mosaikk. (D) viste karyotype 46, XN, -1p (pter→p21.1, ×1) er bare den korte armen av kromosom 1 pter→p21.1 region delesjon, mens (E) viste karyotype 46, XN, +1p (pter→p21.2, ×3), -18(q21.32→qter, ×1) er kort arm av kromosom 1 pter→p21.2 region duplisering og lang arm av kromosom 18 q21.32 → qter region (F) viste balansert kromosomsammensetning. X-aksen betyr 22 autosomer i rødt og blått, y-aksen indikerer kopinummeret til hvert autosom. De grå prikkene er linjalskalaen for kopinummerrespons hvert kassevindu og normal karyotype av kopinummer må være 2. Klikk her for å se en større versjon av denne figuren.
Tabell S1. Suksessratene for DNA-deteksjon alternativ 1 og alternativ 2. Klikk her for å laste ned denne tabellen.
Tabell S2. Samsvar mellom NICS og PGT-A i forskjellige alternativer. Klikk her for å laste ned denne tabellen.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Endringer og feilsøking
Hvis NICS-resultatene er forurenset med foreldrenes genetiske materiale, må du sørge for at alle cumulus-corona radiata-celler fjernes og sørge for at ICSI utføres for befruktning. Feil medium lagring eller malforberedelsesprosesser unngås, noe som kan forringe DNA. Arbeidsområdet ble renset grundig med DNase og RNase dekontamineringsreagenser. For å unngå forurensning fra andre embryoer ble ett embryo alltid dyrket i en enkelt dråpe medium for å unngå krysskontaminering fra dag 4. Fenomenet forurensning minimeres ved forsinkelse av plassering av embryoer i det endelige kulturfallet30,31,32,33. For å minimere maternell forurensning modifiserte Kuznyetsov34 embryokulturprosedyrene fra dag 0 til dag 4, inkludert forsiktig fjerning av gjenværende koronaceller ved pipettering og spyling.
Lane et al.30 viser at når man tar embryokulturmediet fra dag 4 til dag 5, forbedres nøyaktigheten av euploidideteksjon, embryoploidikonsistensen er mer enn 95%, og konsistensen av kjønnskromosomet når 100%. Lledo et al.33 fant at forekomsten mellom dag 3-dagers 5 kulturmedium og TE-prøver var 74,6% og 92,0% når embryoer ble dyrket fra dag 4 til dag 6.
Våre interne data støtter også denne konklusjonen, som vist i tabell S1. Sammenlignet med den konvensjonelle dag 3-dagers 5-dyrkningsmetoden ble granulosacellene ytterligere fjernet på grunn av ytterligere en endring i dyrkningsmedium på dag 4 eller dag 5. Vi leverer interne data (tabell S1) som viser at våre to metoder (alternativ 1 og alternativ 2) har god konsistens sammenlignet med PGT-A, noe som er bedre enn prøvetakingsmetoden uten grundig fjerning av CC.
IF-amplifiserte produkter dukket opp i negativ kontroll, og eksterne DNA-materialer kan ha forurenset reagenset eller arbeidsområdet. Arbeidsområdet skal rengjøres med DNA / RNA som fjerner reagenser, nukleasefrie materialer bør brukes, og reagensene skal alisiteres etter første gangs bruk.
Forskjeller i suksessrater mellom alternativ 1 og alternativ 2 er omtalt i tabell S1 og tabell S2.
Begrensninger ved NICS-analysen
Det er to hovedbegrensninger ved NICS. 1) Før ICSI må alle cumulusceller (vanligvis mors opprinnelse, vanligvis normal kromosomsammensetning) fjernes. Hvis fjerningen er ufullstendig, kan cumuluscellene frigjøre DNA under embryoutvikling og det eksterne DNA forsterkes, noe som kan være årsaken til falsk negativ deteksjon. 2) Det er vanskelig å fjerne sæden festet til zona pellucida, og NICS-prosedyren anbefales sterkt å utføres med ICSI. Selv om regelmessig utskifting av spaltningsmedier på dag 3 kan redusere muligheten for kontaminering på grunn av kumulusceller og overflødige sædceller, må denne kontamineringen minimeres hvis NICS brukes i klinisk IVF. Imidlertid er det utviklet en metode for å oppdage NICS i IVF-embryoer, inkludert funksjonen til å gjenkjenne eksogent DNA, som vil bli demonstrert i nær fremtid.
Denne studien sammenlignet ikke forskjellene mellom ulike medier siden store kliniske studier har sammenlignet kulturmedier. Åtte sentre brukte 4 forskjellige kulturmedier, sekvensielle og kontinuerlige, og 2 forskjellige prosentandeler albumintilskudd (5% og 10%), og disse forskjellene hadde ingen signifikante effekter på nøyaktigheten av embryonale cfDNA-resultater31. Disse funnene støtter den potensielle anvendeligheten av embryonal cfDNA-analyse til alle IVF-laboratorier når de arbeider under den spesifikke protokollen.
Betydning i forhold til eksisterende metoder
NICS-metoden unngår embryobiopsi og forbedrer dermed brukssikkerheten betydelig. Sammenlignet med blastocyster er NICS en enkel, tidsbesparende, sensitiv og reproduserbar preimplantasjonsscreeningteknikk som er egnet for assistert befruktning populasjoner med høy sannsynlighet for aneuploidi. I motsetning til invasiv biopsi, som krever betydelig og faglig kunnskap for blastocystbiopsiprosedyren, kan NICS brukes i stor utstrekning siden den enkle samlingen av brukt medium bare følger den vanlige driften av IVF19 og den ikke krever PGS/PGD-kvalifisering i noen land.
Fremtidige applikasjoner
NICS har potensial for bred anvendelighet for kromosomscreening i klinisk IVF, ikke bare for ICSI, men også for IVF-embryoer. Selv om ICSI er sterkt anbefalt, er det nødvendig med metoder for å fjerne sæd festet til zona pellucida for å forhindre påvirkning av sædceller.
Morfologisk vurdering er en tradisjonell metode for embryoevaluering, men i de fleste tilfeller kan kromosomalt unormale embryoer virke morfologisk lik kromosomalt normale (euploide) embryoer. Å kombinere morfologisk vurdering med NICS-analysen ved overføring av ploide embryoer med god morfologi til livmoren kan forbedre de pågående graviditetsratene og levende fødselsrater. En randomisert klinisk studie vil bli utført for å evaluere den kliniske effekten av enkeltembryooverføring ved bruk av NICS-teknologi.
Kritiske trinn i protokollen
Alle cumulus-corona radiata celler må fjernes fra oocyttene før befruktning. Oocytter ble befruktet ved intracytoplasmatisk spermieinjeksjon (ICSI). Tilsetning av human-avledede proteiner / kosttilskudd til kulturmediet ble unngått. Dyrkningsmediet ble endret på dag 4 og samlet på dag 5-dag 6 da blastocystene ble fullstendig utvidet. Embryoer ble dyrket i individuelle dråper av kulturmedium som begynte på dag 4. Ved oppsamling av kulturmediet ble overføringspipetter mellom prøvene endret for å unngå forurensning.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yaxin Yao, Jieliang Ma, Jing Wang og Sijia Lu er ansatte i Yikon Genomics Co., Ltd.
Acknowledgments
Forfatterne vil gjerne takke Shiping Bo og Shujie Ma for deres hjelp i NGS dataanalyse. Finansiering: dette arbeidet ble støttet av National Key Research and Development Program (Grant No. 2018YFC1003100).
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
References
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Jr Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T. Jr, Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid? Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- Thermo Fisher Scientific. Qubit dsDNA HS Assay Kit. , Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015).
- Miseq system use guide. , Available from: https://support.illumina.com/downloads/miseq_system user _ guide 15027617.html (2016).
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Tags
Kromosomscreening embryoer med menneskelig preimplantasjon brukt kulturmedium prøvetaking kromosomal ploidianalyse in vitro-fertilisering (IVF) preimplantasjonsgenetisk testing for aneuploidi (PGT-A) trohektoderm (TE) indre cellemasse (ICM) falske positive og negativer NICS-teknologi DNA-sekvensering kulturmediumprøvetaking helgenomamplifisering (WGA) bibliotekforberedelse NGS-dataanalyse kryopreservering IVF-laboratoriumErratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021.
Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
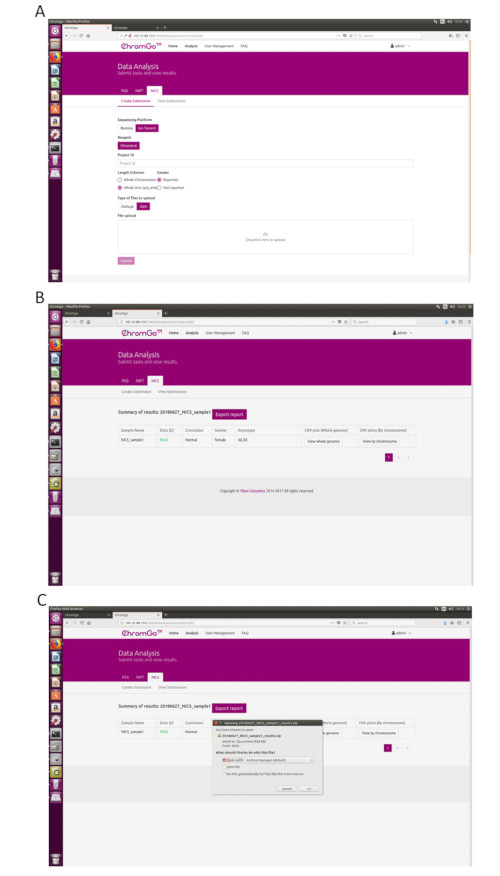
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
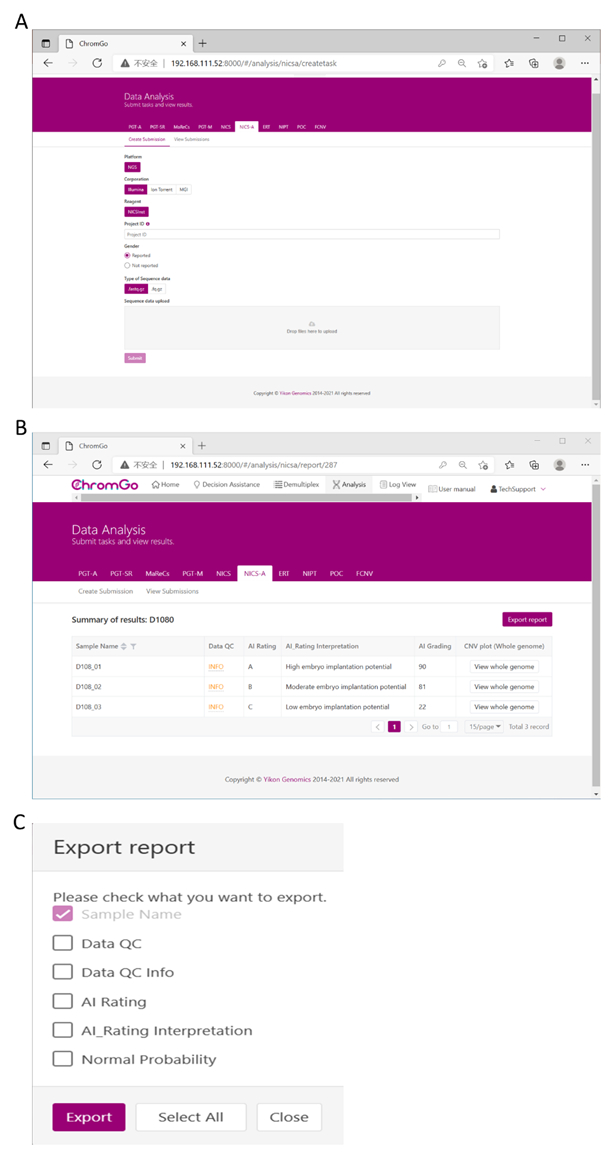
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.



