ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Denne undersøgelse rapporterer en protokol til kromosomscreening af menneskelige embryoner, der bruger brugt dyrkningsmedium, som undgår embryobiopsi og muliggør kromosomploidiidentifikation ved hjælp af NGS. Denne artikel præsenterer den detaljerede procedure, herunder forberedelse af dyrkningsmedium, helgenomamplifikation (WGA), næste generations sekventering (NGS) biblioteksforberedelse og dataanalyse.
Abstract
Ved klinisk in vitro-befrugtning (IVF) kræver den fremherskende metode til PGT-A biopsi af nogle få celler fra trophectoderm (TE). Dette er den slægt, der danner moderkagen. Denne metode kræver imidlertid specialiserede færdigheder, er invasiv og lider af falske positive og negative, fordi kromosomtallene i TE og den indre cellemasse (ICM), som udvikler sig til fosteret, ikke altid er de samme. NICS, en teknologi, der kræver sekventering af DNA, der frigives i kulturmediet fra både TE og ICM, kan tilbyde en vej ud til disse problemer, men har tidligere vist sig at have begrænset effektivitet. Denne undersøgelse rapporterer den fulde protokol for NICS, som inkluderer dyrkningsmediumprøveudtagningsmetoder, helgenomamplifikation (WGA) og biblioteksforberedelse og NGS-dataanalyse ved hjælp af analysesoftware. I betragtning af de forskellige kryopræserveringstider i forskellige embryolaboratorier har embryologer to metoder til indsamling af embryokulturmedium, der kan vælges i henhold til IVF-laboratoriets faktiske forhold.
Introduction
Assisteret reproduktionsteknologi (ART'er) er i stigende grad blevet brugt til behandling af infertilitet. Imidlertid har succesraten for ART, såsom IVF, været begrænset, og graviditetstabsraten er signifikant højere end for den normale befolkning1. Hovedårsagen til disse problemer er kromosomale abnormiteter, som almindeligvis findes i præimplantation menneskelige embryoner2. PGT-A er en effektiv metode til screening af embryoner for kromosombalance før implantation 3,4. Nogle undersøgelser har vist, at PGT-A kan reducere abortraten og forbedre graviditetsraten 5,6,7,8. PGT-A kræver imidlertid kompleks teknisk ekspertise, der kræver specifik træning og erfaring. Den invasive embryobiopsiprocedure kan også potentielt forårsage skade på embryonerne9. Undersøgelser har vist, at blastomerbiopsi kan hindre efterfølgende udvikling, og antallet af biopserede TE'er kan påvirke implantationshastighederne10. Selvom det langsigtede biosikkerhedsproblem ved embryobiopsi endnu ikke er blevet evalueret grundigt hos mennesker, har dyreforsøg vist dets negative indflydelse på embryoudvikling11,12,13.
Tidligere rapporter viste, at spormængder af DNA-materialer blev udskilt i dyrkningsmediet under embryoudvikling, og der er gjort en indsats for at udføre omfattende kromosomscreening (CCS) ved hjælp af brugt embryokulturmedium 14,15,16,17,18. Detektionsraterne og testenes nøjagtighed har imidlertid ikke opfyldt kravene til omfattende klinisk brug. Denne undersøgelse rapporterede en forbedring i NICS-analysen for at øge detektionsraterne samt nøjagtigheden af NICS-testen19. I de senere år er blastocoelevæske (BF) blevet undersøgt som en analytisk prøve af minimalt invasiv PGT-A. Imidlertid varierer andelen af vellykket genom-dækkende amplifikation og detekterbart DNA i blastocystvæskeprøver fra 34,8% til 82%20,21,22. Mængden af BF rapporteret i forskellige undersøgelser varierer fra 0,3 nL til 1 μL. I betragtning af den lave mængde DNA i BF er det muligt at øge mængden af cellefrit DNA ved at blande blastocystvæske og dyrkningsmedium for at forbedre succesraten og konsistensen af detektionen. Kuznyetsov et al.23 og Li et al.24 behandlede zona pellucida med en laser og frigav blastocystvæske i dyrkningsmediet for at forbedre den samlede mængde embryonalt DNA, og amplifikationshastigheden for de kombinerede medium / BF-prøver efter WGA var henholdsvis 100% og 97,5%. Jiao et al.25 opnåede også en 100% forstærkningssuccesrate ved at bruge den samme metode.
Denne undersøgelse rapporterer en detaljeret protokol, der inkluderer forberedelse af brugte medier, NGS-forberedelse og dataanalyse. Ved omhyggeligt at fjerne cumulusceller fra oocytter udførte denne undersøgelse intracytoplasmatisk enkelt sædinjektion (ICSI) og blastocystkultur. Dag 4-dages 5/dag 6 brugt medium blev indsamlet til WGA og NGS bibliotek forberedelse. Ved hjælp af NICS-teknologi strømlinede denne undersøgelse forberedelsestrinnene til WGA- og NGS-biblioteket på ca. 3 timer og opnåede CCS-resultater noninvasivt på ca. 9 timer.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Etisk tilladelse blev erhvervet fra den etiske komité for Peking University Third Hospital.
1. Forberedelse
BEMÆRK: De nødvendige materialer og udstyr er angivet i Materialetabel.
- Reagenser
- Forvarm og ligevægt (afbalanceret) 20-30 μL gametmedium/befrugtningsmedium og spaltnings-/blastocyst-stadium kulturmedium (dækket med mineralolie) og hyaluronidase (i et tæt lukket rør) ved 37 °C, 5% CO2 og 5 %O2 i en tri-gas inkubator natten over før brug.
- Forvarm hyaluronidase til 37 °C på en arbejdsflade i en stinkhætte.
- Forbered forglasningsbuffer og prøveopsamlingsreagenser i henhold til producentens anvisninger.
- Værktøjer
- Forbered prøveopsamlings- og overførselspipetter (indvendig diameter på ~200 til 250 μm), denudations-/stripperpipetter (indvendig diameter på ≥150 μm, ~130-140 μm og ~120 μm) og pipetter til vask (indvendig diameter på ~150 μm) ved at trække Pasteur-pipetter af glas for at generere brandpolerede åbne fine spidser.
BEMÆRK: De pipetter, der bruges til prøveindsamling/overførsel, denudation og vask, kan købes direkte. Holdenålene og injektionsnålene kan også købes direkte.
- Forbered prøveopsamlings- og overførselspipetter (indvendig diameter på ~200 til 250 μm), denudations-/stripperpipetter (indvendig diameter på ≥150 μm, ~130-140 μm og ~120 μm) og pipetter til vask (indvendig diameter på ~150 μm) ved at trække Pasteur-pipetter af glas for at generere brandpolerede åbne fine spidser.
2. Protokol nr. 1: Indsamling af prøver
- Forbehandling af oocyt-corona-cumulus-kompleks (OCCC'er) før fordøjelse med hyaluronidase
- Opnå ovariestimulation med både follikelstimulerende hormon (FSH) og humane menopausale gonadotropin (hMG) præparater. Når blyfolliklen er >18 mm, skal du bruge 10.000 IE choriongonadotropin (hCG) til endelig oocytmodning.
- Udfør oocythentning 36 timer efter triggerskud. Opsaml og overfør oocytter til vævskulturskåle med 2,5 ml forvarmet m-HTF dækket af mineralolie.
- OCCC'erne overføres hurtigt til den centrale brønd i en orgelkulturskål, der indeholder 1 ml befrugtningsmedium ved hjælp af en overførselspipette, og inkuberes derefter med oocytterne ved 37 °C i en 5% CO 2 og 5%O2 inkubator i2-4 timer.
- OCCC'er fordøjes med hyaluronidase ved at tilsætte 1 ml forvarmet hyaluronidase (80 IE/ml) til det centrale hul i en orgelkulturskål, der indeholder OCCC'er (trin 2.1.3). Hold den endelige koncentration af hyaluronidase på 40 IE/ml og bland grundigt.
- OCCC'erne inkuberes på en 37 °C termisk platform i 2 min. Overhold ændringerne under et mikroskop hver 30. s, indtil der kun var 1-2 lag granulosaceller tilbage.
- Denudation af granulosaceller
- Overfør hurtigt de fordøjede OCCC'er i dyrkningsskålen til oocythåndtering og dæk med mineralolie i hvert hul.
- Overhold de adskilte granulosaceller under et mikroskop. Aspirer forsigtigt og frigør oocytterne 5 gange for at fjerne resterende granulosaceller omkring oocytterne.
- Gentag det foregående trin i de resterende 3 brønde for helt at fjerne granulosacellerne.
BEMÆRK: Ovenstående trin (2.1-2.3) kan udføres i henhold til rutinedriften i hvert laboratorium.
- Evaluering af oocytten
- Evaluer fuldstændigheden af granulosa cellefjernelse ved hjælp af et mikroskop. Hvis cellerne ikke kunne fjernes fuldstændigt, er tilbageholdelsen af 5 eller færre granulosaceller acceptabel på dette tidspunkt.
BEMÆRK: Hvis cumulusceller stadig er bundet til oocytten, kan resten fjernes senere på dag 3, før embryoet overføres fra spaltningsstadiets dyrkningsmedium til blastocyst-stadiets dyrkningsmedium.
- Evaluer fuldstændigheden af granulosa cellefjernelse ved hjælp af et mikroskop. Hvis cellerne ikke kunne fjernes fuldstændigt, er tilbageholdelsen af 5 eller færre granulosaceller acceptabel på dette tidspunkt.
- Efter udførelse af intracytoplasmatisk sædinjektion (ICSI)26 overføres oocytterne til 20-30 μL spaltningsembryokulturmediummikrodråber (en oocyt svarer til en mikrodråbe) ved hjælp af overførselspipetter og inkuberes i en 37 °C, 5% CO2 og 5%O2 inkubator.
- Registrer dagen for ICSI som dag 0. Kontroller embryonerne og score i henhold til Istanbul-konsensusworkshoppen om embryovurdering af dag 1 til befrugtning (ca. 18 timer), dag 2 (ca. 45 timer) og dag 3 (ca. 68 timer) til embryospaltning27.
- Embryo vask
- Der fremstilles 20-30 μL blastocystkulturmediummikrodråber for hvert embryo, der er dækket med mineralolie i vævskulturskåle på dag 2 i en inkubator på 37 °C, 5% CO2 og 5 %O2 .
- Forbered yderligere tre mikrodråber dækket med mineralolie, og mærk de nye vævskulturskåle til vask nr. 1-3.
- Overfør dag 3 embryoner til vaskemikrodråberne. Aspirer forsigtigt og frigiv embryonerne 3 gange i hver dråbe ved hjælp af denudationspipetter.
BEMÆRK: Denne procedure kan også hjælpe med at fjerne de resterende granulære celler, der er fastgjort til embryoet. - Observere og evaluere embryonerne under et mikroskop på dag 3, før mediet blev ændret fra spaltningsstadiet kulturmedium til blastocystkulturmedium til morfologisk scoring. Hvis cumulusceller stadig var fastgjort til embryoet, pipetteres passende op og ned i en anden forvarmet og ekvilibreret blastocystkulturmediumdråbe dækket med mineralolie med en stripperpipette, indtil cumuluscellerne var helt fjernet.
BEMÆRK: Alle vedhæftede cumulusceller skulle fjernes fuldstændigt på dag 3, før embryoet blev overført fra spaltningstrinnets kulturmediumplade til blastocyst-trins kulturmediumpladen. Eventuelle resterende cumulusceller vil forstyrre den endelige analyse og give falske negative resultater.
- To muligheder for indsamling af kulturmedium
BEMÆRK: IVF-centret kan vælge mellem en af to metoder til indsamling af kulturmedium baseret på centrets ressourcer, krav og præferencer.- Mulighed 1: Embryovask og -dyrkning
BEMÆRK: Denne mulighed er for IVF-laboratorier, der udfører vitrifikation om morgenen på dag 5.- Embryoet overføres til forvarmede (37 °C) mikrodråber af dyrkningsmedium, og hvert embryon vaskes forsigtigt serielt i 3 mikrodråber ved pipettering på dag 4 eftermiddag.
- Hvert embryon overføres til en unik forvarmet (37 °C) enkelt mikrodråbe dyrkningsmedium til prøveopsamling. Volumenet af en enkelt dråbe dyrkningsmedium må ikke overstige 25 μL.
- Der udføres blastocystembryodyrkning på dag 5/dag 6 ved 37 °C, 5 % CO2 og 5 %O2.
- Mulighed 2: Embryovask og -dyrkning
BEMÆRK: Denne mulighed er for IVF-laboratorier, der udfører vitrifikation på dag 5 eftermiddag eller dag 6.- Embryoet overføres til forvarmede (37 °C) mikrodråber med 10-15 μL dyrkningsmedium, og hvert embryon vaskes forsigtigt serielt i 3 mikrodråber ved pipettering på dag 5.
- Hvert embryon overføres til en unik forvarmet (37 °C) enkelt mikrodråbe dyrkningsmedium til prøveopsamling. Volumenet af en enkelt dråbe dyrkningsmedium må ikke overstige 15 μL.
- Der udføres blastocystembryodyrkning på dag 5/dag 6 ved 37 °C og 5% CO2.
- Mulighed 1: Embryovask og -dyrkning
- Indsamling af prøver
- Juster forsigtigt ICM i en betydelig afstand fra laserstrålens målrettede punkt, som fokuserer på trophectodermens cellekryds for at generere et lille hul i trophectoderm for at frigive væsken fra blastocoelhulen. Derefter flyttes embryoner til fryseopløsning til kryopræservering i henhold til den konventionelle proces.
- Overfør dyrkningsmedium fra hvert dyrket embryo til et RNase/DNase-frit PCR-rør indeholdende 5 μL cellelysebuffer.
- Saml den samme mængde dyrkningsmedium uden at blive brugt til embryokultur som en negativ kontrol. Alle indsamlede prøver fryses straks i flydende nitrogen og opbevares derefter ved -80 °C efter opsamling, indtil de udsættes for NICS-assayet.
- Udfør vitrifikation som beskrevet i protokollen.
3. Protokol nr. 2: Opførelse af biblioteker
- Kultur medium lysis
- 1 μL positiv kontrol (10 ng humant gDNA) fortyndes med 199 μl frisk næringssubstrat. Bland grundigt og centrifuger røret kort (200 x g i 5 s).
- Overfør 10 μL blastocystdyrkningssubstrat fra dag 5 dage 6, fortyndet positiv kontrol og frisk dyrkningsmedium til nye 0,2 ml PCR-reagensglas.
- Der tilsættes 1 μL MT Enzyme Mix til hvert PCR-glas og blandes grundigt ved pipettering og centrifugering straks i 2-3 s ved 200 x g.
- Sæt PCR-røret (-rørene) fra trin 3.1.3 i en forvarmet NICS-prøveforberedelsesstation, og kør lysisprogrammet som følger: 10 min ved 75 ° C; 4 minutter ved 95 °C; opbevares ved 22 °C.
BEMÆRK: Prøveforberedelsesstationen kan sammenlignes med en standard PCR-maskine.- Klik på ikonet Lysis for at åbne opsætningsskærmen.
- Vælg Tube for kontroltilstand; input 10 μL for prøvevolumen; vælg Til for Hotlid-styring, og indtast 105 °C som temperatur. Vælg Nej for Pause ved første seg. Klik på OK for at fortsætte.
- Vent, indtil Resterende tid viser --:- -:-:--, som angiver slutningen af programmet, og klik derefter på Stop for at afslutte programmet.
- Stop programmet, når processen er afsluttet. Fortsæt straks til næste trin.
- Forberedelse før biblioteket
- Pre-Lib-bufferen optøs til RT. Bland grundigt ved pipettering og centrifuger straks i 2-3 s ved 200 x g.
- Forbered en masterblanding til præbiblioteksreaktion som følger: Tilsæt 2 μL Pre-Lib Enzyme Mix til 60 μL Pre-Lib Buffer, bland reaktionen grundigt og centrifuger kort.
- Der tilsættes 60 μL forbiblioteksreaktionsblanding i hver forbehandlet mediumprøve fra det foregående trin. Bland grundigt ved pipettering og centrifuger straks i 2-3 s ved 200 x g.
- Anbring PCR-røret (-rørene) fra trin 3.2.3 i prøveforberedelsesstationen, og kør forbiblioteksprogrammet som følger: 95 °C i 2 minutter; 12 cyklusser på 15 °C i 40 s, 22 °C i 40 s, 33 °C i 30 s, 65 °C i 30 s, 72 °C i 40 s, 95 °C i 10 s og 63 °C i 10 s og opbevares ved 4 °C.
- Klik på ikonet Pre_Lib for at åbne opsætningsskærmen.
- Vælg Tube for kontroltilstand; input 70 μL for prøvevolumen; vælg Til for Kontrol af varmt låg, og indtast 105 °C som temperatur. Vælg Nej for Pause ved første seg. Klik på OK for at fortsætte.
- Vent, indtil Resterende tid vises --:- -:-:--, hvilket angiver slutningen af programmet, og klik på Stop for at afslutte programmet.
- Stop programmet, når processen er færdig. Fortsæt straks til næste trin.
- Forberedelse af biblioteket
- Optø biblioteksbufferen til RT. Bland grundigt ved pipettering og centrifuger straks i 2-3 s ved 200 x g.
- Forbered en masterblanding til biblioteksreaktion som følger: Tilsæt 1,6 μL Library Enzyme Mix til 60 μL Library Buffer, bland reaktionen grundigt og centrifuger kort.
- Der tilsættes 60 μL biblioteksreaktionsblanding og 2 μL stregkodeprimer til hvert forbiblioteksprodukt fra trin 3.2.3. Bland reaktionen grundigt og centrifuger kort.
- PCR-røret/-rørene fra trin 3.2.3 anbringes i varmecyklisten, og bibliotekets forberedelsesprogram køres på følgende måde: 94 °C i 30 s; 17 cyklusser på 94 °C i 25 s, 62 °C i 30 s og 72 °C i 45 s) og hold derefter ved 4 °C.
- Klik på ikonet Lib_Prep for at åbne opsætningsskærmen.
- Vælg Tube for kontroltilstand; input 130 μL for prøvevolumen; vælg Til for Hotlid-styring, og indtast 105 °C for den tilsvarende temperatur. Vælg Nej for Pause ved første seg. Klik på OK for at fortsætte.
- Vent, indtil Resterende tid vises --:- -:-:--, hvilket angiver slutningen af programmet, og klik på Stop for at afslutte programmet.
- Rensning af biblioteket
- Tag Magbeads ud af opbevaring ved 2-8 °C i mindst 20 minutter før rensningstrinnet. Vortex og bland Magbeads i 20 s. Dispenser nok perler til rensningstrinnet i et nyt 1,5 ml mikrocentrifugerør og varme perler til RT.
- Tilføj 1x Magbeads i hvert bibliotek. Bland ved pipettering op og ned ≥10 gange og inkuber ved RT i 5 min.
BEMÆRK: Tilføj f.eks. 100 μL Magbeads til 100 μL bibliotekseksempel. - Efter inkubation centrifugeres røret kort og anbringes på et magnetisk stativ.
- Vent i ca. 5 minutter, indtil opløsningen bliver klar. Mens du holder røret på magnetstativet, skal du forsigtigt aspirere opløsningen og kassere.
- Tilsæt 200 μL frisklavet 80% ethanol til røret. Der inkuberes ved RT i 30 sek., og supernatanten fjernes forsigtigt. Gentag endnu en gang.
- Fjern ethanolen så fuldstændigt som muligt. Lufttør perlerne på magnetstativet i ca. 5-10 min ved RT.
- Fjern røret fra magnetstativet, tilsæt 17,5 μL elueringsbuffer, og hvirvel røret for at resuspendere perlerne. Centrifuger glasset kort og inkuber ved RT i 5 min.
- Placer røret på magnetstativet, og vent, indtil opløsningen bliver klar. 15 μL supernatant overføres forsigtigt til et nyt glas.
- Kvantificering af biblioteket
- Kvantificer oprensede biblioteker ved hjælp af fluorometeret i henhold til brugervejledningen til qubit dsDNA HS Assay kits28. Udbyttet af bibliotekerne varierer fra ~ 15 til 300 ng.
- Sammenlægning af biblioteker
- Brug 10 nanogram af hver biblioteksprøve til pooling.
- Sekventering
- Se brugervejledningen til sekvensering (15027617 v01)29.
- Oprensede bibliotekssekvenser på 50 bp i en enkelt ende på platformen gav ca. 2 millioner læsninger for hver prøve, og en sekventeringsdybde på 0,03 × blev anbefalet.
- Analyse af data
- Indtast brugernes navn og adgangskode på login-siden
- Når du er logget ind på systemet, skal du klikke på Analyse , og en ny side vises. Klik på Opret indsendelse under fanen NICS-A. Vælg derefter NGS for platformen, vælg selskab, vælg NICSInst for reagenset, indtast projektoplysningerne i feltet under Projekt-id, indstil analysepræferencerne og upload filerne. Når alle sekventeringsfiler er uploadet, skal du klikke på Send for at starte analysen (figur 3A).
- Klik på Vis indsendelser for at få vist listen over indsendte projekter. Når analysen er fuldført, bliver status for et projekt Fuldført, og knappen Vis vises i rapportfeltet. Klik på knappen Vis for at få vist oversigtstabellen over NICS-analyse (figur 3B).
- Klik på knappen Eksportér rapport for at gemme rapporterne (Figur 3C).
BEMÆRK: Tre typer filer eksporteres for hver analyse. En grafisk fil, der indeholder alle kopier nummer variation (CNV) plots for hvert kromosom og hele genomet, som vil blive gemt under "graf" mappe; et regneark, der indeholder QC-eksempeloplysningerne for denne analysekørsel en dokumentfil, der indeholder de NICS-rapporter, der er tilpasset af brugeren; og et regneark, der indeholder eksempeloversigtsoplysningerne for denne analysekørsel.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Denne undersøgelse anvendte den foreslåede metode på en patient. IRB-godkendelse og informeret samtykke blev opnået før anvendelsen af NICS-analysen. Den foreliggende undersøgelse opnåede 6 blastocyster fra patienter og udførte NICS på alle 6 embryoner på dag 4 til dag 5 medium. Kromosomabnormiteter forårsaget af forældrenes afbalancerede translokation blev påvist i fem af kromosomerne med NICS-assayet; derfor kunne de ikke bruges til overførsel (figur 4A-E). NICS-resultaterne for de to embryoner viste den samme karyotype 45, og XN og -18 (×1) var begge kromosom 18-deletioner (figur 4A, B). Karyotype 46, XN, -1p (pter→p21.1, ×1) er kun den korte arm af kromosom 1 pter→p21.1 region sletning (figur 4D).
NICS-resultaterne viste karyotype 46, XN, +1p (pter→p21.2, ×3) og -18 (q21.32→qter, ×1), hvilket indikerede, at både den lange arm af kromosom 18 q21.32→qter-regiondeletion og den korte arm af kromosom 1 pter→p21.2-regionen blev duplikeret (figur 4E). Selvom karyotyperne 46, XN, +5q (×4) og -8 (×1, mos) er kromosom 5-duplikationer og viser 8 mosaikforskelle, kan NICS-analysen screene alle 24 kromosomer for aneuploidi. Denne proces giver en ny metode til overførsel af enkelte normale karyotype blastocyster.

Figur 1. Fjernelsen fuldstændighed af cumulusceller. (A) Oocytter med cumulusceller. B) Oocytterne uden cumulusceller fastgjort. Klik her for at se en større version af denne figur.

Figur 2. Cumulusceller fjernes fra et embryo ved D3, før de overføres til BM. Alle vedhæftede cumulusceller skal fjernes, før mediet skifter fra den oprindelige spaltningsembryokulturmediumplade til blastocystkulturmediumpladen, som er på dag 3, efter at embryonerne når 8-cellestadiet. Cumulusceller, der ikke fjernes, vil forstyrre den endelige analyse og give falske negative resultater. Klik her for at se en større version af denne figur.

Figur 3. Dataanalyse. (A) Der er forskellige muligheder for brugerapplikationen. Til sekventeringsplatformselskab kan brugerne vælge Illumina, Ion Torrent eller MGI. Brugerne kan vælge, om kønsoplysningerne skal indberettes. Færdig med ovenstående parameterindstilling, klik på boksen under Filupload og vælg de relevante sekventeringsfiler, der skal uploades. For Illumina skal du vælge filerne med en udvidelse af fastq.gz. Klik på Send for at starte analysen, når den er uploadet. (B) Visningen af den sammenfattende tabel. Oversigtstabellen består af følgende oplysninger: Eksempelnavn: Navnet på hvert NICS-eksempel er angivet; Data QC: Angiver, om sekventeringsfilen passerer QC til NICS-analyse; AI-klassificering: Klassificeringen (A, B eller C) for hver NICS-prøve; AI_Rating Fortolkning: Vurdering af embryonimplantationspotentiale; AI-klassificering: Scoren for hver NICS-prøve; CNV-plot (helgenom): Se CNV-profilerne for alle kromosomer; (c) Siden Gem rapport. Klik på knappen Eksportér rapport ud for Oversigt over resultater. Vælg de oplysninger, du vil have vist i den endelige rapport, og klik på Eksportér. Rapporterne gemmes i mappen Download på din computer. Klik her for at se en større version af denne figur.

Figur 4. Embryoscreening og udvælgelse ved hjælp af NICS fra en patient. I alt seks embryoner udviklede sig med succes til blastocyststadiet, og Day4-Day5 dyrkningsmedium fra hvert embryo blev indsamlet til NICS-assayet. (A) og (B) er NICS-resultaterne af de to blastocystembryoner viste den samme karyotype 45, XN, -18 (×1) er begge kromosom 18 deletion. (C) viste karyotype 46, XN, +5q (×4), -8 (×1, mos) er kromosom 5 duplikering og 8 mosaik. (D) viste karyotype 46, XN, -1p (pter→p21.1, ×1) er kun den korte arm af kromosom 1 pter→p21.1 region deletion, mens (E) viste karyotype 46, XN, +1p (pter→p21.2, ×3), -18(q21.32→qter, ×1) er kort arm af kromosom 1 pter→p21.2 region duplikering og lang arm af kromosom 18 q21.32 → qter region (F) viste afbalanceret kromosomal sammensætning. X-aksen betyder 22 autosomer i rødt og blåt, y-aksen angiver kopinummeret for hvert autosom. De grå prikker er linealskalaen for kopinummerrespons, hvert bin-vindue og normal karyotype af kopinummer skal være 2. Klik her for at se en større version af denne figur.
Tabel S1. Succesraterne for DNA-detektion Mulighed 1 og Mulighed 2. Klik her for at downloade denne tabel.
Tabel S2. Overensstemmelsen mellem NICS og PGT-A i forskellige muligheder. Klik her for at downloade denne tabel.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Ændringer og fejlfinding
Hvis NICS-resultaterne er forurenet med forældrenes genetiske materialer, skal du sørge for, at alle cumulus-corona radiata-celler fjernes, og sørg for, at ICSI udføres til befrugtning. Forkert medium opbevaring eller skabelonforberedelsesprocesser undgås, hvilket kan nedbryde DNA. Arbejdsområdet blev renset grundigt med DNase- og RNase-dekontamineringsreagenser. For at undgå kontaminering fra andre embryoner blev et embryo altid dyrket i en enkelt dråbe medium for at undgå krydskontaminering fra dag 4. Fænomenet forurening minimeres, når placeringen af embryoner i det endelige kulturfaldforsinkes 30,31,32,33. For at minimere moderens kontaminering ændrede Kuznyetsov34 embryokulturprocedurerne fra dag 0 til dag 4, herunder omhyggelig fjernelse af resterende koronaceller ved pipettering og skylning.
Lane et al.30 viser, at når man tager embryokulturmediet fra dag 4 til dag 5, forbedres nøjagtigheden af euploididetektion, embryoploidikonsistensen er mere end 95%, og konsistensen af kønskromosomet når 100%. Lledo et al.33 fandt, at sammenfaldsraten mellem dag 3-dages 5 dyrkningsmedium og TE-prøver var 74,6% og 92,0%, når embryoner blev dyrket fra dag 4 til dag 6.
Vores interne data understøtter også denne konklusion, som vist i tabel S1. Sammenlignet med den konventionelle dag 3-dages 5 dyrkningsmetode blev granulosaccellerne yderligere fjernet på grund af endnu en ændring i dyrkningsmedium på dag 4 eller dag 5. Vi leverer interne data (tabel S1), der viser, at vores to metoder (mulighed 1 og mulighed 2) har god konsistens sammenlignet med PGT-A, hvilket er bedre end prøveudtagningsmetoden uden grundig fjernelse af CC.
IF-amplificerede produkter optrådte i den negative kontrol, og eksterne DNA-materialer kan have forurenet reagenset eller arbejdsområdet. Arbejdsområdet skal rengøres med DNA/RNA-fjernende reagenser, nukleasefrie materialer skal anvendes, og reagenserne skal alikveres efter første brug.
Forskellene i succesraterne mellem løsningsmodel 1 og løsningsmodel 2 behandles i tabel S1 og tabel S2.
Begrænsninger af NICS-analysen
Der er to hovedbegrænsninger ved NICS. 1) Før ICSI skal alle cumulusceller (normalt moderens oprindelse, normalt normal kromosomsammensætning) fjernes. Hvis fjernelsen er ufuldstændig, kan cumuluscellerne frigive DNA under embryoudviklingen, og det eksterne DNA amplificeres, hvilket kan være årsagen til falsk negativ detektion. 2) Det er vanskeligt at fjerne sædcellerne, der er knyttet til zona pellucida, og NICS-proceduren anbefales stærkt at udføres med ICSI. Selvom regelmæssig udskiftning af spaltningsmedier på dag 3 kan reducere muligheden for kontaminering på grund af cumulusceller og overflødige sædceller, skal denne forurening minimeres, hvis NICS anvendes i klinisk IVF. Imidlertid er der udviklet en metode til påvisning af NICS i IVF-embryoner, herunder funktionen til at genkende eksogent DNA, som vil blive demonstreret i den nærmeste fremtid.
Denne undersøgelse sammenlignede ikke forskellene mellem forskellige medier, da store kliniske forsøg har sammenlignet kulturmedier. Otte centre brugte 4 forskellige kulturmedier, sekventielle og kontinuerlige, og 2 forskellige procentdele af albumintilskud (5% og 10%), og disse forskelle havde ikke signifikante virkninger på nøjagtigheden af embryonale cfDNA-resultater31. Disse resultater understøtter den potentielle anvendelighed af embryonal cfDNA-analyse til hvert IVF-laboratorium, når man arbejder under den specifikke protokol.
Betydning i forhold til eksisterende metoder
NICS-metoden undgår embryobiopsi og forbedrer dermed sikkerheden ved brug betydeligt. Sammenlignet med blastocyster er NICS en enkel, tidsbesparende, følsom og reproducerbar præimplantationsscreeningsteknik, der er velegnet til assisterede reproduktive populationer med stor sandsynlighed for aneuploidi. I modsætning til invasiv biopsi, som kræver betydelig og faglig viden til blastocystbiopsiproceduren, kan NICS anvendes i vid udstrækning, da dens enkle indsamling af brugt medium kun følger den regelmæssige drift af IVF19 , og det kræver ikke PGS / PGD-kvalifikation i nogle lande.
Fremtidige applikationer
NICS har potentiale til bred anvendelighed til kromosomscreening i klinisk IVF, ikke kun for ICSI, men også for IVF-embryoner. Selvom ICSI anbefales stærkt, er metoder til fjernelse af sædcellerne, der er knyttet til zona pellucida, nødvendige for at forhindre påvirkning af sædceller.
Morfologisk vurdering er en traditionel metode til embryoevaluering, men i de fleste tilfælde kan kromosomalt unormale embryoner forekomme morfologisk svarende til kromosomalt normale (euploide) embryoner. Kombination af morfologisk vurdering med NICS-analysen ved overførsel af ploide embryoner med god morfologi i livmoderen kan forbedre de igangværende graviditetsrater og levende fødselsrate. Et randomiseret klinisk forsøg vil blive udført for at evaluere den kliniske effekt af enkelt embryooverførsel ved hjælp af NICS-teknologi.
Kritiske trin i protokollen
Alle cumulus-corona radiata-celler skal fjernes fra oocytterne inden befrugtning. Oocytter blev befrugtet ved intracytoplasmatisk sædinjektion (ICSI). Tilsætning af humanafledte proteiner/kosttilskud til dyrkningsmediet blev undgået. Dyrkningsmediet blev skiftet på dag 4 og indsamlet på dag 5-dag 6, når blastocysterne var fuldt udvidet. Embryoner blev dyrket i individuelle dråber af dyrkningsmedium begyndende på dag 4. Ved indsamling af dyrkningsmediet blev overførselspipetter mellem prøver ændret for at undgå kontaminering.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yaxin Yao, Jieliang Ma, Jing Wang og Sijia Lu er ansatte i Yikon Genomics Co., Ltd.
Acknowledgments
Forfatterne vil gerne takke Shiping Bo og Shujie Ma for deres hjælp med NGS-dataanalyse. Finansiering: Dette arbejde blev støttet af National Key Research and Development Program (bevilling nr. 2018YFC1003100).
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
References
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Jr Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T. Jr, Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid? Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- Thermo Fisher Scientific. Qubit dsDNA HS Assay Kit. , Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015).
- Miseq system use guide. , Available from: https://support.illumina.com/downloads/miseq_system user _ guide 15027617.html (2016).
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Tags
Kromosomscreening Human præimplantationsembryoner brugt dyrkningsmedium prøveindsamling kromosomal ploidianalyse in vitro-befrugtning (IVF) præimplantationsgenetisk test for aneuploidier (PGT-A) trophectoderm (TE) indre cellemasse (ICM) falske positive og negative NICS-teknologi DNA-sekventering prøveudtagning af kulturmedium helgenomamplifikation (WGA) biblioteksforberedelse NGS-dataanalyse kryopræservering IVF-laboratoriumErratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021.
Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
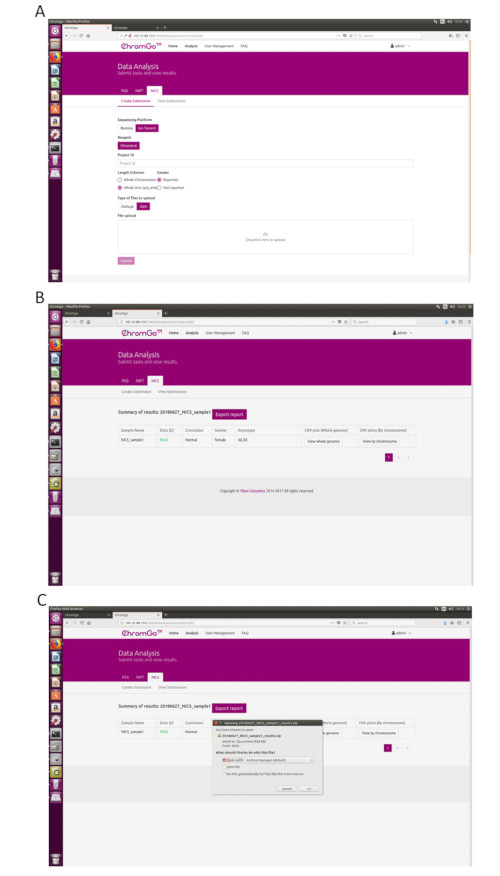
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
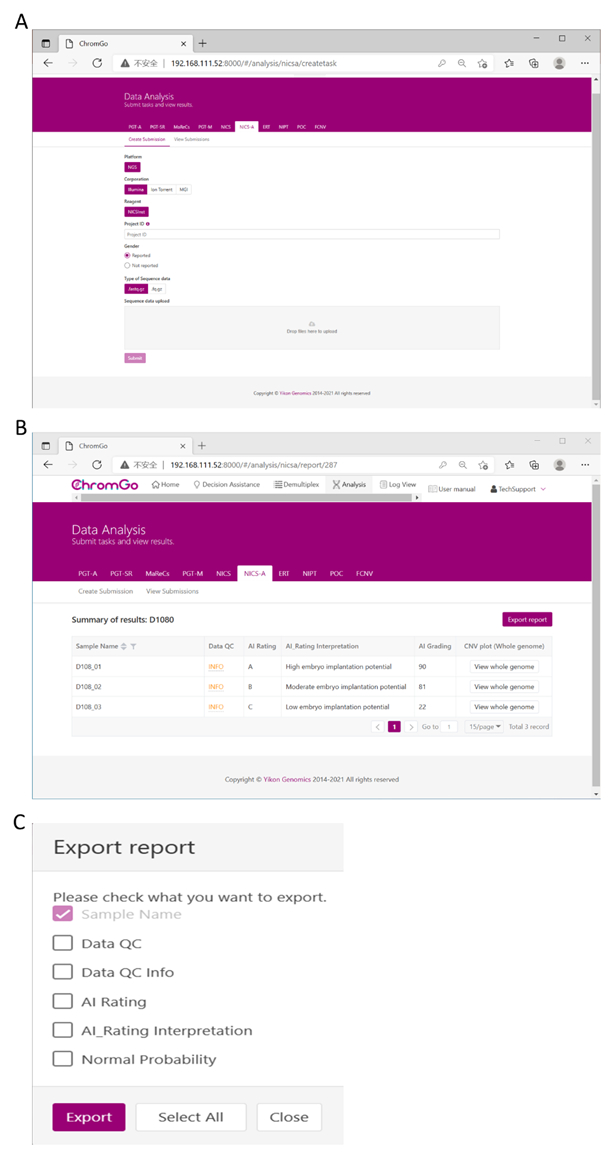
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.



