ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
본 연구는 배아 생검을 피하고 NGS를 사용하여 염색체 ploidy 식별을 가능하게 하는 폐배양 배지를 사용하는 인간 배아의 염색체 스크리닝 프로토콜을 보고합니다. 본 논문에서는 배양액 제조, 전장 유전체 증폭(WGA), 차세대 염기서열분석(NGS) 라이브러리 준비, 데이터 분석 등 자세한 절차를 제시한다.
Abstract
임상 체외 수정(IVF)에서 PGT-A의 일반적인 방법은 영양배엽(TE)에서 몇 개의 세포를 생검해야 합니다. 이것이 태반을 형성하는 계통입니다. 그러나 이 방법은 전문 기술이 필요하고 침습적이며 TE의 염색체 수와 태아로 발달하는 내부 세포 질량(ICM)이 항상 동일하지 않기 때문에 위양성 및 음성이 발생합니다. TE 및 ICM에서 배양 배지로 방출된 DNA의 염기서열 분석이 필요한 기술인 NICS는 이러한 문제를 해결할 수 있는 방법을 제공할 수 있지만 이전에는 효능이 제한적인 것으로 나타났습니다. 본 연구는 배양 배지 샘플링 방법, 전체 게놈 증폭(WGA) 및 라이브러리 준비, 분석 소프트웨어에 의한 NGS 데이터 분석을 포함하는 NICS의 전체 프로토콜을 보고합니다. 배아 실험실마다 동결 보존 시간이 다르다는 점을 고려하여 발생학자는 IVF 실험실의 실제 조건에 따라 선택할 수 있는 배아 배양 배지를 수집하는 두 가지 방법을 가지고 있습니다.
Introduction
보조 생식 기술(ART)은 불임 치료에 점점 더 많이 사용되고 있습니다. 그러나 IVF와 같은 ART의 성공률은 제한적이며, 임신 손실률은 정상 인구에 비해 현저히 높다1. 이러한 문제의 주요 원인은 착상 전 인간 배아에 일반적으로 존재하는 염색체 이상이다2. PGT-A는 착상 전에 배아의 염색체 균형을 스크리닝하는 효과적인 방법입니다 3,4. 일부 연구에서는 PGT-A가 낙태율을 낮추고 임신율을 향상시킬 수 있음을 입증했습니다 5,6,7,8. 그러나 PGT-A에는 특정 교육과 경험이 필요한 복잡한 기술 전문 지식이 필요합니다. 침습적 배아 생검 절차는 또한 잠재적으로 배아에 손상을 줄 수 있다9. 연구에 따르면 할구 생검은 후속 발달을 방해할 수 있으며, 생검된 TE의 수가 착상율에 영향을 미칠 수 있다10. 배아 생검의 장기적인 생물안전성 문제는 아직 인간을 대상으로 철저하게 평가되지 않았지만, 동물 연구에서는 배아 발달에 부정적인 영향을 미치는 것으로 나타났다11,12,13.
이전 보고에 의하면 배아 발달 과정에서 미량의 DNA 물질이 배양 배지로 분비되었으며, 사용된 배아 배양 배지 14,15,16,17,18을 사용하여 포괄적인 염색체 스크리닝(CCS)을 수행하려는 노력이 이루어졌다. 그러나 검사의 검출률과 정확도는 광범위한 임상 사용에 대한 요구 사항을 충족하지 못했습니다. 본 연구는 NICS 테스트의 정확도뿐만 아니라 검출률을 높이기 위한 NICS 분석의 개선을 보고하였다19. 최근 몇 년 동안 배반체액(BF)은 최소 침습 PGT-A의 분석 샘플로 연구되었습니다. 그러나 배반포액 샘플에서 성공적인 게놈 전체 증폭 및 검출 가능한 DNA의 비율은 34.8%에서 82%에 이릅니다20,21,22. 다양한 연구에서 보고된 BF의 부피는 0.3nL에서 1μL 사이입니다. BF에서 DNA의 양이 적다는 점을 고려하여, 배반포액과 배양 배지를 혼합하여 cell-free DNA의 양을 증가시켜 검출의 성공률과 일관성을 향상시킬 수 있습니다. Kuznyetsov 외.23 및 Li et al.24는 zona pellucida를 레이저로 처리하고 배반포액을 배양 배지에 방출하여 배아 DNA의 총량을 개선시켰으며, WGA 후 결합된 배지/BF 샘플의 증폭률은 각각 100% 및 97.5%였습니다. Jiao et al.25도 동일한 방법을 사용하여 100% 증폭 성공률을 얻었습니다.
본 연구는 사용한 배지 샘플 준비, NGS 준비 및 데이터 분석을 포함하는 자세한 프로토콜을 보고합니다. 본 연구는 난모세포에서 적운 세포를 조심스럽게 제거하여 세포질 내 단일 정자 주입(ICSI) 및 배반포 배양을 수행했습니다. 4일-5일/6일 소요 배지를 WGA 및 NGS 라이브러리 준비를 위해 수집했습니다. 본 연구는 NICS 기술을 사용하여 약 3시간 만에 WGA 및 NGS 라이브러리 준비 단계를 간소화하고 약 9시간 만에 비침습적으로 CCS 결과를 얻었습니다.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
북경대학 제3병원 윤리위원회로부터 윤리 허가를 받았다.
1. 준비
알림: 필요한 재료 및 장비는 재료 표에 나열되어 있습니다.
- 시약
- 사용하기 전에 Tri-gas 인큐베이터에서 37°C, 5% CO 2 및 5% O2에서 20-30μL의 배우자 배지/수정 배지 및 분열/배반포 단계 배양 배지(미네랄 오일로 덮임) 및 히알루로니다아제(단단히 뚜껑이 있는 튜브에 있음)를 예열 및 평형(균형)합니다.
- 흄 후드의 작업 표면에서 히알루로니다아제를 37°C로 예열합니다.
- 제조업체의 지침에 따라 유리화 완충액과 시료 채취 시약을 준비합니다.
- 도구
- 유리 파스퇴르 피펫을 당겨 화염 광택 개방형 미세 팁을 생성하여 샘플 수집 및 이송 피펫(내경 ~200-250μm), 탈각/스트리퍼 피펫(내경 ≥150μm, ~130-140μm 및 ~120μm) 및 세척용 피펫(내경 ~150μm)을 준비합니다.
참고: 샘플 채취/이송, 탈취 및 세척에 사용되는 피펫은 직접 구입할 수 있습니다. 고정 바늘과 주사 바늘도 직접 구입할 수 있습니다.
- 유리 파스퇴르 피펫을 당겨 화염 광택 개방형 미세 팁을 생성하여 샘플 수집 및 이송 피펫(내경 ~200-250μm), 탈각/스트리퍼 피펫(내경 ≥150μm, ~130-140μm 및 ~120μm) 및 세척용 피펫(내경 ~150μm)을 준비합니다.
2. 프로토콜 1: 검체 채취
- 히알루로니다아제로 소화하기 전에 난모세포-코로나-적운 복합체(OCCC)의 전처리
- 난포 자극 호르몬(FSH)과 인간 갱년기 성선 자극 호르몬(hMG) 제제로 난소 자극을 달성합니다. 납 난포가 >18mm일 때 최종 난모세포 성숙을 위해 10,000IU의 융모성 성선 자극 호르몬(hCG)을 사용합니다.
- 방아쇠 주사 후 36시간 후에 난모세포 채취를 수행합니다. 난모세포를 채취하여 미네랄 오일로 덮인 2.5mL의 예열된 m-HTF가 있는 조직 배양 접시에 옮깁니다.
- 이송 피펫을 사용하여 OCCC를 1mL의 수정 배지가 들어 있는 장기 배양 접시의 중앙 웰로 빠르게 옮긴 다음 37°C에서 5% CO2 및 5%O2 인큐베이터에서 2-4시간 동안 난모세포와 함께 배양합니다.
- OCCC가 포함된 장기 배양 접시의 중앙 웰에 37°C 예열된 히알루로니다아제(80IU/mL) 1mL를 추가하여 히알루로니다아제로 OCCC를 분해합니다(단계 2.1.3). 히알루로니다아제의 최종 농도를 40IU/mL로 유지하고 잘 섞습니다.
- OCCC를 37°C 열 플랫폼에서 2분 동안 배양합니다. 과립 세포의 1-2 층 만 남을 때까지 30 초마다 현미경으로 변화를 관찰하십시오.
- 과립 세포의 탈질
- 난모세포 처리를 위해 배양 접시에서 소화된 OCCC를 빠르게 옮기고 각 웰에 미네랄 오일을 덮습니다.
- 분리된 과립 세포를 현미경으로 관찰합니다. 부드럽게 흡인하여 난모세포를 5회 방출하여 난모세포 주변에 남아 있는 과립세포를 제거합니다.
- 나머지 3개의 웰에서 이전 단계를 반복하여 과립 세포를 완전히 제거합니다.
알림: 위의 단계(2.1-2.3)는 각 실험실의 일상적인 작업에 따라 수행할 수 있습니다.
- 난모세포의 평가
- 현미경을 사용하여 과립 세포 제거의 완전성을 평가합니다. 세포를 완전히 제거할 수 없는 경우, 이때 5개 이하의 과립 세포의 머무름이 허용됩니다.
참고: 적운 세포가 여전히 난모세포에 부착되어 있는 경우, 배아를 분열 단계 배양 배지에서 배반포 단계 배양 배지로 옮기기 전에 3일째 되는 날에 잔여물을 제거할 수 있습니다.
- 현미경을 사용하여 과립 세포 제거의 완전성을 평가합니다. 세포를 완전히 제거할 수 없는 경우, 이때 5개 이하의 과립 세포의 머무름이 허용됩니다.
- 세포질 내 정자 주입(ICSI)26을 수행한 후 이송 피펫을 사용하여 난모세포를 20-30μL 분열 배아 배지 미세방울(하나의 난모세포는 하나의 미세방울에 해당)로 옮기고 37°C, 5% CO2 및 5%O2 인큐베이터에서 배양합니다.
- ICSI의 날을 0일로 기록합니다. 수정 1일차(약 18시간), 2일차(약 45시간) 및 배아 분열 3일차(약 68시간)의 배아 평가에 대한 이스탄불 합의 워크숍에 따라 배아를 확인하고 점수를 매깁니다.27.
- 배아 세척
- 37 °C, 5 % CO 2 및 5 % O 2 인큐베이터에서2 일째에 조직 배양 접시에서 미네랄 오일로 덮인 각 배아에 대해 20-30 μL의 배반포 배양 배지 미세 방울을 준비합니다.
- 미네랄 오일로 덮인 또 다른 3개의 미세 방울을 준비하고 1-3번 세척을 위해 새 조직 배양 접시에 라벨을 붙입니다.
- 3일째 배아를 세척 미세 방울로 옮깁니다. denudation 피펫을 사용하여 각 방울에서 배아를 3회 부드럽게 흡인하고 방출합니다.
참고: 이 절차는 또한 배아에 부착된 잔류 과립 세포를 제거하는 데 도움이 될 수 있습니다. - 형태학적 채점을 위해 배지를 절단 단계 배양 배지에서 배반포 배양 배지로 변경하기 전 3일째에 현미경으로 배아를 관찰하고 평가합니다. 적운 세포가 여전히 배아에 부착되어 있는 경우, 적운 세포가 완전히 제거될 때까지 스트리퍼 피펫으로 미네랄 오일로 덮인 다른 예열되고 평형화된 배반포 배양 배지 방울에서 위아래로 적절하게 피펫팅합니다.
참고: 배아를 분열 단계 배양 배지 플레이트에서 배반포 단계 배양 배지 플레이트로 옮기기 전에 부착된 모든 적운 세포를 3일째에 완전히 제거해야 했습니다. 남아 있는 적운 세포는 최종 분석을 방해하고 위음성 결과를 제공합니다.
- 배양 배지 수집을 위한 두 가지 옵션
참고: IVF 센터는 센터의 자원, 요구 사항 및 선호도에 따라 두 가지 배양 배지 수집 방법 중 하나를 선택할 수 있습니다.- 옵션 1: 배아 세척 및 배양
참고: 이 옵션은 5일차 아침에 유리화를 수행하는 IVF 실험실을 위한 것입니다.- 배아를 예열된(37°C) 배양 배지의 미세 방울에 옮기고 4일째 오후에 피펫팅하여 각 배아를 3개의 미세 방울로 연속적으로 부드럽게 세척합니다.
- 샘플 수집을 위해 각 배아를 고유한 예열된(37°C) 단일 microdroplet 배양 배지에 옮깁니다. 배양 배지 단일 방울의 부피는 25μL를 초과할 수 없습니다.
- 5일차/6일차에 37°C, 5% CO2 및 5%O2에서 배반포 배아 배양을 수행합니다.
- 옵션 2: 배아 세척 및 배양
참고: 이 옵션은 5일차 오후 또는 6일차에 유리화(vitrification)를 수행하는 IVF 실험실을 위한 것입니다.- 배아를 10-15 μL 배양 배지의 예열된(37°C) 미세방울에 옮기고 5일째에 피펫팅하여 각 배아를 3개의 미세방울로 순차적으로 부드럽게 세척합니다.
- 샘플 수집을 위해 각 배아를 고유한 예열된(37°C) 단일 microdroplet 배양 배지에 옮깁니다. 배양 배지 단일 방울의 부피는 15μL를 초과할 수 없습니다.
- 5일차/6일차에 37°C 및 5%CO2에서 배반포 배아 배양을 수행합니다.
- 옵션 1: 배아 세척 및 배양
- 시료 채취
- 영양배엽의 세포 접합부에 초점을 맞춘 레이저 빔의 목표 지점에서 상당한 거리에서 ICM을 부드럽게 조정하여 영양배엽에 작은 구멍을 생성하여 배반강에서 유체를 방출합니다. 그런 다음 배아는 기존 공정에 따라 냉동 보존을 위해 냉동 용액으로 이동합니다.
- 배양된 각 배아에서 5μL의 세포 용해 완충액이 포함된 RNase/DNase-free PCR 튜브로 배양 배지를 옮깁니다.
- 배아 배양에 사용하지 않고 음성 대조군과 동일한 양의 배양 배지를 수집합니다. 수집된 모든 샘플을 액체 질소에 즉시 동결한 다음 수집 후 NICS 분석이 수행될 때까지 -80°C에서 보관합니다.
- 프로토콜에 설명된 대로 유리화를 수행합니다.
3. 프로토콜 2: 도서관 구축
- 배양 배지 용해
- 1μL의 양성 대조군(10ng 인간 gDNA)을 199μL의 신선한 배양 배지로 희석합니다. 완전히 혼합하고 튜브를 짧게 원심분리합니다(200초 동안 5 x g ).
- 10μL의 5일차 6일 배반포 배양 배지, 희석된 양성 대조군 및 신선한 배양 배지를 새로운 0.2mL PCR 튜브로 옮깁니다.
- 각 PCR 튜브에 1μL의 MT Enzyme Mix를 추가하고 200 x g에서 2-3초 동안 피펫팅 및 원심분리기로 완전히 혼합합니다.
- 3.1.3 단계의 PCR 튜브를 예열된 NICS 샘플 준비 스테이션에 넣고 다음과 같이 용해 프로그램을 실행합니다. 75°C에서 10분; 95°C에서 4분; 22 °C에서 유지합니다.
참고: 시료 전처리 스테이션은 표준 PCR 기계와 비슷합니다.- Lysis 아이콘을 클릭하여 설정 화면으로 들어갑니다.
- 제어 모드로 튜브를 선택하십시오. 시료 부피의 경우 입력 10μL; Hotlid 제어에 대해 On 을 선택하고 온도에 105°C를 입력합니다. 첫 번째 세그먼트에서 일시 중지에 대해 아니요 를 선택합니다. 딸깍 하는 소리 OK 계속합니다.
- 남아 있는 시간에 프로그램 종료를 나타내는 --:--:--가 표시될 때까지 기다린 다음 중지를 클릭하여 프로그램을 종료합니다.
- 프로세스가 완료된 후 프로그램을 중지하십시오. 즉시 다음 단계로 진행하십시오.
- 도서관 사전 준비
- Pre-Lib 버퍼를 RT로 해동합니다. 피펫팅과 원심분리기로 200 x g에서 즉시 2-3초 동안 완전히 혼합합니다.
- Pre-Library 반응을 위한 마스터 믹스를 준비하려면 Pre-Lib Enzyme Mix 2μL를 Pre-Lib Buffer 60μL에 넣고 반응을 완전히 혼합한 후 간단히 원심분리합니다.
- 60μL의 사전 라이브러리 반응 혼합물을 이전 단계의 각 전처리된 배지 샘플에 추가합니다. 피펫팅과 원심분리기로 즉시 200 x g에서 2-3초 동안 완전히 혼합합니다.
- 3.2.3 단계의 PCR 튜브를 Sample Prep Station에 놓고 다음과 같이 prelibrary 프로그램을 실행합니다. 95°C에서 2분; 40초 동안 15°C, 40초 동안 22°C, 30초 동안 33°C, 30초 동안 65°C, 40초 동안 72°C, 10초 동안 95°C, 10초 동안 63°C의 12회 주기; 4 °C에서 유지합니다.
- Pre_Lib 아이콘을 클릭하여 설정 화면으로 들어갑니다.
- 제어 모드로 튜브를 선택하십시오. 샘플 부피의 경우 입력 70 μL; Hot lid control(핫 뚜껑 제어)에서 On(켜기 )을 선택하고 온도로 105°C를 입력합니다. 첫 번째 세그먼트에서 일시 중지에 대해 아니요 를 선택합니다. 딸깍 하는 소리 OK 계속합니다.
- 남은 시간에 프로그램 종료를 나타내는 --:--:--가 표시될 때까지 기다렸다가 중지를 클릭하여 프로그램을 종료합니다.
- 프로세스가 완료되면 프로그램을 중지하십시오. 즉시 다음 단계로 진행하십시오.
- 도서관 준비
- 라이브러리 버퍼를 RT로 해동합니다. 피펫팅과 원심분리기로 완전히 혼합하고 즉시 200 x g에서 2-3초 동안 혼합합니다.
- 라이브러리 반응을 위한 마스터 믹스를 준비하려면 1.6 μL의 Library Enzyme Mix를 60 μL의 Library Buffer에 넣고 반응을 완전히 혼합한 후 간단히 원심분리합니다.
- 60 μL의 라이브러리 반응 혼합물과 2 μL의 바코드 프라이머를 3.2.3단계부터 각 prelibrary 제품에 추가합니다. 반응을 완전히 혼합하고 짧게 원심분리합니다.
- 3.2.3 단계의 PCR 튜브를 열 순환기에 놓고 다음과 같이 라이브러리 준비 프로그램을 실행합니다: 94 °C 동안 30초; 17초 동안 94°C, 62초 동안 30°C, 72초 동안 45°C의 45회 주기); 그런 다음 4°C에서 유지합니다.
- Lib_Prep 아이콘을 클릭하여 설정 화면으로 들어갑니다.
- 제어 모드로 튜브를 선택하십시오. 시료 부피의 경우 입력 130 μL; Hotlid 제어에 대해 On 을 선택하고 해당 온도에 대해 105°C를 입력합니다. 첫 번째 세그먼트에서 일시 중지에 대해 아니요 를 선택합니다. 딸깍 하는 소리 OK 계속합니다.
- 남은 시간에 프로그램 종료를 나타내는 --:--:--가 표시될 때까지 기다렸다가 중지를 클릭하여 프로그램을 종료합니다.
- 도서관 정화
- 정제 단계 전에 최소 20분 동안 2-8°C에서 보관하는 Magbeads를 꺼냅니다. Magbeads를 20초 동안 소용돌이치고 섞습니다. 정제 단계에 필요한 충분한 비드를 새로운 1.5mL 마이크로 원심분리기 튜브에 분주하고 비드를 RT로 데웁니다.
- 각 라이브러리에 1x Magbeads를 추가합니다. 위아래로 ≥10회 피펫팅하여 혼합하고 실온에서 5분 동안 배양합니다.
참고: 예를 들어, 100μL의 Magbeads를 100μL의 라이브러리 샘플에 추가합니다. - 배양 후 튜브를 잠시 원심분리하고 자석 스탠드에 놓습니다.
- 용액이 맑아질 때까지 약 5분 동안 기다립니다. 튜브를 마그네틱 스탠드에 올려 놓은 상태에서 용액을 조심스럽게 흡입하고 버리십시오.
- 갓 준비한 80% 에탄올 200μL를 튜브에 추가합니다. 상온에서 30초 동안 배양하고 상층액을 조심스럽게 제거합니다. 한 번 더 반복합니다.
- 가능한 한 완전히 에탄올을 제거하십시오. 마그네틱 스탠드의 비드를 RT에서 약 5-10분 동안 자연 건조합니다.
- 마그네틱 스탠드에서 튜브를 제거하고 17.5μL의 용출 버퍼를 추가한 다음 튜브를 소용돌이쳐 비드를 재현탁시킵니다. 튜브를 잠시 원심분리하고 실온에서 5분 동안 배양합니다.
- 튜브를 마그네틱 스탠드에 놓고 용액이 투명해질 때까지 기다립니다. 15μL 상층액을 새 튜브로 조심스럽게 옮깁니다.
- 라이브러리 정량화
- qubit dsDNA HS 어세이 키트28의 사용자 가이드에 따라 형광측정기를 사용하여 정제된 라이브러리를 정량화합니다. 라이브러리의 수율은 ~ 15에서 300 ng 사이입니다.
- 라이브러리 풀링
- 풀링을 위해 각 라이브러리 샘플의 10나노그램을 사용합니다.
- 시퀀싱
- 시퀀싱 사용 설명서(15027617 v01)29를 참조하십시오.
- 플랫폼의 단일 말단에서 50bp의 정제된 라이브러리 염기서열은 각 샘플에 대해 약 200만 개의 판독을 생성했으며 0.03 × 염기서열분석 깊이가 권장되었습니다.
- 데이터 분석
- 로그인 페이지에 사용자의 이름과 암호를 입력합니다.
- 시스템에 로그인한 후 분석을 클릭하면 새 페이지가 나타납니다. NICS-A 탭에서 Create Submission(제출 생성 )을 클릭합니다. 그런 다음 플랫폼으로 NGS 를 선택하고, 기업을 선택하고, 시약으로 NICSInst를 선택하고, 프로젝트 ID 아래의 상자에 프로젝트 정보를 입력하고, 분석 기본 설정을 지정하고, 파일을 업로드합니다. 모든 염기서열분석 파일이 성공적으로 업로드되면 Submit(제출 )을 클릭하여 분석을 시작합니다(그림 3A).
- 제출물 보기를 클릭하여 제출된 프로젝트 목록을 표시합니다. 분석이 완료되면 프로젝트 상태가 완료됨이 되고 보고서 필드에 표시 버튼이 나타납니다. Show(표시) 버튼을 클릭하여 NICS 분석의 요약 테이블을 봅니다(그림 3B).
- Export report(보고서 내보내기) 단추를 클릭하여 보고서를 저장합니다(그림 3C).
참고: 각 분석에 대해 세 가지 유형의 파일이 내보내집니다. 각 염색체 및 전체 게놈에 대한 모든 복제 수 변이(CNV) 플롯을 포함하는 그래픽 파일로, "그래프" 폴더에 저장됩니다. 이 분석 실행의 샘플 QC 세부 정보가 포함된 스프레드시트; 사용자가 사용자 지정한 NICS 보고서가 포함된 문서 파일입니다. 및 이 분석 실행의 샘플 요약 정보가 포함된 스프레드시트입니다.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
본 연구는 제안된 방법을 환자에게 적용하였다. NICS 분석을 적용하기 전에 IRB 승인 및 사전 동의를 받았습니다. 본 연구는 환자로부터 6개의 배반포를 채취하고 4일차에서 5일차에 걸쳐 6개의 배아 모두에 대해 NICS를 수행했습니다. 부모의 균형 전위로 인한 염색체 이상은 NICS 분석으로 5개의 염색체에서 발견되었습니다. 따라서 전송에 사용할 수 없습니다(그림 4A-E). 두 배아의 NICS 결과는 동일한 핵형 45를 보여주었고, XN 및 -18(×1)은 모두 18번 염색체 결실이었습니다(그림 4A, B). 핵형 46, XN, -1p(pter→p21.1, ×1)는 염색체 1 pter→p21.1 영역 결실의 짧은 팔일 뿐입니다(그림 4D).
NICS 결과는 핵형 46, XN, +1p(pter→p21.2, ×3) 및 -18(q21.32→qter, ×1)을 보여주었으며, 이는 18번 염색체 q21.32→qter 영역 결실의 긴 팔과 1번 염색체 pter→p21.2 영역의 짧은 팔이 모두 복제되었음을 나타냅니다(그림 4E). 핵형 46, XN, +5q(×4) 및 -8(×1, mos)은 5번 염색체 복제이며 8개의 모자이크 차이를 보이지만 NICS 분석은 24개 염색체 모두를 이수성으로 스크리닝할 수 있습니다. 이 과정은 단일 정상 핵형 배반포를 옮기는 새로운 방법을 제공합니다.

그림 1. 적운 세포의 제거 완전성. (A) 적운 세포가 있는 난모세포. (B) 적운 세포가 부착되지 않은 난모세포. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2. 적운 세포는 BM으로 전달되기 전에 D3의 배아에서 제거됩니다. 부착된 모든 적운 세포는 초기 분열 배아 배양 배지 플레이트에서 배아가 8세포 단계에 도달한 후 3일째인 배반포 배양 배지 플레이트로 배지가 변경되기 전에 제거해야 합니다. 제거되지 않은 적운 세포는 최종 분석을 방해하여 위음성 결과를 제공합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3. 데이터 분석. (A) 사용자 응용 프로그램에는 다양한 옵션이 있습니다. 시퀀싱 플랫폼 회사의 경우 사용자는 Illumina, Ion Torrent 또는 MGI를 선택할 수 있습니다. 사용자는 성별 정보를 보고할지 여부를 선택할 수 있습니다. 위의 매개 변수 설정을 완료하고 파일 업로드 아래의 상자를 클릭한 다음 업로드할 적절한 시퀀싱 파일을 선택합니다. Illumina의 경우 확장자가 fastq.gz 인 파일을 선택하십시오. 업로드에 성공한 후 Submit(제출)을 클릭하여 분석을 시작합니다. (B) 요약 테이블의 보기입니다. 요약 테이블은 다음 정보로 구성됩니다. 샘플 이름: 각 NICS 샘플의 이름이 나열됩니다. 데이터 QC: 시퀀싱 파일이 NICS 분석을 위한 QC를 통과하는지 여부를 나타냅니다. AI 등급: 각 NICS 샘플에 대한 등급(A, B 또는 C)입니다. AI_Rating 해석: 배아 착상 가능성 평가; AI 등급: 각 NICS 샘플에 대한 점수입니다. CNV 플롯(전체 게놈): 모든 염색체의 CNV 프로필을 봅니다. (C) 보고서 저장 페이지. Summary of Results(결과 요약) 옆에 있는 Export report(보고서 내보내기) 버튼을 클릭합니다. 최종 보고서에 표시할 정보를 선택하고 내보내기를 클릭합니다. 보고서는 컴퓨터의 다운로드 폴더에 저장됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4. 환자의 NICS를 사용한 배아 스크리닝 및 선택. 총 6개의 배아가 배반포 단계까지 성공적으로 발달했으며, 각 배아에서 Day4-Day5 배양 배지를 수집하여 NICS 분석을 수행했습니다. (A)와 (B)는 동일한 핵형 45, XN, -18(×1)이 모두 18번 염색체 결실임을 보여주는 두 배반포 배아의 NICS 결과입니다. (C)는 핵형 46, XN, +5q(×4), -8(×1, mos)이 5번 염색체 복제 및 8번 모자이크임을 보여주었다. (D)는 핵형 46, XN, -1p(pter→p21.1, ×1)은 1번 염색체 pter→p21.1 영역 결실의 짧은 팔일 뿐인 반면, (E)는 핵형 46, XN, +1p(pter→p21.2, ×3), -18(q21.32→qter, ×1)은 1번 염색체 pter→p21.2 영역 복제의 짧은 팔이고, qter 영역 (F)→ 18번 염색체 q21.32의 긴 팔은 균형 잡힌 염색체 조성을 보였다. x축은 빨간색과 파란색으로 표시된 22개의 상염색체를 의미하며, y축은 각 상염색체의 복사 번호를 나타냅니다. 회색 점은 복사 번호 응답의 눈금자 눈금자이며 각 빈 창과 복사 번호의 일반 핵형은 2여야 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 S1. DNA 검출 옵션 1 및 옵션 2의 성공률. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 S2. 다양한 옵션에서 NICS와 PGT-A 간의 일치. 이 표를 다운로드하려면 여기를 클릭하십시오.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
수정 및 문제 해결
NICS 결과가 부모의 유전 물질로 오염된 경우 모든 적운-코로나 방사능 세포를 제거하고 수정을 위해 ICSI를 수행해야 합니다. DNA를 저하시킬 수 있는 부적절한 배지 보관 또는 템플릿 준비 프로세스를 피할 수 있습니다. 작업 공간은 DNase 및 RNase 오염 제거 시약으로 철저히 정제되었습니다. 다른 배아로부터의 오염을 피하기 위해, 4일째부터 시작되는 교차 오염을 피하기 위해 하나의 배아를 항상 배지의 단일 방울에서 배양했습니다. 오염 현상은 최종 배양 드롭에서 배아의 배치를 지연시킬 때 최소화됩니다30,31,32,33. 모계 오염을 최소화하기 위해 Kuznyetsov34는 피펫팅(pipetting) 및 플러싱(flushing)을 통해 잔류 코로나 세포를 조심스럽게 제거하는 것을 포함하여 배아 배양 절차를 0일에서 4일째로 수정했습니다.
Lane et al.30 은 4일째부터 5일째까지 배아 배양 배지를 복용할 때 유배체 검출의 정확도가 향상되고 배아 다수체 일관성이 95% 이상이며 성염색체의 일관성이 100%에 도달한다는 것을 보여줍니다. Lledo et al.33 은 배아를 4일째에서 6일째까지 배양했을 때 3일 5일 배양 배지와 TE 샘플 사이의 일치율이 74.6%와 92.0%라는 것을 발견했습니다.
당사의 내부 데이터도 표 S1에서 볼 수 있듯이 이 결론을 뒷받침합니다. 종래의 3일-5일째 배양법과 비교하여, 4일째 또는 5일째에 배양액의 한 번 더 변화로 인해 과립세포를 추가로 제거하였다. 두 가지 방법(옵션 1 및 옵션 2)이 PGT-A에 비해 일관성이 우수하다는 것을 보여주는 내부 데이터(표 S1)를 제공하며, 이는 CC를 완전히 제거하지 않은 샘플링 방법보다 우수합니다.
IF 증폭 산물이 음성 대조군에 나타났으며, 외부 DNA 물질이 시약이나 작업 공간을 오염시켰을 수 있습니다. 작업 공간은 DNA/RNA 제거 시약으로 세척해야 하고, 뉴클레아제가 없는 물질을 사용해야 하며, 시약은 처음 사용한 후 분주해야 합니다.
옵션 1과 옵션 2 간의 성공률 차이는 표 S1 및 표 S2에 설명되어 있습니다.
NICS 분석의 한계
NICS에는 두 가지 주요 제한 사항이 있습니다. 1) ICSI 전에 모든 적운 세포(일반적으로 모계 기원, 일반적으로 정상 염색체 구성)를 제거해야 합니다. 제거가 불완전하면 적운 세포가 배아 발달 중에 DNA를 방출하고 외부 DNA가 증폭되어 위음성 검출의 원인이 될 수 있습니다. 2) zona pellucida에 부착된 정자를 제거하는 것은 어렵기 때문에 ICSI로 NICS 시술을 수행하는 것이 좋습니다. 3일째에 분열 배지를 정기적으로 교체하면 적운 세포 및 중복 정자로 인한 오염 가능성을 줄일 수 있지만, 임상 IVF에서 NICS를 사용하는 경우 이러한 오염을 최소화해야 합니다. 그러나 IVF 배아에서 NICS를 검출하는 방법이 개발되었으며, 여기에는 외인성 DNA를 인식하는 기능이 포함되며 가까운 장래에 입증될 예정입니다.
이 연구는 대규모 임상시험에서 배양 배지를 비교했기 때문에 서로 다른 배지 간의 차이를 비교하지 않았습니다. 8개 센터는 4개의 서로 다른 배양 배지(순차적 및 연속적)와 2가지 다른 비율의 알부민 보충(5% 및 10%)을 사용했으며, 이러한 차이는 배아 cfDNA 결과의 정확도에 유의한 영향을 미치지 않았다31. 이러한 발견은 특정 프로토콜에 따라 작업할 때 모든 IVF 실험실에 배아 cfDNA 분석의 잠재적 적용 가능성을 뒷받침합니다.
기존 방법에 대한 중요성
NICS 방법은 배아 생검을 피하므로 사용 안전성이 크게 향상됩니다. 배반포와 비교했을 때, NICS는 단순하고, 시간을 절약하며, 민감하고, 재현 가능한 착상 전 스크리닝 기술로, 이수성 가능성이 높은 보조 생식 인구에 적합합니다. 배반포 생검 절차에 상당한 전문 지식이 필요한 침습적 생검과 달리, NICS는 사용한 배지의 간단한 수집이 IVF19 의 정규 작동을 따르고 일부 국가에서는 PGS/PGD 자격이 필요하지 않기 때문에 광범위하게 적용할 수 있습니다.
향후 적용 분야
NICS는 ICSI뿐만 아니라 IVF 배아에 대한 임상 IVF의 염색체 스크리닝에 광범위하게 적용될 수 있는 잠재력을 가지고 있습니다. ICSI가 적극 권장되지만 정자의 영향을 방지하기 위해 zona pellucida에 부착된 정자를 제거하는 방법이 필요합니다.
형태학적 평가는 배아 평가를 위한 전통적인 방법이지만, 대부분의 경우 염색체 비정상 배아는 염색체 정상(이수성) 배아와 형태학적으로 유사하게 나타날 수 있습니다. 형태가 양호한 플로이드 배아를 자궁으로 이식할 때 형태학적 평가와 NICS 분석을 결합하면 진행 중인 임신율과 출생률을 개선할 수 있습니다. NICS 기술을 사용하여 단일 배아 이식의 임상적 효능을 평가하기 위해 무작위 임상 시험이 수행됩니다.
프로토콜의 중요한 단계
모든 cumulus-corona radiata 세포는 수정 전에 난모세포에서 제거되어야 합니다. 난모세포는 세포질 내 정자 주입(ICSI)에 의해 수정되었습니다. 배양 배지에 인간 유래 단백질/보충제를 추가하는 것은 피했습니다. 배양 배지는 4일째에 교체하고 배반포가 완전히 팽창하는 5일-6일에 수집했습니다. 배아는 4일째부터 배양 배지의 개별 방울에서 배양되었습니다. 배양 배지를 수집할 때 오염을 방지하기 위해 샘플 간의 이송 피펫을 변경했습니다.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yaxin Yao, Jieliang Ma, Jing Wang 및 Sijia Lu는 Yikon Genomics Co., Ltd.의 직원입니다.
Acknowledgments
저자들은 NGS 데이터 분석에 도움을 준 Shiping Bo와 Shujie Ma에게 감사의 뜻을 전합니다. 자금 지원: 이 연구는 국가 핵심 연구 개발 프로그램(보조금 번호 2018YFC1003100)의 지원을 받았습니다.
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
References
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Jr Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T. Jr, Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid? Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- Thermo Fisher Scientific. Qubit dsDNA HS Assay Kit. , Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015).
- Miseq system use guide. , Available from: https://support.illumina.com/downloads/miseq_system user _ guide 15027617.html (2016).
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Tags
염색체 스크리닝 인간 착상 전 배아 폐 배양 배지 샘플 수집 염색체 배합 분석 체외 수정(IVF) 이수성을 위한 착상 전 유전자 검사(PGT-A) 영양배엽(TE) 내부 세포 질량(ICM) 위양성 및 음성 NICS 기술 DNA 염기서열 분석 배양 배지 샘플링 전체 게놈 증폭(WGA) 라이브러리 준비 NGS 데이터 분석 냉동 보존 IVF 실험실Erratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021.
Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
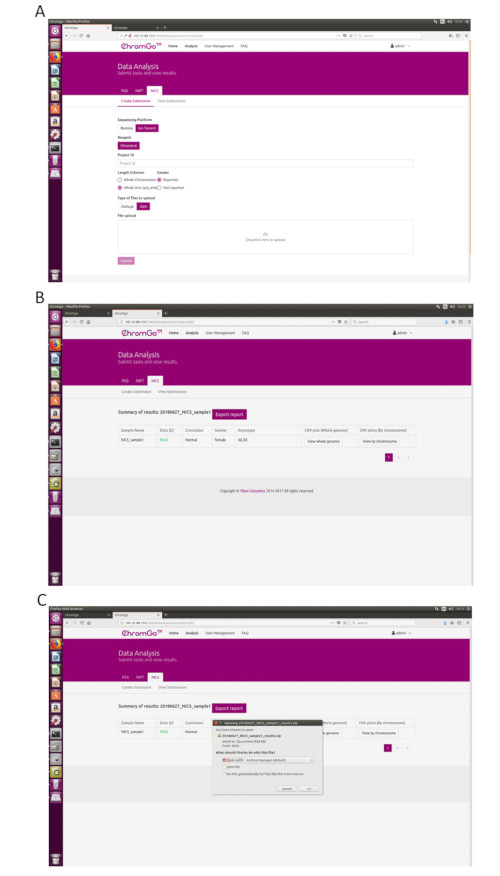
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
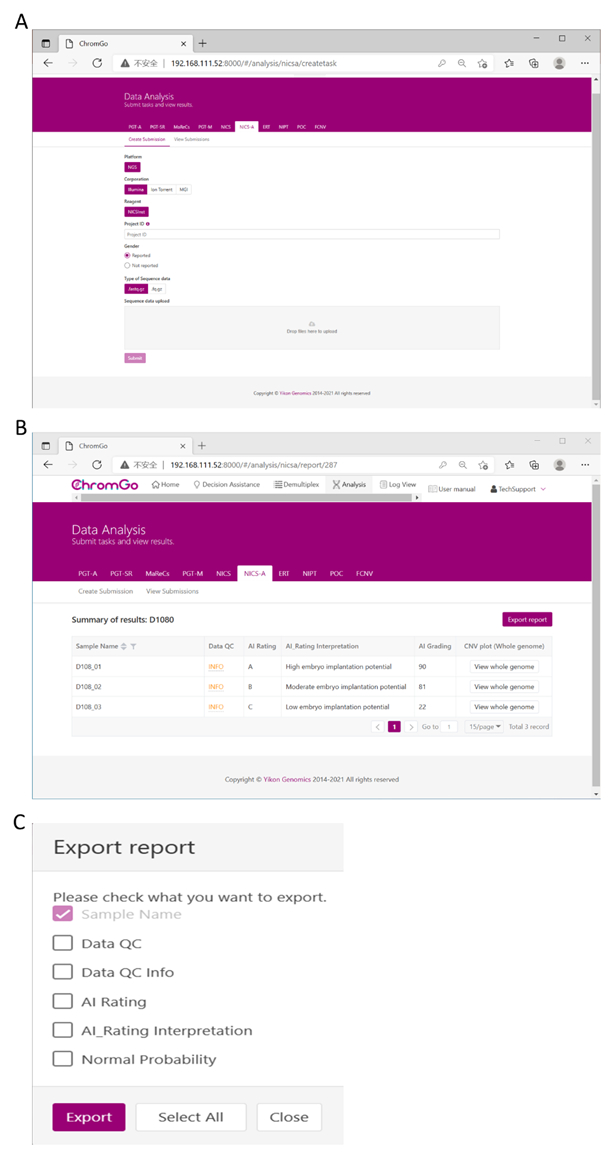
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.



