Introduction
ホスファチジルエタノールアミンメチル(PEMT)酵素は、モノメチルPEを与えるためにPE、モノ-PEまたはジメチル-PE上のメチル基供与体としてのS -adenosylmethionine(SAM)を使用して、1つ以上のメチル基の共有結合を触媒ジメチルPE、および/ またはホスファチジルコリン(PC)。これらの酵素は、動物細胞及び真菌におけるほぼ遍在しています。彼らはまた、いくつかの植物1および細菌の約10%、真核生物2と相互作用する特にで見つけることができます。
PEMTsはだけでなく、他の重要な細胞機能を果たすことにより、動物細胞における主要な脂質クラスのPCの生産に貢献することによって、細胞の生物学に関連します。哺乳類では、PEMTsは、主に、それらは非常に低密度リポタンパク質の正常な分泌のために必要とされる肝臓で発現され、それらはまた、食餌誘発性肥満3、アテローム性動脈硬化4に寄与し、インスリンレジストンス5。さらに、哺乳類のPEMTも低いレベルにあるが、脂肪細胞に発現し、脂肪沈着6、7に参加しています。癌発生8、9アポトーシス、及び細胞増殖10 PEMT役割も実証されています。細菌では、PEMT酵素は、正常な細胞増殖2、毒性2、および宿主植物11との共生のために重要であることが示されています。
本プロトコルの目的及び論理的根拠は、酵素を精製することなく、全細胞抽出物からPEMT活性を測定することです。二つの異なるプロトコルがPEMT活性を測定するために開発されています。最初の、そして最も一般的なものは、この記事のトピックであるPE、上に放射性SAMからトリチウム化メチル基の転移を測定します。このプロトコルは、本来understを得るために12酵母および哺乳動物細胞(肝臓)13からPEMT活性を測定するために開発されましたこれらの酵素の特異性を決定するために、これらの細胞におけるPC生合成の論理積ならびに。以降、この技術は、(15けれどもアッセイの基本的なpH値を使用して)細菌2及び原虫寄生14などの他の細胞型に適用されています。この技術は、全細胞抽出物、ならびに精製された酵素を用いて使用することができ、潜在的に任意の細胞抽出システムにも適用することができます。非放射性のアッセイはまた、Sの -adenosylhomocysteine、SAM 16のトランスメチレーション産物の酵素の定量化に依存しているように設計されています。それは放射能を含まないように後者のアッセイは、より便利かもしれないが、それは精製された酵素にのみ適しています。
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1.細胞抽出物の準備
- 20mMのpH7.4のHEPES、100 U / mlペニシリン、100μg/ mlのストレプトマイシン、5μg/ mlのヘムを補充した1×M199からなる培地中で26ºCで気密キャップで密封滅菌プラスチックボトルにリーシュマニア細胞を増殖させます振盪せずに、0.35グラム/ LのNaCO 2 H、0.1mMのアデニン、および2 / mlのビオプテリン。これらは1〜2×10 7 / mlの細胞密度に達したときに4ºCで5分間1,500 gでの遠心分離によって細胞を回収。
- ピペットで上清を捨て、冷リン酸緩衝生理食塩水(137mMの塩化ナトリウム、2.7mMのKClを文化のボリュームの半分に血清学的ピペットで細胞ペレットを再懸濁することによって細胞を洗浄、10 mMのの Na 2 HPO 4、1.8mMのKH 2 PO 4、pH7.4の)。 BL2の安全ガイドラインに従って、細胞上清を廃棄してください。
- 4ºCで5分間1500グラムで再び遠心細胞。ピペットで上清を捨てます。トンを続行次のステップOまたは(3ヶ月まで)-80ºCで長期保存のために液体窒素中で細胞ペレットを凍結スナップ。
- 2X溶解バッファー(0.5Mスクロース、0.1MのトリスHCl、pH7.5の、2mMのEDTA、および2倍プロテアーゼ阻害剤カクテル)を準備し、氷上で4ºCで保管してください。
- 2X溶解緩衝液の等量(新鮮または凍結した)細胞ペレットを再懸濁します。ガラスビーズの1倍のボリュームを追加します。 10分間4ºCで激しくボルテックス。
- 1×溶解バッファーの2容量を加え、混合します。破壊されていない細胞および核をペレット化するために10分間、4ºCで1,500×gで遠心分離細胞抽出物。
- 新鮮なクールな遠心管にピペットで上清を移し、実験終了まで氷上で細胞抽出物を保持します。
2.このようなビシンコニン酸アッセイなどのタンパク質推定キットを用いて細胞抽出物のタンパク質濃度を決定します
- S(II)、BCAと銅を混合することにより、ビシンコニン酸(BCA)溶液(1ミリリットル/チューブ)を準備49の比でulfate:1(v / v)です。
- BCA溶液1mlアリコートに10 mg / mlのウシ血清アルブミン(BSA)のストック溶液を希釈することによって、0のタンパク質標準を準備10、20、30、40、50、および60μgの/ mlでした。
- 重複でBCA溶液1ml中の細胞抽出物の2μLを加えます。予め温めておいた60℃の水浴中で10分間、標準およびタンパク質サンプルをインキュベートします。
- 転送サンプルを3分間氷に。 562ナノメートルの波長で分光光度計を用いて基準とタンパク質試料の吸光度を測定します。
- 製造業者のプロトコルに記載されているように参照としてBSA標準を使用して細胞抽出物のタンパク質濃度を計算します。 1×溶解緩衝液で10mlのMG /タンパク質濃度まで細胞抽出液を希釈します。
チューブあたり200μl中3酵素アッセイ
注:化学フードで次の手順を実行します。
- 15ミリリットルの重複の各サンプルをテストコニカルチューブ。チューブあたり20μlの1 MトリスHCl pHが7.5を用意し、氷上で保管してください。クロロホルム/メタノール2mlを調製(1:1(V / V)溶液停止)を各チューブのための室温です。
- ピペットで氷の上の各15ミリリットルコニカルチューブ中の1MトリスHCl pH7.5の20μlの。
- ここで上から放射線安全ガイドラインに従ってください。 SAMの50.06μMの合計チューブあたり[メチル- 3 H]アデノシル-L-メチオニンおよび50μM冷たいSAM - 0.06μM(0.2μCiの)Sと同等のものを追加します。冷水μlの追加xここで、x = 200-(20(+細胞抽出物のための20()+寒さと放射性SAMのボリューム)バッファ用)チューブあたり。
- 予め温めておいた30ºCの水槽に各コニカルチューブに移します。反応を開始するために各チューブに細胞抽出物(タンパク質200μgの相当)の20μLを加えます。所望の時間(0〜45分)インキュベートします。
- クロロホルム/メタノール2mlを加えて反応を停止し(1:1、V / V、停止液)を各チューブにします。 RT(20〜25にコニカルチューブを転送6; C)。
4.脂質抽出
注:化学フードで次の手順を実行します。
- 酵素反応試料を含む各チューブに水700μlを添加します。 30秒間激しくボルテックス。 RTで5分間1,500×gで遠心分離し、水相から有機を分離します。
- ピペットで新しい15ミリリットルコニカルチューブに低い有機相を転送します。 30秒間激しくボルテックスチューブとを含む各「下相」への水の1ミリリットルを追加します。遠心分離は、再び5分間1500×gで水相から有機を分離しました。
- ピペットを用いてシンチレーションチューブに低い有機相を転送します。 N 2気流下で乾燥したサンプル。放射線のガイドラインに従って非取り込まれた放射性SAMを含む水相と放射性コニカルチューブを廃棄してください。
- 2ミリリットル/シンチレーション液のチューブを追加します。シンチレーションCOUに取り込まれた放射能を測定しますメーカーのプロトコルと、機器の用途に応じてNTER。
- 以下の一般式を使用して、ナノモル/ mgのタンパク質における酵素活性を計算します。
CPM値は×10 3×[SAM(MM)の合計(放射性およびコールド)濃度]放射性SAM(CI /ミリモル)xの×5特異的活性[放射性SAMの濃度(mM)の]
注:上記のプロトコルは、依存のSAM濃度またはタンパク質依存PEMTアッセイのために変更することができます。 SAM濃度依存的酵素アッセイについては、時間(線形範囲内である15分)、一定に維持され、タンパク質依存PEMTアッセイ、SAM濃度(ここでは0.05 mMのを選んだ)と時刻ながら冷SAMの種々の量をアッセイに添加します(15分間)一定に保たれます。また、緩衝液のpHは、いくつかのPEMT酵素は異なる最適なpH値を持っている場合、必要に応じて変更することができます。
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
図1は、基質としての内因性のPEを使用して、酵素源としてリーシュマニア全細胞抽出物を用いて行った時間依存PEMTアッセイを示します。有機相中の放射能の量をシンチレーション計数によって定量しました。得られた数値を、PEに転写トリチウム化メチル基の量を計算するために利用されました。 PEMT活性は、約20分間のリニアました。それは、それがさらに15分間一定にとどまった後、約30分でプラトーに達しました。予想されるようになし、細胞抽出物は、アッセイ( 図 2)に添加しなかった時、PEMT活性が検出されませんでした。また、この活動は、Lの阻害剤である100μMのオクタデシルトリブロマイドの存在下で廃止されました大手 PEメチルトランスフェラーゼLMJ PEM1およびLMJ PEM2 14。 PEMT活性はまた、タンパク質濃度たdependenトン、そしてこの活動は、酵素アッセイ( 図3)に適用されるタンパク質の量に直線的に比例しました。最後に、SAM濃度依存PEMTアッセイ( 図4)SAMの増加する濃度を試験した中で、行いました。 PEMT活性は、約0.5mmのSAM濃度でプラトーに達しました。すべて一緒に、これら四つのアッセイはPEMT活性は特異的であり、酵素を精製することなく、全細胞抽出物から測定することができることを示しています。
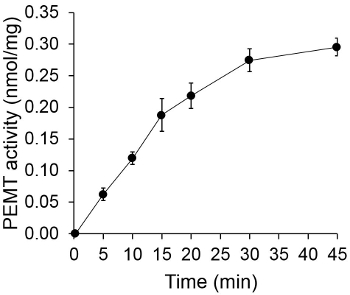
図1時間依存PEMTアッセイ。酵素アッセイは、時間の関数としての全リーシュマニア細胞抽出物の0.2ミリグラムと重複して2回行いました。 PEMT活性は、タンパク質およびp 1mg当たりPEに転写ナノモルのメチル基のように表されますERの時間。 SAMを含有するアッセイ溶液に添加する前に:(1容量、1)時間 "O"は、細胞抽出物は、最初にクロロホルム/メタノールから成る停止溶液2mlと混合しました。標準偏差が示されています。
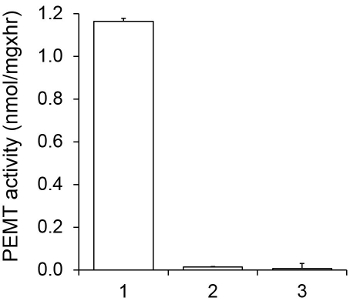
PEMT活性を図2特異性。PEMTアッセイは、0.05 mMのSAMの存在下で15分間、二重に2回行いました。 1、0.2 mgのタンパク質抽出物。 2、無細胞抽出物; 3、0.2mgのタンパク質および0.1mMオクタデシルアンモニウムブロミド。標準偏差が示されています。
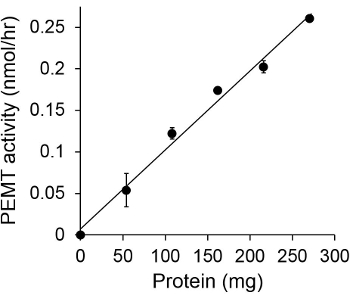
図3タンパク質依存PEMTアッセイは酵素アッセイは2回行いました。15分間0.05 mMのSAMに存在しない状態で(点O '')またはリーシュマニアタンパク質の様々な量の存在を複製します。標準偏差が示されています。
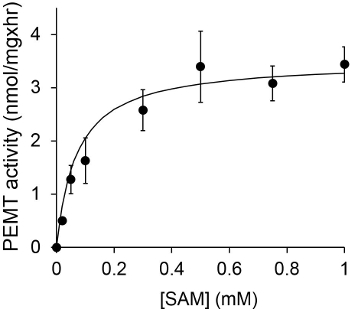
図4は、PEMTアッセイは、15分間のSAMの種々の濃度の存在下で、 リーシュマニア全細胞抽出物の0.2ミリグラムと重複して2回行った。標準偏差が示されています。
Subscription Required. Please recommend JoVE to your librarian.
Discussion
この単純な、迅速なPEMTアッセイは、タンパク質源として、全細胞抽出物を使用して、PE上にSAMからの放射性のメチル基の転移に起因するPEのメチル化形態の定量化を可能にします。これは、高速の敏感な、再現性、および精製された酵素を17にも適しています。興味のメチルトランスフェラーゼは、これらの基板にではなく、PE 12,13,18,19に固有である場合モノメチルまたはジメチル-PEは、アッセイに追加することができます。精製PEMT酵素が使用される場合、PEがアッセイに添加することができます。このプロトコルの制限は、アッセイは、反応生成物(モノ-PE、ジメチル-PE、またはPC)を識別していないということです。 20,21で説明したようにしかし、反応生成物の同一性(モノメチルPE、ジメチルPE、PC)は、さらに、1次元薄層クロマトグラフィーによって分析することができます。さらに、このようなSの -adenosylhomocysteine 及び5'-メチルチオなどのSAMの分解生成物のいくつかはPEMT活性Bを阻害することができますYフィードバック阻害。しかし、 リーシュマニアは、S はアデニンと S -ribosylhomocysteine、およびアデニンとmethylthioribose -1-リン酸23を生成するメチルチオホスへ-adenosylhomocysteine を切断、S -adenosylhomocysteine ラーゼ22を有しています 。しかし、PEMT活性の阻害が生じないようにS -adenosylhomocysteine ヒドロラーゼ及びメチルチオホス活動は、効率的にそれぞれ、Sの -adenosylhomocysteine 及びメチルチオを代謝するのに十分に高いかどうかは不明です。例では、それぞれの精製された組換え酵素の添加は、SAM分解産物24,25によるフィードバック阻害を軽減するためにアッセイに添加することができ、目的の細胞には存在しない-adenosylhomocysteine ヒドロラーゼおよび/ または5'-メチル代謝酵素であり、S、 26。
このプロトコルには4つの重要なステップがあります。ⅰ)プロテアーゼ阻害剤カクテル粉末は、使用直前に溶解緩衝液に(ステップ1.1)に添加しなければなりません。 ii)の全細胞抽出物を、ステップ1.4の後に準備()の後に以下の時間内で利用されるべきです。 iii)の脂質抽出工程(4.2および4.4ステップ)中に、注意が過剰なトリチウム化SAMが含ま相間または水相のいずれかの転送、およびiv)放射性Sをアリコートにしないように適用する必要がある- [メチル- 3 H]アデノシル-L-メチオニン、凍結の繰り返しサイクルとして受信すると、寒さの試薬 とは、不足を説明することができるアデニンと S -pentosylmethionine 26,27,28への加水分解に続いて5'-メチルおよびホモセリンラクトン、にそれを低下させる解凍します測定可能なPEMT活動。悪い全細胞抽出物はまた、無酵素活性に関与し得ます。この場合には、細胞抽出物の品質は、他の既知の酵素活性を測定することによって評価することができます。最後に、PEMT活動の欠如はPEMT専用に依拠することができますモノ-PEまたはジメチル-PEよりもむしろPEに特異的であるザイム。アッセイのモノメチルPEおよび/またはジメチルPE基質の添加は、PEMT活性を回復することができます。
代替的には、非放射性のSAM依存性メチルトランスフェラーゼアッセイは、ホモシステインの酵素的定量化、SAM 29のトランスメチレーション製品に依存すること、開発されました。このプロトコルは、正常PE特定のメチルトランスフェラーゼ活性16を測定するために適用されています。残念ながら、このアッセイは、全細胞抽出物のための精製酵素としないためにのみ適しており、いずれかの反応生成物の同一性を明らかにしません。
本プロトコルは、おそらく任意の細胞型に適用することができます。さらに、このアッセイは、肥満、食事誘導性アテローム性動脈硬化症に対する細菌感染や新規治療薬を戦うために新しい抗菌化合物を試験との関連で関心のPEMTに固有の潜在的な薬をテストするために使用することができます、およびインスリン抵抗性。
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
興味のある競合が宣言されていません
Materials
| Name | Company | Catalog Number | Comments |
| S-[Methyl-3H]adenosyl-L-methionine (specific activity of 5-15 Ci/mMole) | Perkin Elmer | NET155050UC | Aliquot the reagent and freeze at -20 °C; follow radiation safety guidelines while using this reagent |
| Protease inhibitor cocktail | Roche Life Sciences | 11836170001 | dilute it fresh |
| Glass beads, acid washed, 425-600 mm | Sigma Aldrich | G8772 | |
| Bicinchoninic acid solution | Sigma Aldrich | B9643 | |
| Copper (II) sulfate | Sigma Aldrich | C2284 | |
| Scintillation counter MicroBeta2 with 1-detector | Perkin Elmer | 2450-0010 | |
| Spectrophotometer Biomate 3 | Thermo Scientific | 840208300 | |
| BSA stock solution (10 mg/ml) | New England Biolabs | B9001S | |
| Scintillation liquid | Research Product International Corp | 111198 | |
| S-(5'-Adenosyl)-L-methionine chloride (hydrochloride) | Cayman Chemicals | 13956 | dilute the reagent in 20 mM HCl and freeze aliquots at -80 °C |
References
- Keogh, M. R., Courtney, P. D., Kinney, A. J., Dewey, R. E. Functional characterization of phospholipid N-.methyltransferases from Arabidopsis and soybean. J Biol Chem. 284 (23), 15439-15447 (2009).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim Biophys Acta. 1831 (3), 503-513 (2013).
- Gao, X., et al. Decreased lipogenesis in white adipose tissue contributes to the resistance to high fat diet-induced obesity in phosphatidylethanolamine N-.methyltransferase-deficient mice. Biochim Biophys Acta. 1851 (2), 152-162 (2015).
- Zhao, Y., et al. Lack of phosphatidylethanolamine N-.methyltransferase alters plasma VLDL phospholipids and attenuates atherosclerosis in mice. Arterioscler Thromb Vasc Biol. 29 (9), 1349-1355 (2009).
- Vance, D. E. Phospholipid methylation in mammals: from biochemistry to physiological function. Biochim Biophys Acta. 1838 (6), 1477-1487 (2014).
- Nishimaki-Mogami, T., Suzuki, K., Takahashi, A. The role of phosphatidylethanolamine methylation in the secretion of very low density lipoproteins by cultured rat hepatocytes: rapid inhibition of phosphatidylethanolamine methylation by bezafibrate increases the density of apolipoprotein B48-containing lipoproteins. Biochim Biophys Acta. 1304 (1), 21-31 (1996).
- Noga, A. A., Zhao, Y., Vance, D. E. An unexpected requirement for phosphatidylethanolamine N-.methyltransferase in the secretion of very low density lipoproteins. J Biol Chem. 277 (44), 42358-42365 (2002).
- Li, D., et al. Epigenetic repression of phosphatidylethanolamine N-.methyltransferase (PEMT) in BRCA1-mutated breast cancer. Oncotarget. 5 (5), 1315-1325 (2014).
- Cui, Z., Houweling, M., Vance, D. E. Suppression of rat hepatoma cell growth by expression of phosphatidylethanolamine N-.methyltransferase-2. J Biol Chem. 269 (40), 24531-24533 (1994).
- Cui, Z., Shen, Y. J., Vance, D. E. Inverse correlation between expression of phosphatidylethanolamine.N-.methyltransferase-2 and growth rate of perinatal rat livers. Biochim Biophys Acta. 1346 (1), 10-16 (1997).
- Minder, A. C., de Rudder, K. E., Narberhaus, F., Fischer, H. M., Hennecke, H., Geiger, O. Phosphatidylcholine levels in.Bradyrhizobium japonicum. membranes are critical for an efficient symbiosis with the soybean host plant. Mol Microbiol. 39 (5), 1186-1198 (2001).
- Kodaki, T., Yamashita, S. Yeast phosphatidylethanolamine methylation pathway. Cloning and characterization of two distinct methyltransferase genes. J Biol Chem. 262 (32), 15428-15435 (1987).
- Tanaka, Y., Amano, F., Maeda, M., Nishijima, M., Akamatsu, Y. Purification and properties of phosphatidyl-N-.monomethylethanolamine N-.methyltransferase, the enzyme catalyzing the second and the third steps in the phosphatidylethanolamine N-.methyltransferase system, from mouse liver microsomes. Jpn J Med Sci Biol. 43 (3), 59-73 (1990).
- Bibis, S. S., Dahlstrom, K., Zhu, T., Zufferey, R. Characterization of Leishmania major phosphatidylethanolamine methyltransferases LmjPEM1 and LmjPEM2 and their inhibition by choline analogs. Mol Biochem Parasitol. 196 (2), 90-99 (2014).
- deRudder, K. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti. mutants deficient in phospholipid N-.methyltransferase still contain phosphatidylcholine. J Bacteriol. 179 (22), 6921-6928 (1997).
- Aktas, M., Narberhaus, F. In vitro characterization of the enzyme properties of the phospholipid N-.methyltransferase PmtA from Agrobacterium tumefaciens. J Bacteriol. 191 (7), 2033-2041 (2009).
- Ridgway, N. D., Vance, D. E. Phosphatidylethanolamine N-.methyltransferase from rat liver. Methods Enzymol. 209, 366-374 (1992).
- Gaynor, P. M., Carman, G. M. Phosphatidylethanolamine methyltransferase and phospholipid methyltransferase activities from Saccharomyces cerevisiae. Enzymological and kinetic properties. Biochim Biophys Acta. 1045 (2), 156-163 (1990).
- Arondel, V., Benning, C., Somerville, C. R. Isolation and functional expression in Escherichia coli. of a gene encoding phosphatidylethanolamine methyltransferase (EC 2.1.1.17) from Rhodobacter sphaeroides. J Biol Chem. 268 (21), 16002-16008 (1993).
- Wessel, M., Klusener, S., Godeke, J., Fritz, C., Hacker, S., Narberhaus, F. Virulence of Agrobacterium tumefaciens. requires phosphatidylcholine in the bacterial membrane. Mol Microbiol. 62 (3), 906-915 (2006).
- Klusener, S., Aktas, M., Thormann, K. M., Wessel, M., Narberhaus, F. Expression and physiological relevance of Agrobacterium tumefaciens. phosphatidylcholine biosynthesis genes. J Bacteriol. 191 (1), 365-374 (2009).
- Henderson, D. M., et al. Cloning of the gene encoding Leishmania donovani.S.-adenosylhomocysteine hydrolase, a potential target for antiparasitic chemotherapy. Mol Biochem Parasitol. 53 (1-2), 169-183 (1992).
- Koszalka, G. W., Krenitsky, T. A. 5'-Methylthioadenosine (MTA) phosphorylase from promastigote of Leishmania donovani. Purine and Pyrimidine Metabolism in Man V, Adv Exp Med Biol. Nyhan, W. L., Thompson, L. F., Watts, R. W. E. 131, Springer US. 559-563 (1986).
- Biastoff, S., Teuber, M., Zhou, Z. S., Dräger, B. Colorimetric activity measurement of a recombinant putrescine N.-methyltransferase from Datura stramonium. Planta Med. 72 (12), 1136-1141 (2006).
- Hendricks, C. L., Ross, J. R., Pichersky, E., Noel, J. P., Zhou, Z. S. An enzyme-coupled colorimetric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 326 (1), 100-105 (2004).
- Cannon, L. M., Butler, F. N., Wan, W., Zhou, Z. S. A stereospecific colorimetric assay for (S.,S.)-adenosylmethionine quantification based on thiopurine methyltransferase-catalyzed thiol methylation. Anal Biochem. 308 (2), 358-363 (2002).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S.-adenosyl-L-methionine. Biochemistry. 25 (15), 4444-4449 (1986).
- Wu, S. E., Huskey, W. P., Borchardt, R. T., Schowen, R. L. Chiral instability at sulfur of S.-adenosylmethionine. Biochemistry. 22 (12), 2828-2832 (1983).
- Dorgan, K. M., et al. An enzyme-coupled continuous spectrophotometric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 350 (2), 249-255 (2006).



