Introduction
Metiltransferasa fosfatidiletanolamina (PEMA) enzimas catalizan la unión covalente de uno o más grupos metilo utilizando S -adenosylmethionine (SAM) como donante de metilo en PE grupo, monometil o dimetil-PE-PE para dar monometil-PE, dimetil-PE y / o fosfatidilcolina (PC). Estas enzimas son casi omnipresentes en las células animales y hongos. También se pueden encontrar en algunas plantas 1 y aproximadamente el 10% de las bacterias, particularmente aquellos que interactúan con 2 eucariotas.
PEMTs son relevantes para la biología de la célula no sólo contribuyendo a la producción de PC, que es la principal clase de lípidos en las células animales, sino también mediante el cumplimiento de otras funciones celulares importantes. En los mamíferos, PEMTs se expresan principalmente en el hígado donde son requeridas para la secreción normal de la lipoproteína de muy baja densidad y también contribuyen a la obesidad inducida por la dieta 3, 4 aterosclerosis, y la insulina se resistenANCE 5. Además, PEMA mamíferos también se expresan en los adipocitos, aunque a niveles más bajos, y participar en la deposición de grasa 6, 7. PEMA papel en el desarrollo del cáncer 8, 9 apoptosis, y 10 el crecimiento celular también se ha demostrado. En las bacterias, las enzimas PEMA han demostrado ser importantes para el crecimiento normal de las células 2, la virulencia 2, y la simbiosis con la planta huésped 11.
El objetivo y razón de ser del presente protocolo es para medir la actividad PEMA a partir de extractos de células enteras, sin la necesidad de purificar la enzima. Dos protocolos distintos se han desarrollado para medir la actividad PEMA. La primera y más común se mide la transferencia de un grupo metilo tritiada de SAM radiactivo en PE, que es el tema de este artículo. Este protocolo ha sido desarrollado originalmente para medir la actividad de la levadura PEMA 12 y células de mamíferos (hígado) 13 para obtener una understanding de la biosíntesis de PC en estas células, así como para determinar la especificidad de estas enzimas. Más tarde, esta técnica se ha aplicado a otros tipos de células tales como bacterias 2 (utilizando un valor de pH básico para el ensayo aunque 15) y parásitos protozoarios 14. Esta técnica se puede utilizar con extractos de células enteras, así como enzima purificada, y potencialmente se puede aplicar a cualquier sistema de extracto celular. Un ensayo no radiactivo también se ha diseñado que se basa en la cuantificación enzimática de S -adenosylhomocysteine, el producto de transmetilación de SAM 16. El último ensayo puede ser más conveniente, ya que no implica la radiactividad pero sólo es adecuado para las enzimas purificadas.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Preparación de la célula Extracto
- Cultivar las células de Leishmania en una botella de plástico estéril sellado con tapa de aire ajustado a 26 ºC en un medio hecho de 1x M199 suplementado con 20 mM HEPES pH 7,4, 100 U / ml de penicilina, 100 mg / ml de estreptomicina, 5 mg / ml heme , 0,35 g / L NaCO 2 H, adenina 0,1 mM y 2 g / ml biopterina sin agitación. Recoger las células por centrifugación a 1500 g durante 5 min a 4 ºC cuando alcanzan una densidad celular de 1-2 x 10 7 / ml.
- Descartar el sobrenadante con una pipeta y se lavan las células resuspendiendo el sedimento celular con una pipeta serológica en medio del volumen de la cultura de solución salina tamponada con fosfato fría (NaCl 137 mM, 2,7 mM KCl, mM Na 10 2 HPO 4, 1,8 mM KH 2 PO 4, pH 7,4). Deseche sobrenadantes de células de acuerdo con los lineamientos de seguridad BL2.
- Centrifugar las células de nuevo a 1.500 g durante 5 min a 4 ºC. Descartar el sobrenadante con una pipeta. Proceda to el siguiente paso o complemento congelar el sedimento de células en nitrógeno líquido para almacenamiento a largo plazo a -80 ºC (hasta tres meses).
- Preparar tampón de lisis 2x (0,5 M de sacarosa, 0,1 M Tris-HCl, pH 7,5, EDTA 2 mM, y el inhibidor de la proteasa cóctel 2x) y conservarse a 4 ° C en hielo.
- Resuspender el sedimento celular (fresco o congelado) en un volumen igual de tampón de lisis 2x. Añadir 1 x volumen de perlas de vidrio. Vortex vigorosamente a 4 ºC durante 10 min.
- Añadir 2 volúmenes de tampón de lisis 1x y mezclar. Extractos de células se centrifuga a 1500 xga 4 ºC durante 10 minutos para sedimentar las células intactas y núcleos.
- Transferir el sobrenadante con una pipeta en un tubo de centrífuga fresco y mantenerla extractos de células en hielo hasta la finalización del experimento.
2. Determinar la concentración de proteína del extracto celular utilizando el kit de Estimación de proteínas como Ensayo de Ácido bicinconínico
- Preparar la solución de ácido bicinconínico (BCA) (1 ml / tubo) por mezcla de los BCA y cobre (II) sulfate en una proporción de 49: 1 (v / v).
- Preparar los estándares de proteína de 0, 10, 20, 30, 40, 50, y 60 g / ml por dilución de una solución madre de 10 mg / ml de albúmina de suero bovino (BSA) en alícuotas de 1 ml de la solución de BCA.
- Añadir 2 l de extractos de células en 1 ml de solución BCA en los duplicados. Incubar los estándares y muestras de proteína durante 10 min en un baño de agua 60 ° C pre-calentado.
- Muestras de Transferencia de hielo durante 3 min. Medir la absorbancia de los estándares y las muestras de proteínas con un espectrofotómetro a una longitud de onda de 562 nm.
- Calcular la concentración de proteína de los extractos celulares utilizando el estándar de BSA como referencia como se describe en el protocolo del fabricante. Diluir los extractos de células a una concentración de proteína de 10 mg / ml con tampón de lisis 1x.
3. ensayo enzimático en 200 l por tubo
NOTA: Realice los siguientes pasos en una campana química.
- Pruebe cada muestra por duplicado en unos 15 mltubo cónico. Preparar 20 l 1 M Tris HCl pH 7,5 por tubo y mantenerlo en hielo. Preparar 2 ml de cloroformo / metanol (1: 1 (v / v); solución de parada) a TA durante cada tubo.
- Pipetear 20 l de 1 M Tris HCl pH 7.5 en cada tubo cónico de 15 ml en hielo.
- Siga las normas de seguridad radiológica de aquí en adelante. Añadir el equivalente de 0,06 m (0,2 Ci) S - [metil- 3 H] adenosil-L-metionina y 50 mM frío SAM por tubo para un total de 50,06 M de SAM. Añadir x l de agua fría donde x = 200 (20 (de buffer) + 20 (para los extractos celulares) + volumen de frío y radiactivo SAM) por tubo.
- Traslado cada tubo cónico a un baño de agua precalentado 30 ºC. Añadir a cada tubo 20 l de extractos de células (equivalente a 200 g de proteína) para iniciar la reacción. Incubar durante el tiempo deseado (0 a 45 min).
- Detener la reacción mediante la adición de 2 ml de cloroformo / metanol (1: 1; v / v; solución de parada) a cada tubo. Transferir el tubo cónico a RT (20-256; C).
4. Extracción de lípidos
NOTA: Realice los siguientes pasos en una campana química.
- Añadir 700 l de agua a cada tubo que contiene la muestra de reacción enzimática. Vortex vigorosamente durante 30 seg. Centrifugar a 1500 xg durante 5 min a temperatura ambiente para separar el orgánica de la fase agua.
- Transferir la fase orgánica inferior en un nuevo tubo cónico de 15 ml con una pipeta. Añadir 1 ml de agua a cada "fase inferior" que contiene el tubo y agitar vigorosamente durante 30 seg. Centrifugar de nuevo a 1500 xg durante 5 min para separar el orgánica de la fase agua.
- Transferir la fase orgánica inferior en un tubo de centelleo con una pipeta. Las muestras secas bajo una corriente de N2. Deseche las fases de agua que contienen el SAM radiactivo no incorporado y los tubos cónicos radiactivos como por las directrices de radiación.
- Añadir 2 ml / tubo de líquido de centelleo. Medir la radiactividad incorporada con un cou de centelleonter acuerdo con el protocolo del fabricante y el uso del instrumento.
- Calcular la actividad enzimática en mg de proteína / nmol utilizando la siguiente ecuación general:
valor cpm x 10 x 3 [Total (radiactivos y frío) concentración de SAM (mM)] x 5 actividad específica de SAM radiactivo (Ci / mmol) x [concentración de SAM radiactivo (mM)]
NOTA: el protocolo anterior se puede alterar para una concentración SAM dependiente o un ensayo de PEMA dependiente de la proteína. Para la concentración SAM ensayo enzimático dependiente, el tiempo se mantiene constante (15 min, que está en el intervalo lineal) y varias cantidades de frío SAM se añaden al ensayo, mientras que para el ensayo de PEMA dependiente de la proteína, la concentración de SAM (elegimos 0,05 mM) y la hora se mantuvo constante (15 min). También el pH del tampón se puede cambiar según sea necesario si algunas enzimas PEMA tienen un valor de pH óptimo diferente.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
La Figura 1 muestra un ensayo de PEMA dependiente del tiempo, que se llevó a cabo con extracto de células enteras Leishmania como una fuente de enzima usando PE endógena como sustrato. La cantidad de radiactividad en la fase orgánica se cuantificó por recuento de centelleo. Los números resultantes se utilizan para calcular la cantidad de grupos metilo tritiados transferidos a PE. La actividad PEMA fue lineal durante aproximadamente 20 min. A continuación, alcanzó una meseta a alrededor de 30 min, después de lo cual se mantuvo constante durante otros 15 min. Como se esperaba, no se detectó actividad PEMA cuando no se añadieron los extractos de células para el ensayo (Figura 2). Además, esta actividad fue abolida en presencia de 100 mM de bromuro de octadeciltrimetilamonio, que es un inhibidor de L. principales metiltransferasas PE Lmj Pem1 y Lmj PEM2 14. Actividad PEMA también proteínas dependen de concentraciónt, y esta actividad era linealmente proporcional a la cantidad de proteína aplicada para el ensayo enzimático (Figura 3). Por último, un ensayo de PEMA dependiente de la concentración SAM se llevó a cabo, en el que concentraciones crecientes de SAM fueron probados (Figura 4). Actividad PEMA alcanzó una meseta a una concentración de aproximadamente 0,5 SAM mM. Todos juntos, estos cuatro ensayos demuestran que la actividad PEMA es específico y se puede medir a partir de extractos de células enteras y sin la necesidad de purificar la enzima (s).
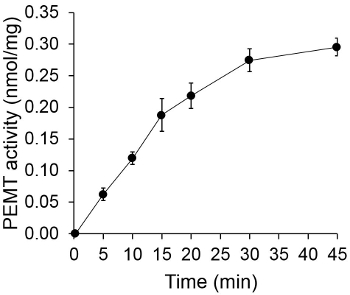
Figura 1. Tiempo de ensayo PEMA dependiente. El ensayo enzimático se realizó dos veces por duplicado con 0,2 mg de extractos de células de Leishmania enteros como una función del tiempo. PEMA actividad se representa como grupos metilo nmol transferidos a PE por mg de proteína y phr er. Para el tiempo de "O", extractos de células se mezclaron primero con 2 ml de solución de parada hecho de cloroformo / metanol (1: 1, en volumen) antes de ser añadido a la solución de ensayo que contiene SAM. Se muestran las desviaciones estándar.
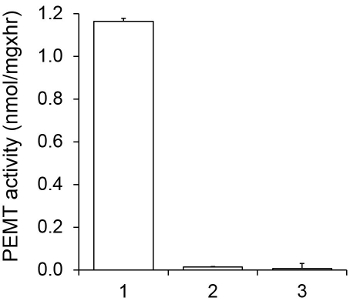
Figura 2. Especificidad de PEMA actividad. PEMA ensayo se llevó a cabo dos veces por duplicado durante 15 min en presencia de 0,05 mM SAM. Extracto de proteína 1, 0,2 mg; 2, ningún extracto celular; 3, 0,2 mg de proteína y 0,1 mM de bromuro de octadeciltrimetilamonio. Se muestran las desviaciones estándar.
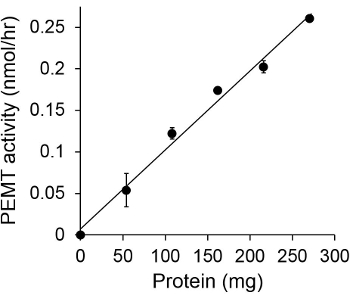
Figura 3. La proteína de ensayo PEMA dependiente. El ensayo enzimático se llevó a cabo dos veces enduplicar en ausencia (punto "O") o la presencia de diversas cantidades de proteínas de Leishmania con 0,05 mM SAM durante 15 min. Se muestran las desviaciones estándar.
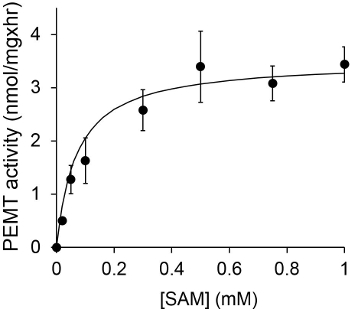
Figura 4. El ensayo se llevó a cabo PEMA dos veces por duplicado con 0,2 mg de extractos de células enteras de Leishmania en presencia de diversas concentraciones de SAM durante 15 min. Las desviaciones estándar se muestran.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Este ensayo PEMA simple, rápida permite la cuantificación de las formas metiladas de PE que resulta de la transferencia de grupos metilo radiactivos de SAM en PE utilizando extracto de células enteras como fuente de proteína. Es rápido, sensible, reproducible, y también es adecuado para las enzimas purificadas 17. Monometil- o dimetil-PE se pueden añadir a el ensayo si el metiltransferasa de interés es específica para estos sustratos en lugar de PE 12,13,18,19. Si se utiliza la enzima purificada PEMA, PE puede ser añadido al ensayo. Una limitación de este protocolo es que el ensayo no identifica los productos de las reacciones (monometil-PE, dimetil-PE, o PC). Sin embargo, la identidad de los productos de reacción (monometil-PE, dimetil-PE, PC) adicionalmente puede ser analizada por uno cromatografía de capa fina dimensional como se describe en 20,21. Además, algunos de los productos de degradación tales como SAM S -adenosylhomocysteine y 5'-metiltioadenosina puede inhibir la actividad de b PEMAla inhibición por retroalimentación y. Sin embargo, Leishmania posee una hidrolasa S -adenosylhomocysteine 22, que escinde S -adenosylhomocysteine en adenina y S -ribosylhomocysteine, y una fosforilasa metiltioadenosina, que produce adenina y metiltiorribosa-1-fosfato 23. Sin embargo, no se sabe si hidrolasa S -adenosylhomocysteine y actividades metiltioadenosina fosforilasa son lo suficientemente altos para metabolizar de manera eficiente S -adenosylhomocysteine y metiltioadenosina, respectivamente, de modo que no se produce inhibición de la actividad PEMA. En el caso de S hidrolasa -adenosylhomocysteine y / o 5'-metiltioadenosina enzima metabólica están ausentes en la célula de interés, además de las respectivas enzimas recombinantes purificados, se puede añadir al ensayo para aliviar la inhibición por retroalimentación por productos de degradación SAM 24,25, 26.
Hay cuatro pasos críticos en este protocolo: i)el polvo de cóctel inhibidor de proteasa tiene que ser añadido al tampón de lisis justo antes del uso (paso 1.1); ii) toda la célula extractos deben ser utilizados dentro de las siguientes horas después de la preparación (después del paso 1.4); iii) durante las etapas de extracción de lípidos (pasos 4.2 y 4.4), la precaución debe aplicarse a no transferir cualquiera de la fase de interfase o el agua, que contiene el exceso de tritiada SAM, y iv) a la parte alícuota radiactivo S - [metil- 3 H] adenosil-L-metionina y el reactivo frío tras la recepción como ciclos repetidos de congelación y descongelación degradar en 5'-metiltioadenosina y homoserina lactona seguido de hidrólisis para la adenina y S -pentosylmethionine 26,27,28, lo que puede explicar la falta de PEMA actividad medible. Extractos de células enteras Bad también pueden ser responsables de ninguna actividad enzimática. En este caso, la calidad del extracto celular se puede evaluar mediante la medición de otra actividad enzimática conocida. Por último, la falta de actividad PEMA puede confiar en el PEMA enZyme ser específico para monometil o dimetil-PE-PE en lugar de PE. La adición de monometil-PE y / o sustratos dimetil-PE en el ensayo puede restaurar la actividad PEMA.
Una alternativa, el ensayo de metiltransferasa dependiente de SAM no radiactivo se ha desarrollado, que se basa en la cuantificación enzimática de homocisteína, los productos de transmetilación de SAM 29. Este protocolo se ha aplicado con éxito para medir PE metiltransferasa actividad específica 16. Desafortunadamente, este ensayo sólo es adecuado para la enzima purificada y no para extracto de células enteras, y no revela la identidad de los productos de reacción tampoco.
La presente protocolo, posiblemente, se puede aplicar a cualquier tipo de célula. Además, este ensayo puede ser utilizado para probar fármacos potenciales específicos de un PEMA de interés en el contexto de pruebas de nuevos compuestos antimicrobianos para combatir infecciones bacterianas o nuevas terapias contra la aterosclerosis inducida por la dieta, la obesidadY resistencia a la insulina.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Ningún conflicto de interés declarado
Materials
| Name | Company | Catalog Number | Comments |
| S-[Methyl-3H]adenosyl-L-methionine (specific activity of 5-15 Ci/mMole) | Perkin Elmer | NET155050UC | Aliquot the reagent and freeze at -20 °C; follow radiation safety guidelines while using this reagent |
| Protease inhibitor cocktail | Roche Life Sciences | 11836170001 | dilute it fresh |
| Glass beads, acid washed, 425-600 mm | Sigma Aldrich | G8772 | |
| Bicinchoninic acid solution | Sigma Aldrich | B9643 | |
| Copper (II) sulfate | Sigma Aldrich | C2284 | |
| Scintillation counter MicroBeta2 with 1-detector | Perkin Elmer | 2450-0010 | |
| Spectrophotometer Biomate 3 | Thermo Scientific | 840208300 | |
| BSA stock solution (10 mg/ml) | New England Biolabs | B9001S | |
| Scintillation liquid | Research Product International Corp | 111198 | |
| S-(5'-Adenosyl)-L-methionine chloride (hydrochloride) | Cayman Chemicals | 13956 | dilute the reagent in 20 mM HCl and freeze aliquots at -80 °C |
References
- Keogh, M. R., Courtney, P. D., Kinney, A. J., Dewey, R. E. Functional characterization of phospholipid N-.methyltransferases from Arabidopsis and soybean. J Biol Chem. 284 (23), 15439-15447 (2009).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim Biophys Acta. 1831 (3), 503-513 (2013).
- Gao, X., et al. Decreased lipogenesis in white adipose tissue contributes to the resistance to high fat diet-induced obesity in phosphatidylethanolamine N-.methyltransferase-deficient mice. Biochim Biophys Acta. 1851 (2), 152-162 (2015).
- Zhao, Y., et al. Lack of phosphatidylethanolamine N-.methyltransferase alters plasma VLDL phospholipids and attenuates atherosclerosis in mice. Arterioscler Thromb Vasc Biol. 29 (9), 1349-1355 (2009).
- Vance, D. E. Phospholipid methylation in mammals: from biochemistry to physiological function. Biochim Biophys Acta. 1838 (6), 1477-1487 (2014).
- Nishimaki-Mogami, T., Suzuki, K., Takahashi, A. The role of phosphatidylethanolamine methylation in the secretion of very low density lipoproteins by cultured rat hepatocytes: rapid inhibition of phosphatidylethanolamine methylation by bezafibrate increases the density of apolipoprotein B48-containing lipoproteins. Biochim Biophys Acta. 1304 (1), 21-31 (1996).
- Noga, A. A., Zhao, Y., Vance, D. E. An unexpected requirement for phosphatidylethanolamine N-.methyltransferase in the secretion of very low density lipoproteins. J Biol Chem. 277 (44), 42358-42365 (2002).
- Li, D., et al. Epigenetic repression of phosphatidylethanolamine N-.methyltransferase (PEMT) in BRCA1-mutated breast cancer. Oncotarget. 5 (5), 1315-1325 (2014).
- Cui, Z., Houweling, M., Vance, D. E. Suppression of rat hepatoma cell growth by expression of phosphatidylethanolamine N-.methyltransferase-2. J Biol Chem. 269 (40), 24531-24533 (1994).
- Cui, Z., Shen, Y. J., Vance, D. E. Inverse correlation between expression of phosphatidylethanolamine.N-.methyltransferase-2 and growth rate of perinatal rat livers. Biochim Biophys Acta. 1346 (1), 10-16 (1997).
- Minder, A. C., de Rudder, K. E., Narberhaus, F., Fischer, H. M., Hennecke, H., Geiger, O. Phosphatidylcholine levels in.Bradyrhizobium japonicum. membranes are critical for an efficient symbiosis with the soybean host plant. Mol Microbiol. 39 (5), 1186-1198 (2001).
- Kodaki, T., Yamashita, S. Yeast phosphatidylethanolamine methylation pathway. Cloning and characterization of two distinct methyltransferase genes. J Biol Chem. 262 (32), 15428-15435 (1987).
- Tanaka, Y., Amano, F., Maeda, M., Nishijima, M., Akamatsu, Y. Purification and properties of phosphatidyl-N-.monomethylethanolamine N-.methyltransferase, the enzyme catalyzing the second and the third steps in the phosphatidylethanolamine N-.methyltransferase system, from mouse liver microsomes. Jpn J Med Sci Biol. 43 (3), 59-73 (1990).
- Bibis, S. S., Dahlstrom, K., Zhu, T., Zufferey, R. Characterization of Leishmania major phosphatidylethanolamine methyltransferases LmjPEM1 and LmjPEM2 and their inhibition by choline analogs. Mol Biochem Parasitol. 196 (2), 90-99 (2014).
- deRudder, K. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti. mutants deficient in phospholipid N-.methyltransferase still contain phosphatidylcholine. J Bacteriol. 179 (22), 6921-6928 (1997).
- Aktas, M., Narberhaus, F. In vitro characterization of the enzyme properties of the phospholipid N-.methyltransferase PmtA from Agrobacterium tumefaciens. J Bacteriol. 191 (7), 2033-2041 (2009).
- Ridgway, N. D., Vance, D. E. Phosphatidylethanolamine N-.methyltransferase from rat liver. Methods Enzymol. 209, 366-374 (1992).
- Gaynor, P. M., Carman, G. M. Phosphatidylethanolamine methyltransferase and phospholipid methyltransferase activities from Saccharomyces cerevisiae. Enzymological and kinetic properties. Biochim Biophys Acta. 1045 (2), 156-163 (1990).
- Arondel, V., Benning, C., Somerville, C. R. Isolation and functional expression in Escherichia coli. of a gene encoding phosphatidylethanolamine methyltransferase (EC 2.1.1.17) from Rhodobacter sphaeroides. J Biol Chem. 268 (21), 16002-16008 (1993).
- Wessel, M., Klusener, S., Godeke, J., Fritz, C., Hacker, S., Narberhaus, F. Virulence of Agrobacterium tumefaciens. requires phosphatidylcholine in the bacterial membrane. Mol Microbiol. 62 (3), 906-915 (2006).
- Klusener, S., Aktas, M., Thormann, K. M., Wessel, M., Narberhaus, F. Expression and physiological relevance of Agrobacterium tumefaciens. phosphatidylcholine biosynthesis genes. J Bacteriol. 191 (1), 365-374 (2009).
- Henderson, D. M., et al. Cloning of the gene encoding Leishmania donovani.S.-adenosylhomocysteine hydrolase, a potential target for antiparasitic chemotherapy. Mol Biochem Parasitol. 53 (1-2), 169-183 (1992).
- Koszalka, G. W., Krenitsky, T. A. 5'-Methylthioadenosine (MTA) phosphorylase from promastigote of Leishmania donovani. Purine and Pyrimidine Metabolism in Man V, Adv Exp Med Biol. Nyhan, W. L., Thompson, L. F., Watts, R. W. E. 131, Springer US. 559-563 (1986).
- Biastoff, S., Teuber, M., Zhou, Z. S., Dräger, B. Colorimetric activity measurement of a recombinant putrescine N.-methyltransferase from Datura stramonium. Planta Med. 72 (12), 1136-1141 (2006).
- Hendricks, C. L., Ross, J. R., Pichersky, E., Noel, J. P., Zhou, Z. S. An enzyme-coupled colorimetric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 326 (1), 100-105 (2004).
- Cannon, L. M., Butler, F. N., Wan, W., Zhou, Z. S. A stereospecific colorimetric assay for (S.,S.)-adenosylmethionine quantification based on thiopurine methyltransferase-catalyzed thiol methylation. Anal Biochem. 308 (2), 358-363 (2002).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S.-adenosyl-L-methionine. Biochemistry. 25 (15), 4444-4449 (1986).
- Wu, S. E., Huskey, W. P., Borchardt, R. T., Schowen, R. L. Chiral instability at sulfur of S.-adenosylmethionine. Biochemistry. 22 (12), 2828-2832 (1983).
- Dorgan, K. M., et al. An enzyme-coupled continuous spectrophotometric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 350 (2), 249-255 (2006).



