Overview
Source: Michael S. Lee1 and Tonya J. Webb1
1 Department of Microbiology and Immunology, University of Maryland School of Medicine and the Marlene and Stewart Greenebaum Comprehensive Cancer Center, Baltimore, Maryland 21201
Immunohistochemistry (IHC) and immunocytochemistry (ICC) are techniques used to visualize the expression and localization of specific antigens using antibodies. The first published use of IHC was in 1941 when Albert Coons used the technique to visualize the presence of pneumococcal antigen in tissue sections from mice infected with Pneumococcus (1). The name, immunohistochemistry, is derived from the roots "immuno-," in reference to antibodies, and "histo-," in reference to the tissue sections used in IHC. The root "cyto-" in immunocytochemistry highlights the key difference between ICC and IHC. Whereas IHC uses sections of whole tissue, ICC uses cells that have been isolated from tissue or grown in culture. The difference in samples used means sample preparation technically differs between IHC and ICC, but otherwise the protocols for ICC and IHC are identical and one will find the terms are frequently used interchangeably.
In both IHC and ICC, antibodies with either chemical or fluorescent tags, such as peroxidase or rhodamine, respectively, are used to visualize the distribution of any antigen of interest through specific binding of the tagged antibody to the antigen. In the case of IHC, thin slices of tissue are immobilized on a slide to maintain the structure of the tissue before being stained, allowing the visualization of antigens in the context of whole tissues (Figure 1). In the case of ICC, cells are distributed evenly on a slide before being stained, allowing the visualization of antigen distribution within individual cells but not within the structure of any specific tissue. Due to the similarities between the two protocols, this protocol will focus on IHC to address the additional complexities of sample preparation involved in IHC.
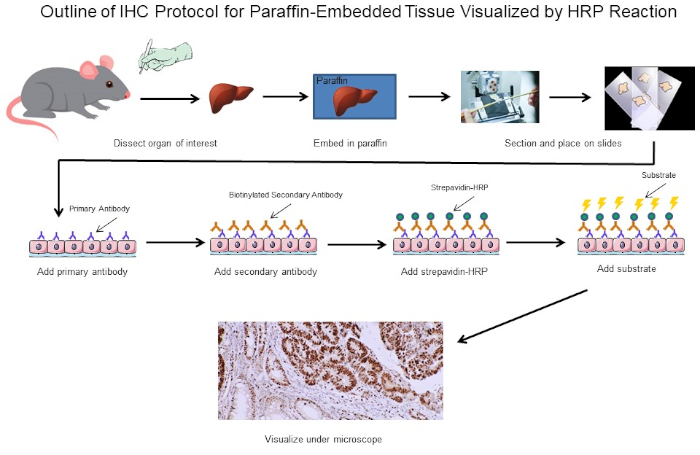
Figure 1: Outline of IHC Protocol. Visual outline of an IHC protocol for paraffin-embedded tissue dissected from a mouse. This protocol uses a biotinylated secondary antibody and strepavidin-HRP to visualize the location of antibody binding. Other options, such as fluorescently tagged antibodies, are also possible. Please click here to view a larger version of this figure.
The first major decision when performing IHC is how to prepare the tissue sections in order to maintain the structure of the tissue throughout the staining process. The two main choices are formalin-fixed sections of paraffin-embedded tissue or fresh sections of frozen tissue. There is no simple answer as to which method to use as it depends on what downstream analysis will be conducted. Formalin-fixation of paraffin embedded tissues is generally thought to better preserve tissue morphology for optimal imaging while freezing fresh tissue can preserve protein function for subsequent assays outside of IHC. In addition, fresh frozen tissue sections have been shown to be more suitable for gene expression analysis (2). A third consideration is whether or not the antibodies for your antigen of interest are suitable for either fixed or frozen tissue sections, as some antibodies have only been optimized for a specific type of section and may not work for others. Finally, one also needs to determine how long they need to store the tissue sections, as fresh frozen samples must be kept at -80°C and may not last beyond one year while fixed sections can be stored for much longer at room temperature. These are a few of the major considerations for determining whether to use formalin-fixed sections of paraffin-embedded tissue or fresh sections of frozen tissue. Ultimately, if one has enough tissue, it may be best just to have some of both.
In this experiment, we set out to determine if cyclin D1 expression was increased in enlarged spleens from a spontaneous mouse model of lymphoma development. Splenic tissue samples were first isolated from either wild-type mice, transgenic mice that do not have lymphoma, or transgenic mice that have spontaneously developed lymphoma. The spleen tissue samples were fixed in paraformaldehyde, embedded in paraffin, sectioned, stained using a mouse anti-cyclin D1 primary antibody followed by a horse anti-mouse secondary antibody, and developed using 3,3-diaminobenzidine (DAB). The sections were then counterstained in Harris Hematoxylin Solution and then the sections were imaged at 20X magnification.
Reagents
Paraffin-Embedded Sections
- 4% Paraformaldehyde (PFA)
- Ethanol (anhydrous denatured, histological grade 100%, 95%, 80%, 75%, and 50%). Can be diluted from 100% stock using double distilled water (ddH2O)
- Xylene
- IHC compatible glass slide to ensure tissue section remains attached throughout the entire procedure. IHC compatible glass slides have a specialized coating and are readily available from multiple retailers. If performing ICC, use a chambered slide. Chambered slides allow for cells to be seeded in the chambers and placed in the incubator until cells attach to the slide and reach proper confluency, at which point the chambers can be removed and staining can proceed in the same manner as IHC.
- Paraffin
- 0.3% Hydrogen peroxide (H2O2)/methanol: To prepare, add 1 mL 30% H2O2 to 99 mL methanol. Store at -20°C
- Antigen retrieval buffer: IHC citrate buffer pH 6.0
Fresh Frozen Sections
- Optimal cutting temperature (OCT) embedding compound
- Optimal fixative: 4% PFA or acetone that has been cooled to -20°C
Staining
- Blocking buffer: Should be determined by user. One example is horse serum diluted in 1X PBS
- Diluted primary antibody: see manufacturer specifications
- Diluted biotinylated secondary antibody: see manufacturer specifications
- Diluted avidin-Horseradish Peroxidase (HRP): Only for peroxidase visualization. See manufacturer specifications.
- DAB or another compatible substrate
- Counterstain (optional)
- Ethanol (anhydrous denatured, histological grade 100% and 95%)
- Xylene
- Organo/Limonene Mount
Procedure
1. Preparation of Cells for Immunocytochemistry
- Seed cells of interest onto chambered slides or chambered coverslips by adding 0.5 mL of cell suspension to the wells of a 24 well culture plate.
Note: Some cells may require growth on treated coverslips, such as coverslips treated with poly-lysine. Optimal treatment conditions should be determined by the user depending on the cell type being used. - Place the plate into a humidified CO2 incubator and allow the cells to grow at 37°C until 50-70% confluent.
- Once the cells reach optimal confluency, remove the culture media from each well and then, fix the cells by incubating them in 0.5 mL of 4% PFA (diluted in 1X PBS) and incubate for 20 min at room temperature.
- Remove the fixative and wash the wells three times with 1 mL of 1X PBS.
- Next, permeabilize the cells by adding 0.5 mL of 0.1% Triton-X-100 in 1X PBS to each well and incubate for 15 min at room temperature.
- Aspirate off the permeabilization buffer and wash the wells thrice with 1 mL of 1X PBS.
- The cells on coverslips are now fixed and permeabilized. Proceed to the staining procedure demonstrated for the following immunohistochemistry example- with the exception that the incubations should be performed within the wells of the 24 well plate rather than directly on a tissue section slide.
2. Preparation of Formalin-Fixed, Paraffin-Embedded Sections for Staining
- Obtain prepared formalin-fixed, paraffin embedded tissue sections.
Deparaffinization
- Immerse the slides in 100% xylene 2 times for 5 min each.
Rehydration
- Immerse the slides in 100% ethanol 2 times for 3 min each.
- Immerse the slides in 95% ethanol for 3 min.
- Immerse the slides in 70% ethanol for 3 min.
- Immerse the slides in 50% ethanol for 3 min.
Blocking Endogenous Peroxidase Activity
- Incubate the slides in 100 mL of 0.3% H2O2 for 30 min at room temperature.
- Wash the slides with 1X PBS 2 times for 5 min each.
Antigen Retrieval
- Immerse the slides in IHC citrate buffer (pH 6) and boil them for 20 min.
- Tissue slides are now ready for staining.
3. Preparation of Fresh-Frozen, OTC-Embedded Sections for Staining
- Place 5 mm of fresh isolated tissue into a mold and add OCT until the section is completely covered.
- Slowly submerge the tissue block into liquid nitrogen until completely frozen. The sample can now be stored at -80°C for up to 1 year.
- Once ready for sectioning, transfer the frozen tissue block to a cryostat and allow the entire setup to come to -20°C.
- Cut 5-10 µm thick tissue sections using a cryostat and use brush to place sections directly onto IHC compatible glass slides.
- Allow slides to dry overnight at room temperature. The slides can also be stored at -80°C.
- Immerse the slides in 250 mL of 4% PFA for 15 min at room temperature to fix the slides prior to staining. Optimal fixation method should be determined by user.
- Immerse the slides in 250 mL of 1X PBS 2 times for 5 min each.
- Immerse the slides in 250 mL of 0.3% H2O2 for 30 min at room temperature in order to block any endogenous peroxidase activity.
- Immerse the slides in 250 mL of 1X PBS 2 times for 5 min each.
- The slides are now ready for staining.
4. Staining
- Circle the tissue with a hydrophobic barrier using a barrier pen.
Blocking
- Using a pipette, place 100 µL of blocking buffer (horse serum diluted in 1X PBS) - over the section for 1 hour at room temperature.
- Remove blocking buffer using a pipette.
Primary Antibody Incubation
- Incubate the encircled tissue with 100 µL diluted primary antibody solution (mouse anti-human cyclin D1 diluted 1:100 in blocking buffer) for 30 min at room temperature.
- Drain the primary antibody off each slide and wash the slides with 1X PBS 2 times for 5 min each.
Secondary Antibody Incubation
- Incubate the sample with 100 µL of diluted biotinylated secondary antibody (biotinylated horse anti-mouse IgG diluted 1:200) for 30 min at room temperature.
- Remove the secondary antibody by draining off the sections and wash with 1X PBS 2 times for 5 min each.
Color Development
- Visualization using HRP: Add 100 µL of avidin-biotin complex (ABC) reagent and incubate the sections in dark in for 30 min at room temperature
Note: Fluorescently tagged antibodies can also be used and visualized using an appropriate microscope. - Wash the slides with 1X PBS 2 times for 5 min each.
- Develop the slides by incubating the sections in 100 µL of DAB for up to 5 min.
- Stop the development by adding distilled water (dH2O) for 5 min at room temperature.
Counterstaining (if desired)
- Immerse the slides briefly in Harris Hematoxylin Solution (or 0.5% methyl green in 0.1M sodium acetate (pH 4.2)) for 10 min.
- Rinse off the counterstain by washing slides in dH2O two times for 5 min each.
Dehydration
- Immerse the slides in 95% ethanol 2 times for 5 min each.
- Immerse the slides in 100% ethanol 2 times for 5 min each.
- Immerse the slides in 100% xylene 2 times for 5 min each.
- Blot the slides with a paper towel.
Mounting and coverslip application
- Add a drop of mounting media such as Organo-Limonene Mount to the slides and place a coverslip over the sections.
Microscopic analysis
- Observe the stained sections under an appropriate microscope for analysis. Here a standard light microscope was used for observation and a mounted digital camera was used for imaging.
Immunocytochemistry and immunohistochemistry are staining methods for a protein of interest in cultured cells and tissues, respectively. The basic principle of both related techniques involves using specific antibodies tagged with a detection system to identify and visualize the protein and determine its location within the cells and tissues, as well as the relative levels. The process in either experiment begins with sample preparation.
For immunocyctochemistry, which specifically visualizes protein or antigen locations in cells, this involves three steps. The first step is plating, which entails culturing the cells in growth media on a cover slip or slide, typically, in the wells of a culture plate. This is followed by fixation, where a precipitating or crosslinking agent like paraformaldehyde is added to the cells to preserve the structural integrity of the proteins and prevent enzyme activity from degrading them. The last step is permeabilization, which involves adding a detergent to make the cell membranes permeable for the staining.
In the counterpart method, immunohistochemistry, proteins or antigens are visualized in tissues and sample preparation has five steps. First, the whole tissue is subjected to fixation, usually with paraformaldehyde. This is followed by embedding of the tissue in a block of paraffin, and then sectioning of this block using a machine called a microtome to cut the tissue into thin slices which can be placed onto slides. Next, the slides are subjected to deparaffinization, or removal of the paraffin from around the tissue slice. Then, an optional antigen retrieval step can be performed. This can either be done using heat or enzymes to unmask epitopes that were cross-linked during fixation making them available for antibody binding. After the appropriate sample preparation, a target-specific primary antibody is added to the cell or tissue sample. This primary antibody should bind to the protein of interest. Next, a secondary antibody is added, which detects and binds to the primary antibody. This secondary antibody is conjugated to, or can bind to, an enzyme called HRP. When its specific substrate, DAB, is added, HRP converts this to an insoluble, brown precipitate. This brown stain marks the location of the target protein. The slides are also stained with hematoxylin, which labels the nuclei in blue and provides a spatial reference point for determining subcellular localization. After that, mounting media is added to the slide, followed by a cover slip in order to seal and preserve the stained sample. Finally, the slides can be imaged on a light microscope.
In this video, you will observe the sample preparation technique for plated cells and tissue sections, followed by immunostaining of the tissue sections.
First, the cells of interest need to be seated onto coverslips. To do this, working in a tissue culture hood, place individual coverslips into the wells of a 24-well plate. Then, close the sash and turn on the UV light to sterilize the coverslips for at least 15 minutes. Next, turn off the UV light. To lift the cells of interest from a confluent 10-centimeter dish, aspirate the media, wash briefly with PBS, and add trypsin to the cells for 2 minutes. Then, tap the side of the plate to ensure the cells have detached and neutralize the trypsin with media. Next, add 0. 5 mL of the cell suspension into each well, making sure to cover the coverslips. Place the plate into a humidified CO2 incubator and allow the cells to grow at 37 degrees celsius until they are 50-70% confluent.
Once the cells reach the optimal confluency, aspirate the culture medium from each well, and then fix the cells by incubating them in . 5 mL of 4% paraformaldehyde diluted in 1X PBS for 20 minutes at room temperature. After removing the fixative, rinse the cells be adding 1 mL of 1X PBS over each coverslip. Immediately aspirate the PBS, then repeat the rinse 2 more times for a total of 3 washes.
Now, permeablize the cells by adding 0.5 mL of 0.1% Triton X-100 in 1X PBS to each well. Leave the plate at room temperature for 15 minutes. Aspirate off the permeabilization buffer and then rinse the cells by adding 1 mL of 1X PBS into each well. Immediately aspirate off the PBS and repeat the rinse 2 more times for a total of 3 washes. Now that the cells on the coverslips are fixed and permeabilized, proceed to the staining procedure demonstrated for the following immunohistochemistry example with the exception that the incubations should be performed within the wells of the 24-well plate rather than directly on a tissue section slide.
To begin, obtain prepared, formalin-fixed, paraffin-embedded tissue sections. Deparaffinize the slides by placing them into a slide rack and then completely immersing them into 250 mL of 100% xylene. Allow the slides to incubate for 5 minutes in the xylene. Then, remove the slides from the container, wipe them off with a paper towel, and place them into a new xylene bath in a fresh container for a further 5 minutes.
Next, rehydrate the sections in a series of graded ethanol solutions starting with 100% ethanol for 3 minutes. Wipe off the slide rack with a paper towel and transfer the slides to a new container of 100% ethanol for another 3 minutes. Continue this cycle of washing, drying with a paper towel, and transferring the slides to a new bath following the indicated concentrations of ethanol for the specified time. After the final ethanol wash, wipe off the rack with a paper towel and incubate the slides in 100 mL of .3% hydrogen peroxide for 30 minutes at room temperature in order to block any endogenous peroxidase activity. Wash the slides in 250 mL of 1X PBS for 5 minutes. Repeat this wash in a container of fresh 1X PBS for an additional 5 minutes.
Next, perform antigen retrieval by immersing the slides in 250 mL of IHC citrate buffer at pH 6.0 and boiling them for 20 minutes. Then, proceed to the staining protocol.
To begin the staining process for IHC, circle the sections with a hydrophobic pen to identify the minimal area that the buffer needs to cover. Then, use a pipette to place 100 microliters of blocking buffer, which in this experiment is horse serum diluted in 1X PBS, over the section. Incubate the slides for 1 hour at room temperature. Following this, remove the blocking buffer using a pipette.
Next, dilute the primary antibody and blocking buffer at a 1:100 dilution by adding 990 microliters of horse serum diluted in 1X PBS into a 1. 5 mL Eppendorf tube, followed by 10 microliters of the primary antibody. Add 100 microliters of the diluted primary antibody to each section, and incubate the slides for 30 minutes at room temperature. When the timer sounds, drain the primary antibody off each slide, and then wash them in 250 mL of 1X PBS for 5 minutes. Repeat this wash once more using fresh 1X PBS.
While the slides are washing in 1X PBS, dilute the secondary antibody to a 1:200 dilution by adding 995 microliters of blocking buffer to a 1.5 mL tube followed by 5 microliters of the secondary antibody, which in this case is biotinylated horse anti-mouse IGG. Add 100 microliters of the diluted secondary antibody to each section, and then incubate the slides for 30 minutes at room temperature. After 30 minutes, remove the secondary antibody by draining it off the sections, then wash the slides in 250 mL of 1X PBS for 5 minutes. Repeat this wash using fresh 1X PBS.
Now, add 100 microliters of avidin-biotin complex reagent, and incubate the sections in the dark for 30 minutes at room temperature. Next, wash the slides by immersing them in 250 mL of 1X PBS for 5 minutes. Similar to previous wash steps, repeat this wash one more time using fresh 1X PBS. Next, develop the slides by incubating the sections in 100 microliters of DAB for up to 5 minutes. Stop the development by immersing the sections in 250 mL of distilled water for 5 minutes.
Now, slides can be counterstained, if desired. To do this, briefly dip the slides in 250 mL of Harris Hematoxylin Solution. Rinse off the counterstain by washing the slides in 250 mL of distilled water for 5 minutes. Repeat this wash 1 more time using fresh distilled water. Next, dehydrate the sections. To do this, first incubate the slides in 95% ethanol for 5 minutes. Blot the slides on a paper towel, and transfer them to a new container of fresh 95% ethanol for another 5 minutes. Continue the cycle of washing, blotting with a paper towel, and transferring the slides to a new bath, following the indicated solutions for 5 minutes each.
After the final incubation, blot the slides with a paper towel, then add a drop of mounting media, such as Organo-Limonene Mount, to the slides. Now, place a coverslip over the sections, taking care not to trap air bubbles. The slides are now ready to be observed under a microscope for analysis.
To observe the stained sections, use a standard light microscope to visualize the stain, and a digital camera to capture the image. In this particular example of IHC, spleen tissues from wild type and spontaneous, double-transgenic, or DTG mice, are compared for studying Dyclin D1 expression in lymphoma. The tissues were paraffin-embedded, sectioned, and stained with anticyclin D1 antibody, and imaged at 20X magnification. Cyclin D1 expressing cells are indicated by the reddish-brown color against the blue tissue background. Comparing the staining intensities among the images from the various mice, the non-enlarged spleens have relatively low amounts of Cyclin D1 expression irrespective of the mouse genotype. In contrast, the enlarged spleen from the DTG mouse, shows increased reddish-brown staining indicating a correlation between cancer development and Cyclin D1 expression in this mouse model.
Subscription Required. Please recommend JoVE to your librarian.
Results
IHC and ICC have a vast range of applications. For example, one use of IHC is to examine the expression of oncogenes in spontaneous mouse models of tumor development. In Figure 2, we set out to determine if cyclin D1 expression was increased in enlarged spleens in a spontaneous mouse model of lymphoma development. Splenic tissue samples were fixed in paraformaldehyde, embedded in paraffin, sectioned, stained using an anti-cyclin D1 antibody (diluted 1:200 in blocking buffer), and then the sections were imaged at 20X magnification. Cyclin D1 expressing cells are indicated by the reddish-brown color against the blue tissue background. These results suggest that cyclin D1 expression was increased in enlarged spleens, indicating a correlation between cancer development and cyclin D1 expression in this model.

Figure 2: Splenic Cyclin D1 Expression in a Spontaneous Double Transgenic (DTG) Mouse Model of Lymphoma. An image of splenic tissue stained with an anti-Cyclin D1 primary antibody, counterstained with methyl green, and visualized using a biotinylated secondary antibody and ABC reagent activated with DAB substrate. The reddish-brown color represents locations where the antibody has bound indicating the presence of Cyclin D1 expressing tumor cells within the structure of splenic tissue that has been counterstained blue. Please click here to view a larger version of this figure.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
Immunohistochemistry (IHC) and immunocytochemistry (ICC) are techniques used to visualize the expression and localization of specific antigens using antibodies. Tissues are first cut into thin sections that maintain the tissue morphology and placed on a slide. The antibodies are then added and will bind the antigen of interest and are equipped with a specific tag that allows them to be visualized under a microscope. Thus, through this basic concept, the distribution of antigens in the context of tissue structure can be visualized and studied. However, while the overarching concept is basic, there are multiple different approaches and variations that have been developed that increase both the complexity and usefulness of these techniques. This paper has covered the basic concept of IHC and ICC, the main decisions that need to be considered when using these techniques, and a detailed step-by-step protocol. The images produced by IHC and ICC are generally the final product and can be published as is to highlight obvious differences in amounts or distribution of staining between different conditions.
Subscription Required. Please recommend JoVE to your librarian.
References
- Coons, A. H. Creech, H. J., Jones, N. and Berliner, E. The Demonstration of Pneumococcal Antigen in Tissues by the Use of Fluorescent Antibody, The Journal of Immunology, 45 (3), 159-170 (1942).
- Ripoli, F. L., Mohr, A., Hammer, S. C., Willenbrock, S., Hewicker-Trautwein, M., Hennecke, S., Escobar, H. M. and Nolte, I. A comparison of fresh frozen vs. Formalin-fixed, paraffin-embedded specimens of canine mammary tumors via branched-DNA assay. International Journal of Molecular Sciences, 17 (5) (2016).




