Summary
Hierbei wird ein Verfahren beschrieben, um die Bindungsaffinität (KD) von radioaktiv markierten Antikörpern an immobilisierte Antigene zu bestimmen. KD ist die Gleichgewichtsdissoziationskonstante, die aus einem Sättigungsbindungsexperiment bestimmt werden kann, indem die gesamte, spezifische und unspezifische Bindung eines radioaktiv markierten Antikörpers in verschiedenen Konzentrationen an sein Antigen gemessen wird.
Abstract
Die Bestimmung der Bindungsaffinität (KD) ist ein wichtiger Aspekt bei der Charakterisierung radioaktiv markierter Antikörper (rAb). Typischerweise wird die Bindungsaffinität durch die Gleichgewichtsdissoziationskonstante KD dargestellt und kann als die Konzentration von Antikörpern berechnet werden, bei der die Hälfte der Antikörperbindungsstellen im Gleichgewicht besetzt ist. Diese Methode kann auf jeden radioaktiv markierten Antikörper oder andere Protein- und Peptidgerüste verallgemeinert werden. Im Gegensatz zu zellbasierten Methoden ist die Wahl immobilisierter Antigene besonders nützlich, um Bindungsaffinitäten nach Langzeitlagerung von Antikörpern zu validieren, Bindungsaffinitäten von Fragment-Antigen-Bindungsregion-Armen (Fab) in bispezifischen Antikörperkonstrukten zu unterscheiden und festzustellen, ob es eine Variabilität in der Antigenexpression zwischen verschiedenen Zelllinien gibt. Bei dieser Methode wird eine feste Menge Antigen in spezifizierte Vertiefungen auf einer zerbrechlichen 96-Well-Platte immobilisiert. Dann wurde die unspezifische Bindung in allen Vertiefungen mit Rinderserumalbumin (BSA) blockiert. Anschließend wurde der rAb in einem Konzentrationsgradienten zu allen Vertiefungen gegeben. Es wurde ein Konzentrationsbereich gewählt, damit der rAb eine Sättigung erreichen kann, d. h. eine Antikörperkonzentration, bei der alle Antigene kontinuierlich an das rAb gebunden sind. In bestimmten Vertiefungen ohne immobilisiertes Antigen kann eine unspezifische Bindung des rAb bestimmt werden. Durch Subtrahieren der unspezifischen Bindung von der Gesamtbindung in den Vertiefungen mit immobilisiertem Antigen kann die spezifische Bindung des rAb an das Antigen bestimmt werden. Das KD des rAb wurde aus der resultierenden Sättigungsbindungskurve berechnet. Als Beispiel wurde die Bindungsaffinität mit radioaktiv markiertem Amivantamab bestimmt, einem bispezifischen Antikörper für epidermale Wachstumsfaktorrezeptoren (EGFR) und zytoplasmatische mesenchymal-epitheliale Übergangsproteine (cMET).
Introduction
Radioaktiv markierte Antikörper (rAb) haben eine Vielzahl von Anwendungen in der Medizin. Während die Mehrheit in der Onkologie als bildgebende und therapeutische Mittel eingesetzt wird, gibt es bildgebende Anwendungen für rheumatologische Entzündungen, Kardiologie und Neurologie1. Imaging rAbs hat eine hohe Empfindlichkeit, Läsionen zu erkennen und hat das Potenzial, bei der Patientenauswahl für die Behandlung zu helfen 2,3,4,5. Sie werden auch wegen ihrer Spezifität für ihre jeweiligen Antigene zur Therapie eingesetzt. In einer Strategie, die als Theranostik bekannt ist, wird derselbe rAb sowohl für die Bildgebung als auch für die Behandlungverwendet 6.
Im Idealfall ist der für die Radiomarkierung gewählte Antikörper einer, der bereits mit nicht radioaktiv markierten Methoden eine hohe Bindungsaffinität und Spezifität aufweist. Die Radiomarkierung von Antikörpern kann durch direkte chemische Modifikation von Antikörpern mit einem Radionuklid, das stabile kovalente Bindungen bildet (z. B. Radiojod), oder indirekt durch Konjugation mit Chelatoren, die sich anschließend mit Radiometallen koordinieren 7,8, erreicht werden. Die direkte Radiomarkierung wie mit Radiojod modifiziert spezifisch Tyrosin- und Histidinreste auf dem Antikörper. Wenn diese Rückstände für die Antigenbindung wichtig sind, dann würde diese Radiokonjugation die Bindungsaffinität verändern. Umgekehrt gibt es mehrere etablierte Protokolle für die Konjugation und indirekte radioaktive Markierung von Antikörpern. Ein gebräuchlicher Chelator, der zur Bindung von Zirkonium-89 (89Zr) für die PET-Bildgebung von Antikörpern verwendet wird, ist beispielsweise das p-Isothiocyanatobenzyl-Desferrioxamin (DFO), das zufällig an Lysinreste des Antikörpers 9,10 konjugiert wird. Wenn Lysinreste in der Antigenbindungsregion vorhanden sind, könnte die Konjugation an diesen Stellen die Antigenbindung sterisch behindern und somit die Antikörper-Antigen-Bindung beeinträchtigen. Daher können die verschiedenen Radiokonjugationsmethoden, die für die indirekte oder direkte Radiomarkierung von Antikörpern verwendet werden, möglicherweise die Immunreaktivität beeinflussen, definiert als die Fähigkeit des Antikörper-Radiokonjugats, an sein Antigen 7,11 zu binden. Ortsspezifische Konjugationsmethoden können diese Einschränkung umgehen, aber diese Techniken erfordern Antikörper-Engineering, um zusätzliche Cysteinreste oder Fachwissen in enzymatischen Reaktionen auf Kohlenhydratrestezu integrieren 12,13,14,15,16. Sobald ein Antikörper radioaktiv markiert ist, ist es wichtig zu testen, ob die Immunreaktivität als Teil der Charakterisierung des rAb erhalten bleibt. Eine Möglichkeit, die Immunreaktivität zu messen, besteht darin, die Bindungsaffinität des rAb zu bestimmen.
Der Zweck dieses Protokolls ist es, ein Verfahren zur Bestimmung der Bindungsaffinität für rAbs unter Verwendung eines etablierten Radioligandensättigungsassays zur Quantifizierung der rAb-Antigenbindung zu beschreiben. Der Bindungstrend ist in Abbildung 1 dargestellt. Die Menge des gebundenen Antigens nimmt zu, wenn mehr rAb zu einer festen Menge an immobilisiertem Antigen hinzugefügt wird. Sobald alle Antigenbindungsstellen gesättigt sind, wird ein Plateau erreicht, und das Hinzufügen weiterer rAbs hat keinen Einfluss auf die Menge des gebundenen Antigens. In diesem Modell ist die Gleichgewichtsdissoziationskonstante (KD) die Konzentration von Antikörpern, die die Hälfte der Antigenrezeptoren einnimmt17. Der K D stellt dar, wie gut ein Antikörper mit einem niedrigeren KD, der einer höheren Bindungsaffinität entspricht, an sein Ziel bindet. Es wurde bereits berichtet, dass ein idealer rAb ein KD von 1 Nanomolar oder weniger18 haben sollte. Neuere rAbs wurden jedoch mit KD im niedrigen nanomolaren Bereich entwickelt und gelten als geeignet für nichtinvasive Bildgebungsanwendungen 19,20,21,22. Ein weiterer Parameter, der im Radioligandensättigungsassay von rAbs bestimmt werden kann, ist Bmax, was der maximalen Menge an Antigenbindung entspricht. Bmax kann verwendet werden, um die Anzahl der Antigenmoleküle bei Bedarf zu berechnen.

Abbildung 1: Repräsentative Sättigungsbindungskurve. Der Prozentsatz des gebundenen Antigens wird gegen steigende Konzentrationen von Antikörpern aufgetragen, die einer festen Menge an Antigen zugesetzt werden. Pop-Outs demonstrieren an verschiedenen Stellen die Bindung. Die Konzentration und Bindung entsprechend KD bzw. Bmax sind dargestellt. Diese Figur wurde mit BioRender.com erstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Dieser Assay ist besonders wichtig für radioaktiv markierte bispezifische Antikörperkonstrukte, um das KD für jeden Fragment Antigen-bindenden Region (Fab) Arm des radioaktiv markierten bispezifischen Antikörpers zu bestimmen, der mit ihren jeweiligen Antigenen bindet. Dieses Protokoll kann verwendet werden, um das KD jedes Fab-Arms separat auf immobilisierten Antigenen zu bestimmen, um unabhängig zu charakterisieren, ob die Bindungsaffinität jedes Fab-Arms für sein jeweiliges Antigen nach der Radiokonjugation beeinflusst wurde. Dieses Protokoll wird durch die Verwendung von radioaktiv markiertem Amivantamab demonstriert, einem bispezifischen Antikörper für epidermale Wachstumsfaktorrezeptoren (EGFR) und zytoplasmatische mesenchymal-epitheliale Übergangsproteine (cMET)19. Radiomarkierte einarmige Antikörper, bei denen ein Fab-Arm an EGFR (α-EGFR) oder cMET (α-cMET) bindet und der andere Fab-Arm eine Isotypkontrolle ist, wurden ebenfalls als Beispiele19 verwendet. Dieses Protokoll ist auch für jeden radioaktiv markierten Antikörper mit einem bekannten Antigen geeignet, der immobilisiert werden kann. In diesem Protokoll wird eine Verdünnungsreihe des rAb zu einer festen Menge immobilisiertem Antigen in bestimmten Vertiefungen zugegeben, die für jeden Fab-Arm des rAb spezifisch sind. Der rAb wird auch zu Vertiefungen gegeben, die nur mit Rinderserumalbumin (BSA) ohne Antigen blockiert wurden, um die unspezifische Bindung zu bestimmen. Um die spezifische Bindung zu bestimmen, wird die unspezifische Bindung an immobilisiertes Antigen von der gesamten rAb-Bindung subtrahiert. Die resultierende Sättigungsbindungskurve wird dann verwendet, um KD zu bestimmen, wie oben beschrieben.
Ein Vorteil dieser Methode ist die höhere Reproduzierbarkeit bei der Verwendung von gereinigten Antigenen im Vergleich zur Verwendung von Zelllinien als Quelle von Antigenen, da die Antigenexpressionsniveaus während der Zellkultur beeinflusst werden können und dass verschiedene Zelllinien unterschiedliche Antigenexpressionsniveaus aufweisen. Bei radioaktiv markierten bispezifischen Antikörpern sind Zelllinien, die nur eines der Antigene ohne das andere exprimieren, möglicherweise nicht verfügbar, was eine Charakterisierung der Bindungsaffinität der einzelnen Fab-Arme sehr schwierig machen würde. Insbesondere ist der Hauptvorteil der Radioligandensättigungsmethode gegenüber nicht radioaktiv markierten Methoden die spezifische Charakterisierung der Bindungsaffinität des rAb ohne den Beitrag der unkonjugierten Fraktion des rAb. Nach bestem Wissen der Autoren gibt es derzeit keine Reinigungstechniken, um den rAb von seinem übergeordneten unkonjugierten Antikörper zu trennen. Angesichts der relativ geringen Größe des Chelators und des Radionuklids ist ihr Beitrag zum Gesamtmolekulargewicht des rAb in der Größenausschlusschromatographie unbedeutend. Daher ist das Produkt, das bei jeder radioaktiven Markierungstechnik erzeugt wird, fast immer eine Mischung aus dem rAb und seinem unkonjugierten Antikörper aus Eltern. Die Charakterisierung der Bindungsaffinität mit dem radioaktiv markierten Sättigungsassay stellt sicher, dass das zu prüfende Produkt ausschließlich das rAb ist.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
HINWEIS: Eine grafische Darstellung des Protokolls finden Sie in Abbildung 2.

Abbildung 2: Schematische Darstellung des Protokolls Zeilen- und Spaltenbeschriftungen werden als Leitfaden für die Einrichtung der zerbrechlichen 96-Well-Platte angegeben. Die erwartete Bindung wird in einem Beispielbrunnen für das Antigen und die BSA gezeigt. Der gekrümmte Pfeil bezeichnet den rAb, von dem erwartet wird, dass er nur mit BSA aus Bohrlöchern ausgewaschen wird. Diese Figur wurde mit BioRender.com erstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
1. Puffervorbereitung
- Es werden 50 ml Immobilisierungspuffer (wässrige Lösung von 50 mMNa2 CO3; pH = 9,0) hergestellt.
- 191 mgNaHCO 3 und 23,9 mgNa2 CO3 werden auf dem Wiegepapier eingewogen und in ein 50 ml konisches Röhrchen überführt. Fügen Sie 40 ml 18 MΩ Wasser und Wirbel hinzu, um sich aufzulösen. Stellen Sie den pH-Wert bei Bedarf auf 9,0 ein, bevor Sie das Gesamtvolumen mit 18 MΩ Wasser auf 50 ml bringen.
- Bereiten Sie etwa 200 ml Waschpuffer (phosphatgepufferte Kochsalzlösung (PBS) mit 0,05% Tween-20) vor, indem Sie 200 ml PBS und dann 100 μL Tween-20 in eine 250 ml Flasche geben.
- Bereiten Sie 50 ml Bindungspuffer vor (PBS mit 0,05% Tween-20 und 0,1% Rinderserumalbumin (BSA)).
- Wiegen Sie 50 mg BSA auf dem Wiegepapier und geben Sie es in ein 50 ml konisches Röhrchen. 50 ml PBS und dann 25 μL Tween-20 in die Tube geben. Vortex vorsichtig zum Mischen.
- Bereiten Sie 50 ml Blockpuffer vor (3% BSA in PBS).
- Wiegen Sie 1,5 g BSA auf dem Wiegepapier und geben Sie es in ein 50 ml konisches Rohr. Fügen Sie 50 ml PBS hinzu und wirbeln Sie vorsichtig vor, um zu mischen.
HINWEIS: Es wird empfohlen, alle Puffer bis zu 1 Woche bei 4 °C zu lagern, um optimale Ergebnisse zu erzielen.
- Wiegen Sie 1,5 g BSA auf dem Wiegepapier und geben Sie es in ein 50 ml konisches Rohr. Fügen Sie 50 ml PBS hinzu und wirbeln Sie vorsichtig vor, um zu mischen.
2. Antigen-Immobilisierung
- Verdünnen Sie das Antigen im Immobilisierungspuffer, um eine Konzentration von 5 μg/ml zu erreichen.
- Fügen Sie 100 μL Antigen pro Vertiefung zum Boden von 24 Vertiefungen einer zerbrechlichen 96-Well-Flachbodenplatte in einem 8 x 3-Array hinzu (Spalten 1-3 für die Zeilen A-H). Decken Sie die Platte mit Dichtband ab.
HINWEIS: Stellen Sie sicher, dass die Oberfläche der Well-Plate behandelt wurde, um die Adsorption von gemischten hydrophoben und hydrophilen Domänen zu maximieren. Diese vorbehandelten Platten sind im Handel erhältlich. - Über Nacht bei 4 °C inkubieren.
- Am nächsten Tag den Teller 3x mit Waschpuffer waschen.
- Drehen Sie die Platte zügig in der Spüle um, um die Flüssigkeit zu entsorgen, und tippen Sie die Platte auf einen Stapel Papierhandtücher, um die überschüssige Flüssigkeit zu entfernen.
- Fügen Sie mit einer Mehrkanalpipette 300 μL Waschpuffer pro Vertiefung zu den Vertiefungen hinzu, die das Antigen enthalten. Entfernen Sie die Flüssigkeit wie in 2.4.1 beschrieben. Wiederholen Sie den Waschschritt insgesamt dreimal.
3. Sperrung von unspezifischen Seiten mit BSA
- Mit einer Mehrkanalpipette fügen Sie den 24 antigenbeschichteten Vertiefungen und 24 leeren Vertiefungen der 96-Well-Platte (Spalten 1-6 für die Zeilen A-H) 300 μL Blockierpuffer pro Vertiefungsloch hinzu.
- Bebrüten Sie die Platte für 1 h bei Umgebungstemperatur.
- Waschen Sie die Platte mit 300 μL pro Vertiefung des Waschpuffers insgesamt dreimal. In Schritt 2.4 finden Sie eine detaillierte Beschreibung des Waschens der Platte.
4. Serielle Verdünnungen und Zugabe der rAb-Lösung
VORSICHT: Die folgenden Schritte beinhalten Radioaktivität. Schritte sollten nur von Personen mit Strahlenschutzausbildung durchgeführt werden. Forscher sollten den Handschuh verdoppeln und Schritte mit angemessener Abschirmung durchführen.
- Synthetisieren Sie den zu untersuchenden rAb mit der Methode Ihrer Wahl. Die als Beispiel verwendeten rAbs wurden wie zuvorbeschrieben synthetisiert 19.
HINWEIS: Dieses Protokoll konzentriert sich auf die Charakterisierung eines rAb nach der radioaktiven Markierung. - Nehmen Sie 8 x 3-fach serielle Verdünnungen (für die Reihen A-H auf der Platte) des rAb im Bindungspuffer vor.
HINWEIS: Die Konzentrationen der seriellen Verdünnungen variieren für jeden rAb. Details werden im Abschnitt Diskussion erläutert. Wenn sich der Verdünnungsfaktor ändert, sollte das für jede Verdünnung benötigte Volumen neu berechnet werden, um ein ausreichendes Volumen für 1) die Bindung des rAb in jeder Vertiefung, 2) das Aussäen der folgenden Verdünnung und 3) das Aliquotieren einer rAb-Standardlösung für die Gammazählung zur Messung der Radioaktivität des gesamten rAb, das jeder Vertiefung zugesetzt wird, zu gewährleisten.- Berechnen Sie das Volumen des Stamm-rAb, das benötigt wird, um eine 1,2-ml-Lösung der ersten Konzentration herzustellen (Etikett als A).
- 800 μL Bindungspuffer in Mikrozentrifugenröhrchen mit B, C, D, ... zu H. 1,2 ml abzüglich des in Schritt 4.2.1 berechneten Volumens des Bindungspuffers in ein Mikrozentrifugenröhrchen mit der Bezeichnung A geben.
- Fügen Sie das in 4.2.1 berechnete Volumen des Lagers rAb zu Röhre A hinzu. Vortex vorsichtig zum Mischen und dann mit einer Mini-Mikrozentrifuge nach unten drehen, um die gesamte Flüssigkeit am Boden des Tubus zu sammeln.
- 400 μL von Röhrchen A zu Röhrchen B geben. Vortex zum Mischen und dann mit einer Mini-Mikrozentrifuge nach unten drehen. Wiederholen Sie das Addieren von B nach C, C nach D, ..., G nach H.
- Fügen Sie 100 μL pro Vertiefung jeder Verdünnung zu drei mit Antigen immobilisierten Vertiefungen und drei nur mit BSA blockierten Vertiefungen hinzu. Fügen Sie beispielsweise Verdünnung A zu den Vertiefungen A1-A3 (Antigen) und A4-A6 (BSA) hinzu.
- 100 μL jeder Verdünnung in Mikrozentrifugenröhrchen mit der Bezeichnung A std - H std. Speichern Sie diese Röhrchen als rAb-Standards, um im Gammazähler untersucht zu werden.
- Bebrüten Sie die Platte für 1 h bei 37 °C mit sanftem Schaukeln.
5. Waschplatten und Assay Radioaktivität
- Beschriften Sie Mikrozentrifugenröhrchen für jede Vertiefung (A1 bis A6, B1 bis B6 ... bis H1-H6). Verwenden Sie zwei verschiedenfarbige Marker, um Proben auf Wunsch farblich zu kennzeichnen - einen für mit Antigen beschichtete Vertiefungen und einen für Vertiefungen nur mit BSA.
- Saugen Sie den rAb mit einem Vakuumsauger aus jeder Vertiefung ab.
- Verwenden Sie eine Mehrkanalpipette und fügen Sie jedem Bohrloch 300 μL Waschpuffer hinzu. Saugen Sie den Waschpuffer ab. Wiederholen Sie die Wäsche insgesamt fünfmal.
- Brechen Sie die Vertiefungen in die entsprechenden Mikrozentrifugenröhrchen auf.
- Zählen Sie die Radioaktivität in den Röhren mit einem Gammazähler. Zählen Sie zuerst die Röhren mit dem Antigen (H1, H2, H3 bis A1, A2, A3) und dann nur die mit BSA (H4, H5, H6 bis A4, A5, A6). Um Interferenzen zu minimieren, zählen Sie die Standards für jede Verdünnung (H std bis A std) zu einem anderen Zeitpunkt separat.
6. Datenanalyse
HINWEIS: Die ergänzenden Dateien enthalten entsprechende Tabellenkalkulations- und statistische Analysevorlagen zur Analyse und Darstellung der Daten.
- Berechnen Sie in einer Kalkulationstabelle die gesamte, spezifische und unspezifische Bindung für jede Stichprobe (siehe die beigefügte Tabellenkalkulationsvorlage als zusätzliche Datei).
- Berechnen Sie "gebundene Aktivität" als die Anzahl pro Minute (CPM) der Probe (aus dem Gammazähler erhalten) dividiert durch den CPM des entsprechenden Standards. Berechnen Sie "% gebunden" als gebundene Aktivität mal 100.
- Berechnen Sie "Total Bound, mol/L", indem Sie "% Bound" mit der Konzentration (mol/L) des hinzugefügten rAb multiplizieren. Berechnen Sie "Total Bound, mol", indem Sie "Total Bound, mol/L" mit dem Volumen von rAb in Litern (0,0001 L) multiplizieren.
- Berechnen Sie "Spezifische Bindung, mol", indem Sie "Total Bound, mol" der BSA-Verdünnungen von den Antigenverdünnungen subtrahieren, so dass A1 mit A4, A2 mit A5, A3 mit A6, B1 mit B4 usw. gepaart wird.
- Berechnen Sie "Unspezifische Bindung, mol", indem Sie "Spezifische Bindung, mol" von "Gesamtbindung, mol" für jedes Bohrloch subtrahieren.
- In der Plottsoftware für die statistische Analyse wird die Konzentration des hinzugefügten rAb (nmol/L) auf der x-Achse gegenüber der Bindung (mol) auf der y-Achse dargestellt. Erstellen Sie separate Gruppen, um in dreifacher Gesamtbindung, spezifischer Bindung und unspezifischer Bindung zu plotten. Führen Sie eine nichtlineare Anpassungsanalyse durch, indem Sie die folgenden Parameter in der verwendeten Software auswählen (Tabelle der Materialien; siehe die angehängte Vorlage für die statistische Analyse als ergänzende Datei).
- Wählen Sie Neue Analyse aus. Wählen Sie unter XY-Analysen die Option Nichtlineare Regression (Kurvenanpassung) aus. Stellen Sie sicher, dass unter Welche Datensätze analysieren? alle Daten ausgewählt sind, und wählen Sie dann OK aus.
- Wählen Sie auf der Registerkarte Modell unter Bindung - Sättigung die Option Eine Site - Spezifische Bindung aus. Wählen Sie auf der Registerkarte " Vertrauen " die Option " Mehrdeutige" Anpassungen identifizieren aus. Lassen Sie alle anderen Parameter als Standard und wählen Sie OK.
HINWEIS: Dadurch werden KD und Bmax für die gesamte, spezifische und unspezifische Bindung berechnet. Das K D der spezifischen Bindung ist das KD in nmol/L des an das Antigen gebundenen rAb.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Diese Methode berechnet die Bindungsaffinität (KD) für einen rAb basierend auf dem Sättigungsbindungsassay, bei dem verschiedene Konzentrationen von rAb zu einer festen Menge immobilisierten Antigens hinzugefügt wurden. Die Bindungskurve sollte dem logarithmischen Wachstum folgen, wo sie zunächst steil ist und dann stagniert, wenn das Antigen gesättigt ist. Um sicherzustellen, dass die bestimmte KD genau ist, müssen die rAb-Konzentrationen hoch genug sein, um eine Sättigung zu erreichen. Für diesen Assay wurden radioaktiv markierte Antikörper mit DFO konjugiert und mit 89Zr radioaktiv markiert, wie zuvor beschrieben19. Abbildung 3 zeigt repräsentative Sättigungsbindungsdiagramme. Radiomarkiertes Amivantamab, ein bispezifischer Antikörper für den epidermalen Wachstumsfaktorrezeptor (EGFR) und das zytoplasmatische mesenchymal-epitheliale Übergangsprotein (cMET), wurde an EGFR (Abbildung 3A) und cMET (Abbildung 3B) Proteinegebunden 19. Sättigungsbindungsdiagramme werden auch für die einarmigen radioaktiv markierten Fabs gezeigt, die an EGFR (Abbildung 3C) und cMET (Abbildung 3D) gebunden sind19. In allen Teilfiguren waren nichtlineare Regressionskurven repräsentativ für die Verdünnungsreihe mit minimalen Ausreißern aus den Kurven. Die spezifische Bindung entsprach dem größten Teil der Gesamtbindung. Während die unspezifische Bindung insgesamt niedrig war, zeigten höhere Konzentrationen von rAb eine größere unspezifische Bindung, was auf einen möglichen Schwellenwert für unspezifische Bindung in diesem Assay hinweist. Die Bindungsaffinitäten der rAbs lagen im erwarteten nanomolaren Bereich. Die K-D-Werte von 8,4 ± 1,7 nM bzw. 4,2 ± 1,5 nM für den einarmigen α-EGFR bzw. α-cMET zeigen eine ähnliche Affinität für das Zielantigen wie der bispezifische rAb mitK-D-Werten von 9,9 ± 2,1 nM bzw. 16,9 ± 5,9 nM für EGFR bzw. cMET.
Suboptimale Experimente zur Bindung von radioaktiv markiertem α-cMET an immobilisierte cMET-Proteine sind in Abbildung 4 dargestellt, um die Ergebnisse von Parametern zu demonstrieren, die optimiert werden müssen. In Abbildung 4A war der Konzentrationsbereich des hinzugefügten rAb zu niedrig, und die Sättigung wurde nicht erreicht, wie die Bindungskurve zeigt, die kein Plateau erreicht. Diese Bedingungen führten zu einem linearen Diagramm der Konzentration von rAb im Vergleich zu gebundenem rAb. Da die Daten nicht zum nichtlinearen Regressionsmodell passen, wurde der KD-Wert in der statistischen Analyse als "mehrdeutig" eingestuft. In Abbildung 4B wurden zu hohe Konzentrationen für radioaktiv markierte α-cMET verwendet. Die Sättigung wurde erreicht, wie durch das horizontale Plateau der Bindungskurve angezeigt. Unspezifische Bindung trägt jedoch spürbar zur Gesamtbindung bei. Diese Bedingungen führen zu einer hohen Unsicherheit, die durch einen KD-Wert von 14,9 nM mit einem großen 95%-Konfidenzintervall von 4,8 - 44,2 nM angezeigt wird. Daher würde die Optimierung des Konzentrationsbereichs des hinzugefügten rAb, wie in Abbildung 3D gezeigt, genauere KD-Werte liefern. Die Sättigungsbindungskurve basiert auf der Michaelis-Menten-Gleichung für die Reaktionskinetik. Eine Kurve ist eine gute Passform, wenn die Punkte zufällig um die Regressionslinie17 verteilt sind. Ein enges Konfidenzintervall zeigt an, dass der KD-Wert genau ist.

Abbildung 3: Sättigungsbindungsdiagramme zur Bestimmung von KD. Bindungskurven von [89 Zr]Zr-DFO-Amivantamab an (A) EGFR- und (B) cMET-Proteine, (C) [89Zr]Zr-DFO-α-EGFR an EGFR-Protein und (D) [89 Zr]Zr-DFO-α-cMET an cMET-Proteine zur Berechnung der Bindungsaffinität. Fehlerbalken stellen die Standardabweichung des gebundenen 89Zr-markierten Antikörpers für jede Konzentration dar. Mittelwert ± SD von KD und Bmax werden aus drei unabhängigen Experimenten für jede Bedingung gezeigt. Diese Abbildung wird mit Genehmigung von Cavaliere et al19 wiederverwendet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
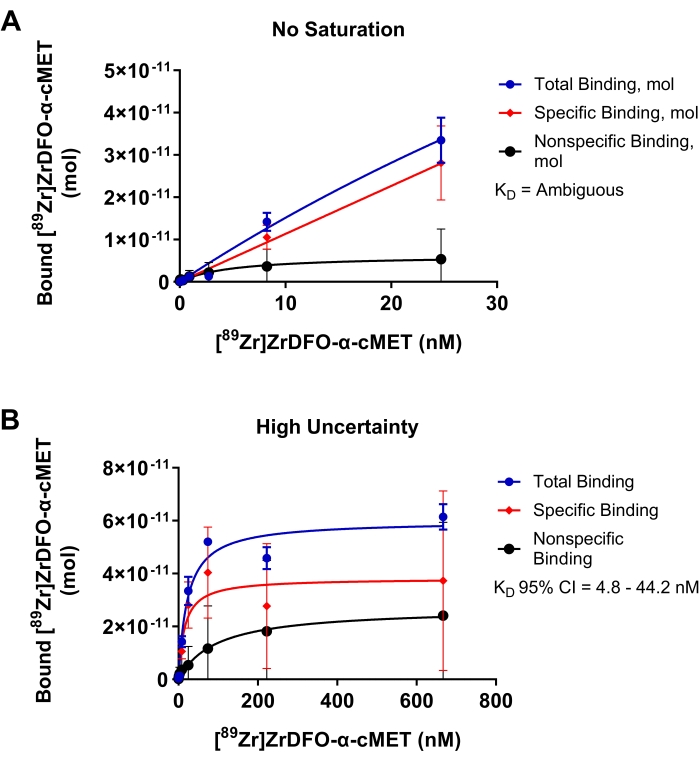
Abbildung 4: Sättigungsbindungsdiagramme, bei denen KD nicht genau bestimmt werden kann. Bindung von [89Zr]Zr-DFO-α-cMET an cMET-Proteine. Fehlerbalken stellen die Standardabweichung der gebundenen [89Zr]Zr-DFO-α-cMET dar. (A) Die Konzentrationen von [89Zr]Zr-DFO-α-cMET sind zu niedrig, und die Sättigung wird nicht erreicht. KD kann nicht berechnet werden, da die Kurve linear ist und nicht zum nichtlinearen Regressionsmodell passt. (B) Eine hohe Unsicherheit für die spezifische Bindung wird durch das breite 95%-Konfidenzintervall (CI) für KD (4,8-44,2 nM) angezeigt. Es gibt auch eine hohe unspezifische Bindung aufgrund hoher Konzentrationen von rAb. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergänzende Dateien. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Im Rahmen der Entwicklung von rAbs ist es wichtig sicherzustellen, dass ein rAb mit hoher Bindungsaffinität spezifisch an sein Ziel bindet. Die Bestimmung der Bindungsaffinität kann darüber informieren, ob die Immunreaktivität des rAb durch Radiokonjugation durch den Radioligandensättigungsassay unter Verwendung eines immobilisierten Antigens beeinflusst wird. Die Bestimmung der rAb-Bindung an BSA kann verwendet werden, um die unspezifische Bindung zu quantifizieren, um die spezifische Bindung an das immobilisierte Antigen genauer zu messen. Diese Methode testet die Bindung verschiedener Konzentrationen von rAb, um eine Sättigungsbindungskurve zu erzeugen, um die KD zu berechnen und die Bindungsaffinität zu bestimmen.
Um diesen Assay durchführen zu können, muss man das Antigen für den zu untersuchenden rAb haben. Das Antigen muss das Epitop haben, an das der Antikörper bindet. In Situationen, in denen neuartige Antikörper unbekannte Bindungsepitope aufweisen, können nicht radioaktiv markierte Methoden zur Bestimmung der Bindungsaffinität besser geeignet sein. Vor Beginn des Experiments wird eine Literaturrecherche empfohlen. Antigene von Interesse, die zuvor in ELISA-basierten Assays verwendet wurden, sind wahrscheinlich für diesen Assay wirksam.
Abhängig von der Halbwertszeit des Radionuklids und der Empfindlichkeit des Gammazählers kann es einige Zeit dauern, bis die Standards auf ein Radioaktivitätsniveau zerfallen, das innerhalb der quantitativen Grenzen des Gammazählers liegt. Wenn erwartet wird, dass der Radioaktivitätsgehalt für den Gammazähler zu hoch ist, verdünnen Sie die Standardproben und multiplizieren Sie sie dann mit dem Verdünnungsfaktor in der Datenanalyse. Dieser Schritt sollte am Tag des Experiments durchgeführt werden, um die Möglichkeit einer Antikörperaggregation oder eines Abbaus zu vermeiden, die zu inhomogenen Standardlösungen und ungenauen Ablesungen führen könnte.
Die Erzeugung einer entsprechend geformten Sättigungskurve basiert auf der gewählten Verdünnungsreihe. Dieser Prozess erfordert Versuch und Irrtum. Die Unfähigkeit, die Konzentrationen von rAbs zu verallgemeinern, ist eine Einschränkung des Protokolls, da von allen rAbs erwartet wird, dass sie unterschiedliche Bindungsaffinitäten für ihre Antigene haben. Dieses Protokoll sollte für jeden rAb optimiert werden. Nach der Optimierung sollte das Protokoll für den spezifischen rAb reproduzierbar sein. Sowohl der Konzentrationsbereich als auch der Verdünnungsfaktor müssen möglicherweise basierend auf der Form der Kurve angepasst werden. Angesichts des erwarteten nanomolaren Bereichs für die Antikörperbindung wird es möglich, mit einer möglichst breiten Darstellung dieses Bereichs zu beginnen, um einen engeren Konzentrationsbereich zu vergrößern, der für zukünftige Studien von Interesse ist. Anstelle von acht seriellen Verdünnungen würden sechzehn mehr Datenpunkte liefern, um den Bindungsbereich zu verstehen. Wenn die KD des unkonjugierten Antikörpers bekannt ist, wird empfohlen, diesen Wert als mittlere Konzentration des Bereichs zu verwenden, wenn Sie die Studie zum ersten Mal entwerfen. Abhängig von der Verfügbarkeit von Materialien können mehrere laufende Versuche mit unterschiedlichen Konzentrationsbereichen und Verdünnungsfaktoren das Protokoll relativ schneller optimieren. Die Inkubationszeit kann angepasst werden, um sicherzustellen, dass die Sättigung erreicht wird. Wenn die Bindung auch bei Sättigung niedrig ist, gibt es möglicherweise nicht genügend Signalbereich, um den KD genau zu berechnen. Eine Optimierung der Konzentration von immobilisiertem Antigen kann erforderlich sein. Eine Vorstudie kann mit unterschiedlichen Konzentrationen von immobilisiertem Antigen und der Stammkonzentration von rAb durchgeführt werden, um das maximale Bindungssignal zu bestimmen.
Der Radioligandensättigungsassay ist eine etablierte Methode und hat im Vergleich zu anderen nicht radioaktiv markierten Methoden wie Durchflusszytometrie oder Oberflächenplasmonenresonanz (SPR) viele Vorteile bei der Bestimmung der Bindungsaffinität von rAbs. Methoden, die den Nachweis von nicht radioaktiv markierten Antikörpern verwenden, bestimmen die Bindungsaffinität einer gemischten Probe, die sowohl aus den konjugierten als auch aus den unkonjugierten Antikörpern besteht. Im Radioligandensättigungsassay ist das einzige registrierte Signal das des radioaktiv markierten Antikörperkonjugats, das eine genauere KD für die radioaktiv markierten Spezies liefert, die die biomedizinische Anwendung beeinflussen würde. Diese Überlegung macht die Ergebnisse des Radioligandensättigungstests und die Ergebnisse von nicht radioaktiv markierten Techniken praktisch unvergleichbar, da sie verschiedene Proben messen - rAb vs. rAb plus Elternantikörper. Eine Studie verglich die Bindungsaffinität von nicht radioaktiv markiertem Antikörper-Chelator-Konjugat und dem elterlichen bispezifischen Antikörper unter Verwendung von SPR auf immobilisierte Proteine. Es gab einen fünffachen Unterschied zwischen denK-D-Werten des Antikörper-Chelator-Konjugats und des elterlichen Antikörpers. Der Unterschied in den K D-Werten, der zwischen SPR und einem Sättigungsassay auf mehreren Zelllinien gefunden wurde, reichte von 10- bis 1.000-fach23. Der nicht radioaktiv markierte Antikörper-Chelator ist eine Mischung aus konjugierten und unkonjugierten Antikörpern, während die durch den Sättigungsassay bestimmtenK-D-Werte nur aus dem radioaktiv markierten konjugierten Antikörper stammen. Es gibt auch Unterschiede in der Antigenexpression zwischen den verwendeten Zelllinien. Daher können nicht radioaktiv markierte Techniken nicht verwendet werden, um den Radioligandensättigungsassay zu validieren. Sobald jedoch die KD des rAb bestimmt wurde, kann ein kompetitiver Bindungsassay durchgeführt werden, um Unterschiede in der Bindung zwischen der elterlichen und der konjugierten Variante des Antikörpers17,21 zu bestimmen.
Der Radioligandensättigungsassay kann erweitert werden, um die Sättigungsbindung des rAb an Zellen anstelle eines immobilisierten Antigens zu bestimmen. Mit den gleichen Konzepten können verschiedene Konzentrationen von rAb zu einer festen Anzahl von Zellen hinzugefügt werden. Die im Protokoll beschriebene Datenanalyse kann dann verwendet werden, um die Bindungsaffinität des rAb zu den Zellen zu bestimmen, was auch Bmax bestimmen würde, um die Anzahl der auf der interessierenden Zelllinie vorhandenen Antigenmoleküle zu berechnen. Diese Methode kann auch auf andere radioaktiv markierte Protein- und Peptidgerüste verallgemeinert werden, um ihre spezifische Bindung an ihre Ziele zu bestimmen. Zusammenfassend beschreibt dieses Protokoll den Prozess der Bestimmung der Bindungsaffinität von rAbs an immobilisierte Proteinantigene. Der Prozess verwendet grundlegende Labortechniken, um serielle Verdünnungen des rAb vorzunehmen, um dem immobilisierten Antigen hinzuzufügen. Die größte Herausforderung besteht darin, die Konzentrationen des rAb zu bestimmen, um eine entsprechend geformte Sättigungsbindungskurve zu erzeugen. Dieser Prozess erfordert Versuch und Irrtum bei der Entwicklung des Assays für einzelne rAbs, wäre dann aber reproduzierbar, sobald er etabliert ist. Die berechnete KD kann als Metrik der Immunreaktivität als Kriterium verwendet werden, um mit bildgebenden oder therapeutischen Anwendungen fortzufahren oder die Antikörper-Chelat-Konjugationsreaktion zu verfeinern.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Die Autoren haben keine Interessenkonflikte.
Acknowledgments
Die Autoren danken 3D Imaging für die Produktion von [89Zr]Zr-Oxalat und Dr. Sheri Moores von Janssen Pharmaceuticals für die Bereitstellung von Antikörpern.
Materials
| Name | Company | Catalog Number | Comments |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A9647 | |
| Gamma Counter | Hidex | Hidex Automatic Gamma Counter | |
| GraphPad Prism Software | GraphPad | version 9.2; used for statistical analyses in this study | |
| Immuno Breakable MaxiSorp 96-well plates | Thermo Scientific | 473768 | |
| Microplate Sealing Tape | Corning | 4612 | |
| Microsoft Excel | Microsoft | ||
| Phosphate Buffered Saline (PBS) | Gibco | 14190144 | |
| Sodium Bicarbonate | JT Baker | 3506-01 | |
| Sodium Carbonate | Sigma-Aldrich | S7795 | |
| Tween-20 | Sigma-Aldrich | P7949 |
References
- Krecisz, P., Czarnecka, K., Krolicki, L., Mikiciuk-Olasik, E., Szymanski, P. Radiolabeled Peptides and Antibodies in Medicine. Bioconjugate Chemistry. 32 (1), 25-42 (2021).
- Dun, Y., Huang, G., Liu, J., Wei, W. ImmunoPET imaging of hematological malignancies: From preclinical promise to clinical reality. Drug Discovery Today. 27 (4), 1196-1203 (2022).
- Lohrmann, C., et al. Retooling a Blood-Based Biomarker: Phase I assessment of the high-affinity CA19-9 antibody HuMab-5B1 for immuno-pet imaging of pancreatic cancer. Clinical Cancer Research. 25 (23), 7014-7023 (2019).
- Pandit-Taskar, N., et al. A phase I/II study for analytic validation of 89Zr-J591 immunoPET as a molecular imaging agent for metastatic prostate cancer. Clinical Cancer Research. 21 (23), 5277-5285 (2015).
- Rousseau, C., et al. Initial clinical results of a novel immuno-PET theranostic probe in human epidermal growth factor receptor 2-negative breast cancer. Journal of Nuclear Medicine. 61 (8), 1205-1211 (2020).
- Moek, K. L., et al. Theranostics using antibodies and antibody-related therapeutics. Journal of Nuclear Medicine. 58 (2), 83-90 (2017).
- Chomet, M., van Dongen, G., Vugts, D. J. State of the art in radiolabeling of antibodies with common and uncommon radiometals for preclinical and clinical immuno-PET. Bioconjugate Chemistry. 32 (7), 1315-1330 (2021).
- Kumar, K., Ghosh, A. Radiochemistry, production processes, labeling methods, and immunoPET imaging pharmaceuticals of Iodine-124. Molecules. 26 (2), 414 (2021).
- Vosjan, M. J., et al. Conjugation and radiolabeling of monoclonal antibodies with zirconium-89 for PET imaging using the bifunctional chelate p-isothiocyanatobenzyl-desferrioxamine. Nature Protocols. 5 (4), 739-743 (2010).
- Zeglis, B. M., Lewis, J. S. The bioconjugation and radiosynthesis of 89Zr-DFO-labeled antibodies. Journal of Visualized Experiments: JoVE. (96), e52521 (2015).
- Wei, W., et al. ImmunoPET: concept, design, and applications. Chemical Reviews. 120 (8), 3787-3851 (2020).
- Tavaré, R., et al. An effective immuno-PET imaging method to monitor CD8-dependent responses to immunotherapy. Cancer Research. 76 (1), 73-82 (2016).
- Tavaré, R., et al. Engineered antibody fragments for immuno-PET imaging of endogenous CD8+ T cells in vivo. Proceedings of the National Academy of Sciences. 111 (3), 1108-1113 (2014).
- Zeglis, B. M., et al. Chemoenzymatic strategy for the synthesis of site-specifically labeled immunoconjugates for multimodal PET and optical imaging. Bioconjugate Chemistry. 25 (12), 2123-2128 (2014).
- Zeglis, B. M., et al. Enzyme-mediated methodology for the site-specific radiolabeling of antibodies based on catalyst-free click chemistry. Bioconjugate Chemistry. 24 (6), 1057-1067 (2013).
- Kristensen, L. K., et al. Site-specifically labeled 89Zr-DFO-trastuzumab improves immuno-reactivity and tumor uptake for immuno-PET in a subcutaneous HER2-positive xenograft mouse model. Theranostics. 9 (15), 4409-4420 (2019).
- Maguire, J. J., Kuc, R. E., Davenport, A. P. Radioligand binding assays and their analysis. in Receptor Binding Techniques. Davenport, A. P. , Humana Press. 31-77 (2012).
- Davenport, A. P., Russell, F. D. Radioligand bindsing assays: theory and practice. Current Directions in Radiopharmaceutical Research and Development. Mather, S. J. , Springer. Netherlands. 169-179 (1996).
- Cavaliere, A., et al. Development of [89Zr]ZrDFO-amivantamab bispecific to EGFR and c-MET for PET imaging of triple negative breast cancer. European Journal of Nuclear Medicine and Molecular Imaging. 48 (2), 383-394 (2021).
- Marquez, B. V., et al. Evaluation of (89)Zr-pertuzumab in breast cancer xenografts. Molecular Pharmaceutics. 11 (11), 3988-3995 (2014).
- Marquez-Nostra, B. V., et al. Preclinical PET imaging of glycoprotein non-metastatic melanoma B in triple negative breast cancer: feasibility of an antibody-based companion diagnostic agent. Oncotarget. 8 (61), 104303-104314 (2017).
- Ghai, A., et al. Development of [(89)Zr]DFO-elotuzumab for immunoPET imaging of CS1 in multiple myeloma. European Journal of Nuclear Medicine and Molecular Imaging. 48 (5), 1302-1311 (2021).
- McKnight, B. N., et al. Imaging EGFR and HER3 through (89)Zr-labeled MEHD7945A (Duligotuzumab). Scientific Reports. 8 (1), 1-13 (2018).



