Summary
यह प्रोटोकॉल डाउनस्ट्रीम माइक्रोस्कोपी अध्ययनों के लिए एस-चरण कोशिकाओं की कुशलतापूर्वक पहचान करने के लिए एक गैर-आक्रामक विधि का वर्णन करता है, जैसे लेजर माइक्रो-विकिरण द्वारा डीएनए मरम्मत प्रोटीन भर्ती को मापना।
Abstract
डीएनए क्षति की मरम्मत अत्यधिक प्रतिक्रियाशील वातावरण में कोशिकाओं की आनुवंशिक अखंडता को बनाए रखती है। कोशिकाएं मेटाबोलिक गतिविधियों या यूवी विकिरण जैसे अंतर्जात और बहिर्जात स्रोतों दोनों के कारण विभिन्न प्रकार के डीएनए क्षति को जमा कर सकती हैं। डीएनए की मरम्मत के बिना, सेल के आनुवंशिक कोड से समझौता हो जाता है, प्रोटीन की संरचनाओं और कार्यों को कम करता है और संभावित रूप से बीमारी पैदा करता है।
डीएनए क्षति मरम्मत के क्षेत्र में विभिन्न कोशिका चक्र चरणों में विभिन्न डीएनए मरम्मत मार्गों की स्थानिक गतिशीलता को समझना महत्वपूर्ण है। वर्तमान फ्लोरोसेंट माइक्रोस्कोपी तकनीक डीएनए क्षति प्रेरण के बाद विभिन्न मरम्मत प्रोटीन की भर्ती काइनेटिक्स को मापने के लिए महान उपकरण प्रदान करती है। कोशिका चक्र के एस चरण के दौरान डीएनए संश्लेषण डीएनए की मरम्मत के बारे में सेल भाग्य में एक अजीब बिंदु है। यह गलतियों के लिए पूरे जीनोम स्क्रीन करने के लिए एक अनूठी खिड़की प्रदान करता है । इसके साथ ही डीएनए संश्लेषण त्रुटियां डीएनए अखंडता के लिए भी खतरा पैदा करती हैं जो गैर-विभाजित कोशिकाओं में सामने नहीं आती है । इसलिए, कोशिका चक्र के अन्य चरणों की तुलना में एस चरण में डीएनए मरम्मत प्रक्रियाओं में काफी अंतर है, और उन मतभेदों को खराब समझा जाता है।
निम्नलिखित प्रोटोकॉल सेल लाइनों की तैयारी और स्थानीय रूप से प्रेरित डीएनए क्षति साइटों पर एस चरण में डीएनए मरम्मत प्रोटीन की गतिशीलता की माप का वर्णन करता है, एक लेजर स्कैनिंग कॉन्फोकल माइक्रोस्कोप का उपयोग करके एक 405 एनएम लेजर लाइन से लैस है। टैग किए गए पीसीएनए (mPlum के साथ) का उपयोग एस चरण में डीएनए क्षति भर्ती को मापने के लिए एक AcGFP-लेबल मरम्मत प्रोटीन ऑफ इंटरेस्ट (यानी, EXO1b) के साथ संयुक्त सेल चक्र मार्कर के रूप में किया जाता है।
Introduction
कोशिकाओं में उत्पन्न होने वाले विभिन्न प्रकार के डीएनए घावों को संबोधित करने के लिए कई डीएनए मरम्मत मार्ग विकसित हुए हैं, जिनमें से सभी अंतरिक्ष और समय दोनों में अत्यधिक विनियमित हैं। कोशिका चक्र के सबसे कमजोर समय में से एक एस चरण है, जब डीएनए संश्लेषण होता है। जबकि प्रसार जीवन के लिए मौलिक है, यह भी एक बड़ी चुनौती प्रदान करता है । कोशिकाओं को अपने जीनोम की वफादार प्रतिकृति सुनिश्चित करने के लिए उत्परिवर्तन से बचने के लिए भविष्य की पीढ़ियों के लिए नीचे पारित किया जाना चाहिए । नतीजतन, प्रसार हस्तक्षेप का एक चिकित्सीय बिंदु प्रदान करता है जिसे ऑन्कोलॉजी के क्षेत्र में चिकित्सीय दृष्टिकोणों के विकास के लिए नियोजित किया गया है।
डीएनए घावों पर प्रोटीन भर्ती का अध्ययन करने के लिए उपयोग की जाने वाली सभी प्रमुख तकनीकों की अपनी ताकत और सीमाएं हैं। माइक्रो-विकिरण में बेहतर स्थानिक और लौकिक संकल्प1 है जैसे कि आयनीकरण विकिरण-प्रेरित फोसी (आईआरआईएफ), क्रोमेटिन-इम्यूनोप्रिपिपिटेशन (सीआईपी), या जैव रासायनिक अंशों की इम्यूनोफ्लोरोसेंट इमेजिंग जैसे अधिकांश वैकल्पिक तरीकों की तुलना में। हालांकि, माइक्रो-विकिरण उपरोक्त तकनीकों की मजबूती को लाख देता है जो एक ही समय में बड़ी संख्या में कोशिकाओं का नमूना ले सकता है।
एस चरण में डीएनए मरम्मत की जांच करने के लिए, एक अतुलकृणाक कोशिका संस्कृति आबादी में एस चरण कोशिकाओं को अलग करने में सक्षम होना चाहिए । इसे संबोधित करने के लिए कई प्रसिद्ध तरीके हैं, जिनमें कोशिकाओं का सिंक्रोनाइजेशन, या विभिन्न कोशिका चक्र चरणों का दृश्य शामिल है। हालांकि, दोनों दृष्टिकोण महत्वपूर्ण चुनौतियों और संभावित कलाकृतियों का परिचय देते हैं। प्रारंभिक एस चरण (उदाहरण के लिए, डबल थाइमिडीन ब्लॉक, एफिडिकोलिन और हाइड्रोक्सीयूरिया उपचार) में कोशिकाओं को समृद्ध करने के लिए व्यापक रूप से उपयोग किए जाने वाले रासायनिक सिंक्रोनाइजेशन तरीके प्रतिकृति तनाव के प्रेरण के माध्यम से सिंक्रोनाइजेशन प्राप्त करते हैं और अंततः डीएनए स्वयं को नुकसान पहुंचाते हैं। यह एस चरण2में डीएनए मरम्मत प्रक्रियाओं का अध्ययन करने के लिए इन तरीकों के उपयोग को सीमित करता है । सीरम भुखमरी और रिलीज के माध्यम से सिंक्रोनाइजेशन केवल सीमित संख्या में सेल लाइनों पर लागू होता है, मोटे तौर पर कैंसर सेल लाइनों को छोड़कर जो गैर-परिवर्तित सेल लाइनों की तुलना में सेल-चक्र प्रगति के लिए विकास कारकों पर कम भरोसा करते हैं। फ्लोरेसेंस सर्वास्थितिन सेल चक्र संकेतक (FUCCI) प्रणाली सेल चक्र का अध्ययन करने के लिए एक विशेष रूप से उपयोगी उपकरण है, लेकिन एस औरजी-2सेल-चक्र चरणों 3 के बीच अंतर करते समय इसकी एक मौलिक सीमा होती है।
यहां यह दिखाया गया है कि फ्लोरोसेंटी टैग पीसीएनए को एस चरण के लिए गैर-इनवेसिव मार्कर के रूप में टैग किया गया है, जो एफसीआईसीआई सिस्टम की तुलना में अधिक विशिष्टता और लचीलेपन की अनुमति देते हुए रासायनिक सेल-चक्र सिंक्रोनाइजेशन विधियों की कमियों को सीमित करता है। एक मार्कर के रूप में, न केवल पीसीएनए एक अतुल्यकालिक आबादी में एस-चरण कोशिकाओं को उजागर कर सकता है, बल्कि यह एस चरण (यानी, प्रारंभिक, मध्य, या देर से एस-चरण)4के भीतर कोशिकाओं की सटीक प्रगति भी दिखा सकता है। बहिर्जात, टैग किए गए पीसीएनए का कम अभिव्यक्ति स्तर सेल चक्र प्रगति और डीएनए मरम्मत प्रक्रियाओं दोनों के साथ न्यूनतम हस्तक्षेप सुनिश्चित करता है। महत्वपूर्ण बात यह है कि पीसीएनए उचित डीएनए क्षति प्रेरण के लिए एक आंतरिक नियंत्रण के रूप में भी कार्य करता है क्योंकि यह कई डीएनए घावों की मरम्मत में शामिल है और इसे स्थानीय रूप से प्रेरित डीएनए क्षति स्थलों1,4 में भर्ती कियाजाताहै।
यहां प्रस्तुत किए गए प्रयोगों से यह प्रदर्शित होता है कि एस चरण में EXO1b की भर्ती गतिशीलता को कैसे मापना है और यह अच्छी तरह से स्थापित PARP अवरोधक, ओलापरिब से कैसे प्रभावित होता है। EXO1b नाभिक गतिविधि बेमेल मरम्मत (एमएमआर), न्यूक्लियोटाइड एक्ससेशन रिपेयर (एनईआर), और डबल-फंसे ब्रेक (डीएसबी) मरम्मत सहित डीएनए मरम्मत मार्गों की एक विस्तृत श्रृंखला के लिए प्रासंगिक है। एस चरण में, EXO1b डीएनए रिसेक्शन5के दौरान 3 ' एसएसडीएनए ओवरहैंग के गठन के माध्यम से मुताबिक़ पुनर्संयोजन (एचआर) में एक प्रमुख भूमिका निभाता है । EXO1b को डीएनए प्रतिकृति में और फंसाया गया है, जिसमें चेकपॉइंट एक्टिवेशन में भूमिकाओं के साथ रुकी हुई डीएनए कांटे को फिर से शुरू करने के साथ ही प्राइमर हटाने और ओकाजाकी टुकड़ा परिपक्वता को प्रतिकृति 5 में स्ट्रैंड विस्थापन केदौरानपिछड़ने वाले स्ट्रैंड पर फिर से शुरू किया गया है । क्षतिग्रस्त डीएनए साइटों के लिए EXO1b भर्ती पाली (एडीपी-रिबोस) (PAR)6,7के साथ सीधी बातचीत से विनियमित है । EXO1b के कई सेल-चक्र विशिष्ट प्रभावों के कारण, यह पीसीएनए का उपयोग करके एस-चरण विशिष्ट भर्ती अध्ययनों के लिए एक उत्कृष्ट विकल्प है।
Protocol
1. मानव ऑस्टियोसारकोमा-व्युत्पन्न कोशिकाओं की खेती (U-2 OS)
नोट: U-2 OS कोशिकाएं इन अध्ययनों के लिए आदर्श हैं क्योंकि उनके पास एक फ्लैट आकृति विज्ञान, बड़े नाभिक हैं और कांच सहित कई सतहों से दृढ़ता से जुड़े हुए हैं। इसी तरह की विशेषताओं के साथ अन्य सेल लाइनों का भी उपयोग किया जा सकता है।
- U-2 ओएस सेल लाइनों की खेती के लिए, 10% भ्रूण गोजातीय सीरम (FBS) और एंटीबायोटिक दवाओं (१०० यू/एमएल पेनिसिलिन और १०० μg/mL स्ट्रेप्टोमाइसिन) के साथ पूरक McCoy के 5A माध्यम का उपयोग करें । 5% सीओ2वाले आर्द्र वातावरण में 37 डिग्री सेल्सियस पर इनक्यूबेट कोशिकाएं। माइक्रोस्कोपी अध्ययन के लिए, पर्याप्त सेल काउंट प्रदान करने के लिए 10 सेमी डिश में सेल कल्चर बनाए रखें।
- जब कोशिकाएं 90% कन्फ्लेम (~ 7 x 106 कोशिकाओं/10 सेमी डिश) से संपर्क करती हैं, तो कोशिकाओं को विभाजित करती हैं।
- पीबीएस के साथ कोशिकाओं कुल्ला दूर ट्राइप्सिन अवरोधकों को धोने के लिए सीरम के भीतर निहित ।
- ट्रिपसिन-ईडीटीए के 1 एमसीएल जोड़ें और सुनिश्चित करें कि सेल लेयर समान रूप से कवर किया गया है।
- 37 डिग्री सेल्सियस पर इनक्यूबेट जब तक सेल परत प्लेट से उठा लिया जाता है (लगभग 6 मिनट)।
- ट्राइप्सिन को निष्क्रिय करने के लिए मीडिया युक्त सीरम में ट्राइसिनाइज्ड कोशिकाओं को फिर से रीसुस्ल करें और पूरक विकास माध्यम के 10 एमएल वाले एक नए 10 सेमी प्लेट में वॉल्यूम (~ 0.7 x10 6 कोशिकाओं) का 1/10th जोड़ें।
- प्रयोग से पहले, निर्माता की सिफारिश के बाद यूनिवर्सल माइकोप्लाज्मा डिटेक्शन किट का उपयोग करके माइकोप्लाज्मा संदूषण के लिए नियमित रूप से कोशिकाओं का परीक्षण करें।
2. रेट्रोवायरल संक्रमण
नोट: बीएसएल-2 सुरक्षा उपायों के लिए और पुनर्संयोजन वायरस के साथ काम करते समय, कृपया देखें: एनआईएच दिशानिर्देश, धारा III-D-3: ऊतक संस्कृति में Recombinant वायरस ।
- बीज 4 x 106 HEK293T कोशिकाओं को एक 10 सेमी संस्कृति पकवान में चढ़ाना के बाद 24 घंटे के भीतर ~ 60% योग्यता प्राप्त करने के लिए।
- HEK293T की खेती के लिए कृपया इस प्रोटोकॉल के 1.1-1.3 में वर्णित U-2 OS की खेती के कदमों का पालन करें। HEK293T स्थानापन्न McCoy के लिए डीएमईएम के लिए 5A माध्यम के लिए । वे ऊतक संस्कृति प्लेटों को कमजोर रूप से देते हैं के रूप में हमेशा धीरे से HEK293T कोशिकाओं को धोने के लिए सुनिश्चित करें।
- प्लाज्मिड की वायरल पैकेजिंग के लिए लिपिड-आधारित ट्रांसफैक्शन रिएजेंट का उपयोग करके ट्रांसफेक्ट HEK293T कोशिकाएं।
- रेट्रोवायरल वैक्टर के लिए, वीएसवी-जी (Addgene #8454) के 1.5 माइक्रोग्राम और PUMVC (Addgene #8449) पैकेजिंग वैक्टर के 1.5 माइक्रोन गठबंधन 3 माइक्रोग्राम के साथ ब्याज के जीन (प्यूरोमाइसिन प्रतिरोध के साथ एक रेट्रोवायरल वेक्टर बैकबोन में) ऑप्टी-एमईएम के 250 माइक्रोनिल में माइक्रोसेंट्रफ्यूज ट्यूब में सीरम मीडिया कम हो गया। ऑप्टी-एमईएम/डीएनए मिश्रण (इस मामले में 6 माइक्रोन) में जोड़े गए डीएनए के प्रत्येक माइक्रोन के लिए P3000 रिएजेंट का 1 माइक्रोन जोड़ें और टैप करके धीरे-धीरे मिलाएं। भंवर या पिपेट को ऊपर-नीचे न करें।
- एक अन्य माइक्रोसेंट्रफ्यूज ट्यूब में, ऑप्टी-एमईएम के 250 माइक्रोन के साथ ट्रांसफेक्शन रीएजेंट के 2 माइक्रोन प्रति माइक्रोन डीएनए (इस मामले में 12 माइक्रोन) को मिलाएं।
- दो मिश्रण (500 μL संयुक्त, भंवर नहीं है, केवल कोमल दोहन से मिश्रण) गठबंधन और यह कमरे के तापमान पर 15 मिनट के लिए इनक्यूबेट करते हैं।
- ध्यान से, कोशिकाओं को अलग किए बिना वरीयता प्राप्त HEK293T कोशिकाओं में मिश्रण ड्रॉपवाइज जोड़ें। प्लेटों को धीरे-धीरे चक्कर लगाते हैं।
- वायरल संक्रमण स्थिर कोशिका रेखाओं को उत्पन्न करने के लिए।
- ट्रांसफैक्शन के बाद HEK293T कोशिकाओं 72 घंटे से सुपरनैंट युक्त वायरस को हटा दें। सावधानी से सेल मलबे और अलग कोशिकाओं को हटाने के लिए एक 0.45 माइक्रोन फिल्टर के साथ समाधान फ़िल्टर। वैकल्पिक रूप से, वायरल संक्रमण की सुविधा के लिए वायरल सुपरनेट में 8 μg/एमएल पॉलीब्रेन जोड़ें।
- 10 सेमी डिश (~ 3 x 106 कोशिकाओं) में ~ 50% कन्फ्यूशी पर यू-2 ओएस कोशिकाओं में सुपरनैंट युक्त वायरस जोड़ें। यू-2 ओएस कोशिकाओं को एक दिन पहले बीज ।
- वायरस युक्त सुपरनैंट को हटाने और त्यागने से पहले 6-16 घंटे के लिए संक्रमित करें।
नोट: ब्याज के जीन के लिए अतिव्यक्तता की वांछित राशि प्राप्त करने के लिए, समय की एक निश्चित राशि के लिए वायरल कमजोर पड़ने की एक श्रृंखला इनक्यूबेट । पश्चिमी दाग के साथ प्रत्येक नव स्थापित सेल लाइन में ट्रांसजीन के अभिव्यक्ति के स्तर की जांच करें यह अंतर्जात स्तर की तुलना । - कोशिकाओं को उपयुक्त एंटीबायोटिक दवाओं की उपस्थिति में चयन करने की अनुमति दें (2 μg/mL अंतिम एकाग्रता पर प्यूरोमाइसिन के मामले में 3-4 दिनों के लिए) और एक माइक्रोस्कोप के तहत ब्याज के फ्लोरोसेंट प्रोटीन टैग जीन की अभिव्यक्ति को सत्यापित करें।
- डबल लेबल सेल लाइनों को उत्पन्न करने के लिए इन चरणों को दोहराएं। यहां प्रस्तुत प्रयोगों में mPlum-PCNA एक रेट्रोवायरल वेक्टर (pBABE) EXO1B-AcGFP के साथ संयुक्त से व्यक्त किया गया था, यह भी एक रेट्रोवायरल वेक्टर (pRetroQ-AcGFP1-N1) से व्यक्त की ।
3. माइक्रो विकिरण के लिए कोशिकाओं की तैयारी
- चढ़ाना कोशिकाओं: प्रयोग से पहले 24 घंटे, नंबर 1.5 बोरोसिलिकेट ग्लास बॉटम के साथ चार अच्छी तरह से कक्षीकृत कवरग्लास पर मीडिया के 500 माइक्रोल-1 एमएल (लगभग 70% आंतरसदी के बीच) के बीच एक मात्रा में कुल 8.0 x 104 कोशिकाओं की प्लेट करें जो उच्च-आवर्धन कॉन्मोिकल माइक्रोस्कोपी और लेजर माइक्रो-इमाडियमेशन के लिए आदर्श परिणाम प्रदान करता है। एक उच्च कोशिका की घुलन शक्ति देखने के एक क्षेत्र (FOV) में मापा और अधिक कोशिकाओं के लिए अनुमति देता है; हालांकि पूरी तरह से ढुलमुल स्लाइड सेल चक्र अनियमितताओं का परिचय देंगे।
- इमेजिंग मीडिया: माइक्रो-विकिरण से एक घंटे पहले, फ्लोरोब्रिटे डीएमईएम के लिए नियमित विकास माध्यम का आदान-प्रदान 10% एफबीएस, 100 यू/एमएल पेनिसिलिन और 100 माइक्रोग्राम/एमएल स्ट्रेप्टोमाइसिन, 15 एमएएम एचईपीपी (पीएच = 7.4) और 1 एमएम सोडियम-पायरेट के साथ पूरक है। यह इमेजिंग मीडिया सिग्नल-टू-शोर अनुपात को अधिकतम करने में मदद करता है जिससे बहुत मंद फ्लोरेसेंस का पता लगाया जा सकता है। चूंकि इसमें एचईपीईएस होता है, इसलिए यह 5% सीओ2 वातावरण के अभाव में पीएच को भी स्थिर करता है।
- इस चरण में इमेजिंग से पहले कोई भी अतिरिक्त उपचार लागू करें। यहां प्रस्तुत किए गए प्रयोगों में, कोशिकाओं को या तो ओलापरिब (PARP अवरोधक, 1 μM अंतिम एकाग्रतापर) या एक वाहन नियंत्रण (DMSO) 1,8,9के साथ इमेजिंग से एक घंटे पहले से इलाज किया गया ।
4. माइक्रोस्कोप तैयार करना और इमेजिंग के लिए एस चरण कोशिकाओं का चयन करना।
- एक कॉन्फोकल सिस्टम का उपयोग करें जिसमें समान गुण हैं जैसा कि सिस्टम ने सर्वोत्तम परिणामों के लिए यहां उल्लिखित किया है। यहां प्रस्तुत किए गए प्रयोगों को एक उल्टे माइक्रोस्कोप स्टैंड (सामग्री की तालिकादेखें) पर घुड़सवार एक कॉन्फोकल माइक्रोस्कोप का उपयोग करके किया गया था।
नोट: यहां इस्तेमाल किया गया माइक्रोस्कोप 50 mW 405 एनएम एफआरएपी लेजर मॉड्यूल और 60x 1.4 एनए तेल योजना-एपोक्रोमैट उद्देश्य से सुसज्जित था। कॉन्फोकल स्कैनहेड में दो स्कैनर विकल्प थे: एक गैल्वानो स्कैनर (उच्च रिज़ॉल्यूशन के लिए) और सुनाई देने वाला स्कैनर (हाई-स्पीड इमेजिंग के लिए)।- एक सॉफ्टवेयर नियंत्रित XY galvano डिवाइस के माध्यम से नमूने के लिए फोटोब्लैचिंग (FRAP) लेजर के बाद फ्लोरेसेंस वसूली का परिचय दें। एक्यूआइपी को उत्तेजित करने के लिए 488 एनएम लेजर लाइन और 561 एनएम या 594 एनएम लेजर लाइन का उपयोग करें ताकि mPlum को उत्तेजित किया जा सके।
नोट: निम्नलिखित फ़िल्टर संयोजन इष्टतम परिणाम देता है: 560 एनएम लंबे पास फ़िल्टर का उपयोग करके, 560 एनएम से कम तरंगदैर्ध्य के साथ उत्सर्जन प्रकाश को एसीजीएफपी के लिए 525/50 एनएम उत्सर्जन फिल्टर के माध्यम से पारित किया गया था, जबकि 560 एनएम से अधिक तरंगदैर्ध्य के साथ उत्सर्जन प्रकाश को एमएलपीएमएल के लिए 595/50 एनएम उत्सर्जन फिल्टर के माध्यम से पारित किया गया था। किसी भी उपयुक्त फिल्टर सेट (जैसे, FITC/TRITC, GFP/mCherry, FITC/TxRed) है कि ंयूनतम फ्लोरेसेंस खून के माध्यम से सुनिश्चित करता है इस्तेमाल किया जा सकता है ।
- एक सॉफ्टवेयर नियंत्रित XY galvano डिवाइस के माध्यम से नमूने के लिए फोटोब्लैचिंग (FRAP) लेजर के बाद फ्लोरेसेंस वसूली का परिचय दें। एक्यूआइपी को उत्तेजित करने के लिए 488 एनएम लेजर लाइन और 561 एनएम या 594 एनएम लेजर लाइन का उपयोग करें ताकि mPlum को उत्तेजित किया जा सके।
- पर्यावरण कक्ष और माइक्रोस्कोप घटकों को चालू करें।
- स्थिर छवि अधिग्रहण के लिए थर्मल संतुलन सुनिश्चित करने के लिए प्रयोग की शुरुआत से पहले हीटिंग (चरण, उद्देश्य, और पर्यावरण कक्ष जब संभव हो), सीओ2 आपूर्ति और आर्द्रता नियामक को कम से कम 4 घंटे चालू करें।
- माइक्रोस्कोप में कोशिकाओं के हस्तांतरण से पहले लेजर लाइनों के साथ प्रकाश स्रोतों को शुरू करें।
- फ्लोरोसेंटी टैग पीसीएनए को मार्कर के रूप में टैग करने का उपयोग करके एक अतुल्कालिक आबादी में एस-चरण कोशिकाओं का चयन करें। नीचे दिए गए चरणों का पालन करके ऐसा करें।
- एस चरण में mPlum-टैग पीसीएनए के अद्वितीय स्थानीयकरण पैटर्न के लिए देखो इस सेल चक्र चरण की पहचान संभव बना रही है । पीसीएनए में नाभिक से बाहर रहते हुए कोशिका चक्र के जी 1 और जी2 चरणों में नाभिक में पूरी तरह से सजातीय वितरण होता है । एस-चरण में, पीसीएनए नाभिक में रिपलिसोम के स्थान पर फोसी बनाता है। चित्रा 1 एस-चरण में पीसीएनए फोसी के विभिन्न पैटर्न दिखाता है, जिससे जल्दी, मध्य और देर से एस-चरण में अंतर करना संभव हो जाता है।
- एक FOV का चयन करने के लिए नेत्र के माध्यम से देखें जिसमें माइक्रो-विकिरण के लिए पर्याप्त एस-चरण कोशिकाएं हैं। अतुलकीय यू-2 ओएस कोशिकाओं में आमतौर पर एस चरण में उनकी आबादी का 30-40% होता है।
- इस मामले में EXO1b-AcGFP, जो प्रयोगात्मक कलाकृतियों के लिए नेतृत्व कर सकता है, दोनों PCNA और ब्याज के प्रोटीन (POI) के लिए अभिव्यक्ति के स्तर (उज्ज्वल और मंद कोशिकाओं एक जैसे) में चरम सीमाओं से बचने की कोशिश करो ।
- एक उपयुक्त FOV खोजने के दौरान, फोटोब्लैचिंग और अवांछित डीएनए क्षति को कम करने के लिए लंबे समय तक क्षेत्र को स्कैन करने से बचने की कोशिश करें।
- माइक्रो-विकिरण के लिए ब्याज के वांछित क्षेत्र (आरओआई) निर्धारित करें। संबद्ध सॉफ़्टवेयर (सामग्रियों की तालिकादेखें) का उपयोग करके, पहले बाइनरी लाइनों को डालने के द्वारा वांछित आरओआई सेट करें (लाइनों और रिक्ति की वांछित संख्या निर्धारित करें)। बाइनरीपर क्लिक करें, फिर इन्सॉलेज लाइन | पर क्लिक करें सर्किल | एलिप्से लाइनों की वांछित संख्या को आकर्षित करने के लिए।
- इन बाइनरी लाइनों को आरओआई में परिवर्तित करें और अंत में इन आरओआई को उत्तेजना आरओआई में परिवर्तित करें। ऐसा करने के लिए, पहले आरओआईपर क्लिक करें, फिर बाइनरी को आरओआईपर क्लिक करें, फिर किसी भी आरओआई पर सही क्लिक करें और उत्तेजना आरओआई के रूप में उपयोग का चयन करें: S1। कोशिकाओं के नाभिक से गुजरने के लिए इन रेखाओं को एफओवी में रखें। पूरे FOV को फैलाने वाले 1024 पिक्सल की लंबाई वाले आरओआई का उपयोग पूरे प्रोटोकॉल में किया गया था।
5. इम्यूनोफ्लोरेसेंस धुंधला या समय चूक इमेजिंग के लिए माइक्रो विकिरण।
- इष्टतम माइक्रो-विकिरण सेटिंग्स का निर्धारण करना।
- कोशिकाओं के सूक्ष्म विकिरण से पहले, बाद में विश्लेषण के लिए पीसीएनए फोसी की पहचान करने के लिए एफओवी की एक उच्च रिज़ॉल्यूशन छवि लें। अनुक्रमिक स्कैनिंग के बजाय, दो तरंगदैर्ध्य में स्कैनिंग के बीच सेल आंदोलन से बचने के लिए, एक साथ उपयोग किए गए दोनों ऑप्टिकल चैनल (हरे और लाल) को रिकॉर्ड करें। फोसी के उचित रिज़ॉल्यूशन के लिए 2x औसत के साथ 1x जूम (इमेजिंग सिस्टम पर 0.29 माइक्रोन पिक्सल साइज) के साथ कम से कम 1024 x 1024 पिक्सल/फील्ड रेजोल्यूशन का इस्तेमाल किया गया है। एक बार जब इन मापदंडों A1 LFOV कॉम्पैक्ट जीयूआई और A1 LFOV स्कैन क्षेत्र खिड़कियों में सेट कर रहे हैं, FOV रिकॉर्ड करने के लिए कैप्चर बटन मारा ।
नोट: तुलनीय परिणाम सुनिश्चित करने के लिए पूरे प्रयोगों में एक ही पिक्सेल आकार बनाए रखना महत्वपूर्ण है। - माइक्रो-विकिरण स्थापित करने के लिए, टाइम शेड्यूल (A1 LFOV/Galvano डिवाइस) विंडो का उपयोग करने के लिए इमेजिंग सॉफ्टवेयर में एनडी उत्तेजना टैब खोलें। यह पूर्व-उत्तेजना छवियों की एक श्रृंखला प्राप्त करने के लिए गैल्वानो स्कैनर का उपयोग करता है, उत्तेजित (LUN-F 50 mW 405 एनएम एफआरएपी लेजर का उपयोग करके), और फिर गैल्वानो स्कैनर का उपयोग करके फिर से पोस्ट-उत्तेजना छवियों की एक श्रृंखला प्राप्त करता है। सबसे पहले समय सारणी विंडो में तीन चरण निर्धारित किए। Acq/Stim कॉलम में अधिग्रहण | का चयन करें ब्लीचिंग | क्रमशः तीन चरणों के लिए अधिग्रहण। ब्लीचिंग चरण के लिए, S1 को आरओआई के रूप में सेट करें।
नोट: यहां प्रस्तुत प्रयोग में, उत्तेजना चरण के दौरान कोई छवियां प्राप्त नहीं की गईं । - Galvano XY विंडोमें, माइक्रो-विकिरण के लिए प्रमुख कारकों की स्थापना: 405 एनएम लेजर पावर आउटपुट, निवास समय (पुनरावृत्ति इस प्रणाली पर डिफ़ॉल्ट रूप से 1 है)। यहां प्रस्तुत प्रयोगों में, कोशिकाओं को 405 एनएम एफआरएपी लेजर (फाइबर टिप पर 50 मिलियन) के साथ 100% बिजली उत्पादन पर 1000-3000 माइक्रोन निवास समय के साथ विकिरणित किया गया था।
नोट: क्योंकि लेजर निवास समय प्रति पिक्सेल आधार पर है, जब तक पिक्सेल का आकार समान रहता है, तब तक निवास समय और बिजली घनत्व के बीच संबंध विभिन्न FOVs के बीच तुलनीय होगा। चित्रा 2A विशिष्ट क्षति प्रेरण के लिए लेजर पावर सेटिंग्स को अनुकूलित करने के लिए डीएनए क्षति प्रतिक्रिया (डीडीआर) मार्ग विशिष्ट प्रोटीन (डीएसबी के लिए FBXL10 और ऑक्सीडेटिव बेस क्षति के लिए एनटीएच1) का उपयोग दिखाता है। प्रोटोकॉल की धारा 2 के बाद वायरल संक्रमण के साथ ये स्थिर सेल लाइनें उत्पन्न हुई थीं।
- कोशिकाओं के सूक्ष्म विकिरण से पहले, बाद में विश्लेषण के लिए पीसीएनए फोसी की पहचान करने के लिए एफओवी की एक उच्च रिज़ॉल्यूशन छवि लें। अनुक्रमिक स्कैनिंग के बजाय, दो तरंगदैर्ध्य में स्कैनिंग के बीच सेल आंदोलन से बचने के लिए, एक साथ उपयोग किए गए दोनों ऑप्टिकल चैनल (हरे और लाल) को रिकॉर्ड करें। फोसी के उचित रिज़ॉल्यूशन के लिए 2x औसत के साथ 1x जूम (इमेजिंग सिस्टम पर 0.29 माइक्रोन पिक्सल साइज) के साथ कम से कम 1024 x 1024 पिक्सल/फील्ड रेजोल्यूशन का इस्तेमाल किया गया है। एक बार जब इन मापदंडों A1 LFOV कॉम्पैक्ट जीयूआई और A1 LFOV स्कैन क्षेत्र खिड़कियों में सेट कर रहे हैं, FOV रिकॉर्ड करने के लिए कैप्चर बटन मारा ।
- समय चूक इमेजिंग।
- समय अनुसूची, A1 LFOV कॉम्पैक्ट जीयूआई और A1 LFOV स्कैन क्षेत्र खिड़कियों का उपयोग कर वांछित समय खिड़की और अंतराल के लिए समय चूक इमेजिंग सेट करें। यहां प्रस्तुत प्रयोगों में, EXO1b और PCNA की भर्ती 12 मिनट के लिए छवि थी, FOV हर 5 सेकंड स्कैनिंग, १०२४ x १०२४ पिक्सल/फील्ड पर, 1x ज़ूम का उपयोग कर (इमेजिंग सिस्टम पर ०.२९ माइक्रोन पिक्सेल आकार में जिसके परिणामस्वरूप यहां इस्तेमाल किया) ०.३५ फ्रेम/स्कैनिंग गति (१.४५ μs/पिक्सेल) के साथ फोटो-ब्लीचिंग को कम करने के लिए औसत के बिना ।
- A1 LFOV कॉम्पैक्ट जीयूआई विंडो में इमेजिंग के दौरान फोटो-ब्लीचिंग को कम करने के लिए लेजर पावर %, लाभ और ऑफसेट सेटिंग्स का अनुकूलन करें। यदि किसी का उद्देश्य पीओआई और पीसीएनए दोनों को मापना है, तो दो अलग-अलग फ्लोरोफोरस के लिए क्षेत्र को स्कैन करने के बीच सेल आंदोलन से बचने के लिए अनुक्रमिक स्कैनिंग के बजाय एक साथ स्कैनिंग का उपयोग करें।
- इमेजिंग सिस्टम का उपयोग निम्नलिखित सेटिंग्स के साथ किया गया था। 488 एनएम लेजर लाइन (20 एमडब्ल्यू) के लिए: 7% लेजर पावर, लाभ: 45 (जीएएएसपी डिटेक्टर) के साथ और 2 की ऑफसेट, 561 एनएम लेजर लाइन (20 एमडब्ल्यू) के लिए: 4% लेजर पावर, लाभ 40 (GaAsP डिटेक्टर) के साथ और 2 की ऑफसेट।
- प्रोटीन की गतिज के आधार पर, छवियों या कुल समय चूक की अवधि के बीच अंतराल का विस्तार या छोटा करें। समय अनुसूची विंडो में, तीसरे चरण के अधिग्रहण पंक्ति के लिए वांछित अंतराल और अवधि निर्धारित करें।
- माइक्रो विकिरण और बाद के समय चूक इमेजिंग को निष्पादित करने के लिए अब प्रेस रन करें।
- समय चूक इमेजिंग के अंत में, उत्तेजना आरओआई को अलग छवियों के रूप में बचाएं, जो विश्लेषण के लिए उपयोग किए जाने वाले किसी भी डाउनस्ट्रीम सॉफ्टवेयर में माइक्रो-विकिरण के निर्देशांक की पहचान करने के लिए एक उपयोगी सहायता होगी।
- इम्यूनोफ्लोरेसेंस धुंधला।
नोट: चरण 5.1.3 और चित्रा 2A माइक्रो-विकिरण द्वारा शुरू किए गए डीएनए घावों के प्रकारों का आकलन करने के लिए ज्ञात डीएनए मरम्मत प्रोटीन के उपयोग को दर्शाता है। कोशिकाओं को ठीक करने के बाद विशिष्ट एंटीबॉडी का उपयोग करके कुछ डीएनए घावों का भी पता लगाया जा सकता है। अंतर्जात प्रोटीन का एंटीबॉडी डिटेक्शन करके पीओआई की भर्ती का पता लगाना भी संभव है। डीएसबी की जांच करने के लिए γH2A.X का दृश्य नीचे दिखाया गया है(चित्रा 2B)। चित्रा 3 दोनों अंतर्जात और बहिर्जात टैग पीसीएनए के लिए सेल चक्र भर में PCNA स्थानीयकरण और भर्ती की निरंतरता से पता चलता है ।- चरण 5.1.3 के बाद, mPlum-PCNA की भर्ती के आधार पर उचित एफआरएपी घटना सुनिश्चित करने के लिए माइक्रो-विकिरण के बाद सिर्फ एक छवि लें। इम्यूनोफ्लोरेसेंट लेबलिंग के बाद बाद में क्षेत्र को खोजने के लिए एफओवी के सटीक निर्देशांक पर ध्यान दें।
- 5-10 मिनट के लिए 5% सीओ2 युक्त आर्द्र वातावरण में माइक्रोस्कोप और इनक्यूबेट कोशिकाओं को 37 डिग्री सेल्सियस पर सेल कल्चर चैंबर से बाहर निकालें।
नोट: पैराफॉर्मलडिहाइड (पीएफए) विषाक्त है, और काम एक अच्छी तरह से हवादार क्षेत्र या एक धुएं हुड में किया जाना चाहिए। सभी बाद धोने और इनक्यूबेशन 4 अच्छी तरह से चैंबर स्लाइड में ०.५ एमएल की मात्रा के साथ किया जाएगा । इनक्यूबेशन समय के बाद, पीबीएस (137 mM NaCl, 2.7 mM KCl, 8 m Na 2 HPO 4, और 2 m MKH2पीओ4)के 0.5 एमएल के साथ कोशिकाओं को धोएं और कमरे के तापमान (आरटी) पर 10 मिनट के लिए पीबीएस में 4% पीएफए के 0.5 एमएल के साथ ठीक करें। - कोशिकाओं को एक बार पीबीएस के साथ धोएं, फिर अवशिष्ट पीएफए को बुझाने के लिए उन्हें 50 m M NH4सीएल से धोएं।
- पीबीएस में 0.1% ट्राइटन एक्स-100 के साथ आरटी में 15 मिनट के लिए कोशिकाओं को पार करें।
- ब्लॉकिंग बफर (5% एफबीएस, 3% बीएसए, पीबीएस में 0.05% ट्राइटन एक्स-100) के साथ 1 एच के लिए नमूनों को ब्लॉक करें।
- अवरुद्ध समाधान निकालें और आरटी में 1 घंटे के लिए बफर को अवरुद्ध करने में पतला प्राथमिक एंटीबॉडी (एंटी-γH2A एक्स, 1:2000) जोड़ें।
- कुओं को अवरुद्ध बफर 3 x 10 मिनट के साथ धोएं।
- आरटी में 1 घंटे के लिए बफर को अवरुद्ध करने में पतला माध्यमिक एंटीबॉडी (एंटी-माउस एलेक्सा 488 प्लस संयुग्म, 1:2000) जोड़ें।
- कुओं को अवरुद्ध बफर 3 x 10 मिनट के साथ धोएं।
- 15 मिनट के लिए पीबीएस में 1 μg/mL DAPI समाधान के साथ नाभिक प्रतिदाठ ।
- पीबीएस के साथ एक बार कोशिकाओं को धो लें। इमेजिंग सीधे पीबीएस या एंटीफैड रिएजेंट्स (जैसे, AFR3) के साथ एक पीबीएस समाधान में किया जा सकता है ताकि फोटोब्लैचिंग को कम किया जा सके।
6. भर्ती विश्लेषण
नोट: चित्रा 4A DMSO या olaparib की उपस्थिति में Exo1b और PCNA भर्ती के प्रतिनिधि छवियों से पता चलता है । चित्रा 4B डेटा विश्लेषण के लिए एक प्रतिनिधि छवि दिखाता है। फिजी का उपयोग करके विभिन्न टाइमपॉइंट्स में एमपुलम-पीसीएनए (ए, पीले आयत) द्वारा हाइलाइट किए गए लेजर ट्रैक के साथ एक आयत का उपयोग करके मतलब एकेजीएफपी तीव्रता को मापकर मतलब फ्लोरेसेंस मूल्यों की गणना की गई थी। पीसीएनए आरओआई निर्देशांक के साथ सफल विकिरण को उजागर करने के लिए एक आंतरिक नियंत्रण के रूप में काम कर सकता है। इसी तरह, मतलब AcGFP फ्लोरेसेंस मूल्यों की गणना नाभिक (बी, नीले आयत) के अक्षतिग्रस्त क्षेत्रों के लिए भी की गई थी। पृष्ठभूमि संकेत तीव्रता को अआबादी वाले क्षेत्रों (सी, लाल आयत) में मापा गया था और मतलब फ्लोरोसेंट मूल्यों(चित्रा ए और बी)से घटाया गया था। इस प्रकार, प्रत्येक डेटा संग्रह बिंदु के लिए सापेक्ष मतलब फ्लोरोसेंट इकाई (आरएफयू) की गणना समीकरण आरएफयू = (ए−सी)/(बी−सी)8,9द्वारा की गई थी । माइक्रो-विकिरणित क्षेत्र के परिणामस्वरूप आरएफयू मूल्यों को माइक्रो-विकिरण से पहले आरएफयू मूल्यों के लिए सामान्यीकृत किया जाता है।
- सूक्ष्म विकिरणित साइट के क्षेत्र एक को परिभाषित करने के लिए, नाभिक क्षेत्रों, प्रतिकृति फोसी, और कोशिका के अनियमित परमाणु क्षेत्रों को माप से बाहर करें। फिजी में दो आरओआई को एक के रूप में दो अलग-अलग क्षेत्रों में खींचने के बीच में शिफ्ट कुंजी रखें।
नोट: प्रोटीन भर्ती विभिन्न जीन और विकिरण की स्थिति के बीच भिन्न होंगे; इस प्रकार, क्षेत्र एक के आकार व्यक्तिगत रूप से निर्धारित किया जाना चाहिए । एक बार क्षेत्र एक की पिक्सेल चौड़ाई निर्धारित किया जाता है, यह किसी भी तुलनात्मक भर्तियों के लिए स्थिर रहना चाहिए । यहां प्रस्तुत प्रयोगों में 7 पिक्सेल चौड़ाई आयतों का इस्तेमाल किया गया। - विश्लेषण से रिकॉर्ड किए गए वीडियो की अवधि के दौरान स्थानांतरित होने वाली कोशिकाओं को बाहर करें। अत्यधिक मोबाइल कोशिकाओं को शामिल करने के लिए, वर्णित विश्लेषण फ्रेम-बाय-फ्रेम किया जाना चाहिए।
- भर्ती प्रोफ़ाइल की कल्पना करने के लिए, सांख्यिकीय सॉफ्टवेयर का उपयोग करके समय के खिलाफ सामान्यीकृत आरएफयू मूल्यों को प्लॉट करें।
- मान-व्हिटनी परीक्षण का उपयोग करके डीएमएसओ और ओलापरिब (एन = 31) उपचार के बीच एक संकेतित समय-बिंदु पर अंतर की गणना करें।
Representative Results
कोशिकाएं प्रत्येक प्रकार के डीएनए घाव को एक विशिष्ट तरीके से संबोधित करती हैं जो इस बात पर भी निर्भर करती है कि वे किस कोशिका चक्र चरण में हैं। उदाहरण के लिए, माइक्रो-विकिरण के बाद, डबल-फंसे ब्रेक (डीएसबी) को सेल चक्र चरण के आधार पर गैर-मुताबिक़ अंत में शामिल होने (एनएचईजे) या एचआर द्वारा संसाधित किया जाएगा। कोशिका चक्र के एस और जी-2 चरणों के दौरान सबसे बड़े पैमाने पर काम करने वाले नाभिक डीएनए ओवरहैंग बनाते हैं जो उचित मानव संसाधन के लिए महत्वपूर्ण हैं। एस चरण में कोशिकाओं के मूल्यांकन को बढ़ावा देने के लिए, पीसीएनए को एकल रंग सेल चक्र मार्कर के रूप में नियोजित किया गया था। चित्रा 1A सेल चक्र प्रगति के दौरान mPlum-PCNA के स्थानीयकरण प्रोफ़ाइल से पता चलता है । PCNA जी 1 और जी 2 चरण में नाभिक में एक पूरी तरह से सजातीय वितरण किया है (जबकि भी ज्यादातर नाभिक से बाहर रखा जा रहा है) । एस चरण में, पीसीएनए डीएनए प्रतिकृति की साइटों के लिए स्थानीयकरण करता है, जिसे नाभिक में उज्ज्वल धब्बे के रूप में कल्पना की जा सकती है। प्रारंभिक एस चरण कोशिकाओं में, धब्बे अपेक्षाकृत छोटे होते हैं और कोशिका के नाभिक में समान रूप से वितरित होते हैं। मध्य एस चरण में प्रगति, धब्बे धुंधला हो जाते हैं और नाभिक और नाभिक की परिधि की ओर अधिक स्थानीयकरण करते हैं। देर से एस चरण में, धब्बे संख्या में कम हो जाते हैं, लेकिन तेजी से बड़े हो जाते हैं क्योंकि पीसीएनए देर से प्रतिकृति साइटों(चित्रा 1B)पर केंद्रित होता है। महत्वपूर्ण बात यह है कि pBABE वेक्टर रीढ़ से बहिर्जात पीसीएनए अभिव्यक्ति अंतर्जात स्तर से कम थी लेकिन माइक्रोस्कोपी द्वारा पता लगाने के लिए पर्याप्त थी जो सेल चक्र प्रगति और डीडीआर में संभावित कलाकृतियों को कम करती है। चित्रा 1C अंतर्जात स्तर की तुलना में पीसीएनए ओवरएक्सप्रेसेशन की सीमा को दर्शाता है। कृपया ध्यान दें कि mPlum-PCNA के अनुरूप बैंड अपने बड़े आकार के कारण धीमी गति से प्रवास करता है।
हमने एस चरण में इन घावों के लिए EXO1b की PARP1/2-निर्भर भर्ती की जांच करने के लिए माइक्रो-विकिरण के दौरान DSBs शुरू करने का लक्ष्य है । चित्रा 2A से पता चलता है कि ऊर्जा की कम खुराक (1000 माइक्रोन निवास समय) EGFP-FBXL10 की भर्ती को प्रेरित नहीं करते हैं, एक डीएसबी उत्तरदाता (एफआरयूसीसी कॉम्प्लेक्स 8का घटक), जबकि यह एनटीएचएल 1-एमएचरी की भर्ती को प्रेरित करने के लिए पर्याप्त था, एक आधार एक्सिसेशन रिपेयर (बीईआर) पाथवे प्रोटीन, ऑक्सीडेटिव डीएनए क्षति10, 11,12की साइटों पर भर्ती। 3000 μs पर समय रहता है, दोनों EGFP-FBXL10 और NTHL1-mCherry भर्ती, एक लेजर उत्पादन है कि दोनों ऑक्सीडेटिव घावों और DSBs उत्पन्न करता है प्रदर्शन । इन परिणामों को मजबूत बनाने, चित्रा 2B γH2A.X (DSB मार्कर) के खिलाफ इम्यूनोफ्लोरेसेंस दाग दिखाता है, जो उच्च ऊर्जा खुराक का उपयोग करते समय स्पष्ट रूप से अधिक स्पष्ट है । पीसीएनए एक सेल चक्र मार्कर और सफल माइक्रो-विकिरण के लिए एक मार्कर दोनों के रूप में कार्य करता है, क्योंकि यह पर्याप्त रूप से दोनों लेजर निवास समय सेटिंग्स के साथ भर्ती करता है। महत्वपूर्ण बात यह है कि इस रिपोर्टर फंक्शन के लिए एक्सोजेनस और/या एंडोजेनस फ्लोरोसेंट प्रोटीन टैग किए गए पीसीएनए दोनों का इस्तेमाल किया जा सकता है क्योंकि वे इसी तरह का व्यवहारकरते हैं (चित्रा 3)। एंडोजेनोसली टैग पीसीएनए को पीसीएनए लोकस13 के एक एलील में पहले एक्सोन के साथ फ्रेम में mRuby डालकर इंजीनियर किया गया था (सेल लाइन जोर्ग मैन्सफेल्ड का एक प्रकार का उपहार था)।
चित्रा 4A और चित्रा 4C एस चरण कोशिकाओं में AcGFP टैग EXO1b की भर्ती से पता चलता है । EXO1b 1 मिनट के आसपास माइक्रो-विकिरण साइटों पर संचय के अधिकतम स्तर तक पहुंचता है और फिर धीरे-धीरे बाद में डीएनए घावों से अलग होने लगता है। माइक्रो-विकिरण साइटों पर संवर्धन ग्राफ पर एक > 1 सापेक्ष फ्लोरेसेंस इकाई द्वारा दर्शाया जाता है। ओलापरिब की उपस्थिति में, 1 मिनट पर लेजर धारी पर EXO1b का संचय वाहन नियंत्रण की तुलना में काफी कम है। ये परिणाम साहित्य6,7के साथ सहमत हैं . चित्रा 4B प्रोटोकॉल में बिंदु 6 में वर्णित मात्राकरण (क्षेत्र ए, बी और सी) के लिए प्रतिनिधि क्षेत्रों को दर्शाता है। चित्रा 4D माइक्रो-विकिरण के लिए उपयोग की जाने वाली कोशिकाओं में अंतर्जात EXO1b और बहिर्जात EXO1b-AcGFP के तुलनीय अभिव्यक्ति स्तर को दर्शाता है।
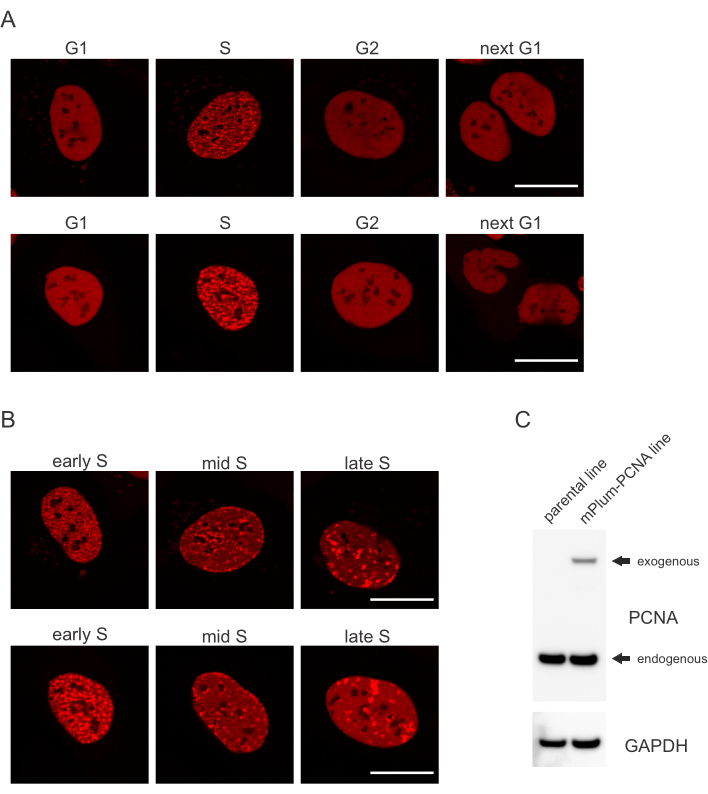
चित्रा 1:पीसीएनए का स्थानीयकरण पैटर्न। (A)छवियां यू-2 ओएस कोशिकाओं में सेल चक्र के दौरान स्थिर एकीकृत, बहिर्जात पीसीएनए के स्थानीयकरण पैटर्न को दिखाती हैं । (ख)छवियां यू-2 ओएस कोशिकाओं में एस चरण (अर्ली, मिड और लेट) के विभिन्न चरणों में पीसीएनए फोसी पैटर्न दिखाती हैं । (ग)इमेजिंग के लिए इस्तेमाल होने वाली यू-2 ओएस कोशिकाओं में पीसीएनए के अंतर्जात और बहिर्जात स्तर को दिखाने वाला पश्चिमी दाग । स्केल बार 20 माइक्रोन का प्रतिनिधित्व करता है । कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।

चित्र 2:अनुकूलित लेजर पावर आउटपुट के माध्यम से डीएसबी को शामिल करना। (ए)लेजर सेटिंग्स को डीएनए क्षति के विभिन्न रूपों को प्रेरित करने के लिए अनुकूलित किया जा सकता है। यू-2 ओएस कोशिकाओं ने क्रमशः डीएसबी और ऑक्सीडेटिव घावों की साइटों की पहचान करने के लिए EGFP-FBXL10 और NTHL1-mCherry दोनों को व्यक्त किया था । 405 एनएम लेजर लाइन के साथ माइक्रो-विकिरण को समकालिक U-2 ओएस कोशिकाओं पर या तो 1000 μs या 3000 माइक्रोन निवास समय के साथ किया गया था। स्केल बार 20 माइक्रोन का प्रतिनिधित्व करता है।(ख)γH2A.X के खिलाफ इम्यूनोफ्लोरेसेंट धुंधला मानव रेटिना वर्णक एपिथेलियल कोशिकाओं (hTERT RPE-1) mRuby टैग एंडोजेनस पीसीएनए होने पर किया गया था । कोशिकाओं को तय किया गया था और या तो 1000 μs या 3000 μs निवास समय के साथ माइक्रो विकिरण के बाद 5 मिनट संसाधित किया गया। स्केल बार 20 माइक्रोन का प्रतिनिधित्व करता है । कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।

चित्रा 3:1000 माइक्रोन या 3000 माइक्रोन लेजर निवास समय पर सूक्ष्म विकिरण स्थलों के लिए अंतर्जात mRuby-PCNA और बहिर्जात mPlum-PCNA की तुलनीय भर्ती। दोनों अंतर्जात और बहिर्जात टैग पीसीएनए फार्म प्रतिकृति foci एस चरण के दौरान । कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।

चित्रा 4:एस चरण में EXO1b की PARP1/2-निर्भर भर्ती । U-2 OS कोशिकाओं को स्थिर रूप से EXO1b-AcGFP और mPlum-PCNA व्यक्त ४०५ एनएम FRAP लेजर लाइन के साथ ३० μs निवास समय का उपयोग कर माइक्रो विकिरणित थे । (A)वाहन नियंत्रण (डीएमएसओ) या ओलापरिब (1 माइक्रोएम) के साथ पूर्व-उपचार के बाद संकेतित समय बिंदुओं पर सूक्ष्म विकिरणित कोशिकाओं की प्रतिनिधि छवियां। स्केल बार भर्ती विश्लेषण के लिए ए,बीऔर सी क्षेत्रों के परिभाषित क्षेत्रों की 20 माइक्रोन का प्रतिनिधित्व करता है। स्केल बार 20 माइक्रोन का प्रतिनिधित्व करता है ।(C)डीएनए क्षति भर्ती गतिशीलता लाइव सेल इमेजिंग द्वारा कब्जा कर लिया गया था । सापेक्ष मतलब फ्लोरेसेंस मूल्यों और छवियों को 12 मिनट के लिए हर 5 एस का अधिग्रहण किया गया था। प्रत्येक स्थिति के लिए, ≥30 कोशिकाओं का मूल्यांकन किया गया । मतलब सापेक्ष फ्लोरेसेंस मान (ठोस काली रेखाएं) और मानक त्रुटि (छायांकित क्षेत्र द्वारा कल्पना की गई सीमा) को समय के खिलाफ प्लॉट किया गया था। धराशायी लाइन माइक्रो-विकिरण के बाद 1 मिनट पर भर्ती मूल्यों को दर्शाती है । डीएमएसओ (एन = 32) और ओलापरिब (एन = 31) उपचार के बीच के अंतर की गणना मान-व्हिटनी परीक्षण का उपयोग करके की गई थी। एस्टरिक्स पी<0.0001 को दर्शाता है। (घ)पश्चिमी दाग सूक्ष्म विकिरण के लिए उपयोग की जाने वाली कोशिकाओं में अंतर्जात EXO1b और बहिर्जात EXO1b-AcGFP के अभिव्यक्ति के स्तर की तुलना करता है । कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
Discussion
महत्वपूर्ण कदम और संभावित प्रोटोकॉल समस्या निवारण/संशोधन
सूक्ष्म विकिरण के लिए उचित ऊतक संस्कृति पोत सफलता के लिए महत्वपूर्ण है। अधिकांश उच्च-रिज़ॉल्यूशन इमेजिंग सिस्टम 0.17 मिमी कवर ग्लास मोटाई के लिए अनुकूलित हैं। उच्च या कम मोटाई इमेजिंग कक्षों या प्लास्टिक पॉलिमर से बने लोगों का उपयोग करना (405 एनएम इमेजिंग के लिए अनुकूलित नहीं), छवि की गुणवत्ता को काफी कम कर सकता है। कांच की सतहों का उपयोग करते समय, सुनिश्चित करें कि वे ऊतक-संस्कृति सेल आसंजन को बढ़ाने के लिए इलाज कर रहे हैं। यदि वे ऊतक-संस्कृति का इलाज नहीं कर रहे हैं, इन कक्षों को लेपित करने की आवश्यकता होगी, उदाहरण के लिए, कोशिकाओं को बोने से पहले पॉली-डी-lysine के साथ । जब कक्षित कवरग्लास में कोशिकाओं चढ़ाना, आदर्श कोशिका घनत्व कोशिका चक्र अनियमितताओं और कोशिकाओं के लिए अतिरिक्त तनाव से बचने के लिए सर्वोपरि है । एक स्थिर तापमान बनाए रखने के लिए प्रयोग से पहले माइक्रोस्कोप घटकों का उचित थर्मल संतुलन समय चूक इमेजिंग भर में ध्यान बनाए रखने के लिए महत्वपूर्ण है और समय और नमूनों में एक सजातीय डीडीआर सुनिश्चित करने के लिए भी आवश्यक है।
यह महत्वपूर्ण है कि कोशिकाओं को एक स्वस्थ हालत में है माइक्रो विकिरण से पहले आर्टिफैक्युअल डेटा को कम करने के लिए । यदि कोशिकाओं में संक्रमण के बाद अनियमित आकृति विज्ञान है/चयन, कोशिकाओं को कई मार्ग के माध्यम से प्रगति करने की अनुमति जब तक आकृति विज्ञान सामांय करने के लिए रिटर्न । हमेशा सुनिश्चित करें कि उपयोग की जाने वाली कोशिकाएं माइकोप्लाज्मा संदूषण से मुक्त हैं। माइकोप्लाज्मा संक्रमण के कई प्रतिकूल प्रभावों में, यह मेजबान कोशिकाओं को डीएनए क्षति भी पहुंचाता है और उनके डीडीआर रास्तों को प्रभावित कर सकता है14,15। सेल संस्कृति में माइकोप्लाज्मा का पता लगाने का सबसे संवेदनशील तरीका पीसीआर (बनाम डीएपीआई या होचस्ट के साथ पता लगाना) के माध्यम से है।
ब्याज की मरम्मत प्रोटीन का इष्टतम अतिव्यवसंन अंतर्जात स्तरों के बराबर होना चाहिए, हालांकि, पता लगाने के लिए पर्याप्त उच्च। वायरल वैक्टर पर उपयोग किए जाने वाले प्रमोटर, संक्रमण के दौरान वायरल टिटर, और संक्रमण के समय की लंबाई सभी को आदर्श अभिव्यक्ति के स्तर के लिए समायोजित किया जा सकता है। लगातार परिणामों के लिए, सजातीय अभिव्यक्ति के स्तर और सामान्य सेल आकृति विज्ञान सुनिश्चित करने के लिए व्यक्तिगत सेल क्लोन को अलग करें। वेक्टर निर्माणों का उपयोग करने की सिफारिश की जाती है जो उचित सेल-चक्र और डीएनए मरम्मत मार्कर फ़ंक्शन के लिए अंतर्जात स्तर से अधिक टैग पीसीएनए को अधिक नहीं करते हैं। यहां तक कि पीसीएनए ओवरएक्सप्रेशन का निम्न स्तर एस-चरण कोशिकाओं को भेदभाव करने के लिए पर्याप्त है। इस उद्देश्य के लिए रेट्रोवायरल पीबीएई वैक्टर का सफलतापूर्वक उपयोग किया गया है (Addgene #1764, #1765, #1766, #1767)। पीसीएनए को किसी भी मोनोमेरिक रेड(जैसे, एमपलम, मचेरी, एमरूबी, आदि) या मोनोमेरिक ग्रीन फ्लोरोसेंट प्रोटीन (जैसे, एमईजीएफपी, एसीजीएफपी, एमवाबी, एमईओएनग्रीन, एमईमेरल्ड आदि) के साथ टैग किया जा सकता है, जिसे तब बारी-बारी से टैग किया जा सकता है। फ्लोरोसेंटली टैग किए गए पीओआई की कुछ सीमाएं और विचार हैं। फ्लोरोसेंट टैग सामान्य प्रोटीन फ़ंक्शन और स्थानीयकरण को बाधित कर सकता है। इस प्रकार, टैग (एन या सी-टर्मिनल) के स्थान पर विचार किया जाना चाहिए। हमेशा मोनोमेरिक फ्लोरोसेंट प्रोटीन का उपयोग करें, क्योंकि गैर-मोनोमेरिक वेरिएंट का ओलिगोमेराइजेशन पीओआई के कार्य को प्रभावित कर सकता है।
लेजर सेटिंग्स प्रत्येक इमेजिंग प्रणाली के लिए निर्धारित किया जाना चाहिए के रूप में ऑप्टिकल पथ के कई घटकों वास्तविक कोशिकाओं में दिया शक्ति को प्रभावित करेगा । लेजर माइक्रो-विकिरण उत्तेजन तरंगदैर्ध्य, एफईआरपी लेजर के बिजली उत्पादन और यदि किसी पूर्व-संवेदीकरण एजेंटों (ब्रोमोडेऑक्सीयूरिडीन या होचस्ट की तरह) का उपयोग किया जाता है, तो कई प्रकार के डीएनए घावों का कारण बन सकता है। 405 एनएम लेजर ऑक्सीडेटिव डीएनए क्षति, एकल और डबल फंसे ब्रेक16,17का कारण बन सकते हैं। उच्च लेजर आउटपुट सेटिंग्स का उपयोग करके, डीएसबी की मात्रा बढ़ जाती है। इस प्रोटोकॉल में पूर्व संवेदीकरण विधियों का उपयोग नहीं किया गया था, लेकिन इन तकनीकों को साहित्य में बहुत शामिल किया गया है और नीचे चर्चा में फिर से छाया हुआ है। हमारी राय में, सबसे अच्छा तरीका है परीक्षण करने के लिए अगर वांछित घाव उत्पन्न होता है ज्ञात डीएनए क्षति मार्ग विशिष्ट जीन की भर्ती के लिए परीक्षण से है । बीईएलआर पाथवे के घटकों एनटीएचएल 1 या ओजीजी 1 की भर्ती से पता चलता है कि ऑक्सीकृत डीएनए बेस10,11,17, 18,19को शामिल किया गया है, जबकि FBXL10 या XRCC5 डीएसबी8,20, 21की उपस्थिति का संकेत देता है। एक्सआरसीसी1 की भर्ती ऑक्सीडाइज्ड डीएनए बेस और सिंगल फंसे ब्रेक (एसएसबी)22, 23की उपस्थिति दोनों को इंगित कर सकती है । एक्सपीसी (यानी, RAD4) एनईआर का एक अच्छा संकेतक है जो पराबैंगनी प्रकाश (यूवी)17,24द्वारा उत्पन्न भारी डीएनए एडक्ट्स को हटा देता है। क्योंकि एक्सोजेनस प्रोटीन की भर्ती से कुछ अनियमितताएं शुरू हो सकती हैं, अंतर्जात डीएनए मरम्मत प्रोटीन या मार्कर (जैसे डबल फंसे ब्रेक के लिए γH2A.X) की इम्यूनोफ्लोरोसेंट धुंधला विशिष्ट डीएनए घावों की उपस्थिति की पुष्टि कर सकता है। वैकल्पिक रूप से, डीएनए घावों के विशिष्ट प्रकार के खिलाफ उठाए गए एंटीबॉडी का भी उपयोग किया जा सकता है। दिया लेजर शक्ति को समायोजित करने के लिए, दोनों निवास समय और लेजर शक्ति बदला जा सकता है।
गणितीय मॉडलिंग की मदद से, एक विस्तृत गतिज विश्लेषण किया जा सकता है जो पीओआई के भर्ती गुणों में मूल्यवान अंतर्दृष्टि प्रदान कर सकता है (उदाहरण के लिए, कई डीएनए बाध्यकारी डोमेन का योगदान, विभिन्न सिग्नलिंग घटनाओं के प्रति संवेदनशीलता, आदि)। 1,25मजबूत वर्कफ्लो बनाने के लिए स्वचालित भर्ती मूल्यांकन और सेल ट्रैकिंग को जोड़ा जा सकता है ।
डीएनए पूर्व संवेदीकरण के फायदे और सीमाएं
माइक्रो-विकिरण से पहले डीएनए का पूर्व-संवेदीकरण डीएनए मरम्मत प्रोटीन भर्ती16, 17के लिए आमतौर पर उपयोग कियाजानेवाला उपकरण है। माइक्रो-विकिरण से पहले डीएनए को संवेदनशील बनाने से यह डीएसबी के लिए अतिसंवेदनशील होता है। डीएनए पूर्व संवेदीकरण के लिए दो सबसे आम तरीके या तो ब्रोमोडेऑक्सीयूडिडीन (BrdU) या Hoechst डाई के साथ कोशिकाओं के पूर्व उपचार कर रहे हैं । उच्च लेजर शक्तियों पर सूक्ष्म विकिरण में सक्षम नहीं प्रणालियों के लिए, इन तरीकों DSBs की तरह डीएनए घावों को प्रेरित करने के लिए आवश्यक हो सकता है । इसके अतिरिक्त, एक संचारित प्रकाश डिटेक्टर या एक फ्लोरोसेंट संकेत के अभाव में सेल नाभिक पर प्रकाश डाला (उदाहरण के लिए, जब अतप्त अंतहीन डीएनए मरम्मत प्रोटीन की भर्ती का अध्ययन), Hoechst दोनों एक पूर्व उपकरण और एक फ्लोरोसेंट दाग के रूप में कार्य करता है हालांकि, डीएनए पूर्व संवेदीकरण महत्वपूर्ण जटिलताओं को पेश कर सकता है। BrdU (10 μM की अंतिम एकाग्रता में इस्तेमाल किया) कोशिकाओं में जोड़ा जाना चाहिए 24 घंटे (या सेल लाइन में एक पूर्ण कोशिका चक्र के बराबर समय) ठीक से डीएनए में शामिल करने के लिए और सेल चक्र हस्तक्षेप26पैदा कर सकता है । Hoechst 33342 (1 μg/mL की अंतिम एकाग्रता में उपयोग किया जाता है) लंबे समय इनक्यूबेशन अवधि के बाद साइटोटॉक्सिक होता है लेकिन डाई के साथ नाभिक को संतृप्त करने के लिए पर्याप्त समय की आवश्यकता होती है। इसलिए, इसे केवल माइक्रो-विकिरण से 15-20 मिनट पहले लागू किया जाना चाहिए; अन्यथा, भर्ती के आंकड़े सुसंगत नहीं होंगे । इस तरह से दागदार कोशिकाओं को27 , 28को कुछ घंटों से अधिक समय तक संस्कृति में नहीं रखा जा सकता . सुनिश्चित करें कि Hoechst 33358 का उपयोग न करें, जो होचस्ट 33342 डाई के रूप में पारम करने योग्य सेल के रूप में नहीं है। पूर्व संवेदीकरण भी प्रयोगों के बीच अनावश्यक विचरण शुरू कर सकते है और प्रयोग और भी अधिक सेल घनत्व में मतभेदों के प्रति संवेदनशील बनाता है (के रूप में यह शामिल डाई/सेल की मात्रा को प्रभावित करेगा) ।
कंफोकल माइक्रोस्कोपी के फायदे और सीमाएं
वाइडफील्ड माइक्रोस्कोपी की तुलना में कॉन्फोकल माइक्रोस्कोपी की इमेजिंग गति सीमित हो सकती है। हालांकि, एक गूंजता स्कैनर से लैस एक कॉन्फोकल माइक्रोस्कोप कताई-डिस्क माइक्रोस्कोपी की गति के करीब आने वाली इमेजिंग गति (संकल्प की कीमत पर) में काफी सुधार कर सकता है। तीन विशेषताएं A1R HD25 कॉन्फोकल सिस्टम को यहां प्रस्तुत प्रोटोकॉल के लिए एक उत्कृष्ट विकल्प बनाती हैं। सबसे पहले, सिस्टम के 25 मिमी एफओवी एक स्कैन किए गए क्षेत्र (नियमित सेटअप में 5-10 कोशिकाओं बनाम) में 15-20 कोशिकाओं के बीच छवि करना संभव बनाता है, सांख्यिकीय विश्लेषण के लिए पर्याप्त कोशिकाओं को प्राप्त करने के लिए आवश्यक अधिग्रहण की संख्या को सीमित करता है। दूसरा, एफईआरपी मॉड्यूल और दो स्कैनहेड कोशिकाओं को एक साथ छवि और माइक्रो-विकिरणित करना संभव बनाते हैं, न कि केवल क्रमिक रूप से। अंत में, दोनों सुनाई देती है और galvano स्कैनर होने का लचीलापन आसानी से असाधारण गति के साथ उच्च लौकिक संकल्प इमेजिंग के बीच स्विच करने की क्षमता प्रदान करता है जो फ्लोरोफोरेस की शमन को कम करता है, और उच्च स्थानिक संकल्प इमेजिंग जो शोर अनुपात के लिए एक उच्च संकेत के साथ छवियों का उत्पादन करने के लिए धीमी स्कैनिंग गति का उपयोग करता है । जबकि उपरोक्त लचीलेपन के लिए उपयोग की गई प्रणाली, अधिक व्यापक रूप से उपलब्ध कॉन्फोकल माइक्रोस्कोप विन्यास के समान होती है, केवल गैलवानो स्कैनर का उपयोग प्रस्तुत प्रयोगों (माइक्रो-विकिरण और बाद में इमेजिंग दोनों के लिए) में किया जाता था।
सूक्ष्म विकिरण के फायदे और सीमाएं
जबकि माइक्रो-विकिरण बेजोड़ स्थानिक और लौकिक संकल्प प्रदान करता है, यह सीमाओं के बिना नहीं है। लेजर माइक्रो विकिरण द्वारा डीएनए क्षति स्वाभाविक रूप से होने वाली हानिकारक एजेंटों की तुलना में नाभिक के विशिष्ट भागों के लिए अत्यधिक संकुल है । इस प्रकार, सूक्ष्म विकिरण के कारण क्रोमेटिन प्रतिक्रिया सजातीय रूप से वितरित क्षति की तुलना में भिन्न हो सकती है। इसके अतिरिक्त, माइक्रो-विकिरण समय लेने वाला है और केवल कुछ दर्जन कोशिकाओं पर आयोजित किया जा सकता है, जबकि बड़ी जनसंख्या आधारित जैव रासायनिक विधियां (क्रोमेटिन आंशिकता, इम्यूनोप्रिपेशन, सीआईपी) एक समय में हजारों कोशिकाओं का अध्ययन करके बढ़ी हुई मजबूती प्रदान कर सकती हैं। पारंपरिक जैव रासायनिक तकनीकों के साथ माइक्रो-विकिरण द्वारा की गई टिप्पणियों की पुष्टि करना विश्वसनीय निष्कर्षों के लिए एक प्रभावी रणनीति है। हालांकि एक निश्चित FOV में कई कोशिकाओं के एक साथ माइक्रो विकिरण संभव है, इमेजिंग प्रणाली को कार्य करने के लिए और अधिक समय की आवश्यकता होगी । इसलिए, प्रोटीन की गतिशीलता को मापने कि डीएनए घावों के लिए बहुत तेजी से भर्ती माइक्रो विकिरण के लिए संभव आरओआई की संख्या एक साथ इस्तेमाल सीमा । इस प्रोटोकॉल के लिए उपयोग की जाने वाली इमेजिंग प्रणाली पर, एकल 1024 पिक्सेल लंबे आरओआई का माइक्रो-विकिरण 1000 माइक्रोन निवास समय का उपयोग करके 1032 एमएस लेता है और 3000 माइक्रोन का उपयोग करके 3088 एमएस पूरा करने के लिए समय रहता है। आरओआई की कई लाइनों का उपयोग करने से माइक्रो-विकिरण को खत्म करने के लिए आवश्यक समय में काफी वृद्धि होगी (उदाहरण के लिए, 7 x 1024 पिक्सेल लंबा आरओआई 14402 एमएस का उपयोग करके 1000 μs निवास समय और 21598 एमएस का उपयोग करके 3000 माइक्रोन निवास समय लेता है)। यह समय छवि अधिग्रहण से खो गया है और इस पर विचार किया जाना चाहिए । जब तेजी से भर्ती की घटनाओं इमेजिंग, सबसे कम आरओआई संभव है और एक समय में केवल माइक्रो विकिरण एक सेल का उपयोग करें ।
सिंक्रोनाइजेशन विधियों पर फायदे और सीमाएं
सेल चक्र विशिष्ट अध्ययनों के लिए, मौजूदा तरीकों में कोशिकाओं को विशिष्ट कोशिका चक्र चरणों में सिंक्रोनाइजेशन या कोशिका के विशिष्ट कोशिका चक्र चरण की पहचान करने के लिए फ्लोरोसेंट रिपोर्टर्स का उपयोग करना शामिल है। हालांकि, इन तरीकों में से प्रत्येक अपनी चुनौतियों और सीमाओं प्रदान करता है ।
FUCCI प्रणाली3 (फ्लोरोसेंट प्रोटीन पर निर्भर सीडीटी 1 और Geminin के कटा हुआ रूपों टैग) सेल चक्र अध्ययन के लिए एक विशेष रूप से उपयोगी उपकरण है, लेकिन सीमाएं है जब यह सेल चक्र के एस और G2 चरणों के बीच अंतर करने के लिए आता है । मिथुन का स्तर पहले से ही मध्य एस चरण से अधिक है और एम चरण तक उच्च रहते हैं, जिससे इन चरणों को अलग करना मुश्किल हो जाता है। FUCCI प्रणाली का उपयोग भी मतलब है कि माइक्रोस्कोप के दो ऑप्टिकल चैनलों POI इमेजिंग के लिए इस्तेमाल नहीं किया जा सकता है ।
गैर कैंसर सेल लाइनों सीरम (सीरम भुखमरी) में पाया विकास कारकों को हटाने के द्वारा G0 में सिंक्रोनाइज्ड किया जा सकता है कोशिकाओं को कम या कोई डीएनए क्षति के कारण । हालांकि, अधिकांश कैंसर सेल लाइनें आंशिक रूप से अपने मीडिया में सीरम की पर्याप्त मात्रा के बिना भी सेल चक्र के माध्यम से प्रगति जारी रखेगी। इसके अतिरिक्त, कोशिकाएं आंशिक रूप से देर से G1, प्रारंभिक एस चरण तक सिंक्रोनाइजेशन खोना शुरू कर देती हैं। सीरम भुखमरी के अलावा, सेल चक्र सिंक्रोनाइजेशन को प्राप्त करने के लिए कई रासायनिक तरीके हैं। हाइड्रोक्सीयूरिया, एफिडिकोलिन और थायरिडीन ब्लॉक कोशिकाओं को प्रारंभिक एस चरण में सिंक्रोनाइज़ करने के लिए डीएनए प्रतिकृति को रोकने के तरीके हैं। जबकि इन तरीकों सस्ते और सरल हैं, वे प्रतिकृति तनाव है जो डीएनए क्षति में परिणाम परिचय । इन डीएनए प्रतिकृति अवरोधकों को H2A के फॉस्फोरिलेशन को प्रेरित करने के लिए दिखाया गया है । एक्स, डीएसबी2, 29का एक प्रसिद्ध मार्कर । एस-चरण कोशिकाओं के लिए एक मार्कर के रूप में टैग-पीसीएनए का उपयोग करने की विधि रासायनिक सिंक्रोनाइजेशन के कारण कलाकृतियों के लिए क्षमता को कम करती है और सीरम भुखमरी की तुलना में सेल लाइनों की एक विस्तृत श्रृंखला के लिए लागू किया जा सकता है ।
समाप्ति
डीएनए क्षति आनुवंशिक रोगों के लिए एक प्रेरक शक्ति है जहां उत्परिवर्तनीय घावों कोशिकाओं के घातक परिवर्तन के लिए नेतृत्व कर सकते हैं । डीएनए संश्लेषण मशीनरी को लक्षित करना कैंसर जैसी हाइपरप्रोलाइफेरेटिव बीमारियों के उपचार में एक मौलिक चिकित्सीय रणनीति है। इन बीमारियों का इलाज ज्यादा लक्षित तरीके से करने के लिए हमें डीएनए घावों की मरम्मत करने वाले प्रोटीन की बेहतर समझ की जरूरत होती है । यहां वर्णित प्रोटोकॉल संभावित कलाकृतियों को कम करने और प्रयोगों की प्रजनन क्षमता बढ़ाने के लिए पारंपरिक सिंक्रोनाइजेशन विधियों द्वारा प्रस्तुत चुनौतियों को कम करके एस चरण में सूक्ष्म विकिरण आधारित अध्ययनों में मदद करता है।
Disclosures
लेखकों का कहना है कि प्रस्तुत कार्य का प्रकाशन निकॉन कॉर्पोरेशन द्वारा प्रायोजित किया गया था। लेखकों की घोषणा है कि कोई प्रतिस्पर्धी हितों मौजूद हैं ।
Acknowledgments
लेखक एम पगानो को उनके निरंतर समर्थन के साथ-साथ डी सिमोनेची, ए मर्जियो और जी तांग को पांडुलिपि की महत्वपूर्ण समीक्षा के लिए धन्यवाद देते हैं । बी मिवातानी-मिंटर ने आर मिवातानी और बी मिंटर को उनके निरंतर समर्थन के लिए धन्यवाद दिया । जी रोना धन्यवाद के रोणे जुराज़ और जी रोना ने अपने निरंतर समर्थन के लिए।
Materials
| Name | Company | Catalog Number | Comments |
| Ammonium chloride | Sigma-Aldrich | A9434-500G | For quenching formaldehyde |
| Anti-EXO1 Rabbit Polyclonal Antibody | Proteintech | 16253-1-AP | primary antibody |
| Anti-phospho-Histone H2A.X (Ser139) Antibody, clone JBW301 | Millipore | 05-636 | primary antibody |
| Bovine Serum Albumin | Sigma-Aldrich | 3117332001 | BSA for blocking |
| BrdU (5-Bromo-2'-deoxyuridine) | Merck | 19-160 | pre-sensitizing agent |
| Citifluor™ Mountant Solution AFR3 | Electron Microscopy Sciences | 17973-10 | antifade containing PBS solution for imaging |
| DAPI | Sigma-Aldrich | D9542-1MG | nucleic acid stain |
| DMEM Medium | Thermo Fisher Scientific | 10569010 | Cell culture medium for HEK293T cells |
| DMSO | Sigma-Aldrich | D2650-100ML | Vehichle control and dissolution solvent |
| EGFP-FBXL10 | Addgene | #126542 | viral expression vector for EGFP-FBXL10 |
| EXO1b-AcGFP (in pRetroQ) | custom cloning | na | EXO1b cDNA was cloned in the NheI, BamHI sites of pRetroQ-AcGFP1-N1 vector. |
| Fetal Bovine Serum | Gibco | 16140071 | Media supplement |
| FluoroBrite DMEM | Thermo Fisher Scientific | A1896701 | Phenol red free medium for microscopy |
| Goat anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 488 | Thermo Fisher Scientific | A32723 | secondary antibody |
| HEK293T cells | ATCC | ATCC CRL-3216 | Cell line for viral packaging |
| HEPES | Sigma-Aldrich | H0887-100ML | Buffering agent to supplement live cell imaging medium |
| Hoechst 33342 | Thermo Fisher Scientific | H3570 | pre-sensitizing agent |
| Lipofectamine 3000 | Thermo Fisher Scientific | L3000015 | Transfection reagent |
| McCoy’s 5A (Modified) Medium | Life Technologies | 16600-108 | Cell culture medium for U-2 OS cells |
| mCherry-PCNA | Addgene | #55117 | non-viral PCNA construct suitable for cell cycle marker |
| mPlum-PCNA | Addgene | #55994 | non-viral PCNA construct suitable for cell cycle marker |
| mPlum-PCNA (in pBABE) | custom cloning | na | mPlum-PCNA cDNA was cloned from Addgene #55994 in the BamHI, SalI sites of pBABE (puro) |
| Nikon A1R-HD25 Confocal Scanhead and Controller | Nikon | na | confocal imaging system |
| Nikon LUN4 laser unit | Nikon | na | excitation system |
| Nikon LUN-F 50 mW 405 nm FRAP laser unit | Nikon | na | FRAP laser unit |
| Nikon NIS Elements Confocal Controller Software | Nikon | na | Confocal controlling software |
| Nikon Ti2-E Inverted Microscope | Nikon | na | inverted epifluorescent microscope base |
| Nikon Ti2-LAPP Modular Illumination System | Nikon | na | illumination system |
| NTHL1-mCherry (in pRetroQ) | custom cloning | na | NTHL1 cDNA was cloned in the NheI, SalI sites of pRetroQ-mCherry-N1 vector. |
| Nunc Lab-Tek II Chambered Coverglass (4 well) | Thermo Fisher Scientific | 155382PK | Live cell microscopy cell culture chamber |
| Olaparib | Selleck Chemicals | S1060 | PARP inhibitor |
| Opti-MEM reduced serum media | Thermo Fisher Scientific | 31985062 | Dilution medium for transient transfection |
| Paraformaldehyde aqueous solution (32%) | Thermo Fisher Scientific | 50-980-494 | Fixative |
| pBABE (hygro) | Addgene | #1765 | retroviral expression vector (for low expression levels) |
| pBABE (neo) | Addgene | #1767 | retroviral expression vector (for low expression levels) |
| pBABE (puro) | Addgene | #1764 | retroviral expression vector (for low expression levels) |
| pBABE (zeo) | Addgene | #1766 | retroviral expression vector (for low expression levels) |
| PCNA Antibody (PC10) | Santa Cruz | sc-56 | primary antibody |
| Penicillin-Streptomycin-Glutamine (100x) | Gibco | 10378016 | Media supplement |
| polybrene | Sigma-Aldrich | TR-1003 | Increase viral infection efficiency |
| pRetroQ-AcGFP-C1 | Takara | 632506 | retroviral expression vector |
| pRetroQ-AcGFP-N1 | Takara | 632505 | retroviral expression vector |
| pRetroQ-mCherry-C1 | Takara | 632567 | retroviral expression vector |
| pRetroQ-mCherry-N1 | Takara | 632568 | retroviral expression vector |
| pUMVC | Addgene | #8449 | Viral packaging vector |
| Sodium-pyruvate | Thermo Fisher Scientific | 11360070 | Supplement for live cell imaging medium |
| Triton X-100 aqueous solution (10%) | Sigma-Aldrich | 11332481001 | Dilute in PBS for cell permeabilization buffer |
| Trypsin-EDTA Solution 10X | Sigma-Aldrich | 59418C-100ML | Dilute in PBS to split cells |
| U-2 OS Cells | ATCC | HTB-96 | Optimal cell line for microscopy experiments |
| Universal Mycoplasma Detection Kit | ATCC | 30-1012K | PCR based Mycoplasma detection kit |
| VSV-G | Addgene | #8454 | Viral protein envelope vector |
References
- Aleksandrov, R., et al. Protein dynamics in complex DNA lesions. Molecular Cell. 69 (6), 1046-1061 (2018).
- Darzynkiewicz, Z., Halicka, H. D., Zhao, H., Podhorecka, M. Cell synchronization by inhibitors of DNA replication induces replication stress and DNA damage response: Analysis by flow cytometry. Methods in Molecular Biology. 761, 85-96 (2011).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Herce, H. D., Rajan, M., Lattig-Tunnemann, G., Fillies, M., Cardoso, M. C. A novel cell permeable DNA replication and repair marker. Nucleus. 5 (6), 590-600 (2014).
- Keijzers, G., et al. Human exonuclease 1 (EXO1) regulatory functions in dna replication with putative roles in cancer. International Journal of Molecular Sciences. 20 (1), (2018).
- Cheruiyot, A., et al. Poly(ADP-ribose)-binding promotes Exo1 damage recruitment and suppresses its nuclease activities. DNA Repair (Amsterdam). 35, 106-115 (2015).
- Zhang, F., Shi, J., Chen, S. H., Bian, C., Yu, X. The PIN domain of EXO1 recognizes poly(ADP-ribose) in DNA damage response. Nucleic Acids Research. 43 (22), 10782-10794 (2015).
- Rona, G., et al. PARP1-dependent recruitment of the FBXL10-RNF68-RNF2 ubiquitin ligase to sites of DNA damage controls H2A.Z loading. elife. 7, (2018).
- Young, L. M., et al. TIMELESS forms a complex with PARP1 distinct from its complex with TIPIN and plays a role in the dna damage response. Cell Reports. 13 (3), 451-459 (2015).
- Kong, X., et al. Laser microirradiation to study in vivo cellular responses to simple and complex dna damage. Journal of Visualized Experiments. (131), e56213 (2018).
- Kong, X., et al. Condensin I recruitment to base damage-enriched DNA lesions is modulated by PARP1. PLoS One. 6 (8), 23548 (2011).
- Lan, L., et al. Novel method for site-specific induction of oxidative DNA damage reveals differences in recruitment of repair proteins to heterochromatin and euchromatin. Nucleic Acids Research. 42 (4), 2330-2345 (2014).
- Zerjatke, T., et al. Quantitative cell cycle analysis based on an endogenous all-in-one reporter for cell tracking and classification. Cell Reports. 19 (9), 1953-1966 (2017).
- Ji, Y., Karbaschi, M., Cooke, M. S. Mycoplasma infection of cultured cells induces oxidative stress and attenuates cellular base excision repair activity. Mutation Research. 845, 403054 (2019).
- Sun, G., et al. Mycoplasma pneumoniae infection induces reactive oxygen species and DNA damage in A549 human lung carcinoma cells. Infection and Immunity. 76 (10), 4405-4413 (2008).
- Gassman, N. R., Wilson, S. H. Micro-irradiation tools to visualize base excision repair and single-strand break repair. DNA Repair (Amsterdam). 31, 52-63 (2015).
- Muster, B., Rapp, A., Cardoso, M. C. Systematic analysis of DNA damage induction and DNA repair pathway activation by continuous wave visible light laser micro-irradiation. AIMS Genetics. 4 (1), 47-68 (2017).
- Ikeda, S., et al. Purification and characterization of human NTH1, a homolog of Escherichia coli endonuclease III. Direct identification of Lys-212 as the active nucleophilic residue. Journal of Biological Chemistry. 273 (34), 21585-21593 (1998).
- Rosenquist, T. A., Zharkov, D. O., Grollman, A. P. Cloning and characterization of a mammalian 8-oxoguanine DNA glycosylase. Proceedings of the National Academy of Science U. S. A. 94 (14), 7429-7434 (1997).
- Reid, D. A., et al. Organization and dynamics of the nonhomologous end-joining machinery during DNA double-strand break repair. Proceedings of the National Academy of Science U. S. A. 112 (20), 2575-2584 (2015).
- Taccioli, G. E., et al. Ku80: product of the XRCC5 gene and its role in DNA repair and V(D)J recombination. Science. 265 (5177), 1442-1445 (1994).
- Marsin, S., et al. Role of XRCC1 in the coordination and stimulation of oxidative DNA damage repair initiated by the DNA glycosylase hOGG1. Journal of Biological Chemistry. 278 (45), 44068-44074 (2003).
- Thompson, L. H., Brookman, K. W., Jones, N. J., Allen, S. A., Carrano, A. V. Molecular cloning of the human XRCC1 gene, which corrects defective DNA strand break repair and sister chromatid exchange. Molecular and Cell Biology. 10 (12), 6160-6171 (1990).
- Scharer, O. D. Nucleotide excision repair in eukaryotes. Cold Spring Harbor Perspective Biology. 5 (10), 012609 (2013).
- Oeck, S., et al. High-throughput evaluation of protein migration and localization after laser micro-irradiation. Science Reports. 9 (1), 3148 (2019).
- Mistrik, M., et al. Cells and stripes: A novel quantitative photo-manipulation technique. Science Reports. 6, 19567 (2016).
- Durand, R. E., Olive, P. L. Cytotoxicity, mutagenicity and dna damage by hoechst 33342. Journal of Histochemistry and Cytochemistry. 30 (2), 111-116 (1982).
- Tobey, R. A., Oishi, N., Crissman, H. A. Cell cycle synchronization: reversible induction of G2 synchrony in cultured rodent and human diploid fibroblasts. Proceedings of the National Academy of Science U. S. A. 87 (13), 5104-5108 (1990).
- Podhorecka, M., Skladanowski, A., Bozko, P. H2AX phosphorylation: Its role in DNA damage response and cancer therapy. Journal of Nucleic Acids. 2010, (2010).



