Summary
תאים חיידקיים במרחב מאוד מאורגנים. כדי לבצע את הארגון הזה לאורך זמן בתאים Myxococcus xanthus גדל לאט, פותחה מלכודת קרינה פלואורסצנטית הדמיה לחיות תאים עם רזולוציה גבוהה ייתכן במשך מספר דורות. באמצעות שיטה זו, ייתכן הדינמיקה של חלבונים חשובים על כרומוזום סגרגציה ועל חלוקת התא יכול להיקבע.
Abstract
קרינה פלואורסצנטית הדמיה לחיות תאים תאים חיידקיים היא שיטה מפתח בניתוח הדינמיקה יכולות של חלבונים ו כרומוזומים שבבסיס אירועים מרכזי מחזור התא. עם זאת, הדמיה של מולקולות אלה במייצג חיידקים הגדלים לאט אתגר בשל photobleaching של fluorophores ו phototoxicity במהלך ייבוא תמונות. כאן, אנו מתארים את פרוטוקול פשוט כדי לעקוף את מגבלות אלה במקרה של Myxococcus xanthus (אשר זמן דור של 4-6 שעות). למטרה זו, מ xanthus התאים גדלים על משטח אגר מזין המכיל עבה סביבה לחים טמפרטורה מבוקרת. בתנאים אלה, אנחנו לקבוע את זמן הכפלה של תאים בודדים על-ידי ביצוע את הצמיחה של תאים בודדים. יתר על כן, סלולרי מפתח תהליכים כגון כרומוזום סגרגציה, חלוקת התא יכול לדימות על-ידי קרינה פלואורסצנטית הדמיה לחיות תאים תאים המכילים חלבונים סמן fluorescently שכותרתו רלוונטיים כגון ParB-YFP FtsZ-GFP, mCherry-PomX מעל מרובות תא מחזורים. לאחר מכן, הדימויים רכשה מעובדים ליצירת montages ו/או סרטים.
Introduction
תאים חיידקיים מאורגנים במרחב מאוד עם הרבה חלבונים לוקליזציה בצורה א-סימטרית בתוך תאים סלולריים1,2,3,4. לוקליזציה הזה הוא לעתים קרובות מאוד דינמי ומשתנה לאורך זמן בתגובה מחזור התא רמזים או אותות חיצוניים. באותה מידה, כרומוזום חיידקי במרחב מאוד מאורגן עם לוקוסים בודדים מיקומו למיקומים ספציפיים subcellular לפני ובמהלך תהליך ההפרדה5. ארגון מרחבי דינמי זה חשוב עבור הצמיחה, חלוקה, ברגולציה מחזור התא, בידול, תנועתיות, אותות וכן כרומוזום הארגון ושל הפרדה; לפיכך, הוא משפיע למעשה בכל ההיבטים של הפונקציה חיידקי.
ייתכן הדינמיקה של תהליכים תאיים אלה נבחנים במגוון מינים חיידקים שונים עם Escherichia coli, Bacillus subtilis, ויבריו choleraeו Caulobacter crescentus המגישה חשוב מודל אורגניזמים. עם זאת, אלו ארבעת המינים לכסות רק קשת קטנה של חיידקים המגוון העצום, אולי באופן לא מפתיע בהתחשב המרחק פילוגנטי גדולים בין המינים הללו, המנגנונים התאיים הארגון, קיטוב הם שונים אלה חיידקים. זה מעלה את הצורך ללמוד מינים חיידקים נוספים תוכל לחלץ בסופו של דבר את עקרונות כלליים שבבסיס ייתכן הדינמיקה של תאים חיידקיים.
גראם שליליים הדלתא-proteobacterium מ xanthus הוא אורגניזם מודל במחקר של התנהגויות חברתיות ושיתוף הפעולה חיידקים6. מ xanthus אובליגטורי קפדנית, בנוכחות חומרים מזינים, טפסים המושבות שבו תאים להפיץ ממכסה ב מתואמת מאוד, שורצים אופנה, הטרף על מיקרואורגניזמים אחרים7. בתגובה לרעב מזין, תאים ליזום תוכנית התפתחותית גורמת להיווצרות של גופים fruiting המורכב אלפי תאים, בתוך אשר, תאי תאי מוט בצורת להבדיל כדורית עד8נבגים דיפלואידי. שני סוגי התנהגויות, קרי, שורצים ו fruiting הגוף והמבנה, מתבצעות רק על ידי תאים המונחים על משטח יציב. יתר על כן, תחת שני תנאים מזין, תאים לעסוק בתהליכים המערבות אנשי הקשר ישיר תאים תאים כולל חילופי lipoproteins הממברנה החיצונית עשויה לעורר את תנועתיות או לתפקד כמו רעלים ב הנמען9,10 , חילופי LPS11, גירוי של תנועתיות מאת exopolysaccharides על השכנה תאים12והמערכת איתות בתא השטח-מעוגן איתות חלבון13,14.
לאחרונה, מ xanthus הפכה גם אורגניזם מודל לומד המנגנונים תנועתיות, את תקנה15, חלוקת התא16,17,18, וארגון כרומוזום19 20, ,21. קריטית נכנס מ xanthus מחזור התא יש נותחו בפירוט על-ידי קרינה פלואורסצנטית מיקרוסקופ באמצעות תמונות ירו snap- או קצר הקלטות בצילום מואץ על זנים נושא רלוונטי חלבונים שכותרתו fluorescently16, 17,18,19,20. באופן אידיאלי, תאים רבים צריך להיות אחריו עם רזולוציה תא בודד על-ידי קרינה פלואורסצנטית תא חי הדמיה עבור מחזור התא מלא אחד לפחות לקבל נתונים כמותיים על מחזור התא בפרמטרים. עם זאת, זהו אתגר במקרה של מ xanthus בשל הזמן שלה הדור ארוכות יחסית של 4-6 h בתנאי מעבדה סטנדרטיים, בשל photobleaching של fluorophores ו phototoxicity במהלך ייבוא תמונות.
כאן, אנו מתארים את פרוטוקול לעקוב מ xanthus תאים עם רזולוציה תא בודד על-ידי קרינה פלואורסצנטית לחיות תאים הדמיה לפחות 24 שעות וכיסוי תא כמה מחזורים. חשוב, במהלך פרוטוקול כולו, תאים נשמרים על רכיב pad אגר, להתקרב קשר המאפשר פעילות תלויי-הקשר חיוני סגנון החיים החברתיים של מ xanthus. הפרוטוקול גם מאפשר למשתמשים צג צורה, גודל, חטיבות של הגששים פלורסנט ברזולוציה הטמפורלית גבוהה ועם רזולוציה תא בודד, לפיכך, מאפשר כימות של השתנות לתא מתאמים אירועי מחזור התא.
Protocol
1. הכנה וצמיחה של מ xanthus זנים
הערה: ראה טבלה 1 ו- 2 בטבלה.
- להכין 1% casitone מרק (CTT) צמיחה בינוני 1% (w/v) הלבלב תקציר של קזאין (למשל, מה נשארתי casitone), 10 מ"מ טריס-HCl pH 8.0, 1 מ מ ח'2PO4 pH 7.6, 8 מ"מ MgSO422, בתוספת kanamycin (50 µg/mL) או oxytetracycline (10 µg/mL). להוסיף gentamycin (10 µg/mL) כל המדיה כדי להפחית את הסיכון של זיהום עם חיידקים אחרים, מאז מ xanthus תאים עמידים באופן טבעי לזה.
- לחסן 5 מ של 1% CTT המכיל הרלוונטי antibiotic(s) עם מושבה בודדת בוגר טרי של הפרוע הקלד DK1622 (WT) 23, SA4420 (ΔmglA)24, SA4797 (ΔmglA, ΔpomX/PpomZ mCherry-pomX )16, SA8241 (ΔmglA, ftsZ+/PnatftsZ-gfp), או SA4749 (ΔmglA, parB+/PnatparB-yfp) ב- בוקר של יום ראשון.
- Resuspend אחד מ xanthus המושבה ב 500 µL של 1% CTT בתוספת אנטיביוטיקה בשפופרת סטרילי ולהעביר המתלה כולו 50 מ ל Erlenmeyer בקבוק המכיל 5 מ של 1% מחסניות.
הערה: השתמש של הבקבוק Erlenmeyer עם 10 פעמים נפח של התרבות כדי להבטיח aeriation מספיק וצמיחה אופטימלית.
- Resuspend אחד מ xanthus המושבה ב 500 µL של 1% CTT בתוספת אנטיביוטיקה בשפופרת סטרילי ולהעביר המתלה כולו 50 מ ל Erlenmeyer בקבוק המכיל 5 מ של 1% מחסניות.
- לגדל את התאים במשך שמונה דורות (כ 40-48 שעות עם זמן דור של 4-6 שעות)-32 מעלות צלזיוס, רועדת-220 סל"ד, בחושך. לשמור על תאים בשלב הגידול המעריכי (OD550 < 1.2) ולמנוע מהם להגיע לשלב נייח. אם יש צורך, לדלל את התאים לתוך טרי בינוני מחסניות של 1%, המכיל את antibiotic(s) הרלוונטי יתר550 של 0.1 - 0.2.
הערה: האופטימלית OD550 עבור מיקרוסקופיה תא בודד הוא 0.5 - 0.7. בשעה זו יתר550, מספר מספיק של התאים קיים לכל תמונה כדי לאפשר כמת, כמו גם ניתוח סטטיסטי של פרמטרים הסלולר.
2. הכנת דוגמאות מיקרוסקופ
הערה: התאים להיות שנצפו על ידי מיקרוסקופ הם מניחים על coverslip מיקרוסקופ, ואז מכוסה על ידי רכיב pad agarose המכיל חומרים מזינים. Coverslip מודבקים פלסטיק או מסגרת מתכת לספק מכני תומכים. כהכנה מיקרוסקופ, כרית גדולה של 1% agarose/TPM/0.2% מחסניות צריך להיות מוכן מראש כמתואר בצעדים 2.1-2.3. בבקשה להתייחס גם הטבלה של חומרים עבור מוצרים ספציפיים המשמש כאן.
- הכנת 500 מ של ה-TPM מאגר (10 מ מ טריס-HCl pH 7.6, 1 מ מ ח'2PO4 pH 7.6, 8 מ"מ MgSO4) ולחטא אוטוקלב או מסנן באמצעות מסנן בראש הבקבוק.
הערה: המאגר סטרילי ניתן לאחסן במשך מספר חודשים בטמפרטורת החדר. - להכין 1%-agarose-מיקרוסקופיה-פתרון המכיל 0.2% CTT (mix 1g של agarose עם 80 מ של מאגר ה-TPM ו- 20 מ של 1% CTT בינוני). מחממים תנור מיקרוגל עד agarose מותך.
הערה: 0.2% CTT מספיקה לאפשר לתאים לגדול ולמנוע רעב. ריכוז גבוה של מחסניות במדיום מיקרוסקופ תגרום רקע זריחה. - למלא צלחת פטרי עם agarose מותכת לעובי 0.5 ס מ (עבור משבצת 11.5 ס"מ x 11.5 ס"מ פטרי, כ- 60 מ של agarose מותכת נדרשת) ולתת לזה להתקרר עד לטמפרטורת החדר.
הערה: משטח agarose ניתן לאחסן ב 4 ° C בסביבת לחים עד 2 ימים.- לחמם את הפנקס מחסניות agarose/TPM/0.2% 1%-32 מעלות צלזיוס למשך לפחות 15 דקות לפני השימוש.
הערה: כדי להכין את התאים במיקרוסקופ, בצע את שלבים 2.4-2.8.
- לחמם את הפנקס מחסניות agarose/TPM/0.2% 1%-32 מעלות צלזיוס למשך לפחות 15 דקות לפני השימוש.
- מקום coverslip של זכוכית סטריליים (60 מ מ x 22 מ מ, עובי: 0.7 מ מ) על מסגרת פלסטיק או מתכת, שיש לו חור באמצע (איור 1א'); מסגרת זו משמש תמיכה מכני עבור coverslip דק והוא מסייע להפחית להיסחף במהלך מיקרוסקופ. לתקן את coverslip על המסגרת עם הקלטת.
- כדי להכין את המסגרת, לגזור מסגרת 75 מ מ × 25 מ מ מ לוחית מתכת בעובי 1 מ מ, ואז לחתוך חור בגודל מתאים (20 מ"מ × 30 מ"מ בניסוי זה) באמצע.
- להוסיף 10-20 µL של גדל באופן אקספוננציאלי מ xanthus תאים coverslip.
- הוסף פלורסנט-0.5 מיקרומטר microspheres כסמני fiducial לתאים כדי לפשט את המעקב של תאים או חלבונים בהקלטות זמן לשגות.
- לדלל את בטחונות microspheres במאגר ה-TPM ולאחסן ב 4 ° C עבור אפילו מספר חודשים. לנער היטב לפני השימוש ולהוסיף 5-10 µL של microspheres מדוללת התאים.
הערה: כאן microspheres אשר נמצאים פלורסנט כל נפוצות כחול, ירוק, צהוב ואדום ערוצי פלורסנט שימשו.
- לדלל את בטחונות microspheres במאגר ה-TPM ולאחסן ב 4 ° C עבור אפילו מספר חודשים. לנער היטב לפני השימוש ולהוסיף 5-10 µL של microspheres מדוללת התאים.
- לגזור כרית קטנה בערך בגודל coverslip של גדולה ומחוממת מראש 1% agarose/TPM/0.2% מחסניות כרית ומניחים אותו על גבי התאים (איור 1B). מניחים את coverslip מעל 1% agarose/TPM/0.2% מחסניות agarose משטח כדי למנוע אידוי וכדי לשמור על תאים בסביבה לחה.
הערה: coverslip לבד ימנע אידוי משמעותי כבר לפחות שעתיים. להקלטות עוד זמן לשגות, 1% agarose/TPM/0.2% מחסניות הכריך pad ו- coverslip צריך להחתם עם פרפין סרט כדי למנוע אידוי. - דגירה הדגימה במיקרוסקופ-32 מעלות צלזיוס למשך 15-20 דקות לשחרר את התאים לצרף התחתון של משטח agarose. אז מתחילים ההקלטות מיקרוסקופ זמן לשגות.
3. מיקרוסקופ הקמה ורכישה בצילום מואץ
הערה: פרוטוקול המתוארים כאן פותחה עבור מיקרוסקופ widefield הפוכה עם פוקוס אוטומטי, 100 X / נה 1.30 שמן PH3 המטרה, X, Y מוטורי הבמה, מצלמה sCMOS, מקור אור, מסננים עבור ירוק-פלורסנט, פלורסנט-אדום או צהוב-פלורסנט חלבונים, תא הדגירה מבוקרת טמפרטורה. החדר הזה שומר על תאי מוגן ומהן אור בטמפרטורה קבועה.
- מראש בחום החדר דגירה, המיקרוסקופ עד 32 ° C עבור h ~ 1-2 לפני הפעלת מיקרוסקופ.
הערה: בהתאם הסידור מיקרוסקופ, חימום עשוי להתארך. חימום מקדים חיוני כדי להפחית את הסחף ומייצב מערכת הבקרה פוקוס אוטומטי. - . הפעילי את המיקרוסקופ ולהתחיל את תוכנת שליטה מיקרוסקופ. בחר את המטרה הנכונה והשלב מראות הנכון ומסננים לרכוש תמונות חדות, כמו גם תמונות של חלבונים ירוק-פלורסנט, פלורסנט-אדום או צהוב-פלורסנט.
הערה: מיקרוסקופ מסופק בדרך כלל עם התוכנה המועדפת עבור רכישת שליטה ואת תמונת מיקרוסקופ. הנה תוכנה זמינים מסחרית (ראה את הטבלה של חומרים) נעשה שימוש כדי לשלוט הרכישה מיקרוסקופ ותמונה. - להוסיף טיפה של שמן טבילה באיכות גבוהה על גבי העדשה של המטרה, לחלק התחתון של המדגם מודגרות מראש ב- 32 מעלות צלזיוס. מקם את המטרה במיקום הנמוך ביותר האפשרי Z-כדי למנוע נזק לעדשה המטרה כאשר המדגם מניחים על הבמה מיקרוסקופ. מקם את המתכת-מסגרת הדגימה לבמה מיקרוסקופ, עם "החור-הצד" לקראת המטרה. הדקו את הדגימה בצורה מאובטחת ב בעל הבמה.
- מתמקדים התאים על ידי הזזת השלב בכיוון Z-קרוב יותר אל המטרה. להעביר את הבמה איטי כאשר השמן טיפות על הצד התחתון מדגם העדשה המטרה להפוך קשר. לעבור את השלב ה-X / Y לכיוון עד מרובות תאים בודדים גלויים באזור של נוף, כאשר התאים נמצאים ב מישור מוקד. ודא שזה ננו-פלורסנט ספירה אחת לפחות נמצא באזור של תצוגה כדי אחר כך ליישר תמונות נרכשות.
הערה: בתנאים אופטימליים, צפיפות התאים של 15-30 תאים לכל אזור של נוף (2,048 x 2,048 פיקסלים או 133.1 x 133.1 מיקרומטר) צריך להגיע. - פתח את אשף Multi-Dimensional רכישה של התוכנה בקרת מיקרוסקופ כדי להגדיר ניסוי מעניין זמן המאפשר המיקרוסקופ לרכוש תמונות במספר אורכי גל ועמדות הבמה במידת הצורך.
- בכרטיסיה ' ראשי ' להפעיל Timelapse ואת מספר אורכי גל. כרטיסיות נוספות יופיע בצד שמאל של החלון.
- לחץ על הכרטיסיה שמירה ועל בחירת ספריה כדי לבחור תיקיה ריקה בכונן הקשיח המחשב כדי לשמור את התמונות רכשה. הפעל תוספת קבועה שם הבסיס, אם קיים קובץ כדי לוודא כי datasets רצופים לא יחליפו הקודמות. אז תן הניסוי שם עם תאריך, שם זן או כותרת של הניסוי.
- לחץ על הכרטיסייה ' Timelapse ' כדי להתאים את הפרמטרים זמן לשגות. הגדרת משך 24 שעות ביממה והגדר מרווח הזמן 20 דקות. מספר של נקודות זמן ישתנו אוטומטית.
הערה: מרווח הזמן האופטימלי תלוי הניסוי ואת הפונקציה תאית וינתח. רכישות התמונה בתדירות גבוהה עלולה לגרום photobleaching. לפיכך, פשרה בין רזולוציה טמפורלית photobleaching מדעית יימצא. בזמן ההכפלה של 4-6 שעות, תמונות ניתן בקלות לרכוש במרווח של 5 דקות (או אפילו קטנה יותר מרווחי במידת הצורך) עבור שלב ניגודיות מיקרוסקופ. אם מיקרוסקופ זריחה מעל הזמן-קורס של 24 שעות רצוי תמונות כדאי שיירשמו במרווח כ- 15-30 דקות. - לחץ על הכרטיסיה אורכי גל בחר מספר אורכי גל לרכוש עבור כל תמונה בכל נקודה בזמן על-ידי שינוי המספר.
הערה: עבור כל אורך גל, בכרטיסיה חדשה מופיע בצד שמאל של Multi-Dimensional רכישה " אשף של אורכי הגל ניתן לרכוש לפי הסדר מלמעלה למטה. עבור כל אורך גל, ההגדרות רכישה יכול להיות שונה בנפרד. - לחץ על הכרטיסיה הגל הראשון מלמעלה. בחר שלב ניגודיות ברשימה הנפתחת תאורה . בחר 100 ms לחשיפה ובחר בכל פעם נקודת ברשימה הנפתחת ידרשו . לבטל אוטומטית לחשוף על-ידי בחירה באפשרות אף פעם ברשימה הנפתחת.
- חזור על צעד 3.5.5 לכל אורך גל שצריך לרכוש בכל נקודה בזמן. עבור חלבונים fluorescently שכותרתו תיאר הקמה ניסיוני, השתמש בפרמטרים הבאים עבור חשיפה: ms 250 mCherry פיוז'ן חלבונים, 200 ms YFP פיוז'ן חלבונים ו- ms 1000 GFP פיוז'ן חלבונים.
הערה: ההגדרות תאורה אופטימלית עבור כל זן של חלבון פלואורסצנטי צריך להיקבע מראש על-ידי שינוי עוצמת המנורה ואת הזמן רכישת תמונות עבור כל אורך גל. תמונה יותר מדי זמן רכישת פעמים להגביר את האפקט פוטוטוקסי, בסופו של דבר להוביל למוות של מעצר התא צמיחה. לכן, צריך להיות מושגת פשרה בין הכדאיות תא ואיכות התמונה. - לרכוש תמונות מזוויות הבמה מרובים כדי להגדיל את מספר התאים טלקוה על אותו ניסוי.
- כדי לרכוש תמונות מתוך מספר מיקומים הבמה, להפעיל את עמדות הבמה מרובות בכרטיסיה ' ראשי '. לאחר מכן לחץ על הכרטיסייה הבמה ולחץ לחצן Live להסתכל על שדה הראייה.
- הזז את הבמה בכיוון X/Y-עד אזור בעל עניין (ROI) נמצא שדה הראייה. שמור X-Y הקואורדינטות ועל ידי לחיצה "+" היחידים הבמה להזיז הבמה שוב בכיוון X/Y-עד רועי חדש ושמור את נקודות הציון שוב על-ידי לחיצה על "+". . קדימה, עד מספר אזורים הרצוי לשמירת
הערה: במקרה של ייבוא תמונות פלורסצנטיות, ודא אזורים מעניינים (ROIs) הם לא יותר מדי אחד לשני כדי למזער את phototoxicity.
- לבדוק פעם נוספת כי התאים הם בפוקוס על-ידי לחיצה על השמורה X - ו Y-עמדות שונות ולהתחיל פוקוס אוטומטי של חומרה על-ידי לחיצה AFC החזק כדי לשמור על המיקום-Z שנשמר קבוע במהלך הניסוי.
- להתחיל ההקלטות בצילום מואץ תוכנת שליטה מיקרוסקופ על ידי לחיצה על רכוש של אשף Multi-Dimensional רכישה .
הערה: חלון אחד יופיע עבור כל אורך גל זה נרכש, חלון נוסף יופיע זה מציג את מספר נקודות זמן רכשה ואת הזמן עד רכישת התמונה הבאה. - בדוק כי התאים נמצאים עדיין בפוקוס לאחר כמה הזמן-נקודות הראשונות בהקלטות בצילום מואץ על מנת למקסם את איכות התמונות למקד מחדש אם יש צורך.
4. דור של יישור תמונות וסרטים בצילום מואץ
הערה: מספר חבילות תוכנה מסחרית ללא תשלום הינם זמינים עבור ייבוא תמונות וניתוח התמונה. אנו משתמשים תוכנה זמינים מסחרית (ראה את הטבלה של חומרים) עם מספר תוספים מותקנים מראש וכלים נוספים.
- להציל את התמונות הבודדות מן הקלטות בצילום מואץ על מחשב בעל התוכנה ניתוח/עיבוד תמונה אינו מותקן.
- להפעיל את התוכנה ולפתוח תמונות כמו ערימה על ידי לחיצה נתונים Multi-Dimensional סקירה | בחר קובץ בסיס | בחר ספריית. פתח את התיקייה עם נתונים רב-ממדיים. לבדוק את ערכת הנתונים, לחץ על תצוגה; ערכת הנתונים יוצגו כפי תמונות יחיד מן הזמן נקודה אחת עד הסוף. להפעיל את אורך הגל (עבור יצירת ערימה), בחר את כל התמונות צריך להיות בערימה ולחץ על עומס כתמונות. חזור על שלב זה עבור כל אורכי הגל ולשמור ערימות שהושלמו.
- (אופציונלי) לפתוח את כל התמונות הדרושות עבור הסרט באמצעות קובץ | פתוח.
הערה: מומלץ לפתוח תמונות מאת גל רכשה אחד בכל פעם כדי לא להאט את המחשב אם חישובית כוח מוגבל. אם צריך להיות תדלג על חלקים מסוימים של ההקלטות זמן לשגות, למשל, ההתחלה, הסיום או במספר נקודות זמן, אז זה יכול להיות מותאם בסרט שהושלמו. - הפעל את ערימת תמונות דורש תיקון עבור סחיפה. פתח את הכלי יישור מאת אפליקציות | Auto Align. בדיקת מחסנית כמקור עבור תמונות המטוס הראשון/זמן הצבע כמו המטוס הפניה. בחר את המחסנית עם לחצן מקור מחסנית ' ולחץ על ' החל'.
הערה: יישור אוטומטי ייקח קצת זמן ואנרגיה חישובית אך היא דרך טובה כדי לתקן ערימות גדולות עבור סחיפה של הסידור מיקרוסקופ. יישור אוטומטי זה עובד טוב אם microspheres נכללים אך יכול גם לעבוד בלעדיהם. - לשמור בה את מחסנית מיושר.
- השתמש ROIs.
הערה: מיקרוסקופ פלואורסצנטי זמן לשגות יוצר בקלות קבוצות גדולות של קבצי נתונים לוקח המון כוח חישובית ולהאט את העיבוד במורד הזרם של הסרטים האלה. לכן מומלץ כדי לזהות ROIs לבודד תאים כדי לעבוד עם קבצים קטנים יותר.- בחרו בכלי האזור המלבני . צור רועי מסביב לתאים של עניין על ידי ידנית ציור רועי על שלב חדות התמונה. ודא כי התאים עניין יהיו גלויים ולא בפוקוס לאורך כל הסרט כל זמן לשגות.
- פתח את הסרט בצילום מואץ של אורך הגל השני של ערכת הנתונים זהה. כדי להעביר את רועי תמונות חדות שלב פלורסצנטיות תמונות של הגל השני השתמש בכלי העברת אזורים עם אזורים | העברת אזורים. בחר את ערכת הנתונים ניגודיות שלב בתמונת המקור , את ערכת הנתונים אורך הגל השני כמו בתמונת היעד. בחר בכל האזורים ' ולחץ על ' אישור'.
- חזור על צעד 4.6.2 לכל אורך גל רכשה עבור ערכת הנתונים זהה.
- בחר את רועי ולשכפל אותו כמו ערימה עם עריכה | שכפול | מחסנית. או הקש על מקשי Shift + Ctrl + D . ואז לשמור בה את מחסנית המשוכפלת עם קובץ | להציל את לתוך באותה תיקיה שבה הנתונים המקוריים.
- חזור על שלב 4.6.4 עבור כל רועי כל הגל רכשה עבור ערכת הנתונים אותו
- כדי להפיק סרט בתבניות MOV או AVI, פתח את הפונקציה Make Movie ויה מחסנית | את הסרט. בחר את הקלטות בצילום מואץ עם לחצן מקור מחסנית . בחר את תבנית הפלט, את קצב המסגרות, מספר המסגרות ולחץ על שמור.
Representative Results
מ xanthus הוא חיידק גדל לאט הנע על-גבי משטחים מוצקים. כדי לבדוק את הגדרת הניסוי שלנו, ביצענו ניסוי בצילום מואץ עם תאי תאי DK1622 WT. תמונות חדות שלב נרכשו במרווחים של 5 דקות במשך 24 שעות ביממה (איור 2א, ב'). רוב התאים מיושר בקבוצות. כצפוי, התאים מוצג תנועתיות ועבר בעיקר בקבוצות. בהמשך הבחנו כי תאים ולעיתים הפוך לכיוון התנועה. ממצאים אלה מראים כי תאים WT בתנאים שנבדקו להתנהג בדרך כלל מבחינת תנועתיות תא. עם זאת, גם כאשר תאים נרשמים כל 5 דקות, הזיהוי של תאים בודדים היא קשה. יתר על כן, מאחר תאים הם תאי, תאים רבים להימלט או הזן את שדה הראיה ומקשה לעקוב אחר תאים לתקופות ממושכות.
כדי לאתר את אותו הדבר מ xanthus תאים עבור מספר מחזורים של מחזור התא על ידי הדמיה לחיות תאים, זנים בודדים ניתן למחוק עבור הגן mglA , אשר חיוני תנועתיות25. פעולה זו מונעת תאים עוזב את שדה הראייה במהלך פרוטוקול הדמיה. במסגרת מחיקות נוצרים כפי שתואר על ידי שי ואח. 26
כצפוי, ב שלב ניגודיות הדמיה לחיות תאים עם Δ שאינם תאי תאיmglA (איור 3), התאים לא יציג תנועה פעיל. הצלחנו לעקוב אחר גדילה וחלוקה של תאים בודדים במהלך היווצרות microcolony. בהתבסס על ההקלטות זמן לשגות בו נרכשו תמונות במרווחי זמן של 5 דקות במשך 24 שעות ביממה, ניתן היה לכמת את הזמן interdivision (הזמן בין שני אירועים חלוקת התא) ברזולוציה של תא בודד. תאים של המוטציהmglA Δ היה מועד חלוקה בין של 235 ± 50 min (n = תאים 97). עם 4h, הפעם interdivision דומה זמן ההכפלה נמדד בתרבויות ההשעיה WT בתאים. זה מספק ראיות זה מ xanthus תאים לגדול באופן מיטבי בתנאים אלה ניסיוני.
כדי לבדוק אם הגדרת שלנו מאפשר לתאים לגדול בדרך כלל תוך מעקב אחר התווית על-ידי YFP חלבונים על פני תקופות ארוכות, ביצענו זריחה זמן לשגות הדמיה עם מ xanthus תאים המבטאים חלבון מתויג YFP. לשם כך, עקבנו ParB-YFP כסמן עבור המקור של שכפול (אורי). ParB הוא רכיב של מערכת ParABS ב מ xanthus ומאגד האתרים parS הפרוקסימלית אורי; לכן, מקור שכפול של כרומוזום ההפרדה ניתן בעקבות19,20,21. עם תמונה רכישה (שלב ניגודיות, זריחה, 200 ms רכישת זמן בערוץ YFP) כל 20 דקות, תאים גדל, לחלק גם להציג צמיחה אפילו ב 24 שעות (איור 4א). בתחילת ההקלטות, ParB-YFP נוצרו שני אשכולות באזורים subpolar ברוב המכריע של תאים (איור 4א). זמן קצר לפני או אחרי חלוקת התא, subpolar של ParB-YFP אשכול בקוטב תא הישן משוכפלים. באחד האשכולות שני נשארו בקוטב תא הישנה תוך העותק השני translocated אל הקוטב תא חדש, שהגיע מעמדה subpolar הסופי לאחר כ 40-60 דקות (איור 4A, B). תצפיות אלה הם מסכים עם הנתונים הקודמים המופקים קצר הקלטות בצילום מואץ באמצעות רפידות אגר דק19. אנו מסיקים כי הגדרת הניסוי הזה מאפשר פלורסצנטיות מיקרוסקופ זמן לשגות למעקב סגרגציה כרומוזום על פני כמה מחזורים תא גדל לאט מ xanthus תאים, ללא perturbing צמיחת תאים או המנגנון סגרגציה כרומוזום.
בניסוי דומה, חיפשנו לעקוב אחר סמנים עבור חלוקת התא על-ידי קרינה פלואורסצנטית זמן לשגות מיקרוסקופ. דומה כמעט כל חיידקים אחרים, מ xanthus דורש FtsZ, GTPase טובולין, כמו חיידקים, חלוקת התא16,17,18. FtsZ יוצר מבנה טבעתי-midcell, מה שנקרא Z-הטבעת, המסייעת לגייס אחרים כל החלבונים הדרושים עבור חלוקת התא27,28. ב מ xanthus, היווצרות של Z-הטבעת והמיקום שלו ב- midcell הוא מגורה על ידי שלוש PomXYZ חלבונים16,17. חלבונים אלה שלושה יוצרים קומפלקס כרומוזום-הקשורים מעבירה ברחבי נוקלאואיד מהאתר של חלוקת התא בתא "אמא" אמצע נוקלאואיד בתאי הבת שני. אמצע נוקלאואיד עולה בקנה אחד עם midcell, לפני כרומוזום סגרגציה, וכאן המגויסים מורכבים PomXYZ FtsZ ומעוררת היווצרות Z-טבעת.
כאן, קודם עקבנו בתאים שאינם תאי לבטא ftsZ-gfp. מכיוון FtsZ-GFP הכוללת מראה אות פלורסצנטיות חלש יותר מאשר ParB-YFP, הגדלנו זמן החשיפה 5 פי 1 s בערוץ ה-GFP. כצפוי, חזקה הצטברות FtsZ-GFP נצפתה רק ב midcell, זו לוקליזציה מוכתב המיקום של חלוקת התא הכיווץ (איור 5א). FtsZ-GFP נוצרו בעיקר אשכול ב midcell תא ארוך יותר. היה זה גם ניכר כי האשכול התחזקו ונשמעו לאורך זמן. לאחר חלוקת התא, הבחנו כי FtsZ-GFP הצטברו מחדש midcell של בתו שני תאים בערך שעתיים מאוחר יותר (איור 5B). . זה תואם את הממצא כי כ- 50% של תאים באוכלוסיה להציג לוקליזציה FtsZ midcell מבוסס על ניתוח ירו snap-16,17.
בניסוי השני, עקבנו תאיםmglA Δ שאינם תאי במשך 24 שעות ביממה לבטא mCherry-pomX. כחלק מערכת PomXYZ, PomX עוזר מדריך Z-טבעת צורה ומיקום, ובכך מגרה חלוקת התא midcell16. האות זריחה של mCherry-PomX היא חזקה, מאפשרת זמן החשיפה של ערוץ זריחה של גב' 250 חשוב, כל התאים גדלה במידה ויוצג אירוע חלוקת התא במהלך הניסוי, ויוצרים microcolonies לאחר 24 שעות ( איור 6A). כפי שדווחה בעבר16, כמעט כל התאים הכיל אשכול mCherry-PomX. רוב אלה מקומי ב midcell ואשכולות מן midcell translocated כדי midcell במהלך הניסוי. במהלך חלוקות תאים, נפרדו אשכולות mCherry-PomX, כאשר כל תא הבת קבלת אשכול. בניגוד FtsZ-GFP, mCherry-PomX מקומי ב midcell 80-90% של מחזור התא, להגיע עמדה זו מיד לאחר חלוקת התא (איור 6B).
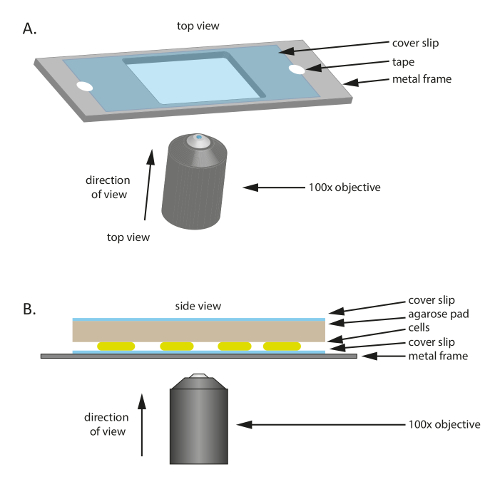
איור 1 : סכימטי של הסידור ניסיוני המשמש לאורך כל המחקר הזה. (א) A ממתכת או פלסטיק משמש מסגרת תמיכה עבור המדגם. Coverslip קבוע למסגרת מתכת עם קלטת כדי להפחית את התנועה של המדגם. (B) צד הצג של קביעת מדגם ניסיוני. התאים הם רכובים על גבי coverslip המוצגים באותיות (A). משטח agarose המספק חומרי הזנה ולחות על התאים מונחת על גבי התאים. משטח agarose מכוסה על ידי coverslip נוספים כדי להקטין התאדות. לתמונות באיכות גבוהה, משמש 100 X שמן טבילה שלב ניגודיות מטרה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 2 : שלב ניגודיות מיקרוסקופ בצילום מואץ של WT מ xanthus תאים. התאים היו במעקב במשך 24 שעות ביממה, תמונות נרכשו כל 5 דק (A) מוצגות תמונות נציג של שדה הראייה זהה בכל 5 דקות. חצים צבעוניים מצביעים על כיוון התנועה של תאים בודדים. באותו הצבע מסמן באותו התא לאורך זמן. המספרים מציינים זמן בדקות. סרגל קנה מידה: 5 מיקרומטר. (B) תמונות של אותו שדה הראייה לאחר כל שעה מוצגים. שימו לב כי שדה הראייה אותו מוצג, אבל כי התאים עוברים, תאים ללא הרף הזנת עוזב את שדה הראיה. המספרים מציינים זמן בשעות. סרגל קנה מידה: 5 מיקרומטר. PH: שלב חדות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 3 : שלב ניגודיות מיקרוסקופ בצילום מואץ של הלא-תאי מ xanthus תאים. ΔmglA תאים עקבו עבור ה 24 תמונות נרכשו בכל 5 דקות ומוצגים להחליפן בתמונות לאחר כל שעה. חלוקת התא הנבחר constrictions מסומנים עם חיצים כתומים. המספרים מציינים זמן בשעות. PH: שלב חדות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
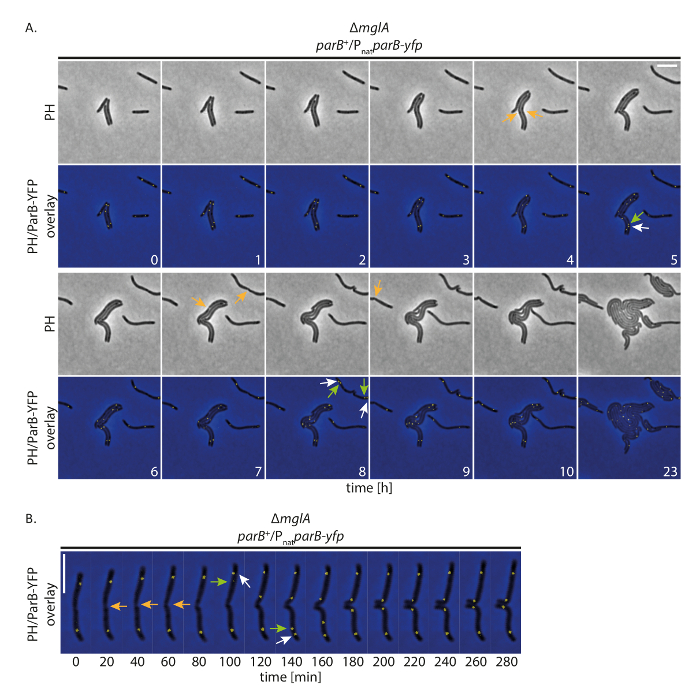
איור 4 : זריחה מיקרוסקופ בצילום מואץ של ParB-YFP בתאי מ xanthus תאים. תאים של ΔmglA מוטציהparB לביטוי - yfp בנוכחות מקורי parB (SA4749; ΔmglA; parB +נת/PparB-yfp) היו במעקב במשך 24 שעות ביממה על ידי מיקרוסקופ ניגודיות וכן קרינה פלואורסצנטית שלב. תמונות (A) נרכשו בכל 20 דקות, נציג תמונות כל שעה עד 10 h מוצגים, יחד עם אותם התאים לאחר ה 24 תמונות מוצגות לעומת שלב (PH) וכפי הכיסוי של שלב ניגוד ו YFP את האות. חלוקות תאים שנבחרו מסומנות עם חיצים כתומים. לבן וירוק חיצים מציינים ParB-YFP אשכול שכפול אירועים, עם החצים הירוקים מסמנים את האשכול translocating. המספרים מציינים זמן בשעות. סרגל קנה מידה: 5 מיקרומטר. (B) תמונות נרכשו כמו (A), אך מוצגים ברזולוציה הטמפורלית גבוה יותר. המספרים מציינים זמן בדקות. . החצים הם כמו (A). סרגל קנה מידה: 5 מיקרומטר. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 5 : זריחה מיקרוסקופ בצילום מואץ של FtsZ-GFP בתאי מ xanthus תאים. תאים של מוטציהmglA Δ לבטא ftsZ-gfp בנוכחות של יליד ftsZ (SA8241; ΔmglA; ftsZ +נת/PftsZ-gfp) היו במעקב במשך 24 שעות ביממה על ידי מיקרוסקופ ניגודיות וכן קרינה פלואורסצנטית שלב. תמונות (A) נרכשו בכל 20 דקות, להחליפן בתמונות כל שעה עד 10 h מוצגים יחד עם אותם התאים לאחר ה 24 תמונות מוצגות שלב ניגודיות (PH), כמו שכבת-על שלב ניגודיות ושידור GFP. חלוקות תאים שנבחרו מסומנות עם חיצים כתומים. חיצים לבנים מציינים FtsZ-GFP אשכולות midcell. המספרים מציינים זמן בשעות. סרגל קנה מידה: 5 מיקרומטר. (B) תמונות נרכשו כמו (A), אך מוצגים ברזולוציה הטמפורלית גבוה יותר. המספרים מציינים זמן בדקות. חצים ירוק ולבן מארק FtsZ-GFP אשכולות בתאים ימינה ושמאלה, בהתאמה. כתום חיצים מציינים חלוקות תאים. סרגל קנה מידה: 5 מיקרומטר. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 6 : זריחה מיקרוסקופ בצילום מואץ של mCherry-PomX, שאינם תאי מ xanthus תאים. Δ Non-תאיpomX תאים צבירת mCherry-PomX (SA4797; ΔmglA; ΔpomX/PpomZ mCherry-pomX) היו במעקב במשך 24 שעות ביממה על ידי מיקרוסקופ ניגודיות וכן קרינה פלואורסצנטית שלב כל 20 דקות (A) נציג תמונות מוצגות כל שעה עד 10 h, יחד עם אותם התאים לאחר ה 24 תמונות מוצגות שלב ניגודיות (PH), כמו שכבת-על שלב ניגודיות, mCherry אות. חלוקות תאים שנבחרו מסומנות עם חיצים כתומים. לבן וירוק חיצים מציינים mCherry-PomX אשכולות לפני ואחרי פיצול אירועים, בהתאמה. המספרים מציינים זמן בשעות. סרגל קנה מידה: 5 מיקרומטר. (B) תמונות נרכשו כמו (A), מוצגים ברזולוציה הטמפורלית גבוה יותר. . החצים הם כמו (A). סרגל קנה מידה: 5 מיקרומטר. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
| זן חיידקי | רלוונטי גנוטיפ1 | הפניה |
| DK1622 | Wildtype | 23 |
| SA4420 | ΔmglA | 24 |
| SA4749 | ΔmglA; parB+/attB:: PnatparB yfp (pAH7) | מחקר זה |
| SA4797 | ΔmglA; ΔpomX / attB::PpomZ mCherry-pomX (pAH53) | 16 |
| SA8241 | ΔmglA; ftsZ+/ mxan18-19::Pנת-ה-gfp-ftsZ (pDS150) | מחקר זה |
| פלסמידים בסוגריים מרובעים מכילים גנים המצוין fusions, היו intergated לכל האתרים המצוינים על הגנום. פלסמידים משולב באתר attB או האזור intergenic mxan18-19 שבאו לידי ביטוי מ יזם מקורי שלהם (Pnat) או האמרגן מקורית של pomZ (PpomZ). |
||
טבלה 1: רשימת זני חיידקים השתמשו במחקר זה.
| פלסמידים | מאפיינים רלוונטיים | הפניה |
| pAH7 | NatPparB-yfp; Mx8 attP; טR | 19 |
| pAH53 | PpomZ mCherry-pomX; Mx8 attP ; קR | 16 |
| pDS150 1 | NatPftsZ-gfp ; mxan18-19 ; טR | מחקר זה |
| pMR3691 | פלסמיד על ביטוי גנים inducible vanillate | 18 |
| pKA51 | NatPftsZ-gfp ; Mx8 attP; טR | 17 |
| 1 pDS150: pDS150 היא נגזרת של pKA51 שבו האתר Mx8 attP הוחלף האזור intergenic mxan18-19 . זה האזור intergenic mxan18-19 היה מוגבר של pMR3691 עם תחל Mxan18-19 fwd BsdRI (GCGATCATTGCGCGCCAGACGATAACAGGC), rev Mxan18-19 BlpI (GCGGCTGAGCCCGCGCCGACAACCGCAACC), משובטים לתוך pKA51. |
||
טבלה 2: רשימת פלסמידים השתמשו במחקר זה.
Discussion
הדמיה לחיות תאים פלורסצנטיות הפך להיות כלי רב עוצמה כדי ללמוד את הדינמיקה ייתכן של תאים חיידקיים. זריחה זמן לשגות מיקרוסקופיה של חיידקים הגדלים לאט ובטוח תאי כגון מ xanthus, עם זאת, כבר מאתגר, בוצעה רק עבור משך זמן קצר. כאן, אנו מציגים שיטה קלה לשימוש וחזק עבור הדמיה לחיות תאים של מ xanthus על-ידי קרינה פלואורסצנטית זמן לשגות מיקרוסקופ. שיטה זו מאפשרת למשתמש לעקוב אחר תאים וחלבונים fluorescently עם תוויות עבור מספר מחזורים של מחזור התא ברזולוציה של תא בודד.
מספר תנאים מוקדמים המשפיעים על ההצלחה של הדמיה לחיות תאים של גידול איטי מ xanthus תאים כולל: 1) משטח יציב עבור התא מצורף; 2) הזמינות של חומרים מזינים וחמצן; 3) קבוע חום ולחות; . ו-4) אופטימיזציה של תנאים ניסיוני כגון תדירות רכישת התמונה וזמן החשיפה.
ב הגדרת הניסוי שלנו, אנו משתמשים agarose עבה רפידות בתוספת חומרים מזינים. באמצעות רפידות עבה agarose לעומת microfluidic התקנים כדי לעקוב אחר תאים בודדים יש כמה יתרונות בסיסיים אבל גם מספר חסרונות. ראשית, משטח agarose לא רק מספק משטח עבור מ xanthus תא המצורף, תנועה אך גם מספיק חומרים מזינים לצמיחה לפחות 24 שעות. שנית, הצמד ירה ניתוח נפוץ ללמוד תאיים לוקליזציה של חלבונים fluorescently שכותרתו נעשה בעבר על אותו סוג של agarose רפידות16,17,29. לכן, הנתונים מניתוחי ירו snap ניתן ישירות להשוות נתונים שהושגו עם השיטה המתוארת כאן. שלישית, רפידות agarose ניתן בקלות לשנות, בתוספת אנטיביוטיקה או תוספי מזון אחרים כגון CuSO4 , vanillate זה משמשות עבור ג'ין ביטוי אינדוקציה18,30. בסופו של דבר, כי תאים מותר טופס microcolonies במהלך ניסוי, זה גם מאפשר ללמוד את השפעת האינטראקציות תאים תאים ישירה על הפרמטר מסוים שעוברים ניתוח. היבט זה חשוב במיוחד במקרה של מ xanthus בגלל חיידק זה מציג מספר האינטראקציות תלויי-הקשר. החיסרון העיקרי של שיטה זו הוא כי התנאים ניסיוני מוגדרות מראש לתקופת ניסוי. לעומת זאת, התקני microfluidic בדרך כלל מאפשרים שינוי התנאים ניסיוני במהלך ניסוי על-ידי הוספת למשל אנטיביוטיקה31.
חבילות תוכנה חופשית (למשל, MicrobeJ, Oufti) זמינים באופן אוטומטי לנתח את הצמיחה של תאים בודדים ולוקליזציה חלבון בתוך תאים בודדים. עם זאת, תוכנות אלה הן רק מתאים היטב הניתוח של תאים בודדים או קבוצות קטנות של תאים. לכן, זה נשאר אתגר לנתח באופן אוטומטי את הנתונים שהופקו עבור הקלטות 24 שעות המתוארים כאן.
לסיכום, אנו המתואר פרוטוקול קל לשימוש, לשחזור כדי לבצע הדמיה לחיות תאים עם איטי הגוברת מ xanthus חיידקים. אנו מראים כי רפידות agarose בתוספת חומרי מזון פשוטים מספיקים לקיים צמיחה לפחות 24 שעות וגם מאפשרים התבוננות וניתוח חלבון לוקליזציה צמיחה ברזולוציה של תא בודד לאורך כמה דורות.
Disclosures
המחברים מצהירים כי יש להם אינטרסים כלכליים אין מתחרים.
Acknowledgments
עבודה זו נתמכה על ידי הגרמנית למחקר המועצה (DFG) במסגרת Transregio 174 "ייתכן הדינמיקה של תאים חיידקיים", חברת מקס פלנק.
Materials
| Name | Company | Catalog Number | Comments |
| DMI6000B with AFC | Leica microsystems | 11888945 | Automated inverted widefield fluorescence microscope with adaptive focus control |
| Universal mounting frame | Leica microsystems | 11532338 | Stage holder for different sample sizes |
| HCX PL FLUOTAR 100x/1.30 oil PH3 | Leica microsystems | 11506197 | Phase contrast objective |
| Orca Flash 4.0 camera | Hamamatsu | 11532952 | 4.0 megapixel sCMOS camera for picture aquisition |
| Filter set TXR ET, k | Leica microsystems | 11504170 | Fluorescence filter set, Ex: 560/40 Em: 645/75 |
| Filter set L5 ET, k | Leica microsystems | 11504166 | Fluorescence filter set, Ex: 480/40 Em: 527/30 |
| Filter set YFP ET, k | Leica microsystems | 11504165 | Fluorescence filter set, Ex: 500/20 Em: 535/30 |
| ProScan III | Prior | H117N1, V31XYZEF, PS3J100 | Microscope automation controller with interactive control center |
| EL 6000 light source | Leica microsystems | 11504115 | External fluorescence light source |
| Incubator BLX Black | Pecon | 11532830 | Black incubation chamber surrounding the microscope |
| Tempcontrol 37-2 digital | Leica microsystems | 11521719 | Automated temperature control for incubation chamber |
| Gentmycin sulphate | Carl Roth | 0233.4 | Gentamycin |
| Oxytetracylin dihydrate | Sigma Aldrich | 201-212-8 | Oxytetracyclin |
| Kanamycin sulphate | Carl Roth | T832.3 | Kanamycin |
| Filtropur BT25 0.2 bottle top filter | Sarstedt | 831,822,101 | Bottle top filter for sterilization of buffers |
| Deckgläser | VWR | 630-1592 | Glass cover slip (60 x 22 mm, thickness: 0.7 mm) |
| Seakem LE agarose | Lonza | 50004 | Agarose for microscopy slides |
| Leica Metamorph AF | Leica microsystems | 11640901 | Microscope control software and software for picture analysis |
| Tetraspeck Microsperes, 0.5 µm | ThermoFisher | T7281 | Fluorescent microspheres |
| petri dish | Greiner Bio-one | 688102 | 120 mm x 120 mm x 17 mm squared petri dish for agarose pads |
| BD Bacto Casitone | Becton Dickinson | 225930 | Casitone |
| Parafilm M | VWR | 291-1213 | Parafilm |
| Tris(hydroxymethyl)-aminomethane | Carl Roth | AE15.2 | Tris |
| Magnesium sulphate heptahydrate | Carl Roth | P027.2 | Magnesium sulphate |
| Potassium dihydrogen phosphate p.a. | Carl Roth | 3904.1 | Potassium dihydrogen phosphate |
| 1% CTT medium: 1 % (w/v) BD Bacto™ casitone, 10 mM Tris-HCl ph 8.0, 1 mM potassium phosphate buffer pH 7.6, 8 mM MgSO4 | Cultivation medium for M.xanthus | ||
| TPM buffer: 10 mM Tris-HCl ph 8.0, 1 mM potassium phosphate buffer pH 7.6, 8 mM MgSO4 | Buffer for preparation of microscopy slides for M.xanthus |
References
- Shapiro, L., McAdams, H. H., Losick, R. Why and how bacteria localize proteins. Science. 326 (5957), 1225-1228 (2009).
- Treuner-Lange, A., Søgaard-Andersen, L. Regulation of cell polarity in bacteria. J Cell Biol. 206 (1), 7-17 (2014).
- Laloux, G., Jacobs-Wagner, C. Spatiotemporal control of PopZ localization through cell cycle-coupled multimerization. J Cell Biol. 201, 827-841 (2013).
- Rudner, D. Z., Losick, R. Protein subcellular localization in bacteria. Cold Spring Harb. Perspect. Biol. 2 (4), 000307 (2010).
- Badrinarayanan, A., Le, T. B. K., Laub, M. T. Bacterial chromosome organization and segregation. Annu Rev Cell Dev Biol. 31 (1), 171-199 (2015).
- Munoz-Dorado, J., Marcos-Torres, F. J., Garcia-Bravo, E., Moraleda-Munoz, A., Perez, J. Myxobacteria: Moving, Killing, Feeding, and Surviving Together. Front Microbiol. 7, 781 (2016).
- Berleman, J. E., Kirby, J. R. Deciphering the hunting strategy of a bacterial wolfpack. FEMS Microbiol Rev. 33 (5), 942-957 (2009).
- Konovalova, A., Petters, T., Søgaard-Andersen, L. Extracellular biology of Myxococcus xanthus. FEMS Microbiol. Rev. 34, 89-106 (2010).
- Nudleman, E., Wall, D., Kaiser, D. Cell-to-cell transfer of bacterial outer membrane lipoproteins. Science. 309, 125-127 (2005).
- Vassallo, C. N., et al. Infectious polymorphic toxins delivered by outer membrane exchange discriminate kin in myxobacteria. eLife. 6, 29397 (2017).
- Vassallo, C., et al. Cell rejuvenation and social behaviors promoted by LPS exchange in myxobacteria. Proc Natl Acad Sci USA. 112 (22), 2939-2946 (2015).
- Li, Y., et al. Extracellular polysaccharides mediate pilus retraction during social motility of Myxococcus xanthus. Proc. Natl. Acad. Sci. USA. 100, 5443-5448 (2003).
- Kim, S. K., Kaiser, D. Cell alignment required in differentiation of Myxococcus xanthus. Science. 249, 926-928 (1990).
- Lobedanz, S., Søgaard-Andersen, L. Identification of the C-signal, a contact dependent morphogen coordinating multiple developmental responses in Myxococcus xanthus. Genes Dev. 17, 2151-2161 (2003).
- Schumacher, D., Søgaard-Andersen, L. Regulation of cell polarity in motility and cell division in Myxococcus xanthus. Annu Rev Microbiol. 71 (1), 61-78 (2017).
- Schumacher, D., et al. The PomXYZ proteins self-organize on the bacterial nucleoid to stimulate cell division. Dev Cell. 41 (3), 299-314 (2017).
- Treuner-Lange, A., et al. PomZ, a ParA-like protein, regulates Z-ring formation and cell division in Myxococcus xanthus. Mol Microbiol. 87 (2), 235-253 (2013).
- Iniesta, A. A., Garcia-Heras, F., Abellon-Ruiz, J., Gallego-Garcia, A., Elias-Arnanz, M. Two systems for conditional gene expression in Myxococcus xanthus inducible by isopropyl-beta-D-thiogalactopyranoside or vanillate. J Bacteriol. 194 (21), 5875-5885 (2012).
- Harms, A., Treuner-Lange, A., Schumacher, D., Søgaard-Andersen, L. Tracking of chromosome and replisome dynamics in Myxococcus xanthus. reveals a novel chromosome arrangement. PLoS Genet. 9 (9), 1003802 (2013).
- Iniesta, A. A. ParABS system in chromosome partitioning in the bacterium Myxococcus xanthus. PLoS One. 9 (1), 86897 (2014).
- Lin, L., Osorio Valeriano, M., Harms, A., Søgaard-Andersen, L., Thanbichler, M. Bactofilin-mediated organization of the ParABS chromosome segregation system in Myxococcus xanthus. Nat Commun. 8 (1), 1817 (2017).
- Hodgkin, J., Kaiser, D. Cell-to-cell stimulation of movement in nonmotile mutants of Myxococcus. Proc Natl Acad Sci U S A. 74 (7), 2938-2942 (1977).
- Kaiser, D. Social gliding is correlated with the presence of pili in Myxococcus xanthus. Proc Natl Acad Sci USA. 76 (11), 5952-5956 (1979).
- Miertzschke, M., et al. Structural analysis of the Ras-like G protein MglA and its cognate GAP MglB and implications for bacterial polarity. EMBO J. 30 (20), 4185-4197 (2011).
- Hodgkin, J., Kaiser, D. Genetics of gliding motility in Myxococcus xanthus. (Myxobacterales): Two gene systems control movement. Mol Gen Genet. 171, 177-191 (1979).
- Shi, X., et al. Bioinformatics and experimental analysis of proteins of two-component systems in Myxococcus xanthus. J Bacteriol. 190 (2), 613-624 (2008).
- Bi, E. F., Lutkenhaus, J. FtsZ ring structure associated with division in Escherichia coli. Nature. 354 (6349), 161-164 (1991).
- Lutkenhaus, J., Pichoff, S., Du, S. Bacterial cytokinesis: From Z ring to divisome. Cytoskeleton. 69 (10), 778-790 (2012).
- McLoon, A. L., et al. MglC, a Paralog of Myxococcus xanthus GTPase-Activating Protein MglB, Plays a Divergent Role in Motility Regulation. J Bacteriol. 198 (3), 510-520 (2015).
- Gomez-Santos, N., et al. Comprehensive set of integrative plasmid vectors for copper-inducible gene expression in Myxococcus xanthus. Appl Environ Microbiol. 78 (8), 2515-2521 (2012).
- Treuner-Lange, A., et al. The small G-protein MglA connects to the MreB actin cytoskeleton at bacterial focal adhesions. J Cell Biol. 210 (2), 243-256 (2015).



