Summary
बैक्टीरियल कोशिकाओं को स्थानिक उच्च संगठित कर रहे हैं । धीमी गति से बढ़ती Myxococcus xanthus कोशिकाओं में समय के साथ इस संगठन का पालन करने के लिए, कई पीढ़ियों से उच्च spatiotemporal संकल्प के साथ प्रतिदीप्ति लाइव सेल इमेजिंग के लिए एक सेट अप विकसित किया गया था. इस विधि का उपयोग करना, गुणसूत्र अलगाव और कोशिका विभाजन के लिए महत्वपूर्ण प्रोटीन की spatiotemporal गतिशीलता निर्धारित किया जा सकता है ।
Abstract
प्रतिदीप्ति लाइव-कोशिकाएं बैक्टीरियल कोशिकाओं के इमेजिंग प्रोटीन और केंद्रीय कोशिका चक्र की घटनाओं अंतर्निहित गुणसूत्रों के स्थानिक और लौकिक गतिशीलता के विश्लेषण में एक महत्वपूर्ण तरीका है । हालांकि, धीमी गति से बढ़ते बैक्टीरिया में इन अणुओं की इमेजिंग छवि अधिग्रहण के दौरान fluorophores और phototoxicity के photobleaching के कारण एक चुनौती का प्रतिनिधित्व करता है । यहां, हम एक सरल प्रोटोकॉल का वर्णन करने के लिए Myxococcus xanthus के मामले में इन सीमाओं को दरकिनार (जो 4-6 ज की पीढ़ी का समय है) । यह अंत करने के लिए, M. xanthus कोशिकाओं को तापमान नियंत्रित आर्द्र वातावरण में एक मोटी पोषक तत्व युक्त आगर पैड पर उगाया जाता है । इन शर्तों के तहत, हम एकल कक्षों की वृद्धि का अनुसरण करके व्यक्तिगत कक्षों के दोहरीकरण का समय निर्धारित करते हैं. इसके अलावा, इस तरह के गुणसूत्र अलगाव और कोशिका विभाजन के रूप में प्रमुख सेलुलर प्रक्रियाओं प्रतिदीप्ति जी द्वारा imaged किया जा सकता है, प्रासंगिक फ्लोरोसेंट जैसे ParB-YFP, FtsZ-GFP, और mCherry-PomX पर कई के रूप में मार्कर प्रोटीन लेबल युक्त कोशिकाओं के सेल इमेजिंग सेल चक्र । इसके बाद, अधिग्रहीत छवियों montages और/या फिल्मों उत्पन्न करने के लिए संसाधित कर रहे हैं ।
Introduction
बैक्टीरियल कोशिकाओं को विशेष रूप से अत्यधिक सेलुलर डिब्बों1,2,3,4के भीतर विषम स्थानीयकृत कई प्रोटीन के साथ आयोजित कर रहे हैं । यह स्थानीयकरण अक्सर अत्यधिक गतिशील और सेल चक्र cues या बाहरी संकेतों के जवाब में समय के साथ बदलता है । समान रूप से, बैक्टीरियल गुणसूत्र विशेष रूप से अत्यधिक व्यक्तिगत loci के साथ आयोजित किया जा रहा है पहले और अलगाव की प्रक्रिया के दौरान5विशिष्ट सेलुलर स्थानों के लिए तैनात । इस गतिशील स्थानिक संगठन विकास, विभाजन, सेल चक्र विनियमन, भेदभाव, गतिशीलता, संकेत transduction के रूप में के रूप में अच्छी तरह से गुणसूत्र संगठन और अलगाव के लिए महत्वपूर्ण है; इस प्रकार, यह जीवाणु समारोह के अनिवार्य रूप से सभी पहलुओं को प्रभावित करता है ।
इन सेलुलर प्रक्रियाओं के spatiotemporal गतिशीलता ई कोलाई, बैसिलस सबटिलिस, Vibrio हैजा, और Caulobacter crescentus के रूप में महत्वपूर्ण सेवारत के साथ विभिन्न जीवाणु प्रजातियों की एक किस्म में विश्लेषण किया जा रहा है मॉडल जीवों । हालांकि, इन चार प्रजातियों में भारी बैक्टीरियल विविधता के केवल एक छोटे स्पेक्ट्रम को कवर और, शायद आश्चर्य की बात इन प्रजातियों के बीच बड़े वंशावली दूरी दिया, सेलुलर संगठन और ध्रुवीकरण तंत्र इन में अलग हैं बैक्टीरिया. यह अतिरिक्त बैक्टीरियल प्रजातियों के अध्ययन के लिए अंततः सामांय बैक्टीरियल कोशिकाओं के spatiotemporal गतिशीलता अंतर्निहित सिद्धांतों को निकालने में सक्षम होने के लिए की जरूरत उठाती है ।
चना-निगेटिव डेल्टा-proteobacterium एम. xanthus 6बैक्टीरिया में सामाजिक व्यवहार और सहयोग के अध्ययन में एक मॉडल जीव है । M. xanthus एक सख्त aerobe है और पोषक तत्वों की उपस्थिति में, यह कालोनियों जिसमें कोशिकाओं को एक उच्च समंवित, बजबजा फैशन और अंय सूक्ष्मजीवों7पर शिकार में बाहर फैल रूपों । पोषक तत्वों भुखमरी के जवाब में, कोशिकाओं के एक विकास कार्यक्रम है कि फल निकायों के गठन में परिणाम है कि कोशिकाओं के हजारों के होते हैं, और जिसके अंदर, रॉड के आकार वाले गतिशील कोशिकाओं गोलाकार द्विगुणित बीजाणुओं को अलग करने के लिए शुरू8. व्यवहार के दोनों प्रकार, यानी, बजबजा रही है और शरीर के गठन फलने, केवल कोशिकाओं है कि एक ठोस सतह पर रखा जाता है द्वारा निष्पादित कर रहे हैं । इसके अलावा, दोनों पोषक तत्वों की स्थिति के तहत, कोशिकाओं प्रक्रियाओं है कि बाहरी झिल्ली लिपो कि गतिशीलता या प्राप्तकर्ता9,10 में विषाक्त पदार्थों के रूप में समारोह को उत्तेजित कर सकते है के आदान प्रदान सहित सीधे सेल सेल संपर्कों को शामिल में संलग्न ,11एलपीएस के आदान-प्रदान, गतिशीलता के पड़ोसी कोशिकाओं12पर exopolysaccharides द्वारा उत्तेजना, और एक सेल की सतह से संकेतन-13,14संकेत प्रोटीन लंगर ।
हाल ही में, एम xanthus भी गतिशीलता अंतर्निहित तंत्र का अध्ययन करने के लिए एक मॉडल जीव बन गया है और इसके विनियमन15, सेल प्रभाग16,17,18, और गुणसूत्र संगठन19 ,20,21. एम. xanthus सेल चक्र में महत्वपूर्ण कदम प्रतिदीप्ति माइक्रोस्कोपी द्वारा स्नैप-शॉट छवियों या कम समय पर चूक रिकॉर्डिंग का उपयोग करके विस्तार से विश्लेषण किया गया है प्रासंगिक फ्लोरोसेंट को ले जाने उपभेदों पर प्रोटीन्स लेबल16, 17,18,19,20. आदर्श रूप में, कक्ष चक्र पैरामीटर्स पर संतुलित मात्रात्मक डेटा प्राप्त करने के लिए कम से एक पूर्ण कक्ष चक्र के लिए प्रतिदीप्ति लाइव सेल इमेजिंग द्वारा एकल-कक्ष रिज़ॉल्यूशन के साथ कई कक्षों का पालन किया जाना चाहिए. हालांकि, यह मानक प्रयोगशाला शर्तों के तहत 4-6 एच की अपनी अपेक्षाकृत लंबी पीढ़ी के समय के कारण और छवि अधिग्रहण के दौरान fluorophores और phototoxicity के photobleaching के कारण एम xanthus के मामले में एक चुनौती है ।
यहाँ, हम प्रतिदीप्ति लाइव सेल इमेजिंग द्वारा एकल सेल संकल्प के साथ एम xanthus कोशिकाओं का पालन करने के लिए एक प्रोटोकॉल का वर्णन कम से कम 24 घंटे के लिए और कई सेल चक्र को कवर. महत्वपूर्ण बात, पूरे प्रोटोकॉल के दौरान, कोशिकाओं को एक आगर पैड पर बनाए रखा और निकट संपर्क में एम. xanthusके सामाजिक जीवन शैली के लिए आवश्यक संपर्क-निर्भर गतिविधियों के लिए अनुमति दे रहे हैं । प्रोटोकॉल भी उपयोगकर्ताओं को आकार, आकार, विभाजन पर नजर रखने के लिए अनुमति देता है, और एक उच्च लौकिक संकल्प और एक सेल संकल्प के साथ फ्लोरोसेंट जांच, और इस प्रकार, सेल के ठहराव-सेल परिवर्तनशीलता और सेल चक्र की घटनाओं के सहसंबंध सक्षम बनाता है ।
Protocol
1. M. xanthus उपभेदों की तैयारी और वृद्धि
नोट: तालिका 1 और तालिका 2देखें ।
- तैयार 1% casitone शोरबा (CTT) विकास मध्यम 1% (डब्ल्यू/) अग्नाशय डाइजेस्ट कैसिइन के (जैसे, Bacto casitone), 10 मिमी Tris-एचसीएल पीएच ८.०, 1 मिमी KH2पीओ4 पीएच ७.६, 8 मिमी MgSO422, कनमीसिन के साथ पूरक (५० µ g/एमएल) या ओक्षयत्टे्रास्यकलने (10 µ g/mL) । अंय बैक्टीरिया के साथ संक्रमण के जोखिम को कम करने के लिए सभी मीडिया के लिए गेन्तमयसीं (10 µ जी/एमएल) जोड़ें, क्योंकि एम xanthus कोशिकाओं को स्वाभाविक रूप से यह प्रतिरोधी रहे हैं ।
- Inoculate 5 मिलीलीटर 1% CTT युक्त एंटीबायोटिक (ओं) जंगली प्रकार की एक ताजा उगाई कॉलोनी के साथ (WT) DK1622 23, SA4420 (Δ mglA)24, SA4797 (ΔmglA, ΔpomX/PpomZ mCherry-pomX )16, SA8241 (ΔmglA, ftsZ+/PनातftsZ-gfp), या SA4749 (ΔmglA, parB+/PनातparB-yfp) दिन की सुबह 1.
- 1% CTT के ५०० µ एल में एक एकल एम. xanthus कॉलोनी reसस्पेंड एक बाँझ ट्यूब में एंटीबायोटिक दवाओं के साथ पूरक और एक ५० मिलीलीटर Erlenmeyer कुप्पी के पूरे निलंबन हस्तांतरण 1% CTT की 5 मिलीलीटर युक्त ।
नोट: पर्याप्त aeriation और इष्टतम विकास की गारंटी के लिए संस्कृति के 10 गुना मात्रा के साथ एक Erlenmeyer कुप्पी का उपयोग करें ।
- 1% CTT के ५०० µ एल में एक एकल एम. xanthus कॉलोनी reसस्पेंड एक बाँझ ट्यूब में एंटीबायोटिक दवाओं के साथ पूरक और एक ५० मिलीलीटर Erlenmeyer कुप्पी के पूरे निलंबन हस्तांतरण 1% CTT की 5 मिलीलीटर युक्त ।
- आठ पीढ़ियों के लिए कोशिकाओं को विकसित (लगभग ४०-४८ एच 4-6 एच की एक पीढ़ी के समय के साथ) ३२ डिग्री सेल्सियस पर, में मिलाते हुए २२० rpm, अंधेरे में । घातीय वृद्धि चरण (आयुध डिपो५५० < 1.2) में कोशिकाओं को बनाए रखने और उंहें स्थिर चरण तक पहुंचने से रोकने के । यदि आवश्यक हो, कोशिकाओं को ताजा 1% CTT में प्रासंगिक एंटीबायोटिक (एस) के एक आयुध डिपो ०.१-०.२ के५५० से युक्त मध्यम ।
नोट: एक इष्टतम एक एकल कोशिका माइक्रोस्कोपी के लिए५५० आयुध डिपो ०.५-०.७ है । इस आयुध डिपो में५५०, कोशिकाओं की एक पर्याप्त संख्या छवि प्रति मौजूद है ठहराव के रूप में के रूप में अच्छी तरह से सेलुलर मापदंडों के सांख्यिकीय विश्लेषण की अनुमति है ।
2. माइक्रोस्कोपी नमूनों की तैयारी
नोट: कोशिकाओं माइक्रोस्कोपी द्वारा देखा जा करने के लिए एक खुर्दबीन coverslip पर रखा जाता है और फिर एक agarose पोषक तत्वों युक्त पैड द्वारा कवर किया जाता है । coverslip यांत्रिक समर्थन प्रदान करने के लिए एक प्लास्टिक या धातु फ्रेम करने के लिए चिपके है । माइक्रोस्कोपी के लिए तैयार करने में, 1% agarose/TPM/0.2% CTT के एक बड़े पैड पहले चरण २.१-२.३ में वर्णित के रूप में तैयार किया जाना चाहिए । कृपया यहां प्रयुक्त विशिष्ट उत्पादों के लिए सामग्री तालिका का भी उल्लेख करें ।
- ५०० मिलीलीटर बफ़र (10 मिमी Tris-एचसीएल पीएच ७.६, 1 मिमी KH2पीओ4 पीएच ७.६, 8 मिमी MgSO4) और आटोक्लेव या फिल्टर एक बोतल शीर्ष फिल्टर का उपयोग कर बंध्याकरण की तैयारी करें ।
नोट: बाँझ बफर कमरे के तापमान पर कई महीनों के लिए संग्रहीत किया जा सकता है । - 1% agarose माइक्रोस्कोपी युक्त समाधान तैयार करें जिसमें ०.२% CTT (agarose के 1 ग्राम TPM बफर और 1% CTT मीडियम की 20 मिलीलीटर के साथ मिश्रण) । एक माइक्रोवेव ओवन में गर्मी जब तक agarose पिघला हुआ है ।
नोट: ०.२% CTT कोशिकाओं को बढ़ने और भुखमरी को रोकने के लिए अनुमति देने के लिए पर्याप्त है । सूक्ष्म मध्यम में CTT के उच्च सांद्रता उच्च पृष्ठभूमि प्रतिदीप्ति में परिणाम होगा । - पिघला हुआ agarose के साथ एक पेट्री पकवान ०.५ सेमी की एक मोटाई के लिए भरें (एक ११.५ सेमी x ११.५ सेमी स्क्वायर पेट्री डिश के लिए, पिघला हुआ agarose के लगभग ६० मिलीलीटर की आवश्यकता है) और यह कमरे के तापमान के लिए नीचे शांत करते हैं ।
नोट: agarose पैड को 2 दिनों तक आर्द्र वातावरण में 4 डिग्री सेल्सियस पर संग्रहित किया जा सकता है ।- पूर्व गर्म 1% agarose/TPM/0.2% CTT पैड का उपयोग करने से पहले कम से कम 15 मिनट के लिए ३२ डिग्री सेल्सियस पर ।
नोट: माइक्रोस्कोप के लिए कोशिकाओं को तैयार करने के लिए, चरणों का पालन करें २.४-२.८.
- पूर्व गर्म 1% agarose/TPM/0.2% CTT पैड का उपयोग करने से पहले कम से कम 15 मिनट के लिए ३२ डिग्री सेल्सियस पर ।
- एक प्लास्टिक या धातु फ्रेम कि बीच में एक छेद है पर एक बाँझ ग्लास coverslip (६० मिमी x 22 मिमी, मोटाई: ०.७ मिमी) प्लेस (चित्रा 1ए); इस फ्रेम पतली coverslip के लिए एक यांत्रिक समर्थन के रूप में कार्य करता है और माइक्रोस्कोपी के दौरान बहाव को कम करने में मदद करता है । टेप के साथ फ्रेम करने के लिए coverslip को ठीक करें ।
- फ्रेम तैयार करने के लिए, एक 1 मिमी मोटी धातु की थाली से एक ७५ मिमी × 25 मिमी फ्रेम में कटौती, तो बीच में एक उचित आकार छेद (इस प्रयोग में 20 मिमी × 30 मिमी) में कटौती ।
- coverslip पर तेजी से उगी मी. xanthus कोशिकाओं के 10-20 µ l को जोड़ें ।
- कोशिकाओं या समय चूक रिकॉर्डिंग में प्रोटीन की ट्रैकिंग को सरल बनाने के लिए फिड्यूशियल मार्करों के रूप में फ्लोरोसेंट ०.५ µm microspheres जोड़ें ।
- microspheres 1:100 TPM बफ़र में और कई महीनों के लिए 4 ° c पर संग्रह को पतला करें । उपयोग करने से पहले अच्छी तरह से हिलाएं और पतला microspheres के 5-10 µ l को कोशिकाओं में जोड़ें ।
नोट: यहां microspheres कि सभी आम नीले, हरे, पीले, और लाल फ्लोरोसेंट चैनलों में फ्लोरोसेंट इस्तेमाल किया गया ।
- microspheres 1:100 TPM बफ़र में और कई महीनों के लिए 4 ° c पर संग्रह को पतला करें । उपयोग करने से पहले अच्छी तरह से हिलाएं और पतला microspheres के 5-10 µ l को कोशिकाओं में जोड़ें ।
- एक छोटा सा पैड लगभग बड़े पूर्व के coverslip के आकार में कटौती 1% agarose/TPM/0.2% CTT पैड और यह कोशिकाओं के शीर्ष पर जगह (चित्रा 1ख) । वाष्पीकरण को रोकने के लिए और आर्द्र वातावरण में कोशिकाओं को बनाए रखने के लिए 1% agarose/TPM/0.2% CTT agarose पैड के शीर्ष पर एक coverslip रखें ।
नोट: अकेले coverslip के लिए महत्वपूर्ण वाष्पीकरण को रोकने जाएगा कम से 2 ज । लंबे समय तक चूक रिकॉर्डिंग के लिए, 1% agarose/TPM/0.2% CTT पैड और coverslip सैंडविच तेल फिल्म के साथ बंद कर दिया जाना चाहिए वाष्पीकरण को रोकने के लिए । - agarose पैड के नीचे करने के लिए कोशिकाओं को देते हैं करने के लिए 15-20 मिनट के लिए ३२ ° c पर माइक्रोस्कोप नमूना मशीन । फिर समय चूक माइक्रोस्कोपी रिकॉर्डिंग शुरू करते हैं ।
3. माइक्रोस्कोप सेट अप और समय चूक अधिग्रहण
नोट: प्रोटोकॉल यहां वर्णित के साथ एक औंधा widefield माइक्रोस्कोप के लिए विकसित किया गया था, एक 100X/1.30 न तेल PH3 उद्देश्य, एक एक्स, वाई मोटर चालित चरण, एक sCMOS कैमरा, एक प्रकाश स्रोत, ग्रीन के लिए फिल्टर-फ्लोरोसेंट, लाल-फ्लोरोसेंट, या पीले-फ्लोरोसेंट प्रोटीन, और एक तापमान नियंत्रित गर्मी चैंबर । यह कक्ष प्रकाश से और स्थिर तापमान पर कोशिकाओं को सुरक्षित रखता है ।
- पूर्व, सूक्ष्मता शुरू करने से पहले मशीन चैंबर और ३२ ° c के लिए माइक्रोस्कोप ~ 1-2 h गर्मी ।
नोट: माइक्रोस्कोप सेट अप पर निर्भर करता है, हीटिंग अधिक समय लग सकता है । पूर्व हीटिंग बहाव को कम करने के लिए आवश्यक है और फोकस नियंत्रण प्रणाली स्थिर । - माइक्रोस्कोप पर स्विच और माइक्रोस्कोप नियंत्रण सॉफ्टवेयर शुरू करते हैं । सही उद्देश्य का चयन करें और सही दर्पण और फिल्टर चरण कंट्रास्ट छवियों के रूप में के रूप में अच्छी तरह से हरी-फ्लोरोसेंट, लाल-फ्लोरोसेंट, या पीले-फ्लोरोसेंट प्रोटीन की छवियों को प्राप्त करने के लिए ।
नोट: एक खुर्दबीन आमतौर पर माइक्रोस्कोप नियंत्रण और छवि अधिग्रहण के लिए एक पसंदीदा सॉफ्टवेयर के साथ आपूर्ति की है । यहां एक व्यावसायिक रूप से उपलब्ध सॉफ्टवेयर ( सामग्री की तालिकादेखें) माइक्रोस्कोप और छवि अधिग्रहण को नियंत्रित करने के लिए इस्तेमाल किया गया था । - उद्देश्य के लेंस पर उच्च गुणवत्ता विसर्जन तेल की एक बूंद जोड़ें और नमूना पूर्व के नीचे करने के लिए ३२ डिग्री सेल्सियस पर मशीन । सबसे कम संभव Z-स्थिति उद्देश्य लेंस हानिकारक जब नमूना माइक्रोस्कोप मंच पर रखा गया है से बचने के लिए पर उद्देश्य प्लेस । धातु फ्रेम माइक्रोस्कोप मंच पर नमूना के साथ और उद्देश्य की ओर "होल साइड" के साथ प्लेस । नमूना सुरक्षित रूप से स्टेज धारक में जकड़ना ।
- उद्देश्य के लिए करीब Z-दिशा में मंच ले जाकर कोशिकाओं पर ध्यान केंद्रित । धीमी गति से कदम जब तेल नमूना नीचे की ओर और उद्देश्य लेंस से संपर्क करें पर गिरता है । जब कक्ष फोकल विमान में हों, तब तक एकाधिक एकल कक्ष दृश्य के क्षेत्र में दृश्यमान होते हैं, जब तक चरण X/ करने के लिए बाद में अधिग्रहीत छवियों को संरेखित करने के लिए एक फ्लोरोसेंट microsphere को देखने के क्षेत्र में है कि सुनिश्चित करें ।
नोट: इष्टतम शर्तों के तहत, दृश्य (२,०४८ x २,०४८ पिक्सेल या १३३.१ x १३३.१ µm) के प्रति क्षेत्र 15-30 कक्षों का एक कक्ष घनत्व तक पहुंच जाना चाहिए । - माइक्रोस्कोप नियंत्रण सॉफ्टवेयर के बहु आयामी अधिग्रहण जादूगर को खोलने के लिए एक समय चूक प्रयोग है कि कई तरंग दैर्ध्य और मंच पदों पर छवियों को प्राप्त करने के लिए अगर आवश्यक अनुमति देता है सेट ।
- मुख्य टैब में डीकॉक और कई तरंग दैर्ध्यको सक्रिय करें । अतिरिक्त टैब्स विंडो के बाईं ओर दिखाई देंगे ।
- बचत टैब पर क्लिक करें और चुनें निर्देशिका अधिग्रहीत छवियों को बचाने के लिए कंप्यूटर हार्ड ड्राइव पर एक खाली फ़ोल्डर का चयन करने के लिए । यह सुनिश्चित करने के लिए कि लगातार डेटासेट पहले वाले अधिलेखित न करें फ़ाइल मौजूद है, तो वृद्धि आधार नाम सक्रिय करें । फिर प्रयोग को तारीख और तनाव का नाम या प्रयोग के शीर्षक के साथ एक नाम दे ।
- समय चूक मापदंडों को समायोजित करने के लिए डीकॉक टैब पर क्लिक करें । अवधि को 24 घंटे के लिए सेट करें और 20 मिनट के लिए समय अंतराल सेट करें । समय बिंदुओं की संख्या स्वचालित रूप से बदल जाएगी ।
नोट: इष्टतम समय अंतराल प्रयोग और सेलुलर समारोह पर निर्भर करता है विश्लेषण किया जाना है । लगातार छवि अधिग्रहण photobleaching कारण हो सकता है । इस प्रकार, लौकिक संकल्प और photobleaching के बीच एक व्यापार बंद पाया empirically होना चाहिए । 4-6 एच के दोहरीकरण के समय में, छवियों को आसानी से 5 मिनट के अंतराल पर प्राप्त कर सकते है (या भी छोटे अंतराल अगर वांछित) चरण कंट्रास्ट माइक्रोस्कोपी के लिए । यदि एक समय पर प्रतिदीप्ति माइक्रोस्कोपी-24 एच के पाठ्यक्रम वांछित छवियों लगभग 15-30 मिनट के अंतराल पर दर्ज किया जाना चाहिए है । - तरंग दैर्ध्य टैब पर क्लिक करें । संख्या बदलकर प्रत्येक समय बिंदु पर प्रत्येक छवि के लिए प्राप्त करने के लिए तरंग दैर्ध्य की संख्या चुनें ।
नोट: प्रत्येक तरंग दैर्ध्य के लिए, एक नया टैब बहु-आयामी अधिग्रहण " जादूगर और तरंग दैर्ध्य के बाईं ओर दिखाई देगा ऊपर से नीचे तक के क्रम में प्राप्त किया जाएगा । प्रत्येक तरंग दैर्ध्य के लिए, अधिग्रहण सेटिंग्स अलग से संशोधित किया जा सकता है । - ऊपर से प्रथम तरंग दैर्ध्य टैब क्लिक करें । रोशनी ड्रॉप डाउन सूची में चरण कंट्रास्ट का चयन करें । जोखिम के लिए १०० ms का चयन करें और प्राप्त ड्रॉप-डाउन सूची में हर समय बिंदु का चयन. निष्क्रिय ऑटो कभी ड्रॉप डाउन सूची में चयन करके बेनकाब ।
- प्रत्येक समय बिंदु पर प्राप्त की जाने वाली प्रत्येक तरंग दैर्ध्य के लिए चरण 3.5.5 दोहराएँ. प्रायोगिक सेट अप और फ्लोरोसेंट लेबल प्रोटीन के लिए यहां वर्णित है, जोखिम के लिए निंनलिखित मापदंडों का उपयोग करें : २५० mCherry फ्यूजन प्रोटीन के लिए एमएस, २०० YFP फ्यूजन प्रोटीन के लिए एमएस, और १,००० GFP फ्यूजन प्रोटीन के लिए एमएस ।
नोट: प्रत्येक तनाव और फ्लोरोसेंट प्रोटीन के लिए इष्टतम रोशनी सेटिंग्स पहले से दीपक तीव्रता और प्रत्येक तरंग दैर्ध्य के लिए छवि अधिग्रहण समय बदलकर निर्धारित किया जाना चाहिए । बहुत लंबी छवि अधिग्रहण बार phototoxic प्रभाव में वृद्धि होगी और अंततः विकास की गिरफ्तारी और सेल मौत के लिए सीसा । इसलिए, छवि गुणवत्ता और कोशिका व्यवहार्यता के बीच एक व्यापार बंद हासिल किया जाना चाहिए । - एक ही प्रयोग में दर्ज कोशिकाओं की संख्या को बढ़ाने के लिए कई चरण पदों से छवियों को प्राप्त करें ।
- कई चरण की स्थितियों से छवियां प्राप्त करने के लिए, मुख्य टैब में एकाधिक चरण स्थितियां सक्रिय करें । फिर स्टेज टैब पर क्लिक करें और देखने के क्षेत्र में देखने के लिए लाइव बटन पर क्लिक करें ।
- चरण को X/Y-दिशा में तब तक ले जाएं जब तक कि ब्याज का क्षेत्र (ROI) दृश्य के क्षेत्र में न हो । x-और y-निर्देशांक सहेजें चरण टैब में "+" पर क्लिक करके एक नया ROI पाया जाता है जब तक कि चरण फिर से x/Y-दिशा में ले जाएं और "+" पर क्लिक करके निर्देशांक फिर से सहेजें । क्षेत्रों की वांछित संख्या सहेजे जाने तक पर जाएं ।
नोट: प्रतिदीप्ति छवि अधिग्रहण के मामले में, सुनिश्चित करें कि ब्याज के क्षेत्रों (ROIs) भी phototoxicity को कम करने के लिए एक दूसरे के पास नहीं हैं ।
- एक बार और अधिक है कि कोशिकाओं को अलग बचाया एक्स पर क्लिक करके ध्यान में है और Y-पदों की जांच करें और एएफसी पकड़ो प्रयोग के पाठ्यक्रम पर बचाया जेड स्थिति लगातार रखने के लिए क्लिक करके हार्डवेयर फोकस शुरू करते हैं ।
- बहु-आयामी अधिग्रहण विज़ार्ड में प्राप्त क्लिक करके माइक्रोस्कोप नियंत्रण सॉफ्टवेयर में समय चूक रिकॉर्डिंग शुरू करो ।
नोट: प्रत्येक के लिए एक विंडो दिखाई देगा तरंग दैर्ध्य प्राप्त किया है और एक अतिरिक्त विंडो दिखाई देगा जो प्राप्त किए गए समय बिंदुओं की संख्या और अगले चित्र प्राप्ति तक समय दिखाता है । - जांच करें कि कोशिकाओं को समय चूक रिकॉर्डिंग में पहले कुछ समय अंक के बाद ध्यान में अभी भी कर रहे है छवियों की गुणवत्ता को अधिकतम करने के लिए और यदि आवश्यक refocus ।
4. समय चूक फिल्मों और छवि संरेखण की पीढ़ी
नोट: कई वाणिज्यिक और मुफ्त सॉफ्टवेयर संकुल छवि अधिग्रहण और छवि विश्लेषण के लिए उपलब्ध हैं । हम कई पूर्व स्थापित plugins और अतिरिक्त उपकरणों के साथ एक व्यावसायिक रूप से उपलब्ध सॉफ्टवेयर ( सामग्री की तालिकादेखें) का उपयोग करें ।
- छवि विश्लेषण/संसाधन सॉफ़्टवेयर स्थापित है जो किसी कंप्यूटर पर समय-चूक रिकॉर्डिंग से व्यक्तिगत छवियाँ सहेजें ।
- बहु-आयामी डेटा की समीक्षा करें क्लिक करके एक स्टैक के रूप में सॉफ़्टवेयर और खुली छवियां प्रारंभ करें | आधार फ़ाइल का चयन करें | निर्देशिका का चयन करें। बहु-आयामी डेटा के साथ फ़ोल्डर खोलें । डेटासेट जांचें और देखेंक्लिक करें; डेटासेट एकल छवियों के रूप में समय बिंदु एक से अंत तक दिखाया जाएगा । तरंग दैर्ध्य सक्रिय (एक ढेर बनाने के लिए), सभी छवियां है कि ढेर में होना चाहिए और लोड छवि (ओं)को क्लिक करें । सभी तरंग दैर्ध्य के लिए इस चरण को दोहराएँ और पूर्ण स्टैक सहेजें ।
- वैकल्पिक सभी फ़ाइल का उपयोग कर फिल्म के लिए आवश्यक छवियां खोलें । खुला।
नोट: यह एक समय में एक तरंग दैर्ध्य को प्राप्त करने के लिए कंप्यूटर को धीमा नहीं अगर अभिकलनी शक्ति सीमित है द्वारा छवियों को खोलने के लिए सिफारिश की है । यदि समय चूक रिकॉर्डिंग के कुछ भागों, जैसे, शुरू, अंत, या कई समय अंक छोड़ दिया जाना चाहिए, तो यह पूरा फिल्म में समायोजित किया जा सकता है । - बहाव के लिए सही किया जा करने की जरूरत है कि छवियों के ढेर को सक्रिय करें. Apps द्वारा संरेखण उपकरण खोलें । स्वत: संरेखित करें... । संदर्भ विमान के रूप में छवियों और पहले विमान/समय बिंदु के लिए स्रोत के रूप में स्टैक जांचें । स्रोत स्टैक बटन के साथ स्टैक का चयन करें और लागूकरेंक्लिक करे ।
नोट: स्वचालित संरेखण कुछ समय और गणना शक्ति ले जाएगा, लेकिन माइक्रोस्कोप सेट अप के बहाव के लिए बड़े ढेर सही करने के लिए एक अच्छा तरीका है. इस स्वचालित संरेखण अच्छी तरह से काम करता है अगर microspheres शामिल हैं, लेकिन उनके बिना भी काम हो सकता है । - संरेखित स्टैक सहेजें ।
- ROIs का उपयोग करें ।
ध्यान दें: फ्लोरोसेंट समय चूक माइक्रोस्कोपी आसानी से डेटा फ़ाइलों के बड़े सेट है कि गणना शक्ति का एक बहुत ले और नीचे इन फिल्मों के बहाव प्रसंस्करण धीमा बनाता है । इसलिए हम ROIs की पहचान करने और छोटी फ़ाइलों के साथ कार्य करने के लिए कक्षों को अलग करने की पुरजोर अनुशंसा करते हैं ।- आयताकार क्षेत्र उपकरण का चयन करें । मैंयुअल रूप से चरण विपरीत छवि पर एक रॉय ड्राइंग द्वारा ब्याज की कोशिकाओं के आसपास एक रॉय बनाएं । सुनिश्चित करें कि ब्याज की कोशिकाओं को दिखाई और पूरे समय चूक फिल्म भर में ध्यान में हैं ।
- एक ही डेटासेट की दूसरी तरंग दैर्ध्य की समय-चूक फिल्म खोलें. के लिए चरण विपरीत चित्रों से रॉय दूसरी तरंग दैर्ध्य के प्रतिदीप्ति छवियों को हस्तांतरण क्षेत्रों के साथ स्थानांतरण क्षेत्रों उपकरण का उपयोग करें । स्थानांतरण क्षेत्र। चरण कन्ट्रास्ट dataset स्रोत छवि और दूसरी तरंग दैर्ध्य dataset गंतव्य छविके रूप में का चयन करें । सभी क्षेत्रों का चयन करें और ठीकदबाएं ।
- एक ही dataset के लिए प्राप्त प्रत्येक तरंग दैर्ध्य के लिए चरण 4.6.2 दोहराएँ ।
- रॉय का चयन करें और इसे संपादित करें के साथ एक स्टैक के रूप में डुप्लिकेट । डुप्लिकेट । पोट... या Shift + ctrl + D कुंजियां दबाएं । फिर डुप्लिकेट स्टैक फ़ाइल के साथ सहेजें । मूल डेटा के रूप में एक ही फ़ोल्डर में सहेजें ।
- एक ही dataset के लिए प्राप्त प्रत्येक तरंग दैर्ध्य के प्रत्येक ROI के लिए चरण 4.6.4 दोहराएँ
- MOV या AVI स्वरूपों में कोई चलचित्र जनरेट करने के लिए, स्टैक के माध्यम से चलचित्र बनाएं फ़ंक्शन खोलें । फिल्म बनाओ। स्रोत स्टैक बटन के साथ समय-चूक रिकॉर्डिंग का चयन करें । आउटपुट स्वरूप, फ़्रेम दर, फ़्रेंस की संख्या, और सहेजेंक्लिक करें ।
Representative Results
M. xanthus एक धीमी गति से बढ़ रही जीवाणु है कि ठोस सतहों पर चलता है । हमारे प्रायोगिक सेट-अप का परीक्षण करने के लिए, हमने gram DK1622 WT कक्षों के साथ समय-चूक प्रयोग किया । चरण कंट्रास्ट छवियां 5 मिनट के अंतराल पर 24 घंटे के लिए अधिग्रहीत की गई (चित्रा 2ए, बी) । कक्षों के अधिकांश समूह में संरेखित । आशा के रूप में, कोशिकाओं गतिशीलता प्रदर्शित और मुख्य रूप से समूहों में ले जाया गया । हम आगे कहा कि कोशिकाओं के आंदोलन की दिशा कभी-कभार उलट । इन निष्कर्षों का सुझाव है कि परीक्षण शर्तों के तहत WT कोशिकाओं कोशिका गतिशीलता के मामले में सामान्य रूप से व्यवहार. हालांकि, यहां तक कि जब कोशिकाओं हर 5 मिनट दर्ज कर रहे हैं, व्यक्तिगत कोशिकाओं की पहचान मुश्किल है । इसके अलावा, क्योंकि कोशिकाओं को गतिशील हैं, कई कोशिकाओं को भागने या विस्तारित अवधि के लिए कोशिकाओं का पालन करने के लिए मुश्किल बना देखने के क्षेत्र में प्रवेश ।
लाइव सेल इमेजिंग द्वारा सेल चक्र के कई दौर के लिए एक ही एम. xanthus कोशिकाओं का पता लगाने के क्रम में, व्यक्तिगत उपभेदों mglA जीन है, जो25गतिशीलता के लिए आवश्यक है के लिए नष्ट किया जा सकता है । यह कक्ष इमेजिंग प्रोटोकॉल के दौरान दृश्य के फ़ील्ड से बाहर ले जाने से रोकता है । में फ्रेम विलोपन शि एट अलद्वारा वर्णित के रूप में उत्पंन कर रहे हैं । 26
अपेक्षा के अनुरूप, चरण विपरीत लाइव-सेल इमेजिंग गैर-gram ΔmglA कोशिकाओं (चित्रा 3) के साथ, कोशिकाओं सक्रिय आंदोलन प्रदर्शित नहीं किया. हम microcolony गठन के दौरान व्यक्तिगत कोशिकाओं के विकास और विभाजन का पालन करने में सक्षम थे । समय-चूक रिकॉर्डिंग में छवियों को 24 घंटे के लिए 5 मिनट के अंतराल पर अधिग्रहीत किया गया था के आधार पर, यह एक सेल रिज़ॉल्यूशन के साथ विभाजन के समय (दो कक्ष विभाजन घटनाओं के बीच का समय) यों तो संभव था । ΔmglA उत्परिवर्ती के कक्ष एक अंतर २३५ ± ५० मिनट (n = ९७ कोशिकाओं) के विभाजन का समय था । लगभग 4 एच के साथ, विभाजन के समय WT कोशिकाओं के लिए निलंबन संस्कृतियों में मापा दोहरीकरण समय के समान है । यह सबूत है कि एम xanthus कोशिकाओं इन प्रयोगात्मक शर्तों के तहत बेहतर हो जाना प्रदान करता है ।
की जांच करने के लिए कि हमारे सेट अप कोशिकाओं को सामांय रूप से बढ़ने की अनुमति देता है, जबकि लंबे समय से अधिक YFP-लेबल प्रोटीन ट्रैकिंग, हम एम xanthus कोशिकाओं के साथ प्रतिदीप्ति समय चूक इमेजिंग प्रदर्शन किया है कि एक YFP-टैग प्रोटीन व्यक्त करते हैं । इस अंत करने के लिए, हम ParB-YFP प्रतिकृति (ओरि) की उत्पत्ति के लिए एक मार्कर के रूप में फ़ॉलो किया । ParB एम. xanthus में परबS प्रणाली के घटक के रूप में है और पार्स साइटों के लिए समीपस्थ की ओरिमें बांधता है; इसलिए, मूल दोहराव और गुणसूत्र अलगाव19,20,21के बाद किया जा सकता है । छवि अधिग्रहण के साथ (चरण कंट्रास्ट और प्रतिदीप्ति, २०० YFP चैनल में एमएस अधिग्रहण समय) हर 20 मिनट, कोशिकाओं बढ़ी, विभाजित है, और 24 घंटे में भी वृद्धि प्रदर्शित (चित्रा 4ए) । रिकॉर्डिंग के शुरू में, ParB-YFP कोशिकाओं के बहुमत में उपध्रुवीय क्षेत्रों में दो समूहों का गठन (चित्रा 4ए). कुछ ही समय पहले या सेल डिवीजन के बाद, उपध्रुवीय ParB-YFP क्लस्टर पुराने सेल पोल पर दोहराया । दो समूहों में से एक पुराने सेल पोल पर बनी हुई है जबकि दूसरी कॉपी नए सेल पोल करने के लिए translocated, लगभग ४०-६० मिनट (चित्रा 4ए, बी) के बाद अपने अंतिम उपध्रुवीय स्थिति तक पहुंचने । ये अवलोकन कम समय चूक रिकॉर्डिंग का उपयोग कर पतली आगर पैड19से उत्पन्न पिछले डेटा के साथ समझौते में हैं । हम निष्कर्ष निकालना है कि इस प्रयोगात्मक सेट अप प्रतिदीप्ति समय चूक के लिए धीमी गति से बढ़ती एम xanthus कोशिकाओं में कई सेल चक्र पर गुणसूत्र अलगाव ट्रैक करने की अनुमति देता है, perturbing सेल विकास या गुणसूत्र अलगाव मशीनरी के बिना ।
एक समान प्रयोग में, हम समय चूक प्रतिदीप्ति माइक्रोस्कोपी द्वारा सेल विभाजन के लिए मार्करों का पालन करने की मांग की । लगभग सभी अंय बैक्टीरिया के समान, M. xanthus FtsZ की आवश्यकता है, एक जीवाणु tubulin की तरह GTPase, सेल प्रभाग16,17,18के लिए । FtsZ midcell पर एक अंगूठी की तरह संरचना रूपों, तथाकथित Z-अंगूठी, कि अंय सभी सेल प्रभाग27,28के लिए आवश्यक प्रोटीन की भर्ती में मदद करता है । एम. xanthusमें, Z-अंगूठी के गठन और midcell पर अपनी स्थिति तीन PomXYZ16,17प्रोटीन से प्रेरित है । इन तीन प्रोटीन एक गुणसूत्र-संबद्ध जटिल है कि सेल प्रभाग की साइट से nucleoid भर में स्थानांतरण "मां" सेल में दो बेटी की कोशिकाओं में nucleoid के बीच में । nucleoid के मध्य midcell के साथ मेल खाती है, गुणसूत्र अलगाव से पहले, और यहां PomXYZ परिसर रंगरूटों FtsZ और जेड-अंगूठी गठन को उत्तेजित करता है ।
यहां, हम पहले गैर-gram ftsZ-gfpएक्सप्रेस कोशिकाओं के बाद । क्योंकि FtsZ-GFP समग्र ParB-YFP से एक कमजोर प्रतिदीप्ति संकेत दिखाता है, हम जोखिम समय में वृद्धि 5-GFP चैनल में 1 एस के लिए गुना । के रूप में की उंमीद है, FtsZ के मजबूत संचय-GFP केवल midcell में मनाया गया था और इस स्थानीयकरण सेल विभाजन कसना (चित्रा 5ए) की स्थिति तय की । FtsZ-GFP मुख्य रूप से अब सेल में midcell पर एक क्लस्टर का गठन किया । यह भी स्पष्ट है कि इस क्लस्टर समय के साथ तीव्रता में वृद्धि हुई थी । सेल विभाजन के बाद, हमने देखा है कि FtsZ-GFP फिर से दो बेटी कोशिकाओं में लगभग 2 ज बाद में midcell में जमा (चित्रा 5बी) । इस खोज के साथ संगत है कि लगभग ५०% की जनसंख्या में कक्षों का प्रदर्शन FtsZ स्थानीयकरण midcell पर स्नैप-शॉट विश्लेषण के आधार पर16,17.
एक दूसरे प्रयोग में, हम 24 ज कि एक्सप्रेस mCherry-pomXके लिए गैर-gram ΔmglA कोशिकाओं का पालन किया । PomXYZ प्रणाली के भाग के रूप में, PomX Z-अंगूठी गठन और स्थिति, जिससे midcell16में सेल डिवीजन उत्तेजक मार्गदर्शन करने में मदद करता है । mCherry के प्रतिदीप्ति संकेत-PomX मजबूत है और २५० ms. के प्रतिदीप्ति चैनल में एक जोखिम समय की अनुमति देता है महत्वपूर्ण बात, सभी कोशिकाओं के आकार में वृद्धि हुई है और प्रयोग के पाठ्यक्रम पर एक सेल डिवीजन घटना प्रदर्शित, 24 घंटे के बाद microcolonies बनाने ( चित्रा 6ए). के रूप में पहले16रिपोर्ट, लगभग सभी कक्षों में एक mCherry-PomX क्लस्टर शामिल है । इन midcell और समूहों में स्थानीय midcell translocated से दूर midcell के दौरान प्रयोग के पाठ्यक्रम के दौरान स्थानीयकृत के बहुमत । सेल डिवीजनों के दौरान, mCherry-PomX क्लस्टर प्रत्येक बेटी सेल एक क्लस्टर प्राप्त करने के साथ विभाजित किया गया था । FtsZ-GFP के विरोध के रूप में, mCherry-PomX midcell ८०-सेल चक्र के ९०% पर स्थानीयकृत और सेल विभाजन (चित्रा 6बी) के बाद जल्द ही इस स्थिति तक पहुंच गया ।
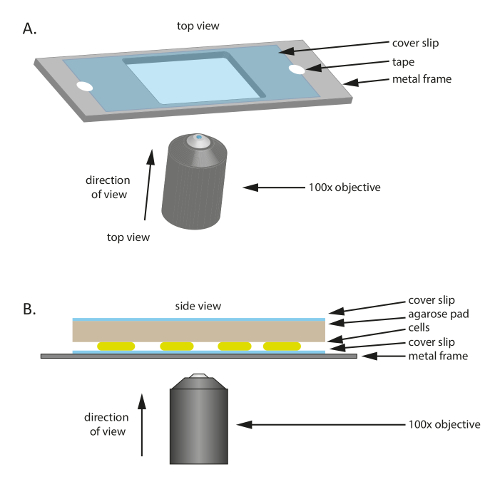
चित्रा 1 : प्रयोगात्मक सेट अप इस अध्ययन के दौरान इस्तेमाल की योजनाबद्ध । (क) एक धातु या प्लास्टिक के फ्रेम के नमूने के लिए एक समर्थन के रूप में कार्य करता है । एक coverslip नमूना की गति को कम करने के लिए टेप के साथ धातु फ्रेम करने के लिए तय हो गई है । (ख) प्रयोगात्मक नमूना सेट अप का पक्ष देखें । कक्ष (A) में दिखाए गए coverslip पर माउंट किए जाते हैं । agarose पैड है कि कोशिकाओं को पोषक तत्वों और नमी की आपूर्ति कोशिकाओं के शीर्ष पर रखा गया है । agarose पैड वाष्पीकरण को कम करने के लिए एक अतिरिक्त coverslip द्वारा कवर किया जाता है । उच्च गुणवत्ता वाले चित्रों के लिए, एक 100X तेल विसर्जन चरण कंट्रास्ट उद्देश्य का प्रयोग किया जाता है । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।

चित्रा 2 : WT M. xanthus कोशिकाओं के चरण कंट्रास्ट समय चूक माइक्रोस्कोपी । कोशिकाओं को 24 घंटे के लिए पीछा किया गया और छवियों को हर 5 मिनट का अधिग्रहण किया गया. (क) हर 5 मिनट के एक ही क्षेत्र के प्रतिनिधि छवियों को दिखाया जाता है । रंगीन तीर व्यक्तिगत कोशिकाओं के आंदोलन की दिशा संकेत मिलता है । समान रंग समय के साथ एक ही कक्ष को चिह्नित करता है । संख्याएँ मिनटों में समय का संकेत देती हैं. स्केल बार: 5 µm. (ख) हर घंटे के बाद देखने के एक ही क्षेत्र के चित्र दिखाए जाते हैं । ध्यान दें कि दृश्य का एक ही फ़ील्ड दिखाया गया है लेकिन क्योंकि कक्ष बढ़ रहे हैं, कक्ष लगातार प्रवेश कर रहे हैं और दृश्य के फ़ील्ड को छोड़ रहे हैं. संख्या घंटे में समय का संकेत है । स्केल बार: 5 µm. PH: चरण कंट्रास्ट । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।

चित्रा 3 : चरण विपरीत समय चूक गैर की माइक्रोस्कोपी-gram M. xanthus कोशिकाओं । ΔmglA कोशिकाओं के लिए पीछा किया गया था 24 घंटे के बाद हर 5 मिनट और प्रतिनिधि छवियों का अधिग्रहण किया गया छवियों को दिखाया गया है । चयनित कक्ष विभाजन कसना नारंगी तीर के साथ चिह्नित कर रहे हैं । संख्या घंटे में समय का संकेत है । पीएच: चरण कंट्रास्ट । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।
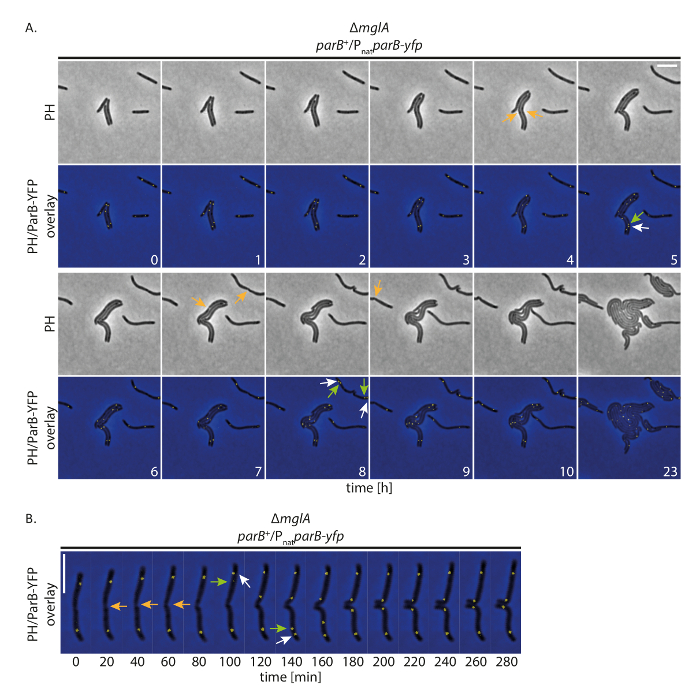
चित्र 4 : गैर-gram M. xanthus कोशिकाओं में ParB-YFP की प्रतिदीप्ति समय-चूक माइक्रोस्कोपी. एक ΔmglA उत्परिवर्ती व्यक्त की कोशिकाओं देशी parB की उपस्थिति मेंparB-yfp (SA4749; ΔmglA; parB +/PnatparB-yfp) चरण कंट्रास्ट और प्रतिदीप्ति माइक्रोस्कोपी द्वारा 24 ज के लिए पीछा किया गया । (A) छवियां हर घंटे के प्रत्येक 20 मिनट और प्रतिनिधि छवियों को अधिग्रहीत कर रहे थे जब तक 10 h दिखाए जाते हैं, एक साथ 24 घंटे के बाद एक ही कोशिकाओं के साथ (PH) और चरण कंट्रास्ट और YFP संकेत के ओवरले के रूप में दिखाए जाते हैं । चयनित कक्ष डिवीजनों नारंगी तीर के साथ चिह्नित कर रहे हैं । सफेद और हरे तीर ParB-YFP क्लस्टर दोहराव घटनाओं, translocating क्लस्टर अंकन हरे तीर के साथ इंगित करते हैं । संख्या घंटे में समय का संकेत है । स्केल बार: 5 µm. (ख) छवियों में (क) के रूप में प्राप्त कर रहे थे लेकिन उच्च लौकिक संकल्प पर दिखाए जाते हैं । संख्याएँ मिनटों में समय का संकेत देती हैं. तीर के रूप में कर रहे है (एक) । स्केल बार: 5 µm. कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण देखने के लिए ।

चित्रा 5 : गैर-gram M. xanthus कोशिकाओं में FtsZ-GFP की प्रतिदीप्ति समय-चूक माइक्रोस्कोपी. एक ΔmglA उत्परिवर्ती व्यक्त ftsZ-gfp की उपस्थिति में देशी ftsZ की कोशिकाओं (SA8241; ΔmglA; ftsZ +/PnatftsZ-gfp) चरण कंट्रास्ट और प्रतिदीप्ति माइक्रोस्कोपी द्वारा 24 ज के लिए पीछा किया गया । (A) छवियां हर घंटे के प्रत्येक 20 मिनट और प्रतिनिधि छवियों को अधिग्रहीत किया गया जब तक 10 ज, एक साथ 24 के बाद एक ही कोशिकाओं के साथ दिखाया जाता है h. images चरण कंट्रास्ट (PH) और चरण कंट्रास्ट और GFP संकेत के ओवरले के रूप में दिखाए जाते हैं । चयनित कक्ष डिवीजनों नारंगी तीर के साथ चिह्नित कर रहे हैं । सफेद तीर midcell पर FtsZ-GFP क्लस्टर्स इंगित करते हैं । संख्या घंटे में समय का संकेत है । स्केल बार: 5 µm. (ख) छवियों में (क) के रूप में प्राप्त कर रहे थे लेकिन उच्च लौकिक संकल्प पर दिखाए जाते हैं । संख्याएँ मिनटों में समय का संकेत देती हैं. हरे और सफेद तीर बाएँ और दाएँ कोशिकाओं में FtsZ-GFP समूहों, क्रमशः चिह्नित. नारंगी तीर कक्ष विभाजन इंगित करते हैं । स्केल बार: 5 µm. कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण देखने के लिए ।

चित्रा 6 : गैर-gram M. xanthus कोशिकाओं में mCherry-PomX की प्रतिदीप्ति समय-चूक माइक्रोस्कोपी. Non-gram ΔpomX cells जमते mCherry-pomX (SA4797; ΔmglA; ΔpomX/PpomZ mCherry-pomX) चरण कंट्रास्ट और प्रतिदीप्ति माइक्रोस्कोपी हर 20 मिनट द्वारा 24 ज के लिए पीछा किया गया । (क) प्रतिनिधि छवियां हर घंटे जब तक 10 ज, एक साथ 24 के बाद एक ही कोशिकाओं के साथ दिखाया ज. छवियां चरण कंट्रास्ट (PH) और चरण कंट्रास्ट और mCherry संकेत के ओवरले के रूप में दिखाए जाते हैं । चयनित कक्ष डिवीजनों नारंगी तीर के साथ चिह्नित कर रहे हैं । सफेद और हरे तीर mCherry-PomX समूहों से पहले और बंटवारे की घटनाओं के बाद क्रमशः इंगित करते हैं । संख्या घंटे में समय का संकेत है । स्केल बार: 5 µm. (ख) छवियों में (क) के रूप में प्राप्त कर रहे थे और उच्च लौकिक संकल्प पर दिखाए जाते हैं । तीर के रूप में कर रहे है (एक) । स्केल बार: 5 µm. कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण देखने के लिए ।
| बैक्टीरियल तनाव | प्रासंगिक जीनोटाइप1 | संदर्भ |
| DK1622 | Wildtype | 23 |
| SA4420 | ΔmglA | 24 |
| SA4749 | ΔmglA; parB+/attB::PnatparB-yfp (pAH7) | इस अध्ययन |
| SA4797 | ΔmglA; ΔpomX/attB::PpomZ mCherry-pomX (pAH53) | 16 |
| SA8241 | ΔmglA; ftsZ+/mxan18-19::पणतftsZ-gfp (pDS150) | इस अध्ययन |
| कोष्ठक में Plasmids संकेत जीन फ्यूजन होते है और जीनोम पर संकेत साइटों पर थे । Plasmids attB स्थल पर एकीकृत या mxan18-19 intergenic क्षेत्र से व्यक्त की गई उनके मूल प्रवर्तक (पीnat) या pomZ (ppomZ) के मूल प्रवर्तक । |
||
तालिका 1: इस अध्ययन में प्रयुक्त बैक्टीरियल उपभेदों की सूची ।
| Plasmids | प्रासंगिक विशेषताओं | संदर्भ |
| pAH7 | PणतparB-yfp; Mx8 attP; Tetआर | 19 |
| pAH53 | PpomZ mCherry-pomX; Mx8 attP ; किमीआर | 16 |
| pDS150 १ | PणतftsZ-gfp ; mxan18-19 ; Tetआर | इस अध्ययन |
| pMR3691 | vanillate inducible जीन अभिव्यक्ति के लिए प्लाज्मिड | 18 |
| pKA51 | PणतftsZ-gfp ; Mx8 attP; Tetआर | 17 |
| 1 pDS150: pDS150 pKA51 के एक व्युत्पंन है जिसमें Mx8 attP साइट mxan18-19 intergenic क्षेत्र के साथ प्रतिस्थापित किया गया था । इसके लिए mxan18-19 intergenic क्षेत्र को प्राइमरियों के साथ pMR3691 से प्रवर्धित किया गया mxan18-19 fwd BsdRI (GCGATCATTGCGCGCCAGACGATAACAGGC) और Mxan18-19 rev BlpI (GCGGCTGAGCCCGCGCCGACAACCGCAACC) और pKA51 में क्लोन । |
||
तालिका 2: इस अध्ययन में प्रयुक्त plasmids की सूची ।
Discussion
प्रतिदीप्ति लाइव सेल इमेजिंग बैक्टीरियल कोशिकाओं के spatiotemporal गतिशीलता का अध्ययन करने के लिए एक शक्तिशाली उपकरण बन गया है । इस तरह के एम xanthusके रूप में गतिशील और धीमी गति से बढ़ते बैक्टीरिया की समय चूक प्रतिदीप्ति माइक्रोस्कोपी, हालांकि, चुनौतीपूर्ण है और केवल कम समय अवधि के लिए प्रदर्शन किया गया था । यहाँ, हम समय-चूक प्रतिदीप्ति माइक्रोस्कोपी द्वारा एम. xanthus के लाइव सेल इमेजिंग के लिए एक आसान करने के लिए उपयोग और मजबूत विधि प्रस्तुत करते हैं । इस विधि की कोशिकाओं का पालन करने के लिए उपयोगकर्ता की अनुमति देता है और फ्लोरोसेंट एक सेल संकल्प के साथ सेल चक्र के कई दौर के लिए प्रोटीन लेबल ।
वहां कई पूर्वापेक्षाएं है कि धीमी गति से बढ़ते एम. xanthus कोशिकाओं के लाइव सेल इमेजिंग की सफलता को प्रभावित कर रहे है सहित: 1) सेल लगाव के लिए एक ठोस सतह; 2) पोषक तत्वों और ऑक्सीजन की उपलब्धता; 3) लगातार आर्द्रता और तापमान; और 4) ऐसे जोखिम समय और छवि अधिग्रहण आवृत्ति के रूप में प्रयोगात्मक शर्तों का अनुकूलन ।
हमारे प्रयोगात्मक सेट अप में, हम मोटी agarose पोषक तत्वों के साथ पूरक पैड का उपयोग करें । एकल कोशिकाओं का पालन करने के लिए microfluidic उपकरणों के विरोध के रूप में मोटी agarose पैड का उपयोग कुछ मौलिक लाभ है, लेकिन यह भी कुछ कमियां हैं । सबसे पहले, agarose पैड न केवल M. xanthus सेल लगाव और आंदोलन के लिए एक सतह प्रदान करता है, लेकिन यह भी विकास के लिए पर्याप्त पोषक तत्वों से कम 24 ज । दूसरा, तस्वीर शॉट विश्लेषण आमतौर पर फ्लोरोसेंट लेबल प्रोटीन के intracellular स्थानीयकरण का अध्ययन करने के लिए इस्तेमाल किया पहले agarose पैड16,17,29के एक ही प्रकार पर किया गया था । इसलिए, स्नैप शॉट विश्लेषण से डेटा सीधे यहां वर्णित विधि के साथ प्राप्त आंकड़ों की तुलना में किया जा सकता है । तीसरे, agarose पैड आसानी से संशोधित किया जा सकता है और एंटीबायोटिक दवाओं या ऐसे CuSO4 और vanillate है कि आमतौर पर जीन अभिव्यक्ति प्रेरण18,30के लिए इस्तेमाल कर रहे है के रूप में अंय की खुराक के साथ पूरक । अंत में, क्योंकि कोशिकाओं को एक प्रयोग के दौरान microcolonies फार्म की अनुमति दी जाती है, इस सेट अप भी विशेष पैरामीटर पर सीधे सेल-सेल बातचीत के प्रभाव का अध्ययन करने की अनुमति देता है विश्लेषण किया जा रहा है । यह पहलू xanthus के मामले में विशेष रूप से महत्वपूर्ण है क्योंकि यह जीवाणु कई संपर्क-निर्भर इंटरैक्शन प्रदर्शित करता है. इस विधि का मुख्य दोष यह है कि प्रयोगात्मक शर्तों के एक प्रयोग की अवधि के लिए पूर्व निर्धारित कर रहे हैं । इसके विपरीत, microfluidic उपकरणों आम तौर पर उदाहरण एंटीबायोटिक दवाओं के लिए जोड़कर एक प्रयोग के पाठ्यक्रम के दौरान प्रयोगात्मक शर्तों को बदलने की अनुमति31।
मुफ्त सॉफ्टवेयर संकुल (जैसे, MicrobeJ, Oufti) स्वचालित रूप से एकल कोशिकाओं और व्यक्तिगत कोशिकाओं के भीतर प्रोटीन स्थानीयकरण के विकास का विश्लेषण करने के लिए उपलब्ध हैं । हालांकि, इन सॉफ्टवेयर केवल एकल कोशिकाओं या कोशिकाओं के छोटे समूहों के विश्लेषण के लिए अच्छी तरह से अनुकूल हैं । इस प्रकार, यह स्वचालित रूप से 24 घंटे यहां वर्णित रिकॉर्डिंग के लिए उत्पंन डेटा का विश्लेषण करने के लिए एक चुनौती बनी हुई है ।
संक्षेप में, हम एक आसान उपयोग करने के लिए और reproducible प्रोटोकॉल का वर्णन करने के लिए जी धीमी गति से बढ़ते एम. xanthus बैक्टीरिया के साथ सेल इमेजिंग प्रदर्शन । हम बताते है कि सरल पोषक तत्व-पूरक agarose पैड को कम से 24 घंटे के लिए विकास को बनाए रखने और देख और कई पीढ़ियों से अधिक एकल सेल संकल्प के साथ प्रोटीन स्थानीयकरण और विकास का विश्लेषण करने के लिए अनुमति देने के लिए पर्याप्त हैं ।
Disclosures
लेखकों की घोषणा है कि वे कोई प्रतिस्पर्धा वित्तीय हितों की है ।
Acknowledgments
यह काम जर्मन अनुसंधान परिषद (DFG) Transregio १७४ "बैक्टीरियल कोशिकाओं के Spatiotemporal गतिशीलता" और मैक्स प्लैंक सोसायटी के ढांचे के भीतर द्वारा समर्थित किया गया था ।
Materials
| Name | Company | Catalog Number | Comments |
| DMI6000B with AFC | Leica microsystems | 11888945 | Automated inverted widefield fluorescence microscope with adaptive focus control |
| Universal mounting frame | Leica microsystems | 11532338 | Stage holder for different sample sizes |
| HCX PL FLUOTAR 100x/1.30 oil PH3 | Leica microsystems | 11506197 | Phase contrast objective |
| Orca Flash 4.0 camera | Hamamatsu | 11532952 | 4.0 megapixel sCMOS camera for picture aquisition |
| Filter set TXR ET, k | Leica microsystems | 11504170 | Fluorescence filter set, Ex: 560/40 Em: 645/75 |
| Filter set L5 ET, k | Leica microsystems | 11504166 | Fluorescence filter set, Ex: 480/40 Em: 527/30 |
| Filter set YFP ET, k | Leica microsystems | 11504165 | Fluorescence filter set, Ex: 500/20 Em: 535/30 |
| ProScan III | Prior | H117N1, V31XYZEF, PS3J100 | Microscope automation controller with interactive control center |
| EL 6000 light source | Leica microsystems | 11504115 | External fluorescence light source |
| Incubator BLX Black | Pecon | 11532830 | Black incubation chamber surrounding the microscope |
| Tempcontrol 37-2 digital | Leica microsystems | 11521719 | Automated temperature control for incubation chamber |
| Gentmycin sulphate | Carl Roth | 0233.4 | Gentamycin |
| Oxytetracylin dihydrate | Sigma Aldrich | 201-212-8 | Oxytetracyclin |
| Kanamycin sulphate | Carl Roth | T832.3 | Kanamycin |
| Filtropur BT25 0.2 bottle top filter | Sarstedt | 831,822,101 | Bottle top filter for sterilization of buffers |
| Deckgläser | VWR | 630-1592 | Glass cover slip (60 x 22 mm, thickness: 0.7 mm) |
| Seakem LE agarose | Lonza | 50004 | Agarose for microscopy slides |
| Leica Metamorph AF | Leica microsystems | 11640901 | Microscope control software and software for picture analysis |
| Tetraspeck Microsperes, 0.5 µm | ThermoFisher | T7281 | Fluorescent microspheres |
| petri dish | Greiner Bio-one | 688102 | 120 mm x 120 mm x 17 mm squared petri dish for agarose pads |
| BD Bacto Casitone | Becton Dickinson | 225930 | Casitone |
| Parafilm M | VWR | 291-1213 | Parafilm |
| Tris(hydroxymethyl)-aminomethane | Carl Roth | AE15.2 | Tris |
| Magnesium sulphate heptahydrate | Carl Roth | P027.2 | Magnesium sulphate |
| Potassium dihydrogen phosphate p.a. | Carl Roth | 3904.1 | Potassium dihydrogen phosphate |
| 1% CTT medium: 1 % (w/v) BD Bacto™ casitone, 10 mM Tris-HCl ph 8.0, 1 mM potassium phosphate buffer pH 7.6, 8 mM MgSO4 | Cultivation medium for M.xanthus | ||
| TPM buffer: 10 mM Tris-HCl ph 8.0, 1 mM potassium phosphate buffer pH 7.6, 8 mM MgSO4 | Buffer for preparation of microscopy slides for M.xanthus |
References
- Shapiro, L., McAdams, H. H., Losick, R. Why and how bacteria localize proteins. Science. 326 (5957), 1225-1228 (2009).
- Treuner-Lange, A., Søgaard-Andersen, L. Regulation of cell polarity in bacteria. J Cell Biol. 206 (1), 7-17 (2014).
- Laloux, G., Jacobs-Wagner, C. Spatiotemporal control of PopZ localization through cell cycle-coupled multimerization. J Cell Biol. 201, 827-841 (2013).
- Rudner, D. Z., Losick, R. Protein subcellular localization in bacteria. Cold Spring Harb. Perspect. Biol. 2 (4), 000307 (2010).
- Badrinarayanan, A., Le, T. B. K., Laub, M. T. Bacterial chromosome organization and segregation. Annu Rev Cell Dev Biol. 31 (1), 171-199 (2015).
- Munoz-Dorado, J., Marcos-Torres, F. J., Garcia-Bravo, E., Moraleda-Munoz, A., Perez, J. Myxobacteria: Moving, Killing, Feeding, and Surviving Together. Front Microbiol. 7, 781 (2016).
- Berleman, J. E., Kirby, J. R. Deciphering the hunting strategy of a bacterial wolfpack. FEMS Microbiol Rev. 33 (5), 942-957 (2009).
- Konovalova, A., Petters, T., Søgaard-Andersen, L. Extracellular biology of Myxococcus xanthus. FEMS Microbiol. Rev. 34, 89-106 (2010).
- Nudleman, E., Wall, D., Kaiser, D. Cell-to-cell transfer of bacterial outer membrane lipoproteins. Science. 309, 125-127 (2005).
- Vassallo, C. N., et al. Infectious polymorphic toxins delivered by outer membrane exchange discriminate kin in myxobacteria. eLife. 6, 29397 (2017).
- Vassallo, C., et al. Cell rejuvenation and social behaviors promoted by LPS exchange in myxobacteria. Proc Natl Acad Sci USA. 112 (22), 2939-2946 (2015).
- Li, Y., et al. Extracellular polysaccharides mediate pilus retraction during social motility of Myxococcus xanthus. Proc. Natl. Acad. Sci. USA. 100, 5443-5448 (2003).
- Kim, S. K., Kaiser, D. Cell alignment required in differentiation of Myxococcus xanthus. Science. 249, 926-928 (1990).
- Lobedanz, S., Søgaard-Andersen, L. Identification of the C-signal, a contact dependent morphogen coordinating multiple developmental responses in Myxococcus xanthus. Genes Dev. 17, 2151-2161 (2003).
- Schumacher, D., Søgaard-Andersen, L. Regulation of cell polarity in motility and cell division in Myxococcus xanthus. Annu Rev Microbiol. 71 (1), 61-78 (2017).
- Schumacher, D., et al. The PomXYZ proteins self-organize on the bacterial nucleoid to stimulate cell division. Dev Cell. 41 (3), 299-314 (2017).
- Treuner-Lange, A., et al. PomZ, a ParA-like protein, regulates Z-ring formation and cell division in Myxococcus xanthus. Mol Microbiol. 87 (2), 235-253 (2013).
- Iniesta, A. A., Garcia-Heras, F., Abellon-Ruiz, J., Gallego-Garcia, A., Elias-Arnanz, M. Two systems for conditional gene expression in Myxococcus xanthus inducible by isopropyl-beta-D-thiogalactopyranoside or vanillate. J Bacteriol. 194 (21), 5875-5885 (2012).
- Harms, A., Treuner-Lange, A., Schumacher, D., Søgaard-Andersen, L. Tracking of chromosome and replisome dynamics in Myxococcus xanthus. reveals a novel chromosome arrangement. PLoS Genet. 9 (9), 1003802 (2013).
- Iniesta, A. A. ParABS system in chromosome partitioning in the bacterium Myxococcus xanthus. PLoS One. 9 (1), 86897 (2014).
- Lin, L., Osorio Valeriano, M., Harms, A., Søgaard-Andersen, L., Thanbichler, M. Bactofilin-mediated organization of the ParABS chromosome segregation system in Myxococcus xanthus. Nat Commun. 8 (1), 1817 (2017).
- Hodgkin, J., Kaiser, D. Cell-to-cell stimulation of movement in nonmotile mutants of Myxococcus. Proc Natl Acad Sci U S A. 74 (7), 2938-2942 (1977).
- Kaiser, D. Social gliding is correlated with the presence of pili in Myxococcus xanthus. Proc Natl Acad Sci USA. 76 (11), 5952-5956 (1979).
- Miertzschke, M., et al. Structural analysis of the Ras-like G protein MglA and its cognate GAP MglB and implications for bacterial polarity. EMBO J. 30 (20), 4185-4197 (2011).
- Hodgkin, J., Kaiser, D. Genetics of gliding motility in Myxococcus xanthus. (Myxobacterales): Two gene systems control movement. Mol Gen Genet. 171, 177-191 (1979).
- Shi, X., et al. Bioinformatics and experimental analysis of proteins of two-component systems in Myxococcus xanthus. J Bacteriol. 190 (2), 613-624 (2008).
- Bi, E. F., Lutkenhaus, J. FtsZ ring structure associated with division in Escherichia coli. Nature. 354 (6349), 161-164 (1991).
- Lutkenhaus, J., Pichoff, S., Du, S. Bacterial cytokinesis: From Z ring to divisome. Cytoskeleton. 69 (10), 778-790 (2012).
- McLoon, A. L., et al. MglC, a Paralog of Myxococcus xanthus GTPase-Activating Protein MglB, Plays a Divergent Role in Motility Regulation. J Bacteriol. 198 (3), 510-520 (2015).
- Gomez-Santos, N., et al. Comprehensive set of integrative plasmid vectors for copper-inducible gene expression in Myxococcus xanthus. Appl Environ Microbiol. 78 (8), 2515-2521 (2012).
- Treuner-Lange, A., et al. The small G-protein MglA connects to the MreB actin cytoskeleton at bacterial focal adhesions. J Cell Biol. 210 (2), 243-256 (2015).



