Summary
Bakterieceller anordnas rumsligt mycket. För att följa denna organisation över tid i långsam växande Myxococcus xanthus celler, utvecklades en set-up för fluorescens live-cell avbildning med hög spatiotemporal upplösning över flera generationer. Med den här metoden kunde spatiotemporal dynamics av viktiga proteiner för kromosomsegregering och celldelning bestämmas.
Abstract
Fluorescens live-cell avbildning av bakterieceller är en viktig metod i analysen av den rumsliga och tidsmässiga dynamiken av proteiner och kromosomer underliggande centrala cellcykeln händelser. Dock avbildning av dessa molekyler i långsamt växande bakterier representerar en utmaning på grund av fotoblekning fluorophores och fototoxicitet under bild förvärv. Här beskriver vi ett enkelt protokoll för att kringgå dessa begränsningar när det gäller Myxococcus xanthus (som har en generationstid på 4-6 h). I detta syfte M. xanthus celler odlas på en tjock som innehåller näringsämne agar pad i en temperatur-kontrollerad fuktiga miljö. Under dessa förhållanden bestämma vi fördubbling tid av enskilda celler genom att följa tillväxten av enstaka celler. Dessutom processer viktiga cellulära som kromosomsegregering och celldelning kan avbildas av fluorescens live-cell avbildning av celler som innehåller relevanta fluorescently märkt markör proteiner såsom ParB-YFP, FtsZ-GFP och mCherry-PomX över flera cell cykler. Därefter bearbetas förvärvade bilderna för att generera montage eller filmer.
Introduction
Bakterieceller organiseras rumsligt mycket med många proteiner lokalisera asymmetriskt inom cellulär fack1,2,3,4. Denna lokalisering är ofta mycket dynamiska och förändras över tid som svar på cellcykelns ledtrådar eller externa signaler. Lika, bakteriell kromosomen är rumsligt mycket organiserad med enskilda loci placeras till specifika subcellulär platser före och under den segregation process5. Denna dynamiska rumslig organisation är viktig för tillväxt, division, cellcykelns reglering, differentiering, motilitet, signaltransduktion samt kromosom organisation och segregation; det påverkar således, i princip alla aspekter av bakterie funktion.
Spatiotemporal dynamiken i dessa cellulära processer analyseras i en mängd olika bakteriearter med Escherichia coli, Bacillus subtilis, Vibriocholeraeoch Caulobacter crescentus som serverar lika viktigt modellorganismer. Men dessa fyra arter täcker bara ett litet spektrum av en enorm bakteriell mångfald och kanske föga förvånande gett stora fylogenetiska avståndet mellan dessa arter, cellulära mekanismer som organisation och polarisering är olika i dessa bakterier. Detta aktualiserar behovet för att studera ytterligare bakteriearter ska kunna så småningom extrahera de allmänna principerna spatiotemporal dynamiken i bakterieceller.
Den gramnegativa delta-proteobacterium M. xanthus modellorganism i studiet av sociala beteenden och samarbete i bakterier6. M. xanthus är en strikt aerobe och i närvaro av näringsämnen, det bildar kolonier där celler spridit sig utåt i en starkt samordnad, myllrande mode och bytesorganismer på andra mikroorganismer7. Svar på näringsämnen svält, celler inleda ett utvecklande program som leder till bildandet av fruktkroppar som består av tusentals celler, och inuti som stavformade motila celler differentiera till sfärisk diploida sporer8. Båda typer av beteenden, dvs, svärma och fruiting kropp bildandet, utförs endast av celler som placeras på en fast yta. Dessutom både näringsämnen villkor celler engagera sig i processer som inbegriper direkt cell-cell kontakter inklusive utbyte av yttre membran lipoproteiner som kan stimulera motilitet eller fungera som gifter i mottagarens9,10 , utbyte av LPS11, stimulering av motilitet genom exopolysaccharides på angränsande celler12och intercellulära signalering av en cell surface-förankrade signalering protein13,14.
Nyligen, M. xanthus har också blivit en modellorganism för att studera mekanismerna bakom motilitet och dess förordning15, celldelning16,17,18och kromosom organisation19 ,20,21. Kritiska steg i den M. xanthus cellcykeln har analyserats i detalj av fluorescensmikroskopi använder snap-shot bilder eller korta time-lapse inspelningar på stammar som transporterar relevanta fluorescently märkta proteiner16, 17,18,19,20. Många celler bör helst följas med encelliga upplösning av fluorescens levande cell imaging för minst en full cellcykeln att få robust kvantitativa uppgifter om cellcykelns parametrar. Detta är dock en utmaning för M. xanthus på grund av dess relativt lång tid 4-6 h under standard laboratorieförhållanden och fotoblekning fluorophores och fototoxicitet under bild förvärv.
Här beskriver vi ett protokoll att följa M. xanthus celler med enstaka cell upplösning av fluorescens live-cell imaging under minst 24 h och täcker flera cell cykler. Allt under hela protokollet, celler upprätthålls på en agar-pad och i nära kontakt möjliggör kontakt-beroende aktiviteter viktigt för den sociala livsstilen i M. xanthus. Protokollet också tillåt förbrukaren till ordningsmanen form, storlek, divisioner och fluorescerande sonder med hög temporal upplösning och med enstaka cell upplösning, och således möjliggör kvantifiering av cell-till-cell variabilitet och korrelationer av cellcykeln händelser.
Protocol
1. beredning och tillväxt av M. xanthus stammar
Obs: Se tabell 1 och tabell 2.
- Förbereda 1% casitone buljong (CTT) tillväxt medium 1% (w/v) bukspottskörteln digest kasein (t.ex., Bacto casitone), 10 mM Tris-HCl pH 8,0, 1 mM KH2PO4 pH 7,6, 8 mM MgSO422, kompletterat med kanamycin (50 µg/mL) eller oxytetracyklin (10 µg/mL). Lägg till gentamycin (10 µg/mL) till alla media för att minska risken för kontaminering med andra bakterier, sedan M. xanthus celler är naturligt resistenta mot den.
- Inokulera 5 mL 1% CTT som innehåller de relevanta antibiotic(s) med en enda färska odlade koloni av wild typ (WT) DK1622 23, SA4420 (ΔmglA)24, SA4797 (ΔmglA, ΔpomX/PpomZ mCherry-pomX )16, SA8241 (ΔmglA, ftsZ+/PnatftsZ-gfp), eller SA4749 (ΔmglA, parB+/PnatparB-yfp) i den morgon dag 1.
- Återsuspendera en enda M. xanthus koloni i 500 µL av 1% CTT kompletteras med antibiotika i ett sterilt rör och överför hela suspensionen till en 50 mL Erlenmeyerkolv, innehållande 5 mL 1% CTT.
Obs: Använd en Erlenmeyerkolv med 10 gånger volymen av kultur för att garantera tillräcklig aeriation och optimal tillväxt.
- Återsuspendera en enda M. xanthus koloni i 500 µL av 1% CTT kompletteras med antibiotika i ett sterilt rör och överför hela suspensionen till en 50 mL Erlenmeyerkolv, innehållande 5 mL 1% CTT.
- Odla cellerna åtta generationer (cirka 40-48 h med en generationstid på 4-6 h) vid 32 ° C, skaka på 220 rpm, i mörkret. Upprätthålla celler i exponentiella tillväxtfasen (OD550 < 1.2) och hindra dem från att nå den stationära fasen. Vid behov späd cellerna till färska 1% CTT medium som innehåller den relevanta antibiotic(s) till en OD550 av 0,1 - 0,2.
Obs: En optimal OD550 för en enda cell mikroskopi är 0,5 - 0,7. Vid denna OD550är ett tillräckligt antal celler närvarande per bild att möjliggöra kvantifiering samt statistisk analys av cellulära parametrar.
2. beredning av mikroskopi prover
Obs: Celler att ses av mikroskopi placeras på ett Mikroskop täckglas och sedan täckas av en agaros pad som innehåller näringsämnen. Täckglaset är limmade till en plast eller metall ram att ge mekanisk stöd. Förberedelse för microscopyen, en stor pad av 1% agarose/TPM/0.2% CTT bör förberedas i förväg enligt beskrivningen i steg 2.1-2.3. Se också Tabell av material för specifika produkter används här.
- Bereda 500 mL TPM-buffert (10 mM Tris-HCl pH 7,6, 1 mM KH2PO4 pH 7,6, 8 mM MgSO4) och autoklav eller filter sterilisera använder en flaska övre filter.
Obs: Det steril buffert kan lagras i flera månader i rumstemperatur. - Bered 1% agaros mikroskopi lösning som innehåller 0,2% CTT (mix 1 g av agaros med 80 mL TPM buffert och 20 mL 1% CTT medium). Värm i mikrovågsugn tills Agarens är smält.
Obs: 0,2% CTT är tillräcklig för att celler att växa och förhindra svält. Högre koncentrationer av CTT mikroskopi medium kommer att resultera i hög bakgrund fluorescens. - Fyll en petriskål med den smälta agarosen till en tjocklek av 0,5 cm (för en 11,5 x 11,5 cm fyrkant petriskål, cirka 60 mL smälta agarosen krävs) och låt den svalna ner till rumstemperatur.
Obs: Agaros pad kan lagras vid 4 ° C i en fuktig miljö för upp till 2 dagar.- Pre varma 1% agarose/TPM/0.2% CTT pad vid 32 ° C i minst 15 minuter före användning.
Obs: För att förbereda cellerna för mikroskopi, Följ steg 2,4-2,8.
- Pre varma 1% agarose/TPM/0.2% CTT pad vid 32 ° C i minst 15 minuter före användning.
- Placera en steril täckglas (60 mm x 22 mm, tjocklek: 0,7 mm) på en plast eller metall ram som har ett hål i mitten (figur 1A). denna ram fungerar som ett mekaniskt stöd för det tunna täckglaset och hjälper till att minska drift vid mikroskopi. Fixa täckglaset till ramen med tejp.
- Att förbereda ramen, klipp ut en 75 mm × 25 mm ram från en 1 mm tjock metallplatta och sedan klippa ut en lagom stora hål (20 mm × 30 mm i detta experiment) i mitten.
- Tillsätt 10-20 µL av exponentiellt odlade M. xanthus celler på täckglaset.
- Lägga till fluorescerande 0,5 µm mikrosfärer som relaterat markörer i cellerna för att förenkla spårningen av celler eller proteiner i time-lapse inspelningar.
- Späd den mikrosfärer 1: 100 i TPM buffert och förvaras vid 4 ° C i upp till flera månader. Skaka ordentligt innan användning och tillsätt 5-10 µL utspädda mikrosfärerna i cellerna.
Obs: Här mikrosfärer som är fluorescerande i alla vanliga blå, grön, gul och röd fluorescerande kanaler användes.
- Späd den mikrosfärer 1: 100 i TPM buffert och förvaras vid 4 ° C i upp till flera månader. Skaka ordentligt innan användning och tillsätt 5-10 µL utspädda mikrosfärerna i cellerna.
- Klipp ut en liten pad ungefär storleken av täckglaset av stora pre värmde 1% agarose/TPM/0.2% CTT pad och placera den ovanpå cellerna (figur 1B). Placera ett täckglas ovanpå 1% agarose/TPM/0.2% CTT agaros pad att förhindra avdunstning och upprätthålla celler i en fuktig miljö.
Obs: Det ensam täckglaset förhindrar betydande avdunstning för minst 2 h. För längre time-lapse inspelningar, ska 1% agarose/TPM/0.2% CTT pad och täckglas smörgåsen tätas med paraffin film att förhindra avdunstning. - Inkubera mikroskopi provet vid 32 ° C i 15-20 min att låta cellerna fäster till botten av agaros pad. Starta sedan time-lapse mikroskopi inspelningarna.
3. Mikroskop Set-up och Time-lapse förvärv
Obs: Det protokoll som beskrivs här utvecklades för en inverterad widefield Mikroskop med autofokus, en 100 X / 1,30 NA olja PH3 mål, ett X, Y motoriserade scenen, en sCMOS kamera, en ljuskälla, filter för grön fluorescerande, röd fluorescerande eller gul fluorescerande proteiner, och en temperatur kontrollerade inkubation kammare. Denna kammare håller celler skyddas från ljus och vid konstant temperatur.
- Förvärm inkubation kammaren och mikroskopet till 32 ° C för ~ 1-2 h innan mikroskopi.
Obs: Beroende på mikroskopet set-up, värme kan ta längre tid. Förvärmning är viktigt att minska drift och stabiliserar autofokus kontrollsystemet. - Slå på mikroskopet och starta programvaran Mikroskop kontroll. Välj det rätta målet och korrekt speglar och filter att förvärva fas kontrast bilder samt bilder av grönt fluorescerande, röd fluorescerande eller gul fluorescerande proteiner.
Obs: Mikroskop levereras normalt med en rekommenderad programvara för Mikroskop kontroll och bild förvärv. Här användes en inköpt programvara (se Tabell för material) för att styra mikroskopet och bild förvärv. - Tillsätt en droppe olja för nedsänkning av hög kvalitet på objektivet av målet och till botten av provet före inkuberas vid 32 ° C. Placera målet vid lägsta möjliga Z-position att undvika att skada objektivet när provet placeras på Mikroskop scenen. Placera den metall-ram med provet på Mikroskop scenen och med ”hål-sidan” mot målet. Fäst provet ordentligt i hållaren scenen.
- Fokusera på cellerna genom att flytta scenen i Z-riktningen närmare målet. Flytta stadiet långsammare när oljan droppar på prov undersidan och objektivet gör kontakt. Förflytta bordet i X / Y riktning tills flera enstaka celler är synliga i regionen i vyn när cellerna är i fokalplanet. Kontrollera att minst en fluorescerande microsphere är i regionen av vy för att senare anpassa förvärvade bilderna.
Obs: Under optimala förhållanden, en cell densiteten av 15-30 celler per regionen view (2 048 x 2 048 pixlar eller 133.1 x 133.1 µm) bör nås. - Öppna guiden Multi-Dimensional förvärv av programvaran Mikroskop kontroll att ställa in en tid förflutit experiment som tillåter mikroskopet att förvärva bilder på flera våglängder och scenen positioner vid behov.
- I Main fliken aktivera Timelapse och Flera våglängder. Ytterligare flikar visas på vänster sida av fönstret.
- Klicka på fliken Spara och Välj katalog att välja en tom mapp på datorhårddisk att spara förvärvade bilderna. Aktivera Increment basnamn om filen finns kontrollerar att på varandra följande datamängder inte skriva över tidigare. Sedan ge experimentet ett namn med datum och stam namn eller titel av experimentet.
- Klicka på fliken Timelapse att justera time-lapse parametrar. Ange varaktighet för 24 h och ange Tidsintervallet till 20 min. De Nummer av tidpunkter ändras automatiskt.
Obs: Det optimala tidsintervallet beror på experimentet och cellulär funktion ska analyseras. Frekventa bild förvärv kan orsaka fotoblekning. Således måste en avvägning mellan temporal upplösning och fotoblekning hittas empiriskt. På en fördubbling tid av 4-6 h, kan bilder enkelt förvärvas med ett intervall på 5 min (eller ännu mindre intervall om så önskas) för fas kontrast mikroskopi. Om fluorescensmikroskopi över en tid-loppet av 24 h önskas bilder bör registreras i ett intervall på cirka 15-30 min. - Klicka på fliken våglängder Välj antal våglängder att förvärva för varje bild som vid varje tidpunkt genom att ändra värdet.
Obs: för varje våglängd, visas en ny flik på vänster sida av den Multi-Dimensional förvärv ” guiden och våglängder kommer att förvärvas i ordning från topp till botten. För varje våglängd, kan förvärv inställningarna ändras separat. - Klicka på fliken första våglängd från toppen. Välj fas kontrast i belysning nedrullningsbara listan. Välj 100 ms för exponering och Varje tidpunkt i förvärva nedrullningsbara listan. Avaktivera Auto exponera genom att välja aldrig i den nedrullningsbara listan.
- Upprepa steg 3.5.5 för varje våglängd som måste förvärvas vid varje tidpunkt. För experimental set-up och fluorescently märkta proteiner beskrivs här, använda följande parametrar för exponering: 250 ms för mCherry fusionsproteinerna, 200 ms för YFP fusionsproteinerna och 1000 ms för god Jordbrukarsed fusionsproteinerna.
Obs: Optimal belysning inställningarna för varje stam och fluorescerande protein bör fastställas i förväg genom att ändra lampans intensitet och bild förvärv tid för varje våglängd. Gånger för lång bild förvärv kommer att öka effekten fototoxisk och i slutändan leda till tillväxt gripandet och cell död. Därför bör en avvägning mellan bild kvalitet och cell lönsamhet uppnås. - Hämta bilder från flera scenen positioner att öka antalet celler registreras i samma experiment.
- För att förvärva bilder från flera scenen positioner, aktivera Flera scenen positioner i huvudfliken . Klicka sedan på fliken scenen och klicka på knappen Live att titta på synfältet.
- Förflytta bordet i X/Y-riktningen tills en region av intresse (ROI) är i synfältet. Spara de X - och Y-koordinater genom att klicka flytta på ”+” scenen på fliken scenen igen i X/Y-riktning tills en ny ROI hittas och spara koordinaterna igen genom att klicka på ”+”. Gå på tills önskat antal regioner har sparats.
Obs: Vid fluorescens bild förvärvandet, se till att regionerna av intresse (ROIs) inte är för nära varandra för att minimera fototoxicitet.
- Kontrollera återigen att cellerna är i fokus genom att klicka på de olika sparade X - och Y-position och starta hårdvara autofokus genom AFC håll för att hålla den sparade Z-positionen konstant under loppet av experimentet.
- Starta time-lapse inspelningarna i Mikroskop kontroll programvaran genom att klicka på Skaffa i guiden Multi-Dimensional förvärv .
Obs: Ett fönster visas för varje våglängd som förvärvas och en ytterligare fönster visas som visar antalet förvärvade tidpunkter och tiden fram till nästa bild förvärv. - Kontrollera att cellerna är fortfarande i fokus efter de första några tidpunkter i time-lapse inspelningar att maximera kvaliteten på bilderna och fokusera om det behövs.
4. generering av Time-lapse filmer och Bildjustering
Obs: Flera kommersiella och gratis programvarupaket finns tillgängliga för bild förvärv och bildanalys. Vi använder en kommersiellt tillgänglig programvara (se Tabell för material) med flera förinstallerade plugins och ytterligare verktyg.
- Spara enskilda bilder från time-lapse inspelningar på en dator som har bild analys/bearbetning programvara installerad.
- Starta programmet och öppna bilder som stack genom att klicka Granska Multi-Dimensional Data | Välj bas fil | Välj katalog. Öppna mappen med flerdimensionella data. Kontrollera datamängden och klicka på Visa; datamängden kommer att visas som enstaka bilder från tid punkt ett fram till slutet. Aktivera våglängden (för att skapa en stack), markera alla bilder som ska vara i stacken och klicka Ladda bilderna. Upprepa detta för alla våglängder och spara ifyllda staplar.
- (Valfritt) Öppna alla bilder krävs för film med hjälp av fil | Öppna.
Obs: Det rekommenderas att öppna bilder av en förvärvad våglängd samtidigt för att inte sakta ner datorn om beräkningskraft är begränsad. Om vissa delar av time-lapse inspelningarna, t.ex., start, slut eller flera tidpunkter ska hoppas över, kan då detta justeras i den färdiga filmen. - Aktivera i en bunt med bilder som måste korrigeras för avdrift. Öppna verktyget för justering av appar | Automatiskt justera... kontrollera Stack som källa för bilder och första planet/tid pekar som referensplanet. Välj stacken med knappen Source stack och klicka på Verkställ.
Obs: Den automatiska justeringen kommer att ta lite tid och beräkningskraft men är ett bra sätt att korrigera stora stackar för drift av mikroskopet set-up. Denna automatiska justering fungerar bra om mikrosfärer ingår, men kan också fungera utan dem.. - Spara anpassade stacken.
- Använd ROIs.
Obs: Fluorescerande time-lapse mikroskopi enkelt skapar stora uppsättningar datafiler som tar upp en hel del datorkraft och nedströms handläggningen av dessa filmer. Vi rekommenderar därför starkt att identifiera ROIs och isolera cellerna att arbeta med mindre filer.- Välj verktyget Rektangulärt område . Skapa en ROI runt celler av intresse genom att manuellt rita en ROI på fas kontrast bilden. Se till att cellerna i intresse är synlig och i fokus genom hela hela time-lapse filmen.
- Öppna den time-lapse filmen av andra våglängden av samma datamängd. För att överföra ROI från fas kontrast bilderna till fluorescensen bilder av andra våglängden med verktyget Överföring regioner med regioner | Överföra regioner. Välj fas kontrast datamängden som Källbilden och andra våglängd datamängden som Målbilden. Alla regioner och trycker på OK.
- Upprepa steg 4.6.2 för varje våglängd som förvärvats för samma datamängd.
- Välj ROI och duplicera det som stack med Redigera | Duplicera | Stack... eller tryck på Skift + Ctrl + D nycklar. Spara sedan den duplicerade stacken med fil | Spara till samma mapp som den ursprungliga datan.
- Upprepa steg 4.6.4 för varje ROI för varje våglängd som förvärvats för samma datamängd
- Generera en film i MOV- eller AVI-format, öppna funktionen Skapa film via Stack | Göra film. Välj time-lapse inspelningarna med knappen Source Stack . Välj utdataformat, bildfrekvens, antalet ramar, och klicka på Spara.
Representative Results
M. xanthus är en långsamt växande bakterie som rör sig på fasta ytor. För att testa våra experimentella set-up, utfört vi en time-lapse experiment med rörliga DK1622 WT celler. Fas kontrast bilder förvärvades med 5 min intervall för 24 h (figur 2A, B). Flertalet celler linje i grupper. Som förväntat, celler visas motilitet och flyttade huvudsakligen i grupper. Vi konstaterade vidare att celler ibland vänds rörelseriktning. Dessa fynd tyder på att WT celler de testade villkor beter sig normalt när det gäller cell motilitet. Men även när celler registreras varje 5 min, är identifiering av enskilda celler svårt. Dessutom eftersom celler är rörliga, många celler fly eller ange synfältet vilket gör det svårt att följa celler under längre perioder.
För att spåra samma M. xanthus celler för flera omgångar av cellcykeln genom live-cell imaging, enskilda stammar kan tas bort för mglA genen, vilket är viktigt för motilitet25. Detta hindrar celler från att flytta ut ur synfältet under imaging protokollet. I-frame borttagningar genereras som beskrivs av Shi et al. 26
Som förväntat, i fas kontrast live-cell imaging med icke-rörliga ΔmglA celler (figur 3), framvisade celler inte aktiv rörelse. Vi har kunnat följa tillväxt och avdelningen för enskilda celler under microcolony bildande. Baserat på time-lapse inspelningarna där förvärvades bilder med 5 min intervall för 24 h, var det möjligt att kvantifiera den interdivision tid (tiden mellan två celldelning händelser) med enstaka cell upplösning. Celler ΔmglA muterade hade en mellan division tid 235 ± 50 min (n = 97 celler). Med ca 4 h är interdivision tiden liknande till fördubbling tid mätt i suspension kulturer för WT celler. Detta ger bevis för att M. xanthus celler växa optimalt under dessa försöksbetingelser.
För att undersöka om vår installation tillåter celler att växa normalt medan spårning YFP-märkt proteiner under långa perioder, vi utfört fluorescens time-lapse imaging med M. xanthus celler som uttrycker ett YFP-taggade protein. Därför följde vi ParB-YFP som markör för beskärningen av replikering (ori). ParB är som komponent i liknelsenS systemet i M. xanthus och binder till parS platser proximalt ori; ursprung dubbelarbete och kromosom segregation kan därför följt19,20,21. Med bild förvärv (faskontrast och fluorescens, 200 ms förvärv tiden i YFP kanal) varje 20 min, celler växte, uppdelad och visade tillväxt även vid 24 h (figur 4A). I början av inspelningarna bildade ParB-YFP två kluster i de subpolara områdena i flertalet celler (figur 4A). Strax före eller efter celldelning, den subpolara ParB-YFP kluster på den gamla cell Polen dupliceras. En av de två klustren återstod på den gamla cell Polen medan den andra kopian flyttad till den nya cellen pol, når sin slutliga subpolara position efter cirka 40-60 min (figur 4A, B). Dessa observationer är ense med tidigare data som genereras från kort time-lapse inspelningar med tunn agar pads19. Vi konstatera att denna experimentella set-up tillåter time-lapse fluorescensmikroskopi att spåra kromosomsegregering över flera cell cykler i långsamt växande M. xanthus celler, utan störande celltillväxt eller kromosom segregation maskiner.
I ett liknande experiment försökte vi följa markörer för celldelning av time-lapse fluorescensmikroskopi. Liknar nästan alla andra bakterier, M. xanthus kräver FtsZ, en bakteriell tubulin-liknande GTPase, för celldelning16,17,18. FtsZ bildar en ringliknande struktur på midcell, de så kallade Z-ring, som hjälper till att rekrytera alla andra proteiner som behövs för celldelningen27,28. I M. xanthus, bildandet av Z-ringen och dess placering på midcell stimuleras av de tre PomXYZ proteiner16,17. Dessa tre proteiner bildar en kromosom-associerade komplex som överför över nucleoid från platsen för celldelning i cellen ”mamma” till mitten av nucleoid i de två dottercellerna. I mitten av nucleoid sammanfaller med midcell, innan kromosomsegregering, och här PomXYZ komplexa rekryna FtsZ och stimulerar Z-ring bildas.
Här, följde vi första icke-motila celler som uttrycker ftsZ-gfp. Eftersom FtsZ-GFP övergripande visar en svagare fluorescens signal än ParB-YFP, ökade vi exponeringstiden 5 gånger till 1 s i GFP-kanal. Som förväntat, stark ackumulering av FtsZ-GFP observerades endast vid midcell och denna lokalisering dikterade positionen för celldelning sammandragning (figur 5A). FtsZ-GFP bildas huvudsakligen ett kluster på midcell i längre cell. Det var också uppenbart att detta kluster ökade i intensitet över tid. Efter celldelning observerade vi att FtsZ-GFP åter samlat på midcell i två dotter celler ca 2 h senare (figur 5B). Detta är förenligt med konstaterandet att cirka 50% av cellerna i en befolkning visar FtsZ lokalisering på midcell baserat på snap-shot analys16,17.
I ett andra experiment följde vi icke-rörliga ΔmglA celler för 24 h som uttrycker mCherry-pomX. Som en del av det PomXYZ systemet, PomX hjälper till att vägleda Z-ring bildandet och positionering, därmed stimulera celldelning midcell16. MCherry-PomX fluorescens signal är stark och tillåter en exponeringstid i kanalen fluorescens av 250 ms. ännu viktigare, alla celler växte i storlek och visas en celldelning händelse under loppet av experimentet, bildar microcolonies efter 24 h ( Figur 6A). Som tidigare rapporterats16innehöll nästan alla celler ett mCherry-PomX kluster. Flesta av dessa lokaliserade på midcell och kluster från midcell flyttad till midcell under experimentet. Under celldelningar delades mCherry-PomX kluster, med varje dotter cell får ett kluster. I motsats till FtsZ-GFP, mCherry-PomX lokaliserad till midcell 80-90% av cellcykeln och nådde denna ståndpunkt snart efter celldelning (figur 6B).
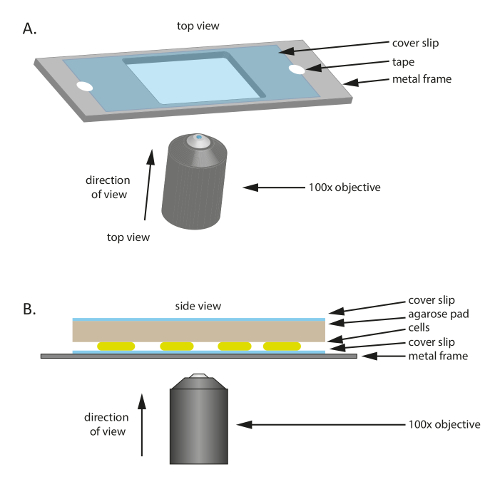
Figur 1 : Schematisk av den experimentella set-up som används i denna studie. (A) A metall eller plast ram fungerar som ett stöd för provet. Ett täckglas fixeras till metallramen med tejp för att minska rörelse av provet. (B) Side Visa av experimentella provet set-up. Celler är monterade på den täckglas visas i (A). Agaros pad som levererar näringsämnen och fukt till cellerna är placerad ovanpå cellerna. Agaros pad omfattas av en extra täckglas att minska avdunstning. För bilder av hög kvalitet används en 100 X fas kontrast oljeimmersionsobjektivet. Klicka här för att se en större version av denna siffra.

Figur 2 : Fas kontrast time-lapse mikroskopi av WT M. xanthus celler. Celler följdes under 24 h och bilder förvärvades varje 5 min. (A) representativa bilder av samma synfält var 5 minut visas. Färgade pilarna indikerar riktningen på rörlighet för enskilda celler. Samma färg markerar samma cell över tid. Siffrorna anger tiden i minuter. Skalstapeln: 5 µm. (B) bilder av samma synfältet efter varje timme visas. Observera att samma synfält visas men eftersom cellerna är rörliga, celler ständigt kommer in och lämnar synfältet. Siffrorna anger tiden i timmar. Skalstapeln: 5 µm. PH: fas kontrast. Klicka här för att se en större version av denna siffra.

Figur 3 : Fas kontrast time-lapse mikroskopi av icke-rörliga M. xanthus celler. ΔmglA celler följdes för 24 h. bilder förvärvades var 5 minut och representativa bilder efter varje timme visas. Valda celldelning sammandragningar är markerade med orange pilar. Siffrorna anger tiden i timmar. PH: faskontrast. Klicka här för att se en större version av denna siffra.
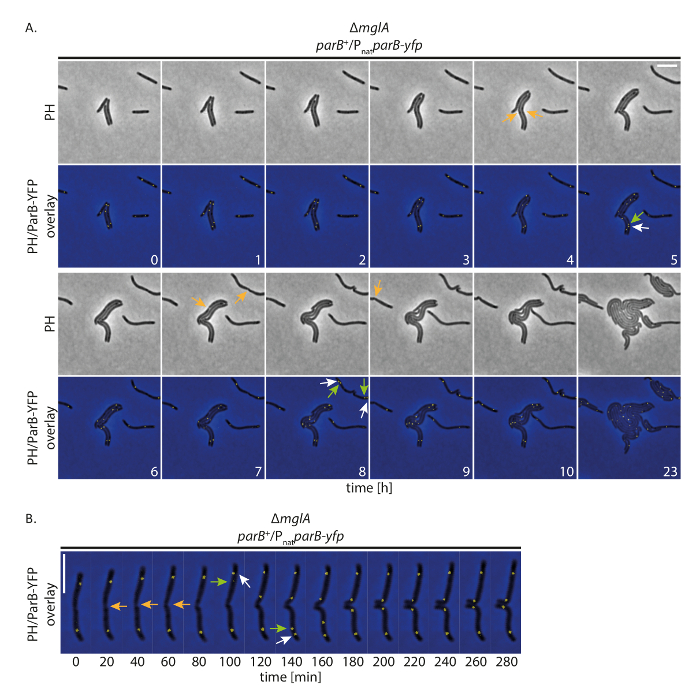
Figur 4 : Fluorescensmikroskopi vid time-lapse av ParB-YFP i icke-rörliga M. xanthus celler. Cellerna i en ΔmglA mutantuttrycker parB-yfp i närvaro av infödda parB (SA4749; ΔmglA; parB +/PnatparB-yfp) följdes under 24 h av fas kontrast och fluorescence mikroskopi. (A) bilder förvärvades varje 20 min och representativa bilder varje timme tills 10 h visas, tillsammans med samma celler efter 24 h. bilder visas i faskontrast (PH) samt som överlagring av faskontrast och YFP signal. Valda celldelningar markeras med orange pilar. Vita och gröna pilar indikerar ParB-YFP klusterhändelser dubbelarbete, med de gröna pilarna märkning translocating klustret. Siffrorna anger tiden i timmar. Skalstapeln: 5 µm. (B) bilder förvärvades som i (A) men visas på högre temporal upplösning. Siffrorna anger tiden i minuter. Pilarna är som i (A). Skalstapeln: 5 µm. vänligen klicka här för att visa en större version av denna siffra.

Figur 5 : Fluorescensmikroskopi vid time-lapse av FtsZ-GFP i icke-rörliga M. xanthus celler. Cellerna i en ΔmglA mutant uttrycker ftsZ-gfp i närvaro av infödda ftsZ (SA8241; ΔmglA; ftsZ +/PnatftsZ-gfp) följdes under 24 h av fas kontrast och fluorescence mikroskopi. (A) bilder förvärvades varje 20 min och representativa bilder varje timme tills 10 h visas, tillsammans med samma celler efter 24 h. bilder visas i faskontrast (PH) och som överlagring av faskontrast och god Jordbrukarsed signal. Valda celldelningar markeras med orange pilar. Vita pilar visar FtsZ-GFP-kluster på midcell. Siffrorna anger tiden i timmar. Skalstapeln: 5 µm. (B) bilder förvärvades som i (A) men visas på högre temporal upplösning. Siffrorna anger tiden i minuter. Gröna och vita pilar markerar FtsZ-GFP kluster i vänster och höger celler, respektive. Orange pilar indikerar celldelningar. Skalstapeln: 5 µm. vänligen klicka här för att visa en större version av denna siffra.

Figur 6 : Time-lapse fluorescensmikroskopi av mCherry-PomX i icke-rörliga M. xanthus celler. Icke-rörliga ΔpomX celler ackumulerar mCherry-PomX (SA4797; ΔmglA; ΔpomX/PpomZ mCherry-pomX) följdes under 24 h av fas kontrast och fluorescence mikroskopi varje 20 min. (A) representant bilder varje timme tills 10 h visas, tillsammans med samma celler efter 24 h. bilder visas i faskontrast (PH) och som överlagring av faskontrast och mCherry signal. Valda celldelningar markeras med orange pilar. Vita och gröna pilarna anger mCherry-PomX kluster före och efter dela händelser, respektive. Siffrorna anger tiden i timmar. Skalstapeln: 5 µm. (B) bilder förvärvades som i (A) och visas med högre temporal upplösning. Pilarna är som i (A). Skalstapeln: 5 µm. vänligen klicka här för att visa en större version av denna siffra.
| Bakteriestam | Relevanta genotyp1 | Referens |
| DK1622 | Vildtyp | 23 |
| SA4420 | ΔmglA | 24 |
| SA4749 | ΔmglA; parB+/attB:: PnatparB-yfp (pAH7) | Denna studie |
| SA4797 | ΔmglA; ΔpomX / attB::PpomZ mCherry-pomX (pAH53) | 16 |
| SA8241 | ΔmglA; ftsZ+/ mxan18-19::PnatftsZ-gfp (pDS150) | Denna studie |
| Plasmider inom parentes innehåller angivna genfusioner och var intergated på angivna platser på genomet. Plasmider är integrerade på attB platsen eller regionen mxan18-19 intergenic uttrycktes från deras infödda arrangören (Pnat) eller infödda arrangören av pomZ (PpomZ). |
||
Tabell 1: Lista över bakteriestammar som används i denna studie.
| Plasmider | Relevanta egenskaper | Referens |
| pAH7 | PnatparB-yfp; Mx8 oakstreet; TetR | 19 |
| pAH53 | PpomZ mCherry-pomX; Mx8 oakstreet ; KmR | 16 |
| pDS150 1 | PnatftsZ-gfp ; mxan18-19 ; TetR | Denna studie |
| pMR3691 | Plasmid för vanillate inducerbara genuttryck | 18 |
| pKA51 | PnatftsZ-gfp ; Mx8 oakstreet; TetR | 17 |
| 1 pDS150: pDS150 är ett derivat av pKA51 där webbplatsen Mx8 oakstreet ersattes med regionen mxan18-19 intergenic. För detta förstärktes regionen mxan18-19 intergenic från pMR3691 med grundfärg Mxan18-19 fwd BsdRI (GCGATCATTGCGCGCCAGACGATAACAGGC) och Mxan18-19 rev BlpI (GCGGCTGAGCCCGCGCCGACAACCGCAACC) och klonade in i pKA51. |
||
Tabell 2: Lista av plasmider som används i denna studie.
Discussion
Fluorescens live-cell imaging har blivit ett kraftfullt verktyg att studera spatiotemporal dynamiken i bakterieceller. Time-lapse fluorescensmikroskopi av motila och långsamt växande bakterier såsom M. xanthus, dock har varit utmanande och utfördes endast för kort tid varaktighet. Här presenterar vi en lätt-till-använda och robust metod för live-cell avbildning av M. xanthus av time-lapse fluorescensmikroskopi. Denna metod tillåter användaren att följa celler och fluorescently märkta proteiner för flera omgångar av cellcykeln med enstaka cell upplösning.
Det finns flera förutsättningar som påverkar framgången för live-cell avbildning av långsamt växande M. xanthus celler inklusive: 1) en fast yta för cell fastsättning; 2) tillgången till näringsämnen och syre. (3) konstant luftfuktighet och temperatur. och 4) optimering av experimentella förhållanden såsom exponering tid och bild förvärv frekvens.
I våra experimentella set-up använder vi tjocka agaros pads kompletteras med näringsämnen. Använda agaros som tjocka kuddar till skillnad från mikroflödessystem enheter för att följa enstaka celler har vissa grundläggande fördelar men också vissa nackdelar. Först, agaros pad ger inte bara en yta för M. xanthus cell fastsättning och rörelse men också tillräckligt näringsämnen för tillväxt under minst 24 h. För det andra, snap shot analyser används vanligen för att studera intracellulära lokalisering av fluorescently märkta proteiner gjordes tidigare på samma typ av agaros pads16,17,29. Data från snap shot analyser kan därför jämföras direkt till uppgifter som erhållits med den metod som beskrivs här. För det tredje, agaros kuddar kan ändras lätt och kompletteras med antibiotika eller andra kosttillskott såsom CuSO4 och vanillate som används vanligen för gen uttryck induktion18,30. Slutligen, eftersom celler tillåts form microcolonies under loppet av ett experiment, detta upplägg kan också studera effekten av direkt cell-cell interaktioner på viss parameter som analyseras. Denna aspekt är särskilt viktigt vid M. xanthus eftersom denna bakterie visar flera kontakt-beroende interaktioner. Den största nackdelen med denna metod är att de experimentella förhållandena är förinställda för varaktigheten av ett experiment. Däremot tillåter mikroflödessystem enheter generellt ändra de experimentella förhållandena under ett experiment genom att lägga till exempelvis antibiotika31.
Fri programvarupaket (t.ex., MicrobeJ, Oufti) finns att automatiskt analysera tillväxten av enstaka celler och protein lokalisering inom enskilda celler. Men dessa program är endast lämpade för analys av enstaka celler eller små grupper av celler. Således återstår det en utmaning att automatiskt analysera de data som genereras för 24 h inspelningarna beskrivs här.
I sammanfattning, beskrev vi en lätt-till-använda och reproducerbara protokoll för att utföra live-cell imaging med långsamt växande M. xanthus bakterier. Vi visar att enkla näringsämne-kompletteras agaros pads är tillräcklig för att upprätthålla tillväxten under minst 24 h och tillåta för att observera och analysera protein lokalisering och tillväxt med enstaka cell upplösning över flera generationer.
Disclosures
Författarna förklarar att de har inga konkurrerande finansiella intressen.
Acknowledgments
Detta arbete fick stöd av den tyska forskning rådet (DFG) inom ramen för Transregio 174 ”Spatiotemporal dynamiken i bakterieceller” och i Max Planck-sällskapet.
Materials
| Name | Company | Catalog Number | Comments |
| DMI6000B with AFC | Leica microsystems | 11888945 | Automated inverted widefield fluorescence microscope with adaptive focus control |
| Universal mounting frame | Leica microsystems | 11532338 | Stage holder for different sample sizes |
| HCX PL FLUOTAR 100x/1.30 oil PH3 | Leica microsystems | 11506197 | Phase contrast objective |
| Orca Flash 4.0 camera | Hamamatsu | 11532952 | 4.0 megapixel sCMOS camera for picture aquisition |
| Filter set TXR ET, k | Leica microsystems | 11504170 | Fluorescence filter set, Ex: 560/40 Em: 645/75 |
| Filter set L5 ET, k | Leica microsystems | 11504166 | Fluorescence filter set, Ex: 480/40 Em: 527/30 |
| Filter set YFP ET, k | Leica microsystems | 11504165 | Fluorescence filter set, Ex: 500/20 Em: 535/30 |
| ProScan III | Prior | H117N1, V31XYZEF, PS3J100 | Microscope automation controller with interactive control center |
| EL 6000 light source | Leica microsystems | 11504115 | External fluorescence light source |
| Incubator BLX Black | Pecon | 11532830 | Black incubation chamber surrounding the microscope |
| Tempcontrol 37-2 digital | Leica microsystems | 11521719 | Automated temperature control for incubation chamber |
| Gentmycin sulphate | Carl Roth | 0233.4 | Gentamycin |
| Oxytetracylin dihydrate | Sigma Aldrich | 201-212-8 | Oxytetracyclin |
| Kanamycin sulphate | Carl Roth | T832.3 | Kanamycin |
| Filtropur BT25 0.2 bottle top filter | Sarstedt | 831,822,101 | Bottle top filter for sterilization of buffers |
| Deckgläser | VWR | 630-1592 | Glass cover slip (60 x 22 mm, thickness: 0.7 mm) |
| Seakem LE agarose | Lonza | 50004 | Agarose for microscopy slides |
| Leica Metamorph AF | Leica microsystems | 11640901 | Microscope control software and software for picture analysis |
| Tetraspeck Microsperes, 0.5 µm | ThermoFisher | T7281 | Fluorescent microspheres |
| petri dish | Greiner Bio-one | 688102 | 120 mm x 120 mm x 17 mm squared petri dish for agarose pads |
| BD Bacto Casitone | Becton Dickinson | 225930 | Casitone |
| Parafilm M | VWR | 291-1213 | Parafilm |
| Tris(hydroxymethyl)-aminomethane | Carl Roth | AE15.2 | Tris |
| Magnesium sulphate heptahydrate | Carl Roth | P027.2 | Magnesium sulphate |
| Potassium dihydrogen phosphate p.a. | Carl Roth | 3904.1 | Potassium dihydrogen phosphate |
| 1% CTT medium: 1 % (w/v) BD Bacto™ casitone, 10 mM Tris-HCl ph 8.0, 1 mM potassium phosphate buffer pH 7.6, 8 mM MgSO4 | Cultivation medium for M.xanthus | ||
| TPM buffer: 10 mM Tris-HCl ph 8.0, 1 mM potassium phosphate buffer pH 7.6, 8 mM MgSO4 | Buffer for preparation of microscopy slides for M.xanthus |
References
- Shapiro, L., McAdams, H. H., Losick, R. Why and how bacteria localize proteins. Science. 326 (5957), 1225-1228 (2009).
- Treuner-Lange, A., Søgaard-Andersen, L. Regulation of cell polarity in bacteria. J Cell Biol. 206 (1), 7-17 (2014).
- Laloux, G., Jacobs-Wagner, C. Spatiotemporal control of PopZ localization through cell cycle-coupled multimerization. J Cell Biol. 201, 827-841 (2013).
- Rudner, D. Z., Losick, R. Protein subcellular localization in bacteria. Cold Spring Harb. Perspect. Biol. 2 (4), 000307 (2010).
- Badrinarayanan, A., Le, T. B. K., Laub, M. T. Bacterial chromosome organization and segregation. Annu Rev Cell Dev Biol. 31 (1), 171-199 (2015).
- Munoz-Dorado, J., Marcos-Torres, F. J., Garcia-Bravo, E., Moraleda-Munoz, A., Perez, J. Myxobacteria: Moving, Killing, Feeding, and Surviving Together. Front Microbiol. 7, 781 (2016).
- Berleman, J. E., Kirby, J. R. Deciphering the hunting strategy of a bacterial wolfpack. FEMS Microbiol Rev. 33 (5), 942-957 (2009).
- Konovalova, A., Petters, T., Søgaard-Andersen, L. Extracellular biology of Myxococcus xanthus. FEMS Microbiol. Rev. 34, 89-106 (2010).
- Nudleman, E., Wall, D., Kaiser, D. Cell-to-cell transfer of bacterial outer membrane lipoproteins. Science. 309, 125-127 (2005).
- Vassallo, C. N., et al. Infectious polymorphic toxins delivered by outer membrane exchange discriminate kin in myxobacteria. eLife. 6, 29397 (2017).
- Vassallo, C., et al. Cell rejuvenation and social behaviors promoted by LPS exchange in myxobacteria. Proc Natl Acad Sci USA. 112 (22), 2939-2946 (2015).
- Li, Y., et al. Extracellular polysaccharides mediate pilus retraction during social motility of Myxococcus xanthus. Proc. Natl. Acad. Sci. USA. 100, 5443-5448 (2003).
- Kim, S. K., Kaiser, D. Cell alignment required in differentiation of Myxococcus xanthus. Science. 249, 926-928 (1990).
- Lobedanz, S., Søgaard-Andersen, L. Identification of the C-signal, a contact dependent morphogen coordinating multiple developmental responses in Myxococcus xanthus. Genes Dev. 17, 2151-2161 (2003).
- Schumacher, D., Søgaard-Andersen, L. Regulation of cell polarity in motility and cell division in Myxococcus xanthus. Annu Rev Microbiol. 71 (1), 61-78 (2017).
- Schumacher, D., et al. The PomXYZ proteins self-organize on the bacterial nucleoid to stimulate cell division. Dev Cell. 41 (3), 299-314 (2017).
- Treuner-Lange, A., et al. PomZ, a ParA-like protein, regulates Z-ring formation and cell division in Myxococcus xanthus. Mol Microbiol. 87 (2), 235-253 (2013).
- Iniesta, A. A., Garcia-Heras, F., Abellon-Ruiz, J., Gallego-Garcia, A., Elias-Arnanz, M. Two systems for conditional gene expression in Myxococcus xanthus inducible by isopropyl-beta-D-thiogalactopyranoside or vanillate. J Bacteriol. 194 (21), 5875-5885 (2012).
- Harms, A., Treuner-Lange, A., Schumacher, D., Søgaard-Andersen, L. Tracking of chromosome and replisome dynamics in Myxococcus xanthus. reveals a novel chromosome arrangement. PLoS Genet. 9 (9), 1003802 (2013).
- Iniesta, A. A. ParABS system in chromosome partitioning in the bacterium Myxococcus xanthus. PLoS One. 9 (1), 86897 (2014).
- Lin, L., Osorio Valeriano, M., Harms, A., Søgaard-Andersen, L., Thanbichler, M. Bactofilin-mediated organization of the ParABS chromosome segregation system in Myxococcus xanthus. Nat Commun. 8 (1), 1817 (2017).
- Hodgkin, J., Kaiser, D. Cell-to-cell stimulation of movement in nonmotile mutants of Myxococcus. Proc Natl Acad Sci U S A. 74 (7), 2938-2942 (1977).
- Kaiser, D. Social gliding is correlated with the presence of pili in Myxococcus xanthus. Proc Natl Acad Sci USA. 76 (11), 5952-5956 (1979).
- Miertzschke, M., et al. Structural analysis of the Ras-like G protein MglA and its cognate GAP MglB and implications for bacterial polarity. EMBO J. 30 (20), 4185-4197 (2011).
- Hodgkin, J., Kaiser, D. Genetics of gliding motility in Myxococcus xanthus. (Myxobacterales): Two gene systems control movement. Mol Gen Genet. 171, 177-191 (1979).
- Shi, X., et al. Bioinformatics and experimental analysis of proteins of two-component systems in Myxococcus xanthus. J Bacteriol. 190 (2), 613-624 (2008).
- Bi, E. F., Lutkenhaus, J. FtsZ ring structure associated with division in Escherichia coli. Nature. 354 (6349), 161-164 (1991).
- Lutkenhaus, J., Pichoff, S., Du, S. Bacterial cytokinesis: From Z ring to divisome. Cytoskeleton. 69 (10), 778-790 (2012).
- McLoon, A. L., et al. MglC, a Paralog of Myxococcus xanthus GTPase-Activating Protein MglB, Plays a Divergent Role in Motility Regulation. J Bacteriol. 198 (3), 510-520 (2015).
- Gomez-Santos, N., et al. Comprehensive set of integrative plasmid vectors for copper-inducible gene expression in Myxococcus xanthus. Appl Environ Microbiol. 78 (8), 2515-2521 (2012).
- Treuner-Lange, A., et al. The small G-protein MglA connects to the MreB actin cytoskeleton at bacterial focal adhesions. J Cell Biol. 210 (2), 243-256 (2015).



