Overview
Fonte: Laboratórios do Dr. Ian Pepper e Dr. Charles Gerba - Universidade do Arizona
Autor de Demonstração: Alex Wassimi
Os vírus são um grupo único de entidades biológicas que infectam organismos eucarióticos e procarióticos. São parasitas obrigatórios que não têm capacidade metabólica, e para se replicarem, dependem do metabolismo hospedeiro para produzir partes virais que se auto-montam dentro das células hospedeiras.
Os vírus são ultramicroscópicos — pequenos demais para serem vistos com o microscópio de luz, visível apenas com a maior resolução do microscópio eletrônico. Uma partícula viral consiste em um genoma ácido nucleico, seja DNA ou RNA, cercado por uma camada de proteína, conhecida como capsid, composta de subunidades proteicas ou capsomers. Em alguns vírus mais complexos, o capsídeo é cercado por um envelope lipídedo adicional, e alguns têm apêndices de superfície ou caudas.
Vírus que infectam o trato intestinal de humanos e animais são conhecidos como vírus entéricos. Eles são excretados em fezes e podem ser isolados de águas residuais domésticas. Os vírus que infectam bactérias são conhecidos como bacteriófagos, e aqueles que infectam bactérias coliformes são chamados de colifagos (Figura 1). Os phages de bactérias coliformes são encontrados em qualquer lugar que as bactérias coliformes são encontradas.
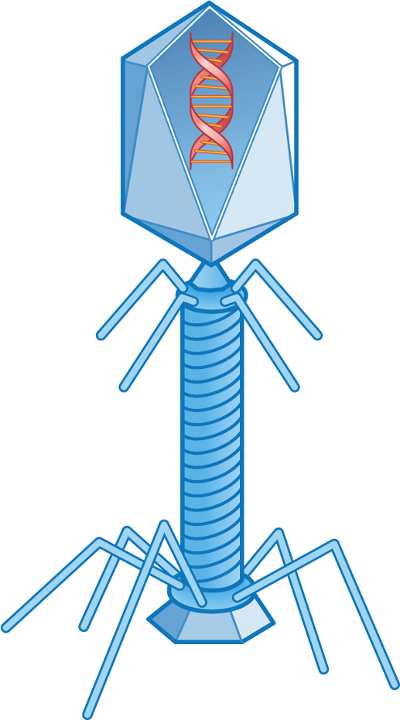
Figura 1. Coliphage T2.
Principles
Bacteriófagos são estudados em ciências ambientais por serem um componente crítico dos sistemas biológicos. Eles são a entidade biológica mais abundante do mundo e são importantes porque ajudam a controlar populações bacterianas, processos alimentares, ciclos biogeoquímicos, bem como melhorar a diversidade procariótica através da transferência horizontal de genes. Há também evidências de que os phages são indicadores substitutos confiáveis para vírus entéricos causadores de doenças que também são transmitidos fecalmente, mas difíceis de avaliar. A disponibilidade de métodos relativamente rápidos e baratos para enumerar bacteriófagos torna-os uma ferramenta atraente para a avaliação da contaminação fecal em amostras ambientais.
Colifagos na água são avaliados pela adição de uma amostra para ágar macio ou sobreposto, juntamente com uma cultura de E. coli na fase de tronco do crescimento. O phage se liga à célula bacteriana e lise as bactérias. As bactérias produzem um gramado confluente de crescimento, exceto para áreas onde a praga cresceu e liseu as bactérias. Essas áreas claras resultantes são conhecidas como placas. Uma sobreposição de ágar macio é usada para restringir a difusão física dos vírus para que, ao sair de uma bactéria, eles só possam se espalhar para células bacterianas vizinhas.
Para obter a formação ideal da placa é importante que a bactéria hospedeira esteja em fase de registro de crescimento. Isso garante que toda a praga se conecte a bactérias vivas e produza progênere. Isso exige que uma cultura de bactérias hospedeiras seja preparada a cada dia que um ensaio seja realizado. Normalmente, uma cultura é incubada um dia antes do ensaio para que ela esteja na fase estacionária. No dia do ensaio, a cultura é usada para inocular um caldo, que é incubado para obter bactérias hospedeiras suficientes na fase de tronco para o ensaio (isso geralmente requer 2-3h de incubação em um banho de água tremendo a 35-37 °C).
Subscription Required. Please recommend JoVE to your librarian.
Procedure
- Obtenha uma amostra de esgoto ou água contendo colifago.
- Diluir a amostra 1:10 e 1:100 usando tampão Tris. Faça isso transferindo 1,0 mL de cultura para 9 mL de buffer Tris, e depois fazendo uma segunda diluição de 10 vezes.
- Derreta três tubos de ágar macio (0,7% de ágar nutriente ou ágar de soja de tripática por tubo de 3 mL) colocando-os em um banho de vapor ou autoclave.
- Coloque o ágar em um banho de água a 45-48 °C por 15 minutos para permitir que a temperatura do ágar se ajuste a 45 °C.
- Ao primeiro tubo, adicione 1 mL de uma cultura de caldo de fase de tronco de E. coli1 e 1 mL de amostra não diluída.
- Retire o tubo do banho de água e balance suavemente entre as mãos para misturar a suspensão por 2-3 s.
- Limpe a água do tubo com uma toalha de papel e despeje o ágar sobre uma placa de Petri previamente preparada contendo ágar inferior (ágar de nutrientes regular ou ágar de soja de tripática).
- Gire rapidamente a placa para espalhar o ágar superior. Certifique-se de que o ágar cubra toda a superfície.
- Repita as etapas 5-8 com os outros dois tubos de ágar macio, utilizando 1 mL de bactérias e 1 mL de cada diluição amostral(Figura 2).
- Depois que o ágar se solidificar, inverta as placas de Petri e incubar a 37 °C por 48 h. Tire qualquer umidade da tampa da placa de Petri. Se uma gota de umidade cair sobre uma placa, fará com que o vírus se espalhe pela superfície do ágar.
- Após a incubação, conte o número de placas em cada diluição(Figura 3) e calcule a concentração de phage na amostra original. Regisso regissário de diferenças no tamanho ou aparência das placas.

Figura 2. Procedimento para a preparação de um gramado bacteriano utilizando ágar superior para enumeração colifa.

Figura 3. Placas de phage em um gramado bacteriano.
1E. coli strain ATCC 15597 geralmente produzirá o maior número de placas a partir de amostras de esgoto. Deve ser cultivado durante a noite em um frasco de Erlenmeyer de 250 mL contendo 100 mL de nutrientes ou caldo de soja de trippticase e incubado em condições de agitação a 35 °C. 3 h antes do ensaio de phage inocular um mL desta cultura em um frasco fresco contendo 100 mL de nutrientes ou caldo de soja tripática e colocar em um banho de água tremendo a 35-37 °C. Isso garantirá que as bactérias estejam na fase de registro do crescimento.
Os vírus são partículas biológicas infecciosas que são responsáveis por muitas doenças, resfriado e gripe, para hepatite e HIV.
Vírus são partículas biológicas que consistem em um DNA ou genoma de RNA, envoltos dentro de uma camada de proteína conhecida como capsídeo, às vezes com um envelope lipídico adicional. Os vírus não têm capacidade metabólica ou reprodutiva por conta própria, e devem invadir células vivas e sequestrar suas máquinas celulares para fazer mais cópias de si mesmos.
Ambas as células procarióticas, como bactérias, e células eucarióticas, como as de humanos, podem ser infectadas por classes específicas de vírus. Especificamente, bacteriófagos são vírus que infectam bactérias. Por exemplo, colifagos são aqueles que infectam e. coli, uma bactéria intestinal comum, algumas cepas das quais podem causar intoxicação alimentar, e que é uma indicação de contaminação fecal do abastecimento de água.
Embora os phages em si não sejam geralmente conhecidos por serem patogênicos em humanos, há evidências de que eles são indicadores de substituto confiáveis para enterovírus causadores de doenças que também são transmitidos fecalmente, mas difíceis de avaliar. A disponibilidade de métodos relativamente rápidos e baratos para enumerar bacteriófagos torna-os uma ferramenta atraente para a avaliação da contaminação fecal em amostras ambientais.
Este vídeo introduzirá os princípios por trás da enumeração da phage; demonstrar um protocolo para quantificar phages, conhecido como ensaio de placa; e, finalmente, explorar várias aplicações de ciência ambiental para a detecção e contagem de phages e outros vírus.
Bacteriófagos, como todos os vírus, devem parasitar células vivas, neste caso bactérias, a fim de se reproduzirem.
Phages fazem isso pousando e anexando à superfície celular bacteriana e injetando seus materiais genéticos na célula. Uma vez dentro da célula, o genoma viral é replicado e os componentes proteicos do capsídeo viral são produzidos, ambos usando a máquina bioquímica da célula hospedeira. Uma vez que as partículas de praga são montadas, elas são liberadas das bactérias, muitas vezes lisendo o hospedeiro e estourando, matando a célula hospedeira no processo.
Um método amplamente utilizado para determinar a concentração de phage em uma amostra aproveita essa atividade lítica. Nesta técnica, o phage da amostra é misturado com bactérias e ágar macio. Esta mistura é derramada em pratos petri com ágar regular como um substrato, e a camada superior forma uma sobreposição.
As bactérias estão em uma concentração tão alta que formam um gramado contínuo. As bactérias devem ser obtidas a partir da cultura que está na fase de registro do crescimento, para garantir que cada bactéria que os phages infectam esteja viva e permita que o phage produza prole.
Quando uma partícula de phage infecta e lise uma bactéria, a prole phage se espalhará para células bacterianas próximas e continuará a infecção. O ágar macio restringe a difusão das partículas de praga. Eventualmente, uma área de desmatamento, conhecida como placa, será formada.
Se a praga for diluída a uma concentração baixa o suficiente, então discretas, placas individuais podem ser observadas no gramado bacteriano. Estes podem ser contados e usados para calcular o número de unidades formadoras de placas, ou PFUs, de phage por mL da amostra original.
Agora que você entende como as pragas infectam bactérias e como essa atividade pode ser usada para medir a concentração de phage, vamos passar por um protocolo para usar um ensaio de placa para enumerar phages em amostras de água ambiental.
Um dia antes de realizar o ensaio, inocular uma colônia de E. coli escoar ATCC 15597 em 100 mL de caldo de soja de tripca em um frasco de Erlenmeyer de 250 mL. Incuba-o com agitação a 35 °C durante a noite.
3 h antes do ensaio, inocular 1 mL da cultura E. coli durante a noite em um fresco 100 mL de caldo. Coloque esta nova cultura em um banho de água tremendo a uma temperatura entre 35 e 37 °C. Isso garante que as bactérias estejam na fase de registro do crescimento quando o ensaio começar.
Para iniciar o ensaio da placa, faça diluições seriais de 10 e 100 vezes da amostra de água, usando 9 mL de tampão Tris.
Usando um banho de vapor, derreta três tubos de 5 mL de 0,7% de ágar superior macio, ou soja de trippticase ou ágar nutriente. Uma vez derretido, coloque em um banho de água a 45 a 48 °C por pelo menos 15 minutos para que a temperatura do ágar caia para 45 °C.
Ao primeiro tubo de ágar macio, adicione 1 mL da cultura E. coli de fase de tronco previamente preparada e 1 mL da amostra de água não diluída. Retire o tubo do banho de água e balance suavemente entre as mãos por 2-3 s para misturar a suspensão.
Despeje o ágar macio sobre uma placa de Petri previamente preparada com soja de trippticase ou ágar nutriente. Gire rapidamente a placa para se espalhar, certificando-se de que ela cobre toda a superfície.
Repita a inoculação e o revestimento para os outros dois tubos, utilizando 1 mL de cada uma das amostras diluídas.
Uma vez que o ágar superior tenha solidificado, inverta os pratos e incuba-os a 37 °C por 48 h.
Após a incubação, conte o número de pragas em cada prato. A partir da contagem, calcule a concentração de phage na amostra original
Por exemplo, se 9 placas foram obtidas da placa de diluição de 10 vezes, então há 9 vezes 10 divididas por 1 mL, ou 90 PFUs/mL de colifago na amostra de água original.
Agora que você viu como os ensaios da placa de phage são realizados, vamos ver como os ensaios de placa podem ser usados para enumerar phage e outros tipos de vírus de uma variedade de fontes.
Métodos baseados em ensaios de placa podem ser usados para isolar bacteriófago de diferentes amostras ambientais, como o solo. Neste exemplo, os pesquisadores primeiro coletaram phage do solo por filtragem. A praga foi então usada para infectar a bactéria do solo comum Arthrobacter em um ensaio de placa. Phages foram colhidos a partir de placas individuais e listrados em novas placas de ágar, e depois sobrepostos com ágar superior contendo bactérias. A concentração de fáragem diminui ao longo do comprimento da raia, de modo que placas discretas, provavelmente formadas por um único tipo de phage, possam ser obtidas. Essas placas individuais poderiam então ser escolhidas para analisar melhor os phages dentro.
Além dos bacteriófagos, ensaios de placas também podem ser realizados com outros vírus, incluindo aqueles, como a gripe, que infectam mamíferos. Para isso, as células de mamíferos são cultivadas pela primeira vez como monocamadas em pratos de cultura tecidual. As mídias que contêm os vírus são então adicionadas às células para permitir que a infecção ocorra, antes que as células sejam sobrepostas com um meio imobilizador, como a agarose semelhante ao gel. Após um período de incubação que pode durar até duas semanas, as células infectadas são fixas e manchadas para permitir que as placas sejam visualizadas e contadas.
Finalmente, além das amostras coletadas do ambiente, os ensaios de placas são úteis para detectar e enumerar vírus em amostras de tecidos de indivíduos infectados. Aqui, os pesquisadores obtiveram e homogeneizaram tecidos pulmonares de camundongos infectados com herpesvírus gama. Este homogenate contendo vírus foi então usado para infectar a cultura celular dos mamíferos. O número de placas poderia então ser contado para fornecer uma estimativa para o título viral nos tecidos pulmonares infectados.
Você acabou de assistir ao vídeo do JoVE sobre a detecção de bacteriófagos em amostras ambientais. Agora você deve entender a biologia básica dos phages, como realizar um ensaio de placa para quantitar phages em uma amostra ambiental, e como ensaios de placa podem ser usados para estudar phages e outros vírus em amostras ambientais ou clínicas. Como sempre, obrigado por assistir!
Subscription Required. Please recommend JoVE to your librarian.
Results
Diluição da amostra de esgoto = 10-1
Número de placas obtidas = 9
Portanto, concentração de phage na amostra de esgoto
= 10 x 9 ÷ 1 mL
= 90 unidades formadoras de placas / mL
O esgoto bruto normalmente contém 103 – 104 colifagos por mL, com um intervalo de 102 – 108 por mL.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
Existem muitas aplicações potenciais de colifagos como indicadores ambientais. Estes incluem seu uso como indicadores de contaminação de esgoto, eficiência do tratamento de água e esgoto, e sobrevivência de vírus e bactérias entéricas no meio ambiente. O uso de bacteriófagos como indicadores da presença e comportamento de bactérias entéricas e vírus animais sempre foi atrativo devido à facilidade de detecção e baixo custo associado aos ensaios de phage. Além disso, podem ser quantificados em amostras ambientais dentro de 24h em comparação com dias ou semanas para vírus entéricos.
1E. coli strain ATCC 15597 geralmente produzirá o maior número de placas a partir de amostras de esgoto. Deve ser cultivado durante a noite em um frasco de Erlenmeyer de 250 mL contendo 100 mL de nutrientes ou caldo de soja de trippticase e incubado em condições de agitação a 35 °C. 3 h antes do ensaio de phage inocular um mL desta cultura em um frasco fresco contendo 100 mL de nutrientes ou caldo de soja tripática e colocar em um banho de água tremendo a 35-37 °C. Isso garantirá que as bactérias estejam na fase de registro do crescimento.
Subscription Required. Please recommend JoVE to your librarian.




