형광 기반 프라이머 확장 기술은 전사 시작 포인트 및 RNases의 쪼개짐 사이트를 확인하는<em> 생체</em
Summary
We here describe a fluorescence based primer extension method to determine transcriptional starting points from bacterial transcripts and RNA processing in vivo using an automated gel sequencer.
Abstract
형광 계 프라이머 신장 (FPE)는 전사 시작점 또는 RNA 분자의 프로세싱 사이트를 결정하기 위해 분자 방법이다. 이것은 특정 형광 표지 된 프라이머 및 폴리 아크릴 아미드 겔 전기 영동을 변성하여 수득 된 cDNA 단편의 후속 분석을 이용하여 관심있는 RNA의 역전사에 의해 달성된다. 동시에, 전통적인 생거 시퀀싱 반응은 자신의 정확한 해당 기지의 cDNA 단편의 단부를 매핑 겔상에서 실행된다. 제품은 복제되어야 복수 후보 서열 5'-RACE (cDNA를 엔드 신속한 증폭), 대조적으로, 프라이머 신장에 의해 생성 된 cDNA 단편의 대부분은 동시에 하나 겔 실행에서 검출 할 수있다. 또한, (결과를 최종 분석에 역전사에서) 모든 절차는 하나의 작업 일에 완료 할 수 있습니다. 형광 표지 된 프라이머를 사용함으로써, 유해 방사성 동위 원소의 사용은 표지 된 시약을회피 할 수 있고, 제품은 전기 영동 과정에서 검출 될 수 있기 때문에 처리 시간이 감소된다.
다음의 프로토콜에서는 확실하고 신속 S.에서 (예를 들어, 독소 항독소 시스템 구성 요소) 시작점 및 RNA 프로세싱 사이트 전사을 추론의 RNA의 5 '말단을 검출하는 생체 내 형광 프라이머 신장 방법을 서술 구균, E. 대장균 및 기타 세균.
Introduction
프라이머 신장도 1은 하나의 기본 해상도까지 특정 RNA 분자의 5 '말단을 결정하기 위해 분자 방법이다. 이러한 5'-RACE (cDNA의 신속한 증폭이 종료 됨) 같은 다른 방법의 장점은 빠른 처리 시간을 용이 RNA 분자의 서로 다른 길이의 혼합물을 분석 할 수있는 능력이다.
이 방법은 RNA 분자가 특정 길이의 cDNA 단편을 생성하는 특정 형광 프라이머를 사용하여 전사 반응을 반대로 실시함으로써 작동한다. 이들의 cDNA 분자 변성 폴리 아크릴 아마이드 겔에 전통적인 생거 시퀀싱 반응이 함께 실행 의한 형광 표지 된 프라이머를 사용하여 그 형광을 검출 할 수있다. 된 cDNA 단편의 길이는 5 '말단 RNA의 맵핑을 허용 시퀀싱 사다리 비교에 의해 평가된다.
전통적으로, 프라이머 신장 반응을 함께 사용방사성 동위 원소와 X 선 필름에의 cDNA 분자들을 검출한다. 그들의 감도가 약간 낮다이라도 인해 건강 위험, 폐기물 처리 문제 및 취급의 용이성, 새로운 프로토콜은 자동화 된 시퀀서와 프라이머 신장의 검출을위한 형광을 이용한다. 형광 프라이머 (로모 손 년 이상) 장기간 안정적으로 형광 표지 된 프라이머를 사용하여 무선 표지 반복 절차가 생략 될 수있다.
우리는 여기에서 설명하는 방법은 자동화 겔 시퀀서를 이용하지만, 약간의 수정과, 모세관 시퀀서는 cDNA의 분리 및 검출 3에 사용될 수있다. 겔 분석 병렬 특성은 가능한 RNA 절단 또는 프로세싱 심지어 적은 양을 검출 할 수있다. 또 다른 이점은 검출 될 수 단말 절단 또는 심지어 하나의베이스 처리로서,이 방법의 고해상도이다.
RNA 절단 또는 가공, t의 검출에 관련하여프라이머 확장 ypically 두 가지 종류가 구별된다. 다른 경우에, 처리가 생체 내에서 수행하고, 생성 된 RNA 정제 반면 한 경우, 효소 처리, 정제 된 RNA 정제 효소를 사용하여 시험 관내에서 수행된다. 프라이머 신장 시험 관내에서 수행에 두 경우 RNA가 RNA의 소스에 따라, 그러나, 실시되고, 상기 방법은 시험 관내 또는 생체 내 프라이머 신장이라고 하나. 여기 제시 프로토콜에서는 때문에 사용 용이성 (정제없이 필요한 단백질)과 동시에 전사 시작점 및 처리를 결정하는 가능성 단독 생체 프라이머 신장에 초점. 그러나, 시험 관내 프라이머 확장은 동일한 방법으로 설정 원칙적으로,이 프로토콜은 시작점을 제공 할 수있다.
여기에 예시 된 방법은 그것들이 높은 의무만큼 많은 세균 종에 적용될 수있다핵산의 순도 높은 수율 준비.
우리 실험실 연구 독소 항독소 (맞대기) 시스템 4,5, 프라이머 신장 방법은 광범위하게 사용되는 필드의 규정 범위에 초점을 맞춘다. TA-시스템은 안정적이고 내생 활성 독성 단백질과 독성 6,7 중화 대부분 불안정한 단백질 또는 RNA 항독소로 구성 원핵 생물의 게놈에 존재하는 작은 유전 적 요소입니다. 독소 활성은 때때로하는 RNase 활성 8,9 의해 대부분 복제, 세포벽 합성 또는 다른 메커니즘의 억제에 의해 발휘되지만. 일반적으로, RNA 분해 효소 특이성 프라이머 신장 방법 중 하나는 다른 테스트를 수행하여 결정된다. 벽개 및 전체 길이 단편의 혼합물을 동시에 그들의 5 '끝나는 결정하기 위해 분석 될 수있는 프라이머 신장 반응,이 애플리케이션에 매우 적합하다. 시험 관내 및 생체 프라이머 확장에 믹스를 사용하여,특정 독소의 RNase 절단은, 예를 들어, 서열 특이성 10-13 결정될 수있다.
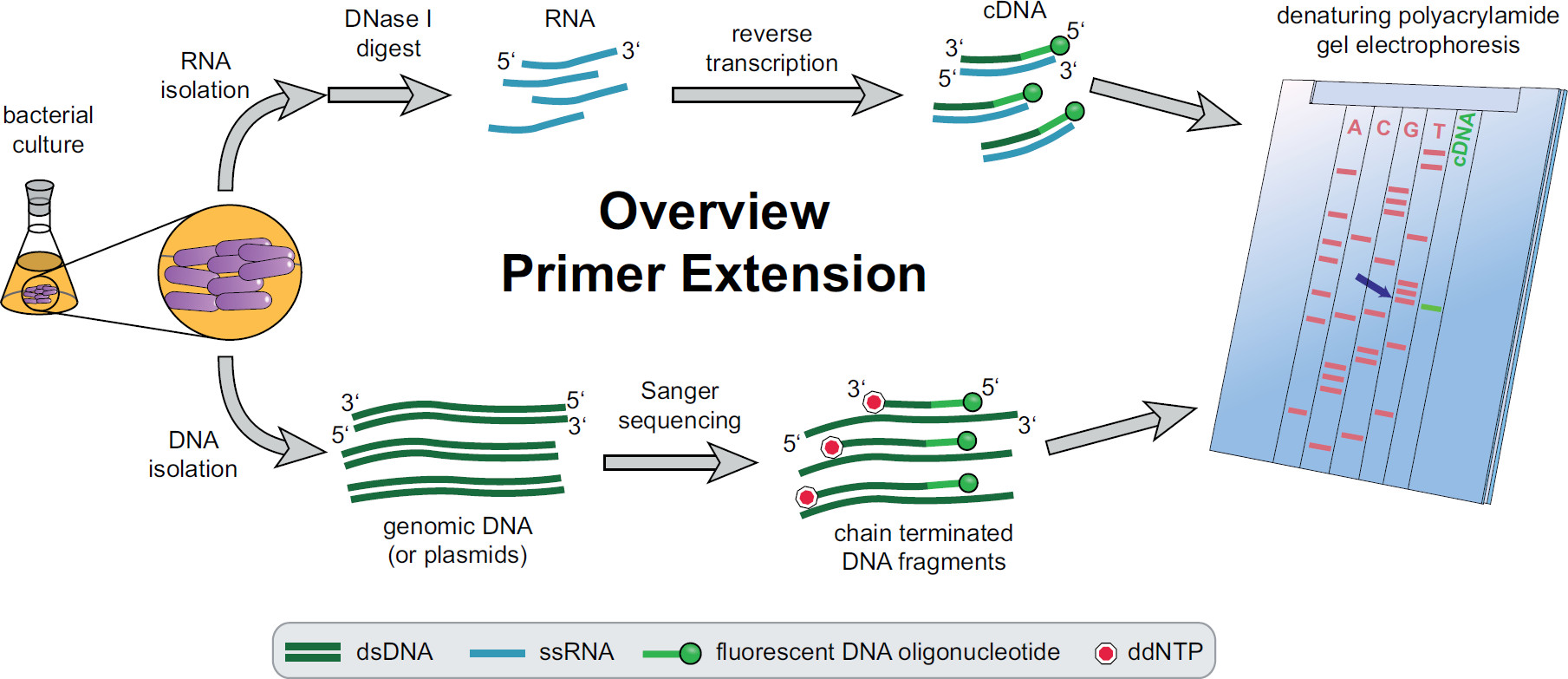
. 프라이머 확장 절차 1. 개요 그림 세균 배양 실험의 필요에 따라 배양 및 처리됩니다. 총 RNA는 세포의 cDNA를 수득 대상 특정 형광 DNA 프라이머를 사용하여 역전사 반응에 DNA의 흔적을 제거하기의 DNase I로 처리하고 실시로부터 추출된다. 게놈 DNA 또는 플라스미드를 추출하여 이후의 cDNA 조각과 크기 비교를 위해 형광 생거 시퀀싱 반응에 사용된다. 프라이머 신장 산물은 변성 우레아 폴리 아크릴 아미드 겔상에서 생거 시퀀싱 제품을 함께 실행하고 자동화 레이저 현미경으로 분석한다. 라 cDNA를 밴드와 함께 선까지입니다 시퀀싱베이스세인트 5 '의 cDNA 끝 (파란색 화살표)의 기본. Fekete 추가 정보, 등. (3) 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
전체 프라이머 신장 절차의 개요는도 1에서 발견 될 수있다. 요약하면, 박테리아 세포 수확, 배양, 세포 펠렛을 용해 및 RNA 추출. 정제 된 RNA이어서 역전사 템플릿으로 동작 할 수있다 DNA 분자의 흔적을 제거하기의 DNase I로 처리된다. 특정 형광 프라이머는 관심 영역에 교배와 이후 단일 가닥 보완 DNA (cDNA를) 결과, 전사 역, RNA에 추가됩니다. 시퀀싱 사다리 형광 프라이머를 이용하는 전통적인 생거 시퀀싱에 의해 생성 및 프라이머 신장 된 cDNA 단편의 나란히 변성 폴리 아크릴 아미드 겔상에서 분리된다. 결과겔은 관심의 5 '말단의 식별을 허용 형광 밴드를 비교하여 분석된다. 전사 시작 지점 및 처리 사이트는 다음 서열 비교에 의해 개별적으로 평가된다.
Protocol
Representative Results
Discussion
형광 프라이머 신장은 TSP- 또는 RNA 이차 가공 식별 어느 쪽의 RNA의 5 '말단을 결정하기위한 간단하고 신속한 방법이다. 때문에 형광 프라이머의 사용, 반응이 설정 될 수 있으며, (방사성 표지 된 프라이머 경우와 달리) 추가 보안 조치없이 실행합니다. 샘플은 형광으로 검출되는 바와 같이 전기 영동 X 선 필름은 일반적으로 사용되는 방사성 방법에 비해 신속한 분석을 허용 진행되는 동안, 이들…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Anne Wochele for her assistance in the laboratory and Vera Augsburger for help with the automated gel sequencer. We thank the Deutsche Forschungsgemeinschaft for funding by grants BE4038/2 and BE4038/5 within the “priority programmes” SPP1316 and SPP1617.
Materials
| Name | Supplier | Catalog Number(s) | Comment |
| AMV Reverse Transcriptase (20-25 U/µl) | NEB / Roche | NEB: M0277-T / Roche: 10109118001 | |
| DNase I (RNase free) | Ambion (life technologies) | AM2222 | |
| FastPrep-24 Instrument | MPBio | 116004500 | |
| Fluorescently labeled primers | Biomers | n/a | 5’ DY-681 modification of “ordinary” DNA oligonucleotides. Compatible dyes such as the LICOR IRDye 700/800 are also available from other suppliers such as IDTdna. |
| Li-Cor 4200 Sequencer incl. ImagIR Data collection software | Li-Cor | Product discontinued | |
| NanoDrop 2000 | Thermo Scientific | ||
| Nuclease free water | Ambion (life technologies) | AM9915G | |
| Plasmid mini preparation kit | QIAGEN | 12125 | |
| RapidGel-XL-40% Concentrate | USB | US75863 | |
| RNA STORAGE BUFFER | Ambion (life technologies) | AM7000 | |
| Roti-Aqua-P/C/I | Carl Roth | X985.3 | Alternative: “Acid-Phenol:Chloroform, pH 4.5 (with IAA, 125:24:1)” from Ambion (AM9720) |
| SUPERase•In RNase Inhibitor | Ambion (life technologies) | AM2696 | |
| Thermo Sequenase fluorescently labelled primer cycle sequencing kit with 7-deaza-dGTP | GE Healthcare | RPN2538 | |
| TRIzol reagent | life technologies | 15596-026 | |
| Zirconia/Silica Beads 0.1 mm | BioSpec | 11079101z |
References
- Simpson, C. G., Brown, J. W. Primer extension assay. Methods Mol. Biol. 49, 249-256 (1995).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74, 5463-5467 (1977).
- Fekete, R. A., Miller, M. J., Chattoraj, D. K. Fluorescently labeled oligonucleotide extension: a rapid and quantitative protocol for primer extension. Biotechniques. 35, 90-94 (2003).
- Schuster, C. F., et al. Characterization of a mazEF Toxin-Antitoxin Homologue from Staphylococcus equorum. J. Bacteriol. 195, 115-125 (2013).
- Nolle, N., Schuster, C. F., Bertram, R. Two paralogous yefM-yoeB loci from Staphylococcus equorum encode functional toxin-antitoxin systems. Microbiology. 159, 1575-1585 (2013).
- Yamaguchi, Y., Park, J. H., Inouye, M. Toxin-antitoxin systems in bacteria and archaea. Annu. Rev. Genet. 45, 61-79 (2011).
- Schuster, C. F., Bertram, R. Toxin-antitoxin systems are ubiquitous and versatile modulators of prokaryotic cell fate. FEMS Microbiol. Lett. 340, 73-85 (2013).
- Goeders, N., Van Melderen, L. Toxin-antitoxin systems as multilevel interaction systems. Toxins. 6, 304-324 (2014).
- Yamaguchi, Y., Inouye, M. mRNA interferases, sequence-specific endoribonucleases from the toxin-antitoxin systems. Progress in molecular biology and translational science. 85, 467-500 (2009).
- Park, J. H., Yamaguchi, Y., Inouye, M. Bacillus subtilis MazF-bs (EndoA) is a UACAU-specific mRNA interferase. FEBS Lett. 585, 2526-2532 (2011).
- Zhu, L., et al. et al.Staphylococcus aureus MazF specifically cleaves a pentad sequence, UACAU, which is unusually abundant in the mRNA for pathogenic adhesive factor SraP. J. Bacteriol. 191, 3248-3255 (2009).
- Zhu, L., et al. The mRNA interferases, MazF-mt3 and MazF-mt7 from Mycobacterium tuberculosis target unique pentad sequences in single-stranded RNA. Mol. Microbiol. 69, 559-569 (2008).
- Fu, Z., Donegan, N. P., Memmi, G., Cheung, A. L. Characterization of MazFSa, an endoribonuclease from Staphylococcus aureus. J. Bacteriol. 189, 8871-8879 (2007).
- Marmur, J. A procedure for the isolation of deoxyribonucleic acid from micro-organisms. J. Mol. Biol. 3, 208-218 (1961).
- Emory, S. A., Belasco, J. G. The ompA 5′ untranslated RNA segment functions in Escherichia coli as a growth-rate-regulated mRNA stabilizer whose activity is unrelated to translational efficiency. J. Bacteriol. 172, 4472-4481 (1990).
- Cole, S. T., Bremer, E., Hindennach, I., Henning, U. Characterisation of the promoters for the ompA gene which encodes a major outer membrane protein of Escherichia coli. Mol. Gen. Genet. 188, 472-479 (1982).
- Schleifer, K. H., Kilpper-Bälz, R., Devriese, L. Staphylococcus arlettae sp. nov., S. equorum sp. nov. and S. kloosii sp. nov.: Three New Coagulase-Negative, Novobiocin-Resistant Species from Animals. Syst. Appl. Microbiol. 5, 501-509 .
- Yu, H., Goodman, M. F. Comparison of HIV-1 and avian myeloblastosis virus reverse transcriptase fidelity on RNA and DNA templates. J. Biol. Chem. 267, 10888-10896 (1992).
- Ying, B. W., Fourmy, D., Yoshizawa, S. Substitution of the use of radioactivity by fluorescence for biochemical studies of RNA. RNA. 13, 2042-2050 (2007).

