Fluorescens Baseret Primer Extension teknik til at bestemme Transkriptionelle udgangspunkter og kløvningsstederne RNaser<em> In Vivo</em
Summary
We here describe a fluorescence based primer extension method to determine transcriptional starting points from bacterial transcripts and RNA processing in vivo using an automated gel sequencer.
Abstract
Fluorescens baserede primerforlængelse (FPE) er en molekylær metode til at bestemme transkriptionelle udgangspunkter eller forarbejdning lokaliteter af RNA-molekyler. Dette opnås ved revers transkription af RNA af interesse under anvendelse af specifikke fluorescensmærkede primere og efterfølgende analyse af de resulterende cDNA-fragmenter ved denaturerende polyacrylamidgelelektroforese. Samtidig er en traditionel Sanger sekventering reaktion kørt på gelen for at kortlægge enderne af cDNA-fragmenterne til deres nøjagtige tilsvarende baser. I modsætning til 5'-RACE (Rapid Amplifikation af cDNA Ends), hvor produktet skal klones og flere kandidater sekventeret, kan hovedparten af cDNA-fragmenter genereret ved primerforlængelse samtidigt detekteres i en gel løb. Derudover kan hele proceduren (fra revers transkription til endelig analyse af resultaterne) udfyldes på én arbejdsdag. Ved hjælp af fluorescens-mærkede primere, brug af farlige radioaktive isotop mærkede reagenserkan undgås, og sagsbehandlingstider er nedbragt så produkterne kan blive fundet under elektroforese procedure.
I den følgende protokol beskriver vi en in vivo fluorescerende primerforlængelse metode til pålideligt og hurtigt påvise 5 'enderne af RNA'er til at udlede transkriptionel udgangspunkter og RNA-forarbejdning sites (fx ved toksin-antitoksin systemkomponenter) i S. aureus, E. coli og andre bakterier.
Introduction
Primerforlængelse 1 er en molekylær metode til bestemmelse af 5 'enderne af specifikke RNA-molekyler op til én base opløsning. Fordelen ved at andre metoder såsom 5'-RACE (rapid amplifikation af cDNA-ender) er den hurtige behandlingstid og evnen til let at analysere en blanding af forskellige længder af RNA-molekyler.
Denne metode fungerer ved at underkaste RNA-molekyler til at vende transkriptionsreaktioner anvendelse af specifikke fluorescerende primere generere cDNA-fragmenter af visse længder. Disse cDNA-molekyler er sideløbende traditionelle Sanger sekventeringsreaktioner 2 på denaturerende polyacrylamidgeler og kan påvises ved deres fluorescens som følge af anvendelsen af fluorescens-mærkede primere. Længderne af de cDNA-fragmenter vurderes derefter ved sammenligning med den sekventeringsstige, tillader kortlægning af 5'-RNA-ender.
Traditionelt er primerforlængelsesreaktioner anvendes i forbindelsemed radioaktive isotoper til påvisning af cDNA-molekyler på X-ray film. Grundet sundhedsfarer, bortskaffelse af affald spørgsmål og lette håndtering, nyere protokoller udnytte fluorescens til påvisning af primerforlængelsen med automatiserede sequencere, omend deres følsomhed er lidt lavere. Brug af fluorescens-mærkede primere, kan den tilbagevendende procedure radio-mærkning undlades, som fluorescerende primere er stabile i lang tid (mere end et år i vores hænder).
Fremgangsmåden beskrevet her anvender en automatiseret gel sequencer, men med små modifikationer kan kapillære sequencers også anvendes til cDNA-separation og detektion 3. Den parallelle karakter gel analyse gør det muligt at detektere selv en lille mængde RNA spaltning eller forarbejdning. En anden fordel er den høje opløsning af denne fremgangsmåde, som terminal spaltning eller forarbejdning af endnu en base kan detekteres.
Med hensyn til påvisning af RNA-spaltningen eller forarbejdning, typically to forskellige typer af primerforlængelser skelnes. I et tilfælde er den enzymatiske behandling udføres in vitro ved anvendelse af oprenset RNA og oprenset enzym, mens i det andet tilfælde er behandlingen udføres in vivo, og den resulterende RNA renses. I begge tilfælde RNA underkastes et primerforlængelsesprodukt udføres in vitro, dog afhængigt af kilden til RNA, er fremgangsmåden enten kaldes en in vitro eller in vivo primerforlængelse. I protokollen præsenterer vi her fokuserer vi udelukkende på in vivo primerforlængelse grund af brugervenlighed (ingen oprensede proteiner nødvendigt) og muligheden for at bestemme transkriptionelle udgangspunkter og forarbejdning på samme tid. Men i vitro primerforlængelser i princippet oprettet på samme måde og denne protokol kan tjene som udgangspunkt.
Fremgangsmåden er illustreret her, kan anvendes til mange bakteriearter, så længe de er medgørlige til højforberedelse renhed og højt udbytte af nukleinsyrer.
Forskningen i vores laboratorium fokuserer på den regulerende rækkevidde af toksin-antitoksin (TA) systemer 4,5, et område, hvor primerekstensionsproduktet metode benyttes flittigt. TA-systemer er små genetiske elementer til stede i prokaryote genomer, der består af en stabil og endogent aktivt toksisk protein og en hovedsagelig ustabilt protein eller RNA antitoksin, der modvirker toksicitet 6,7. Toksin-aktivitet er undertiden udøves ved hæmning af replikation, cellevægsyntese eller andre mekanismer, men oftest af RNase-aktivitet 8,9. Typisk er RNase specificitet bestemmes ved at udføre forskellige test, hvoraf den ene er primerekstensionsproduktet metode. Primerforlængelsesreaktioner er velegnede til denne anvendelse, som en blanding af spaltede og fuld længde fragmenter samtidig kan analyseres for at bestemme deres 5'-ender. Ved hjælp af en blanding af in vitro og in vivo primerforlængelser, denspecifikke toksin RNase-spaltning, f.eks sekvens specificitet kan bestemmes 10-13.
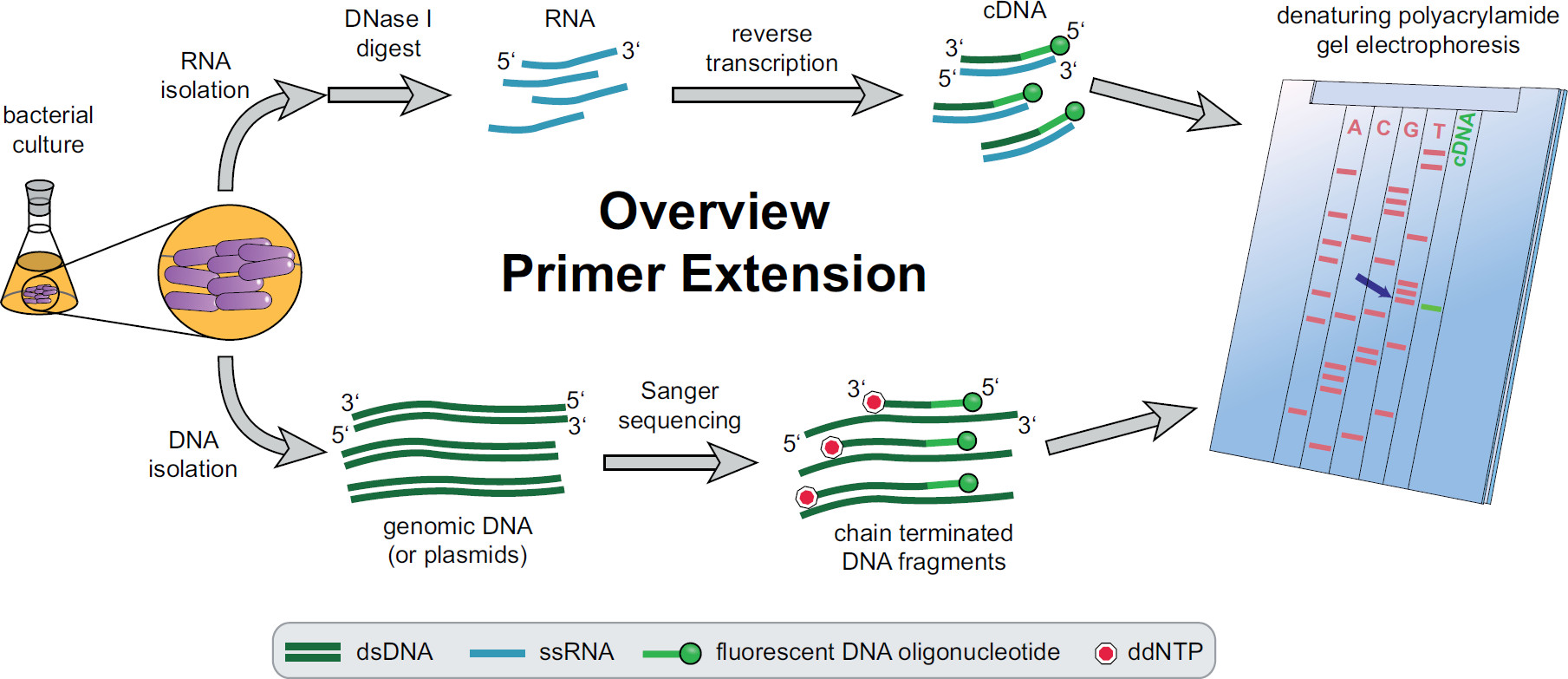
Figur 1. Oversigt over primerforlængelse procedure. Bakteriekulturer inkuberes og behandles i henhold til de eksperimentelle behov. Totalt RNA ekstraheres fra cellerne, der er behandlet med DNase I for at fjerne DNA-spor og underkastet en revers transkription under anvendelse af målspecifikke fluorescerende DNA-primere, der giver cDNA. Genomisk DNA eller plasmider ekstraheres og derefter anvendes til fluorescerende Sanger sekventeringsreaktioner for størrelse sammenligning med cDNA-fragmenter. Primerforlængelsesprodukter køres sideløbende Sanger sekventering produkter på en denaturerende polyacrylamidgel urinstof og analyseret med et automatiseret laser og mikroskop. Den sekventering base, der flugter med cDNA bandet er last base af 5'-cDNA-ende (blå pil). Mere information i Fekete, et al. 3 Klik her for at se en større udgave af dette tal.
Et overblik over hele primerforlængelse procedure kan findes i figur 1. Kort fortalt bakterieceller dyrkes, høstes, cellepelleten lyseret, og RNA ekstraheret. Oprenset RNA behandles dernæst med DNase I for at fjerne spor af DNA-molekyler, som kunne fungere som skabeloner for revers transkriptase. Specifikke fluorescerende primere tilsættes til RNA hybridiseret til regionen af interesse og efterfølgende revers transkriberet, resulterer i enkeltstrenget komplementært DNA (cDNA). En sekventeringsstige er skabt af traditionelle Sanger-sekventering anvender fluorescerende primere og separeret på en denaturerende polyacrylamidgel ved siden af primerekstensionsproduktet cDNA-fragmenter. Den resulterendegel analyseres ved at sammenligne de fluorescerende bånd, som muliggør identifikation af 5 'enderne af interesse. Transkriptionelle udgangspunkter og forarbejdning sites derefter vurderes individuelt i sekvens sammenligninger.
Protocol
Representative Results
Discussion
Fluorescent primerforlængelse er en enkel og hurtig metode til bestemmelse af 5 'enderne af RNA, enten til TSP- eller sekundær RNA-bearbejdning identifikation. På grund af anvendelsen af fluorescerende primere, kan reaktionerne blive oprettet og drives uden yderligere sikkerhedsforanstaltninger (i modsætning til i tilfælde af radioaktivt mærkede primere). Da prøverne påvises ved fluorescens, kan de afbildes mens elektroforese er i gang, som tillader hurtig analyse sammenlignet med radioaktive metoder, …
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Anne Wochele for her assistance in the laboratory and Vera Augsburger for help with the automated gel sequencer. We thank the Deutsche Forschungsgemeinschaft for funding by grants BE4038/2 and BE4038/5 within the “priority programmes” SPP1316 and SPP1617.
Materials
| Name | Supplier | Catalog Number(s) | Comment |
| AMV Reverse Transcriptase (20-25 U/µl) | NEB / Roche | NEB: M0277-T / Roche: 10109118001 | |
| DNase I (RNase free) | Ambion (life technologies) | AM2222 | |
| FastPrep-24 Instrument | MPBio | 116004500 | |
| Fluorescently labeled primers | Biomers | n/a | 5’ DY-681 modification of “ordinary” DNA oligonucleotides. Compatible dyes such as the LICOR IRDye 700/800 are also available from other suppliers such as IDTdna. |
| Li-Cor 4200 Sequencer incl. ImagIR Data collection software | Li-Cor | Product discontinued | |
| NanoDrop 2000 | Thermo Scientific | ||
| Nuclease free water | Ambion (life technologies) | AM9915G | |
| Plasmid mini preparation kit | QIAGEN | 12125 | |
| RapidGel-XL-40% Concentrate | USB | US75863 | |
| RNA STORAGE BUFFER | Ambion (life technologies) | AM7000 | |
| Roti-Aqua-P/C/I | Carl Roth | X985.3 | Alternative: “Acid-Phenol:Chloroform, pH 4.5 (with IAA, 125:24:1)” from Ambion (AM9720) |
| SUPERase•In RNase Inhibitor | Ambion (life technologies) | AM2696 | |
| Thermo Sequenase fluorescently labelled primer cycle sequencing kit with 7-deaza-dGTP | GE Healthcare | RPN2538 | |
| TRIzol reagent | life technologies | 15596-026 | |
| Zirconia/Silica Beads 0.1 mm | BioSpec | 11079101z |
References
- Simpson, C. G., Brown, J. W. Primer extension assay. Methods Mol. Biol. 49, 249-256 (1995).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74, 5463-5467 (1977).
- Fekete, R. A., Miller, M. J., Chattoraj, D. K. Fluorescently labeled oligonucleotide extension: a rapid and quantitative protocol for primer extension. Biotechniques. 35, 90-94 (2003).
- Schuster, C. F., et al. Characterization of a mazEF Toxin-Antitoxin Homologue from Staphylococcus equorum. J. Bacteriol. 195, 115-125 (2013).
- Nolle, N., Schuster, C. F., Bertram, R. Two paralogous yefM-yoeB loci from Staphylococcus equorum encode functional toxin-antitoxin systems. Microbiology. 159, 1575-1585 (2013).
- Yamaguchi, Y., Park, J. H., Inouye, M. Toxin-antitoxin systems in bacteria and archaea. Annu. Rev. Genet. 45, 61-79 (2011).
- Schuster, C. F., Bertram, R. Toxin-antitoxin systems are ubiquitous and versatile modulators of prokaryotic cell fate. FEMS Microbiol. Lett. 340, 73-85 (2013).
- Goeders, N., Van Melderen, L. Toxin-antitoxin systems as multilevel interaction systems. Toxins. 6, 304-324 (2014).
- Yamaguchi, Y., Inouye, M. mRNA interferases, sequence-specific endoribonucleases from the toxin-antitoxin systems. Progress in molecular biology and translational science. 85, 467-500 (2009).
- Park, J. H., Yamaguchi, Y., Inouye, M. Bacillus subtilis MazF-bs (EndoA) is a UACAU-specific mRNA interferase. FEBS Lett. 585, 2526-2532 (2011).
- Zhu, L., et al. et al.Staphylococcus aureus MazF specifically cleaves a pentad sequence, UACAU, which is unusually abundant in the mRNA for pathogenic adhesive factor SraP. J. Bacteriol. 191, 3248-3255 (2009).
- Zhu, L., et al. The mRNA interferases, MazF-mt3 and MazF-mt7 from Mycobacterium tuberculosis target unique pentad sequences in single-stranded RNA. Mol. Microbiol. 69, 559-569 (2008).
- Fu, Z., Donegan, N. P., Memmi, G., Cheung, A. L. Characterization of MazFSa, an endoribonuclease from Staphylococcus aureus. J. Bacteriol. 189, 8871-8879 (2007).
- Marmur, J. A procedure for the isolation of deoxyribonucleic acid from micro-organisms. J. Mol. Biol. 3, 208-218 (1961).
- Emory, S. A., Belasco, J. G. The ompA 5′ untranslated RNA segment functions in Escherichia coli as a growth-rate-regulated mRNA stabilizer whose activity is unrelated to translational efficiency. J. Bacteriol. 172, 4472-4481 (1990).
- Cole, S. T., Bremer, E., Hindennach, I., Henning, U. Characterisation of the promoters for the ompA gene which encodes a major outer membrane protein of Escherichia coli. Mol. Gen. Genet. 188, 472-479 (1982).
- Schleifer, K. H., Kilpper-Bälz, R., Devriese, L. Staphylococcus arlettae sp. nov., S. equorum sp. nov. and S. kloosii sp. nov.: Three New Coagulase-Negative, Novobiocin-Resistant Species from Animals. Syst. Appl. Microbiol. 5, 501-509 .
- Yu, H., Goodman, M. F. Comparison of HIV-1 and avian myeloblastosis virus reverse transcriptase fidelity on RNA and DNA templates. J. Biol. Chem. 267, 10888-10896 (1992).
- Ying, B. W., Fourmy, D., Yoshizawa, S. Substitution of the use of radioactivity by fluorescence for biochemical studies of RNA. RNA. 13, 2042-2050 (2007).

