Fluoreszenzbasierten Primer Extension-Technik, um Transkriptionsstartpunkte und Schnittstellen von RNasen Bestimmen<em> In Vivo</em
Summary
We here describe a fluorescence based primer extension method to determine transcriptional starting points from bacterial transcripts and RNA processing in vivo using an automated gel sequencer.
Abstract
Fluoreszenzbasierten Primer-Extension (FPE) ist ein Molekular Methode, um Transkriptionsstartpunkte oder Verarbeitungsstätten von RNA-Molekülen zu bestimmen. Dies wird durch die reverse Transkription der RNA von Interesse mit spezifischen fluoreszenzmarkierten Primern und anschließender Analyse der resultierenden cDNA-Fragmente durch denaturierende Polyacrylamid-Gelelektrophorese erreicht. Gleichzeitig wird eine traditionelle Sanger-Sequenzierungsreaktion auf dem Gel laufen gelassen, um die Enden der cDNA-Fragmente anzeigen, um deren genaue entsprechenden Basen. Im Gegensatz zu 5'-RACE (Rapid Amplification von cDNA-Enden), wobei das Produkt muss kloniert und sequenziert mehreren Kandidaten kann die Masse der cDNA-Fragmente durch Primerverlängerung erzeugt werden gleichzeitig in einem Gel laufen detektiert werden. Zusätzlich kann das gesamte Verfahren (von der reversen Transkription der endgültigen Analyse der Ergebnisse) an einem Arbeitstag abgeschlossen sein. Durch die Verwendung fluoreszierend markierter Primer, die Verwendung von gefährlichen radioaktiven Isotop markierten Reagenzienvermieden werden und die Bearbeitungszeit verringert werden als Produkte können während der Durchführung der Elektrophorese nachgewiesen werden.
In dem folgenden Protokoll beschreiben wir ein in vivo Fluoreszenz Primer-Extension-Methode auf das 5'-Ende der RNA schnell und zuverlässig zu erfassen, abzuleiten Transkriptionsstartpunkte und RNA-Verarbeitungsstellen (zB durch Toxin-Antitoxin-Systemkomponenten) in S. aureus, E. coli und anderen Bakterien.
Introduction
Primerverlängerungs 1 ist ein Molekular Methode, um die 5'-Enden der spezifischen RNA-Moleküle bis zu einer eine Basisauflösung bestimmen. Der Vorteil gegenüber anderen Verfahren, wie 5'-RACE (schnelle Amplifikation von cDNA-Enden) ist die schnelle Durchlaufzeit und die Fähigkeit, leicht zu analysieren, die eine Mischung von verschiedenen Längen von RNA-Molekülen.
Diese Methode funktioniert, indem RNA-Moleküle, die Transkription Reaktionen Reverse mit spezifischen fluoreszierenden Primer, Erzeugung cDNA-Fragmente bestimmter Längen. Diese cDNA-Moleküle werden neben der traditionellen Sanger-Sequenzierungsreaktionen 2 auf denaturierenden Polyacrylamid-Gelen und durch ihre Fluoreszenz aufgrund der Verwendung von fluoreszenzmarkierten Primern nachgewiesen werden. Die Längen der cDNA-Fragmente werden dann durch Vergleich mit der Sequenzierungsleiter beurteilt, so dass die Zuordnung der 5'-RNA-Ende.
Traditionell werden Primer-Verlängerungsreaktionen in Verbindung verwendet werdenmit radioaktiven Isotopen, um cDNA-Moleküle auf Röntgenfilmen zu erkennen. Wegen der Gesundheitsrisiken, Entsorgungsprobleme und der einfachen Handhabung, neuere Protokolle zu nutzen Fluoreszenz zum Nachweis der Primer-Extension mit automatisierten Sequenzer, wenn ihre Empfindlichkeit ist etwas niedriger. Mit fluoreszenzmarkierten Primern kann die wiederkehrende Radiomarkierungsverfahren verzichtet werden, als fluoreszierende Primer sind für eine lange Zeit (mehr als ein Jahr in unseren Händen) stabil.
Die Methode, die wir hier beschreiben, nutzt ein automatisiertes Gel Sequenzer, aber mit leichten Modifikationen können Kapillare Sequenzern auch für die cDNA Trennung und Detektion 3 verwendet werden. Die Parallelität der Gel-Analyse ermöglicht es, auch eine kleine Menge der RNA-Spaltung oder Verarbeitung zu erfassen. Ein weiterer Vorteil ist die hohe Auflösung dieses Verfahrens als terminale Spaltung oder Verarbeitung von nur einer Basis detektiert werden.
In Bezug auf den Nachweis von RNA-Spaltung oder Verarbeitung, typically zwei verschiedene Arten von Primer-Extensions unterschieden. In einem Fall wird die enzymatische Behandlung in vitro durchgeführt unter Verwendung gereinigter RNA und gereinigtes Enzym, wohingegen in dem anderen Fall, wird die Verarbeitung in vivo durchgeführt und die resultierende RNA wird gereinigt. In beiden Fällen wird die RNA unterzogen, um eine Primer-Extension durchgeführt in vitro, jedoch in Abhängigkeit von der Quelle der RNA wird das Verfahren entweder bezeichnet eine in vitro oder in vivo Primerverlängerung. In dem Protokoll wir hier konzentrieren wir uns lediglich auf der in-vivo-Primerverlängerung, aufgrund der Einfachheit der Nutzung (keine gereinigten Proteine notwendig) und die Möglichkeit zur Transkriptionsstartpunkte und die Verarbeitung in der gleichen Zeit zu bestimmen. Allerdings sind in vitro Primerverlängerungen im Prinzip die gleiche Weise eingestellt und dieses Protokoll kann als Ausgangspunkt dienen.
Das hier dargestellte Verfahren kann auf viele Bakterienarten, solange sie zugänglich sind hoch angewendetReinheit und High-Yield-Vorbereitung von Nukleinsäuren.
Die Forschung in unserem Labor konzentriert sich auf den Regelungsbereich von Toxin-Antitoxin (TA) Systeme 4,5, einem Bereich, in dem die Primer-Extension-Methode wird intensiv genutzt. TA-Systeme sind kleiner in prokaryotischen Genom, das aus einem stabilen und endogen aktive toxische Protein und einer meist instabil Protein oder RNA-Antitoxin, die Toxizität entgegen 6,7 bestehen vorliegenden genetischen Elemente. Toxinaktivität wird manchmal durch Hemmung der Replikation, der Zellwandsynthese oder andere Mechanismen, die von RNase-Aktivität 8,9 ausgeübt wird, aber meistens. Typischerweise wird RNase Spezifität durch die Durchführung verschiedener Tests, von denen der eine der Primer-Extension-Methode bestimmt. Primer-Verlängerungsreaktionen sind für diese Anwendung gut geeignet, da eine Mischung aus gespaltenen und in voller Länge Fragmente können gleichzeitig analysiert werden, um festzustellen ihren 5'-Enden. Verwendung einer Mischung von in vitro und in vivo Primerverlängerungen, diespezifische Toxin RNase-Spaltung, zB Sequenzspezifität bestimmt 10-13 werden.
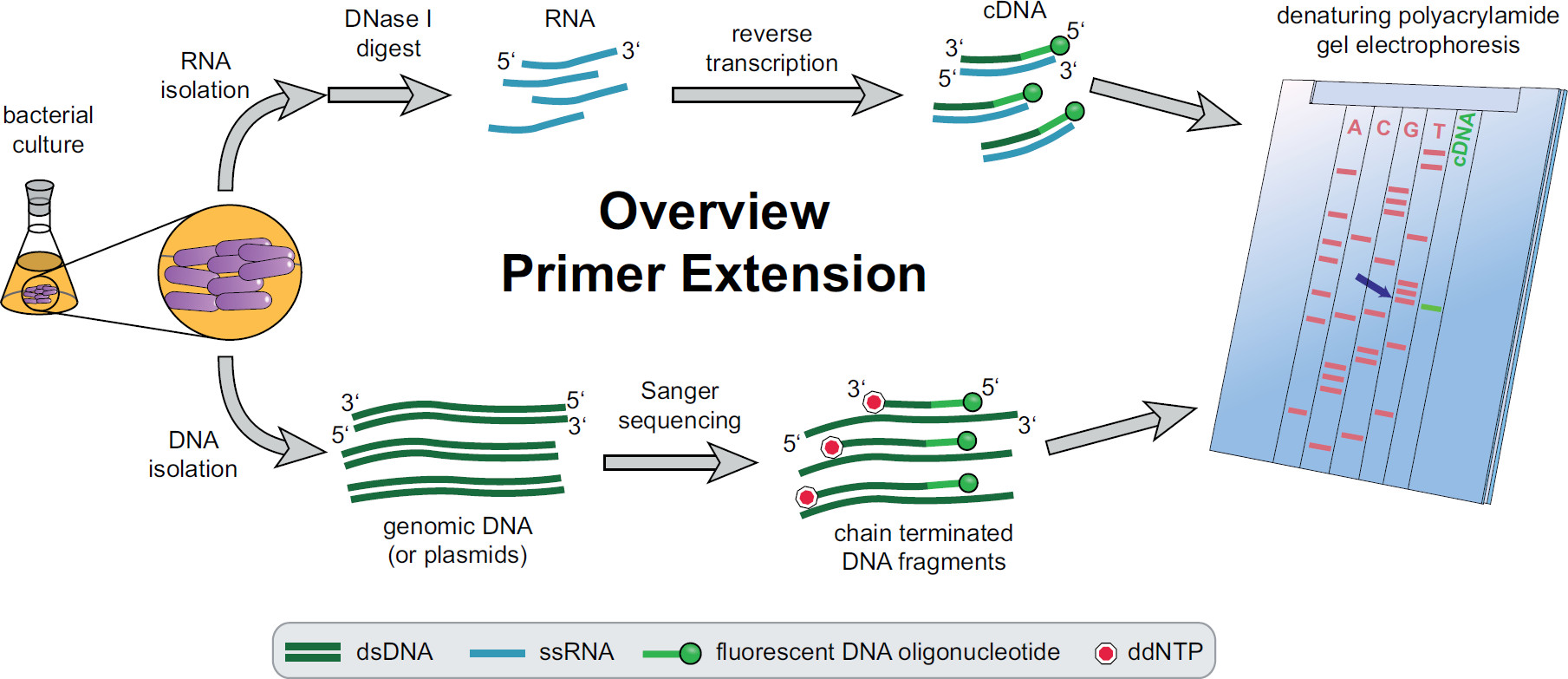
Abbildung 1. Übersicht der Primerverlängerungsverfahren. Bakterielle Kulturen werden nach den experimentellen Anforderungen inkubiert und behandelt. Gesamt-RNA wird aus den Zellen mit DNase I behandelt, um DNA-Spuren zu entfernen, und einer Umkehrtranskriptionsreaktion unter Verwendung von Ziel-DNA spezifischen Fluoreszenz Primer wodurch cDNA unterworfen extrahiert. Genomische DNA oder Plasmide extrahiert und anschließend zur Fluoreszenz Sanger-Sequenzierungsreaktionen zum Größenvergleich mit den cDNA-Fragmenten verwendet. Primerverlängerungsprodukte sind neben Sanger-Sequenzierungsprodukte auf einem denaturierenden Harnstoff-Polyacrylamidgel laufen und mit einem automatisierten Laser und Mikroskop analysiert. Die Sequenzierung Basis, die Linie mit dem cDNA-Band ist die last Basis des 5 'cDNA Ende (blauer Pfeil). Weitere Informationen im Fekete et al. 3 Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Eine Übersicht über den gesamten Primerverlängerungsverfahren kann in 1 gefunden werden. Kurz gesagt, kultiviert, geerntet sind bakterielle Zellen, das Zellpellet lysiert und die RNA extrahiert. Gereinigte RNA wird dann mit DNase I behandelt, um Spuren von DNA-Molekülen, die als Matrizen für die reverse Transkriptase wirken könnten, zu entfernen. Spezifische fluoreszierende Primer an die RNA gegeben, um die Region von Interesse hybridisiert und anschließend revers transkribiert, wodurch einzelsträngige komplementäre DNA (cDNA). Ein Sequenzierleiter wird von traditionellen Sanger-Sequenzierung erstellt die Verwendung von Fluoreszenz Primer und auf einem denaturierenden Polyacrylamid-Gel neben der Primerverlängerung cDNA-Fragmente getrennt. Das resultierendeGel wird durch den Vergleich der Fluoreszenzbanden, die eine Identifizierung des 5'-Enden von Interesse analysiert. Transkriptionsstartpunkte und Verarbeitungsstellen werden dann einzeln durch Sequenzvergleiche geprüft.
Protocol
Representative Results
Discussion
Fluoreszierende Primerverlängerung ist ein einfaches und schnelles Verfahren zur Bestimmung der 5'-Enden der RNAs, entweder für TSP- oder sekundäre RNA-Prozessierung Identifikation. Durch die Verwendung von fluoreszierenden Primern können die Umsetzungen eingerichtet werden und ausgeführt werden, ohne zusätzliche Sicherheitseinrichtungen (anders als im Fall von radioaktiv markierten Primern). Da die Proben durch Fluoreszenz detektiert, können sie abgebildet werden, während der Elektrophorese durchgeführt wi…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Anne Wochele for her assistance in the laboratory and Vera Augsburger for help with the automated gel sequencer. We thank the Deutsche Forschungsgemeinschaft for funding by grants BE4038/2 and BE4038/5 within the “priority programmes” SPP1316 and SPP1617.
Materials
| Name | Supplier | Catalog Number(s) | Comment |
| AMV Reverse Transcriptase (20-25 U/µl) | NEB / Roche | NEB: M0277-T / Roche: 10109118001 | |
| DNase I (RNase free) | Ambion (life technologies) | AM2222 | |
| FastPrep-24 Instrument | MPBio | 116004500 | |
| Fluorescently labeled primers | Biomers | n/a | 5’ DY-681 modification of “ordinary” DNA oligonucleotides. Compatible dyes such as the LICOR IRDye 700/800 are also available from other suppliers such as IDTdna. |
| Li-Cor 4200 Sequencer incl. ImagIR Data collection software | Li-Cor | Product discontinued | |
| NanoDrop 2000 | Thermo Scientific | ||
| Nuclease free water | Ambion (life technologies) | AM9915G | |
| Plasmid mini preparation kit | QIAGEN | 12125 | |
| RapidGel-XL-40% Concentrate | USB | US75863 | |
| RNA STORAGE BUFFER | Ambion (life technologies) | AM7000 | |
| Roti-Aqua-P/C/I | Carl Roth | X985.3 | Alternative: “Acid-Phenol:Chloroform, pH 4.5 (with IAA, 125:24:1)” from Ambion (AM9720) |
| SUPERase•In RNase Inhibitor | Ambion (life technologies) | AM2696 | |
| Thermo Sequenase fluorescently labelled primer cycle sequencing kit with 7-deaza-dGTP | GE Healthcare | RPN2538 | |
| TRIzol reagent | life technologies | 15596-026 | |
| Zirconia/Silica Beads 0.1 mm | BioSpec | 11079101z |
References
- Simpson, C. G., Brown, J. W. Primer extension assay. Methods Mol. Biol. 49, 249-256 (1995).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74, 5463-5467 (1977).
- Fekete, R. A., Miller, M. J., Chattoraj, D. K. Fluorescently labeled oligonucleotide extension: a rapid and quantitative protocol for primer extension. Biotechniques. 35, 90-94 (2003).
- Schuster, C. F., et al. Characterization of a mazEF Toxin-Antitoxin Homologue from Staphylococcus equorum. J. Bacteriol. 195, 115-125 (2013).
- Nolle, N., Schuster, C. F., Bertram, R. Two paralogous yefM-yoeB loci from Staphylococcus equorum encode functional toxin-antitoxin systems. Microbiology. 159, 1575-1585 (2013).
- Yamaguchi, Y., Park, J. H., Inouye, M. Toxin-antitoxin systems in bacteria and archaea. Annu. Rev. Genet. 45, 61-79 (2011).
- Schuster, C. F., Bertram, R. Toxin-antitoxin systems are ubiquitous and versatile modulators of prokaryotic cell fate. FEMS Microbiol. Lett. 340, 73-85 (2013).
- Goeders, N., Van Melderen, L. Toxin-antitoxin systems as multilevel interaction systems. Toxins. 6, 304-324 (2014).
- Yamaguchi, Y., Inouye, M. mRNA interferases, sequence-specific endoribonucleases from the toxin-antitoxin systems. Progress in molecular biology and translational science. 85, 467-500 (2009).
- Park, J. H., Yamaguchi, Y., Inouye, M. Bacillus subtilis MazF-bs (EndoA) is a UACAU-specific mRNA interferase. FEBS Lett. 585, 2526-2532 (2011).
- Zhu, L., et al. et al.Staphylococcus aureus MazF specifically cleaves a pentad sequence, UACAU, which is unusually abundant in the mRNA for pathogenic adhesive factor SraP. J. Bacteriol. 191, 3248-3255 (2009).
- Zhu, L., et al. The mRNA interferases, MazF-mt3 and MazF-mt7 from Mycobacterium tuberculosis target unique pentad sequences in single-stranded RNA. Mol. Microbiol. 69, 559-569 (2008).
- Fu, Z., Donegan, N. P., Memmi, G., Cheung, A. L. Characterization of MazFSa, an endoribonuclease from Staphylococcus aureus. J. Bacteriol. 189, 8871-8879 (2007).
- Marmur, J. A procedure for the isolation of deoxyribonucleic acid from micro-organisms. J. Mol. Biol. 3, 208-218 (1961).
- Emory, S. A., Belasco, J. G. The ompA 5′ untranslated RNA segment functions in Escherichia coli as a growth-rate-regulated mRNA stabilizer whose activity is unrelated to translational efficiency. J. Bacteriol. 172, 4472-4481 (1990).
- Cole, S. T., Bremer, E., Hindennach, I., Henning, U. Characterisation of the promoters for the ompA gene which encodes a major outer membrane protein of Escherichia coli. Mol. Gen. Genet. 188, 472-479 (1982).
- Schleifer, K. H., Kilpper-Bälz, R., Devriese, L. Staphylococcus arlettae sp. nov., S. equorum sp. nov. and S. kloosii sp. nov.: Three New Coagulase-Negative, Novobiocin-Resistant Species from Animals. Syst. Appl. Microbiol. 5, 501-509 .
- Yu, H., Goodman, M. F. Comparison of HIV-1 and avian myeloblastosis virus reverse transcriptase fidelity on RNA and DNA templates. J. Biol. Chem. 267, 10888-10896 (1992).
- Ying, B. W., Fourmy, D., Yoshizawa, S. Substitution of the use of radioactivity by fluorescence for biochemical studies of RNA. RNA. 13, 2042-2050 (2007).

