Floresan Bazlı Astar Uzatma Tekniği Transkripsiyonal Başlangıç Noktalarını ve RNases yarılması Siteleri belirleme<em> In Vivo</em
Summary
We here describe a fluorescence based primer extension method to determine transcriptional starting points from bacterial transcripts and RNA processing in vivo using an automated gel sequencer.
Abstract
Flüoresan göre primer uzatma (FPE) transkripsiyonel başlangıç noktalarının ya da RNA moleküllerinin işlenmesi bölgelerini belirlemek için moleküler bir yöntemdir. Bu özel floresanla işaretlenmiş primerler ve poliakrilamid jel elektroforezi ile denaturasyon ile elde edilen cDNA parçaları daha sonra analizi kullanılarak, ilgi konusu RNA'nın ters transkripsiyonu ile elde edilir. Aynı zamanda, geleneksel bir Sanger dizileme Reaksiyon bunların tam karşılık gelen tabanlarına cDNA fragmanlarının uçları eşlemek için jel üzerinde çalıştırılır. Ürün klonlanmış olması ve çok sayıda aday dizilenmiştir 5'-RACE (cDNA uçlarının hızlı büyütülmesi), aksine, primer uzama yolu ile üretilen bir cDNA fragmanlarının toplu eşzamanlı olarak bir jel vadede tespit edilebilir. Buna ek olarak, (sonuçların son analizde, ters transkripsiyon ile ilgili) bütün işlem bir iş günü içinde tamamlanabilir. Floresanla işaretlenmiş primerler kullanılarak, tehlikeli radyoaktif izotop kullanımı reaktifler etiketliönlenebilir ve ürünler elektroforez işlemi sırasında tespit edilebilir işlem süreleri azaltılır.
Aşağıdaki protokol, güvenilir ve hızlı S. (örneğin, toksin antitoksin sistem bileşenleri ile) bir başlangıç noktası ve RNA işleme the transkripsiyonel anlamak için RNA'ların 5 'uçlarını tespit etmek için, bir in vivo floresan primer uzatma yöntemi tarif S. aureus, E. E. coli ve diğer bakteriler.
Introduction
Primer uzatma 1 bir taban çözünürlüğe kadar özel bir RNA moleküllerinin 5 'ucunun saptanması için moleküler bir yöntemdir. 5'-RACE (cDNA uçlarının hızlı yükseltilmesi) gibi diğer yöntemlerin bir avantajı, hızlı bir geri dönüş süresi ve kolay bir RNA moleküllerinin farklı uzunluklarının bir karışımı analiz yeteneğidir.
Bu yöntem, RNA molekülleri, belirli uzunluktaki cDNA fragmanlarının üretilmesi, özel bir flüoresan primerler kullanılarak transkripsiyon reaksiyonlarını ters tabi tutarak çalışır. Bu cDNA molekülleri denatüre edici poliakrilamid jelleri üzerinde geleneksel Sanger dizileme reaksiyonlarının 2 yanına uygulanır ve bağlı floresan etiketli primerlerin kullanımı ile fluoresansları ile tespit edilebilir. CDNA fragmanlarının uzunlukları 5 'RNA uçlarının eşleşmesini sağlayan bir sıralama merdiven karşılaştırılarak değerlendirilir.
Geleneksel olarak, primer uzatma reaksiyonları bağlantılı olarak kullanılanradyoaktif izotoplar X-ray filmlerde cDNA molekülleri tespit etmek. Onların duyarlılığı biraz daha düşük de olsa dolayı sağlık tehlikeleri, atık bertaraf sorunları ve kullanım kolaylığı, yeni protokoller, otomatik dizicilerle ile primer uzatma tespiti için floresan kullanmaktadır. Floresan primerler (bizim elimizde bir yıldan daha fazla), uzun bir süre için kararlı olduğu gibi, floresanla işaretlenmiş primerler kullanılarak, tekrar eden radyo-etiketleme prosedürü, ihmal edilebilir.
Burada açıklandığı gibi bir yöntem otomatik bir jel sekanslayıcı kullanır, ancak küçük değişiklikler yapılarak, kapillerli da cDNA ayrılması ve keşfi 3 için kullanılabilir. Jel analizi paralel yapısı mümkün RNA bölünme ve işlem daha az miktarda tespit edilebilir. Diğer bir avantajı tespit edilebilir terminali bölünme ya da bir bazın işleme gibi, bu yöntemin yüksek çözünürlüktür.
RNA bölünme ya da işlem, t tespiti ile ilgili olarakprimer uzantıları ypically iki farklı tür ayırt edilir. Diğer bir durumda, işlem, in vivo olarak yapılması ve sonuçta elde edilen RNA saflaştırılmıştır ise Bir durumda, olan enzimatik işlem, saflaştırılmış RNA ve arıtılmış enzim kullanılarak in vitro olarak yapılır. Bir primer uzatma in vitro olarak, her iki durumda da RNA RNA kaynağına bağlı olarak, ancak, tabi tutulur, bu yöntem, bir in vitro veya in vivo olarak, primer genişlemesini, adlandırılır ya. Burada mevcut protokol, bunların kullanım kolaylığı (herhangi bir saflaştırılmış protein için) ve aynı zamanda kopyalama başlangıç noktaları ve belirlemek için olasılığı, sadece, in vivo bir primer uzatma odaklanır. Bununla birlikte in vitro primer uzantıları ile aynı şekilde ayarlanır, prensip olarak ve bu protokol, bir başlangıç noktası olarak görev yapabilir.
Burada gösterilen yöntem sürece yüksek giriş oranlı gibi pek çok bakteri türleri uygulanabilirNükleik asitlerin saflığı ve yüksek verimli hazırlanması.
Laboratuarımızda araştırma toksin antitoksin (TA-) sistemler 4,5, primer uzatma yöntemi yaygın olarak kullanıldığı bir alanın düzenleme kapsamında odaklanmaktadır. TA-sistemleri istikrarlı ve endojen aktif toksik protein ve toksisite 6,7 zıt bir çoğunlukla kararsız protein ya da RNA antitoksin oluşmaktadır prokaryotik genomları mevcut küçük genetik unsurlardır. Toksin aktivitesi bazen RNaz etkinliğinin 8,9 En sık replikasyonu, hücre çeperi sentezi veya başka mekanizmalar inhibisyonu tarafından uygulanan, ancak. Tipik haliyle, RNaz özgüllük primer uzatma yöntemdir biri farklı testler, iletken ile belirlenir. Parçalanır ve tam uzunlukta parçalarının bir karışımı, aynı zamanda, 5 'ucunun saptanması için analiz edilebilir Primer uzatma reaksiyonları, bu uygulama için uygundur. In vitro ve in vivo primeri uzantılarında bir karışımını kullanarak,spesifik toksin RNaz ayrılma, örneğin, sekans özgüllük 10-13 belirlenebilir.
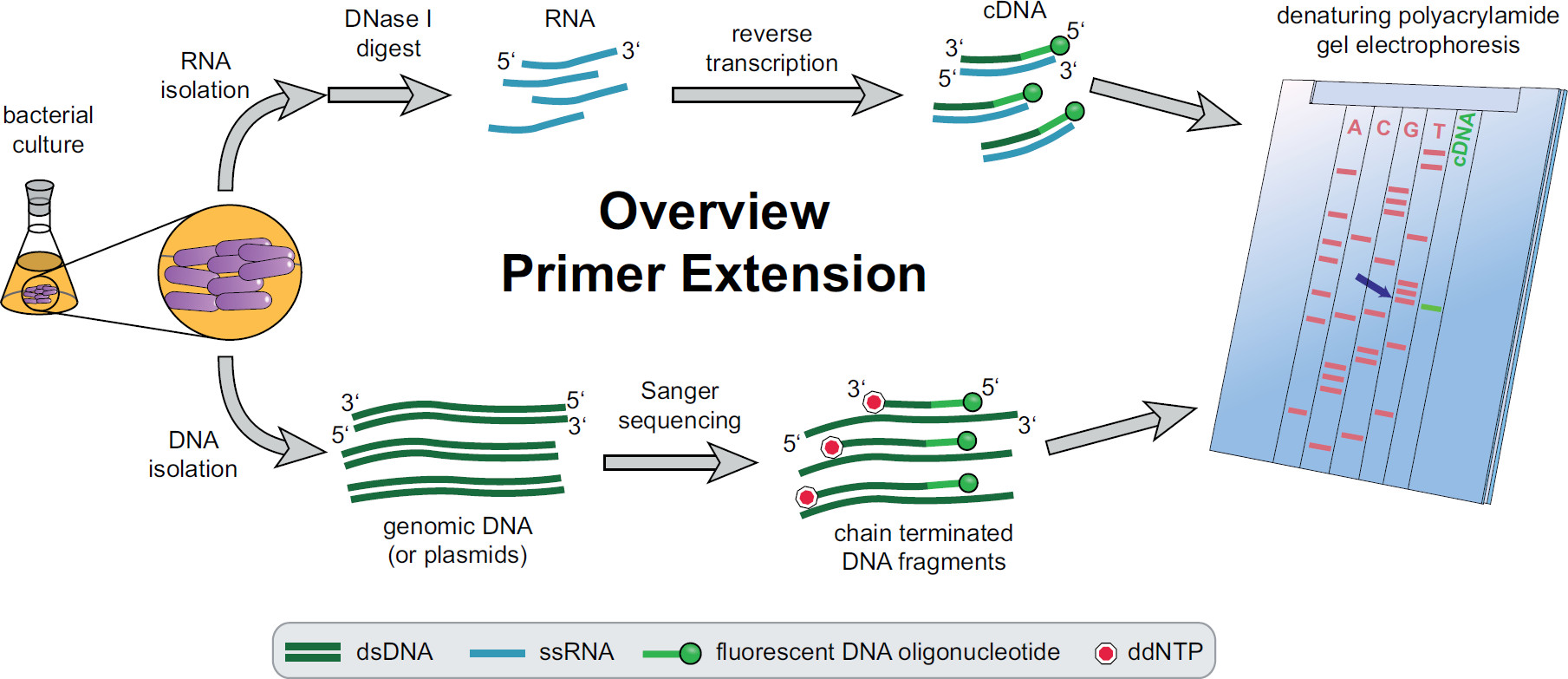
. Primer uzatma prosedürünün 1. Genel Bakış Şekil Bakteriyel kültürler deneysel ihtiyaçlarına göre kuluçkaya ve tedavi edilir. Toplam RNA, hücre cDNA elde edildi hedefe özgü floresan DNA primerler kullanılarak bir ters transkripsiyon reaksiyonu, DNA izlerini ortadan kaldırmak için DNase I ile işleme tabi tutuldu ve tabi elde edilir. Genomik DNA ya da plazmidler ekstre edilmiş ve daha sonra cDNA fragmanları ile boyut karşılaştırma için floresan Sanger dizileme reaksiyonları için kullanılır. Primer uzatma ürünleri denatüre üre poliakrilamid jel üzerinde Sanger dizileme ürünlerin yanında çalıştırmak ve otomatik lazer ve mikroskop ile analiz edilir. La cDNA bandı ile çizgiler kadar olan sıralama bazSt olarak 5 'cDNA ucu (mavi ok) tabanı. Fekete daha fazla bilgi, vd. 3 Bu rakamın büyük bir versiyonunu görmek için lütfen buraya tıklayınız.
Bütün primer uzatma işlemi ile ilgili bilgi, Şekil 1 'de bulunabilir. Kısaca, bakteri hücreleri, hasat edilir, kültürlenir, hücre topağı eritilir ve RNA ekstre edildi. Arındırılmış RNA daha sonra ters transkriptaz için şablon olarak hareket edebilir DNA moleküllerinin izlerini ortadan kaldırmak için DNase I ile işleme tabi tutulur. Özel flüoresan primerler ilgi bölgeye hibridize ve daha sonra tek sarmallı Tamamlayıcı DNA (cDNA) 'de elde edilen, ters transkribe edilmiş RNA ilave edilir. Bir sıralama merdiven floresan primer kullanılarak, geleneksel Sanger dizileme ile oluşturulan ve primer uzatma cDNA parçaları ile birlikte bir denatüre edici poliakrilamid jeli üzerinde ayrılır. Elde edilenJel ilgi 5 'uçlarının tanımlanmasına izin veren, floresan bantları karşılaştırılarak analiz edilir. Transkripsiyonal başlangıç noktaları ve işleme siteleri daha sonra sırası karşılaştırmaları tek tek değerlendirilir.
Protocol
Representative Results
Discussion
Floresan primer uzatma TSP- veya ikincil bir RNA işleme tanımlanması için iki, RNA'ların 5 'uçlarını belirlemek için basit ve hızlı bir yöntemdir. Dolayı floresan primerlerin kullanımı ile reaksiyonlar, ayarlanabilir ve (radyoaktif olarak etiketlenmiş primerler aksine olarak) ilave güvenlik önlemleri olmadan çalışabilir. Numuneler floresan ile tespit edilir elektroforez X-ışını filmleri yaygın olarak kullanılan radyoaktif yöntemlerle karşılaştırıldığında hızlı bir analizine …
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Anne Wochele for her assistance in the laboratory and Vera Augsburger for help with the automated gel sequencer. We thank the Deutsche Forschungsgemeinschaft for funding by grants BE4038/2 and BE4038/5 within the “priority programmes” SPP1316 and SPP1617.
Materials
| Name | Supplier | Catalog Number(s) | Comment |
| AMV Reverse Transcriptase (20-25 U/µl) | NEB / Roche | NEB: M0277-T / Roche: 10109118001 | |
| DNase I (RNase free) | Ambion (life technologies) | AM2222 | |
| FastPrep-24 Instrument | MPBio | 116004500 | |
| Fluorescently labeled primers | Biomers | n/a | 5’ DY-681 modification of “ordinary” DNA oligonucleotides. Compatible dyes such as the LICOR IRDye 700/800 are also available from other suppliers such as IDTdna. |
| Li-Cor 4200 Sequencer incl. ImagIR Data collection software | Li-Cor | Product discontinued | |
| NanoDrop 2000 | Thermo Scientific | ||
| Nuclease free water | Ambion (life technologies) | AM9915G | |
| Plasmid mini preparation kit | QIAGEN | 12125 | |
| RapidGel-XL-40% Concentrate | USB | US75863 | |
| RNA STORAGE BUFFER | Ambion (life technologies) | AM7000 | |
| Roti-Aqua-P/C/I | Carl Roth | X985.3 | Alternative: “Acid-Phenol:Chloroform, pH 4.5 (with IAA, 125:24:1)” from Ambion (AM9720) |
| SUPERase•In RNase Inhibitor | Ambion (life technologies) | AM2696 | |
| Thermo Sequenase fluorescently labelled primer cycle sequencing kit with 7-deaza-dGTP | GE Healthcare | RPN2538 | |
| TRIzol reagent | life technologies | 15596-026 | |
| Zirconia/Silica Beads 0.1 mm | BioSpec | 11079101z |
References
- Simpson, C. G., Brown, J. W. Primer extension assay. Methods Mol. Biol. 49, 249-256 (1995).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74, 5463-5467 (1977).
- Fekete, R. A., Miller, M. J., Chattoraj, D. K. Fluorescently labeled oligonucleotide extension: a rapid and quantitative protocol for primer extension. Biotechniques. 35, 90-94 (2003).
- Schuster, C. F., et al. Characterization of a mazEF Toxin-Antitoxin Homologue from Staphylococcus equorum. J. Bacteriol. 195, 115-125 (2013).
- Nolle, N., Schuster, C. F., Bertram, R. Two paralogous yefM-yoeB loci from Staphylococcus equorum encode functional toxin-antitoxin systems. Microbiology. 159, 1575-1585 (2013).
- Yamaguchi, Y., Park, J. H., Inouye, M. Toxin-antitoxin systems in bacteria and archaea. Annu. Rev. Genet. 45, 61-79 (2011).
- Schuster, C. F., Bertram, R. Toxin-antitoxin systems are ubiquitous and versatile modulators of prokaryotic cell fate. FEMS Microbiol. Lett. 340, 73-85 (2013).
- Goeders, N., Van Melderen, L. Toxin-antitoxin systems as multilevel interaction systems. Toxins. 6, 304-324 (2014).
- Yamaguchi, Y., Inouye, M. mRNA interferases, sequence-specific endoribonucleases from the toxin-antitoxin systems. Progress in molecular biology and translational science. 85, 467-500 (2009).
- Park, J. H., Yamaguchi, Y., Inouye, M. Bacillus subtilis MazF-bs (EndoA) is a UACAU-specific mRNA interferase. FEBS Lett. 585, 2526-2532 (2011).
- Zhu, L., et al. et al.Staphylococcus aureus MazF specifically cleaves a pentad sequence, UACAU, which is unusually abundant in the mRNA for pathogenic adhesive factor SraP. J. Bacteriol. 191, 3248-3255 (2009).
- Zhu, L., et al. The mRNA interferases, MazF-mt3 and MazF-mt7 from Mycobacterium tuberculosis target unique pentad sequences in single-stranded RNA. Mol. Microbiol. 69, 559-569 (2008).
- Fu, Z., Donegan, N. P., Memmi, G., Cheung, A. L. Characterization of MazFSa, an endoribonuclease from Staphylococcus aureus. J. Bacteriol. 189, 8871-8879 (2007).
- Marmur, J. A procedure for the isolation of deoxyribonucleic acid from micro-organisms. J. Mol. Biol. 3, 208-218 (1961).
- Emory, S. A., Belasco, J. G. The ompA 5′ untranslated RNA segment functions in Escherichia coli as a growth-rate-regulated mRNA stabilizer whose activity is unrelated to translational efficiency. J. Bacteriol. 172, 4472-4481 (1990).
- Cole, S. T., Bremer, E., Hindennach, I., Henning, U. Characterisation of the promoters for the ompA gene which encodes a major outer membrane protein of Escherichia coli. Mol. Gen. Genet. 188, 472-479 (1982).
- Schleifer, K. H., Kilpper-Bälz, R., Devriese, L. Staphylococcus arlettae sp. nov., S. equorum sp. nov. and S. kloosii sp. nov.: Three New Coagulase-Negative, Novobiocin-Resistant Species from Animals. Syst. Appl. Microbiol. 5, 501-509 .
- Yu, H., Goodman, M. F. Comparison of HIV-1 and avian myeloblastosis virus reverse transcriptase fidelity on RNA and DNA templates. J. Biol. Chem. 267, 10888-10896 (1992).
- Ying, B. W., Fourmy, D., Yoshizawa, S. Substitution of the use of radioactivity by fluorescence for biochemical studies of RNA. RNA. 13, 2042-2050 (2007).

