Overview
Fonte: Tilde Andersson1, Rolf Lood1
1 Departamento de Ciências Clínicas Lund, Divisão de Medicina de Infecção, Centro Biomédico, Universidade de Lund, 221 00 Lund, Suécia
Aparentemente impossível de determinar, a biodiversidade microbiana é verdadeiramente surpreendente com uma estimativa de um trilhão de espécies coexistindo (1,2). Embora climas particularmente severos, como o ambiente ácido do estômago humano (3) ou os lagos subglaciais da Antártida (4), possam ser dominados por uma espécie específica, as bactérias são tipicamente encontradas em culturas mistas. Como cada cepa pode influenciar o crescimento de outra (5), a capacidade de separar e cultivar colônias "puras" (consistindo apenas de um tipo) tornou-se essencial em ambientes clínicos e acadêmicos. Culturas puras permitem mais exames genéticos (6) e proteômicos (7), análise da pureza amostral e, talvez mais notável, a identificação e caracterização de agentes infecciosos a partir de amostras clínicas.
As bactérias têm uma ampla gama de requisitos de crescimento e existem inúmeros tipos de mídia de nutrientes projetadas para sustentar tanto as espécies não exigentes quanto as meticulosas (8). A mídia de crescimento pode ser preparada na forma líquida (como um caldo) ou em uma forma sólida tipicamente baseada em ágar (um agente de gelling derivado de algas vermelhas). Considerando que a inoculação direta no caldo traz o risco de gerar uma população bacteriana geneticamente diversa ou mesmo mista, chapeamento e re-streaking cria uma cultura mais pura onde cada célula tem uma composição genética altamente semelhante. A técnica da placa de raia baseia-se na diluição progressiva de uma amostra(Figura 1),com o objetivo de separar células individuais umas das outras. Qualquer célula viável (doravante referida como uma unidade formadora de colônias, CFU) sustentada pela mídia e ambiente designado pode posteriormente encontrar uma colônia isolada de células filhas através de fissão binária. Apesar das rápidas taxas de mutação dentro das comunidades bacterianas, esse grupo celular é geralmente considerado clonal. A colheita e re-streaking dessa população, consequentemente, garante que o trabalho subsequente envolva apenas um único tipo de bactéria.
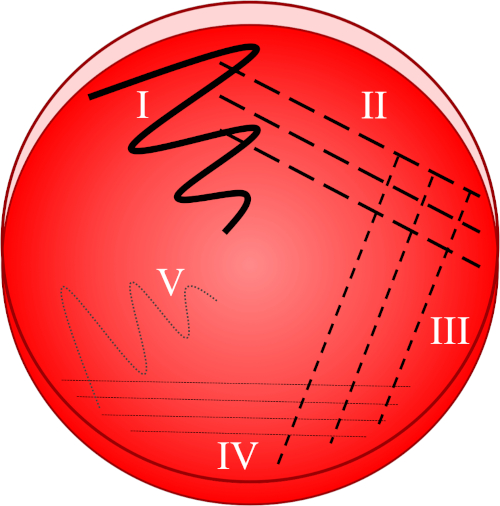
Figura 1: Uma placa de raia é baseada na diluição progressiva da amostra original. I) O inóculo é inicialmente disperso usando um movimento em zig-zag, criando uma área com uma população bacteriana relativamente densa. II-IV) As listras são extraídas da área anterior, usando um laço de inoculação estéril cada vez, até que o quarto quadrante seja atingido. V) Um movimento final em zigue-zague direcionado para o meio da placa forma uma região onde o inóculo foi marcadamente diluído, permitindo que as colônias apareçam separadas umas das outras.
A técnica da placa de raia também pode ser combinada com o uso de mídias seletivas e/ou diferenciais. Um meio seletivo inibirá o crescimento de certos organismos ( porexemplo, através da adição de antibióticos), enquanto um meio diferencial só ajudará a distinguir um do outro (por exemplo, através da hemolise em placas de ágar sanguíneo).
Por trás de todo o trabalho em microbiologia está o uso de técnicas assépticas (estéreis). Toda cultura bacteriana deve ser considerada potencialmente patogênica, pois há risco de crescimento não intencional de cepas traiçoeiras, formação de aerossol e contaminação de equipamentos/pessoal. Para minimizar esses riscos, todas as mídias, plásticos, metal e vidro são tipicamente esterilizados através de autoclaving antes e depois do uso, submetendo-os a vapor saturado de alta pressão em torno de 121°C que efetivamente elimina quaisquer células persistentes. O espaço de trabalho é geralmente desinfetado usando etanol antes e depois do uso. Jaleco e luvas são sempre usados durante o trabalho com agentes infecciosos.
Procedure
1. Configurar
- Todos os micróbios devem ser tratados como se fossem perigosos. Use sempre um jaleco e luvas, amarre os cabelos longos e certifique-se de que todas as feridas estejam particularmente bem protegidas.
- Prepare o espaço de trabalho esterilizando-o usando 70% de etanol.
- Certifique-se de que as placas de ágar, a solução de amostra(s) e uma caixa de laços de inoculação de plástico pré-esterilizados ou uma alça metálica mais uma chama bunsen, estão próximas. Alças de plástico descartáveis são tipicamente pré-esterilizadas. As alças metálicas devem ser mergulhadas em 70% de etanol, e depois mantidas perto da área azul de uma chama bunsen e aquecidas até que aqueçam fogo. Deixe o fio esfriar levantando a tampa da placa (apenas ligeiramente para evitar contaminação) e batendo-o contra o meio solidificado.
- Finalize cada procedimento com uma esterilização repetida do espaço de trabalho e uma lavagem/esterilização completa das mãos e pulsos.
2. Protocolo
- Preparação de mídia
- Identifique e prepare um meio sólido (tipicamente contendo 1,5% (p/v) ágar) que sustentará as espécies/cepas bacterianas utilizadas. Misture o meio em uma garrafa capaz de segurar o dobro do volume final para evitar o estouro ao autoclavar.
- Esterilize a mídia colocando a garrafa, com uma tampa semi-apertada, em uma autoclave fixada em 121°C por 20 min.
- Feche a tampa corretamente assim que a garrafa for removida da autoclave. Se a mídia for usada em breve, coloque a garrafa em um banho de água definido para 45°C para preservá-la em um estado líquido. O ágar se solidificará a nada menos que 32-40°C, e mais tarde pode ser reaquecido (normalmente usando um micro-ondas) a um ponto de fusão a 85°C.
- Preparação de placas de cultura
- Marque a base de placas estéreis de Petri (tipicamente 100 x 15 mm) na lateral ou na parte inferior com o nome do experimentador, a data e o tipo de mídia.
- Despeje 20-25 mL de 45°C de cultura de ágar (previamente preparado) em cada uma das placas rotuladas. Caso a espuma apareça ao longo das bordas, esta deve ser rapidamente removida usando uma pipeta regular e uma ponta estéril.
- Coloque imediatamente todas as tampas de volta nos pratos para evitar contaminação.
- Deixe o ágar solidificar por aproximadamente 2h em temperatura ambiente ou durante a noite a 4°C. Uma vez definidas, as placas de cultura bacteriana devem ser armazenadas de cabeça para baixo a 4°C para minimizar a condensação na superfície média.
- Chapeamento de raias
- Submerque um laço estéril no inóculo desejado e disperse imediatamente a amostra coletada no primeiro quadrante da placa usando um movimento zig-zag(Figura 1, I).
- Feche a tampa e reestericione o laço de inoculação ou colete um novo laço descartável estéril.
- Faça 3-4 traçados irradiando do primeiro quadrante (contendo uma população bacteriana relativamente densa) em direção ao segundo quadrante da placa(Figura 1, II).
- Feche a tampa e reestericione o laço de inoculação ou descarte o laço descartável e colete um novo estéril.
- Repita esta raia de 3-4 tacadas do segundo para o terceiro quadrante, e depois do terceiro ao quarto quadrante, usando um laço estéril cada vez(Figura 1, III - IV).
- Usando um laço estéril, faça um último golpe em um padrão zig-zag do quarto quadrante em direção ao meio da placa(Figura 1, V). A prevalência bacteriana será menor nesta área, permitindo idealmente que colônias individuais sejam estabelecidas a partir de uma única célula-mãe viável.
- Feche a tampa e (se necessário pelas espécies bacterianas) sela com parafilme para evitar o fluxo de ar.
- Dependendo da espécie/cepa bacteriana, coloque a placa de cultura de lado para baixo em um ambiente adequado e incubar até que colônias bacterianas sejam visíveis (colônias segregadas podem aparecer em qualquer área da placa, pois a concentração inicial pode variar).
- Para gerar uma população bacteriana clonal, escroto outra placa, trocando o inóculo banhado na placa original por células isoladas de uma única colônia da placa original.
Em uma placa de Petri, se uma única bactéria sofrer múltiplas rodadas de reprodução assexuada, levará à formação de uma colônia clonal. No entanto, obter uma única bactéria de uma amostra mista, como uma suspensão do solo, pode ser difícil. Se um loopful desta cultura heterogênea é tomado, ele pode conter até um trilhão de bactérias individuais. Para espalhar tantas bactérias para a superfície de uma placa de ágar e obter uma única colônia, mesmo usando um padrão zig-zag, o laço precisaria ser arrastado continuamente sobre a superfície de placas suficientes definidas lado a lado para cercar toda a ilha da Liberdade. Obviamente, os cientistas não usam tantas placas. Em vez disso, eles usam uma técnica chamada chapeamento de raia.
A técnica da placa de raia é baseada na diluição progressiva de uma amostra bacteriana, e é realizada sobre a superfície de mídia sólida de uma única placa de Petri. Para começar, a superfície da mídia é visualmente dividida em cinco seções, atribuindo quatro fragmentos da circunferência como as quatro primeiras seções, e o centro da placa como o quinto. Isso efetivamente criará cinco placas de mídia a partir de uma única placa de Petri. Em seguida, usando um loopful de inóculo desejado, a primeira seção é listrada usando um padrão zig-zag. Em seguida, ou um novo laço descartável é usado, ou no caso de um laço de fio, ele é esterilizado com um queimador Bunsen, flamejando-o até que esteja vermelho quente ao longo do comprimento do fio. Este uso de um novo laço, ou laço esterilizador de chama, remove todas as células bacterianas restantes, auxiliando na diluição das bactérias. O laço quente é então resfriado no ar por alguns segundos antes de ser arrastado pela primeira seção para criar três a quatro linhas separadas, cada uma carregando apenas uma fração de bactérias para a segunda seção. As seções restantes são listradas da mesma maneira, usando um laço estéril cada vez, e uma única passagem através da raia anterior.
Usando este ciclo de listras e esterilização, a concentração bacteriana em cada seção subsequente deve ser diluída de modo que a seção final contenha apenas algumas bactérias discretamente localizadas. Após a incubação, essas bactérias discretas se multiplicam para produzir colônias clonais isoladas de células filhas, que são chamadas de Unidades formadoras de colônias, ou UFC. Estes podem ser colhidos e re-listrados para garantir que o trabalho subsequente envolva apenas um único tipo bacteriano, referido como uma cultura pura. Além de isolar colônias únicas de uma cultura mestideira, a técnica de chapeamento de raias também é usada para selecionar cepas específicas da mídia, determinar morfologia de colônias bacterianas ou identificar diferentes espécies bacterianas. Neste vídeo, demonstraremos como isolar colônias unibacterianas de uma suspensão de amostra de amostra de amostra de bactérias mistas através da técnica de chapeamento de listras.
Para começar, coloque luvas de laboratório e um jaleco. Em seguida, esterilize o espaço de trabalho usando 70% de etanol. Em seguida, selecione um meio adequado que sustente as espécies bacterianas ou cepas utilizadas e comece a preparar a mídia. Aqui, o ágar LB comum é preparado pesando dez gramas de mídia pré-formulada, em pó e 7,5 gramas de ágar. Adicione os componentes pesados e secos a uma garrafa de vidro que é capaz de segurar o dobro do volume final para evitar o transbordamento. Em seguida, adicione 500 mililitros de água à garrafa, e tampe-a semi-firmemente. Esterilize a mídia colocando a garrafa em uma autoclave fixada a 121 graus Celsius por vinte minutos. Após a conclusão, use luvas à prova de calor ou uma almofada quente para remover a mídia da máquina e, em seguida, imediatamente torça a tampa da garrafa para fechá-la firmemente.
Para o uso do mesmo dia, deixe a mídia esfriar colocando a garrafa em um banho de água aquecido a aproximadamente 45 graus Celsius, para preservar a mídia em um estado líquido. Alternativamente, a mídia pode ser deixada à temperatura ambiente para armazenar em estado sólido. Quando necessário, micro-ondas a garrafa com a tampa ligeiramente aberta para derreter a mídia, e permitir que a mídia esfrie usando um banho de água de 45 graus Celsius.
Em seguida, pegue uma manga de pratos petri estéreis, e com um marcador permanente, rotule-os com o investigador e nomes da mídia, bem como a data. Em seguida, transfira o volume necessário de mídia para um vaso estéril e adicione antibióticos ou outros componentes sensíveis, se necessário. Aqui, 50 mililitros de mídia são misturados com 100 microliters de Kanamycin para uma concentração final de 25 microgramas por mililitro. Gire o tubo para garantir a distribuição uniforme dos componentes adicionados em toda a mídia. Lentamente, de modo a evitar a formação de bolhas, despeje de 20 a 25 mililitros de aproximadamente 45 graus Celsius de cultura média em cada uma das placas. Se aparecerem bolhas ou espuma, remova rapidamente usando uma pipeta regular e uma ponta estéril. Em seguida, substitua imediatamente todas as tampas para evitar contaminação. Deixe o ágar solidificar à temperatura ambiente por pelo menos duas horas ou durante a noite. Uma vez solidificado, armazene as placas de cultura de cabeça para baixo a quatro graus Celsius para minimizar a condensação na superfície do meio.
Para ver a cultura da escolha, primeiro pegue uma placa de cultura limpa e remova a tampa. Trabalhando rapidamente, submergir um laço descartável e estéril no inóculo desejado e, em seguida, imediatamente esfregar o laço sobre o primeiro quadrante da placa usando um movimento zig-zag. Substitua a tampa do prato, descarte o laço de inoculação usado e selecione um novo laço estéril. Usando o novo loop, faça de três a quatro golpes cruzando a linha original do cotonete irradiando do primeiro quadrante, que deve conter uma população relativamente densa de bactérias no segundo quadrante. Feche a tampa mais uma vez e descarte o laço. Com um novo loop, repita esta ação novamente, mas desta vez se espalhando do segundo para o terceiro quadrante. Em seguida, com um novo loop novamente, faça outra raia do terceiro para a quarta seção da placa. Finalmente, com um loop fresco, faça um último golpe em um padrão zig-zag do quarto quadrante em direção ao centro da placa. A prevalência bacteriana será menor nesta área, permitindo idealmente que colônias individuais sejam estabelecidas a partir de uma única célula materna viável.
Substitua a tampa da placa e, se for o caso, para as espécies bacterianas, sele a placa com para película para evitar o fluxo de ar. Vire a placa de cultura de cabeça para baixo para evitar gotejamentos de condensação e, em seguida, coloque em uma temperatura adequada para o crescimento. Aqui, uma incubadora está programada para 37 graus Celsius. Deixe a placa incubar até que as colônias bacterianas sejam visíveis. Para gerar uma população bacteriana clonal, selecione uma colônia discreta desta placa. Agora, com o laço estéril, toque na colônia alvo, e como antes, faça uma raia no primeiro quadrante de uma nova placa. Continue esterilizando alternadamente o laço e listrar os quadrantes restantes da placa como demonstrado anteriormente, terminando com o zig-zague para o centro. Feche a placa e coloque-a para incubar até formar colônias discretas. Uma vez que essas colônias são cultivadas, elas normalmente representarão cepas clonais puras.
A placa de listras iniciais pode conter colônias originárias de células de diferentes espécies bacterianas ou células com diferentes maquiagens genéticas, dependendo da pureza da amostra. Através do isolamento subsequente de uma única colônia, onde todas as unidades são derivadas de uma célula-mãe comum, o segundo procedimento de listras gera uma população bacteriana relativamente clonal, adequada para caracterização ou inoculação em caldo.
Subscription Required. Please recommend JoVE to your librarian.
Results
A placa de listras inicial pode conter colônias originárias de células com diferentes maquiagens genéticas ou (dependendo da pureza da amostra) de diferentes espécies bacterianas(Figura 2A).
Através do isolamento subsequente de uma única colônia, onde todas as unidades são derivadas de uma célula-mãe comum, o segundo procedimento de estria gera uma população bacteriana relativamente clonal, adequada para caracterização ou inoculação adicional em caldo(Figura 2B).

Figura 2: Uma cultura pura pode ser gerada a partir de uma amostra mista através do isolamento de uma única colônia isolada. A) O crescimento de uma única célula bacteriana (UFC) gerou uma colônia clonal, separada das de outras espécies e cepas. Esta UFC foi usada para posterior estiliar em uma nova placa B) Uma segunda placa, onde a população bacteriana consiste unicamente de células derivadas da UFC inicial.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
A capacidade de obter e cultivar uma colônia bacteriana pura é essencial, tanto em ambientes clínicos quanto acadêmicos. O revestimento de raia permite o isolamento de uma população celular relativamente clonal, originária de uma UFC compartilhada, que pode ser de particular interesse durante o diagnóstico ou para caracterização adicional do isolado. Uma amostra é listrada em um meio de nutrientes à base de ágar adequado e incubada até que as colônias se tornem visíveis. Uma colônia isolada é posteriormente colhida e re-listrada em uma segunda placa.
Subscription Required. Please recommend JoVE to your librarian.
References
- The Human Microbiome Project C. Structure, Function and Diversity of the Healthy Human Microbiome. Nature. 486:207-214. (2012)
- Locey KJ, Lennon JT. Scaling laws predict global microbial diversity. Proceedings of the National Academy of Sciences. 113 (21) 5970-5975 (2016)
- Skouloubris S, Thiberge JM, Labigne A, De Reuse H. The Helicobacter pylori UreI protein is not involved in urease activity but is essential for bacterial survival in vivo. Infection and Immunity. 66:4517-21. (1998)
- Mikucki JA, Auken E, Tulaczyk S, Virginia RA, Schamper C, Sørensen KI, Doran PT, Dugan H, Foley N. Deep groundwater and potential subsurface habitats beneath an Antarctic dry valley. Nature Communications. 6:6831. (2015)
- Mullineaux-Sanders C, Suez J, Elinav E, Frankel G. Sieving through gut models of colonization resistance. Nature Microbiology. 3:132-140. (2018)
- Fournier PE, Drancourt M, Raoult D. Bacterial genome sequencing and its use in infectious diseases. Lancet Infectious Diseases. 7:711-23 (2007)
- Yao Z, Li W, Lin Y, Wu Q, Yu F, Lin W, Lin X. Proteomic Analysis Reveals That Metabolic Flows Affect the Susceptibility of Aeromonas hydrophila to Antibiotics. Scientific Reports. 6:39413 (2016)
- Medina D, Walke JB, Gajewski Z, Becker MH, Swartwout MC, Belden LK. Culture Media and Individual Hosts Affect the Recovery of Culturable Bacterial Diversity from Amphibian Skin. Frontiers in Microbiology. 8:1574 (2017)




