Overview
Quelle: Tilde Andersson1, Rolf Lood1
1 Institut für Klinische Wissenschaften Lund, Abteilung für Infektionsmedizin, Biomedical Center, Universität Lund, 221 00 Lund, Schweden
Viren, die prokaryotische Organismen infizieren, bakteriopische oder einfach Phagen genannt, wurden Anfang des20. Jahrhunderts durch Twort (1) und d'Hérelle (2) unabhängig voneinander identifiziert. Phages sind seitdem weithin anerkannt für ihren therapeutischen Wert (3) und ihren Einfluss auf den Menschen (4) sowie globale Ökosysteme (5). Die derzeitigen Bedenken haben ein erneutes Interesse an der Verwendung von Phagen als Alternative zu modernen Antibiotika bei der Behandlung von Infektionskrankheiten geschürt (6). Im Wesentlichen beruht die gesamte Phagenforschung auf der Fähigkeit, Viren zu reinigen und zu quantifizieren, auch als viraler Titer bekannt. Ursprünglich 1952 beschrieben, war dies der Zweck des Plaque-Assays (7). Jahrzehnte und mehrere technologische Fortschritte später, bleibt der Plaque-Assay eine der zuverlässigsten Methoden zur Bestimmung von viralem Titer (8).
Bakteriophagen erbestehen, indem sie ihr genetisches Material in Wirtszellen injizieren, die Maschinen für die Produktion neuer Phagenpartikel entführen und schließlich dazu führen, dass der Wirt zahlreiche Nachkommenvirionen durch Zelllyse freisetzt. Aufgrund ihrer winzigen Größe können Bakteriophagen nicht allein mit der Lichtmikroskopie beobachtet werden; Daher ist eine Rasterelektronenmikroskopie erforderlich (Abbildung 1). Darüber hinaus können Phagen nicht auf Ernährungs-Agar-Platten wie Bakterien kultiviert werden, da sie Wirtszellen benötigen, um Beute zu machen.
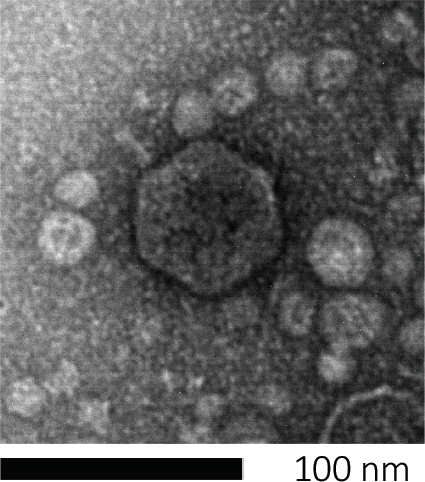
Abbildung 1: Die Morphologie eines Bakteriophagens, hier am Beispiel eines E. coli-Phagens, kann mit Derkelektronenmikroskopie untersucht werden. Die meisten Bakteriophagen gehören zu Caudoviralen (tailed bacteriophages). Dieser besondere Phagen hat eine sehr kurze Schwanzstruktur und einen Ikosaederkopf, der ihn in die Familie der Podoviren eingibt.
Der Plaque-Assay (Abbildung 2) basiert auf der Einbindung von Wirtszellen, bevorzugt im Logphasenwachstum, in das Medium. Dies schafft eine dichte, trübe Schicht von Bakterien in der Lage, virales Wachstum zu erhalten. Ein isolierter Phagen kann anschließend eine Zelle infizieren, replizieren und lysieren. Mit jeder lysierten Zelle werden mehrere benachbarte Zellen sofort infiziert. Mehrere Zyklen in, eine klare Zone (eine Plaque) kann in der ansonsten trüben Platte beobachtet werden (Abbildung 2B/Abbildung 3A), was auf das Vorhandensein eines anfangs einzelnen Bakteriophagenpartikels hinweist. Die Anzahl der Plaqueformeinheiten pro Volumen(d.h. PFU/ml) einer Probe kann somit anhand der Anzahl der erzeugten Plaques bestimmt werden.
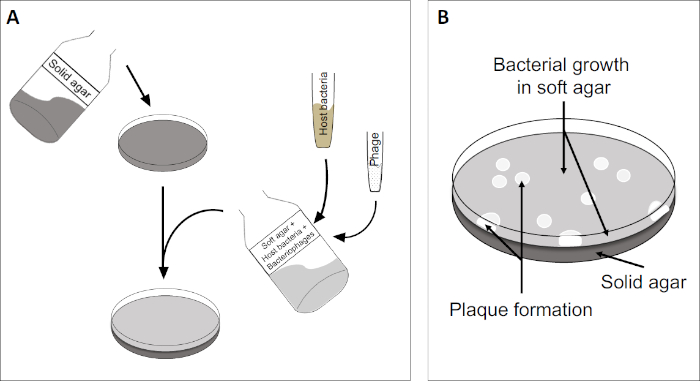
Abbildung 2: Tests auf Plaqueforming Units (PFU) ist eine gängige Methode zur Bestimmung der Anzahl der Bakteriophagen in einer Probe. (A) Die Basis einer sterilen Petrischale ist mit einem geeigneten festen Nährmedium bedeckt, gefolgt von einer Mischung aus weichen Medien, anfälligen Wirtszellen und einer Verdünnung der ursprünglichen Bakteriophagenprobe. Beachten Sie, dass die Phagensuspension in einigen Fällen auch gleichmäßig über die Oberfläche des bereits erstarrten weichen Agars verteilt werden könnte. (B) Das Wachstum der Wirtsbakterien bildet einen Rasen von Zellen in der oberen Agarschicht. Die Bakteriophagenreplikation erzeugt klare Zonen oder Plaques, die durch die Leierzelllyse verursacht werden.
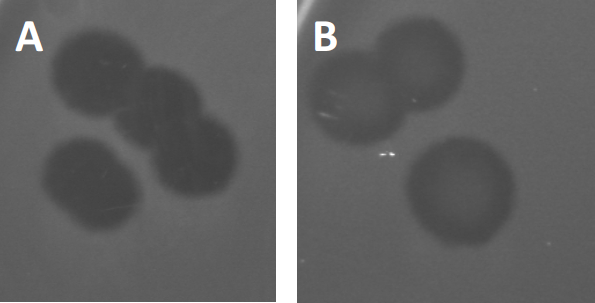
Abbildung 3: Die Ergebnisse der PFU-Tests zeigen mehrere Plaques, die durch Bakteriophagen erzeugt werden. Aufgrund der Lyse empfänglicher Wirtszellen können Plaques als Clearingzonen im bakteriellen Rasen angesehen werden, entweder mit (A) voller Clearance oder (B) teilweisem Nachwachsen, das durch die Erzeugung resistenter Bakterien (oder möglicherweise durch gemäßigte Phagen in der lysogene Zyklus).
Bestimmte gemäßigte Phagen können zusätzlich zu dem früher beschriebenen lytischen Wachstum einen sogenannten lysogenen Lebenszyklus annehmen. In der Lysogenesie nimmt das Virus einen latenten Zustand durch DieInintegration seines genetischen Materials in das Genom der Wirtszelle (9) an, was häufig Resistenzen gegen weitere Phageninfektionen verleiht. Dies zeigt sich manchmal durch eine leichte Trübung der Plaque (Abbildung 3B). Es ist jedoch erwähnenswert, dass Plaques auch durch das Wiederwachstum von Bakterien, die Resistenz gegen den Phagen unabhängig von früheren Phageninfektionen entwickelt haben, verschwommen erscheinen können.
Viren können nur an eine begrenzte Anzahl von Wirtsbakterien anhängen oder adsorbieren (10). Die Wirtsbereiche werden durch intrazelluläre antivirale Strategien wie das CRISPR-Cas-System (11) weiter begrenzt. Die Resistenz/Empfindlichkeit gegenüber bestimmten Phagen, die von bakteriellen Untergruppen angezeigt werden, wurde in der Vergangenheit verwendet, um Bakterienstämme in verschiedene Phagentypen zu kategorisieren (Abbildung 4). Obwohl die Wirksamkeit dieser Methode nun durch neuartige Sequenzierungstechniken übertroffen wurde, kann die Phagentypisierung dennoch wertvolle Informationen über Bakterien-Phagen-Wechselwirkungen liefern, z. B. was die Entwicklung eines Phagencocktails für den klinischen Einsatz erleichtert. .
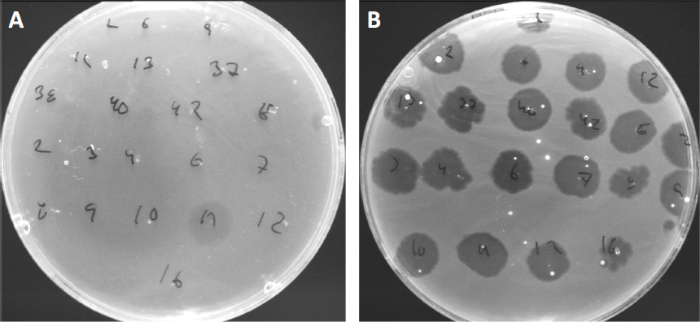
Abbildung 4: Phage-Empfindlichkeit verschiedener Bakterienstämme. Weiche Agarplatten mit Cutibacterium acnes Stamm (A) AD27 und (B) AD35, wurden mit 21 verschiedenen C. acnes Bakteriophagen gesichtet. Nur Phagen 11 waren in der Lage, AD27 zu infizieren und zu töten, während Stamm AD35 Empfindlichkeit gegenüber allen Phagen zeigte. Diese Technik, die als Phagentypisierung bezeichnet wird, kann verwendet werden, um Bakterienarten und Stämme in verschiedene Untergruppen zu unterteilen, basierend auf der Phagenanfälligkeit.
Procedure
1. Einrichtung
- Bevor Sie mit einer Arbeit mit Mikroben zu tun haben, stellen Sie sicher, dass der Arbeitsraum sterilisiert ist(z. B. mit 70% Ethanol abgewischt). Tragen Sie immer einen Labormantel und Handschuhe, halten Sie lange Haare zurück gebunden, und stellen Sie sicher, dass alle Wunden besonders gut geschützt sind.
- Nach fertigstellungen Jahren alle Oberflächen sterilisieren und Hände und Handgelenke gründlich waschen/sterilisieren.
2. Protokoll
- LB Medienvorbereitung
Hinweis: Je nach dem wirtsbakteriellen Stamm und dem Bakteriophagen kann ein anderes flüssiges Medium für die anfängliche Kultivierung des Wirts-Bakterienstamms besser geeignet sein oder ein anderes festes Medium für das spätere Wachstum besser geeignet sein. Lysogenese Brühe (LB) wird in diesem Protokoll für die Brühe und den Agar verwendet.- Mischen Sie 4 g LB in 200 ml destilliertem Wasser, in Dreifacher Ausführung, für die LB-Brühe, den festen Unteren Agar und den weichen Top-Agar. Alle Lösungen sollten in Containern vorbereitet werden, die das Doppelte des Endvolumens aufnehmen können, um einen Überlauf beim Autoklavieren zu verhindern.
Hinweis: Wenn Sie den Test in dreifacher Ausfertigung durchführen, bereiten Sie die doppelte Menge an LB-Bodenagar vor. - Stellen Sie den pH-Wert aller drei Lösungen mit NaOH oder HCl entsprechend auf 7,4 ein.
- Fügen Sie 3 g Agar-Power auf die untere Agar-Flasche, um eine 1,5% Agar-Lösung für den festen Agar zu machen
- Fügen Sie 1,2 g Agar-Power in die obere Agar-Flasche, um eine 0,6% Agar-Lösung für weiche Agar zu machen.
- Legen Sie die Flaschen mit halbfestangezogenen Kappen in einen Autoklaven, der auf 121°C für 20 min eingestellt ist, um die Lösungen zu sterilisieren. Schließen Sie die Kappen, sobald der Lauf beendet ist, um eine Kontamination zu verhindern.
- Wenn das LB-Medium eine Temperatur von ca. 45-50°C erreicht hat, fügen Sie 450 l sterile 1 M CaCl2 zu allen drei der 200 ml LB-Lösungen hinzu, um eine Endkonzentration von 2,25 mM zu erreichen.
- 15 ml Aliquots des festen Agarmediums in sterile Petrischalen gießen (Schütteln vermeiden, um Schaumbildung zu verhindern) und lassen Sie den Agar für ein paar Stunden oder über Nacht bei Raumtemperatur erstarren (Abbildung 4A). Platten können bei 4°C mehrere Tage auf dem Kopf gelagert werden.
- Mischen Sie 4 g LB in 200 ml destilliertem Wasser, in Dreifacher Ausführung, für die LB-Brühe, den festen Unteren Agar und den weichen Top-Agar. Alle Lösungen sollten in Containern vorbereitet werden, die das Doppelte des Endvolumens aufnehmen können, um einen Überlauf beim Autoklavieren zu verhindern.
- Kultivieren von Wirtszellen
- Einen Tag vor dem Test 10 L E. coli-Kultur zu 10 ml LB-Brühe hinzufügen.
- Inkubieren Sie die Bakterien bei 37°C über Nacht bei 160 Rpm in einem Schüttelbrutzentrum.
- Am Morgen des Assays, fügen Sie 0,5 ml der Nachtkultur zu 10 ml frische LB.
Hinweis: Wenn Sie den Test in dreifacher Ausfertigung durchführen, bereiten Sie die doppelte Menge dieser Kultur vor. - Inkubieren Sie die bei 37°C bei 160 Rpm in einem Schüttelinkubator, bis sich die Bakterienkultur im Wachstum der Logphase befindet. Dies kann spektrophotometrisch durch eine OD600 von 0,5-0,7 bestimmt werden.
- Halten Sie die Kultur bei Raumtemperatur, bis die Bakterien dem oberen LB-Agar hinzugefügt werden sollen.
- 10-fache serielle Verdünnung von Bakteriophagen
- 180 L LB-Brühe in sieben Brunnen in der ersten Reihe einer 96-Well-Platte hinzufügen
Hinweis: Es wird vorgeschlagen, die Verdünnung in Dreifacharbeit durchzuführen, um ihre statistische Zuverlässigkeit zu erhöhen. Bereiten Sie dazu zusätzliche Verdünnungen des Bakteriophagens in der zweiten und dritten Reihe der Platte vor. - Wirbeln Sie vorsichtig die ursprüngliche Bakteriophagenprobe, um homogene Zusicherung zu gewährleisten und übertragen Sie 20 l in den ersten Brunnen.
- Mischen Sie die Probe gut durch Pipettieren und nach unten.
- Übertragen Sie 20 l der resultierenden Suspension in den zweiten Brunnen.
- Setzen Sie die serielle Verdünnung fort, indem Sie 20 l der Lösung im zweiten Brunnen in den dritten Brunnen übertragen, und so weiter, bis zum sechsten Brunnen, so dass der siebte Brunnen sowie eine negative Kontrolle, zu der keine Phagen-Suspension hinzugefügt wird. Dadurch wird ein Verdünnungsbereich von 10-1-10-6erstellt.
- 180 L LB-Brühe in sieben Brunnen in der ersten Reihe einer 96-Well-Platte hinzufügen
- versilberung
- Beschriften Sie die Basis der Petrischalen (zuvor in Schritt 2.1.7) mit Name, Datum und einer kurzen Probenbeschreibung (einschließlich des Verdünnungsfaktors der Phagenprobe). Die Petrischalen im 37°C-Inkubator eine Stunde vor dem Assay vorheizen.
- Schmelzen Sie das erstarrte weiche Agar-Medium (typischerweise mit einer Mikrowelle; erstarrter Agar schmilzt bei 85°C), und lassen Sie es auf etwa 45°C kommen. Das beheizte Medium kann in einem 45°C-Wasserbad für 1h platziert werden, um eine entsprechende Temperatur zu erreichen. Es sollte heiß genug sein, um in flüssiger Form zu bleiben, aber kühl genug, um zugesetzte Bakterien nicht abzutöten.
- 4 ml Bakterienkultur (ab Schritt 2.2) mit 35 ml LB Soft Agar (bei 45°C) mischen. Wirbeln Sie, um die Zellen gleichmäßig zu verteilen, aber vermeiden Sie Schütteln, um Schaumbildung zu verhindern (Abbildung 4A).
- Etikettieren Sie ein steriles Reagenzglas für jeden der seriellen Verdünnungsschritte und eines für die Kontrollprobe für insgesamt 7 beschriftete Reagenzgläser. 5 ml Aliquots der Bakterienkultur/Soft-Agar-Mischung aus Schritt 2.4.3 in die 7 Röhren geben. Arbeiten Sie schnell durch Schritt 2.4.6, da solche kleinen Mengen von agar-basierten Medien schnell bei Raumtemperatur verfestigen.
- Fügen Sie 100 l der Kontrollprobe (ab Schritt 2.3) in das Kontrollprüfrohr und wirbeln Sie vorsichtig. Entsorgen Sie die verwendete Pipettenspitze und übertragen Sie das gleiche Volumen von jeder der seriell verdünnten Bakteriophagenproben (Schritt 2.3) auf ihre jeweiligen Reagenzgläser, wirbeln, um sie zu mischen.
- Die 5 ml-Mischungen sofort auf die beschrifteten, vorgewärmten, festen Agarplatten geben (Abbildung 4A). Wirbeln Sie die Platten sanft, um die Mischungen gleichmäßig zu verteilen.
Hinweis: Wenn Sie den Test in dreifacher Ausfertigung durchführen, wiederholen Sie die Schritte 2.4.3-6 zwei weitere Male. - Versiegeln Sie jede Platte mit Laborfolie, und lassen Sie beide Schichten bei Raumtemperatur (ca. 15 Minuten) richtig erstarren, bevor Sie sie einen 37°C-Inkubator nach oben legen, was das Wachstum sowohl der Bakterien als auch des Phagens anregt, für 24 Stunden oder bis zu Plaques entwickeln, Es dauert in der Regel etwa 1-5 Tage für Plaques erscheinen (Abbildung 4B), aber das Timing variiert erheblich je nach Inkubationsbedingungen, Medium, und die Bakterienarten.
3. Datenanalyse und Ergebnisse
-
Zähltafeln
- Stellen Sie sicher, dass keine Plaques in den Schildern mit der Aufschrift "Kontrolle" sichtbar sind, da dies auf eine Viruskontamination hindeuten würde.
- Beginnen Sie mit den Platten mit der Bezeichnung 10-6, die die am meisten verdünnte Bakteriophagenprobe enthalten. Zählen Sie die Plaketten, ohne den Deckel zu entfernen, und markieren Sie sie mit einem Stift, während Sie gehen, um anzugeben, welche Plaketten bereits gezählt wurden.
- Zählen Sie die restlichen Platten. Einige Platten haben möglicherweise zu wenige oder zu viele Plaketten, um zu zählen. Verwenden Sie die Platten mit 10-150 Plaques für die weitere Analyse.
-
Berechnung von PFU
- Dividieren Sie die Anzahl der Plaques durch den Verdünnungsfaktor (z.B. 10-6 für die am meisten verdünnte Probe), um die Anzahl der Plaque forming Units (PFU) in 100 l phagengemisch zu erhalten.
Hinweis: Wenn Sie den Test in Dreifacharbeit durchführen, verwenden Sie die durchschnittliche Anzahl der Plaques aus den drei Platten. - Um die Konzentration (in PFU/ml) der Ausgangsprobe zu bestimmen, multiplizieren Sie sie mit einem zusätzlichen Verdünnungsfaktor von 10, da nur 100 l der Probe plattiert wurden. (dh
 )
) - Berechnen Sie den Mittelwert der PFU/ml für alle Verdünnungen, die zwischen 10 und 150 Plaques hatten, um ein zuverlässigeres Ergebnis zu erzielen.
- Dividieren Sie die Anzahl der Plaques durch den Verdünnungsfaktor (z.B. 10-6 für die am meisten verdünnte Probe), um die Anzahl der Plaque forming Units (PFU) in 100 l phagengemisch zu erhalten.
Bakteriophagen, auch Phagen genannt, sind Viren, die gezielt Bakterien infizieren, und wir können ihre Anwesenheit bestätigen und sie mit einem Werkzeug namens Plaque-Assay quantifizieren. Bakteriophagen infizieren ihre anfälligen Wirte, indem sie sich zuerst an der bakteriellen Zellwand anheften und ihr genetisches Material injizieren. Dann entführen sie die biosynthetische nalytokische Maschinerie der Zelle, um ihre DNA zu replizieren und zahlreiche Nachkommen-Phagenpartikel zu produzieren, die sie dann durch Lysing und Tötung der Wirtszelle freisetzen.
Diese lytische Aktivität ist die Grundlage einer weit verbreiteten Phagen-Enumerierungstechnik, die als Plaque-Assay oder Doppel-Agar-Schicht-Assay bekannt ist. Hierbei wird zunächst eine Bakteriophagenmischung in einer geschmolzenen Nährwertbrühe mit niedrigkonzentriertem Agar hergestellt. Alle Bakterien, die in der Mischung verwendet werden, sollten lebendig sein und aktiv in der Protokollphase ihres Wachstums teilen, was sicherstellt, dass ein großer Prozentsatz der Bakterien lebensfähig ist und in der Lage ist, einen dichten Rasen um die Plaques herum zu bilden. Als nächstes wird diese geschmolzene bakterielle Agarmischung über ein festeres, konzentrierteres Agar-Nährstoffmedium verteilt, das bereits auf einer Petrischale verfestigt ist. Bei inkubationstemperatur verfestigt sich auch die niedrigkonzentrierte Agar-Phagen-Bakterienbrühe zu einem weichen Agar-Overlay.
Hier können die Bakterienzellen zusätzliche Nährstoffe aus der unteren Schicht ableiten und sollten sich schnell vermehren, um einen konfluenten Rasen von Bakterien zu produzieren. Da jedoch phagenpartikel auch in der weichen Schicht vorhanden sind, werden diese ihr genetisches Material innerhalb der Bakterien infizieren und replizieren, was in einer Zelllyse gipfelt, die mehrere Nachkommen freisetzt. Diese Phagennachkommen infizieren dann die benachbarten Zellen, da der halbfeste Zustand der Bakterien-Phagenschicht ihre Bewegung auf weiter entfernt eitel gelegene Wirtszellen beschränkt. Dieser Zyklus von Infektion und Lyse setzt sich über mehrere Runden fort und tötet eine große Anzahl von Bakterien in einem lokalisierten Gebiet. Die Wirkung der benachbarten Zellen zerstört werden, ist es, eine einzige kreisförmige klare Zone zu produzieren, genannt eine Plaque, die mit dem bloßen Auge gesehen werden kann, effektiv verstärken die bakterielle lytische Aktivität des Phagens und ermöglicht ihre Aufzählung.
Die Anzahl der Plaques auf einer Petrischale wird als Plaque-Forming Units oder PFUs bezeichnet und sollte, sofern die anfängliche Bakteriophagenkonzentration ausreichend verdünnt war, direkt der Anzahl der infektiösen Phagenpartikel in der ursprünglichen Probe entsprechen. Diese Technik kann auch zur Charakterisierung der Plaquemorphologie, zur Identifizierung von Phagentypen oder zur Isolierung von Phagenmutanten verwendet werden. In diesem Labor erfahren Sie, wie Sie den Plaque-Test zur Aufzählung von Phagen durchführen, indem Sie als Beispiel den T7-Phagen von E. coli verwenden.
Identifizieren Sie zunächst ein geeignetes Medium für die Kultivierung der Wirtsbakterienzellen und des Bakteriophagens. Hier wurde Lysogeny Brühe, oder LB Medium, verwendet, um E. coli und den T7 Phagen zu kultur. Als nächstes nehmen Sie drei saubere Glasflaschen und beschriften sie mit Medien, Namen, und dann die erste als LB-Broth, die zweite als LB-Bottom Agar, und die dritte als LB-Top Agar. Jetzt wägen Sie vier Gramm vorformuliertes LB-Pulver in drei Sätzen ab und übertragen Sie dann einen Satz gewogener getrockneter Medien in jede Flasche. Fügen Sie 200 Milliliter Wasser in die erste Flasche. Mischen Sie den Inhalt mit einem magnetischen Rührbalken.
Dann, mit einem pH-Meter und ständigem Rühren, bringen Sie den endgültigen pH-Wert auf 7,4 durch Zugabe von Natriumhydroxid oder Salzsäure. Wiederholen Sie die Wasserzugabe und pH-Einstellung für die anderen beiden verbleibenden Flaschen. Jetzt wiegen Sie drei Gramm Agarpulver und fügen Sie es der zweiten Flasche hinzu, um einen 1,5 % unteren Agar zu machen. Schließlich wiegen 1. 2 Gramm Agar und fügen Sie es auf die dritte Flasche, um die .6 % LB Top Agar zu machen. Der Brühezustand in der Flasche eins braucht keine Agarzugabe. Die Flasche halbfest verschließen und dann die Medien sterilisieren, indem Sie 20 Minuten lang bei 121 Grad Celsius autoklavieren. Nach dem Abschluss die Medienflaschen aus dem Autoklaven entfernen und die Flaschenverschlüsse sofort verdrehen, um sie vollständig zu schließen, um eine Kontamination zu verhindern. Halten Sie die LB-Broth und LB-Top Agar Medien für den späteren Gebrauch auf der Bank. Legen Sie den LB-Bottom Agar in einem Auflagenbad von ca. 45 Grad Celsius abkühlen.
Wenn der LB-Bottom Agar etwa 45 Grad Celsius erreicht, überträgt er ihn auf die Arbeitsbank. Als Nächstes sterilisieren Sie den Arbeitsbereich mit 70 % Ethenol. Als nächstes fügen Sie 450 Mikroliter steriles ein molares Calciumchlorid in den geschmolzenen Bodenagar, um eine endgültige Konzentration von 2,25 Millimolar zu machen. Mischen Sie die Flasche vorsichtig zum Mischen. Dann legen Sie sieben saubere Petri-Gerichte. Beschriften Sie jedes Gericht auf der Unterseite mit dem Mediennamen und dem Zubereitungsdatum. Dann gießen Sie 15 Milliliter des unteren Agars in jedes der sieben Petri-Gerichte. Lassen Sie die Platten für ein paar Stunden oder über Nacht bei Raumtemperatur einstellen. Einmal gesetzt, können die Kulturplatten bei Bedarf mehrere Tage bei vier Grad Celsius gelagert werden, um die Kondensation auf den Kopf zu stellen. Übertragen Sie die Petri-Gerichte vom Vier-Grad-Kühlschrank auf einen 37 Grad Celsius-Brutkasten eine Stunde vor dem Assay.
Am Tag vor dem Assay sollte der E. coli kultiviert werden. Hier wurden 10 Mikroliter E. coli-Kultur in 10 Milliliter LB-Broth geimpft. Platzieren Sie die Bakterien, um über Nacht in einem schütteren Inkubator zu wachsen, der auf 37 Grad Celsius bei 160 RPM eingestellt ist. Dann, am Tag des Assays, entfernen Sie die Bakterienkultur aus dem Brutkasten. Samen Sie eine frische 10 Milliliter frische LB Brühe mit 0,5 Milliliter der Übernachtungskultur. Platzieren Sie diese Zellen, um zu einem schüttelnden Inkubator zu wachsen, der bei 160 RPM auf 37 Grad Celsius eingestellt ist. Als Nächstes verwenden Sie ein Spektralphotometer, um zu überprüfen, wann diese Kultur das Wachstum der Logphase erreicht, was durch eine optische Dichte von 0,5 bis 0,7 angezeigt wird. Sobald die OD diese Ebene erreicht hat, stoppen Sie die Inkubation, indem Sie die Zellkultur auf die Bank übertragen. Sie sind nun bereit, für Phagen-Overlay-Assay verwendet zu werden.
Phage-Titers können exponentiell zwischen verschiedenen Phagentypen und -proben variieren. Um sie effektiv zu zählen, sollten sie verdünnt werden, um eine breite Palette von Phagenkonzentrationen zu erzeugen. Am Tag des Assays, erzeugen Eine Reihe von Phagen verdünnungen von einem Zehntel bis ein Millionstel Konzentrationen, nach einer 10-fachen Verdünnungstechnik. Um statistisch signifikante und genaue Daten zu erhalten, führen Sie die serielle Verdünnung in Dreifacharbeit durch.
Als nächstes schmelzen Sie den erstarrten LB-Top-Agar mit einer Mikrowelle. Dann legen Sie es in ein Wasserbad, das bei 45 Grad Celsius für eine Stunde voreingestellt ist. Nach einer Stunde die Petri-Gerichte mit der unteren Agarschicht aus dem Brutkasten sammeln. Beschriften Sie die Platten mit Phagenkonzentration und Assay-Datum. Legen Sie dann sieben saubere Reagenzgläser auf. Beschriften Sie jedes Reagenzglas mit der serienmäßigen Phagenverdünnungsnummer und benennen Sie eines als Steuerung.
Wenn der LB-Top-Agar 45 Grad Celsius erreicht, überträgt er ihn auf die Arbeitsbank. Fügen Sie nun 450 Mikroliter eines Molaren-Calciumchlorids in den 200-Milliliter-Agar ein, um eine Endkonzentration von 2,25 Millimolar zu erreichen. Mischen Sie die Flasche vorsichtig zum Mischen. Als nächstes fügen Sie 35 Milliliter LB-Top-Agar und vier Milliliter bakterielle Suspension zu einem sterilen konischen Rohr hinzu. Wirbeln Sie sanft, um die Zellen gleichmäßig zu verteilen, aber vermeiden Sie Schütteln, um Schaumbildung zu verhindern.
Nun, aliquot fünf Milliliter dieser Bakterien-Top-Agar-Mischung zu jedem der sieben Reagenzgläser. Dann übertragen Sie 100 Mikroliter der seriell verdünnten Bakteriophagenproben und Kontrollmedien, die einfach Medien ohne Bakteriophagen sein sollten, auf die respektvoll beschrifteten Reagenzgläser. Wirbeln Sie die Mischung vorsichtig, um eine ordnungsgemäße Mischung zu gewährleisten. Fünf Milliliter Bakteriophage-Mischung vorsichtig auf die jeweilige Petriplatte geben. Gleichmäßig die Mischung über die gesamte Oberfläche verteilen, indem sie sanft die Petriplatte wirbeln.
Sobald alle Petriplatten mit der Mischung geschichtet sind, erlauben Sie eine Erstarrung der obersten Schicht, indem Sie bei Raumtemperatur für 15 Minuten inkubieren. Nach Abschluss dieser Schritte wiederholen Sie den Prozess für den zweiten und dann den dritten Satz der Petrischalen mit den verbleibenden zwei Sätzen von Phagen verdünnungen. Versiegeln Sie jede Schale mit Parafilm und brüten Bei Raumtemperatur für 15 Minuten. Stellen Sie die Kulturplatte 24 Stunden lang bei einer geeigneten Temperatur auf den Kopf oder bis sich Plaques entwickeln. Hier wurden Platten für einen Tag in einem 37 Grad Celsius Inkubator platziert, eine stimulierende Wachstumsbedingung für E. coli und den T7-Phagen.
Plaques erscheinen nach ein bis fünf Tagen Inkubation, abhängig von der Bakterienart, Inkubationsbedingungen und der Wahl des Mediums. Hier waren Plaques nach einem Tag der Inkubation bei 37 Grad Celsius sichtbar. Beginnen Sie, indem Sie die gekennzeichneten Platten kontrollieren und sicherstellen, dass keine Plaques in diesen Platten gebildet wurden, da dies auf eine Viruskontamination hinweisen würde. Um den Phagen-Titer in der Originalprobe zu bestimmen, beginnen Sie zuerst mit den Platten, die die am meisten verdünnte Phagenprobe enthalten, und zählen Sie die Plaques, ohne die Deckel zu entfernen, und markieren Sie sie, um anzugeben, welche bereits gezählt wurden. Wiederholen Sie die Zählung für jede Platte in jedem Satz. Einige Platten haben möglicherweise zu viele oder zu wenige Plaketten, um gezählt zu werden. Betrachten Sie 10 bis 150 als ideale Plaquezahl.
Generieren Sie als Nächstes eine Tabelle mit den Plaquenummernwerten für die verschiedenen Verdünnungen und Replikationen. Berechnen Sie dann die mittleren Plaquezahlenwerte für die Verdünnungsplatten, die die ideale Anzahl von Plaqueanzahlen enthielten. In diesem Beispiel handelte es sich um die durchschnittliche Anzahl der Plaques, die in der 10 bis minus drei und 10 zu den minus vier Verdünnungsplatten gebildet wurden. Passen Sie nun den Phagenverdünnungsfaktor an, indem Sie die erhaltenen mittleren Plaquewerte durch die jeweiligen Phagenverdünnungsfaktoren dividieren. Hier bei, die durchschnittliche Anzahl der Plaques, die zu den 10 bis minus drei und 10 zu den minus vier Verdünnungsplatten gebildet wurden, wurden durch ihre jeweiligen Verdünnungsfaktoren geteilt, um die Anzahl der Plaquebildenden Einheiten oder PFUs in 100 Mikroliter Phagengemisch zu erhalten. Um den Wert in PFU pro Milliliter umzuwandeln, multiplizieren Sie die erzeugten Werte mit 10, da während des Bakteriophagen-Overlay-Vorbereitungsschritts nur 100 Mikroliter Phagenverdünnungsmischung verwendet wurden, wodurch ein zusätzlicher Verdünnungsfaktor von 10 erzeugt wurde. Berechnen Sie schließlich den Durchschnitt der Werte, die aus den verschiedenen Verdünnungsplatten ermittelt werden. Dadurch wird die durchschnittliche Anzahl der PFUs pro Milliliter angibt. Die Anzahl der PFUs entspricht der Anzahl der infektiösen Phagenpartikel in der Originalprobe.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
Trotz mehrfacher technologischer Fortschritte bleiben Plaque-Assays der Goldstandard für die Bestimmung von viralem Titer (als PFU) und für die Isolierung reiner Bakteriophagenpopulationen unerlässlich. Anfällige Wirtszellen werden in der Deckschicht einer zweischichtigen Agarplatte kultiviert und bilden ein homogenes Bett, das eine virale Replikation ermöglicht. Das ausgangse Ereignis, bei dem ein isoliertes Bakteriophagen im lytischen Lebenszyklus eine Zelle infiziert, sich in ihr repliziert und sie schließlich lysiert, ist zu klein, um sie zu beobachten. Die freigesetzten Virionen werden jedoch benachbarte Zellen infizieren, was anschließend zu einer Clearingzone oder Plaque führt, die das Vorhandensein einer einzigen PFU bezeichnet.
Subscription Required. Please recommend JoVE to your librarian.
References
- Twort, F. An investigation on the nature of ultra-microscopic viruses. Lancet. 186 (4814): 1241-1243. (1915)
- d'Hérelle, F. An invisible antagonist microbe of dysentery bacillus. Comptes Rendus Hebdomadaires Des Seances De L Academie Des Sciences. 165: 373-375. (1917)
- Cisek AA, Dąbrowska I, Gregorczyk KP, Wyżewski Z. Phage Therapy in Bacterial Infections Treatment: One Hundred Years After the Discovery of Bacteriophages. Current Microbiology. 74 (2):277-283. (2017)
- Mirzaei MK, Maurice CF. Ménage à trois in the human gut: interactions between host, bacteria and phages. Nature Reviews Microbiology. 15 (7):397. (2017)
- Breitbart M, Bonnain C, Malki K, Sawaya NA. Phage puppet masters of the marine microbial realm. Nature Microbiology. 3 (7):754-766. (2018)
- Leung CY, Weitz JS. Modeling the synergistic elimination of bacteria by phage and the innate immune system. Journal of Theoretical Biology. 429:241-252. (2017)
- Dulbecco R. Production of Plaques in Monolayer Tissue Cultures by Single Particles of an Animal Virus. Proceedings of the National Academy of Sciences of the United States of America. 38 (8):747-752. (1952)
- Juarez D, Long KC, Aguilar P, Kochel TJ, Halsey ES. Assessment of plaque assay methods for alphaviruses. J Virol Methods. 187 (1):185-9. (2013)
- Clokie MRJ, Millard AD, Letarov AV, Heaphy S. 2011. Phages in nature. Bacteriophage. 1 (1):31-45. (2011)
- Moldovan R, Chapman-McQuiston E, Wu XL. On kinetics of phage adsorption. Biophys J. 93 (1):303-15. (2007)
- Garneau JE, Dupuis M-È, Villion M, Romero DA, Barrangou R, Boyaval P, Fremaux C, Horvath P, Magadán AH, Moineau S.. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320):67. (2010)





 )
)