Overview
Fonte: Tilde Andersson1, Rolf Lood1
1 Departamento de Ciências Clínicas Lund, Divisão de Medicina de Infecção, Centro Biomédico, Universidade de Lund, 221 00 Lund, Suécia
Vírus que infectam organismos procarióticos, chamados bacteriófagos ou simplesmente fálticos, foram identificados no início do séculoXX por Twort (1) e d'Hérelle (2) de forma independente. Desde então, os phages têm sido amplamente reconhecidos por seu valor terapêutico (3) e sua influência sobre os humanos (4), bem como os ecossistemas globais (5). As preocupações atuais têm alimentado um interesse renovado no uso de phages como alternativa aos antibióticos modernos no tratamento de doenças infecciosas (6). Essencialmente, todas as pesquisas de phage dependem da capacidade de purificar e quantificar vírus, também conhecido como um titer viral. Inicialmente descrito em 1952, este era o propósito do ensaio da placa (7). Décadas e múltiplos avanços tecnológicos depois, o ensaio da placa continua sendo um dos métodos mais confiáveis para a determinação do título viral (8).
Bacteriophages subsistem injetando seu material genético em células hospedeiras, sequestrando as máquinas para produção de novas partículas de phage, e eventualmente fazendo com que o hospedeiro libere numerosos virions progêneres através da lise celular. Devido ao seu tamanho minucioso, os bacteriófagos não podem ser observados usando apenas microscopia leve; portanto, é necessária a microscopia eletrônica de varredura (Figura 1). Além disso, os phages não podem ser cultivados em placas de ágar nutricional como bactérias, pois precisam de células hospedeiras para se aproveitarem.
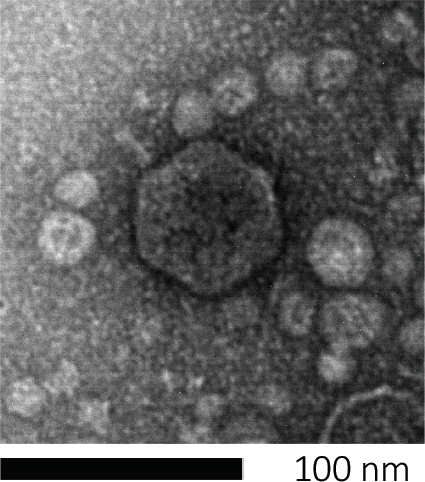
Figura 1: A morfologia de um bacteriófago, aqui exemplificado por um phage E. coli, pode ser estudado usando microscopia eletrônica de varredura. A maioria dos bacteriófagos pertence a Caudovirales (bacteriófagos tailed). Este phage em particular tem uma estrutura de cauda muito curta e uma cabeça icosaedral, colocando-a na família de Podovírus.
O ensaio da placa (Figura 2) baseia-se na incorporação de células hospedeiras, preferencialmente em crescimento de fase de registro, no meio. Isso cria uma densa camada turva de bactérias capazes de sustentar o crescimento viral. Uma praga isolada pode infectar, replicar dentro e lise uma célula. A cada célula lise, várias células adjacentes ficam imediatamente infectadas. Vários ciclos dentro, uma zona clara (uma placa) pode ser observada na placa turva (Figura 2B/Figura 3A), indicando a presença do que inicialmente era uma única partícula bacteriófago. O número de unidades formadoras de placas por volume (ou seja, PFU/mL) de uma amostra, pode ser determinado a partir do número de placas geradas.
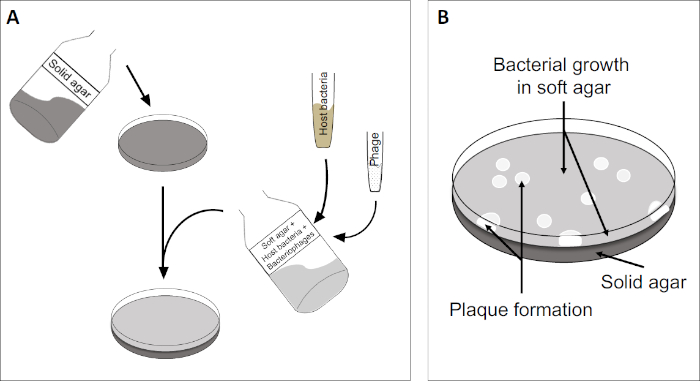
Figura 2: O teste para unidades de formação de placas (PFU) é um método comum para determinar o número de bacteriófagos em uma amostra. (A) A base de uma placa de Petri estéril é coberta com um meio de nutrientes sólidos apropriado, seguido por uma mistura de mídia macia, células hospedeiras suscetíveis e uma diluição da amostra original de bacteriófago. Note que a suspensão de phage poderia, em alguns casos, também ser uniformemente espalhada pela superfície de ágar macio já solidificado. (B) O crescimento das bactérias hospedeiras forma um gramado de células na camada superior do ágar. A replicação do bacteriófago gera zonas claras, ou placas, causadas pela lise celular hospedeira.
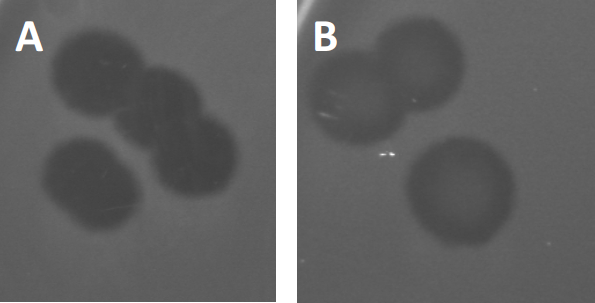
Figura 3: Os resultados do teste de PFU mostram múltiplas placas geradas por bacteriófagos. Devido à lise de células hospedeiras suscetíveis, as placas podem ser vistas como zonas de compensação no gramado bacteriano, seja com (A) desobstrução total, ou (B) re-crescimento parcial causado pela geração de bactérias resistentes (ou possivelmente por phages temperados no ciclo lisogênico).
Certos fáticos temperados podem adotar o que é chamado de ciclo de vida lisogênico, além do crescimento lítico descrito anteriormente. Na lysogenia, o vírus assume um estado latente através da incorporação de seu material genético no genoma da célula hospedeira (9), muitas vezes conferindo resistência a novas infecções por pragas. Isso às vezes é revelado através de uma leve nebulosidade da placa (Figura 3B). Vale ressaltar, porém, que as placas também podem aparecer borradas devido ao reesúde de bactérias que evoluíram resistência ao phage independente de infecções anteriores.
Os vírus podem se conectar, ou adsorb, a apenas uma gama limitada de bactérias hospedeiras (10). As faixas de host são ainda limitadas por estratégias antivirais intracelulares, como o sistema CRISPR-Cas (11). A resistência/sensibilidade para phages específicos exibidos por subgrupos bacterianos tem sido historicamente usada para categorizar cepas bacterianas em diferentes tipos de phage (Figura 4). Embora a eficácia deste método tenha sido agora superada por novas técnicas de sequenciamento, a digitação de phage ainda pode fornecer informações valiosas sobre interações bactérias-phage, por exemplo, facilitando o design de um coquetel phage para uso clínico.
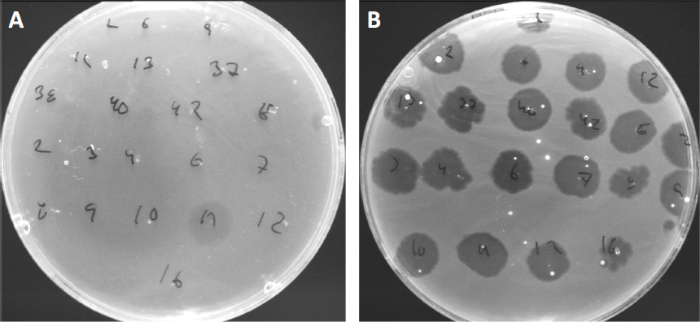
Figura 4: Sensibilidade à praga de diferentes cepas bacterianas. Placas de ágar macio com cepa cutibacterium acnes (A) AD27 e (B) AD35, foram avistadas com 21 bacteriófagos C. acnes diferentes. Apenas o phage 11 foi capaz de infectar e matar AD27, enquanto a cepa AD35 mostrou sensibilidade para todas as pragas. Esta técnica, denominada digitação phage, pode ser usada para dividir espécies bacterianas e cepas em diferentes subgrupos com base na suscetibilidade de phage.
Procedure
1. Configuração
- Antes de iniciar qualquer trabalho envolvendo micróbios, certifique-se de que o espaço de trabalho seja esterilizado (por exemplo, limpo com 70% de etanol). Use sempre um jaleco e luvas, mantenha os cabelos longos amarrados para trás e certifique-se de que todas as feridas estejam particularmente bem protegidas.
- Quando terminar, esterilize todas as superfícies e lave/esterilize completamente as mãos e pulsos.
2. Protocolo
- Preparação de mídia LB
Nota: Dependendo da cepa bacteriana hospedeira e do bacteriófago, um meio líquido diferente pode ser mais adequado para a cultura inicial da cepa bacteriana hospedeira ou um meio sólido diferente pode ser mais adequado para o crescimento subsequente. O caldo de lysogeny (LB) é usado neste protocolo para o caldo e o ágar.- Misture 4 g LB em 200 mL de água destilada, em triplicado, para o caldo LB, o ágar de fundo sólido e o ágar superior macio. Todas as soluções devem ser preparadas em recipientes capazes de conter o dobro do volume final para evitar o estouro enquanto se autoclavar.
Nota: Se realizar o ensaio em triplicado, prepare o dobro da quantidade de ágar inferior LB. - Ajuste o pH das três soluções para 7.4 usando NaOH ou HCl conforme apropriado.
- Adicione 3 g de potência de ágar à garrafa de ágar inferior para fazer uma solução de 1,5% de ágar para o ágar sólido
- Adicione 1,2 g de potência de ágar à garrafa de ágar superior para fazer uma solução de 0,6% de ágar para ágar macio.
- Coloque as garrafas, com tampas semi-apertadas, em uma autoclave fixada em 121°C por 20 minutos para esterilizar as soluções. Feche as tampas assim que a corrida estiver concluída para evitar contaminação.
- Quando a mídia LB atingir uma temperatura de aproximadamente 45-50°C, adicione 450 μL de cacl2 estéril a todas as três soluções lb de 200 mL para fazer uma concentração final de 2,25 mM.
- Despeje 15 mL de alíquotas do meio de ágar sólido em placas de Petri estéreis (evite tremer para evitar espuma) e deixe que o ágar solidifique por algumas horas ou durante a noite à temperatura ambiente(Figura 4A). As placas podem ser armazenadas de cabeça para baixo a 4°C por vários dias.
- Misture 4 g LB em 200 mL de água destilada, em triplicado, para o caldo LB, o ágar de fundo sólido e o ágar superior macio. Todas as soluções devem ser preparadas em recipientes capazes de conter o dobro do volume final para evitar o estouro enquanto se autoclavar.
- Cultivo de células hospedeiras
- Um dia antes do ensaio, adicione 10 μL de cultura E. coli a 10 mL de caldo LB.
- Incubar a bactéria a 37°C durante a noite a 160 rpm em uma incubadora de agitação.
- Na manhã do ensaio, adicione 0,5 mL da cultura da noite para o dia a 10 mL de LB fresco.
Nota: Se você está realizando o ensaio em triplicado, prepare o dobro da quantidade desta cultura. - Incubar os a 37°C a 160 rpm em uma incubadora de agitação até que a cultura bacteriana esteja em crescimento em fase de registro. Isso pode ser determinado espectrofotometricamente por um OD600 de 0,5-0,7.
- Mantenha a cultura em temperatura ambiente até que as bactérias sejam adicionadas ao ágar LB superior.
- Diluição serial de 10 vezes do bacteriófago
- Adicione 180μL de caldo LB a sete poços na primeira linha de uma placa de 96 poços
Nota: Sugere-se realizar a diluição em triplicado, a fim de aumentar sua confiabilidade estatística. Para isso, prepare diluições adicionais do bacteriófago na segunda e terceira fileiras da placa. - Cuidadosamente vórtice a amostra original de bacteriófago para garantir a homogeneidade e transferir 20 μL para o primeiro poço.
- Misture bem a amostra por pipetação e para baixo.
- Transferir 20 μL da suspensão resultante para o segundo poço.
- Continue a diluição serial transferindo 20 μL da solução no segundo poço para o terceiro poço, e assim por diante, até o sexto bem, deixando o sétimo bem como um controle negativo ao qual nenhuma suspensão phage será adicionada. Isso criará uma faixa de diluição de 10-1-10-6.
- Adicione 180μL de caldo LB a sete poços na primeira linha de uma placa de 96 poços
- Chapeamento
- Rotule a base das placas de Petri (previamente preparadas na etapa 2.1.7) com nome, data e uma breve descrição da amostra (incluindo o fator de diluição da amostra de phage). Pré-aqueça as placas de Petri na incubadora de 37°C uma hora antes do ensaio.
- Derreta o ágar-médio macio solidificado (normalmente feito usando um micro-ondas; o ágar solidificado derrete a 85°C), e deixe-o chegar a cerca de 45°C. A mídia aquecida pode ser colocada em um banho de água de 45°C por ~1h para atingir uma temperatura apropriada. Deve ser quente o suficiente para permanecer em forma líquida, mas frio o suficiente para não matar bactérias adicionadas.
- Misture cultura bacteriana de 4 mL (a partir do passo 2.2) com ágar macio de 35 mL LB (a 45°C). Redemoinho para distribuir uniformemente as células, mas evite tremer para evitar espuma(Figura 4A).
- Rotule um tubo de ensaio estéril para cada uma das etapas de diluição serial e outro para a amostra de controle, para um total de 7 tubos de ensaio rotulados. Coloque 5 mL aliquots da cultura bacteriana/mistura de ágar macio da etapa 2.4.3 para os 7 tubos. Trabalhe rapidamente através da etapa 2.4.6 porque esses pequenos volumes de mídia à base de ágar se solidificarão rapidamente à temperatura ambiente.
- Adicione 100 μL da amostra de controle (da etapa 2.3) ao tubo de ensaio de controle e gire cuidadosamente. Descarte a ponta de pipeta usada e transfira o mesmo volume de cada uma das amostras de bacteriófago diluídas em série (etapa 2.3) para seus respectivos tubos de ensaio, girando para misturar.
- Transfira imediatamente as misturas de 5 mL para as placas de ágar rotuladas, pré-aquecidas e sólidas(Figura 4A). Gire suavemente as placas para até mesmo espalhar as misturas.
Nota: Se você estiver executando o ensaio em triplicado, repita as etapas 2.4.3-6 mais duas vezes. - Sele cada placa com filme de laboratório, e permita que ambas as camadas se solidifiquem adequadamente à temperatura ambiente (aproximadamente 15 minutos) antes de colocá-las de lado para baixo em uma incubadora de 37°C, estimulando o crescimento tanto da bactéria quanto da praga, por 24 horas ou até que as placas se desenvolvam, normalmente leva cerca de 1-5 dias para que as placas apareçam(Figura 4B), mas o tempo varia consideravelmente dependendo das condições de incubação, média e as espécies bacterianas.
3. Análise e Resultados de Dados
-
Contando placas
- Certifique-se de que nenhuma placa esteja visível nas placas marcadas como "controle", pois isso indicaria contaminação viral.
- Comece com as placas rotuladas 10-6,contendo a amostra de bacteriófago mais diluída. Conte as placas sem remover a tampa, marcando-as com uma caneta enquanto você vai para indicar quais placas já foram contadas.
- Conte as placas restantes. Algumas placas podem ter poucas ou muitas placas para contar. Use as placas com placas de 10 a 150 para análise posterior.
-
Cálculo pfu
- Divida o número de placas pelo fator de diluição (ex. 10-6 para a amostra mais diluída) paraobentar o número de Unidades de Formação de Placa (PFU) em 100 μL de mistura de phage.
Nota: Se realizar o ensaio em triplicado, use o número médio de placas das três placas. - Para determinar a concentração (em PFU/mL) da amostra original, multiplique por um fator adicional de diluição de 10, uma vez que apenas 100 μL de amostra foram emplacados.
 (ie)
(ie) - Calcule o valor médio do PFU/mL para todas as diluições que tinham entre 10 e 150 placas para obter um resultado mais confiável.
- Divida o número de placas pelo fator de diluição (ex. 10-6 para a amostra mais diluída) paraobentar o número de Unidades de Formação de Placa (PFU) em 100 μL de mistura de phage.
Bacteriófagos, também chamados de phages, são vírus que infectam especificamente bactérias e podemos confirmar sua presença e quantificá-las usando uma ferramenta chamada ensaio de placa. Bacteriófagos infectam seus hospedeiros suscetíveis, primeiro anexando-se à parede celular bacteriana e injetando seu material genético. Então, eles sequestram as máquinas biossintéticas da célula para replicar seu DNA e produzir numerosas partículas de phage progênero, que eles então liberam lise e matando a célula hospedeira.
Esta atividade lítica é a base de uma técnica de enumeração de phage amplamente utilizada conhecida como ensaio de placa ou ensaio de camada de ágar duplo. Aqui, uma mistura de bacteriófago é preparada pela primeira vez em um caldo de nutriente derretido contendo ágar de baixa concentração. Todas as bactérias utilizadas na mistura devem estar vivas e ativamente se dividindo na fase de tronco de seu crescimento, o que garantirá que uma grande porcentagem das bactérias sejam viáveis e capazes de formar um gramado denso ao redor das placas. Em seguida, esta mistura de ágar bacteriana-phage derretida é espalhada sobre um meio de nutrientes ágar mais sólido e concentrado que já está solidificado em uma placa de Petri. Na incubação à temperatura ambiente, o caldo de baixa concentração de ágar-phage-bactérias também se solidifica para formar uma sobreposição de ágar macia.
Aqui, as células bacterianas podem derivar nutrientes adicionais da camada inferior e devem se multiplicar rapidamente para produzir um gramado confluente de bactérias. No entanto, como as partículas de phage também estão presentes na camada macia, elas infectarão e replicarão seu material genético dentro das bactérias, culminando na lise celular, que libera múltiplas progêneres. Essas descendentes de fábulas então infectam as células vizinhas, já que o estado semissólido da camada de bactérias-phage restringe seu movimento a células hospedeiras mais distantes. Esse ciclo de infecção e lise continua ao longo de várias rodadas, matando um grande número de bactérias em uma área localizada. O efeito das células vizinhas serem destruídas, é produzir uma única zona circular clara, chamada de placa, que pode ser vista a olho nu, amplificando efetivamente a atividade bacteriana da praga e permitindo sua enumeração.
O número de placas em uma placa de Petri são referidos como Unidades formadoras de placas, ou PFUs, e, desde que a concentração inicial de bacteriófago fosse suficientemente diluída, deve corresponder diretamente ao número de partículas de phage infecciosas na amostra original. Esta técnica também pode ser usada para caracterização da morfologia da placa, para auxiliar na identificação de tipos de phage, ou para isolar mutantes de phage. Neste laboratório, você aprenderá como realizar o ensaio de placa para enumerar phages, usando o phage T7 de E. coli como exemplo.
Primeiro, identifique um meio adequado para acultura das células bacterianas hospedeiras e do bacteriófago. Aqui o caldo de lysogeny, ou meio LB, foi usado para cultivar E. coli e o phage T7. Em seguida, pegue três garrafas de vidro limpas e rotule-as com mídia, nome, e depois a primeira como LB-Broth, a segunda como LB-Bottom Agar, e a terceira como LB-Top Agar. Agora, pese quatro gramas de pó LB pré-formulado em três conjuntos e, em seguida, transfira um conjunto de mídia seca pesada em cada garrafa. Adicione 200 mililitros de água à primeira garrafa. Misture o conteúdo usando uma barra de agitação magnética.
Em seguida, utilizando um medidor de pH e agitação constante, leve o pH final para 7,4 através da adição de hidróxido de sódio ou ácido clorídrico. Repita a adição de água e o ajuste de pH para as outras duas garrafas restantes, também. Agora, pese três gramas de ágar em pó e adicione-o à segunda garrafa para fazer um ágar inferior de 1,5%. Finalmente, pesar 1. 2 gramas de ágar e adicione-o à terceira garrafa para fazer o ágar superior de 0,6 % LB. A condição do caldo na garrafa não precisa de adição de ágar. Tampe a garrafa semi-firmemente e, em seguida, esterilize a mídia autoclaving a 121 graus Celsius por 20 minutos. Uma vez concluída, remova as garrafas de mídia da autoclave e torça imediatamente as tampas das garrafas para fechá-las completamente para evitar contaminação. Mantenha a mídia LB-Broth e LB-Top Agar no banco para uso posterior. Coloque o Ágar LB-Bottom para esfriar em um banho de água que é predefinido a aproximadamente 45 graus Celsius.
Quando o ágar LB-Bottom atingir aproximadamente 45 graus Celsius, transfira-o para o banco de trabalho. Em seguida, esterilize o espaço de trabalho usando 70 % de ethenol. Em seguida, adicione 450 microliters de cloreto de cálcio molar estéril ao ágar de fundo derretido para fazer uma concentração final de 2,25 milimililar. Gire suavemente a garrafa para misturar. Então, coloque sete pratos de Petri limpos. Rotule cada prato na parte inferior com o nome da mídia e data de preparação. Em seguida, despeje 15 mililitros do ágar inferior em cada uma das sete placas de Petri. Deixe as placas fixar por algumas horas ou durante a noite em temperatura ambiente. Uma vez definidas, as placas de cultura podem ser armazenadas a quatro graus Celsius por vários dias, se necessário, de cabeça para baixo para minimizar a condensação. Transfira as placas de Petri da geladeira de quatro graus Celsius para uma incubadora de 37 graus Celsius uma hora antes do ensaio.
Um dia antes do ensaio ser pré-formado, o E. coli deve ser cultivado. Aqui, 10 microliters da cultura E. coli foram inoculados em 10 mililitros de CALdo LB. Coloque as bactérias para crescer durante a noite em uma incubadora de agitação fixada a 37 graus Celsius a 160 RPM. Então, no dia do ensaio, remova a cultura bacteriana da incubadora. Semente um fresco 10 mililitros de caldo LB fresco com 0,5 mililitros da cultura da noite para o dia. Coloque essas células para crescer em uma incubadora de agitação a 37 graus Celsius a 160 RPM. Em seguida, use um espectrômetro para verificar quando essa cultura atinge o crescimento da fase de registro, indicada por uma densidade óptica de 0,5 a 0,7. Uma vez que o OD atinja esse nível, pare a incubação transferindo a cultura celular para o banco. Eles estão agora prontos para serem usados para ensaio de sobreposição de phage.
Os titulantes de phage podem variar exponencialmente entre diferentes tipos de phage e amostras. Assim, a fim de contá-los efetivamente, eles devem ser diluídos para gerar uma ampla gama de concentrações de phage. No dia do ensaio, gerar uma série de diluições de phage variando de um décimo a um milionésimo de concentrações, seguindo uma técnica de diluição de 10 vezes. Para obter dados estatisticamente significativos e precisos, realize a diluição serial em triplicado.
Em seguida, derreta o ágar lb-top solidificado usando um micro-ondas. Em seguida, coloque-o em um banho de água que é predefinido a 45 graus Celsius por uma hora. Após uma hora, colete as placas de Petri contendo a camada de ágar inferior da incubadora. Rotule as placas com concentração de phage e data do ensaio. Então, coloque sete tubos de ensaio limpos. Rotule cada tubo de ensaio com o número de diluição de phage serial e designe um como controle.
Quando o ágar lb-top atingir 45 graus Celsius, transfira-o para o banco de trabalho. Agora, adicione 450 microliters de um cloreto de cálcio molar ao ágar de 200 mililitros para fazer uma concentração final de 2,25 mililitros. Gire suavemente a garrafa para misturar. Em seguida, adicione 35 mililitros de ágar lb-top e quatro mililitros de suspensão bacteriana a um tubo cônico estéril. Gire suavemente para distribuir uniformemente as células, mas evite tremer para evitar espuma.
Agora, alíquota de cinco mililitros desta mistura de ágar de topo para cada um dos sete tubos de ensaio. Em seguida, transfira 100 microliters das amostras de bacteriófago diluídas em série e mídia de controle, que deve ser simplesmente mídia sem bacteriófago, para os tubos de ensaio respeitosamente rotulados. Gire a mistura suavemente para garantir a mistura adequada. Transfira suavemente cinco mililitros de mistura de bacteriófago para a respectiva placa de Petri. Espalhe uniformemente toda a superfície girando suavemente a placa de Petri.
Uma vez que todas as placas de Petri estejam em camadas com a mistura, permita a solidificação da camada superior incubando à temperatura ambiente por 15 minutos. Após a conclusão dessas etapas, repita o processo para a segunda e, em seguida, os terceiros conjuntos das placas de Petri usando os dois conjuntos restantes de diluições de phage. Sele cada prato com parafilm e incubar em temperatura ambiente por 15 minutos. Coloque a placa de cultura de cabeça para baixo a uma temperatura adequada por 24 horas ou até que as placas se desenvolvam. Aqui, as placas foram colocadas em uma incubadora de 37 graus Celsius por um dia, uma condição de crescimento estimulante para E. coli e o phage T7.
As placas aparecerão após um a cinco dias de incubação, dependendo das espécies bacterianas, condições de incubação e escolha do meio. Aqui, as placas eram visíveis após um dia de incubação a 37 graus Celsius. Comece verificando o controle das placas marcadas e garantindo que não foram formadas placas nessas placas, pois isso indicaria contaminação viral. Para determinar o titulador de phage na amostra original, comece com as placas contendo a amostra de phage mais diluída primeiro e conte as placas sem remover as tampas, marcando-as para indicar quais já foram contadas. Repita a contagem de cada placa em cada conjunto. Algumas placas podem ter muitas ou poucas placas para serem contadas. Considere de 10 a 150 como uma contagem de placas ideal.
Em seguida, gere uma tabela listando os valores do número da placa para as diferentes diluições e réplicas. Em seguida, calcule os valores médios do número da placa para as placas de diluição que continham o número ideal de contagem de placas. Neste exemplo, estes foram o número médio de placas formadas nas 10 para as menos três e 10 para as menos quatro placas de diluição. Agora, ajuste para fator de diluição de phage dividindo os valores médios da placa obtidos pelos respectivos fatores de diluição de fásia. Aqui, o número médio de placas formadas para os 10 a menos três e 10 para as menos quatro placas de diluição, foram divididos por seus respectivos fatores de diluição para obter o número de unidades formadoras de placas, ou PFUs, em 100 microliters de mistura de phage. Para converter o valor em PFU por mililitro, multiplique os valores gerados por 10, pois apenas 100 microliters de mistura de diluição de fágio foram utilizados durante a etapa de preparação de sobreposição de bacteriófago, produzindo um fator adicional de diluição de 10. Por fim, calcule a média dos valores obtidos das diferentes placas de diluição. Isso dará o número médio de PFUs por mililitro. O número de PFUs corresponde ao número de partículas de phage infecciosas na amostra original.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
Apesar dos múltiplos avanços tecnológicos, os ensaios de placas continuam sendo o padrão-ouro para a determinação do título viral (como PFU) e essencial para o isolamento de populações de bacteriófagos puros. Células hospedeiras suscetíveis são cultivadas na camada superior de uma placa de ágar de duas camadas, formando uma cama homogênea que permite a replicação viral. O evento inicial em que um bacteriófago isolado no ciclo de vida lítico infecta uma célula, se replica dentro dela e, eventualmente, a lise, é muito pequena para ser observada. No entanto, os virions liberados infectarão células adjacentes, posteriormente dando origem a uma zona de desobstrução, ou placa, denotando a presença de um único PFU.
Subscription Required. Please recommend JoVE to your librarian.
References
- Twort, F. An investigation on the nature of ultra-microscopic viruses. Lancet. 186 (4814): 1241-1243. (1915)
- d'Hérelle, F. An invisible antagonist microbe of dysentery bacillus. Comptes Rendus Hebdomadaires Des Seances De L Academie Des Sciences. 165: 373-375. (1917)
- Cisek AA, Dąbrowska I, Gregorczyk KP, Wyżewski Z. Phage Therapy in Bacterial Infections Treatment: One Hundred Years After the Discovery of Bacteriophages. Current Microbiology. 74 (2):277-283. (2017)
- Mirzaei MK, Maurice CF. Ménage à trois in the human gut: interactions between host, bacteria and phages. Nature Reviews Microbiology. 15 (7):397. (2017)
- Breitbart M, Bonnain C, Malki K, Sawaya NA. Phage puppet masters of the marine microbial realm. Nature Microbiology. 3 (7):754-766. (2018)
- Leung CY, Weitz JS. Modeling the synergistic elimination of bacteria by phage and the innate immune system. Journal of Theoretical Biology. 429:241-252. (2017)
- Dulbecco R. Production of Plaques in Monolayer Tissue Cultures by Single Particles of an Animal Virus. Proceedings of the National Academy of Sciences of the United States of America. 38 (8):747-752. (1952)
- Juarez D, Long KC, Aguilar P, Kochel TJ, Halsey ES. Assessment of plaque assay methods for alphaviruses. J Virol Methods. 187 (1):185-9. (2013)
- Clokie MRJ, Millard AD, Letarov AV, Heaphy S. 2011. Phages in nature. Bacteriophage. 1 (1):31-45. (2011)
- Moldovan R, Chapman-McQuiston E, Wu XL. On kinetics of phage adsorption. Biophys J. 93 (1):303-15. (2007)
- Garneau JE, Dupuis M-È, Villion M, Romero DA, Barrangou R, Boyaval P, Fremaux C, Horvath P, Magadán AH, Moineau S.. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320):67. (2010)





 (ie)
(ie)