Introduction
Resistencia Myxovirus (Mx) las proteínas son una parte importante de la defensa inmune innata contra los patógenos virales. Estas proteínas son grandes GTPasas-dynamin como que son inducidos por el tipo I y tipo III interferones. Los genes MX correspondientes están presentes en casi todos los vertebrados en una o varias copias y sus productos génicos inhiben una amplia gama de virus, incluyendo Orthomyxoviridae (por ejemplo., Virus de la gripe), Rhabdoviridae (por ejemplo., Virus de la estomatitis vesicular), Bunyaviridae (por ejemplo. , el virus de la crosse) y Retroviridae (por ejemplo, virus de inmunodeficiencia humana-1) 1-4. No está claro cómo estas proteínas reconocen una amplia gama de virus tales, sin ninguna secuencia primaria compartida aparente motivos en estos virus. El análisis de la interacción de proteínas Mx con sus objetivos virales, potencialmente involucra complejos de orden superior con otros factores de la célula huésped, le ayudará a comprender los mecanismos moleculares tsombrero de haber evolucionado en la carrera armamentista entre los virus y sus anfitriones.
La interacción entre las proteínas de mamíferos Mx y objetivos virales se ha estudiado más ampliamente para MxA humano. MxA humano puede inhibir la replicación de muchos virus, incluyendo el ortomixovirus influenza A y virus Thogoto. MxA une los complejos de ribonucleoproteína virus Thogoto (vRNPs), impidiendo de este modo su entrada nuclear, lo que resulta en el bloque 5 de la infección. Esta interacción entre MXa y Thogoto vRNPs virus se ha demostrado con co-sedimentación y co-inmunoprecipitación experimentos 6-9. Cómo proteínas Mx obstaculizan virus de influenza A que está menos claro. Un problema importante es que no es fácil de demostrar una interacción entre una proteína Mx y un producto génico influenza. Un informe mostró una interacción entre MxA humana y la proteína NP de la gripe A células infectadas por virus 10. Esta interacción sólo se pudo demostrar por co-immunoprecipitation si las células habían sido tratados con los ditiobis reactivo de reticulación (propionato de succinimidilo) antes de la lisis, lo que sugiere que la interacción es transitorio y / o débil. Estudios más recientes han demostrado que el diferencial Mx sensibilidad de las distintas cepas de la gripe A se determina por el origen de la proteína NP 11,12. En línea con esto, los virus de la influenza A pueden escapar parte del control Mx mutando residuos específicos en la proteína NP 13. Esto sugiere que el principal objetivo de los virus de la gripe A para el host Mx es la proteína NP, probablemente NP montado en complejos vRNP. Sin embargo, ninguno de estos estudios más recientes demostraron una interacción entre la gripe NP o vRNPs y, o bien MxA humano o de ratón Mx1.
Recientemente hemos demostrado, por primera vez, una interacción entre la influenza NP y la proteína Mx1 de ratón con un protocolo de co-inmunoprecipitación optimizado 14, que se describe aquí en detalle. En general, co-immunoprecipitation es uno de los enfoques bioquímicos utilizados más frecuentemente para investigar las interacciones proteína-proteína. Esta técnica se prefiere a menudo sobre las técnicas alternativas, por ejemplo, dos híbridos de levadura, ya que permite investigar las interacciones proteína-proteína en su entorno natural. Co-inmunoprecipitación se puede llevar a cabo en las proteínas expresadas endógenamente si los anticuerpos contra las proteínas de interés están disponibles. Alternativamente, las proteínas de interés se pueden expresar en la célula a través de la transfección o infección y una etiqueta de afinidad pueden ser utilizados. Además de las ventajas mencionadas anteriormente, el protocolo de co-inmunoprecipitación descrita permite la detección de interacciones débiles y / o transitorios de proteínas. El componente principal de este protocolo optimizado es la adición de N-etilmaleimida (NEM) en el tampón de lisis celular. NEM es un reactivo de alquilación que reacciona con los grupos tiol libres tales como presente en cisteínas, a un pH de 6.5 a 7.5, para formar un éster tio-estable(Figura 1). A mayor pH, NEM también puede reaccionar con grupos amino o someterse a hidrólisis 15. NEM se utiliza típicamente para bloquear los grupos tiol libres, con el fin de prevenir la formación de enlaces disulfuro o inhibir la actividad enzimática. Por ejemplo, NEM se utiliza a menudo para bloquear enzimas desumoylating, que son proteasas de cisteína. En el protocolo de co-inmunoprecipitación descrita, NEM se incluyó en el tampón de lisis porque había sido informado de que la sumoilación de las proteínas de la gripe puede influir en la interacción entre las proteínas virales 16. Inesperadamente, la adición de NEM resultó ser clave para documentar la interacción entre la gripe NP y el ratón Mx1 por co-inmunoprecipitación. No está claro por qué la adición de NEM es crucial para detectar la interacción NP-Mx1. Posiblemente la interacción es demasiado transitoria y / o débil. NEM podría estabilizar la interacción, por ejemplo, mediante la preservación de una conformación específica de Mx1, una proteína viral o incluso un tercero desconocido componente. Tal efecto estabilizador de NEM se ha observado antes, por ejemplo, para la interacción entre la ribonucleótido reductasa M1 y su gemcitabina inhibidor (F2dC) 17. Mx1 y NP ambos contienen múltiples residuos de cisteína que podrían ser modificados por NEM. Por ejemplo, un estudio reciente de Rennie et al. Demostró que una variante sin pecíolo MxA contiene tres residuos de cisteína expuestos de disolventes que pueden ser modificados por yodoacetamida. La mutación de estos residuos de serinas no influyó en la actividad enzimática de MxA, pero impide la agregación mediada por disulfuro de 18. Como estas cisteínas se conservan en Mx1, esto sugiere que las cisteínas análogos en Mx1 pueden ser modificados por NEM y como tal influencia su conformación o solubilidad. Además, NEM también podría afectar a la actividad GTPasa de Mx1, que es esencial para la actividad anti-influenza de Mx1, y con ello estabilizar la interacción entre Mx1 y NP. Sin embargo, un efecto directo de NEM en la GTPasa actividad de Mx1 es improbable, ya que también se requiere NEM para detectar la interacción entre la gripe NP y GTPasa mutantes inactivas de la proteína Mx1 14. Claramente, se necesita más investigación para desentrañar el efecto de NEM en la interacción NP-Mx1.
En resumen, el protocolo de co-inmunoprecipitación descrita permite estudiar la interacción entre la proteína antiviral Mx1 y su diana viral, la proteína NP de la gripe. Este protocolo también podría ser utilizado para estudiar otras interacciones débiles o transitorios que dependen de la estabilización de las conformaciones de proteínas específicas. Interacción proteína-proteína que dependen de conformaciones específicas se han descrito antes, por ejemplo, para las proteínas de unión a calcio tales como calmodulina 19. Por último, el papel beneficioso de NEM también podría ser utilizado en otros métodos que detectan interacciones proteína-proteína, tales como ensayos de co-sedimentación.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Nota: El siguiente protocolo de transfección y co-inmunoprecipitación se establece para un formato de placa de Petri de 9 cm. Otros formatos también son posibles después de escalar el protocolo.
1. Siembra el riñón embrionario humano (HEK) Células 293T
- Sembrar el células HEK293T un día antes de la transfección a 1,2 x 10 6 células por 9 cm placa de Petri en 12 ml de medio de Eagle modificado por Dulbecco (DMEM) suplementado con suero de ternera fetal al 10%, 2 mM L-glutamina, 0,4 mM Na-piruvato, 0,1 mM de aminoácidos no esenciales, 100 U / ml de penicilina y 0,1 mg / ml de estreptomicina.
- Cultivar las células 16 horas a 37 ° C y 5% de CO 2.
- Inspeccione visualmente la morfología y la viabilidad de las células con un microscopio de luz invertida antes de la transfección. Las células necesitan para ser sub-confluente para una óptima eficiencia de transfección.
2.-fosfato de calcio transfección de células HEK293T
Nota: Uso 0,5-1 g de pCAXL-NP o plásmido vacío pCAXL en combinación con 1.3 g de pCAXL-Mx1 por 9 cm plato. Utilice una cantidad igual de ADN plasmídico total en todas las muestras; ajustar con el plásmido vacío si es necesario.
- Prepare los siguientes tampones de transfección:
- Preparar Tris-EDTA (TE) con concentraciones de 1,0 mM Tris-HCl pH 8,0 y EDTA 0,1 mM pH 8,0.
- Preparar BS / HEPES con concentraciones de 25 mM HEPES (5,96 g / L, 4- (2-hidroxietil) -1-piperazinetanosulfónico), NaCl 274 mM (16 g / L), 10 mM KCl (0,74 g / L), 1,5 mM NaHPO 4 · 12H 2 O (0,5 g / L) y dextrosa 11,1 mM (2 g / L). Ajustar el pH a 7,05.
- Preparar CaCl 2 / HEPES con concentraciones de 1,25 M CaCl 2 · 2H 2 O (183,8 g / L) y 125 mM HEPES (29,79 g / L). Ajustar el pH a 7,05 con NaOH.
- Calentar los buffers de transfección a 37 ° C antes de su uso.
- Preparar las muestras de plásmido mediante la dilución del ADN plásmido en 600 l de TE.Preparar estas mezclas en pocillos de una placa de 6 pocillos.
- Añadir 150 l de CaCl 2 / HEPES en un gota a gota de manera que las muestras de plásmidos y mezclar pipeteando 3 veces arriba y abajo.
- Preparar la solución de transfección a gota la adición de la solución sabia plásmido (TE + ADN + CaCl 2 / HEPES; 750 l) a 750 l de tampón BS / HEPES proporcionados en una placa de 6 pocillos fresco. Distribuir el plásmido de manera uniforme sobre el buffer / HEPES BS así que contiene completa.
- Agite la solución de transfección en un agitador de placas durante 90 segundos a 1000 RPM.
- Incubar la mezcla durante 5 min a temperatura ambiente.
- Añadir la solución de transfección (1,5 ml) gota a gota a las células. Utilice una micropipeta P1000 a gotear la solución de transfección en las células. Dispersar la mezcla sobre la completa 9 cm placa de Petri y agitar la placa muy suavemente.
- Se incuban las células a 37 ° C y 5% de CO 2 durante 6 horas. A continuación, retire el medio por aspiración y sustituir de inmediato wiº 12 ml de medio fresco, pre-calentado. Añadir con cuidado el medio fresco a las células para evitar el desprendimiento de células. Para ello, mantenga la punta de la pipeta contra el lado del pozo y empuje suavemente hacia fuera el medio.
- Se incuban las células durante una 16 h adicionales a 37 ° C y 5% de CO 2.
3. Co-inmunoprecipitación
Nota: Realice el coinmunoprecipitación 24 horas después de la transfección.
- Preparación de la baja tampón de lisis sal y tampón de lavado alta en sal.
- Preparar una solución madre de 2 M N-etil-maleimida (NEM) pesando la cantidad de NEM y disolviéndolo en etanol absoluto. Preparar la solución madre NEM fresco antes de su uso.
PRECAUCIÓN: NEM es muy tóxica, preparar y utilizar esta solución madre en una campana de humos. - Preparar baja tampón de lisis a concentraciones de sal mM Tris-HCl 50 pH 8, NaCl 150 mM, ácido etilendiaminotetraacético 5 mM (EDTA), 1% de NP40 y un cóctel inhibidor de la proteasa (1 disolver tablat en tampón de lisis 50 ml). Añadir NEM a una concentración final de 25 mM (es decir, diluir 1:80). Mantener en hielo después de la adición de los inhibidores de la proteasa y NEM.
Nota: Siempre agregue los inhibidores de la proteasa y NEM recién antes de su uso. - Preparar un tampón de lavado de sal alta en concentraciones de 50 mM Tris-HCl pH 8, NaCl 500 mM, EDTA 5 mM y 1% de NP40. Tenga en cuenta que el tampón de lavado alta en sal no contiene NEM.
- Preparar una solución madre de 2 M N-etil-maleimida (NEM) pesando la cantidad de NEM y disolviéndolo en etanol absoluto. Preparar la solución madre NEM fresco antes de su uso.
- Preparación de lisados celulares.
- Retire el medio y se lavan las células con 2 ml de hielo frío buffer fosfato salino (PBS). Añadir Muy suavemente el tampón de lavado, como células HEK293T se desprenden fácilmente.
- Retire el PBS y añadir 600 l de tampón enfriado con hielo bajo lisis sal por 9 cm placa de Petri.
- Se incuban las placas durante 20 minutos en hielo. Asegúrese de que las placas se mantienen horizontal, para asegurar la cobertura completa de la superficie de la placa con tampón de lisis. Agite suavemente las placas cada 5 minutos.
- Recoger el lisado de células en una microcentrífuga de 1,5 mltubo y centrifugar durante 3 min a 4 ° C y 16.000 xg para sedimentar la fracción insoluble.
- Transferencia de la fracción soluble, es decir, el lisado celular a un tubo de microcentrífuga de 1,5 ml y mantenerla en hielo. Inmediatamente continuar con el protocolo de co-inmunoprecipitación, para evitar la disociación de las proteínas que interactúan. Realice todos los pasos siguientes en lo posible en el hielo o a 4 ° C para limitar la actividad proteolítica en los lisados.
- Generación de complejos inmunes.
Nota: En este paso, la proteína de interés está vinculado por el anticuerpo apropiado. Para estudiar la interacción NP-Mx1, utilizar un anticuerpo monoclonal anti-NP ratón.- Para cada muestra, mezclar 135 l de lisado con 2 l de anti-NP anticuerpo monoclonal y 113 l de tampón de lisis de baja sal (volumen total de 250 l). Almacenar el lisado restante a -20 ° C para su posterior análisis como el Western Blot, para documentar los niveles de expresión de la Intera conjeturadosocios cción en las células transfectadas.
Nota: Como alternativa, medir la concentración de proteína del lisado (por ejemplo, con el reactivo de Bradford) y el uso de una cantidad fija de proteína total, por ejemplo, 400 g, para cada lisado. - Se incuba la mezcla de anticuerpos lisado durante 3 horas en una rueda giratoria a 4 ° C. Este paso se puede extender a una incubación durante la noche.
- Para cada muestra, mezclar 135 l de lisado con 2 l de anti-NP anticuerpo monoclonal y 113 l de tampón de lisis de baja sal (volumen total de 250 l). Almacenar el lisado restante a -20 ° C para su posterior análisis como el Western Blot, para documentar los niveles de expresión de la Intera conjeturadosocios cción en las células transfectadas.
- Preparación de la proteína G cuentas.
Nota: Las perlas de proteína G se envían y se almacenan en etanol al 20% para su conservación. El talón suspensión normalmente consiste de 50% de perlas y estas perlas necesitan ser lavados antes de ser utilizados para inmunoprecipitar los complejos inmunes.- Utilice 50 l de perlas, es decir, 100 l de perlas en suspensión, para cada muestra. Lavar la cantidad de perlas necesarios para todas las muestras en el ensayo de co-inmunoprecipitación en un solo tubo. Cortar la punta de una punta de pipeta de 1 ml para facilitar el pipeteado de la perla de la lechada.
- Centrifugar la proteína G perlas suspensión a 8000 xg y 4 ° C durante 30 seg. Retirar la solución de etanol y añadir un volumen igual de tampón de lisis de baja sal. Centrifugar la proteína G bead-suspensión a 8000 xg y 4 ° C durante 30 segundos y retirar el sobrenadante con suavidad. Repita este paso de lavado 3 veces.
Nota: El tampón de lisis de baja sal utilizada para lavar las perlas no necesita contener inhibidores de la proteasa o NEM. - Estimar el volumen de la proteína G cuentas y añadir un volumen igual de tampón de baja lisis sal para hacer un nuevo 50% del grano-suspensión en tampón de baja lisis sal.
- Para cada muestra, la transferencia de 100 l de perlas en suspensión en un tubo de microcentrífuga de 1,5 ml fresco y almacenar en hielo hasta su uso. Tenga cuidado para resuspender la suspensión de perlas antes de dividir, ya que estas perlas se sedimentan rápidamente a la parte inferior del tubo.
- La inmunoprecipitación de los complejos inmunes de perlas de proteína G y su elución.
- Antes de utilizar las proteínas G bolas para la inmunoprecipitación, todos los tubos de centrífuga de 30 segundos a 8000 xg y 4 ° C yverificar mediante inspección visual de que hay una cantidad igual de perlas presente en todas las muestras. Si es necesario ajustar la cantidad de perlas en algunas de las muestras y se centrifuga de nuevo. Deseche los sobrenadantes. Tenga cuidado de no molestar a las proteínas G bolas sedimentadas.
- Centrifugar brevemente los complejos inmunes (es decir., Lisados con el anticuerpo, 250 l) durante 30 segundos a 8000 xg y 4 ° C para recoger la muestra completa en la parte inferior del tubo. La transferencia de los complejos inmunes a las perlas proteína G (50 l).
- Incubar 60 min en una rueda de inflexión a 4 ° C. No incubar estos complejos inmunes más de 75 min con las perlas para reducir la unión no específica de proteínas a la proteína G cuentas.
- Centrifugar las perlas de proteína G (con complejos inmunes ligados) durante 30 segundos a 8000 xg y 4 ° C y retirar los sobrenadantes. Tenga cuidado de no molestar a las proteínas G bolas sedimentadas. Opcional: almacenar estos sobrenadantes a 4 ° C o -20 ° C para su posterior análisis,por ejemplo, para estimar la cantidad de proteína no unida.
- Lavar las proteínas G bolas durante aproximadamente 5 min con 900 l de tampón de lisis de alta sal. Asegúrese de que las cuentas están completamente resuspendidas en el tampón de lavado para el lavado óptimo. Centrifugar las proteínas G bolas durante 30 segundos a 8.000 xg y 4 ° C y descartar el sobrenadante. Repita este paso de lavado 4 veces. Tenga cuidado de no alterar las perlas de proteína G sedimentadas para evitar la pérdida de material inmunoprecipitado.
- Después de la última etapa de lavado, se añaden 50 l de tampón de muestra 2x Laemmli a las perlas y se calienta la suspensión durante 10 min a 95 ° C para eluir los (co) proteínas inmunoprecipitadas.
- Preparar 10 ml de tampón de Laemmli 6x con 1 g de dodecil-sulfato de sodio, 3,5 ml de glicerol, 3,5 ml de 1 M Tris-HCl pH 6,8 y 420 l de β-mercaptoetanol. Ajustar a un volumen total de 10 ml por adición de agua destilada. Diluir 3 veces en agua destilada para obtener tampón Laemmli 2x.
PRECAUCIÓN: &# 946; mercaptoetanol es tóxico, preparar y usar tampón Laemmli en una campana de humos.
- Preparar 10 ml de tampón de Laemmli 6x con 1 g de dodecil-sulfato de sodio, 3,5 ml de glicerol, 3,5 ml de 1 M Tris-HCl pH 6,8 y 420 l de β-mercaptoetanol. Ajustar a un volumen total de 10 ml por adición de agua destilada. Diluir 3 veces en agua destilada para obtener tampón Laemmli 2x.
- Después de calentar, centrifugar las proteínas G bolas durante 30 segundos a 8.000 xg y almacenar las muestras a 4 ° C (corto plazo) o -20 ° C (a largo plazo).
4. Analizar la (co) inmunoprecipitaron proteínas
- Visualizar las proteínas presentes en el lisado de células y eluato de la co-inmunoprecipitación por SDS-PAGE 20 y el oeste de transferencia de 21,22. Normalmente cargar medio del eluido Laemmli en gel. Tenga cuidado de no molestar a los granos G de proteínas sedimentadas al tomar muestras para carga de gel. Mx1 y expresión NP se revelaron con un anti-Mx1 y anticuerpo-NP anti, respectivamente 14. Las bandas se detectaron con quimioluminiscencia basado-HRP y un desarrollador de película de rayos X.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
N-etil-maleimida es un compuesto orgánico que se puede utilizar para modificar irreversiblemente grupos tiol libres, por ejemplo, para inhibir las proteasas de cisteína (Figura 1).
La proteína antiviral Mx1 inhibe la replicación del virus influenza A mediante la interacción con la nucleoproteína viral. El protocolo coinmunoprecipitación optimizado descrito aquí permite estudiar esta interacción NP-Mx1. Células HEK293T fueron transfectadas con vectores de expresión para la proteína antiviral Mx1 en la ausencia o presencia de la proteína NP de la gripe. A continuación, la proteína NP fue tirado hacia abajo a partir de lisados de células totales con un anticuerpo monoclonal NP-específico. Figura 2 muestra que la proteína es sólo Mx1 co-inmunoprecipitado en presencia de co-expresado NP. Posible inespecífica co-inmunoprecipitación de Mx1 en ausencia de NP es causada por cualquiera de inespecífica desplegable de la proteína Mx1 por el anticuerpo anti-NP o por unión no específica de Mx1 a laperlas de proteína G. Por lo tanto, siempre incluir un control negativo para evaluar esta inespecífica co-inmunoprecipitación. Figura 3 muestra que la interacción NP-Mx1 sólo puede ser detectado en presencia de NEM. En este experimento, la unión no específica de Mx1 a la proteína G cuentas se evaluó con una reacción de control co-inmunoprecipitación en ausencia de anticuerpo anti-NP.
Este protocolo también se puede usar para estudiar la interacción entre Mx1 y la proteína NP de la gripe aislado a partir de células infectadas o a partir de viriones. Para esta aplicación, el protocolo anterior fue ligeramente adaptado mediante la combinación de los lisados de células que expresan Mx1 con lisados que contienen la proteína NP viral antes de iniciar el protocolo de co-inmunoprecipitación. La Figura 4 muestra la co-inmunoprecipitación de Mx1 con NP aislado de las células transfectadas, las células infectadas o viriones.
En conclusión, estos resultados muestran que esta prot co-inmunoprecipitaciónocol se puede utilizar para estudiar la interacción entre una proteína antiviral y su diana viral.
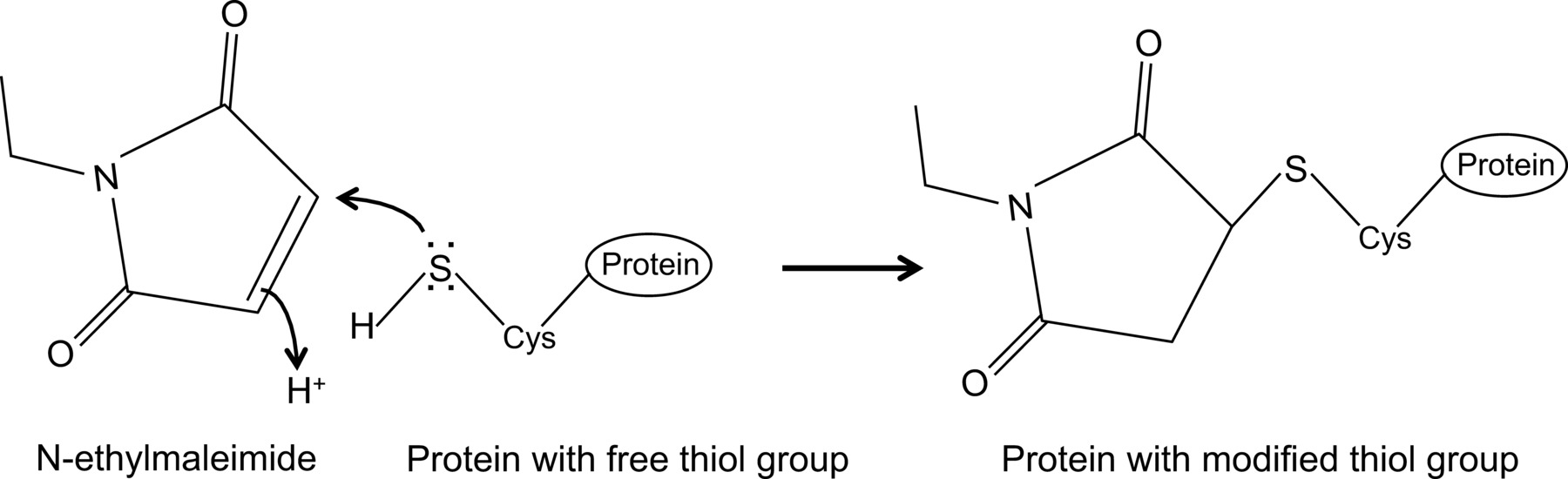
Figura 1:. Modificación irreversible de grupos tiol libres por N-etilmaleimida Haga clic aquí para ver una versión más grande de esta figura.

Figura 2:. Mx1 interactúa con NP Western blot de un experimento de co-inmunoprecipitación con dos muestras: una en la que tanto Mx1 y NP están presentes y una configuración de control en el que la proteína NP está ausente. NP se immunoprecipitated con anti-NP y NP y Mx1 se visualizaron por el Western Blot. Esta cifra se ha modificado from 14.

Figura 3: N-etil-maleimida es importante para detectar la NP - interacción Mx1 análisis de transferencia Western de un experimento de co-inmunoprecipitación realizada en presencia o ausencia de N-etilmaleimida.. NP se immunoprecipitated con anti-NP y NP Mx1 y se visualizaron por el Western Blot.

Figura 4:. Mx1 interactúa con NP aislado de las células infectadas o a partir de viriones que contienen lisados Mx1 se combinaron con lisados que contienen NP de diferentes fuentes: un lisado de control (sin vRNPs), transfectadas vRNPs, vRNPs a partir de células infectadas o vRNPs aislados a partir de viriones de la gripe A . Después de mezclar los lisados, co-immunopreprecipitación con anti-NP se realizó y Mx1 y NP se visualizaron por el Western Blot.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
El estudio de la interacción entre las proteínas antivirales y sus dianas virales es muy importante para entender los detalles del mecanismo antiviral de estas proteínas. Esto puede dar nuevas pistas sobre cómo coevolucionado virus y sus anfitriones y ser la base para el desarrollo de nuevas estrategias antivirales. El protocolo de co-inmunoprecipitación optimizado descrito aquí permite estudiar la interacción entre la proteína Mx1 de ratón y su diana viral, la proteína NP de la gripe. El aspecto más importante de este protocolo es la adición de NEM en el tampón de lisis, como la interacción NP-Mx1 es indetectable en ausencia de NEM (Figura 3). Hasta la fecha, no se sabe por qué la presencia de NEM es esencial para detectar esta interacción. Sin embargo, este protocolo podría ser útil para estudiar otras interacciones débiles y / o transitorios que dependen de la estabilización de las conformaciones de proteínas específicas, especialmente si las cisteínas están involucrados.
Unalimitación importante de los ensayos de co-inmunoprecipitación, en general, es la disponibilidad de anticuerpos específicos y de alta calidad que reconocen epítopos conformacionales en una de las parejas de interacción con alta afinidad. La interacción NP-Mx1 no se pudo demostrar con nuestro en casa producida antisuero policlonal anti-Mx1. Este antisuero también inmunoprecipita la proteína NP de la gripe, incluso en la ausencia de Mx1. Además, el anticuerpo monoclonal anti-NP que se utilizó, reconoce la proteína NP de la A / Puerto Rico / 8/34 cepa de la gripe, pero por desgracia, no es adecuado para tirar abajo NP de cepas del virus de la gripe aviar. Otra limitación de este protocolo de co-inmunoprecipitación es la unión no específica de Mx1 a la proteína G cuentas. Este último unión se puede superar mediante el aumento de la concentración de sal en el tampón de lavado y reducir el tiempo de contacto entre el lisado y la proteína G cuentas. Además, la reducción de la cantidad de perlas utiliza para cada inmunoprecipitaciónreacción de 50 l 25 l, puede disminuir aún más la unión no específica de Mx1 a estas cuentas. En general, el no deseado desplegable de proteínas contaminantes, causado por la unión no específica a la proteína G cuentas, también se puede reducir por otras estrategias. Por ejemplo, estas proteínas pueden ser removidos durante un paso de pre-clara, en la que estas proteínas se eliminan incubando el lisado con perlas de proteína G en ausencia de anticuerpo. Las proteínas contaminantes son eliminados junto con las perlas y el lisado y pre-autorizada se utiliza para la co-inmunoprecipitación. Esta estrategia sólo es ventajoso si las proteínas contaminantes son diferentes de la proteína (s) en estudio. Alternativamente, los sitios de unión no específica sobre la proteína G cuentas podrían ser bloqueadas con BSA. Sin embargo, esta estrategia sólo se recomienda si las perlas se recubrieron primero con el anticuerpo, como BSA también puede reducir la precipitación de complejos inmunes (es decir, de unión de anticuerpo reducido por las perlas recubiertas). Tomadojuntos, es muy importante incluir controles adecuados para excluir la unión no específica de las proteínas de interés a la proteína G cuentas o para los anticuerpos utilizados.
El protocolo de co-inmunoprecipitación descrito puede ser modificado para estudiar la interacción de Mx1 con la proteína NP presentes en un entorno diferente, por ejemplo, en células infectadas o en vRNPs purificados. Mx1 inhibe la expresión de proteínas virales, incluyendo NP, durante la infección por virus de la gripe 23. Por lo tanto, es técnicamente muy difícil de estudiar la interacción NP-Mx1 en las células transfectadas Mx1 que están infectadas posteriormente. Aún así, este protocolo de co-inmunoprecipitación también se puede realizar después de combinar los lisados de células que expresan Mx1 y células infectadas, lo que permite la detección exitosa de la interacción NP-Mx1 (Figura 4). Como no se conoce el objetivo crucial de NEM, se añadió NEM durante la lisis de ambas poblaciones celulares. Si se desea, el pH deel tampón de lisis se puede cambiar. Los experimentos descritos aquí se realizan a pH 8 (como en Turan et al. 10), pero la co-inmunoprecipitación NP-Mx1 también se llevó a cabo con éxito con un tampón de lisis pH 7,2. En realidad se prefiere un pH de 7,2, porque en este NEM pH reacciona exclusivamente con los grupos tiol libres y este pH también aumenta el rendimiento de la extracción de la proteína Mx1. Además, la proteína G cuentas podrían ser reemplazadas por perlas de proteína A, dependiendo de las especies hospedadoras de las que proceden los anticuerpos utilizados. Sin embargo, los resultados más limpios se obtienen con perlas de proteína G. Finalmente, este protocolo también se puede usar para estudiar la interacción entre Mx1 y otras proteínas de la gripe, por ejemplo, PB2 14. En este caso un epítopo V5 marcado se utilizó PB2, lo que podría, en principio, combinarse con gel de afinidad de agarosa anti-V5 disponible comercialmente.
En experimentos futuros, este protocolo podría ser valiosa para identificar las regiones en Mx1 yNP que son importantes para la interacción NP-Mx1. Una de estas regiones en Mx1 podría ser L4 bucle, tal como este bucle se ha demostrado ser crucial para la interacción entre MxA y virus Thogoto NP 8,9. Si los anticuerpos NP-específicos adecuados estén disponibles, este protocolo también permitirá determinar si el aumento de la sensibilidad de las cepas de influenza aviar A para Mx1 se correlaciona con una mayor interacción NP-Mx1 o no, lo que podría ayudar a comprender el mecanismo de la sensibilidad Mx. Además, la interacción entre Mx1 y componentes de virus que pertenecen a las familias distintas de la Orthomyxoviridae, puede también ser abordado por este protocolo co-inmunoprecipitación adaptado. Por último, más estudios para desentrañar el efecto de NEM en la NP-Mx1 será muy valiosa para comprender mejor esta interacción específica, sino también para obtener conocimientos en las aplicaciones más amplias de la adición de este compuesto durante la lisis celular y co-immunoprecipitation experimentos.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Los autores declaran que no tienen intereses financieros en competencia.
Acknowledgments
Este trabajo fue apoyado por FWO-Vlaanderen, el proyecto IOF IOF10 / stARTT / 027 y la Universidad de Gante Especial de Becas de Investigación BOF12 / GOA / 014.
Materials
| Name | Company | Catalog Number | Comments |
| DMEM high glucose | Gibco | 52100-047 | |
| N-Ethylmaleimide | Sigma | E-3876 | Toxic |
| Igepal CA-630 | Sigma | I-30212 | also known as NP40 |
| Protease inhibitor cocktail | Roche | 11 873 580 001 | |
| anti-NP monoclonal antibody | NIH Biodefense and Emerging Infections Research Resources Repository | NR-4282 | ascites blend of clones A1 and A3 |
| anti-RNP polyclonal serum | NIH Biodefense and Emerging Infections Research Resources Repository | NR-3133 | directed against A/Scotland/840/74 (H3N2) |
| Protein G Sepharose 4FF | GE Healthcare | 17-0618-01 | |
| Hyperfilm ECL 18 x 24 cm | GE Healthcare | 28-9068-36 | |
| ECL western blotting substrate | Pierce | 32106 |
References
- Verhelst, J., Hulpiau, P., Saelens, X. Mx proteins: antiviral gatekeepers that restrain the uninvited. Microbiol Mol Biol Rev. 77 (4), 551-566 (2013).
- Goujon, C., et al. Human MX2 is an interferon-induced post-entry inhibitor of HIV-1 infection. Nature. 502 (7472), 559-562 (2013).
- Kane, M., et al. MX2 is an interferon-induced inhibitor of HIV-1 infection. Nature. 502 (7472), 563-566 (2013).
- Liu, Z., et al. The interferon-inducible MxB protein inhibits HIV-1 infection. Cell Host Microbe. 14 (4), 398-410 (2013).
- Kochs, G., Haller, O. Interferon-induced human MxA GTPase blocks nuclear import of Thogoto virus nucleocapsids. Proc Natl Acad Sci U S A. 96 (5), 2082-2086 (1999).
- Flohr, F., Schneider-Schaulies, S., Haller, O., Kochs, G. The central interactive region of human MxA GTPase is involved in GTPase activation and interaction with viral target structures. FEBS Lett. 463 (1-2), 24-28 (1999).
- Kochs, G., Haller, O. GTP-bound human MxA protein interacts with the nucleocapsids of Thogoto virus (Orthomyxoviridae). J Biol Chem. 274 (7), 4370-4376 (1999).
- Mitchell, P. S., et al. Evolution-guided identification of antiviral specificity determinants in the broadly acting interferon-induced innate immunity factor MxA. Cell Host Microbe. 12 (4), 598-604 (2012).
- Patzina, C., Haller, O., Kochs, G. Structural requirements for the antiviral activity of the human MxA protein against Thogoto and influenza A virus. J Biol Chem. 289 (9), 6020-6027 (2014).
- Turan, K., et al. Nuclear MxA proteins form a complex with influenza virus NP and inhibit the transcription of the engineered influenza virus genome. Nucleic Acids Res. 32 (2), 643-652 (2004).
- Dittmann, J., et al. Influenza A virus strains differ in sensitivity to the antiviral action of Mx-GTPase. J Virol. 82 (7), 3624-3631 (2008).
- Zimmermann, P., Manz, B., Haller, O., Schwemmle, M., Kochs, G. The viral nucleoprotein determines Mx sensitivity of influenza A viruses. J Virol. 85 (16), 8133-8140 (2011).
- Manz, B., et al. Pandemic influenza A viruses escape from restriction by human MxA through adaptive mutations in the nucleoprotein. PLoS Pathog. 9 (3), e1003279 (2013).
- Verhelst, J., Parthoens, E., Schepens, B., Fiers, W., Saelens, X. Interferon-inducible protein Mx1 inhibits influenza virus by interfering with functional viral ribonucleoprotein complex assembly. J Virol. 86 (24), 13445-13455 (2012).
- Brewer, C. F., Riehm, J. P. Evidence for possible nonspecific reactions between N-ethylmaleimide and proteins. Anal Biochem. 18 (2), 248-255 (1967).
- Wu, C. Y., Jeng, K. S., Lai, M. M. The SUMOylation of matrix protein M1 modulates the assembly and morphogenesis of influenza A virus. J Virol. 85 (13), 6618-6628 (2011).
- Chen, Z., Zhou, J., Zhang, Y., Bepler, G. Modulation of the ribonucleotide reductase M1-gemcitabine interaction in vivo by N-ethylmaleimide. Biochem Biophys Res Commun. 413 (2), 383-388 (2011).
- Rennie, M. L., McKelvie, S. A., Bulloch, E. M., Kingston, R. L. Transient dimerization of human MxA promotes GTP hydrolysis, resulting in a mechanical power stroke. Structure. 22 (10), 1433-1445 (2014).
- Gifford, J. L., Walsh, M. P., Vogel, H. J. Structures and metal-ion-binding properties of the Ca2+-binding helix-loop-helix EF-hand motifs. Biochem J. 405 (2), 199-221 (2007).
- Separating Protein with SDS-PAGE. , JoVE. Available from: http://www.jove.com/science-education/5058/separating-protein-with-sds-page (2014).
- Gallagher, S., Chakavarti, D. Immunoblot analysis. J Vis Exp. (16), (2008).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Pavlovic, J., Haller, O., Staeheli, P. Human and mouse Mx proteins inhibit different steps of the influenza virus multiplication cycle. J Virol. 66 (4), 2564-2569 (1992).



