Summary
Sammenligningen og optimeringen af to planteorganelle DNA-berigelsesmetoder er præsenteret: traditionel differentiel centrifugering og fraktionering af det totale gDNA baseret på methyleringsstatus. Vi vurderer den resulterende DNA-mængde og kvalitet, demonstrerer ydeevne ved kortlæsning af næste generations sekventering og diskuterer potentialet til brug i langlæsende enkeltmolekylære sekventering.
Abstract
Plantorganelle genomer indeholder store, gentagne elementer, der kan undergå parring eller rekombination for at danne komplekse strukturer og / eller sub-genomiske fragmenter. Organellærgener findes også i blandinger inden for en given celle eller vævstype (heteroplasmien), og en overflod af subtyper kan ændre sig under udvikling eller under stress (substøkiometrisk forskydning). Next generation sekvensering (NGS) teknologier er nødvendige for at opnå en dybere forståelse af organellar genom struktur og funktion. Traditionelle sekventeringsundersøgelser anvender adskillige metoder til opnåelse af organellært DNA: (1) Hvis en stor mængde startvæv anvendes, homogeniseres den og underkastes differentiel centrifugering og / eller gradientrensning. (2) Hvis der anvendes en mindre mængde væv ( dvs. hvis frø, materiale eller rum er begrænset), udføres samme fremgangsmåde som i (1) efterfulgt af hel-genom-amplifikation for at opnå tilstrækkeligt DNA. (3) Bioinformatikanalyse kan bruges til at seqUence det totale genomiske DNA og at analysere organellar læser. Alle disse metoder har iboende udfordringer og kompromiser. I (1) kan det være vanskeligt at opnå en sådan stor mængde startvæv; I (2) kunne helgenomdannelse indføre en sekvenseringsforspænding; Og i (3) kunne homologi mellem nukleare og organelle genomer interferere med samling og analyse. I planter med store atomgener er det fordelagtigt at berige for organellært DNA for at reducere sekventeringsomkostninger og sekvenskompleksitet til bioinformatikanalyser. Her sammenligner vi en traditionel differenscentrifugeringsmetode med en fjerde metode, en tilpasset CpG-methyl pulldown-tilgang, for at adskille det totale genomiske DNA i nukleare og organelle fraktioner. Begge metoder giver tilstrækkeligt DNA til NGS, DNA, der er stærkt beriget for organellære sekvenser, omend i forskellige forhold i mitokondrier og chloroplaster. Vi præsenterer optimeringen af disse metoder til hvedebladevæv og diskuterer store fordele og dUlemper ved hver tilgang i forbindelse med prøveindgang, protokollethed og downstream-applikation.
Introduction
Genomsekvensering er et kraftfuldt værktøj til at dissekere det underliggende genetiske grundlag for vigtige planteegenskaber. De fleste genom-sekventeringsundersøgelser fokuserer på det nukleære genomindhold, da størstedelen af gener er placeret i kernen. Organellære genomer, herunder mitokondrierne (på tværs af eukaryoter) og plastider (i planter, den specialiserede form, chloroplasten, arbejder i fotosyntese) bidrager imidlertid til væsentlig genetisk information, der er afgørende for organisationsudvikling, stressrespons og overordnet fitness 1 . Organellærgener er typisk inkluderet i totale DNA-ekstraktioner beregnet til nuklear genom sekventering, selv om metoder til reduktion af organelle tal før DNA-ekstraktion også anvendes 2 . Mange undersøgelser har anvendt sekvenseringsresultater fra totale gDNA-ekstraktioner til at samle organellære genomer 3 , 4 , 5 ,Xref "> 6 , 7. Men når målet for undersøgelsen er at fokusere på organellære genomer, øges sekventeringsomkostningerne, fordi mange læsninger er" tabte "til de nukleare DNA-sekvenser, især i planter med store nukleare genomer På grund af overlapningen og overførslen af organellære sekvenser i det nukleare genom og mellem organeller, er det desuden en af opkøling af den korrekte kortlægningsposition for sekventering, der læser det korrekte genom, bioinformatisk udfordrende 2 , 8. Rensningen af organellære genomer fra det nukleære genom er en Strategi for at reducere disse problemer. Yderligere bioinformatik strategier kan bruges til at adskille læser det kort til regioner af homologi mellem mitokondrier og chloroplaster.
Mens organellærgenerne fra mange plantearter er blevet sekventeret, er der ikke meget kendt om bredden af organellær genom diversitetTilgængelig i vilde populationer eller i dyrkede avlspuljer. Organellære genomer er også kendt for at være dynamiske molekyler, der undergår væsentlig strukturel omlægning på grund af rekombination mellem gentagelsessekvenser 9 . Desuden er flere kopier af organellargenomet indeholdt i hver organel, og flere organeller er indeholdt i hver celle. Ikke alle kopier af disse genomer er identiske, som er kendt som heteroplasmisk. I modsætning til det kanoniske billede af "mastercirkler" er der nu voksende beviser for et mere komplekst billede af organellære genomstrukturer, herunder sub-genomiske cirkler, lineære kromosomer, lineære concatamere og forgrenede strukturer 10 . Samlingen af planteorganelle genomer kompliceres yderligere af deres forholdsvis store størrelser og væsentlige inverterede og direkte gentagelser.
Traditionelle protokoller til organellær isolering, DNA rensning og efterfølgende genom E-sekventering er ofte besværlig og kræver store mængder vævsindgang, med flere gram op til hundrede gram ungt bladvæv, der er nødvendigt som udgangspunkt 11 , 12 , 13 , 14 , 15 , 16 , 17 . Dette gør organellærgenomsekvensering utilgængelig, når væv er begrænset. I nogle situationer er frømængderne begrænsede, såsom når det er nødvendigt at sekvensere på generationsbasis eller i hansterile linjer, der skal opretholdes via krydsning. I disse situationer kan organellært DNA renses og derefter udsættes for hel-genom-amplifikation. Hele-genom-amplifikation kan imidlertid indføre signifikant sekventeringsforspænding, hvilket er et særligt problem ved vurdering af strukturelle variationer, sub-genomiske strukturer og heteroplasmieniveauer> 18. Nylige fremskridt i bibliotekets forberedelse til kortlæsede sekventeringsteknologier har overvundet lavindgangsbarrierer for at undgå hel-genom-amplifikation. For eksempel tillader Illumina Nextera XT-bibliotekets forberedelsessæt så lidt som 1 ng DNA, der skal anvendes som input 19 . Standardbiblioteksforberedelser til langlæsede sekventeringsapplikationer, såsom PacBio eller Oxford Nanopore-sekventeringsteknologier, kræver imidlertid stadig en relativt høj mængde input-DNA, som kan udgøre en udfordring for organellær genom-sekventering. For nylig er der udviklet nye brugerdefinerede, langlæsede sekventeringsprotokoller for at reducere inputmængderne og for at lette genometsekvensering i prøver, hvor opnåelse af mikrogrammængder af DNA er vanskelig 20 , 21 . Imidlertid er opnåelse af højmolekylære, rene organellære fraktioner til føde i disse bibliotekspræparationer fortsat en udfordring.
Vi søgte tO sammenligne og optimere organellære DNA berigelses- og isoleringsmetoder egnede til NGS uden behov for hel-genom-amplifikation. Specifikt var vores mål at bestemme bedste praksis for at berige for organellært DNA med høj molekylvægt fra begrænsede udgangsmaterialer, såsom en subsample af et blad. Dette arbejde præsenterer en komparativ analyse af metoder til at berige for organellært DNA: (1) en modificeret, traditionel differentieringsscentrifugeringsprotokol versus (2) en DNA-fraktioneringsprotokol baseret på anvendelsen af et kommercielt tilgængeligt DNA-CpG-methylbindende domæneprotein-udløbsfremgangsmåde 22 påført plantevæv 23 . Vi anbefaler bedste praksis til isolering af organellært DNA fra hvedebladvæv, som let kan udvides til andre planter og vævstyper.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Generering af plantematerialer til organellarisolering og DNA-ekstraktion
- Standard vækst af hvedeplanter
- Plantefrø i vermikulit i små, firkantede gryder med 4 - 6 frø pr. Hjørne. Overførsel til et drivhus eller vækstkammer med 16 h lyscyklus, 23 ºC dag / 18 ºC nat.
- Vand planterne hver dag. Gød planterne med ¼ tsk granuleret 20-20-20 NPK-gødning ved spiring og efter 7 dage efter spiring.
- Alternativ ætsning af hvedeplanter
- Følg trin 1.1, men placer potterne i et mørkt vækstkammer, 23 ° C i 16 timer / 18 ºC i 8 timer. Alternativt dækker planterne i drivhuset ( f.eks. Med en opbevaringsbeholder, dog skal der sikres korrekt ventilation).
- Vækst og vævsopsamling
- Væk planterne i 12-14 dage. For de fleste hvede genotypeS, 75 - 100 kimplanter giver omkring 10-12 g væv, hvilket er tilstrækkeligt til to organellære ekstraktioner ved anvendelse af differentieringscentrifugeringsmetoden (sektion 2); Kun en plante er nødvendig, hvis man anvender DNA-CpG-methyleringsbaseret pulldown-tilgang til fraktionering af organellar fra nukleært DNA (afsnit 3).
- Hvis man anvender differenscentrifugeringsmetoden, skal man samle væv frisk og fortsætte med at behandle prøverne som beskrevet i afsnit 2.
- Hvis man bruger CpG-methyl-nedlukningstilgangen, høst 20 mg sektioner af ungt bladvæv i mikrocentrifugerør (brug enten standardvokset eller ætioleret væv, se repræsentative resultater ). Snapfryse på flydende nitrogen og frys ved -80 ºC indtil brug. Fortsæt til pulldown fraktionering af DNA, som beskrevet i afsnit 3.
2. Metode nr. 1: DNA-ekstraktion ved anvendelse af differentiel centrifugering (DC)
BEMÆRK: diffErentialcentrifugeringsprotokol blev modificeret fra to publikationer, som optimerede betingelser for at isolere begge organeller, men beriget for mitokondrier 17 , 24 . Den resulterende protokol er mindre tidsintensiv og bruger færre toksiske kemikalier end de tidligere metoder. Specifikt har vi foretaget modifikationer af buffere og vaske trin, herunder tilsætning af polyvinylpyrrolidon (PVP) til STE-ekstraktionsbufferen og eliminering af slutvasketrin i NETF-buffer, der indeholder natriumfluorid (NaF).
Forsigtig: Forberedelse og brug af STE-puffer skal udføres under en kemisk dampplade med passende personlige værnemidler, da denne puffer indeholder 2-mercaptoethanol (BME).
- Ting at gøre før du starter
- Sørg for, at alt udstyr er ekstremt rent og autoklaver alt udstyr, som kan autoklaveres ( f.eks. Slibesylindere, high-speed centriFuge rør osv. ).
BEMÆRK: Filter-tips anbefales til alle trin, der kræver pipettering for at undgå krydskontaminering. - Se listen over nødvendige udstyr og reagenser, og lav de nødvendige buffere og arbejdsstande til Metode # 1 ( Tabel 1 ). Kyl de kryogene slibeblokke til -20 ºC og rotorerne og bufferne til 4 ºC, sæt mikrocentrifugen til 4 ºC, og tænd et 37 ºC vandbad.
- Sørg for, at alt udstyr er ekstremt rent og autoklaver alt udstyr, som kan autoklaveres ( f.eks. Slibesylindere, high-speed centriFuge rør osv. ).
- Isolering af organeller
- Høst 5 g frisk væv og skyll det i koldt, sterilt vand i et kølet bæger på is.
BEMÆRK: Hold altid prøverne på is under alle betjeninger og transporter til og fra centrifuger, dampkåler osv. Alternativt kan du arbejde i et forkølelokale, hvis der er adgang til tilstrækkelig plads og udstyr til at udføre protokollen. - Ved hjælp af saks skæres bladvæv i ~ 1 cm-stykker direkte ind i et 50 ml rør, der indeholder to keramiske slibningercylindre.
BEMÆRK: Rengør eller skift saks mellem prøver for at undgå krydskontaminering. - Hvis der ikke er vævshomogenisator, skal du bruge en mørtel og støv og følg for at erstatte trin 2.2.4 - 2.2.9.
- Skær bladvævet i en forkølet mørtel på is. Grind prøverne i 2 - 3 min i 15 ml STE (i dampdækslet).
- Hæld bufferen ud (efterlad vævet i mørtel) gennem en tragt, der indeholder et lag af for-våd, steril filtreringsklud (~ 22 til 25 μm porestørrelse, se hovedprotokollen for detaljer) i et andet 50 ml rør . Tilsæt yderligere 10 ml STE til mørtel og støv og homogeniser igen.
- Hæld det homogeniserede væv og puffer i samme tragt. Skyl morter og pistle med 10 ml STE og hæld det i tragten. Klem og vrid filtreringskluden ind i tragten for at genoprette så meget væske som muligt.
BEMÆRK: Skift handsker mellem prøver for at undgå krydskontaminering. Fortsæt med proTocol i trin 2.2.10.
- Tilsæt 20 ml STE (i dampdækslet) til hvert 50 ml rør.
- Anbring prøverne i forkølede kryogenkværneblokke i et vævsslibningsapparat og formaler prøverne i 2 x 30 s ved 1.750 omdr./min. Drej prøvepositionerne og læg prøverne på is i ~ 1 min mellem grinds.
BEMÆRK: En morter og pestle, blender eller anden vævsslibning / homogeniseringsindretning kan anvendes i dette trin. Hver metode vil imidlertid påvirke den resulterende DNA-kvalitet i forskellige grader, og derfor bør DNA-længde og kvalitet vurderes, før der fortsættes med downstream-applikationer. - Indsæt en tragt i et rent 50 ml rør placeret i is. Anbring et lag filtreringsklud i tragten og forvædet med 5 ml STE. Forsøg ikke gennemstrømningen.
- Hæld det homogeniserede væv i tragten. Skyl slibestrengen med 15 ml STE, saml og vend røret for at skylle væggene og låget og hæld i funnel.
- Fjern forsigtigt de keramiske sten og tryk derefter og vri filtreringskluden i tragten.
BEMÆRK: Skift handsker mellem prøver for at undgå krydskontaminering. - Sæt rørdækslerne sammen med parafilm for at undgå spild. Centrifuger ved 2.000 xg i 10 minutter ved 4 ºC.
- Sug forsigtigt supernatanten ved hjælp af en serologisk pipette (undgå at forstyrre pelleten) og læg den i et 50 ml højhastighedssentrifugerør (hvis rørene ikke har tætte tætnings pakninger, pakk rørhættene sammen med parafilm for at undgå spild). Kassér pellets.
- Balancer rørene inden for 0,1 g ved hjælp af STE og centrifuger den resulterende supernatant i 20 minutter ved 18.000 xg og 4 ºC. For at afbalancere rørene skal du placere et lille bægte is på balancen, tørre skalaen og veje prøverne på is for at holde dem kolde. Alternativt kan du bruge en balance og en dampplade i et koldt rum.
- Kassér supernatanten. Tilsæt 1 ml ST til pellet og gentag forsigtigt osI en blød pensel. Tilsæt 24 mL ST (slutvolumen på 25 mL) og bland / hvirv ( dvs. tryk på penslen på siden af røret for at fjerne al væske).
- Balancer rørene til inden for 0,1 g ved hjælp af ST. Centrifuge i 20 minutter ved 18.000 xg og 4 ºC. I mellemtiden forberede DNaseI-opløsning (se tabel 1 for lageropløsninger og arbejdsløsningsopskrifter). For hver prøve fremstilles en 200 μl alikvot i et 1,5 ml rør.
- Kassér supernatanten, blottet for røret og genopsæt pelleten (stadig i et centrifugerør med høj hastighed) i 300 μL ST ved hjælp af en blød pensel. Placér penslen i det tidligere fremstillede 1,5 ml rør indeholdende 200 μl DNaseI opløsning og rør penselboringen for at fjerne enhver resterende pellet fast i børsten. Pipetter DNaseI-opløsningen tilbage i højhastighedscentrifugerøret og rør forsigtigt for at blande.
- Inkuber ved 37 ° C i 30 minutter i et vandbad (wrap parafilm omkring toppen af røret for at forhindre kondensvandslækageG ind i hætten). Bland forsigtigt ved at hvirvle 2 gange under inkubation.
- Sæt forsigtigt pelletsblandingen ud af røret ved hjælp af en pipettespids med en bred åbning, og læg den i et 1,5 ml lavt rør. Tilsæt 500 μL 400 mM EDTA, pH 8,0, til centrifugerøret med høj hastighed og forsigtigt pipette for at få hele den resterende pellet ud af røret. Overfør EDTA til det samme 1,5 ml, lavt bindingsrør som pelletsblandingen og bland forsigtigt ved inversion.
- Centrifuger ved 18.000 xg i 20 minutter ved 4 ºC. Kassér supernatanten, blottet for røret og anvend straks til DNA-isolering. Hvis det er nødvendigt, frys pellets ved -20 ºC, men dette kan resultere i en udbytteduktion, da resterende DNaseI kan nedbryde prøve-DNA'et, hvis det ikke umiddelbart behandles.
- Høst 5 g frisk væv og skyll det i koldt, sterilt vand i et kølet bæger på is.
- DNA-ekstraktion fra isolerede organeller under anvendelse af en kommerciel søjlebaseret tilgang
BEMÆRK: Se kithåndbogen for fuld protokol 25 , og se nedenfor for ændringer. prAt overføre direkte fra organellærisolering til DNA-ekstraktion foretrækkes. Gentagen frysning og optøning vil reducere DNA-fragmentstørrelser og føre til DNA-nedbrydning ved resterende DNaseI. Begræns vortexing eller kraftig pipettering, da dette kan forskyde DNA'et. Anvendelsen af lavbindende mikrocentrifugerør anbefales til at maksimere DNA-genopretning.- DNA-ekstraktionsprocedure
BEMÆRK: Læs den detaljerede kommercielle protokol 25, før du begynder at sikre, at bufferne er korrekt lavet / lagret, og at spin-column-procedurerne forstås.- Tilsæt 180 μL Buffer ATL direkte i røret med pellet (optøet, hvis den tidligere er frosset og ækvilibreret til stuetemperatur på benken).
- Fortsæt med trin 3 i protokollen for "DNA-rensninger fra væv" i kithåndbogen med følgende modifikationer: en 30 min lysis i trin 3 indbefatter den valgfrie RNase A-fordøjelse og elueres i 3 x 200 μl AE ( Hver til en seParate tube og derefter kombinere elutions).
- Gem en alikvot (mindst 20 μL) for qPCR (se trin 4.1). For at kvantificere, inden du koncentrerer, gem yderligere 1 μL for kvantificering med høj følsomhed.
- Fortsæt med prøvekoncentration, hvis det ønskes.
- DNA-ekstraktionsprocedure
- Prøvekoncentration med kommercielle filterenheder
BEMÆRK: Se handelsprotokollen 26 for flere detaljer. Afhængig af downstream-brugen er det muligvis ikke nødvendigt at udføre prøvekoncentration ( f.eks. Til slutpunkts-PCR og qPCR-applikationer). For NGS bibliotekskonstruktion vil det sandsynligvis være nødvendigt at koncentrere fortyndet organellært DNA opnået efter DNA-ekstraktion.- Koncentrationssøjleprocedure
- Forsigtig forsigtigt (se Tabel 2 ) den tomme filterenhed (uden rør) på et rent stykke vægpapir på en digital analytisk balance. Optag vægten.
- PiPette de kombinerede elutioner ind i filterenheden og vejr omhyggeligt igen.
BEMÆRK: Den kommercielle manual 26 siger, at filterenhedens maksimale volumen er 500 μL, men op til 575 μL kan tilføjes til enheden på én gang uden overløb. - Anbring forsigtigt den fyldte filterenhed i et rør (forsynet med kolonnerne). Centrifuge ved 500 xg i den ønskede tid for at opnå det krævede koncentratvolumen. For et prøvevolumen på ~ 575 μL vil en 20 minutters spin normalt resultere i et koncentratvolumen på 15-30 μL.
- Fjern filterenheden fra røret og vej igen. Brug tabellen til at bestemme, om det ønskede koncentratvolumen er opnået. Hvis ikke, centrifuger igen ved 500 xg i kortere tid og vejer igen; Gentag indtil det ønskede koncentratvolumen er nået.
- Anbring et nyt rør (forsynet med kolonnerne) oven over filterenheden og vend omvendt. Centrifuge i 3 minutter ved 1.000 xg for at overføre coNcentreres til røret.
- Bestem det genvundne volumen. Dette vil normalt være ~ 3 - 5 μL mindre end det beregnede volumen på grund af filterretention. Hvis overkoncentreret, fortyndes med sterilt vand eller TE for at opnå det ønskede volumen.
- Kvantificer DNA'et ved hjælp af kvantificering med høj følsomhed (pr. Producentens anvisninger).
- Koncentrationssøjleprocedure
3. Metode nr. 2: Metylfraktionering (MF) tilnærmelse til berigelse af organellært DNA fra total genomisk DNA
BEMÆRK: Denne protokol blev modificeret fra en brugerudviklet DNA-ekstraktionsprotokol for genomisk tip-kit til planter og svampe 27 og den kommercielle Microbiome DNA Enrichment Kit-protokol 28 . I teorien kan enhver DNA-isolationsprotokol, som giver DNA med høj molekylvægt, anvendes til nedlukning. Til kortlæsnings-sekventering er enhver ekstraktion, der giver overvejende> 15 kb fragmenter, tilstrækkelig til anvendelse i pulldown. For loNg-læsede sekventering kan større fragmenter være ønskelige. Derfor optimerede vi denne protokol for at give DNA med høj molekylvægt.
- Isolering af total DNA
BEMÆRK: Se listen over krævede udstyr og reagenser, og forbered de nødvendige buffere og arbejdsstande til metode nr. 2 ( tabel 1 ). Tilsæt lyseringsenzymer til lysisbufferbeholdningen for at gøre lysisbufferens arbejdsløsning. Tænd termomixeren og indstil den til 37 ° C. Tænd vandbadet til 50 ° C og læg QF-buffer i badet. Anbring 70% EtOH i fryseren og sæt mikrocentrifugen til 4 ° C.- Total DNA-ekstraktion ved anvendelse af kommercielle DNA-ekstraktionskolonner
BEMÆRK: Før du starter, læs den kommercielle håndbog 29 for detaljerede oplysninger om brugen af gravitetsflowanionbytterkolonnerne. Kolonnerne kan opstilles ved hjælp af en specialstativ eller placeres over rørene ved hjælp af de medfølgende plastringe. Alle trin, herunder gEnomic tips, bør have lov til at fortsætte med tyngdekraft flow, og resterende væske bør IKKE tvunget igennem.- Slib 20 mg frosset væv i flydende nitrogen i et 2 ml lavt bindestrøb ved hjælp af håndholdte slibestøv beregnet til 2 ml rør.
- Tilsæt 2 ml lysis buffer arbejdsløsning (rørene vil være meget fulde).
- Inkuber i en termomixer ved 37 ° C i 1 time med forsigtig omrøring ved 300 omdr./min. Hvis en termomixer ikke er tilgængelig, er inkubation på en varmeblok og blanding ved let blinkning hvert 15. minut et passende alternativ.
- Tilsæt 4 μl RNase A (100 mg / ml, slutkoncentration på 200 μg / ml). Omvendt til blanding og inkubering i en termomixer i 30 minutter ved 37 ° C med forsigtig omrøring ved 300 omdr./min.
- Tilsæt 80 μl proteinase K (20 mg / ml, slutkoncentration på 0,8 mg / ml), omvendt til blanding og inkuber i en termomixer i 2 timer ved 50 ° C med forsigtig omrøring ved 300 omdr./min.
- Centrifuge i 20 minutter ved 4 ° C og 15.000 xg for at pille det uopløselige affald.
- Mens prøverne centrifugeres, ækvilibrere kolonnerne med 1 ml Buffer QBT og lad kolonnen tømme ved tyngdekraftstrømmen.
- Brug en pipettepipe med bredboring til hurtigt at anvende prøven (undgå pellet) til den ækvilibrerede søjle og lad den strømme helt gennem søjlen. Hvis prøven bliver uklar, filtrer eller centrifuger den igen før applikationen til kolonnen (se den kommercielle håndbog for detaljer 29 ).
- Når prøven er helt ind i harpiksen, vaskes søjlen med 4 x 1 ml Buffer QC.
- Suspension kolonnen over et rent, 2 ml lavt bindende mikrocentrifugerør. Eluer det genomiske DNA med 0,8 ml buffer QF forvarmet ved 50 ° C.
- Præcipiterer DNA'et ved at tilsætte 0,56 ml (0,7 volumener elueringsbuffer) af rumtemperaturisopropanol til det eluerede DNA.
- Bland ved inversion (10X) og centrifuger straks i 20 minutter ved 15.000 xg og 4 ° C. OmsorgFjern supernatanten helt uden at forstyrre den glasagtige, løst fastlagte pellet.
- Vask den centrifugerede DNA-pellet med 1 ml kold 70% ethanol. Centrifuger i 10 minutter ved 15.000 xg og 4 ° C.
- Fjern forsigtigt supernatanten (vær også forsigtig med dette trin) uden at forstyrre pelleten. Lufttørre i 5-10 minutter og resuspender DNA'et i 0,1 ml elueringsbuffer (EB). Opløs DNA'et natten over ved stuetemperatur. Undgå pipettering, som kan forskyde DNA'et.
- Kvantificer prøverne ved hjælp af en højfølsom DNA-kvantificeringsanalyse (pr. Producentens instruktioner).
- Total DNA-ekstraktion ved anvendelse af kommercielle DNA-ekstraktionskolonner
- Perlebaseret fraktionering af methyleret og ummethyleret DNA
BEMÆRK: En nylig offentliggørelse demonstrerede brugen af et kommercielt tilgængeligt kit 28, der udnytter en pulldown-fremgangsmåde ved anvendelse af et CpG-specifikt methylbindende domæneprotein fusioneret til det humane IgG Fc-fragment (MBD2-Fc-protein) til fraktionSpiste planteorganelle genomer (ummethyleret) fra nukleart genom (højt methyleret) indhold 23 . Fraktionseringseffektivitet i hvedeprøver blev ikke tidligere testet under anvendelse af dette kommercielle MF kit 28 .- Ting at gøre før du starter
- Tilbered frisk 80% ethanol (mindst 800 μl pr. Reaktion). Sæt 5x bind / vaskebuffer til optøning på is og tilbered 5 ml 1x buffer pr. Prøve (fortyndet 5X-buffer med sterilt nukleasefrit vand og hold på is under protokollen).
- Forbered MBD2-Fc proteinbundne magnetiske perler
- Forbered det ønskede antal perle sæt. Skalere reaktionerne til brug mellem 1 og 2 μg total input DNA, der kræver 160 - 320 μl perler. Bemærk, at reaktionerne nedenfor er for 1 μg af totalt input-DNA, så de kræver 160 μl perler. Skalere reaktionerne efter behov.
- Ved hjælp af brøndboretips pipetteres Protein A Magnetic B forsigtigtEad slurry op og ned for at skabe en homogen suspension. Som et alternativ drejes forsigtigt rørene af perler i 15 minutter ved 4 ° C.
BEMÆRK: Vortex ikke perlerne. - Fortsæt med anvisningerne efter producentens anvisninger 28 .
- Capture methylated nuclear DNA
- For hver enkelt prøve tilsættes 1 μg input DNA til et rør indeholdende 160 μl MBD2-Fc-bundet magnetiske perler.
- Tilsæt 5x bind / vaskebuffer efter behov givet volumenet af DNA-indgangsprøven for en slutkoncentration på 1x (volumen 5x bind / vaskebuffer for at tilføje (μL) = volumen af input-DNA (μL) / 4). Pipetter prøven op og ned et par gange for at blande ved hjælp af en pipette med en bred boring.
- Drej rørene ved stuetemperatur i 15 minutter. Forsigtigt pipetter prøverne med en brøndpipettip og flip prøverne 2 - 3 gange i løbet af inkubationen for at forhindre perlemaskning.
BEMÆRK: Pipettering og flickiNg er kritisk for at sikre effektiv nedlukning af det methylerede DNA.
- Samle beriget, ummethyleret organellært DNA
- Drej kort om røret indeholdende DNA og MBD2-Fc-bundet magnetisk perle blanding. Placer røret på et magnetstativ i mindst 5 minutter for at indsamle perlerne på siden af røret. Opløsningen skal fremgå tydelig.
- Brug brede boretips, fjern forsigtigt den rydde supernatant uden at forstyrre perlerne. Overfør supernatanten (indeholder ikke-methyleret, organellært beriget DNA) til et rent, lavt bindende, 2 ml mikrocentrifugerør. Opbevar denne prøve ved -20 eller -80 ° C, eller fortsæt direkte til trin 3.2.6 til oprensning.
- Elute indfanget nukleært DNA fra de MBD2-Fc-bundne magnetiske perler
- Hvis den nukleare fraktion også ønskes, følg producentens instruktioner 28 for at eluere det nukleare DNA fra de MBD2-Fc-bundne magnetiske perler; Oprensning som beskrevet i trin 3.2.7.
- Perlebaseret nukleinsyreoprensning
- Sørg for, at rensekuglerne er ved stuetemperatur og blandes grundigt. Fortsæt med protokollen efter instruktionerne i MF kit manualen 28 .
BEMÆRK: Prøven kan nu bruges til NGS-bibliotekskonstruktion eller en anden nedstrømsanalyse.
- Sørg for, at rensekuglerne er ved stuetemperatur og blandes grundigt. Fortsæt med protokollen efter instruktionerne i MF kit manualen 28 .
4. Prøvekvantificering og kvalitetskontrol
- QPCR assay til vurdering af organellær berigelse
BEMÆRK: De qPCR-reaktions- og analyseparametre, der er angivet her, blev designet til brug på en Roche LightCycler 480 og kan muligvis indstilles til forskellige udstyr og reagenser. Hvis qPCR ikke er tilgængelig, kan endpoint-PCR og visualisering på en agarosegel anvendes som et kvalitativt mål for prøverenhed ved anvendelse af de samme primere og betingelser beskrevet her. Amplicon størrelser vil være ~ 150 bp for alle primer sæt. Se tabel 3 for primer sekvensCes og parringer.- QPCR reaktionsopsætning
- For at oprette en individuel 20 μl qPCR reaktion pipetteres følgende omhyggeligt i en enkelt brønd i en 96-brønds qPCR plade: 10 μL 2x SYBR Green I Master; 2 μL af 10 μM for- og revers-primerblandingen (for en slutkoncentration på 0,5 μM); 2 μL skabelon (inden for standardkurvens område); og 6 pi sterilt, nukleasefri H2O For at reducere pipetteringsfejl, foretrækkes det at gøre en masterblanding med alle reaktionskomponenter undtagen template. Tilføj master mix til qPCR pladen, og tilføj derefter den skabelon af interesse for hver brønd. Tre tekniske replikater for hver prøve skal udføres for at minimere virkningerne af pipetteringsfejl.
BEMÆRK: I sidste ende sammenlignes forholdet mellem nukleare og organellære kvantifikationscykler mellem prøver, så små forskelle i koncentration er acceptable. DNA-koncentrationerne bør dog være inden for intervallet eaCh andre. - Pak pladen med en højkvalitets qPCR-tætningsfilm. Vortex forsigtigt prøverne og pas på at undgå at skabe bobler. Drej pladen kort i 2 minutter ved 4 ° C for at samle prøven og eliminere eventuelle små bobler.
- Læg pladen i maskinen. Kør qPCR-programmet i henhold til nedenstående retningslinjer.
- For at oprette en individuel 20 μl qPCR reaktion pipetteres følgende omhyggeligt i en enkelt brønd i en 96-brønds qPCR plade: 10 μL 2x SYBR Green I Master; 2 μL af 10 μM for- og revers-primerblandingen (for en slutkoncentration på 0,5 μM); 2 μL skabelon (inden for standardkurvens område); og 6 pi sterilt, nukleasefri H2O For at reducere pipetteringsfejl, foretrækkes det at gøre en masterblanding med alle reaktionskomponenter undtagen template. Tilføj master mix til qPCR pladen, og tilføj derefter den skabelon af interesse for hver brønd. Tre tekniske replikater for hver prøve skal udføres for at minimere virkningerne af pipetteringsfejl.
- QPCR reaktionsparametre
BEMÆRK: Disse er standardparametre, undtagen for forstærkningstrinnets glødningscyklus. Juster denne indstilling for at rumme specifikke primere, hvis de anvendte afviger fra de primere, der præsenteres i denne protokol.- Forinkuber ved 95 ° C i 5 minutter med en rampe på 4,4 ° C / s.
- Udfør 45 amplifikationscykler på (1) 95 ° C i 10 s med en rampefrekvens på 4,4 ° C / s; (2) 60 ° C i 20 s med en rampehastighed på 2,2 ° C / s; Og (3) 72 ° C i 10 s med en rampe på 4,4 ° C / s (data indhentet under (3)).
- Brug en optioNal smeltekurvecyklus på 95 ° C i 5 s med en rampehastighed på 4,4 ° C / s; 65 ° C i 1 min, med en rampe på 2,2 ° C / s; Og 97 ° C, med en kontinuerlig opkøbsmodus.
- Brug en kølecyklus på 40 ° C i 30 s, med en rampe på 1,5 ° C / s.
- Assay parametre
- Vælg SYBR-skabelonen. Kontroller programparametrene i Eksperiment-knappen. Når pladen er indlæst, kan analysen startes, og indstillingerne kan justeres, mens analysen kører.
- Tildel prøver ved hjælp af prøve editoren. Vælg Abs Quant som workflow og betegne prøverne som ukendte, standarder eller negative kontroller. Designér replikater og udfyld prøvenavnene på den første af hver replik. Tilføj koncentrationer og enheder til standarderne.
- Opsæt delmængder til analyse; Disse er tildelt i delværkteditoren.
- Til analyse skal du vælge Abs Quant / 2nd Derivative Max fra listen "Opret ny analyse".Importer den eksternt gemte standardkurve (hvis relevant), og tryk derefter på beregne; Rapporten indeholder de valgte oplysninger.
- For at udføre nøjagtig absolut kvantificering til bestemmelse af kopiantal eller koncentration skal der anvendes en standardkurve, som er repræsentativ for den prøve, der testes ( fx organellært DNA isoleret fra ovennævnte metoder). Da mængden af mitokondrie-DNA, der kræves for at forberede en standardkurve, er for høj til at blive opnået med en rimelig mængde væv, må du ikke benytte kopieringsberegninger tilvejebragt af softwaren, men i stedet undersøge krydsningspunkt (Cp) værdier for at bestemme den relative berigelse Af organellar sammenlignet med nukleært DNA i prøverne. Sammenlign disse relative mængder med dem af total genomisk DNA (se repræsentative resultater ). Prøveprimereffektivitet på fem 1:10 fortyndinger af totalt genomisk DNA fra fuldt lysede, to ugers gamle hvedeplantager (repræsentative effektiviteter rapporteret iE legende af figur 2 ).
- QPCR reaktionsopsætning
- Pulsfeltgelelektroforese (PFGE)
BEMÆRK: Denne protokol er baseret på producentens retningslinjer for at udføre PFGE for at løse DNA med høj molekylvægt. Se materialebordet.- Klargøring af gelen og prøverne
- Følg retningslinjerne for gel- og prøveforberedelse og tilpass dem til det tilgængelige system.
- Kør parametrene
- Følg retningslinjerne for opstilling af elektroforesystemet og brug følgende parametre: starttidspunkt for 2 s, slutafbrydertid på 13 s, løbetid på 15 h og 16 min, V / cm på 6 og inkluderet vinkel på 120 ° .
- Farv og billedet gelen
- Farve gelen med et valg af farve ( f.eks. Ethidiumbromid eller et egnet alternativ) og billede med et egnet gel dokumentationssystem.
- Klargøring af gelen og prøverne
- Brug 1 ng DNA som input til DNA Library Prep Kit, efter producentens anvisninger.
- Stregkode og pool prøverne til sekventering i et enkelt løb. Udfør sekventering i henhold til producentens retningslinjer.
BEMÆRK: Pooling og sekventeringsparametre kan ændres afhængigt af arten af interesse, det ønskede dækningsniveau og platformen der bruges til at sekvensere bibliotekerne. For eksempel har en HiSeq-bane væsentligt mere output end en MiSeq-bane, så mange flere prøver kan multiplekseres. Sekvensere en mindre delmængde af prøver for at bestemme, om dækningsniveauerne for organellære genomerne er tilstrækkelige til nedstrømsanalyse.- Undersøg læsekvaliteten ved hjælp af FastQC 31 for at bestemme omfanget af trimning og filtrering, der kræves for dataene.
- Trim og filtrer den rå læser ved hjælp af Trimmomatic 32 eller et andet sammenligneligt program. Brug følgende indstillinger: ILLUMINACLIP 2:30:10 (for at fjerne adaptere), LEADING 3, TRAILING 3, SLIDINGWINDOW 4:10 og MINLEN 100.
- Kortlægge kvaliteten-filtreret og adapter-klippede parret-ende (PE) læser for kinesisk Spring mitokondrie (NCBI referencesekvens NC_007579.1 33), chloroplast (NCBI referencesekvens NC_002762.1 34), og nukleare 35 referencepunkter genomer under anvendelse Bowtie2 36, Med følgende indstillinger: -I 0 -X 800 - følsom.
- Konverter sam alignment filer til bam format (samtools) og sorter bam filerne. Brug bam-filerne til at beregne genoom-dækkende dækning og per-dækning dækning med bedtools. Visualiser resultaterne med R-plot-funktionen.
- Ting at gøre før du starter
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Protokollerne præsenteret i dette manuskript beskriver to forskellige metoder til at berige for organellært DNA fra plantevæv. De her fremlagte betingelser afspejler optimering for hvedevæv. En sammenligning af vigtige trin i protokollerne, obligatorisk vævsindgang og DNA-output er beskrevet i figur 1 . Trinnene i DC-protokollen, som vi testede, følger lignende betingelser som dem, der er beskrevet tidligere ( figur 1A ). Høstet væv skal behandles frisk og udsættes for differentiel centrifugering og / eller gradienter for at isolere intakte organeller. Det nukleare DNA elimineres, før organellerne lyseres, og til sidst ekstraheres DNA'et og anvendes til downstream-applikationer. I modsætning hertil kan plantevæv i MF-protokollen høstes og opbevares før brug, og intakte organeller er ikke påkrævet. I stedet fraktioneres det nukleare og organelle DNA fra total gDNA baseret på DNA-methyleringsstatusen. Begge protokoller giver omtrent lige store mængder organellært DNA ( figur 1C ). Med hensyn til total organellær DNA-output i forhold til vævsindgang er MF-protokollen fordelagtig, når væv er begrænset, da en lille prøve fra en enkelt plante kan anvendes, og planten må lov til at vokse til yderligere analyse. Typisk kræves der i DC-protokoller alt luftvæv fra mange planteplanter, og disse planter kasseres. DC-metoden kan imidlertid optimeres til specifikt at berige for en organeltype over den anden, hvilket ikke er muligt med MF-tilgangen. Det er værd at nævne, at den samlede tid for hver protokol er stort set ækvivalent, selvom der er mindre praktisk tid i MF-tilgangen.
Begge metoder beriger for organellært DNA, omend med forskellige proportioner af mitokondrier og plastidsekvenser:
"> Meget lave mængder renset organellært DNA opnås ved en hvilken som helst metode (i størrelsesordenen 50-100 ng, figur 1C ). For at vurdere niveauerne af organellært genom-berigelse og atomergenomforurening i DNA isoleret fra både DC og MF Metoder, blev der anvendt et qPCR assay. I dette assay blev de relative mængder af tre amplikoner ( dvs. nukleinspecifikke , ACTIN , mitokondrialspecifikke, NAD3 og Chloroplast-specifik, PSBB ) blev vurderet i totalt genomisk DNA, og den organelle DNA-fraktion blev opnået fra begge metoder ( figur 2 ). Kvantifikationscyklus (C q ) -værdier blev undersøgt for hver prøve ( figur 2A ), og fordi C q er defineret som PCR-cyklen, ved hvilken fluorescensen fra målamplificeringen stiger over baggrundsfluorescensniveauet, har C q og målgennemstrømning en Omvendt forhold. IDC-prøven, C q af NAD3 og PSBB er henholdsvis ~ 17 og ~ 15 cykler tidligere end ACTIN (som har en C q på ~ 36) (se figur 2B for C q værdier og berigelsesniveauer). Dette svarer til teoretiske 167.181 og 47.790 gange berigninger for henholdsvis NAD3 og PSBB , I forhold til ACTIN i DC-prøven ( figur 2B , se legenden om figur 2 til beregningen). I den samlede genomiske DNA-prøve er fold-berigelserne for NAD3 og PSBB i forhold til ACTIN kun 158 og 10701. Det er ikke overraskende at finde en højere overflod af de organellære amplikoner i forhold til det nukleare amplicon i det totale genomiske DNA, idet organellærgenerne findes i større kopiantal pr. Celle end det nukleare genom 37 og at antallet af organeller peCelle kan variere afhængigt af vævstypen eller udviklingsstadiet 38 , 39 . Samlet set indikerer dataene, at DC-metoden fortrinsvis beriger mitokondrier, hvilket man kan forvente, da centrifugeringshastigheder optimeres for selektivt at isolere mitochondrier og reducere nuklear og chloroplast "kontaminering".Den ummethylerede fraktion af MF totalt gDNA viser også betydelig berigelse af begge organellære ampliconer og forventes at bibeholde de native relative mængder af disse mål. Foldberigelserne for NAD3 og PSBB i forhold til ACTIN i den ummethylerede fraktion er henholdsvis 20.551 og 1.703.253 ( Figur 2A og 2B ). I den methylerede fraktion er fold-berigelserne for NAD3 og PSBB i forhold til ACTIN henholdsvis 31 og 823 indiCating, at MBD2-Fc-proteinet er yderst effektivt ved nedlukning af methyleret nukleært DNA. Da chloroplast amplicon har en højere overflod end mitokondrie amplicon i total genomisk DNA (~ 6 C q tidligere), methyleret fraktion (~ 5 C q tidligere) og umethyleret fraktion (~ 6 C q tidligere) prøver dette at Oprindelig overflod af disse ampliconer ændres ikke væsentligt ved MDB2-pulldown. Vi fokuserer her på den ikke-methylerede (organellære) fraktion på grund af interesse i at sekvensere disse genomer specifikt. Hvis nukleærgenomet er den primære interesse, vil MF og efterfølgende sekventering af den methylerede fraktion imidlertid give en langt højere nukleær genomdækning end total genomisk DNA-sekventering på grund af reduktionen i organellær DNA "kontaminering".
Det er værd at bemærke, at hvis qPCR ikke er tilgængelig, giver endpoint-PCR (ved anvendelse af de samme primere som for qPCR) den kvalitaTiv vurdering af organellar renhed. I dette tilfælde vil rene organellære DNA-prøver vise amplifikation for mitokondrie- og plastid-ampliconerne, men ingen påviselig amplifikation af det nukleare amplicon på agarosegelen, hvorimod det totale genomiske DNA viser amplifikation for alle tre primer-sæt som vist i tidligere undersøgelser 11 , 12 .
Organellar DNA isoleret fra begge metoder er egnet til NGS:
Trimmet og rengjort PE sekventering læses (se trin 4.3) blev kortlagt til tidligere offentliggjorte hvedeorganellære referencegener, og mængden af læs anvendt til kortlægning af hver prøve varierede fra ~ 800.000 til 1.100.000 læsninger ( Figur 3I ). Resultater fra kortlægning de novo Illumina sekventering læser til tilgængelige hvide chloroplast og mitokondrier genomer er i overensstemmelse med qPCR resUlts, idet DC-metoden giver DNA, der er mere beriget i mitokondrielt DNA ( Figur 3A og 3B , ~ 80% og ~ 10% af henholdsvis kort til mitokondrie (mt) og chloroplast (cp) genomerne) og MF-metoden Hvilket giver DNA, som sandsynligvis afspejler den oprindelige overflod af de to organellære genomer ( fig. 3A og 3B , ~ 20% og ~ 80% af læs kort til henholdsvis mt og cp genomerne). I begge metoder overstiger den teoretiske dækning (se legenden af figur 3 til beregningen) af begge hvedeorganellære genomer over 100X dækning (og varierer op til ~ 2.000X dækning for chloroplastgenomet i den ikke-methylerede fraktion fra MF-metoden), selv Når 12 biblioteker multiplexeres ( figur 3C og 3D ; de 6 biblioteker, der indgår i denne analyse, blev samlet sammen med yderligere 6 biblioteker til en separat analyse for i alt 12 bibliotekerSamlet i en enkelt sekvenseringsbane). Et mere detaljeret billede af dækningen blev opnået ved at undersøge den fraktion af genomet, der er dækket på specifikke dybder, såvel som ved dækningsniveauer i dæk ( Figur 3E -3I ). For MF-metoden var den gennemsnitlige dækningsbaseret dækning ~ 300 - 450X for mt-genomet og 4.000 - 5.000X for cp-genomet. For DC-metoden var den gennemsnitlige dækning pr. Bundt ~ 900 - 1.300 og ~ 500 - 700X for henholdsvis mt og cp genomerne. Der var imidlertid en lille del af både mt- og cp-genomerne, der havde ekstremt lav eller høj dækning, og dette blev set i organellært DNA afledt af en hvilken som helst metode ( figur 3I ). Regioner med højere end gennemsnittet dækning svarer sandsynligvis til regioner med homologi mellem organellar genomerne og regioner med lav dækning kan angive SNP'er eller andre små varianter mellem de sorter, vi sekventerede og de offentliggjorte referencer. Til støtte for denne forestilling, disse piggeAf høj dækning var mest udtalte for mt DNA afledt af MF-metoden ( figur 3E og 3I ), sandsynligvis på grund af den høje dækning af cp-genomet i denne fremgangsmåde. Upletteligt er dækningen af cp-genomet mere ujævn i MF-metoden end DC-metoden ( Figur 3G og 3H ), hvilket kunne skyldes små forstyrrelser i MBD2-Fc-pulldown langs cp-DNA'et. Yderligere eksperimenter vil være nødvendige for at afgøre, hvorfor dette er tilfældet. Uanset hvorledes mt- og cp-genomerne havde relativt lige dækning med begge metoder og ingen store områder med manglende dækning, hvilket kan påvises ved undersøgelsen af den fraktion af genomer, der sekventeres på en given dybde ( figur 3E -3H ). Dækningsniveauerne for begge genomer anses desuden for tilstrækkelige til nedstrømsanalyse, såsom variantanalyse. Hvis det anses for nødvendigt for analysen af sjældne varianter, reducerer numbeR af poolede prøver ville opnå større dækning. Alternativt kan et langt større antal prøver være samlet på en HiSeq-bane, mens der opnås endnu større sekventeringsdybde, omend ved et offer i sekvenslængde, idet HiSeq-biblioteker i øjeblikket er begrænset til PE150-længden i modsætning til PE300 MiSeq-biblioteker.
For at undersøge niveauerne af forurening af nuklear genomet ved hjælp af en kortlægningsmetode blev PE-læsekortkategorier undersøgt. PE-læsninger kan kortlægge til et referencegenomet i forskellige konfigurationer. Når man læser 1 og 2, henvises der til referencen i en head to head mode med en vis "forventet" afstand mellem de to hjælpere (baseret på bibliotekets gennemsnitsindsatsstørrelse og typisk angivet som input parameter i kortlægningssoftwaren ), Disse PE læser siges at kort "concordantly." I modsætning hertil er "uhensigtsmæssig" kortlægning den situation, hvor vennerne kortlægger en mindre eller større end forventet disTance til referencegenomet eller kortet i alternative konfigurationer (fra top til hale eller fra hale til hale). Hvis kun en kompis indstilles til referencegenomet, siges det, at PE lægges til at kortlægge hverken concordantly eller discordantly til referencegenomet. I alle tre læsekortkategorier kan PE-læsninger tilpasse sig referencegenomet en eller flere gange.
For både DC- og MF-isoleret organellært DNA var læsning af kortlægning til mitokondriegenomet hovedsagelig i den justerede sammenhængende engangskategori ( Figur 4A ), mens læsning kortlægges til chloroplastgenomet i relativt lige store mængder af enstemmigt en gang og i overensstemmelse med mere end En gang ( figur 4B ), sandsynligvis på grund af de store inverterede gentagelser, der er til stede i chloroplastgenomet og også til de ekstremt høje dækningsniveauer. Men færre PE læser kortlagt til det nukleare genom og kortlægges stort set mere end en gang i aHverken concordant eller discordant mode ( dvs. kun en kompis er i stand til at kortlægge). Disse er mest sandsynligt at kortlægge "off-target" til sekvenser i det nukleære genom, som er homologe med organellære genomerne eller misassemblede regioner. Kun en mindre mængde læses (<5%) kortlagt til det nukleare genom på samme måde, hvilket indikerer lave niveauer af nukleargenomforurening i organellært DNA isoleret fra DC eller MF-metoden ( figur 4C ), som også afspejles i qPCR-resultaterne ( figur 2A ). Den nukleare fraktion efter MBD2-Fc pulldown fra kinesiske forår ikke-etiolerede væv blev også sekventeret for at bestemme, hvor effektiv pulldown er ved fjernelse af ummethyleret DNA. Mindre end 1% af læser i det nukleare fraktion-afledte bibliotek kortlagt til organellære referencegener, mens ~ 45% af alle læses kortlagt til det nukleære genom ( figur 4 ). Men de fleste læser kortlagt på en uoverensstemmende måde, wHvilket sandsynligvis afspejler de høje niveauer af misassemblering og fragmentering i hvedekernereferencegenomet. Uanset hvad antydes resultaterne, at MBD2-Fc pulldown er meget effektiv ved fjernelse af ummethyleret organellært DNA fra methyleret nukleært DNA. Det er værd at bemærke, at fordi det organellar-berigede DNA, der er resultatet af disse metoder, indeholder en blanding af mitochondrier og chloroplast-sekvenser, og fordi sekvenslikheder, der skyldes gammel genoverførsel mellem disse organeller, forbliver i deres genomer, lægger den korrekte tildeling af den specifikke Genomer skal løses bioinformatisk.
Etiolation af bladvæv ændrer ikke på nogen måde organelle overflod:
Traditionelt foretrækkes etiolerede væv til plantemitokondrisk DNA-isolering for at reducere niveauerne af phenol og stivelse, hvilket kan forstyrre ekstraktionN eller downstream applikationer 13 . For at bestemme, om organellærgenoms berigelsesniveauer kunne ændres eller forbedres ved vækstbetingelser, blev både etiolerede og ikke-etiolerede væv udsat for MF-protokollen og sekventering. Interessant ændrede etiolation ikke signifikant procentdelen af læsninger, der kortlægges til organellærreferencegenomerne ( figur 3A og 3B ) eller dækningsdækningen ( Figur 3I ) sammenlignet med ikke-etiolerede betingelser. Vi isolerede også organellært DNA ved hjælp af differentiel centrifugering med både etiolerede og ikke-etiolerede væv, og der blev fundet en lille forskel i berigelse mellem de forskellige væv ved anvendelse af qPCR (data ikke vist). Dette antyder, at mere fysiologisk relevante ikke-etiolerede væv kan anvendes til organellære sekventeringsundersøgelser uden nogen mærkbar forandring af berigelse.
Kvalitetskontrol foreslår detMF-DNA er mest velegnet til langlæste sekventering:
Da langlæsning af sekvenser bliver mere tilgængelig for forskere, bliver isoleringen af højmolekylært DNA stadig vigtigere. For at vurdere organellært DNA isoleret med enten fremgangsmåde til intakt og kvalitet blev PFGE anvendt. Total genomisk DNA migrerer typisk som et diffust smear i PFGE, og molekylvægten bestemmes af protokollen og hvordan DNA'et blev opbevaret og håndteret efter ekstraktion. Det samlede genomiske DNA isoleret med genomiske tips bør overstige 50 kb, hvilket blev verificeret ved hjælp af PFGE ( Figur 5 , bane 2). Det samlede genomiske DNA fra de genomiske tips bruges som input til Microbiome Enrichment Kit til fraktionering af kernen fra organellær DNA. Den nukleare fraktion opnået efter fraktionering falder i størrelse, men forbliver centreret omkring 50 kb ( figur 5 , bane 4). Dette er ikke suIdet den forholdsvis hårdere håndtering af den nukleare fraktion som eluering fra MBD2-Fc-bundne perler kræver varme- og proteinase K-fordøjelse. På grund af den begrænsede masse blev organellarfraktionen ikke kørt på PFGE, men efterfølgende analyse med TapeStation indikerede DNA> 50 kb (data ikke vist). Det organellære DNA opnået med differentiel centrifugering har en gennemsnitlig masse på ~ 20 kb, sandsynligvis forårsaget af den udvidede organellære isolationsprotokol og den efterfølgende kolonnebaserede DNA-ekstraktion og -koncentration. Gradientbaseret organellær isolering og alternative DNA-ekstraktionsmetoder kan opretholde større DNA-fragmentstørrelser. Uanset hvad, DNA af den størrelse, der opnås i denne protokol, kan anvendes til at generere 10- eller 15-kb-sekventering, læses, hvis der tages omhu under bibliotekets forberedelse.
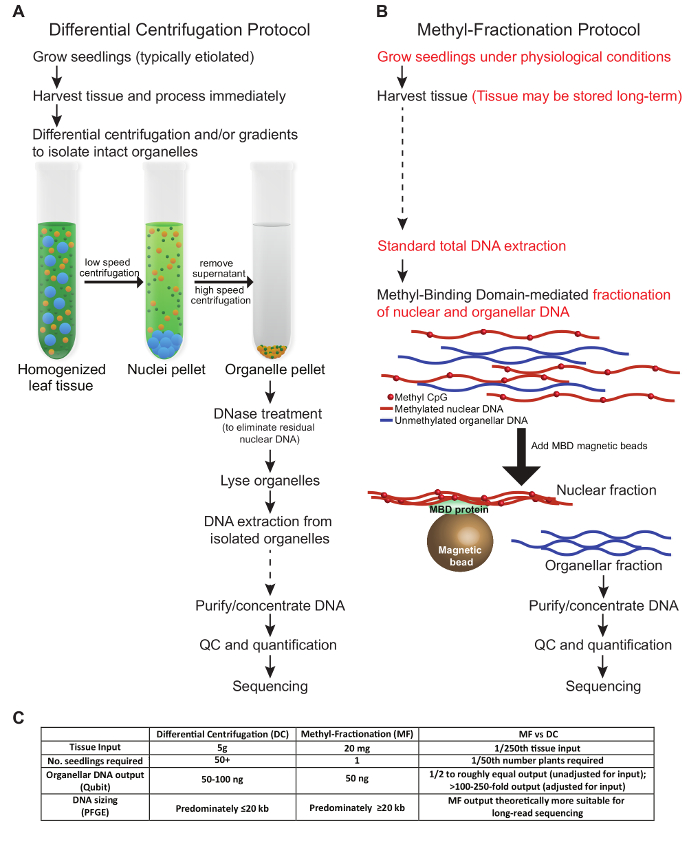
Figur 1: En sammenligningsvisning af Two MethoDs at berige for plantorganellært DNA. En traditionel DC-protokol ( A ) er i modsætning til MF-protokollen ( B ). Det anbefales at undgå frysning og optøning af prøverne. Trin, hvor prøverne kan opbevares på lang sigt, er angivet med stiplede pile ( A og B ). Nøgleforskelle mellem protokollerne fremhæves i rødt ( B ). ( C ) Tabellen sammenligner metoderne i form af vævsindgang, antal planter, der kræves, DNA-udgang og resulterende DNA-størrelse. Klik her for at se en større version af denne figur.

Figur 2: Vurdering af nuklear DNA-kontaminering i organellær DNA isoleret ved anvendelse af to metoder. (
( B ) Tabellen viser C q værdierne, som er vist på grafen i ( A ) og fold berigelsen af de organellære amplikoner i forhold til ACTIN . * Fold berigelse = 2 (Cq ACTIN - Cq Target) . Formlen forudsætter en perfekt effektivitet på 2 for hver primersæt, siden den mindre deviatIon af hver primer fra 2 er ubetydelig og ville have ringe effekt på beregningen og den samlede tendens ( ACTIN = 1.961, NAD3 = 1,95 og PSBB = 1,989). Primereffektiviteten blev evalueret ved at lave en standardkurve med en serie på fem 1:10 fortyndinger af totalt genomisk DNA. Klik her for at se en større version af denne figur.

Figur 3: Læs kortlægning og teoretisk dækning af chloroplast og mitokondrielle genomer. Procentdel af læs kortlagt til mitokondrie ( A ) eller chloroplast ( B ) Kinesiske forårsreferencegener. Tilsvarende teoretisk dækning af den kinesiske forårsmitokondrie ( C ) eller chloroplast ( D ) referencegenoMes, idet der antages at være genomstørrelser på henholdsvis 450 og 135 kb, beregnet ved anvendelse af de samlede læsetal og procentdelen af læsning af kortlægning til de forskellige genomer. Genomfattet distribution af dækning for organellært DNA fra MF-metoden ( E og G ) eller DC-metoden ( F og H ). Dataene i paneler E - H er fra den kinesiske forårs-ætsede prøve, men alle andre prøver viste en lignende tendens. ( I ) Gennemsnitlig, laveste og højeste basisdækning for alle prøver i panelerne A - D. Prøvemærker indbefattende "E" betegner ætiolerede prøver, og "NE" betegner ikke-etiolerede prøver. DC indikerer DNA isoleret med differentieringscentrifugeringsmetoden, og ummethyleret indikerer DNA, som er i den ummethylerede fraktion efter pulldown med MBD2-Fc (MF-protokol). Prøver mærket "Chris" betegner hvede Triticum aestivum'Chris'. CS betegner prøver af hvede Triticum aestivum 'Chinese Spring. Bemærk: På grund af sekvenshomologi mellem kloroplast, mitokondrier og nukleare genomer, der er resultatet af gammel genoverførsel mellem organellargenerne såvel som mellem organellære og nukleare genomer, kan en lille procentdel af rålæser mappe til flere genomer. Derudover læser, der ikke kortlægger til enten organellar referencegenomet, ikke er repræsenteret i denne figur. Således udgør de procentsatser, der vises her ( A og B ), ikke 100%. Klik her for at se en større version af denne figur.

Figur 4: PE Læs kortlægning til hvede kernekernet. Andel af kategorier af PE Læs kortlægningstyper til mitokondrie (A) , chloroplast (B) eller nukleare (C) kinesiske forårshenvisninger. - E betegner ætiolerede prøver og - NE betegner ikke-etiolerede prøver. DC indikerer DNA isoleret med differentieringscentrifugeringsmetoden. Umethyleret indikerer DNA, som er i den ummethylerede fraktion efter pulldown med MBD2-Fc i MF-protokollen, og methyleret betegner nuklearfraktionen efter MBD2-Fc-pulldown. Prøver mærket "Chris" betegner hvede Triticum aestivum 'Chris.' CS betegner prøver af hvede Triticum aestivum 'Chinese Spring.' Umappede læsninger vises ikke. Klik her for at se en større version af denne figur.
Oad / 55528 / 55528fig5.jpg "/>
Figur 5: Undersøgelse af DNA-kvalitet ved brug af PFGE. Hvedetallet genomisk DNA (bane 2), hvedorganellært DNA opnået ved differentiel centrifugering (bane 3) og den nukleare fraktion efter MF med MBD2-Fc pulldown-tilgangen (bane 4) blev udsat for PFGE på en 1% agarosegel med en 1 kb udvidet stige, der bruges som markør (baner 1 og 5). Klik her for at se en større version af denne figur.
| Buffernavn | Opskrift | Noter | Metode |
| STE buffer | 400 mM saccharose, 50 mM Tris pH 7,8, 20 mM EDTA pH 8,0, 0,6% (vægt / volumen) polyvinylpyrrolidon (PVP), 0,2% (vægt / volumen) bovint serumalbumin (BSA), O.1% (vol / vol) p-mercaptoethanol (BME) | Bufferblanding indeholdende kun saccharose, Tris og EDTA kan laves op til en måned i forvejen og holdes ved 4 ° C. PVP, BSA og BME bør tilsættes frisk til en alikvot af den krævede mængde buffer lige før brug. | Metode nr. 1 |
| ST buffer | 400 mM saccharose, 50 mM Tris pH 7,8, 0,6% (vægt / volumen) polyvinylpyrrolidon (PVP), 0,1% (vægt / volumen) bovint serumalbumin (BSA) | Bufferblanding indeholdende kun saccharose og Tris kan laves op til en måned i forvejen og holdes ved 4 ° C. Bemærk, at ST-bufferen ikke indeholder EDTA eller BME, og indeholder en lavere koncentration af BSA. | Metode nr. 1 |
| DNase lager | 2 mg / ml DNase i 0,15 M NaCl til en bestandskoncentration på 2 mg / ml | Opbevar 200 μl alikvoter ved -20 ° C. For at forberede DNase-arbejdsløsning (200 μl DNase-opløsning pr. Prøve) seTabel 1 nedenfor. Se den fulde protokol nedenfor for fuldstændige detaljer om DNase-fordøjelsen. DNase-arbejdsløsningen skal fremstilles frisk. For at standse DNase-reaktionen kræves en 400 mM EDTA pH 8,0 opløsning (slutkoncentration, der er nødvendig for at stoppe reaktionen, er 0,2 M EDTA, se den fulde protokol for detaljer). | Metode nr. 1 |
| DNase arbejdsløsning | 0,25 mg / ml DNase og 20 mM MgCl2 i ST buffer | Forbered frisk, 200 μl pr. Prøve. Viste koncentrationer er til endelig reaktionsvolumen, så blandes: 62,5 pi 2 mg / ml DNase (baseret på endelig 500 pi reaktionsvolumen), 4 pi 1 M MgCI2 (baseret på 200 pi DNase opløsning volumen), og 133,5 pi ST buffer for Et slutvolumen på 200 μl. | Metode nr. 1 |
| Lysis Buffer | 20 mM EDTA pH 8,0; 10 mM Tris pH 7,9; 500 mM guanidin-HCI; 200 mM NaCl; 1% Triton X-100; 0,5 mg / ml lyseringsenzymer fraTrichoderma harzianum | Bland alle ingredienser undtagen lysende enzymer og opbevar ved stuetemperatur. Lysende enzymer skal tilsættes frisk til en lille alikvot til øjeblikkelig anvendelse. | Metode nr. 2 |
Tabel 1: Opskrifter af hjemmelavede buffere og arbejdsstande.
| Koncentrationsark | |||||||
| PRØVNAMN | Tom enhedens vægt (g) | Vægt af den fyldte enhed (g) | Fyldt volumen (ul, fyldt minus tomme vægte) | Vægt efter 1. spin (20 min *, g) | Volumen efter 1. spin (ul, fyldtMinus tomme vægte) | Vægt efter 2. spin (X min *, g) | Volumen efter 2. spin (ul, fyldt minus tomme vægte) |
| Bemærk, at det faktiske genindvindte volumen vil være et par ul mindre end beregnet volumen. | |||||||
Tabel 2: Koncentrationsark.
| Navn | Genomspecificitet | Gen sekvens kilde | Sekvens (5 '- 3') |
| Ta_ACTIN - F | nuklear | Gramene Stillads IWGSC_CSS_1AS_scaff_3272162: 10,663-12,557 | CAGGTATCGCTGACCGTATGA |
| Ta_ACTIN - R | nuklear | Samme som ovenfor | GAAGGTAGGGCTGAACAAGAAAC |
| Ta_NAD3 - F | Mitokondriel | NCBI-tiltrædelse EU534409.1 | GGTGATGCCAGAAGTCGTTT |
| Ta_NAD3 - R | Mitokondriel | Samme som ovenfor | CAGATCAATCTTGTTAGGAGGTACTG |
| Ta_PSBB - F | kloroplast | NCBI tiltrædelse KJ592713.1 | GCTACCTTTGCTTTGCTCTTCT |
| Ta_PSBB - R | kloroplast | Samme som ovenfor | GCTGCCTGTTTCCTTGTAGTT |
Tabel 3: Liste over qPCR Primers.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Til dato er de fleste organellære sekventeringsundersøgelser centreret om traditionelle DC-metoder til at berige for specifikt DNA. Metoder til isolering af organeller fra forskellige planter er blevet beskrevet, herunder mose 40 ; Monocotter såsom hvede 15 og havre 11 ; Og dicots såsom arabidopsis 11 , solsikke 17 og rapsfrø 14 . De fleste protokoller fokuserer på bladvæv 13 , 14 , 15 , 16 , 17 , hvoraf nogle er blevet tilpasset til forskellige vævstyper, herunder frø 11 . Isoleringen af organeller fra protoplaster er også blevet påvist 41 . Dette er imidlertid ikke acceptabelt for alle systemer, og det er heller ikke muligt, når væv af interesse er begrænset. Mange af disse orgaNellære isoleringsmetoder blev designet til at genvinde intakte organeller til specifikke forsøg, såsom fysiologiske undersøgelser. Disse protokoller er besværlige og kræver typisk anvendelse af densitetsgradienter, såsom sucrose eller Percoll-gradienter, som er meget effektive til isolering af specifikke organellære fraktioner, men kræver en stor vævsindgang ( dvs. over 5 g og opad for kilo afhængigt af Vævstypen). DC-metoden kan imidlertid optimeres til at berige for specifikke cellulære fraktioner, såsom mitochondrier eller chloroplast, ved at ændre spinhastigheder og densitetsgradienter. I modsætning hertil kræver MF-tilgangen langt mindre udgangsmateriale (20 mg), men mitokondrie- og plastid-DNA'er vil være til stede pr. Deres relative overflod i vævet anvendt til DNA-ekstraktion. Ikke desto mindre tilbyder MF-protokollen en alternativ tilgang til isolering af blandet organellært DNA og er især gavnligt for at starte med små mængder væv.
T O vurdere prøve renhed efter organelle isolation, de fleste undersøgelser hidtil kun bruge slutpunkt PCR og gel elektroforese 11 , 12 . Dette giver en retfærdig kvalitativ måling af prøve renhed. Imidlertid kan lave amplifikationsniveauer ikke visualiseres på en agarosegel. Få rapporter indeholder mere kvantitative foranstaltninger for kvalitetskontrol, som f.eks. QPCR 14 . For en kvantitativ vurdering af DNA-prøve-renhed isoleret fra begge metoder anvendte vi qPCR og sekventering for at bestemme, hvor meget nukleært DNA forbliver i prøven, såvel som de relative proportioner af mitokondrie versus chloroplast-DNA. Begge metoder, der evalueres her, er effektive til fjernelse af nukleært DNA. Begge metoder giver en blanding af mitochondrial og chloroplast DNA, omend i forskellige proportioner.
Voksende planter i mørket (etiolation) rapporteres at bidrage til at lette organellarisolering på grund af en reduktion af phenolicsRef "> 13. Imidlertid fandt vi i denne sammenligning ikke en mærkbar fordel at arbejde med etioleret væv over lette dyrkede prøver. Selvom andelen af specialiserede chloroplaster sandsynligvis vil være højere, når de er letvoksede, vil det totale plastidtal, som Reflekteret i andelen af læsning af kortlægning til chloroplastgenomet, er uændret under forskellige lysforhold. Derfor anbefales det at udføre genomisk sekventering ved nedstrøms funktionelle analyser, såsom vurdering af heteroplasmien i forskellige væv eller under forskellige stressorer eller ekspressionsanalyser. Planter dyrket under fysiologisk relevante betingelser.
Til anvendelse med kortlæste sekventeringsteknologier giver begge teknikker sammenlignet her tilstrækkelig DNA-mængde og kvalitet. For at opnå lange læsninger på> 20 kb for enkeltmolekylære sekventeringsapplikationer er imidlertid en større mængde DNA af højere kvalitet nødvendig. For eksempel er ideelt,> 1 μg ren orgaNellær hvedeprotein med en molekylvægt> 20 kb er nødvendig for interne, lavindgangsprotokoller til 20 kb insertionsbibliotekspræparater 42 . Nye brugerudviklede lavindgangsprotokoller kan reducere DNA-krav ( dvs. til 50 ng eller endnu mindre 20 ), men udfordringen er fortsat at have højkvalitets DNA med høj molekylvægt, der går ind i bibliotekets præparater. Det er vigtigt, at et flertal af DNA'et er> 20 kb, da mindre fragmenter fortrinsvis indsættes i SMRTbell og smider bibliotekets størrelsesfordeling 43 . Vi forsøgte en række hjemmelavede DNA-ekstraktionsprotokoller og en række kommercielle protokoller til DNA-ekstraktion (ikke vist). For hvedebladevæv blev den bedste balance mellem DNA-kvantitet og kvalitet, især længde, opnået under anvendelse af et kommercielt kit 27 , 29 . Afhængigt af plantearter og væv af interesse, alternatiVe ekstraktionsprotokoller kan være lige egnet eller mere frugtbare. Ikke desto mindre konkluderer vi, at den totale ekstraktion af genomisk DNA med en molekylvægt på> 50 kb, efterfulgt af fraktionering med MBD2-Fc nedadgående tilgang 28 , kan anvendes til langlæsning af sekvensering fra begrænset udgangsmateriale. Fremtidigt arbejde bør afprøve grænserne for udgangsmaterialet, der kræves efter fraktionering til langtidsindstillet biblioteksforberedelse og efterfølgende langlæste sekventering. Kritisk kunne denne fremgangsmåde tilvejebringe en robust metode til at isolere DNA fra en subsample af et enkelt blad, der er egnet til langlæsnings-sekventering uden helgenomdannelse. Vi forventer, at denne tilgang let kan tilpasses til yderligere vævstyper og bredt anvendelig for andre plantearter. Det vil være særligt nyttigt i situationer, hvor vævsmængderne er begrænsende, såsom sekventering ved individuelle generationer i et krydsningsskema eller i sjældnere vævstyper.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Forfatterne erklærer, at de ikke har nogen konkurrerende interesser.
Omtale af handelsnavne eller kommercielle produkter i denne publikation er udelukkende med det formål at give specifikke oplysninger og indebærer ikke anbefaling eller godkendelse fra det amerikanske landbrugsministerium. USDA er en lige muligheder udbyder og arbejdsgiver.
Acknowledgments
Vi vil gerne anerkende finansiering fra USA's Department of Agriculture-Agricultural Research Service og fra National Science Foundation (IOS 1025881 og IOS 1361554). Vi takker R. Caspers for vedligeholdelse af drivhuse og plantepleje. Vi takker også universitetet i Minnesota Genomics Center, hvor Illumina biblioteket forberedelser og sekventering blev udført. Vi er også taknemmelige for kommentarerne fra tidsskriftets redaktører og fire anonyme korrekturlæsere, der yderligere styrker vores manuskript. Vi takker også OECD for et fællesskab til SK at integrere disse protokoller for samarbejdsprojekter med kolleger i Japan.
Materials
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol (beta-mercaptoethanol; BME) | Sigma Aldrich | M3148-100ml | |
| 2-propanol (Isopropyl alcohol/isopropanol), bioreagent | Sigma Aldrich | I9516 | |
| agarose, Bio-Rad Cetified Megabase agarose | Bio-Rad | 1613108 | |
| analytical balance | Mettler Toledo | AB54-S | |
| balance | Mettler Toledo | PB1502-S | |
| bovine serum albumin (BSA) | Sigma Aldrich | B4287-25G | |
| Ceramic grinding cylinders, 3/8in x 7/8in | SPEX SamplePrep | 2183 | |
| Cryogenic Blocks compatible with tissue homogenizer for holding 50 mL tubes | SPEX SamplePrep | 2664 | |
| DNaseI | Sigma | DN25 | |
| ethanol, absolute | Decon Laboratories | 2716 | |
| Ethylenediamine Tetraacetic Acid (EDTA), 0.5 M Solution, pH 8.0 | Fisher | BP2482-500 | |
| gel imaging system | |||
| gel stain | Such as GelRed or Ethidium Bromide | ||
| grinding pestle, wide tip for 2 mL conical tubes | |||
| Guanidine-HCl, 8 M solution | ThermoFisher | 24115 | |
| LightCycler 480 SYBR Green I Master | Roche | 4707516001 | |
| liquid nitrogen | |||
| Lysing enzymes from Trichoderma harzianum | Sigma | L1412 | |
| Magnesium Chloride | G Bioscience | 24115 | |
| magnetic rack | ThermoFisher | A13346 | |
| microcentrifuge tubes, LoBind 1.5 mL | Eppendorf | 22431021 | |
| microcentrifuge tubes, standard nuclease-free 1.5 mL | Eppendorf | ||
| microcentrifuge, refrigerated | Sorvall | Legend X1R | Or equivalent product, must be capable of reaching at least 18,000 x g with rotors for 50 mL tubes, Oak Ridge tubes, and 1.5 mL tubes |
| microcentrifuge, room temperature | Eppendorf | 5424 | Or equivalent product, must be capable of reaching at least 18,000 x g with rotor for 1.5 mL and 2 mL microcentrifuge tubes |
| Microcon DNA Fast Flow Centrifugal Filter Units | EMD Millipore | MRCFOR100 | |
| Miracloth, 1 square per sample cut to fit funnel | EMD Millipore | 475855 | |
| NEBNext Microbiome DNA Enrichment Kit | New England Biolabs | E2612L | |
| parafilm | Parafilm M | PM992 | |
| plastic pots and trays | |||
| polyvinylpyrrolidone (PVP) | Fisher | BP431-100 | |
| Proteinase K | Qiagen | 19131 | |
| Pulsed-Field Gel Electrophoresis rig (e.g. CHEF DR III) | Bio-Rad | 1703697 | |
| purification beads, Agencourt AMpureXP beads | Beckman Coulter | A63881 | |
| QIAamp DNA Mini Kit | Qiagen | 51304 | |
| Qiagen 20/g Genomic Tip DNA Extraction Kit | Qiagen | 10223 | |
| Qiagen Buffer EB (elution buffer) | Qiagen | 19086 | |
| Qiagen DNA Extraction Buffer Set | Qiagen | 19060 | |
| QiaRack | Qiagen | 19015 | |
| qPCR machine (e.g. Roche Light Cycler 480) | Roche | ||
| qPCR plate sealing film | Roche | 4729757001 | |
| qPCR plate, 96 well plate | Roche | 4729692001 | |
| Qubit assay tubes | Life Technologies | Q32856 | |
| Qubit Broad Spectrum assay kit | Life Technologies | Q32850 | |
| Qubit High Sensitivity assay kit | Life Technologies | Q32851 | |
| RNaseA | Qiagen | 19101 | |
| Serological pipettes (20 mL) and pipet-aid | Fisher | 13-678-11 | |
| Small funnels, 1 per sample | |||
| Sodium Chloride | Ambion | AM9759 | |
| Soft paintbrush, 2 per sample | |||
| SPEX SamplePrep 2010 Geno/Grinder or another type of tissue homogenizer | SPEX SamplePrep | Or another comparable tissue homogenizer. If you do not have access to a tissue homogenizer, then grinding in a pre-chilled mortar and pestle will suffice (see protocol for details). However, a homogenizer will give more consistent results and total homogenization time is reduced. | |
| Sucrose | Omnipure | 8550 | |
| TBE | |||
| thermomixer | |||
| Tris | Sigma | T2819-100ml | |
| Triton X-100 | Promega | H5142 | |
| tube rotater | |||
| tubes, 50 mL conical polypropylene | Corning | 352070 | |
| tubes, 50 mL high-speed polypropylene | ThermoScientific/Nalgene | 3119-0050 | e.g. Nalgene Oakridge tubes or equivalent |
| vermiculite | |||
| water bath | |||
| water, sterile and certified Nuclease-free | Fisher | 1481 | |
| water, sterile milliQ |
References
- Liberatore, K. L., Dukowic-Schulze, S., Miller, M. E., Chen, C., Kianian, S. F. The role of mitochondria in plant development and stress tolerance. Free Radic Biol Med. 100, 238-256 (2016).
- Samaniego Castruita, J. A., Zepeda Mendoza, M. L., Barnett, R., Wales, N., Gilbert, M. T. Odintifier--A computational method for identifying insertions of organellar origin from modern and ancient high-throughput sequencing data based on haplotype phasing. BMC Bioinformatics. 16 (232), 1-13 (2015).
- Zhang, T., Zhang, X., Hu, S., Yu, J. An efficient procedure for plant organellar genome assembly, based on whole genome data from the 454 GS FLX sequencing platform. Plant Methods. 7 (38), 1-8 (2011).
- Wambugu, P. W., Brozynska, M., Furtado, A., Waters, D. L., Henry, R. J. Relationships of wild and domesticated rices (Oryza AA genome species) based upon whole chloroplast genome sequences. Sci Rep. 5 (13957), 1-9 (2015).
- Iorizzo, M., et al. De novo assembly of the carrot mitochondrial genome using next generation sequencing of whole genomic DNA provides first evidence of DNA transfer into an angiosperm plastid genome. BMC Plant Biol. 12 (61), 1-17 (2012).
- Park, S., et al. Complete sequences of organelle genomes from the medicinal plant Rhazya stricta (Apocynaceae) and contrasting patterns of mitochondrial genome evolution across asterids. BMC Genomics. 15 (405), 1-18 (2014).
- Skippington, E., Barkman, T. J., Rice, D. W., Palmer, J. D. Miniaturized mitogenome of the parasitic plant Viscum scurruloideum is extremely divergent and dynamic and has lost all nad genes. Proc Natl Acad Sci U S A. 112 (27), E3515-E3524 (2015).
- Wicke, S., Schneeweiss, G. M. Chapter 1. Next Generation Sequencing in Plant Systematics. Hörandl, E., Appelhans, M. , Koeltz Scientific Books. (2015).
- Sloan, D. B. One ring to rule them all? Genome sequencing provides new insights into the 'master circle' model of plant mitochondrial DNA structure. New Phytol. 200 (4), 978-985 (2013).
- Woloszynska, M. Heteroplasmy and stoichiometric complexity of plant mitochondrial genomes--though this be madness, yet there's method in't. J Exp Bot. 61 (3), 657-671 (2010).
- Ahmed, Z., Fu, Y. B. An improved method with a wider applicability to isolate plant mitochondria for mtDNA extraction. Plant Methods. 11 (56), 1-11 (2015).
- Ejaz, M., et al. Comparison of small scale methods for the rapid and efficient extraction of mitochondrial DNA from wheat crop suitable for down-stream processes. Genet Mol Res. 13 (4), 10320-10331 (2014).
- Eubel, H., Heazlewood, J. L., Millar, A. H. Isolation and subfractionation of plant mitochondria for proteomic analysis. Methods Mol Biol. 355, 49-62 (2007).
- Hao, W., Fan, S., Hua, W., Wang, H. Effective extraction and assembly methods for simultaneously obtaining plastid and mitochondrial genomes. PLoS One. 9 (9), e108291 (2014).
- Pomeroy, M. K. Studies on the respiratory properties of mitochondria isolated from developing winter wheat seedlings. Plant Physiol. 53 (4), 653-657 (1974).
- Taylor, N. L., Stroher, E., Millar, A. H. Arabidopsis organelle isolation and characterization. Methods Mol Biol. 1062, 551-572 (2014).
- Triboush, S. O., Danilenko, N. G., Davydenko, O. G. A method for isolation of chloroplast DNA and mitochondrial DNA from Sunflower. Plant Mol Biol Rep. 16 (2), 183-189 (1998).
- Pinard, R., et al. Assessment of whole genome amplification-induced bias through high-throughput, massively parallel whole genome sequencing. BMC Genomics. 7 (216), 1-21 (2006).
- Lamble, S., et al. Improved workflows for high throughput library preparation using the transposome-based Nextera system. BMC Biotechnol. 13 (104), 1-10 (2013).
- Raley, C., et al. Preparation of next-generation DNA sequencing libraries from ultra-low amounts of input DNA: Application to single-molecule, real-time (SMRT) sequencing on the Pacific Biosciences RS II. bioRxiv. , (2014).
- Tsai, Y. C., et al. Resolving the Complexity of Human Skin Metagenomes Using Single-Molecule Sequencing. MBio. 7 (1), e01948 (2016).
- Feehery, G. R., et al. A method for selectively enriching microbial DNA from contaminating vertebrate host DNA. PLoS One. 8 (10), e76096 (2013).
- Yigit, E., Hernandez, D. I., Trujillo, J. T., Dimalanta, E., Bailey, C. D. Genome and metagenome sequencing: Using the human methyl-binding domain to partition genomic DNA derived from plant tissues. Appl Plant Sci. 2 (11), 1-6 (2014).
- Noyszewski, A. K., et al. Accelerated evolution of the mitochondrial genome in an alloplasmic line of durum wheat. BMC Genomics. 15 (67), 1-16 (2014).
- Qiagen. QIAamp DNA Mini and Blood Mini Handbook. , 5th ed, Available from: https://www.qiagen.com/ch/resources/ (2016).
- E.M. Corporation. User Guide: Microcon Centrifugal Filter Devices. , Available from: http://www.emdmillipore.com/US/en/product/Microcon-DNA-Fast-Flow-Centrifugal-Filter-Unit-with-Ultracel-membrane,MM_NF-MRCF0R100 (2013).
- Qiagen. User developed protocol: Isolation of genomic DNA from plants and filamentous fungi using the QIAGEN Genomic-tip - (EN). , Available from: https://www.qiagen.com/ch/resources/ (2001).
- New England BioLabs, Inc.. NEBNext Microbiome DNA Enrichment Kit: Instruction Manual Version 4.0. , Available from: http://www.neb.com/~/media/Catalog/All-Products/371BCB5A557C462D95D1E45E15BBFEA3/Datacards or Manuals/E2612Manual.pdf (2015).
- Qiagen. QIAGEN Genomic DNA Handbook. , Available from: https://www.qiagen.com/ch/resources/ (2012).
- PacificBiosciences. Guidelines for Using the BIO-RAD® CHEF Mapper® XA Pulsed Field Electrophoresis System. , Available from: http://www.pacb.com/wp-content/uploads/Unsupported-Guidelines-Using-BIO-RAD-CHEFMapper-XA-Pulsed-Field-Electrophoresis.pdf (2016).
- Andrews, S. FastQC: A quality control tool for high throughput sequence data. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Ogihara, Y., et al. Structural dynamics of cereal mitochondrial genomes as revealed by complete nucleotide sequencing of the wheat mitochondrial genome. Nucleic Acids Res. 33 (19), 6235-6250 (2005).
- Ogihara, Y., et al. Structural features of a wheat plastome as revealed by complete sequencing of chloroplast DNA. Mol Genet Genomics. 266 (5), 740-746 (2002).
- International Wheat Genome Sequencing Consortium (IWGSC). A chromosome-based draft sequence of the hexaploid bread wheat (Triticum aestivum) genome. Science. 345 (6194), (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nat Methods. 9 (4), 357-359 (2012).
- Bendich, A. J. Why do chloroplasts and mitochondria contain so many copies of their genome? Bioessays. 6 (6), 279-282 (1987).
- Kumar, R. A., Oldenburg, D. J., Bendich, A. J. Changes in DNA damage, molecular integrity, and copy number for plastid DNA and mitochondrial DNA during maize development. J Exp Bot. 65 (22), 6425-6439 (2014).
- Ma, J., Li, X. Q. Organellar genome copy number variation and integrity during moderate maturation of roots and leaves of maize seedlings. Curr Genet. 61 (4), 591-600 (2015).
- Lang, E. G., et al. Simultaneous isolation of pure and intact chloroplasts and mitochondria from moss as the basis for sub-cellular proteomics. Plant Cell Rep. 30 (2), 205-215 (2011).
- Tobin, A. K. Subcellular fractionation of plant tissues. Isolation of chloroplasts and mitochondria from leaves. Methods Mol Biol. 59, 57-68 (1996).
- PacificBiosciences. Procedure & Checklist - 10 kb to 20 kb Template Preparation and Sequencing with Low (100 ng) Input DNA. , Available from: http://www.pacb.com/wp-content/uploads/Procedure-Checklist-10-20kb-Template-Preparation-and-Sequencing-with-Low-Input-DNA.pdf (2015).
- PacificBiosciences. Template Preparation and Sequencing Guide. , Available from: http://www.pacb.com/wp-content/uploads/2015/09/Guide-Pacific-Biosciences-Template-Preparation-and-Sequencing.pdf (2014).



