Summary
दो संयंत्र ऑर्गेनेल डीएनए संवर्धन के तरीकों की तुलना और अनुकूलन प्रस्तुत किए गए हैं: मेथिलिकेशन स्थिति पर आधारित कुल जीडीएनए के पारम्परिक अंतर सेंट्रीफ्यूजेशन और विभाजन। हम परिणामस्वरूप डीएनए मात्रा और गुणवत्ता का आकलन करते हैं, अगली पीढ़ी के शॉर्ट-वाचन में प्रदर्शन को प्रदर्शित करते हैं, और लंबे-पढ़े गए एकल-अणु अनुक्रमणों में उपयोग की संभावनाओं पर चर्चा करते हैं।
Abstract
प्लांट ऑ organellar जीनोम में बड़े, दोहराव वाले तत्व होते हैं जो जटिल संरचनाओं और / या उप-जीनोमिक टुकड़े बनाने के लिए युग्मन या पुन: संयोजन से गुजर सकते हैं। ऑर्गेलेर जीनोम भी किसी दिए गए सेल या टिशू प्रकार (हेटेरोप्लासी) के अंदर प्रवेश में मौजूद होते हैं, और उपप्रकार के बहुतायत में विकास के दौरान या तनाव के दौरान बदल सकता है (उप-स्टेइकीओमेट्रिक स्थानांतरण)। अगली पीढ़ी के अनुक्रमण (एनजीएस) प्रौद्योगिकियों को ऑर्गेनेल जेनोम संरचना और कार्य की गहरी समझ प्राप्त करने के लिए आवश्यक है। ऑर्गेनेल डीएनए प्राप्त करने के लिए पारंपरिक अनुक्रमण के कई तरीकों का इस्तेमाल किया जाता है: (1) यदि ऊतक को शुरू करने की एक बड़ी मात्रा में उपयोग किया जाता है, तो उसे समरूपित किया जाता है और विभेदक केन्द्रोत्पादन और / या ढाल शुद्धि के अधीन होता है। (2) यदि ऊतक की एक छोटी मात्रा का प्रयोग किया जाता है ( यानी, अगर बीज, सामग्री या स्थान सीमित है), तो उसी प्रक्रिया को (1) के रूप में किया जाता है, इसके बाद पूरे जीनोम प्रवर्धन के लिए पर्याप्त डीएनए प्राप्त होता है। (3) जैव सूचना विज्ञान का विश्लेषण सीईसी के लिए किया जा सकता हैकुल जीनोमिक डीएनए का उपयोग करें और ऑर्गेनेल ने पढ़ा। इन सभी विधियों में निहित चुनौतियों और व्यापारिक अवसर हैं (1) में, ऊतक को शुरू करने की इतनी बड़ी मात्रा में प्राप्त करना मुश्किल हो सकता है; (2) में, पूरे जीनोम प्रवर्धन एक अनुक्रमण पूर्वाग्रह परिचय कर सकता है; और (3) में, परमाणु और ऑर्गेनेल जेनोमों के बीच समरूपता विधानसभा और विश्लेषण के साथ हस्तक्षेप कर सकती है। बड़े परमाणु जीनोमों वाले पौधों में ऑक्सीनेल डीएनए के लिए जैव सूचना विज्ञान के विश्लेषण के लिए अनुक्रमण लागत और अनुक्रम जटिलता को कम करने के लिए यह लाभप्रद है। यहां, हम एक चौथाई विधि, एक अनुकूल सीपीजी-मिथाइल पुलडाउन दृष्टिकोण के साथ एक पारंपरिक अंतर सेंटीफ्यूगेशन विधि की तुलना करते हैं, जो कि परमाणु और ऑर्गेनेल अंशों में कुल जीनोमिक डीएनए को अलग करता है। दोनों तरीकों से एनजीएस, डीएनए के लिए पर्याप्त डीएनए प्राप्त होता है जो ऑ organellar अनुक्रमों के लिए अत्यधिक समृद्ध होता है, यद्यपि मिटोचंद्रिया और क्लोरोप्लास्ट्स में विभिन्न अनुपात पर होता है। हम गेहूं के पत्तों के ऊतकों के लिए इन विधियों के अनुकूलन को पेश करते हैं और प्रमुख फायदे और डी पर चर्चा करते हैंनमूना इनपुट, प्रोटोकॉल आराम और डाउनस्ट्रीम एप्लीकेशन के संदर्भ में प्रत्येक दृष्टिकोण के फायदे हैं
Introduction
जीनोम अनुक्रमण महत्वपूर्ण पौधे लक्षणों के अंतर्निहित आनुवंशिक आधार काटना करने के लिए एक शक्तिशाली उपकरण है। अधिकांश जीनोम-अनुक्रमिक अध्ययन परमाणु जीनोम सामग्री पर ध्यान केंद्रित करते हैं, क्योंकि अधिकांश जीन नाभिक में स्थित हैं हालांकि, ऑटोनॉल्टर जीनोम (यूकेरियोट्स में) और प्लास्टिड (पौधों में, विशेष रूप से, क्लोरोप्लास्ट, प्रकाश संश्लेषण में काम करता है), जीव-जंतु विकास, तनाव प्रतिक्रिया और संपूर्ण फिटनेस 1 के लिए महत्वपूर्ण आनुवांशिक जानकारी का योगदान देता है। ऑर्गेलेर जीनोम आम तौर पर परमाणु जीनोम अनुक्रमण के लिए आवश्यक कुल डीएनए निष्कर्षों में शामिल हैं, हालांकि डीएनए निष्कर्षण से पहले ईएनजीई संख्या को कम करने के तरीकों को भी नियोजित किया जाता है 2 । कई अध्ययनों ने ऑर्गेनेल जेनोम्स 3 , 4 , 5 को इकट्ठा करने के लिए कुल जीडीएनए निकासी से अनुक्रमण परिणामों का उपयोग किया है ,Xref "> 6 , 7। हालांकि, जब ऑब्जेनेल जीनोम पर ध्यान केंद्रित करने के लिए अध्ययन का लक्ष्य है, कुल जीडीएनए का उपयोग अनुक्रमण लागत को बढ़ाता है क्योंकि कई पढ़ता है परमाणु डीएनए अनुक्रमों को" खो दिया ", विशेषकर बड़े परमाणु जीनोमों वाले पौधों में इसके अलावा, परमाणु जीनोम में और ऑर्गेनल्स के बीच ऑगनेरल अनुक्रमों के दोहराव और हस्तांतरण के कारण, उचित जीनोम को पढ़ते हुए अनुक्रमण की सही मैपिंग स्थिति को हल करना जैव-रूप से 2 , 8 को चुनौती देने वाला है। परमाणु जीनोम से ऑर्गेनेल जेनोम का शुद्धि एक है इन समस्याओं को कम करने की रणनीति। आगे की जैव सूचना विज्ञान रणनीतियों का उपयोग मितोचोन्रिया और क्लोरोप्लास्ट्स के बीच समरूपता के क्षेत्रों को उस नक्शे को पढ़ा जाने के लिए अलग किया जा सकता है।
जबकि कई पौधों की प्रजातियों के ऑ organellar जीनोम क्रमबद्ध हुए हैं, जबकि ऑ organellar जीनोम विविधता की चौड़ाई के बारे में बहुत कुछ पता हैजंगली आबादी में या खेती के प्रजनन पूल में उपलब्ध है। ऑर्गेनेर जीनोम को गतिशील अणुओं के रूप में भी जाना जाता है जो दोहराए गए अनुक्रम 9 के बीच पुनर्संयोजन के कारण महत्वपूर्ण संरचनात्मक पुनर्व्यवस्था से गुजरते हैं। इसके अलावा, ऑ organeller जीनोम की कई प्रतियां प्रत्येक अंग में समाहित होती हैं, और प्रत्येक कक्ष में कई ऑर्गेनल्स समाहित होते हैं। इन जीनोमों की सभी प्रतियां एक जैसी नहीं हैं, जिसे हेटेरोप्लासिमी के रूप में जाना जाता है। "मास्टर सर्कल्स" की कैनोनिकल तस्वीर के विपरीत, उप-जीनोमिक सर्किल, रैखिक क्रोमोसोम, रैखिक कॉंक्वैमर, और ब्रंकेड स्ट्रक्चर 10 सहित ऑ organeller जीनोम संरचनाओं की अधिक जटिल तस्वीर के लिए अब बढ़ती सबूत हैं। वनस्पति organellar जीनोमों की विधानसभा उनके अपेक्षाकृत बड़े आकारों और पर्याप्त उल्टे और प्रत्यक्ष दोहराव से जटिल है।
ऑर्गेनेल अलगाव, डीएनए शुद्धि और बाद में जेनोम के लिए पारंपरिक प्रोटोकॉल ई अनुक्रमण अक्सर बोझिल होते हैं और टिशू इनपुट के बड़े संस्करणों की आवश्यकता होती है, कई ग्रामों के साथ, प्रारंभिक बिंदु 11 , 12 , 13 , 14 , 15 , 16 , 17 के रूप में आवश्यक युवा पत्ती के ऊतक के सैकड़ों ग्रामों के ऊपर। जब यह ऊतक सीमित होता है तो यह ऑगनलार जीनोम अनुक्रमण को दुर्गम बनाता है। कुछ स्थितियों में, बीज की मात्रा सीमित होती है, जैसे कि जब पीढ़ी के आधार पर या नर बाँझ लाइनों में क्रम अनुक्रम के लिए जरूरी हो, जिसे क्रॉसिंग के जरिए बनाए रखा जाना चाहिए। इन स्थितियों में, ऑ organellar डीएनए को शुद्ध किया जा सकता है और तब पूरे जीनोम प्रवर्धन के अधीन होता है। हालांकि, पूरे जीनोम प्रवर्धन महत्वपूर्ण अनुक्रमण पक्षपात को लागू कर सकता है, जो कि संरचनात्मक विविधता, उप-जीनोमिक संरचनाओं, और हेटेरोप्लासी स्तरों का मूल्यांकन करते समय एक विशेष समस्या है> 18 लघु-पढ़ने वाली अनुक्रमण तकनीक के लिए पुस्तकालय की तैयारी में हालिया प्रगति पूरे जीनोम प्रवर्धन से बचने के लिए कम-इनपुट बाधाओं को दूर करती है। उदाहरण के लिए, इल्यूमिना नेक्टेरा एक्सटी लाइब्रेरी तैयारी किट 1 एनजी डीएनए के रूप में इनपुट 1 के रूप में इस्तेमाल होने की अनुमति देता है। हालांकि, लंबे समय से पढ़े जाने वाले अनुक्रमिक अनुप्रयोगों जैसे पीएसीबीओ या ऑक्सफोर्ड नैनोपोर अनुक्रमण तकनीकों के लिए मानक पुस्तकालय की तैयारी को अभी भी इनपुट डीएनए की अपेक्षाकृत उच्च मात्रा की आवश्यकता होती है, जो ऑ organellar जीनोम अनुक्रमण के लिए एक चुनौती रख सकता है। हाल ही में, नए यूजर-निर्मित, लंबे समय से पढ़े हुए अनुक्रमण प्रोटोकॉल को इनपुट राशियों को कम करने और नमूनों में जीनोम अनुक्रमण की सुविधा के लिए विकसित किया गया है, जहां डीएनए की माइक्रोग्राम मात्रा 20 , 21 मुश्किल है। हालांकि, उच्च-आणविक भार प्राप्त करना, इन पुस्तकालयों की तैयारी में फ़ीड करने के लिए शुद्ध ऑर्गेनेल अंशों को एक चुनौती बनी हुई है।
हमने टी की मांग कीO पूरे जीनोम प्रवर्धन की आवश्यकता के बिना एनजीएस के लिए उपयुक्त organellar डीएनए संवर्धन और अलगाव विधियों की तुलना और अनुकूलन। विशेष रूप से, हमारा लक्ष्य सीमित प्रारंभिक सामग्रियों से उच्च-आणविक वजन ऑनेलवेल डीएनए को समृद्ध करने के लिए सर्वोत्तम तरीकों का निर्धारण करना था, जैसे कि पत्ते का सब्सम्। यह काम ऑर्गेनेल डीएनए के लिए समृद्ध करने के तरीकों का तुलनात्मक विश्लेषण प्रस्तुत करता है: (1) एक संशोधित, पारंपरिक विभेदक सेंट्रिफ्यूजेशन प्रोटोकॉल बनाम (2) एक डीएनए फ्रैक्शन प्रोटोकॉल, जिस पर व्यावसायिक रूप से उपलब्ध डीएनए सीपीजी-मिथाइल बाध्यकारी डोमेन प्रोटीन पुलडाउन दृष्टिकोण 22 संयंत्र ऊतक 23 पर लागू हम गेहूं के पत्तों के ऊतक से organellar डीएनए के अलगाव के लिए सर्वोत्तम प्रथाओं की सलाह देते हैं, जिसे आसानी से अन्य पौधों और ऊतक प्रकारों तक बढ़ाया जा सकता है।
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. ऑगेलर अलगाव और डीएनए निष्कर्षण के लिए संयंत्र सामग्री का उत्पादन
- गेहूं के पौधों की मानक वृद्धि
- वर्मीक्यूलाईट में छोटे, चौड़े बर्तनों में 4-6 बीज प्रति कोने में बीज लगाते हैं। एक 16 घंटे प्रकाश चक्र, 23 डिग्री सेल्सियस / 18 डिग्री सी रात के साथ एक ग्रीनहाउस या विकास चैंबर में स्थानांतरण
- प्रत्येक दिन पौधों को पानी दें कणों के ¼ चम्मच के साथ पौधों को उर्वरक 20-20-20 एनपीके उर्वरक अंकुरण पर और 7 दिनों के बाद अंकुरण पर।
- गेहूं के पौधों की वैकल्पिक भंग
- चरण 1.1 का पालन करें, लेकिन 8 घंटे के लिए 16 घंटे / 18 डिग्री सेल्सियस के लिए 23 डिग्री सेल्सियस गहरा विकास कक्ष में बर्तन रखें। वैकल्पिक रूप से, ग्रीन हाउस में पौधों को कवर करें ( जैसे, भंडारण कंटेनर के साथ, हालांकि, उचित वेंटिलेशन बनाए रखा जाना चाहिए)।
- ग्रोथ एंड टिशू कलेक्शन
- 12-14 दिनों के लिए पौधों को बढ़ाना। अधिकांश गेहूं जीनोटाइप के लिएएस, 75-100 पौधों के ऊतक के लगभग 10-12 ग्राम पैदा होते हैं, जो कि अंतर सेंटीफ्यूगेशन विधि (खंड 2) का उपयोग करने वाले दो ऑर्गेनेल निष्कर्षों के लिए पर्याप्त है; डीएनए सीपीजी-मेथिलैशन-आधारित पुलडाउन दृष्टिकोण का उपयोग विभेदित ऑनेलवेल पर परमाणु डीएनए (खंड 3) से करने के लिए केवल एक संयंत्र आवश्यक है।
- यदि विभेदक केन्द्रोत्सव दृष्टिकोण का उपयोग करते हुए, टिशू ताजा इकट्ठा करें और नमूने को संसाधित करने के लिए तत्काल आगे बढ़ें, जैसा कि अनुभाग 2 में वर्णित है।
- सीपीजी-मिथाइल पुलडाउन दृष्टिकोण का उपयोग करते हुए, सूक्ष्मदर्शीय ट्यूबों में युवा पत्ती के ऊतक के 20 मिलीग्राम वर्गों का फसल (मानक-उगने वाले या ऊतकयुक्त ऊतक का उपयोग करें, प्रतिनिधि परिणाम देखें)। तरल नाइट्रोजन पर स्नैप-फ्रीज और -80 डिग्री सेल्सियस पर फ्रीज तक उपयोग न करें। खंड 3 में वर्णित के अनुसार, डीएनए के पुलडाउन विभाजन को आगे बढ़ें।
2. विधि # 1: डीएनए निष्कर्षण विभेदक केन्द्रापसारक (डीसी) का उपयोग करना
नोट: अंतरईंटेंटियल सेंट्रीफ्यूजेशन प्रोटोकॉल दो प्रकाशनों से संशोधित किया गया था, जो अनुकूलन स्थितियों को दोनों अंगों को अलग करने के लिए , लेकिन मितोचोनड्रिया 17 , 24 के लिए समृद्ध। परिणामस्वरूप प्रोटोकॉल समय-गहन होता है और पिछले तरीकों से कम विषाक्त रसायनों का उपयोग करता है। विशेष रूप से, हमने बफ़र्स में संशोधन किया और स्टीव निष्कर्षण बफर में पॉलीविनालिप्रोलीरोइडोन (पीवीपी) के अतिरिक्त और एनईटीएफ बफर में अंतिम धोने के चरण को समाप्त करने सहित कदम धोने थे, जिसमें सोडियम फ्लोराइड (NaF) शामिल है।
सावधानी: एसईई बफर की तैयारी और उपयोग उचित व्यक्तिगत सुरक्षा उपकरणों के साथ रासायनिक धूआं हुड के तहत किया जाना चाहिए, क्योंकि इस बफर में 2-मर्कैपटोथैनॉल (बीएमई) शामिल हैं।
- शुरू करने से पहले करने के लिए चीजें
- सुनिश्चित करें कि सभी उपकरण बेहद साफ होते हैं, और किसी भी उपकरण को आटोक्लेव कर सकते हैं जिसे आटोक्लेव किया जा सकता है ( जैसे, पीस सिलेंडर, हाई-स्पीड सेंटीFuge ट्यूब, आदि )
नोट: क्रॉस-संदूषण से बचने के लिए pipetting की आवश्यकता के सभी चरणों के लिए फ़िल्टर-टिप्स की अनुशंसा की जाती है। - आवश्यक उपकरण और अभिकर्मकों की सूची देखें और विधि # 1 ( तालिका 1 ) के लिए आवश्यक बफ़र्स और कार्यशील स्टॉक तैयार करें। -20 डिग्री सेल्सियस और रोटार और बफ़र्स को 4 डिग्री सेल्सियस के लिए क्रायोजेनिक पीस ब्लॉकों को ठंडा करें, माइक्रोसिंत्रिफ़्यूज को 4 डिग्री सेल्सियस पर सेट करें और 37 डिग्री सेल्सियस पानी के नहाने को चालू करें।
- सुनिश्चित करें कि सभी उपकरण बेहद साफ होते हैं, और किसी भी उपकरण को आटोक्लेव कर सकते हैं जिसे आटोक्लेव किया जा सकता है ( जैसे, पीस सिलेंडर, हाई-स्पीड सेंटीFuge ट्यूब, आदि )
- ऑर्गेनल्स का अलगाव
- फसल ताजा ऊतक के 5 ग्राम और बर्फ पर एक ठंडा बीकर में ठंड, बाँझ पानी में कुल्ला।
नोट: सेंट्रिफ्यूज, धूआं हुड, आदि के लिए और सभी कार्यों और परिवहन के दौरान हमेशा नमूने को बर्फ में रखें । वैकल्पिक रूप से, एक ठंडे कमरे में काम करें, अगर प्रोटोकॉल को पूरा करने के लिए पर्याप्त स्थान और उपकरण तक पहुंच हो। - कैंची का उपयोग, पत्ती के ऊतक को ~ 1 सेमी सेमी में सीधे 50 एमएल ट्यूब में दो सिरेमिक पीस युक्त युक्त करता हैसिलेंडर।
नोट: क्रॉस-संदूषण से बचने के लिए नमूने के बीच कैंची को साफ या बदलें। - यदि कोई ऊतक होमोजीज़रेटर नहीं है, तो मोर्टार और मूसल का उपयोग करें और चरण 2.2.4 - 2.2.9 को बदलने के लिए अनुसरण करें।
- बर्फ पर एक पूर्व ठंडा मोर्टार में पत्ती के ऊतकों को काट लें। नमूनों को 2 से 3 मिनट के लिए 15 एमएल एसईई (पीस हुड) में पीस लें।
- पूर्व-गीले, बाँझ छानने का कपड़ा (~ 22 से 25 माइक्रोन आकार का आकार, एक अन्य 50 एमएल ट्यूब में मुख्य प्रोटोकॉल देखें) की एक परत वाले फ़नल के माध्यम से बफर बंद करें (मोर्टार में ऊतक को छोड़ दें) । मोर्टार और मूसल के लिए अतिरिक्त 10 मिलीलीटर एसटीई जोड़ें और फिर से होमोजिनायज करें।
- एक ही फ़नल में homogenized ऊतक और बफर डालो। 10 एमएल एसईई के साथ मोर्टार और मूसल को कुल्ला और इसे फ़नल में डाल दें। जितना संभव हो उतना तरल पुनर्प्राप्त करने के लिए फ़नल में छानने का कपड़ा निचोड़ कर डालना।
नोट: पार-संदूषण से बचने के लिए नमूनों के बीच दस्ताने बदलें। प्रो के साथ जारी रखेंचरण 2.2.10 पर टोल करें
- प्रत्येक 50 एमएल ट्यूब में 20 एमएल की एसटीई (धूआं हुड में) जोड़ें।
- नमूनों को पूर्व-ठंडा क्रायोजेनिक पीसने वाले ब्लॉकों में एक ऊतक पीसने वाले यंत्र में रखें और 2 x 30 एस के 1,750 आरपीएम के नमूनों को पीस दें। नमूना स्थितियों को घुमाएं और नमक के टुकड़े को बर्फ के बीच ~ 1 मिनट के लिए ग्राइंड्स में रखें।
नोट: इस चरण में एक मोर्टार और पीले, ब्लेंडर, या अन्य ऊतक पीस / होमोजीनिंग डिवाइस का उपयोग किया जा सकता है। हालांकि, प्रत्येक विधि परिणामस्वरूप डीएनए गुणवत्ता को विभिन्न डिग्री पर प्रभावित करेगा, और इसलिए डीएनए लंबाई और गुणवत्ता का मूल्यांकन डाउनस्ट्रीम अनुप्रयोगों के साथ जारी रखने से पहले किया जाना चाहिए। - बर्फ में रखी एक साफ 50-एमएल ट्यूब में एक फ़नल डालें। फ़नल में एक छानने का कपड़ा की एक परत रखें और 5 एमएल एसईई के साथ इसे पूर्व में गीला रखें। प्रवाह के माध्यम से मत छोड़ो
- होमनलिज्ड टिशू को फ़नल में डालें। 15 एमएल के एसटीई के साथ पीसने वाली ट्यूब कुल्ला, रीकैप और ट्यूबों को दीवारों और ढक्कन कुल्ला करने के लिए पलटना, और म्यान में डालनाएल।
- सावधानी से सिरेमिक पत्थरों को हटा दें और फिर निस्पंदन और छानने का कपड़ा फनल में दबाएं।
नोट: पार-संदूषण से बचने के लिए नमूनों के बीच दस्ताने बदलें। - Spillage से बचने के लिए parafilm के साथ ट्यूब कैप लपेटें। 4 डिग्री सेल्सियस पर 10 मिनट के लिए 2,000 xg पर अपकेंद्रित्र
- सीरोलॉजिकल पिपेट का उपयोग करके सतह पर तैरने वाले को सावधानीपूर्वक महाप्राणित करें और इसे 50 एमएल हाई-स्पीड अपकेंद्रित्र ट्यूब में रखें (यदि ट्यूबों में तंग मोहरी नहीं है, तो स्पिलेज से बचने के लिए पैराफिल के साथ ट्यूब टोपी लपेटें) छर्रों को त्यागें।
- एसटीई का उपयोग करके 0.1 ग्राम के अंदर ट्यूबों को बाधित करें और परिणामस्वरूप सतह पर तैरनेवाला को 18,000 XG और 4 डिग्री सी पर 20 मिनट के लिए अपकेंद्रित करें। ट्यूबों को संतुलित करने के लिए, संतुलन पर बर्फ का एक छोटा बीकर रखें, बड़े पैमाने पर तारे, और बर्फ पर नमूनों का वजन उन्हें ठंडा रखने के लिए करें। वैकल्पिक रूप से, एक ठंडे कमरे में संतुलन और धूआं का उपयोग करें।
- सतह पर तैरनेवाला त्यागें गोली के लिए एसटी के 1 एमएल जोड़ें और धीरे से हमें निलंबित करेंनरम तटरक्षक। 24 एमएल एसटी (25 मिलीलीटर की अंतिम मात्रा) और मिश्रण / झुंड ( यानी, सभी तरल पदार्थ को हटाने के लिए ट्यूब के किनारे पर पेंटब्रश दबाएं) जोड़ें।
- अनुसूचित जनजाति का उपयोग करके 0.1 ग्राम के अंदर ट्यूबों को शेष रखें 18 मिनट और 18 डिग्री सेल्सियस पर 20 मिनट के लिए अपकेंद्रित्र इस बीच, DNaseI समाधान तैयार करें (स्टॉक के लिए तालिका 1 और कार्य समाधान व्यंजनों को देखें)। प्रत्येक नमूने के लिए, 1.5 एमएल ट्यूब में 200 μL विभाज्य बनाओ।
- सतह पर तैरनेवाला को त्याग दें, ट्यूब को ब्लोट करें और नरम तूलिका का उपयोग करके 300 μL ST में गोली को फिर से निलंबित करें (फिर भी उच्च गति वाले अपकेंद्रित्र ट्यूब में)। पेटीब्रश को पहले तैयार 1.5 मिलीलीटर ट्यूब में 200 μL DNaseI समाधान युक्त रखें और ब्रश में फंसे किसी भी अवशिष्ट गोली को हटाने के लिए पेंटब्रश को घुमाएं। डीएनसीआई समाधान को उच्च गति वाले अपकेंद्रित्र ट्यूब में पिपेट करें और धीरे-धीरे मिश्रण को मिलाएं।
- 37 डिग्री सेल्सियस पर पानी के स्नान में 30 मिनट के लिए सेते हैं (ट्यूब के शीर्ष के आसपास लपेटो पैराफिलम को संक्षेपण लेकिनजी को टोपी में) ऊष्मायन के दौरान धीरे-धीरे घूमते हुए 2 बार मिलाएं।
- धीरे से गोली मिश्रण को एक विस्तृत छिद्र के साथ एक विंदुक टिप का उपयोग करके ट्यूब से बाहर निकालना और उसे 1.5 एमएल की कम-बायीं ट्यूब में रखें। 500 एमएल का 400 एमएम EDTA, पीएच 8.0, उच्च गति के अपकेंद्रित्र ट्यूब में जोड़ें और ट्यूब के सभी अवशिष्ट गोली निकालने के लिए धीरे से पिपेट करें। EDTA को उसी 1.5-एमएल, कम बाइंड ट्यूब को गोली मिश्रण के रूप में स्थानांतरित करें और उलटाव से धीरे-धीरे मिश्रण करें।
- 4 डिग्री सेल्सियस पर 20 मिनट के लिए 18,000 xg पर अपकेंद्रित्र सतह पर तैरनेवाला त्यागें, ट्यूब को ब्लोट करें, और एक बार डीएनए अलगाव के लिए उपयोग करें यदि आवश्यक हो, -20 डिग्री सेल्सियस पर फ्रीज छल्ले, लेकिन इसका परिणाम उपज में कमी हो सकता है, क्योंकि डीएनसीआई अवशिष्ट डीएनएआई को तत्काल संसाधित नहीं किया जा सकता है।
- फसल ताजा ऊतक के 5 ग्राम और बर्फ पर एक ठंडा बीकर में ठंड, बाँझ पानी में कुल्ला।
- एक व्यावसायिक कॉलम-आधारित दृष्टिकोण का उपयोग करने वाले अलग-अलग संगठनों से डीएनए निष्कर्षण
नोट: पूर्ण प्रोटोकॉल 25 के लिए किट पुस्तिका देखें, और संशोधनों के लिए नीचे देखें। पीआरडीएनए निष्कर्षण के लिए ऑर्गेनेल अलगाव से सीधे आगे बढ़ना पसंद है। दोहराया गया ठंड और विगलन डीएनए के टुकड़े के आकार को कम करेगा और अवशिष्ट डीएनसीआईआई द्वारा डीएनए गिरावट की ओर ले जाएगा। ऊर्ध्वाधर या जोरदार pipetting सीमा, के रूप में यह डीएनए कर सकते हैं कतरनी। डीएनए वसूली को अधिकतम करने के लिए कम-बाइंड माइक्रोसेंट्रिफ्ज ट्यूबों का उपयोग करने की सलाह दी जाती है।- डीएनए निष्कर्षण प्रक्रिया
नोट: विस्तृत वाणिज्यिक प्रोटोकॉल 25 को यह सुनिश्चित करने के लिए शुरू करें कि बफ़र्स ठीक से बनाए गए / संग्रहीत किए गए हैं और स्पिन-स्तंभ प्रक्रियाओं को समझ लिया गया है।- गोली के साथ 180 μL बफर एटीएल सीधे ट्यूब में जोड़ें (बैक्टीटॉप पर कमरे के तापमान पर पहले से जमी और थका हुआ)।
- निम्नलिखित संशोधनों के साथ, किट पुस्तिका में "टिशू से डीएनए शोधन" के प्रोटोकॉल में चरण 3 के साथ आगे बढ़ें: चरण 3 में एक 30 मिनट की विधि, वैकल्पिक RNase A पाचन शामिल है, और 3 x 200 μL में एई (एई) प्रत्येक में से एकपाराट ट्यूब और फिर एल्यूलेशन को जोड़ते हैं)।
- QPCR के लिए एक विभाज्य (कम से कम 20 μL) सहेजें (चरण 4.1 देखें) ध्यान केंद्रित करने से पहले मात्रा निर्धारित करने के लिए, उच्च संवेदनशीलता मात्रा का ठहराव के लिए अतिरिक्त 1 μL बचाएं।
- यदि वांछित है, तो नमूना एकाग्रता के साथ आगे बढ़ें।
- डीएनए निष्कर्षण प्रक्रिया
- वाणिज्यिक फिल्टर इकाइयों के साथ नमूना एकाग्रता
नोट: अधिक विवरण के लिए वाणिज्यिक प्रोटोकॉल 26 देखें। डाउनस्ट्रीम उपयोग के आधार पर, नमूना एकाग्रता ( उदाहरण के लिए, अंत-बिंदु पीसीआर और qPCR अनुप्रयोगों के लिए) करने के लिए आवश्यक नहीं हो सकता है हालांकि, एनजीएस पुस्तकालय निर्माण के लिए डीएनए निष्कर्षण के बाद पतला ऑग्लानेल डीएनए प्राप्त करने पर ध्यान देने की आवश्यकता होगी।- एकाग्रता स्तंभ प्रक्रिया
- एक डिजिटल विश्लेषणात्मक संतुलन पर वजन कागज के एक साफ टुकड़े पर ध्यान से पूर्व वजन ( तालिका 2 देखें) खाली फिल्टर इकाई (एक ट्यूब के बिना)। वजन दर्ज करें
- अनुकरणीयसंयुक्त इल्यूशन को फिल्टर यूनिट में पट्ट करना और ध्यान से फिर से वजन करना।
नोट: वाणिज्यिक मैनुअल 26 का कहना है कि फिल्टर इकाई की अधिकतम मात्रा 500 μL है, लेकिन 575 μL तक यूनिट में बिना किसी अतिप्रवाह के साथ जोड़ा जा सकता है। - ध्यान से भरे हुए फिल्टर इकाई को एक ट्यूब में रखें (स्तंभों के साथ प्रदान किया गया) आवश्यक ध्यान मात्रा मात्रा हासिल करने के लिए इच्छित समय के लिए 500 xg पर अपकेंद्रित्र। ~ 575 μL की एक नमूना मात्रा के लिए, 20 मिनट की स्पिन आमतौर पर 15 - 30 μL के एक ध्यान मात्रा में होती है।
- ट्यूब से फिल्टर इकाई निकालें और फिर से तौलना यह निर्धारित करने के लिए तालिका का उपयोग करें कि वांछित ध्यान केंद्रित मात्रा हासिल की गई है या नहीं। यदि नहीं, तो समय की एक छोटी लंबाई के लिए 500 xg पर फिर से अपकेंद्रित्र और फिर से तौलना; वांछित ध्यान केंद्रित मात्रा तक पहुंचने तक दोहराएँ।
- फ़िल्टर यूनिट के शीर्ष पर एक नई ट्यूब (स्तंभों के साथ प्रदान की जाती है) डालें और पलटना। 1000 मिनट में 3 मिनट के लिए अपकेंद्रित्र को सह स्थानांतरणनलिका को नसीहत
- मात्रा बरामद निर्धारित करें। फ़िल्टर प्रतिधारण के कारण यह आमतौर पर ~ 3-5 μL की गणना की मात्रा से कम होगा। अधिक मात्रा में केंद्रित होने पर, वांछित मात्रा को प्राप्त करने के लिए बाँझ पानी या ते के साथ पतला।
- उच्च संवेदनशीलता मात्रा का ठहराव (प्रति निर्माता निर्देशों) का उपयोग करके डीएनए को बढ़ाएं।
- एकाग्रता स्तंभ प्रक्रिया
3. विधि # 2: कुल जीनोमिक डीएनए से ऑगेलर डीएनए के लिए समृद्ध करने के लिए मिथाइल-विभाजन (एमएफ) दृष्टिकोण
नोट: यह प्रोटोकॉल पौधों और कवक 27 के लिए एक उपयोगकर्ता-विकसित जीनोमिक टिप किट डीएनए निष्कर्षण प्रोटोकॉल और वाणिज्यिक माइक्रोबाइम डीएनए संवर्धन किट प्रोटोकॉल 28 से संशोधित किया गया था। सिद्धांत रूप में, किसी भी डीएनए अलगाव प्रोटोकॉल को उच्च-आणविक वजन डीएनए की पैदावार के लिए उपयोग किया जा सकता है, पुलडाउन के लिए। शॉर्ट-पढें अनुक्रमण के लिए, किसी भी निष्कर्षण को प्राथमिकता से उत्पन्न किया जाता है> पुलडाउन में उपयोग के लिए 15 केबी टुकड़े पर्याप्त हैं। लो के लिएएनजी-पढ़ना अनुक्रमण, बड़े टुकड़े वांछनीय हो सकते हैं। इसलिए, हमने इस प्रोटोकॉल को उच्च आणविक भार डीएनए प्राप्त करने के लिए अनुकूलित किया है।
- कुल डीएनए का अलगाव
नोट: आवश्यक उपकरण और अभिकर्मकों की सूची देखें और विधि # 2 ( तालिका 1 ) के लिए आवश्यक बफ़र्स और कार्यशील स्टॉक तैयार करें। Lysis बफर स्टॉक को lysing एंजाइम जोड़ें lysis बफर काम कर समाधान बनाने के लिए। थर्मोमिक्सर चालू करें और इसे 37 डिग्री सेल्सियस तक सेट करें पानी के स्नान को 50 डिग्री सेल्सियस पर बारी और स्नान में क्यूएफ बफर रखें। फ्रीज़र में 70% एटोहा रखें और 4 डिग्री सेल्सियस तक माइक्रोसेटर्रिफ्यूज सेट करें।- वाणिज्यिक डीएनए निष्कर्षण स्तंभों का उपयोग कर कुल डीएनए निकासी
नोट: शुरू करने से पहले गुरुत्वाकर्षण प्रवाह के आयनों-विनिमय कॉलम के उपयोग के बारे में विस्तृत जानकारी के लिए वाणिज्यिक पुस्तिका 2 पढ़ें। एक विशेष रैक का उपयोग करके कॉलमों को सेट किया जा सकता है या प्लास्टिक के छल्ले के उपयोग से ट्यूबों पर रखा जा सकता है। जी सहित सभी कदमएनोमिक टिप्स, को गुरुत्वाकर्षण प्रवाह से आगे बढ़ने की अनुमति दी जानी चाहिए, और अवशिष्ट तरल को इसके माध्यम से मजबूर नहीं किया जाना चाहिए।- 2 मिलीलीटर ट्यूबों के लिए डिज़ाइन किए गए हाथ से आयोजित पीसने वाले पेस्टल का उपयोग करके 2 मिलीलीटर की निम्न बाध्य ट्यूब में तरल नाइट्रोजन में 20 मिलीग्राम फ्रोजन टिशू को पीस लें।
- 2 मिलीलीटर का लिसेफेशन बफर काम करने वाला समाधान जोड़ें (ट्यूबें बहुत पूर्ण होंगी)
- 300 आरपीएम पर कोमल आंदोलन के साथ 1 घंटे के लिए 37 डिग्री सेल्सियस पर थर्मोमिक्सर में सेते हैं। यदि एक थर्मोमिक्सर उपलब्ध नहीं है, तो गर्मी ब्लॉक पर इनक्यूबेट करना और हर 15 मिनट में कोमल फिसलने से मिश्रण एक उपयुक्त विकल्प है।
- 4 μL आरएनज़ ए (100 मिलीग्राम / एमएल, 200 माइक्रोग्राम / एमएल की अंतिम एकाग्रता) जोड़ें। थर्मोमिक्सर में 30 मिनट के लिए 37 डिग्री सेल्सियस के मिश्रण और इनक्यूबेट करें, 300 आरपीएम पर कोमल आंदोलन के साथ।
- 300 आरपीएम पर कोमल आंदोलन के साथ, 50 डिग्री सेल्सियस पर 2 घंटे के लिए थर्मोमिक्सर में मिश्रण करने के लिए 80% प्रोटीनेस के (80 मिलीग्राम / एमएल, 0.8 मिलीग्राम / एमएल का अंतिम एकाग्रता) प्रोटीनेस जोड़ें।
- 4 डिग्री सेल्सियस और 1 में 20 मिनट के लिए अपकेंद्रित्रअघुलनशील मलबे को गोली से 5000 XG
- जबकि नमूनों को केन्द्रित किया जाता है, बफर QBT के 1 एमएल के साथ कॉलम को संतुलित करना और कॉलम को गुरुत्वाकर्षण प्रवाह से खाली करने की अनुमति देते हैं।
- नमूना (गोली से बचने) को समसामयिक स्तंभ में तुरंत लागू करने के लिए विस्तृत बोर विंदुक टिप का उपयोग करें और उसे स्तंभ के माध्यम से पूरी तरह से प्रवाह करने की अनुमति दें। नमूना स्तंभ के लिए आवेदन करने से पहले फिर से बादल छाए रहेंगे, फिल्टर या अपकेंद्रित्र हो जाता है (विवरण 29 के लिए वाणिज्यिक पुस्तिका देखें)।
- नमूना पूरी तरह से राल में प्रवेश करने के बाद, 4 x 1 एमएल की बफर क्यूसी के साथ स्तंभ धो लें।
- एक साफ, 2 एमएल, कम बाध्य माइक्रोसेंट्रिफ्यूज ट्यूब पर कॉलम को निलंबित करें। 50 डिग्री सेल्सियस पर पूर्ववर्ती बफर क्यूएफ के 0.8 एमएल के साथ जीनोमिक डीएनए को एल्यूट करें
- डीएनए को डीएनए में कमरे के तापमान के आइसोप्रोणोल के 0.56 एमएल (0.7 मात्रा में अल्युशन बफर) जोड़कर डीएनए की गति बढ़ाएं।
- उलटा (10 एक्स) और 15 मिनट और 15 डिग्री सेल्सियस पर 20 मिनट के लिए तुरंत अपकेंद्रित्र देखभालचकाचौंध, ढीले से जुड़े पैलेट को परेशान किए बिना सतह पर तैरने वाले को पूरी तरह से हटा दें।
- 1 मिलीलीटर ठंड 70% इथेनॉल के साथ सेंटीफ्यूज डीएनए गोली धो लें। 15 मिनट और 10 डिग्री सेल्सियस पर 10 मिनट के लिए अपकेंद्रित्र
- गोली को परेशान किए बिना सतह पर तैरने वाले को ध्यानपूर्वक हटाएं (इस चरण के साथ सावधान रहें) 5-10 मिनट के लिए वायु-सूखा और एल्यूएशन बफर (ईबी) के 0.1 एमएल में डीएनए को पुनः खोलें। कमरे के तापमान पर रात भर डीएनए भंग करें Pipetting से बचें, जो डीएनए को उतार सकते हैं।
- एक उच्च संवेदनशीलता डीएनए मात्रा का ठहराव परख (निर्माता निर्देशों के अनुसार) का इस्तेमाल करते हुए नमूनों को बढ़ाएं।
- वाणिज्यिक डीएनए निष्कर्षण स्तंभों का उपयोग कर कुल डीएनए निकासी
- मेथिलेटेड और अनमाइथिलेटेड डीएनए की मनका-आधारित विभाजन
नोट: एक हालिया प्रकाशन में वाणिज्यिक रूप से उपलब्ध किट 28 के उपयोग का प्रदर्शन किया गया, जो कि सीपीजी-विशिष्ट मिथाइल-बाइंडिंग डोमेन प्रोटीन का इस्तेमाल मानव आईजीजी एफसी टुकड़ा (एमबीडी 2-एफसी प्रोटीन) से भिन्न करने के लिए एक पुलडाउन दृष्टिकोण का लाभ उठाता है।परमाणु जीनोम (अत्यधिक मेथिलेटेड) सामग्री 23 से ऑटवेल प्लांट ऑजेंलेर जीनोम (अनमाइथिलेटेड) इस वाणिज्यिक एमएफ किट 28 का उपयोग करके गेहूं के नमूनों में फ्रेक्सेक्शन दक्षता पहले नहीं की गई थी।- शुरू करने से पहले करने के लिए चीजें
- हौसले से 80% इथेनॉल (प्रति प्रतिक्रिया कम से कम 800 μL) तैयार करें। बर्फ पर पिघलना करने के लिए 5x बाँध / वॉश बफर सेट करें और प्रति नमूना 5 एमएल 1x बफर प्रति तैयार करें (प्रोटूॉल के दौरान बाँझ, नूसलेज़ मुक्त पानी के साथ 5x बफर कम करें और बर्फ पर रखें)।
- एमबीडी 2-एफसी प्रोटीन बाध्य चुंबकीय मोती तैयार करें
- मनका सेटों की आवश्यक संख्या तैयार करें कुल इनपुट डीएनए के 1 और 2 माइक्रोग्राम के बीच उपयोग करने के लिए प्रतिक्रियाओं को स्केल करें, जिसमें मोती के 160-320 μL की आवश्यकता होती है। ध्यान दें कि नीचे दी गई प्रतिक्रियाएं कुल इनपुट डीएनए के 1 माइक्रोग्राम के लिए हैं, इसलिए उन्हें 160 μL मोती की आवश्यकता होती है। जरूरतों के अनुसार प्रतिक्रियाओं को स्केल करें
- चौड़े बोर युक्तियों का प्रयोग, धीरे से प्रोटीन ए चुंबकीय बी विंदुक करेंएक सजातीय निलंबन बनाने के लिए ऊपर और नीचे की ओर घूमना वैकल्पिक रूप से, धीरे-धीरे मोती की ट्यूब 4 डिग्री सेल्सियस पर 15 मिनट के लिए घुमाएं
नोट: मोती भंवर न करें - निर्माता निर्देशों के अनुसार दिशानिर्देशों के साथ आगे बढ़ें 28 ।
- मिथाइलेटेड परमाणु डीएनए कैप्चर करें
- प्रत्येक व्यक्ति के नमूने के लिए, 1 μg इनपुट डीएनए को एक ट्यूब में जोड़ें जिसमें 160 μL एमबीडी 2-एफसी-बद्ध चुंबकीय मोती शामिल हैं।
- 5x बाँध / वॉश बफर को डीएनए इनपुट नमूने की मात्रा 1x (5x बाइंड / वॉश बफर के वॉल्यूम (μL) = इनपुट डीएनए (μL) / 4) में जोड़ने के लिए दी गई मात्रा के अनुसार दी गई है। विस्तृत बोर विंदुक टिप का उपयोग करने के लिए कुछ समय के लिए नमूना को ऊपर और नीचे पिपेट करें।
- ट्यूबों को 15 मिनट के लिए कमरे के तापमान पर घुमाएं धीरे-धीरे बोर विंदुक टिप के साथ नमूनों को विंदुक और नमूनों को झटका दो से 3 बार मनका clumping को रोकने के लिए ऊष्मायन के दौरान।
नोट: pipetting और flickiमिथाइलेटेड डीएनए के कुशल पुलडाउन सुनिश्चित करने के लिए एनजी महत्वपूर्ण है
- समृद्ध, अनमाइथिलेटेड ऑर्गेनेल डीएनए लीजिए
- संक्षेप में डीएनए और एमबीडी 2-एफसी-बाध्य चुंबकीय मनका मिश्रण युक्त ट्यूब स्पिन करें। ट्यूब के किनारे मोतियों को इकट्ठा करने के लिए कम से कम 5 मिनट के लिए एक चुंबकीय रैक पर ट्यूब रखें। समाधान स्पष्ट दिखाई देना चाहिए।
- चौड़े बोर युक्तियों का उपयोग करते हुए, मोतियों को परेशान किए बिना साफ़ सतह पर तैरने वाले को ध्यान से हटा दें। एक स्वच्छ, कम बाँध, 2-एमएल माइक्रोसेंट्रिफ्यूज ट्यूब के लिए सतह पर तैरनेवाला (अनमाइथिलेटेड, ऑगनेलर-समृद्ध डीएनए) को स्थानांतरित करें। यह नमूना -20 या -80 डिग्री सेल्सियस पर स्टोर करें, या शुद्धि के लिए 3.2.6 चरण के लिए सीधे आगे बढ़ें।
- एल्यूट ने एमबीडी 2-एफसी-बाध्य चुंबकीय मोती से परमाणु डीएनए पर कब्जा कर लिया
- परमाणु अंश भी वांछित है, तो निर्माता निर्देश MBD2-एफसी बाध्य चुंबकीय मोतियों से परमाणु डीएनए elute करने के लिए 28 का पालन करें; चरण 3.2.7 में वर्णित अनुसार शुद्ध करें।
- मनका आधारित न्यूक्लिक एसिड शुद्धि
- सुनिश्चित करें कि शुद्धिकरण मोती कमरे के तापमान पर हैं और अच्छी तरह मिश्रित हैं। एमएफ किट मैनुअल 28 में दिए गए निर्देशों के अनुसार प्रोटोकॉल के साथ आगे बढ़ें
नोट: नमूना अब एनजीएस पुस्तकालय निर्माण या अन्य डाउनस्ट्रीम विश्लेषण के लिए इस्तेमाल किया जा सकता है।
- सुनिश्चित करें कि शुद्धिकरण मोती कमरे के तापमान पर हैं और अच्छी तरह मिश्रित हैं। एमएफ किट मैनुअल 28 में दिए गए निर्देशों के अनुसार प्रोटोकॉल के साथ आगे बढ़ें
4. नमूना मात्रा और गुणवत्ता नियंत्रण
- ऑर्गेनेलियर संवर्धन का आकलन करने के लिए qPCR परख
नोट: यहां सूचीबद्ध qPCR रिएक्शन और एटे पैरामीटर को रोश लाइट क्लाक्लर 480 पर उपयोग के लिए डिज़ाइन किया गया था और इसे विभिन्न उपकरणों और अभिकर्मकों के लिए समायोजित करने की आवश्यकता हो सकती है। यदि qPCR अनुपलब्ध है, तो agarose gel पर अंत-बिंदु पीसीआर और विज़ुअलाइज़ेशन को वर्णित एक ही प्राइमरों और शर्तों का उपयोग करते हुए, नमूना शुद्धता के गुणात्मक माप के रूप में उपयोग किया जा सकता है। एम्प्लिकॉन आकार सभी प्राइमर सेटों के लिए ~ 150 बीपी होगा। प्राइमर सीक्वैन के लिए तालिका 3 देखेंसीई और जोड़ी- QPCR प्रतिक्रिया सेटअप
- एक व्यक्तिगत 20 μL qPCR प्रतिक्रिया की स्थापना, एक 96-अच्छी तरह से qPCR प्लेट की एक कूल में निम्नलिखित को ध्यानपूर्वक पिपेट करें: 10 μL 2x SYBR ग्रीन I मास्टर; 10 माइक्रोन के 2 μL आगे और रिवर्स प्राइमर मिश्रण (0.5 माइक्रोन के अंतिम एकाग्रता के लिए); टेम्पलेट के 2 μL (मानक वक्र की सीमा के भीतर); और 6 μL बाँझ, न्युकली-फ्री एच 2 ओ। Pipetting त्रुटियों को कम करने के लिए, टेम्पलेट को छोड़कर सभी प्रतिक्रिया घटकों के साथ एक मास्टर मिश्रण बनाने के लिए बेहतर है। मुख्य मिश्रण को qPCR प्लेट में जोड़ें और फिर प्रत्येक अच्छी तरह से रुचि के टेम्पलेट को जोड़ें। Pipetting त्रुटि के प्रभाव को कम करने के लिए प्रत्येक नमूना के लिए तीन तकनीकी प्रतिकृतियां की जानी चाहिए।
नोट: आखिरकार, ऑक्ज़ेलर मात्रात्मक चक्रीय करने के लिए परमाणु के अनुपात की तुलना नमूने के बीच की जाती है, इसलिए एकाग्रता में मामूली अंतर स्वीकार्य है। हालांकि, ईएनए की सीमा के भीतर डीएनए सांद्रता मोटे तौर पर होनी चाहिएसी अन्य - एक उच्च गुणवत्ता वाले qPCR सीलिंग फिल्म के साथ प्लेट को सील करें धीरे-धीरे नमूने भंवर करें, किसी भी बुलबुले के निर्माण से बचने के लिए सावधानी बरतें। संक्षेप में प्लेट को नीचे 2 मिनट के लिए 4 डिग्री सेल्सियस पर स्पिन करें ताकि नमूना एकत्र कर किसी भी छोटे बुलबुले को समाप्त कर सकें।
- मशीन में प्लेट को लोड करें नीचे सूचीबद्ध दिशानिर्देशों के अनुसार qPCR कार्यक्रम चलाएं।
- एक व्यक्तिगत 20 μL qPCR प्रतिक्रिया की स्थापना, एक 96-अच्छी तरह से qPCR प्लेट की एक कूल में निम्नलिखित को ध्यानपूर्वक पिपेट करें: 10 μL 2x SYBR ग्रीन I मास्टर; 10 माइक्रोन के 2 μL आगे और रिवर्स प्राइमर मिश्रण (0.5 माइक्रोन के अंतिम एकाग्रता के लिए); टेम्पलेट के 2 μL (मानक वक्र की सीमा के भीतर); और 6 μL बाँझ, न्युकली-फ्री एच 2 ओ। Pipetting त्रुटियों को कम करने के लिए, टेम्पलेट को छोड़कर सभी प्रतिक्रिया घटकों के साथ एक मास्टर मिश्रण बनाने के लिए बेहतर है। मुख्य मिश्रण को qPCR प्लेट में जोड़ें और फिर प्रत्येक अच्छी तरह से रुचि के टेम्पलेट को जोड़ें। Pipetting त्रुटि के प्रभाव को कम करने के लिए प्रत्येक नमूना के लिए तीन तकनीकी प्रतिकृतियां की जानी चाहिए।
- QPCR प्रतिक्रिया पैरामीटर
नोट: यह प्रवर्धन अवस्था के annealing चक्र को छोड़कर डिफ़ॉल्ट पैरामीटर हैं। विशिष्ट प्राइमरों को समायोजित करने के लिए इस सेटिंग को समायोजित करें यदि वे इस प्रोटोकॉल में प्रस्तुत प्राइमरों से भिन्न हैं।- 5 डिग्री सेल्सियस के लिए 95 डिग्री सेल्सियस पर पूर्व सेते, 4.4 डिग्री सेल्सियस के रैंप दर के साथ।
- 4.4 डिग्री सेल्सियस के रैंप दर के साथ 10 एस के लिए (1) 95 डिग्री सेल्सियस के 45 प्रवर्धन चक्र करें; (2) 20 डिग्री के लिए 60 डिग्री सेल्सियस, 2.2 डिग्री सेल्सियस के रैंप दर के साथ; और (3) 10 डिग्री के लिए 72 डिग्री सेल्सियस, 4.4 डिग्री सेल्सियस के एक रैंप दर (डेटा (3 के दौरान अधिग्रहण) के साथ)।
- एक ऑप्टिओ का उपयोग करेंएनएएल 5 डिग्री के लिए 95 डिग्री सेल्सियस की वक्र चक्र, 4.4 डिग्री सेल्सियस के रैंप दर से पिघलता है; 65 डिग्री सेल्सियस 1 मिनट, 2.2 डिग्री सेल्सियस के रैंप दर के साथ; और 97 डिग्री सेल्सियस, एक निरंतर अधिग्रहण मोड के साथ।
- 30 डिग्री के लिए 40 डिग्री सेल्सियस के एक ठंडा चक्र का प्रयोग करें, 1.5 डिग्री सेल्सियस के रैंप दर के साथ।
- परख पैरामीटर
- SYBR टेम्प्लेट का चयन करें। प्रयोग बटन में प्रोग्राम पैरामीटर देखें। प्लेट लोड होने पर, परख शुरू किया जा सकता है, और सेटिंग को समायोजित किया जा सकता है, जबकि परख चल रही है।
- नमूना संपादक का उपयोग कर नमूने असाइन करें। अॉब क्वांट को कार्यप्रवाह के रूप में चुनें और अज्ञात, मानकों, या नकारात्मक नियंत्रण के रूप में नमूने निर्दिष्ट करें। Designate प्रत्येक प्रतिकृति के पहले के नमूने नामों की प्रतिकृति और भरें। मानकों को सांद्रता और इकाइयां जोड़ें
- विश्लेषण के लिए सबसेट सेट करें; इन सबसेट एडिटर में नियुक्त किया जाता है।
- विश्लेषण के लिए, "नया विश्लेषण बनाएँ" सूची से एब कोट / 2 डी व्युत्पन्न मैक्स का चयन करें।बाहरी रूप से सहेजी मानक वक्र आयात करें (यदि लागू हो) और फिर हिट गणना करें; रिपोर्ट में चयनित जानकारी शामिल होगी
- प्रति संख्या या एकाग्रता के निर्धारण के लिए सटीक मात्रा का ठहराव करने के लिए, एक मानक वक्र का उपयोग करें, जो नमूना का परीक्षण किया जा रहा है ( उदाहरण के लिए, उपरोक्त तरीकों से अलग ऑर्गेनेल डीएनए)। चूंकि माइटोकॉन्ड्रियल डीएनए की मात्रा एक मानक वक्र तैयार करने के लिए आवश्यक है, ऊतक की एक उचित मात्रा के साथ प्राप्त करने के लिए बहुत अधिक है, इसलिए सॉफ़्टवेयर द्वारा प्रदान किए गए प्रति संख्या की गणना का उपयोग न करें, बल्कि रिश्तेदार संवर्धन को निर्धारित करने के लिए क्रॉसिंग पॉइंट (सीपी) मानों की जांच करें। के नमूने में परमाणु डीएनए की तुलना में organellar की। कुल जीनोमिक डीएनए ( प्रतिनिधि परिणाम देखें) की तुलना में इन रिश्तेदार मात्रा की तुलना करें। पूरी तरह हल्का-उगने वाले, दो सप्ताह के पुराने गेहूं के पौधों से कुल जीनोमिक डीएनए के पाँच 1:10 को भ्रष्टाचार पर टेस्ट प्राइमर की क्षमताएंचित्रा 2 की ई कथा)
- QPCR प्रतिक्रिया सेटअप
- स्पंदित-क्षेत्र जेल वैद्युतकणसंचलन (पीएफजीई)
नोट: उच्च प्रोटोकॉल हाई आणविक भार डीएनए को हल करने के लिए पीएफजीई करने के लिए निर्माता दिशानिर्देशों पर आधारित है। सामग्री तालिका देखें- जेल और नमूनों की तैयारी
- जेल और नमूना तैयार करने के लिए दिशानिर्देशों का पालन करें और उपलब्ध सिस्टम में उन्हें अनुकूलित करें।
- पैरामीटर चलाएं
- वैद्युतकणसंचलन प्रणाली की स्थापना के लिए दिशानिर्देशों का पालन करें और निम्न मापदंडों का उपयोग करें: 2 एस के प्रारंभिक स्विच समय, 13 सेकंड के अंतिम स्विच समय, 15 घंटे और 16 मिनट का समय, 6 के वी / सेमी, और 120 डिग्री का कोण शामिल है ।
- दाग और जेल छवि
- जेल को पसंद की एक डाई ( उदाहरण के लिए, नैथिडियम ब्रोमाइड या उपयुक्त विकल्प) और एक उपयुक्त जेल दस्तावेजीकरण प्रणाली के साथ छवि दागें।
- जेल और नमूनों की तैयारी
- डीएनए लाइब्रेरी तैयारी किट के लिए इनपुट के रूप में 1 एनजी डीएनए का उपयोग करें, निर्माता के निर्देशों के अनुसार।
- बारकोड और एकल रन में अनुक्रमण के लिए नमूनों को पूल। निर्माता दिशानिर्देशों के अनुसार क्रमिक प्रदर्शन करें।
नोट: पूलिंग और अनुक्रमण मापदंडों को हित की प्रजातियों, वांछित कवरेज स्तर, और पुस्तकालयों के अनुक्रम के लिए उपयोग किए गए मंच के आधार पर बदल दिया जा सकता है। उदाहरण के लिए, एक हायसेक लेन में MiSeq लेन की तुलना में काफी अधिक उत्पादन होता है, इसलिए कई नमूने मल्टीप्लेक्स हो सकते हैं। यह निर्धारित करने के लिए नमूने के एक छोटे उपसमूह को अनुक्रमित करें कि ऑगनेरल जीनोम का कवरेज स्तर नीचे की ओर के विश्लेषण के लिए पर्याप्त है या नहीं।- डाटा के लिए आवश्यक ट्रिमिंग और फ़िल्टरिंग की सीमा निर्धारित करने के लिए फास्टक्यूसी 31 का उपयोग करते हुए पढ़ने की गुणवत्ता की जांच करें।
- त्रिकोमिक 32 या किसी अन्य तुलनीय कार्यक्रम का प्रयोग करके कच्चे पढ़ता है और छानिये। निम्नलिखित सेटिंग्स का उपयोग करें: ILLUMIएनएसीएलआईपी 2:30:10 (एडेप्टर निकालने के लिए), लीडिंग 3, ट्रायलिंग 3, स्लिपिंगविंडो 4:10, और मिनलेन 100।
- गुणवत्ता-फ़िल्टर्ड और एडेप्टर-ट्रिम युक्त पेयर-एंड (पीई) को मानचित्रित करें, चीनी वसंत मिटोकॉन्ड्रियल (एनसीबीआई संदर्भ सीक्वेंसी NC_007579.1 33 ), क्लोरोप्लास्ट (एनसीबीआई संदर्भ सीक्वेंस NC_002762.1 34 ), और परमाणु 35 संदर्भ जीनोम बोल्टि 2 36 , निम्नलिखित सेटिंग्स के साथ: I 0 -X 800 - संवेदनशील
- सैम संरेखण फ़ाइलों को बाम प्रारूप (समतोल) में कनवर्ट करें और बैम फ़ाइलों को सॉर्ट करें। जीनोम-व्यापी कवरेज की गणना करने के लिए बेम फ़ाइलों का उपयोग करें और बेड-टूल्स के साथ प्रति-आधार कवरेज आर-प्लॉट फ़ंक्शन के साथ परिणामों को विज़ुअलाइज़ करें
- शुरू करने से पहले करने के लिए चीजें
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
इस पांडुलिपि में प्रस्तुत प्रोटोकॉल संयंत्र के ऊतकों से organellar डीएनए के लिए समृद्ध करने के लिए दो अलग तरीकों का वर्णन करते हैं। यहां प्रस्तुत शर्तों में गेहूं के ऊतकों के अनुकूलन को दर्शाया गया है। प्रोटोकॉल, आवश्यक ऊतक इनपुट, और डीएनए आउटपुट में महत्वपूर्ण कदमों की एक तुलना चित्रा 1 में वर्णित है। डीसी प्रोटोकॉल के चरण हमने पहले वर्णित उन लोगों के समान स्थितियों का अनुसरण किया ( चित्रा 1 ए )। कटाई वाले ऊतक को हाल में संसाधित किया जाना चाहिए और पृथक ऑर्गेनल्स को अलग करने के लिए विभेदक केन्द्रोत्सर्जन और / या ग्रेडिएंट के अधीन किया जाना चाहिए। ऑर्गेनल्स लिशीड होने से पहले परमाणु डीएनए समाप्त हो जाता है, और अंत में, डीएनए निकाला जाता है और डाउनस्ट्रीम अनुप्रयोगों के लिए उपयोग किया जाता है। इसके विपरीत, एमएफ प्रोटोकॉल में, पौधे के ऊतकों का उपयोग करने से पहले काटा और संग्रहीत किया जा सकता है, और बरकरार ऑर्गेनल्स की आवश्यकता नहीं है। इसके बजाय, परमाणु और ऑर्गेनेल डीएनए को कुल जीडीएनए से अलग किया जाता है डीएनए की मेथिलिकेशन स्थिति दोनों प्रोटोकॉल ऑर्गेनेल डीएनए ( चित्रा 1 सी ) की लगभग बराबर मात्रा में उत्पन्न करते हैं। ऊतक इनपुट के मुकाबले कुल organellar डीएनए आउटपुट के संदर्भ में, एमआईएफ प्रोटोकॉल फायदेमंद होता है जब ऊतक सीमित होता है, क्योंकि एक ही संयंत्र से एक छोटा सा नमूना इस्तेमाल किया जा सकता है और संयंत्र को आगे के विश्लेषण के लिए विकसित करने की अनुमति दी जा सकती है। आमतौर पर, डीसी प्रोटोकॉल में, कई पौधों के सभी हवाई ऊतकों की आवश्यकता होती है, और इन पौधों को त्याग दिया जाता है। हालांकि, डीसी पद्धति को दूसरे पर एक ऑग्नेल प्रकार के लिए विशेष रूप से समृद्ध करने के लिए अनुकूलित किया जा सकता है, जो एमएफ दृष्टिकोण के साथ संभव नहीं है। यह उल्लेखनीय है कि प्रत्येक प्रोटोकॉल के लिए कुल समय लगभग समतुल्य है, हालांकि एमएफ दृष्टिकोण में हाथों पर कम समय नहीं है।
ऑब्जेनल डीएनए के लिए दोनों तरीकों को समृद्ध, यद्यपि मिटोकोंड्रिया और प्लॉस्टीड सीक्वेंस के भिन्न अनुपात के साथ:
"> शुद्ध ऑर्गेनेल डीएनए की बहुत कम मात्रा या तो विधि से प्राप्त की जाती है (~ 50 - 100 एनजी; चित्रा 1 सी के आदेश पर)। ऑनेलर जीनोम संवर्धन और डीएनए में परमाणु जीनोम दूषित होने के स्तर का आकलन करने के लिए डीसी और एमएफ तरीकों, एक qPCR परख नियोजित किया गया था। इस परख में, तीन amplicons ( यानी, परमाणु-विशिष्ट, ACTIN ; mitochondrial-specific, NAD3 ; क्लोरोप्लास्ट-विशिष्ट, पीएसबीबी ) का कुल जीनोमिक डीएनए में मूल्यांकन किया गया था, और ऑर्गेनेल डीएनए अंश दोनों तरीकों ( चित्रा 2 ) से प्राप्त किया गया था। प्रत्येक नमूना ( चित्रा 2 ए ) के लिए मात्रा चक्र (सी क्यू ) मूल्यों की जांच की गई और सी q को पीसीआर चक्र के रूप में परिभाषित किया गया है, जिस पर लक्ष्य प्रवर्धन से प्रतिदीप्ति पृष्ठभूमि प्रतिदीप्ति स्तर, सी क्यू और लक्ष्य बहुतायत से ऊपर बढ़ जाता है उलटा नाता। मेंडीसी नमूना, NAD3 और PSBB की सी क्यू हैं, क्रमशः, ~ 17 और ~ 15 चक्र actin (जिनमें से एक सी क्यू ~ 36 है) से पहले (चित्रा 2 बी को देखने के सी क्यू मूल्यों और संवर्धन के स्तर के लिए)। यह NAD3 और PSBB, क्रमशः के लिए सैद्धांतिक 167,181- और 47,790 गुना संवर्धन के बराबर, डीसी नमूने में एटीटीएन के सापेक्ष ( चित्रा 2 बी , गणना के लिए चित्रा 2 की कथा देखें)। कुल जीनोमिक डीएनए नमूने में, actin के लिए NAD3 और PSBB रिश्तेदार गुना संवर्धन केवल 158 और 10,701 क्रमशः। कुल जीनोमिक डीएनए में परमाणु अम्लिपिकन के सापेक्ष ऑग्नेलिक एम्प्लिकॉन के उच्च बहुतायत को खोजने में आश्चर्य की बात नहीं है, यह देखते हुए कि ऑगनेरल जीनोम परमाणु जीनोम 37 की तुलना में प्रति कोशिकाओं की अधिक प्रति संख्या में मौजूद है और यह कि ऑर्गेनेल पे की संख्याआर सेल ऊतक प्रकार या विकास चरण 38 , 3 9 के आधार पर भिन्न हो सकता है। कुल मिलाकर, आंकड़ों से पता चलता है कि डीसी पद्धति मिटोकोंड्रिया के लिए प्राथमिकता से बढ़ती है, जो उम्मीद की जानी चाहिए, क्योंकि केन्द्रापसारक गति चुनिंदा मिटोकोंड्रिया को अलग करने और परमाणु और क्लोरोप्लास्ट "प्रदूषण" को कम करने के लिए अनुकूलित हैं।एमएफ कुल जीडीएनए का अनमाइथिलेटेड अंश ऑर्गेनेल एम्प्लिकॉन दोनों के पर्याप्त संवर्धन को दर्शाता है और इन लक्ष्यों की मूल रिश्तेदार मात्रा को बनाए रखने की उम्मीद है। (चित्रा 2A और 2 बी) के लिए unmethylated अंश में actin के NAD3 और PSBB रिश्तेदार गुना संवर्धन के 20,551 और 1,703,253 क्रमश: कर रहे हैं। मिथाइल अंश में, के लिए actin के NAD3 और PSBB रिश्तेदार गुना संवर्धन के क्रमश: इंडी 31 कर रहे हैं और 823,यह बताते हुए कि एमबीडी 2-एफसी प्रोटीन मैथिलेटेड परमाणु डीएनए के पुलडाउन पर अत्यधिक कुशल है। चूंकि क्लोरोप्लास्ट एम्प्लिकॉन के पास कुल जीनोमिक डीएनए (~ 6 सी क्यू पहले) में मिटोकोलेड्रल एम्पिलिकन, मेथाइलेटेड अंश (~ 5 सी क्यू पहले), और अनमाइथिलेटेड फ्रैक्चर (~ 6 सी क्यूके पहले) नमूनों की तुलना में अधिक प्रचुरता है, इससे पता चलता है कि एमडीबी 2 पुलडाउन द्वारा इन एम्प्लिक्ंसों की मूल बहुतायत काफी हद तक बदली नहीं है हम इन जीनोमों के विशेष रूप से क्रमशः में रुचि के कारण अनमाइथिलेटेड (ऑलंनेल) अंश पर ध्यान देते हैं। हालांकि, अगर परमाणु जीनोम प्राथमिक ब्याज है, एमएफ़ और मेथाइलेटेड अंश की अनुक्रमिक अनुक्रम कुल जीनोमिक डीएनए अनुक्रमण के मुकाबले बहुत अधिक परमाणु जीनोम कवरेज पैदा करेगा, क्योंकि ऑर्गेनेल डीएनए "संदूषण" में कमी के कारण।
यह ध्यान देने योग्य है कि यदि qPCR उपलब्ध नहीं है, अंत-बिंदु पीसीआर (qPCR के लिए एक ही प्राइमर का उपयोग करके) योग्यता प्रदान करता हैऑगेंलेर शुद्धता का मूल्यांकन इस मामले में, शुद्ध organellar डीएनए नमूने मिटोकोन्ड्रियल और प्लैस्टिड एम्प्लिकॉन के लिए प्रवर्धन दिखाएंगे, लेकिन agarose जेल पर परमाणु एम्प्लिकॉन का कोई पता लगाने योग्य प्रवर्धन नहीं होगा, जबकि कुल जीनोमिक डीएनए सभी तीन प्राइमर सेटों के लिए प्रवर्धन दिखाता है, जैसा कि पिछले अध्ययनों में दर्शाया गया है 11 , 12
दोनों तरीकों से अलग ऑर्गेलर डीएनए एनजीएस के लिए उपयुक्त है:
छंटनी और साफ पीई अनुक्रमण को पढ़ता है (चरण 4.3 देखें) पहले प्रकाशित गेहूं ऑग्लेनेल रिरेक्शन जीनोम के लिए मैप किए गए थे, और प्रत्येक नमूना के मानचित्रण के लिए इस्तेमाल किए गए पढ़े जाने की मात्रा ~ 800,000 से 1,100,000 रीडिंग ( चित्रा 3I ) तक होती है। मैपिंग डी नोवो इलुमिना अनुक्रमण से परिणाम उपलब्ध गेहूं क्लोरोप्लास्ट और मिटोचोनड्रिया जीनोम के लिए पढ़ता है qPCR Res के साथ संगत हैएमआईटीओकोड्रियल डीएनए ( चित्रा 3 ए और 3 बी , ~ 80% और ~ 10% में एमसीटी और क्लोरोप्लास्ट (सीपी) जीनोम को क्रमशः मैप्स पढ़ने के लिए डीसी पद्धति से उत्पन्न डीएन पद्धति के साथ) और एमएफ विधि डीएनए से उत्पन्न होने वाली संभावना है कि ऑर्गेनेल जीनोम ( आंकड़े 3 ए और 3 बी , ~ 20% और ~ 80% एमटी और सीपी जीनोम को क्रमशः नक्शे को पढ़ता है) की मूल प्रचुरता को दर्शाता है। दोनों तरीकों में, दोनों गेहूं ऑज़नलार जीनोम के सैद्धांतिक कवरेज (गणना के लिए चित्रा 3 की कथा देखें) 100 एक्स कवरेज से अधिक है (और एमएफ विधि से अनमाइथिलेटेड अंश में क्लोरोप्लास्ट जीनोम के लिए ~ 2,000 एक्स कवरेज तक की दूरी), यहां तक कि जब 12 पुस्तकालयों मल्टिप्लेक्स ( चित्रा 3 सी और 3 डी) हैं , इस विश्लेषण में शामिल 6 पुस्तकालयों को एक अतिरिक्त 6 पुस्तकालयों के साथ अलग विश्लेषण के लिए जमा किया गया था, कुल 12 पुस्तकालयों के लिएएक एकल अनुक्रमण लेन में जमा) कवरेज के एक अधिक विस्तृत विचार विशिष्ट गहराई में शामिल जीनोम के अंश के साथ-साथ प्रति-आधार कवरेज स्तर ( चित्रा 3 ई -3 ई ) की जांच करके प्राप्त किया गया था। एमएफ विधि के लिए, एमटी जीनोम के लिए औसत प्रति-आधार कवरेज ~ 300-450X और सीपी जीनोम के लिए 4,000-5000 एक्स था। डीसी पद्धति के लिए, क्रमशः एमटी और सीपी जीनोम के लिए प्रति-आधार कवरेज ~ 900 - 1,300 और ~ 500-700X थी। हालांकि, एमटी और सीपी जीनोम दोनों का एक छोटा सा अंश था, जो बेहद कम या उच्च कवरेज थे, और यह ऑर्गेनेल डीएनए में देखा गया था, जो किसी भी विधि ( चित्रा 3I ) से प्राप्त हुआ था। ऑर्गेनेल जीनोम के बीच समरूपता के क्षेत्र से अधिक औसत-औसत कवरेज की संभावनाएं, और कम कवरेज वाले क्षेत्रों में एसएनपी या अन्य लघु रूपों का संकेत हो सकता है जो हमने अनुक्रमित और प्रकाशित संदर्भों के बीच किया था। इस धारणा के समर्थन में, ये स्पाइकइस पद्धति में सीपी जीनोम की उच्च कवरेज की वजह से संभवतः एमएफ विधि ( आंकड़े 3 ई और 3 आई ) से प्राप्त एमटी डीएनए के लिए उच्च कवरेज का सबसे ज्यादा स्पष्ट किया गया था। अनजाने में, सीपी जीनोम का कवर डीसी पद्धति ( चित्रा 3 जी और 3 एच ) की तुलना में एमएफ विधि में अधिक असमान है, जो सीपी डीएनए के साथ एमबीडी 2-एफसी पुलडाउन में मामूली पूर्वाग्रहों के कारण हो सकता है। आगे के प्रयोगों को यह निर्धारित करने की आवश्यकता होगी कि ऐसा क्यों है। भले ही, एमटी और सीपी जीनोमों में अपेक्षाकृत दोनों विधियों और गुम कवरेज के बड़े क्षेत्रों के साथ कवरेज होती है, जो किसी दी गई गहराई पर दी गई जीनोम के अंश ( चित्रा 3 ई -3 एच ) की परीक्षा से प्रदर्शित किया जा सकता है। इसके अतिरिक्त, दोनों जीनोमों के लिए कवरेज के स्तर को डाउनस्ट्रीम विश्लेषण के लिए पर्याप्त माना जाता है, जैसे कि वेरिएंट विश्लेषण यदि दुर्लभ संस्करणों के विश्लेषण के लिए आवश्यक समझा, numbe को कम करनेजमा किए गए नमूनों के आर को अधिक कवरेज प्राप्त होगा। वैकल्पिक रूप से, हायसेक लेन पर एक बहुत अधिक संख्या में नमूनों को जमा किया जा सकता है, जबकि अनुक्रम की लंबाई के लिए बलिदान पर यद्यपि अधिक से अधिक अनुक्रमण गहराई प्राप्त होती है, क्योंकि हायसेक पुस्तकालय वर्तमान में पीई 30000 एमईएसईईक पुस्तकालयों के विपरीत PE150 की लंबाई में सीमित हैं।
मैपिंग दृष्टिकोण का उपयोग करके परमाणु जीनोम के प्रदूषण के स्तरों की जांच करने के लिए, पीई पढ़ने वाली मैपिंग श्रेणियों की जांच की गई। पीई पढ़ता है कि विन्यास की एक किस्म में एक संदर्भ जीनोम में मैप कर सकता है। जब 1 और 2 पढ़ता है तो सिर-टू-हेड फैशन में संदर्भ में संरेखित करें, दो साथी के बीच एक निश्चित "अपेक्षित" दूरी के साथ (लाइब्रेरी के औसत डालने के आकार के आधार पर और मैपिंग सॉफ्टवेयर में इनपुट पैरामीटर के रूप में निर्दिष्ट किया जाता है ), इन पीई पढ़ा जाता है कि "समवर्ती" नक्शा कहा जाता है। इसके विपरीत, "असंतोष" मानचित्रण वह स्थिति है जहां साथी कम-या-अधिक-अपेक्षा-अपेक्षित डिस्क के साथ मानचित्र करते हैंवैकल्पिक विन्यास में संदर्भ जीनोम या मैप के लिए तनाव (सिर से पूंछ या पूंछ-पूंछ) यदि केवल एक साथी संदर्भ जीनोम में संरेखित करता है, तो उस पीई को पढ़ने के संदर्भ में जीनोम के संदर्भ में न तो एकसाथ या असुविधाजनक रूप से नक्शा कहा जाता है। सभी तीन पढ़-मैपिंग श्रेणियों में, पीई पढ़ता संदर्भ जीनोम को एक या कई बार संरेखित कर सकता है।
डीसी और एमएफ-पृथक ऑर्गेनेल डीएनए दोनों के लिए, मितोचोन्ड्रियल जीनोम के मानचित्रण को मुख्यतः गठबंधन में एक बार श्रेणी ( चित्रा 4 ए ) में पढ़ना था, जबकि एक समय के समतुल्य रूप से अपेक्षाकृत बराबर अनुपात में मैल किए गए और समरूप रूप से एक बार मैप किया जाता है। एक समय ( चित्रा 4 बी ), संभवतः क्लोरोप्लास्ट जीनोम में मौजूद बड़े उल्टे दोहरावों के कारण और अत्यंत उच्च कवरेज स्तर तक भी। हालांकि, कम पीई परमाणु जीनोम के लिए मैप पढ़ता है और काफी हद तक एक बार में एक से अधिक मैप किया जाता हैन तो संकात्मक और न ही असाधारण फैशन ( यानी, केवल एक दोस्त मानचित्रण करने में सक्षम है)। यह परमाणु जीनोम में अनुक्रमों के लिए "ऑफ-टारगेट" का मैपिंग सबसे अधिक संभावना है, जो ऑर्गेनेल जीनोम या दुघर्टने वाले क्षेत्रों के मुताबिक हैं। परमाणु जीनोम में एकमात्र रूप से पढ़ा जाने वाला मामूली राशि (<5%), डीएनसी या एमएफ विधि ( चित्रा 4 सी ) से पृथक ऑनेलवेल डीएनए में परमाणु जीनोम दूषित कम स्तर का संकेत देता है, जैसा कि qPCR परिणाम ( चित्रा 2 ए )। चीनी स्प्रिंग से गैर-एटिलेटेड ऊतकों के एमबीडी 2-एफसी पुलडाउन के बाद परमाणु अंश भी यह निर्धारित करने के लिए अनुक्रमित किया गया था कि कैसे पुलडाउन बिना मैथिथिलेटेड डीएनए को हटाने में है 1% से कम अणु अंश-व्युत्पन्न लाइब्रेरी में पढ़ा जाता है जो ऑगनेल रेफरेंस जीनोम में मैप किया जाता है, जबकि सभी ~ 45% परमाणु जीनोम ( चित्रा 4 ) के लिए मैप पढ़ता है। हालांकि, सबसे अधिक एक असंतुलित फैशन में मैप पढ़ता है, डब्ल्यूहिक की संभावना गेहूं के परमाणु संदर्भ जीनोम में गलत विभाजन और विखंडन के उच्च स्तर को दर्शाती है। भले ही, परिणाम बताते हैं कि मैथिलेटेड परमाणु डीएनए से अनमाइथिलेटेड ऑगनेरल डीएनए हटाने पर एमबीडी 2-एफसी पुलडाउन अत्यधिक कुशल है। यह ध्यान देने योग्य है, क्योंकि इन तरीकों से उत्पन्न ऑगनेगलर-समृद्ध डीएनए में मिटोकोंड्रिया और क्लोरोप्लास्ट अनुक्रमों का मिश्रण होता है, और क्योंकि इन अंगों के बीच प्राचीन जीन अंतरण के परिणामस्वरूप अनुक्रम समानताएं उनके जीनोम में रहते हैं, उचित असाइनमेंट को पढ़ता है विशिष्ट जीनोमों को बायोइनफॉरमैटिक रूप से हल किया जाना चाहिए।
पत्ता ऊतक का इटोलिशन ऑर्गेनोल ऐंडबन्सेस को अनुकूल नहीं करता है:
पारम्परिक रूप से, फिनोलिक्स और स्टार्च के स्तर को कम करने के लिए एटिलेटेड ऊतक पौधे मिटोकॉन्ड्रियल डीएनए अलगाव के लिए पसंद किया जाता है, जो एक्स्ट्रेसिओ के साथ हस्तक्षेप कर सकता हैN या डाउनस्ट्रीम एप्लिकेशन 13 यह निर्धारित करने के लिए कि ऑ organellar जीनोम संवर्धन के स्तर में वृद्धि की स्थिति में सुधार या सुधार किया जा सकता है, दोनों etiolated और गैर- etiolated ऊतकों एमएफ प्रोटोकॉल और अनुक्रमण के अधीन थे। दिलचस्प बात यह है कि गैर-एटिलेटेड परिस्थितियों के मुकाबले ऑगनेल रेफरेंस जीनोम ( आंकड़े 3 ए और 3 बी ) या प्रति-आधार कवरेज ( चित्रा 3 आई ) में मैप किए गए पढ़े जाने वाले प्रतिशत के लिए अनुकूलता नहीं बदला। हम अलग-अलग और गैर-एटिलेटेड ऊतकों दोनों के साथ विभेदक केन्द्रापूर्णता का उपयोग कर अलग-अलग डीएनए (डीएनए) को भी अलग कर चुके हैं, और qPCR (डेटा नहीं दिखाया गया) का उपयोग करते हुए विभिन्न ऊतकों के बीच संश्लेषण में थोड़ा अंतर पाया गया था। इससे पता चलता है कि अधिक शारीरिक रूप से प्रासंगिक गैर-एटिलेटेड ऊतकों का उपयोग ऑगनेरल अनुक्रमण के अध्ययन के लिए किया जा सकता है, साथ ही संवर्धन का कोई अनुकूल बदलाव नहीं किया जा सकता है।
गुणवत्ता नियंत्रण यह बताता है किएमएफ डीएनए लम्बी-पढ़े हुए सीक्वेंसिंग के लिए सबसे उपयुक्त है:
चूंकि लंबे समय से पढ़े गए अनुक्रम शोधकर्ताओं के लिए अधिक सुलभ हो जाते हैं, उच्च आणविक भार डीएनए का अलगाव तेजी से महत्वपूर्ण होता जा रहा है। अस्थिरता और गुणवत्ता के लिए किसी भी विधि से पृथक ऑलंनेल डीएनए का आकलन करने के लिए, पीएफजीई को नियोजित किया गया था। कुल जीनोमिक डीएनए आम तौर पर पीएफजी में फैलानेवाला धब्बा के रूप में पलायन करता है, और प्रोटोकॉल पर आणविक भार निर्धारित होता है और डीएनए को निष्कासन कैसे संग्रहीत और नियंत्रित किया जाता है। जीनोमिक सुझावों के साथ पृथक कुल जीनोमिक डीएनए 50 केबी से अधिक होना चाहिए, जो कि पीएफजीई ( चित्रा 5 , लेन 2) का प्रयोग करके सत्यापित किया गया था। जीनोमिक युक्तियों के कुल जीनोमिक डीएनए को ऑर्बोबियम एंकरमेंट किट में इनपुट के रूप में प्रयोग किया जाता है ताकि ऑर्गेनेल डीएनए से परमाणु को अलग किया जा सके। विभक्त होने के बाद परमाणु अंश प्राप्त हुआ, आकार में कमी आई है, लेकिन 50 केबी ( चित्रा 5 , लेन 4) के आसपास केंद्रित रहता है। यह सु नहीं हैयह देखते हुए कि एमबीडी 2-एफसी-बाध्य मोतियों से अलौकिक के रूप में परमाणु अंश की अपेक्षाकृत रौप्यता से निपटने के लिए गर्मी और प्रोटीनेस की पाचन आवश्यक है। सीमित द्रव्यमान के कारण, ऑग्नेलवेल अंश को पीएफजीई पर नहीं चलाया गया था, लेकिन टेपस्टेशन के बाद के विश्लेषण में डीएनए> 50 केबी (डेटा नहीं दिखाया गया) दर्शाया गया था। अंतर सेंट्रीफ्यूजेशन के साथ प्राप्त ऑनेलवेल डीएनए का औसत द्रव्यमान ~ 20 केबी होता है, संभवतः विस्तारित ऑगनेल अलगाव प्रोटोकॉल और इसके बाद के कॉलम-आधारित डीएनए निष्कर्षण और एकाग्रता के कारण होता है। ढाल-आधारित आँगेलर अलगाव और वैकल्पिक डीएनए निष्कर्षण विधि बड़े डीएनए टुकड़ा आकार बनाए रख सकते हैं। भले ही, इस प्रोटोकॉल में प्राप्त आकार के डीएनए 10- या 15-kb अनुक्रमण उत्पन्न करने के लिए इस्तेमाल किया जा सकता है यदि पुस्तकालय की तैयारी के दौरान ध्यान रखा जाता है।
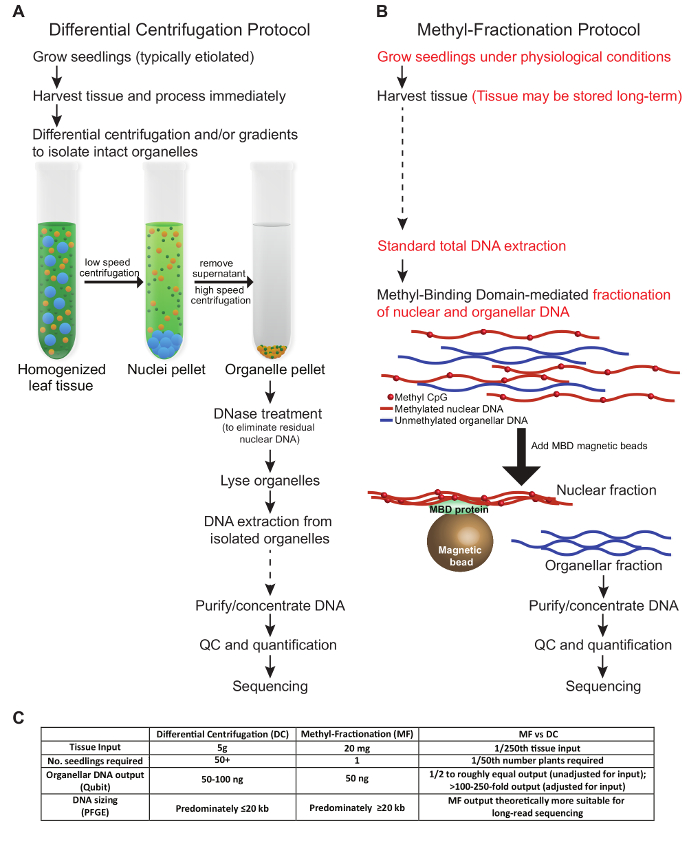
चित्रा 1: दो मेथो का एक तुलनात्मक दृष्टिकोणप्लांट ऑगेलर डीएनए के लिए समृद्ध करने के लिए डीएसए एक पारंपरिक डीसी प्रोटोकॉल ( ए ) एमएफ प्रोटोकॉल ( बी ) के साथ विपरीत है। नमूनों को ठंड और विगलन से बचने के लिए सिफारिश की जाती है; हालांकि, ऐसे कदम जिन पर नमूने लंबे समय तक संग्रहीत किए जा सकते हैं, उन्हें धराशायी तीरों ( ए और बी ) से संकेत मिलता है। प्रोटोकॉल के बीच मुख्य अंतर लाल ( बी ) में हाइलाइट किए गए हैं। ( सी ) तालिका ऊतक इनपुट, आवश्यक पौधों की संख्या, डीएनए उत्पादन, और जिसके परिणामस्वरूप डीएनए आकार के मामले में तरीकों की तुलना करती है। इस आंकड़े के एक बड़े संस्करण को देखने के लिए कृपया यहां क्लिक करें

चित्रा 2 चित्रा 2: दो तरीकों का उपयोग पृथक डीएनए पृथक डीएनए में परमाणु डीएनए संदूषण का आकलन। (
( बी ) तालिका सी क्यू मानों को दर्शाती है, जो एपीटीएन के सापेक्ष ऑर्गेनेल एम्प्लिकॉन के ( ए ) में ग्राफ़ पर दिखाए गए हैं, और गुना संवर्धन * तराजू संवर्धन = 2 ( सीसीएटीटीएन - सीसी लक्ष्य) सूत्र प्रत्येक प्राइमर सेट के लिए 2 की एक आदर्श क्षमता रखता है, चूंकि मामूली देवताप्रत्येक प्राइमर 2 से सेट की आयन (, NAD3 = 1.95, और PSBB = 1.989 actin = 1.961) नगण्य है और गणना और समग्र प्रवृत्ति पर खास असर नहीं होगा। कुल जीनोमिक डीएनए के पाँच 1:10 की मात्रा के साथ एक मानक वक्र बनाकर प्राइमर की क्षमता का मूल्यांकन किया गया। इस आंकड़े के एक बड़े संस्करण को देखने के लिए कृपया यहां क्लिक करें

चित्रा 3: क्लोरोप्लास्ट और मिटोकॉन्ड्रियल जीनोम के मैपिंग और सैद्धांतिक कवरेज पढ़ें। मिटोकॉन्ड्रियल ( ए ) या क्लोरोप्लास्ट ( बी ) चीनी स्प्रिंग संदर्भ जेनोम को मैप किए जाने वाले प्रतिशत का प्रतिशत। चीनी वसंत mitochondrial ( सी ) या क्लोरोप्लास्ट ( डी ) संदर्भ के अनुरूप सैद्धांतिक कवरेजमेस, जो जीनोम के आकार को 450 और 135 केबी मानते हैं, क्रमशः कुल पठन संख्या का उपयोग करते हुए गणना करते हैं और विभिन्न जीनोमों के लिए मानचित्रण पढ़ता है। एमएफ विधि ( ई और जी ) या डीसी पद्धति ( एफ एंड एच ) से ऑर्गेनेल डीएनए के लिए कवरेज का जीनोम-विस्तृत वितरण। पैनल ई - एच में डेटा चीनी स्प्रिंग एटिलाटेड नमूना से है, लेकिन अन्य सभी नमूने एक समान प्रवृत्ति दिखाते हैं। ( I ) पैनल, ए - डी में सभी नमूनों के लिए औसत, सबसे कम, और सर्वोच्च प्रति कवरेज। "ई" नामित नमूनों सहित नमूना लेबल, और "पूर्वोत्तर" गैर-लोचयुक्त नमूनों को निर्दिष्ट करता है। डीसी विभेदक केन्द्रापसारक विधि के साथ पृथक डीएनए को इंगित करता है और अनमाइथिलेटेड डीएनए को इंगित करता है जो एमबीडी 2-एफसी (एमएफ प्रोटोकॉल) के साथ पुलडाउन के बाद अनमाइथिलेटेड अंश में है। नमूने "क्रिस" नामित गेहूं ट्रिटिकम एस्तेविम नामित हैं'क्रिस।' सीएस गेहूं के नमूने को परिभाषित करता है ट्रिटिकम एस्टीवम 'चीनी स्प्रिंग नोट: ऑर्गेनेल जेनोमों के साथ-साथ ऑग्नेलियर और परमाणु जीनोमों के बीच प्राचीन जीन अंतरण के परिणामस्वरूप क्लोरोप्लास्ट, मिटोचोनड्रिया और परमाणु जीनोम के बीच क्रम अनुरूपता के कारण, कच्चे पढ़े जाने का एक छोटा प्रतिशत कई जीनोमों को मैप कर सकता है। इसके अलावा, यह पढ़ा जाता है कि ऑर्गेनेल रेफरेंस जीनोम का नक्शा न करें इस आंकड़े में प्रतिनिधित्व नहीं किया गया है। इसलिए, यहां प्रदर्शित प्रतिशत ( ए और बी ) कुल 100% नहीं हैं इस आंकड़े के एक बड़े संस्करण को देखने के लिए कृपया यहां क्लिक करें

चित्रा 4: पे गे मैंगिंग टू गेट परमाणु जीनोम पीई की श्रेणियों का प्रतिशत मैटोकॉन्ड्रियल (ए) , क्लोरोप्लास्ट (बी) , या परमाणु (सी) चीनी स्प्रिंग संदर्भ जेनोम के मानचित्रण प्रकार पढ़ते हैं। - ई नामित नमूनों को निर्दिष्ट करता है और - एनई गैर-एटिलेटेड नमूनों को निर्दिष्ट करता है। डीसी विभेदक केन्द्रित विधि के साथ अलग डीएनए को इंगित करता है, अनमाइथिलेटेड एमएनडी प्रोटोकॉल में एमबीडी 2-एफसी के साथ पुलडाउन के बाद अनमाइथिलेटेड अंश में डीएनए को इंगित करता है, और मेथिलेटेड एमबीडी 2-एफसी पुलडाउन के बाद परमाणु अंश को निर्दिष्ट करता है। नमूने "क्रिस" नामित गेहूं ट्रिटिकम ऐस्टीवम 'क्रिस।' सीएस ने गेहूं के नमूने को नामित किया है ट्रिटिकम सस्टेवम 'चीनी स्प्रिंग।' अनमेप किए गए पढ़े नहीं दिखाए जाते हैं इस आंकड़े के एक बड़े संस्करण को देखने के लिए कृपया यहां क्लिक करें
ओएड / 55528 / 55528fig5.jpg "/>
चित्रा 5: डीएनए गुणवत्ता का प्रयोग PFGE का उपयोग करना। गेहूं कुल जीनोमिक डीएनए (लेन 2), गेहूं ऑब्जेनेल डीएनए विभेदक केन्द्रण से प्राप्त (लेन 3), और एमबीडी 2-एफसी पुलडाउन दृष्टिकोण (लेन 4) के साथ एमएफ के बाद परमाणु अंश को 1% agarose जेल पर पीएफजी के अधीन किया गया था 1 केबी विस्तारित सीढ़ी का उपयोग मार्कर (लेन 1 और 5) के रूप में किया गया इस आंकड़े के एक बड़े संस्करण को देखने के लिए कृपया यहां क्लिक करें
| बफर का नाम | विधि | टिप्पणियाँ | तरीका |
| एसईई बफर | 400 एमएम सुक्रोज़, 50 एमएम ट्रिएस पीएच 7.8, 20 एमएम ईडीटीए पीएच 8.0, 0.6% (डब्ल्यू / वी) पॉलीविनाइलपीरॉरिओडोन (पीवीपी), 0.2% (वाई / वी) गोजातीय सीरम एल्बूमिन (बीएसए), 0.1% (v / v) β-mercaptoethanol (बीएमई) | केवल सूक्रोज, ट्राइस और ईडीटीए वाले बफर मिश्रण में एक महीने तक अग्रिम रखा जा सकता है और इसे 4 डिग्री सेल्सियस पर रखा जा सकता है। पीवीपी, बीएसए, और बीएमई को इस्तेमाल करने से पहले बफर की आवश्यक राशि के एक विभाज्य को ताज़ा करना चाहिए। | विधि # 1 |
| अनुसूचित जनजाति बफर | 400 एमएम सुक्रोज़, 50 एमएम ट्राइस पीएच 7.8, 0.6% (डब्ल्यू / वी) पॉलीविनैलीप्रोलीओडोन (पीवीपी), 0.1% (डब्ल्यू / वी) गोजातीय सीरम एल्बूमिन (बीएसए) | केवल सूक्रोज युक्त बफर मिश्रण और ट्राइस को एक महीने तक अग्रिम में रखा जा सकता है और इसे 4 डिग्री सेल्सियस पर रखा जा सकता है। ध्यान दें कि अनुसूचित जनजाति बफर में ईडीटीए या बीएमई शामिल नहीं है, और इसमें बीएसए की कम एकाग्रता शामिल है। | विधि # 1 |
| DNase स्टॉक | 2 मिलीग्राम / एमएल के स्टॉक एकाग्रता में 0.15 एम NaCl में 2 मिलीग्राम / एमएल डीएनसे | -20 डिग्री सेल्सियस पर 200 उल अल्कोट्स को स्टोर करें DNase काम कर समाधान (नमूना प्रति DNase समाधान के 200 μl) तैयार करने के लिए देखेंतालिका 1 नीचे। DNase पाचन के पूर्ण विवरण के लिए नीचे दिए गए पूर्ण प्रोटोकॉल देखें। DNase काम कर समाधान ताजा तैयार किया जाना चाहिए DNase प्रतिक्रिया को रोकने के लिए 400 एमएम EDTA पीएच 8.0 समाधान आवश्यक है (प्रतिक्रिया को रोकने के लिए आवश्यक अंतिम एकाग्रता 0.2 एम EDTA है, विवरण के लिए पूर्ण प्रोटोकॉल देखें)। | विधि # 1 |
| DNase काम कर रहे समाधान | एसटी बफर में 0.25 मिलीग्राम / एमएल डीएनसे और 20 मिमी एमजीसीएल 2 | ताजा तैयार करें, प्रति नमूना 200 उल। दिखाया गया सांद्रता अंतिम प्रतिक्रिया मात्रा के लिए है, इसलिए मिश्रण: 62.5 μl 2 मिलीग्राम / मिलीलीटर डीएनसे (अंतिम 500 μl प्रतिक्रिया मात्रा के आधार पर), 4 μl 1M MgCl 2 (200 μl DNase समाधान मात्रा के आधार पर), और 133.5 μl अनुसूचित जनजाति बफर के लिए 200 μl की अंतिम मात्रा | विधि # 1 |
| लिसेस बफ़र | 20 मिमी ईडीटीए पीएच 8.0; 10 मिमी ट्रिएस पीएच 7.9; 500 मिमी Guanidine-HCl; 200 मिमी NaCl; 1% ट्राइटन एक्स -100; 0.5 मिलीग्राम / एमएल लिज़िंग एंजाइम सेट्राइकोडर्मा हर्जियानम | कमरे के तापमान पर लिज़िंग एंजाइम और स्टोर के अलावा सभी अवयवों को मिलाएं। तत्काल उपयोग के लिए लेशिंग एंजाइम्स को एक छोटे से विभाज्य के लिए ताज़ा जोड़ा जाना चाहिए। | विधि # 2 |
तालिका 1: होममेड बफ़र्स और कामकाजी शेयरों की व्यंजन
| एकाग्रता वर्कशीट | |||||||
| नमूना नाम | खाली डिवाइस वजन (जी) | भरा डिवाइस का भार (जी) | भरा हुआ वॉल्यूम (उल, भरा खाली वजन) | 1 स्पिन के बाद वजन (20 मिनट *, छ) | 1 स्पिन के बाद वॉल्यूम (उल, भरेशून्य से खाली वजन) | दूसरे स्पिन के बाद वजन (एक्स मिन *, जी) | दूसरे स्पिन के बाद का वॉल्यूम (उल, से भरा खाली वजन) |
| ध्यान दें कि वास्तविक बरामद किए गए वॉल्यूम की गणना वॉल्यूम से कम होगी। | |||||||
तालिका 2: एकाग्रता वर्कशीट
| नाम | जीनोम विशिष्टता | जीन अनुक्रम स्रोत | अनुक्रम (5 '- 3') |
| टा_एक्टिन - एफ | नाभिकीय | ग्रामीन स्कैफोल्ड IWGSC_CSS_1AS_scaff_3272162: 10,663-12,557 | CAGGTATCGCTGACCGTATGA |
| Ta_ACटीआईएन - आर | नाभिकीय | ऊपर की तरह | GAAGGTAGGGCTGAACAAGAAAC |
| Ta_NAD3 - एफ | mitochondrial | एनसीबीआई अभिग्रहण EU534409.1 | GGTGATGCCAGAAGTCGTTT |
| Ta_NAD3 - आर | mitochondrial | ऊपर की तरह | CAGATCAATCTTGTTAGGAGGTACTG |
| टै_पीएसबीबी - एफ | क्लोरोप्लास्ट | एनसीबीआई परिग्रहण केजे 592713.1 | GCTACCTTTGCTTTGCTCTTCT |
| टा_पीएसबीबी - आर | क्लोरोप्लास्ट | ऊपर की तरह | GCTGCCTGTTTCCTTGTAGTT |
तालिका 3: qPCR प्राइमर की सूची
Subscription Required. Please recommend JoVE to your librarian.
Discussion
तिथि करने के लिए, विशिष्ट डीएनए के लिए समृद्ध करने के लिए परंपरागत डीसी पद्धतियों पर अधिकांश ऑग्नलर सिकेंजिंग अध्ययन केंद्र। विभिन्न पौधों से ऑर्गेनल्स को अलग करने के तरीके का वर्णन किया गया है, जिसमें मॉस 40 ; मोनोकॉट जैसे गेहूं 15 और जई 11 ; और डिकोट्स जैसे कि अरबिडोप्सिस 11 , सूरजमुखी 17 और रेपसीड 14 । ज्यादातर प्रोटोकॉल 13 , 14 , 15 , 16 , 17 के पत्तों के ऊतकों पर ध्यान केंद्रित करते हैं, जिनमें से कुछ को ऊतक प्रकार के 11 प्रकार के बीज के लिए अनुकूलित किया गया है। प्रोटॉप्लेट से ऑर्गेनल्स का अलगाव भी प्रदर्शित किया गया है 41 । हालांकि, यह सभी प्रणालियों के लिए अनुकूल नहीं है, न ही यह संभव है जब ब्याज की ऊतक सीमित है। इनमें से कई यागानेलार अलगाव के तरीकों को विशिष्ट प्रयोगों जैसे कि शारीरिक अध्ययनों के लिए अक्षत organelles को पुनर्प्राप्त करने के लिए डिजाइन किए गए थे। ये प्रोटोकॉल बोझिल होते हैं और आमतौर पर सुक्रोज या पर्सोल ग्रैडिएंस जैसे घनत्व के ढाल के उपयोग की आवश्यकता होती है, जो विशिष्ट ऑग्नेल आंशिकताओं को अलग करने में बहुत ही कुशल होते हैं, लेकिन बड़े टिशू इनपुट ( अर्थात, 5 ग्रा से ऊपर और किलोग्राम के ऊपर, इसके आधार पर ऊतक प्रकार) हालांकि, डीसी पद्धति स्पिन गति और घनत्व ग्रेडियेंट्स को बदलकर, विशिष्ट सेलुलर अंशों जैसे मीइटोकोंड्रिया या क्लोरोप्लास्ट के लिए समृद्ध करने के लिए अनुकूलित हो सकती है। इसके विपरीत, एमएफ दृष्टिकोण के लिए कम से कम प्रारंभिक सामग्री (20 मिलीग्राम) की आवश्यकता होती है, लेकिन डीआईएन निष्कर्षण के लिए उपयोग किए जाने वाले ऊतक में उनके रिश्तेदार बहुतायत के अनुसार मिटोचोनड्रियल और प्लास्टिड डीएनए मौजूद रहेंगे। फिर भी, एमएफ प्रोटोकॉल मिश्रित ऑर्गेनेल डीएनए को अलग करने के लिए एक वैकल्पिक दृष्टिकोण प्रदान करता है और विशेष रूप से ऊतक की थोड़ी मात्रा से शुरू करने के लिए फायदेमंद है।
टी ओग्नेएल अलगाव के बाद नमूना शुद्धता का आकलन करें, ज्यादातर अध्ययनों की समाप्ति केवल अंत-प्वाइंट पीसीआर और जेल वैद्युतकणसंचलन 11 , 12 का उपयोग करती है । यह नमूना शुद्धता का एक उचित गुणात्मक उपाय देता है। हालांकि, एग्रोसेज जेल पर प्रवर्धन का निम्न स्तर नहीं देखा जा सकता है। कुछ रिपोर्टों में गुणवत्ता नियंत्रण के अधिक मात्रात्मक उपाय शामिल हैं, जैसे qPCR 14 दोनों तरीकों से अलग डीएनए नमूना शुद्धता के मात्रात्मक आकलन के लिए, हमने क्वार्पोसीआर और अनुक्रमण का उपयोग किया है कि यह निर्धारित करने के लिए कि कितना परमाणु डीएनए नमूना में रहता है, साथ ही मिटोचोनड्रीअल बनाम क्लोरोप्लास्ट डीएनए के सापेक्ष अनुपात भी। यहां मूल्यांकन किए गए दोनों विधियां, परमाणु डीएनए को हटाने में सक्षम हैं। दोनों तरीकों में मिटोकोन्ड्रियल और क्लोरोप्लास्ट डीएनए का मिश्रण होता है, हालांकि अलग-अलग अनुपात पर।
अंधेरे में पौधों को बढ़ाना (फेरबदल) को फेनोलिक्स की कमी के कारण ऑर्गेनेल अलगाव को सुगम बनाने में मदद करने के लिए सूचित किया गया है13। हालांकि, इस तुलना में, हमें हल्के उगने वाले नमूनों पर एटियलैटेड ऊतक के साथ काम करने के लिए एक सम्माननीय लाभ नहीं मिला.हालांकि, विशेष रूप से क्लोरोप्लास्ट का अनुपात अधिक हो जाएगा, जब हल्के उगले, कुल प्लास्टर संख्या क्लोरोप्लास्ट जीनोम के मानचित्रण को पढ़ने के अनुपात में परिलक्षित होता है, भिन्न प्रकाश स्थितियों के तहत अपरिवर्तित होता है। इसलिए, अलग-अलग ऊतकों में या अलग-अलग तनाव में या अभिव्यक्ति के विश्लेषण के लिए ऊतक के कार्यात्मक विश्लेषण के मूल्यांकन के लिए, हम अनुशंसा करते हैं कि जीनोमिक अनुक्रमण शारीरिक रूप से संबंधित शर्तों के तहत उगने वाले पौधे
शॉर्ट-पढें अनुक्रमण तकनीकों के साथ आवेदन के लिए, यहां की तुलना में दोनों तकनीक पर्याप्त डीएनए मात्रा और गुणवत्ता प्राप्त करते हैं। हालांकि, सिंगल अणु अनुक्रमण अनुप्रयोगों के लिए 20 केबी के लंबे समय से पढ़ता है, उच्च गुणवत्ता वाले डीएनए की अधिक मात्रा आवश्यक है। उदाहरण के लिए, आदर्श रूप से,> 1 μg शुद्ध orgaएक आणविक भार के साथ नालेदार गेहूं डीएनए> 20 केबी की लाइब्रेरी की तैयारी 42 के लिए घर में, कम इनपुट प्रोटोकॉल के लिए 20 केबी आवश्यक है। नए उपयोगकर्ता-विकसित, कम-इनपुट प्रोटोकॉल डीएनए आवश्यकताओं को कम कर सकते हैं ( यानी, 50 एनजी या उससे भी कम 20 ), लेकिन चुनौती के लिए उच्च गुणवत्ता वाला, उच्च आणविक भार डीएनए पुस्तकालय की तैयारी में बनी हुई है। यह आवश्यक है कि ज्यादातर डीएनए> 20 केबी है, क्योंकि छोटे टुकड़े को प्राथमिकता से एसएमआरटीबेल में डाला जाएगा और लाइब्रेरी 43 का आकार वितरण बंद कर दिया जाएगा। हमने डीएनए निष्कर्षण के लिए कई घरेलू डीएनए निष्कर्षण प्रोटोकॉल और कई व्यावसायिक प्रोटोकॉल की कोशिश की है (न दिखाया गया है)। गेहूं के पत्तों के ऊतक के लिए, डीएनए मात्रा और गुणवत्ता के बीच का सबसे अच्छा संतुलन, विशेष रूप से लंबाई, एक व्यावसायिक किट 27 , 29 का उपयोग करके प्राप्त किया गया था। पौधे की प्रजातियों और ब्याज की ऊतक पर निर्भर करता है, वैकल्पिकतानिकाले प्रोटोकॉल समान रूप से अनुकूल या अधिक उपयोगी हो सकते हैं। इसके बावजूद, हम यह निष्कर्ष निकालते हैं कि उच्च आणविक वजन जीनोमिक डीएनए> 50 केबी आकार में एमबीडी 2-एफसी पुलडाउन दृष्टिकोण 28 के साथ विभाजन के बाद, सीमित प्रारंभिक सामग्री से लंबे समय तक पढ़े जाने वाले अनुक्रमण के लिए अनुकूल है। भविष्य के कार्य को लंबे समय से सम्मिलित लाइब्रेरी की तैयारी और बाद में लंबे समय से पढ़े जाने वाले अनुक्रमण के लिए विभाजन के बाद आवश्यक प्रारंभिक सामग्री की सीमाओं का परीक्षण करना चाहिए। क्रिटिक रूप से, इस दृष्टिकोण से पूरे जीनोम प्रवर्धन के बिना, लंबे पठन अनुक्रमण के लिए उपयुक्त एकल पत्ती के एक subsample से डीएनए को अलग करने का एक मजबूत तरीका प्रदान किया जा सकता है। हम आशा करते हैं कि यह दृष्टिकोण अतिरिक्त ऊतक प्रकारों के लिए आसानी से अनुकूल होगा और अन्य पौधों की प्रजातियों के लिए मोटे तौर पर लागू होगा। यह उन परिस्थितियों में विशेष रूप से उपयोगी होगा जहां ऊतक की मात्रा सीमित हो रही है, जैसे कि एक क्रॉसिंग योजना में अलग-अलग पीढ़ियों के क्रम में या दुर्लभ टिशू प्रकार में।
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
लेखक घोषणा करते हैं कि उनकी कोई प्रतियोगी रुचि नहीं है।
इस प्रकाशन में व्यापार के नाम या वाणिज्यिक उत्पादों का उल्लेख केवल विशिष्ट जानकारी प्रदान करने के उद्देश्य के लिए है और अमेरिकी कृषि विभाग द्वारा सिफारिश या अनुमोदन नहीं दर्शाता है। यूएसडीए एक समान अवसर प्रदाता और नियोक्ता है।
Acknowledgments
हम कृषि-कृषि अनुसंधान सेवा के संयुक्त राज्य विभाग से और राष्ट्रीय विज्ञान फाउंडेशन (आईओएस 1025881 और आईओएस 1361554) से धन को स्वीकार करना चाहते हैं। हम ग्रीनहाउस रखरखाव और पौधे देखभाल के लिए आर। कैस्पर का धन्यवाद करते हैं। हम मिनेसोटा यूनिवर्सिटी ऑफ यूनिवर्सिटी सेंटर का भी धन्यवाद करते हैं, जहां इलुमिना पुस्तकालय की तैयारी और अनुक्रमण की गई थी। हम पत्रिका के संपादकों और चार अज्ञात समीक्षकों की टिप्पणियों के लिए भी आभारी हैं जिन्होंने हमारी पांडुलिपि को और मजबूत किया। जापान में सहकर्मियों के साथ सहयोगी परियोजनाओं के लिए इन प्रोटोकॉल को एकीकृत करने के लिए हम एसके से फेलोशिप के लिए ओईसीडी का भी धन्यवाद करते हैं।
Materials
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol (beta-mercaptoethanol; BME) | Sigma Aldrich | M3148-100ml | |
| 2-propanol (Isopropyl alcohol/isopropanol), bioreagent | Sigma Aldrich | I9516 | |
| agarose, Bio-Rad Cetified Megabase agarose | Bio-Rad | 1613108 | |
| analytical balance | Mettler Toledo | AB54-S | |
| balance | Mettler Toledo | PB1502-S | |
| bovine serum albumin (BSA) | Sigma Aldrich | B4287-25G | |
| Ceramic grinding cylinders, 3/8in x 7/8in | SPEX SamplePrep | 2183 | |
| Cryogenic Blocks compatible with tissue homogenizer for holding 50 mL tubes | SPEX SamplePrep | 2664 | |
| DNaseI | Sigma | DN25 | |
| ethanol, absolute | Decon Laboratories | 2716 | |
| Ethylenediamine Tetraacetic Acid (EDTA), 0.5 M Solution, pH 8.0 | Fisher | BP2482-500 | |
| gel imaging system | |||
| gel stain | Such as GelRed or Ethidium Bromide | ||
| grinding pestle, wide tip for 2 mL conical tubes | |||
| Guanidine-HCl, 8 M solution | ThermoFisher | 24115 | |
| LightCycler 480 SYBR Green I Master | Roche | 4707516001 | |
| liquid nitrogen | |||
| Lysing enzymes from Trichoderma harzianum | Sigma | L1412 | |
| Magnesium Chloride | G Bioscience | 24115 | |
| magnetic rack | ThermoFisher | A13346 | |
| microcentrifuge tubes, LoBind 1.5 mL | Eppendorf | 22431021 | |
| microcentrifuge tubes, standard nuclease-free 1.5 mL | Eppendorf | ||
| microcentrifuge, refrigerated | Sorvall | Legend X1R | Or equivalent product, must be capable of reaching at least 18,000 x g with rotors for 50 mL tubes, Oak Ridge tubes, and 1.5 mL tubes |
| microcentrifuge, room temperature | Eppendorf | 5424 | Or equivalent product, must be capable of reaching at least 18,000 x g with rotor for 1.5 mL and 2 mL microcentrifuge tubes |
| Microcon DNA Fast Flow Centrifugal Filter Units | EMD Millipore | MRCFOR100 | |
| Miracloth, 1 square per sample cut to fit funnel | EMD Millipore | 475855 | |
| NEBNext Microbiome DNA Enrichment Kit | New England Biolabs | E2612L | |
| parafilm | Parafilm M | PM992 | |
| plastic pots and trays | |||
| polyvinylpyrrolidone (PVP) | Fisher | BP431-100 | |
| Proteinase K | Qiagen | 19131 | |
| Pulsed-Field Gel Electrophoresis rig (e.g. CHEF DR III) | Bio-Rad | 1703697 | |
| purification beads, Agencourt AMpureXP beads | Beckman Coulter | A63881 | |
| QIAamp DNA Mini Kit | Qiagen | 51304 | |
| Qiagen 20/g Genomic Tip DNA Extraction Kit | Qiagen | 10223 | |
| Qiagen Buffer EB (elution buffer) | Qiagen | 19086 | |
| Qiagen DNA Extraction Buffer Set | Qiagen | 19060 | |
| QiaRack | Qiagen | 19015 | |
| qPCR machine (e.g. Roche Light Cycler 480) | Roche | ||
| qPCR plate sealing film | Roche | 4729757001 | |
| qPCR plate, 96 well plate | Roche | 4729692001 | |
| Qubit assay tubes | Life Technologies | Q32856 | |
| Qubit Broad Spectrum assay kit | Life Technologies | Q32850 | |
| Qubit High Sensitivity assay kit | Life Technologies | Q32851 | |
| RNaseA | Qiagen | 19101 | |
| Serological pipettes (20 mL) and pipet-aid | Fisher | 13-678-11 | |
| Small funnels, 1 per sample | |||
| Sodium Chloride | Ambion | AM9759 | |
| Soft paintbrush, 2 per sample | |||
| SPEX SamplePrep 2010 Geno/Grinder or another type of tissue homogenizer | SPEX SamplePrep | Or another comparable tissue homogenizer. If you do not have access to a tissue homogenizer, then grinding in a pre-chilled mortar and pestle will suffice (see protocol for details). However, a homogenizer will give more consistent results and total homogenization time is reduced. | |
| Sucrose | Omnipure | 8550 | |
| TBE | |||
| thermomixer | |||
| Tris | Sigma | T2819-100ml | |
| Triton X-100 | Promega | H5142 | |
| tube rotater | |||
| tubes, 50 mL conical polypropylene | Corning | 352070 | |
| tubes, 50 mL high-speed polypropylene | ThermoScientific/Nalgene | 3119-0050 | e.g. Nalgene Oakridge tubes or equivalent |
| vermiculite | |||
| water bath | |||
| water, sterile and certified Nuclease-free | Fisher | 1481 | |
| water, sterile milliQ |
References
- Liberatore, K. L., Dukowic-Schulze, S., Miller, M. E., Chen, C., Kianian, S. F. The role of mitochondria in plant development and stress tolerance. Free Radic Biol Med. 100, 238-256 (2016).
- Samaniego Castruita, J. A., Zepeda Mendoza, M. L., Barnett, R., Wales, N., Gilbert, M. T. Odintifier--A computational method for identifying insertions of organellar origin from modern and ancient high-throughput sequencing data based on haplotype phasing. BMC Bioinformatics. 16 (232), 1-13 (2015).
- Zhang, T., Zhang, X., Hu, S., Yu, J. An efficient procedure for plant organellar genome assembly, based on whole genome data from the 454 GS FLX sequencing platform. Plant Methods. 7 (38), 1-8 (2011).
- Wambugu, P. W., Brozynska, M., Furtado, A., Waters, D. L., Henry, R. J. Relationships of wild and domesticated rices (Oryza AA genome species) based upon whole chloroplast genome sequences. Sci Rep. 5 (13957), 1-9 (2015).
- Iorizzo, M., et al. De novo assembly of the carrot mitochondrial genome using next generation sequencing of whole genomic DNA provides first evidence of DNA transfer into an angiosperm plastid genome. BMC Plant Biol. 12 (61), 1-17 (2012).
- Park, S., et al. Complete sequences of organelle genomes from the medicinal plant Rhazya stricta (Apocynaceae) and contrasting patterns of mitochondrial genome evolution across asterids. BMC Genomics. 15 (405), 1-18 (2014).
- Skippington, E., Barkman, T. J., Rice, D. W., Palmer, J. D. Miniaturized mitogenome of the parasitic plant Viscum scurruloideum is extremely divergent and dynamic and has lost all nad genes. Proc Natl Acad Sci U S A. 112 (27), E3515-E3524 (2015).
- Wicke, S., Schneeweiss, G. M. Chapter 1. Next Generation Sequencing in Plant Systematics. Hörandl, E., Appelhans, M. , Koeltz Scientific Books. (2015).
- Sloan, D. B. One ring to rule them all? Genome sequencing provides new insights into the 'master circle' model of plant mitochondrial DNA structure. New Phytol. 200 (4), 978-985 (2013).
- Woloszynska, M. Heteroplasmy and stoichiometric complexity of plant mitochondrial genomes--though this be madness, yet there's method in't. J Exp Bot. 61 (3), 657-671 (2010).
- Ahmed, Z., Fu, Y. B. An improved method with a wider applicability to isolate plant mitochondria for mtDNA extraction. Plant Methods. 11 (56), 1-11 (2015).
- Ejaz, M., et al. Comparison of small scale methods for the rapid and efficient extraction of mitochondrial DNA from wheat crop suitable for down-stream processes. Genet Mol Res. 13 (4), 10320-10331 (2014).
- Eubel, H., Heazlewood, J. L., Millar, A. H. Isolation and subfractionation of plant mitochondria for proteomic analysis. Methods Mol Biol. 355, 49-62 (2007).
- Hao, W., Fan, S., Hua, W., Wang, H. Effective extraction and assembly methods for simultaneously obtaining plastid and mitochondrial genomes. PLoS One. 9 (9), e108291 (2014).
- Pomeroy, M. K. Studies on the respiratory properties of mitochondria isolated from developing winter wheat seedlings. Plant Physiol. 53 (4), 653-657 (1974).
- Taylor, N. L., Stroher, E., Millar, A. H. Arabidopsis organelle isolation and characterization. Methods Mol Biol. 1062, 551-572 (2014).
- Triboush, S. O., Danilenko, N. G., Davydenko, O. G. A method for isolation of chloroplast DNA and mitochondrial DNA from Sunflower. Plant Mol Biol Rep. 16 (2), 183-189 (1998).
- Pinard, R., et al. Assessment of whole genome amplification-induced bias through high-throughput, massively parallel whole genome sequencing. BMC Genomics. 7 (216), 1-21 (2006).
- Lamble, S., et al. Improved workflows for high throughput library preparation using the transposome-based Nextera system. BMC Biotechnol. 13 (104), 1-10 (2013).
- Raley, C., et al. Preparation of next-generation DNA sequencing libraries from ultra-low amounts of input DNA: Application to single-molecule, real-time (SMRT) sequencing on the Pacific Biosciences RS II. bioRxiv. , (2014).
- Tsai, Y. C., et al. Resolving the Complexity of Human Skin Metagenomes Using Single-Molecule Sequencing. MBio. 7 (1), e01948 (2016).
- Feehery, G. R., et al. A method for selectively enriching microbial DNA from contaminating vertebrate host DNA. PLoS One. 8 (10), e76096 (2013).
- Yigit, E., Hernandez, D. I., Trujillo, J. T., Dimalanta, E., Bailey, C. D. Genome and metagenome sequencing: Using the human methyl-binding domain to partition genomic DNA derived from plant tissues. Appl Plant Sci. 2 (11), 1-6 (2014).
- Noyszewski, A. K., et al. Accelerated evolution of the mitochondrial genome in an alloplasmic line of durum wheat. BMC Genomics. 15 (67), 1-16 (2014).
- Qiagen. QIAamp DNA Mini and Blood Mini Handbook. , 5th ed, Available from: https://www.qiagen.com/ch/resources/ (2016).
- E.M. Corporation. User Guide: Microcon Centrifugal Filter Devices. , Available from: http://www.emdmillipore.com/US/en/product/Microcon-DNA-Fast-Flow-Centrifugal-Filter-Unit-with-Ultracel-membrane,MM_NF-MRCF0R100 (2013).
- Qiagen. User developed protocol: Isolation of genomic DNA from plants and filamentous fungi using the QIAGEN Genomic-tip - (EN). , Available from: https://www.qiagen.com/ch/resources/ (2001).
- New England BioLabs, Inc.. NEBNext Microbiome DNA Enrichment Kit: Instruction Manual Version 4.0. , Available from: http://www.neb.com/~/media/Catalog/All-Products/371BCB5A557C462D95D1E45E15BBFEA3/Datacards or Manuals/E2612Manual.pdf (2015).
- Qiagen. QIAGEN Genomic DNA Handbook. , Available from: https://www.qiagen.com/ch/resources/ (2012).
- PacificBiosciences. Guidelines for Using the BIO-RAD® CHEF Mapper® XA Pulsed Field Electrophoresis System. , Available from: http://www.pacb.com/wp-content/uploads/Unsupported-Guidelines-Using-BIO-RAD-CHEFMapper-XA-Pulsed-Field-Electrophoresis.pdf (2016).
- Andrews, S. FastQC: A quality control tool for high throughput sequence data. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Ogihara, Y., et al. Structural dynamics of cereal mitochondrial genomes as revealed by complete nucleotide sequencing of the wheat mitochondrial genome. Nucleic Acids Res. 33 (19), 6235-6250 (2005).
- Ogihara, Y., et al. Structural features of a wheat plastome as revealed by complete sequencing of chloroplast DNA. Mol Genet Genomics. 266 (5), 740-746 (2002).
- International Wheat Genome Sequencing Consortium (IWGSC). A chromosome-based draft sequence of the hexaploid bread wheat (Triticum aestivum) genome. Science. 345 (6194), (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nat Methods. 9 (4), 357-359 (2012).
- Bendich, A. J. Why do chloroplasts and mitochondria contain so many copies of their genome? Bioessays. 6 (6), 279-282 (1987).
- Kumar, R. A., Oldenburg, D. J., Bendich, A. J. Changes in DNA damage, molecular integrity, and copy number for plastid DNA and mitochondrial DNA during maize development. J Exp Bot. 65 (22), 6425-6439 (2014).
- Ma, J., Li, X. Q. Organellar genome copy number variation and integrity during moderate maturation of roots and leaves of maize seedlings. Curr Genet. 61 (4), 591-600 (2015).
- Lang, E. G., et al. Simultaneous isolation of pure and intact chloroplasts and mitochondria from moss as the basis for sub-cellular proteomics. Plant Cell Rep. 30 (2), 205-215 (2011).
- Tobin, A. K. Subcellular fractionation of plant tissues. Isolation of chloroplasts and mitochondria from leaves. Methods Mol Biol. 59, 57-68 (1996).
- PacificBiosciences. Procedure & Checklist - 10 kb to 20 kb Template Preparation and Sequencing with Low (100 ng) Input DNA. , Available from: http://www.pacb.com/wp-content/uploads/Procedure-Checklist-10-20kb-Template-Preparation-and-Sequencing-with-Low-Input-DNA.pdf (2015).
- PacificBiosciences. Template Preparation and Sequencing Guide. , Available from: http://www.pacb.com/wp-content/uploads/2015/09/Guide-Pacific-Biosciences-Template-Preparation-and-Sequencing.pdf (2014).



