Summary
ההשוואה ואופטימיזציה של שתי שיטות העשרה DNA DNA organellar צמח מוצגים: צנטריפוגה ההפרש הדיפרנציאלי ואת החלוקה של gDNA הכולל מבוסס על מצב מתילציה. אנו מעריכים את כמות הדנ"א וכתוצאה מכך, מדגימים ביצועים ברצף של הדור הבא, וקוראים את הפוטנציאל לשימוש ברצף המולקולה החד-פעמית.
Abstract
גנומים צמחיים מכילים רכיבים גדולים וחוזרים על עצמם, שעשויים לעבור זיווג או רקומבינציה ליצירת מבנים מורכבים ו / או קטעים גנומיים. גנומים של Organellar קיימים גם בתוספות בתוך תא נתון או סוג רקמות (heteroplasmy), ושפע של תת-סוגים עשויים להשתנות במהלך הפיתוח או כאשר נמצאים תחת לחץ (תת-סטויצ'יומטרי). הדור הבא רצף (NGS) טכנולוגיות נדרשים כדי להשיג הבנה עמוקה יותר של מבנה הגנום organellar ותפקוד. מחקרי רצף מסורתיים משתמשים במספר שיטות כדי להשיג DNA DNA: (1) אם נעשה שימוש בכמות גדולה של רקמה מתחילה, הוא הומוגני ונתון לצנטריפוגה דיפרנציאלית ו / או לטיהור הדרגתי. (2) אם נעשה שימוש בכמות קטנה יותר של רקמות ( כלומר, אם הזרעים, החומר או החלל מוגבלים), אותו תהליך מבוצע כמו (1), ולאחר מכן הגברה של הגנום כולו כדי להשיג מספיק DNA. (3) ניתוח ביואינפורמטיקה ניתן להשתמש כדי seqאת הדנ"א הגנומי הכולל ולנתח את הקריאה. לכל השיטות הללו יש אתגרים ופריצות. ב (1), זה יכול להיות קשה להשיג כזה כמות גדולה של רקמת החל; ב (2), הגברה הגנום כולו יכול להציג הטיה רצף; (3), הומולוגיה בין גנומים גרעיניים ו organellar יכול להפריע הרכבה וניתוח. במפעלים עם גנומים גרעיניים גדולים, זה יתרון להעשיר עבור DNA organellar כדי להפחית את עלויות הרצף ואת המורכבות רצף עבור ניתוחים ביואינפורמטיקה. כאן, אנו משווים שיטת צנטריפוגה דיפרנציאלית מסורתית בשיטה הרביעית, שיטה מתואמת CpG-methyl הנפתחת, כדי להפריד בין ה- DNA הגנומי הכולל לשברים גרעיניים ואורגניים. שתי השיטות מניבות די-אן-אי מספיק ל- NGS, DNA המועשר מאוד עבור רצפים אורגניים, אם כי ביחסים שונים במיטוכונדריה ובצ'לורופלסטים. אנו מציגים את האופטימיזציה של שיטות אלה עבור רקמות עלה חיטה ולדון היתרונות העיקריים דחסרונות של כל גישה בהקשר של קלט המדגם, קלות הפרוטוקול, ואת היישום במורד הזרם.
Introduction
רצף הגנום הוא כלי רב עוצמה לנתח את הבסיס הגנטי הבסיסי של תכונות המפעל חשוב. רוב מחקרי הגנום-רצף מתמקדים בתוכן הגנום הגרעיני, שכן רוב הגנים נמצאים בגרעין. עם זאת, גנומים אורגנואר, כולל המיטוכונדריה (על פני eukaryotes) ופלסטידים (בצמחים, הצורה המקצועית, הכלורופלסט, עובד בפוטוסינתזה) לתרום מידע גנטי משמעותי חיוני לפיתוח אורגניזמים, תגובת מתח, ואת הכושר הכללי 1 . גנומים של Organellar כלולים בדרך כלל בחריצות דנ"א הכוללות רצף גנומי גרעיני, למרות ששיטות להפחתת מספר האורגנים לפני החילוץ של דנ"א מועסקים גם 2 . מחקרים רבים השתמשו תוצאות רצף מתוך סך gDNA חילוץ להרכיב genelles organellar 3 , 4 , 5 ,Xref "> 6 , 7. עם זאת, כאשר המטרה של המחקר היא להתמקד genomes organellar, באמצעות gDNA הכולל מגדילה את עלויות רצף כי רבים קורא" אבודים "רצפי DNA גרעיני, במיוחד צמחים עם גנומים גרעיניים גדולים יתר על כן, בשל שכפול והעברת רצפים organellar לתוך הגנום הגרעיני בין האברונים, פתרון המיקום הנכון מיפוי של רצף קורא לגנום הנכון הוא מאתגר ביואינפורמטית 2 , 8. טיהור genomes organellar מן הגנום הגרעיני הוא אחד אסטרטגיות ביואינפורמטיקה נוספות ניתן להשתמש כדי להפריד קורא את המפה לאזורים של הומולוגיה בין המיטוכונדריה ו chloroplasts.
בעוד גנומים organellar ממין צמחים רבים כבר רצף, מעט ידוע על רוחב של מגוון גנום organellarזמין באוכלוסיות בר או בבריכות גידול מעובדות. גנומים Organellar ידועים גם להיות מולקולות דינמיות שעוברות סידור מבניים משמעותי עקב רקומבינציה בין רצפים חוזרים 9 . יתר על כן, עותקים מרובים של הגנום organellar כלולים בתוך כל אברון, ואת האברונים מרובים נמצאים בתוך כל תא. לא כל העותקים של גנומים אלה זהים, הידועה בשם הטרופלסמי. בניגוד לתמונה הקאנונית של "חוגי אב", יש כיום ראיות גוברות לתמונה מורכבת יותר של מבני גנום, כולל חוגים תת-גנומיים, כרומוזומים ליניאריים, קונקאמרים לינאריים ומבנים מסועפים 10 . ההרכבה של הגנום צמח organellar הוא מסובך עוד יותר על ידי גדלים גדולים יחסית שלהם חוזר משמעותי הפוך ישיר.
פרוטוקולים מסורתיים לבידוד organellar, טיהור דנ"א, ואת הגנום הבאים רצף דואר הם לעתים קרובות מסורבל ודורשים כמויות גדולות של קלט רקמות, עם כמה גרם כלפי מעלה של מאות גרם של רקמת עלה צעיר צורך כנקודת התחלה 11 , 12 , 13 , 14 , 15 , 16 , 17 . זה עושה רצף הגנום organellar נגיש כאשר הרקמה מוגבלת. במצבים מסוימים, כמויות זרע מוגבלות, כגון כאשר יש צורך רצף על בסיס דורי או בקווים סטריליים זכרים כי צריך להישמר דרך חציית. במצבים אלה, DNA יכול להיות מטוהרים ולאחר מכן חשוף הגברה של הגנום כולו. עם זאת, הגברה שלמה הגנום יכול להציג הטיה רציף רצף, אשר היא בעיה מסוימת בעת הערכת וריאציה מבנית, מבנים גנומיים, ורמות heteroplasmy22 ההתקדמות האחרונה בהכנה לספריה לטווחים קצרים לקריאה של טכנולוגיות יש להתגבר על חסמי כניסה נמוכים כדי למנוע הגברה של הגנום כולו. לדוגמה, ערכת הכנה של ספריית NXTERA XT של Illumina XT מאפשרת מעט כמו 1 ng של DNA שישמש כקלט 19 . עם זאת, ההכנות הספריות סטנדרטיים עבור יישומים רצף לקרוא ארוך, כגון PacBio או אוקספורד Nanopore טכנולוגיות רצף, עדיין דורשים כמות גבוהה יחסית של ה- DNA קלט, אשר יכול להוות אתגר עבור רצף הגנום organellar. לאחרונה, חדש שפותח על ידי המשתמש, ארוך לקרוא פרוטוקולי רצף פותחו כדי להפחית את כמויות קלט כדי לעזור להקל על רצף הגנום בדגימות שבו קבלת מיקרוגרם כמויות של DNA קשה 20 , 21 . עם זאת, קבלת משקל מולקולרי גבוה, שברים organellar טהור להאכיל את ההכנות הספרייה הזאת נותרה אתגר.
חיפשנו לאO להשוות לייעל DNA העשרה organelar ושיטות בידוד מתאים NGS ללא צורך הגברה של הגנום כולו. באופן ספציפי, המטרה שלנו היתה לקבוע את שיטות העבודה המומלצות להעשרת עבור DNA גבוהה מולקולרית משקל organellar מחומרים החל מוגבל, כגון תת המדגם של עלה. עבודה זו מציגה ניתוח השוואתי של שיטות להעשיר עבור DNA organellar: (1) פרוטוקול צנטריפוגה דיפרנציאלית מסורתית שונה לעומת (2) פרוטוקול חלוקה DNA מבוסס על השימוש של דנ"א זמין מסחרית CpG-methyl- מחייב תחום גישה הנפתח חלבון 22 הוחל על רקמת הצמח 23 . אנו ממליצים על שיטות עבודה מומלצות לבידוד דנ"א אורגני מחיידק עלה חיטה, אשר ניתן להרחיב בקלות צמחים אחרים ורקמות סוגים.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. הדור של חומרים צמחיים בידוד Organellar ו - DNA החילוץ
- גידול סטנדרטי של שתילי חיטה
- זרעי הצמח vermiculite קטן, מרובע סירים עם 4 - 6 זרעים לפינה. העברה לחממה או תא צמיחה עם מחזור אור 16 שעות, 23 מעלות צלזיוס יום / 18 מעלות צלזיוס.
- להשקות את הצמחים בכל יום. לדשן את הצמחים עם כפית ¼ של דשן 20-20-20 NPK גרעיני על נביטה ב 7 ימים לאחר נביטה.
- אלטרנטיבי אלטרנטיבי של שתילי חיטה
- בצע את שלב 1.1, אבל במקום את הסירים בתא צמיחה כהה, 23 מעלות צלזיוס במשך 16 שעות / 18 מעלות צלזיוס במשך 8 שעות. לחלופין, לכסות את הצמחים בחממה ( למשל, עם מיכל אחסון, עם זאת, אוורור ראוי חייב להישמר).
- גידול וגידול רקמות
- לגדל את הצמחים עבור 12 - 14 ימים. עבור רוב גנוטיפ חיטהS, 75-100 שתילים תשואה סביב 10 - 12 גרם של רקמה, אשר מספיקה עבור שני extractellar לחלץ בשיטת צנטריפוגה ההפרש (סעיף 2); רק מפעל אחד הוא הכרחי אם ניצול דנ"א CpG- מתילציה מבוסס הנפתח גישה organellar מפוצל מ DNA גרעיני (סעיף 3).
- אם באמצעות גישת צנטריפוגה ההפרש, לאסוף רקמות טריות ולהמשיך מיד לעיבוד דגימות, כמתואר בסעיף 2.
- אם ניצול הגישה CpG-methyl הנפתח, הקציר 20 מ"ג סעיפים של רקמת עלה צעיר לתוך צינורות microcentrifuge (להשתמש גם רגיל או גדל רקמה etiolated, לראות את תוצאות נציג ). הצמד להקפיא על חנקן נוזלי להקפיא ב -80 מעלות צלסיוס עד לשימוש. המשך לפריצה הנפתחת של דנ"א, כמתואר בסעיף 3.
2. שיטה # 1: מיצוי DNA באמצעות צנטריפוגה דיפרנציאלית (DC)
הערה: הבדלפרוטוקול צנטריפוגה erential שונה משני פרסומים כי אופטימיזציה התנאים לבודד את שני האברונים אבל להעשיר עבור המיטוכונדריה 17 , 24 . הפרוטוקול שנוצר הוא פחות זמן אינטנסיבי ומשתמש בכימיקלים רעילים פחות מאשר בשיטות הקודמות. באופן ספציפי, עשינו שינויים במאגר ו לשטוף צעדים, כולל תוספת של polyvinylpyrrolidone (PVP) למאגר החילוץ STE ואת חיסול השלב לשטוף הסופי במאגר NETF, המכיל פלואוריד נתרן (NaF).
זהירות: הכנה ושימוש במאגר STE צריך להתבצע מתחת למכסה קטר כימי עם ציוד הגנה אישי ראוי, כמו מאגר זה מכיל 2-mercaptoethanol (BME).
- דברים לעשות לפני שמתחילים
- ודא כי כל הציוד הוא נקי מאוד, החיטוי כל הציוד שניתן autoclaved ( למשל, צילינדרים שחיקה, מהירות גבוהה centriצינורות fuge, וכו ' ).
הערה: עצות סינון מומלצות לכל השלבים המחייבים את pipetting כדי למנוע זיהום צולב. - ראה רשימה של הציוד הנדרש ריאגנטים ולהכין את המאגרים הנדרשים ומלאי עבודה לשיטה # 1 ( טבלה 1 ). צינת בלוקים שחיקה קריוגני כדי -20 מעלות צלזיוס ואת הרוטורים מאגרים ל 4 מעלות צלזיוס, להגדיר את microcentrifuge ל 4 מעלות צלזיוס, ולהפעיל על אמבט מים 37 מעלות צלזיוס.
- ודא כי כל הציוד הוא נקי מאוד, החיטוי כל הציוד שניתן autoclaved ( למשל, צילינדרים שחיקה, מהירות גבוהה centriצינורות fuge, וכו ' ).
- בידוד האברונים
- קציר 5 גרם של רקמות טריות ולשטוף אותו במים קרים, סטרילי בכוס צונן על הקרח.
הערה: תמיד לשמור על דגימות על הקרח במהלך כל פעולות הובלות אל וממנו צנטריפוגות, ברדסים ברדסים, וכו ' לחילופין, לעבוד בחדר קר אם יש גישה מספיק מקום וציוד לבצע את הפרוטוקול. - באמצעות מספריים, לחתוך רקמת עלה לתוך ~ 1 ס"מ חתיכות ישירות לתוך צינור 50 מ"ל המכיל שני שחיקה קרמיצילינדרים.
הערה: נקה או שנה את המספריים בין דגימות כדי למנוע זיהום צולב. - אם אין homogenizer רקמות, להשתמש מרגמה העלי ועקוב אחר צעדים 2.2.4 - 2.2.9.
- חותכים את רקמת העלה לתוך מרגמה טרום צונן על הקרח. טוחנים את הדגימות עבור 2 - 3 דקות ב 15 מ"ל של STE (במנדף קטר).
- יוצקים את המאגר (להשאיר את הרקמה של מרגמה) דרך משפך המכיל שכבה אחת של בד טרום רטוב, סטרילי סינון (~ ~ 25 עד 25 מיקרומטר גודל הנקבוביות, לראות את הפרוטוקול העיקרי לפרטים) לתוך צינור אחר 50 מ"ל . מוסיפים עוד 10 מ"ל של STE על המלט ואת העלי ו homogenize שוב.
- יוצקים את הרקמה הומוגנית למאגר לתוך משפך זהה. שוטפים את המלט ואת העלי עם 10 מ"ל של STE ושופכים אותו לתוך משפך. לחץ וסחט את בד הסינון לתוך המשפך כדי לשחזר כמה שיותר נוזלים.
הערה: לשנות כפפות בין דגימות, כדי למנוע זיהום צולבות. המשך עם המקצועTocol at שלב 2.2.10.
- הוסף 20 מ"ל של STE (במנדף קטר) לכל צינור 50 מ"ל.
- מניחים את דגימות לתוך טרום צוננים חוסם cryogenic בלוקים במנגנון רקמה שחיקה לטחון דגימות עבור 2 x 30 s ב 1,750 סל"ד. סובב את עמדות המדגם במקום דגימות על הקרח במשך דקות 1 ~ בין grinds.
הערה: ניתן להשתמש בשלב זה במכתש ועלי, בלנדר או מכשיר אחר לטחנת רקמות / הומוגניות. עם זאת, כל שיטה ישפיע על איכות ה- DNA שנוצר מעלות שונות, ולכן אורך DNA ואיכות צריך להיות מוערך לפני המשך עם יישומים במורד הזרם. - הכנס משפך לתוך צינור נקי 50 מ"ל להציב בקרח. מניחים שכבה אחת של בד סינון לתוך המשפך מראש רטוב עם 5 מ"ל של STE. אל תשליך את הזרימה.
- יוצקים את הרקמה הומוגנית לתוך משפך. שוטפים את הצינור שחיקה עם 15 מ"ל של STE, לסכם ולהפוך את הצינור לשטוף את הקירות ואת המכסה, ושופכים לתוך funnEl.
- בזהירות להסיר את אבני החרס ולאחר מכן לסחוט ולסחוט את בד הסינון לתוך המשפך.
הערה: לשנות כפפות בין דגימות, כדי למנוע זיהום צולבות. - לעטוף את פקקי צינור עם parafilm כדי למנוע שפיכה. צנטריפוגה ב 2000 xg במשך 10 דקות ב 4 מעלות צלזיוס.
- בזהירות לשאוב supernatant באמצעות פיפטה סרולוגית (למנוע הפרעה גלולה) ומניחים אותו צינור צנטריפוגה 50 מ"ל במהירות גבוהה (אם צינורות אין אטמים אטום הדוק, לעטוף את כובעי צינור עם parafilm כדי למנוע שפיכה). מחק את כדורי.
- מאזן את צינורות לתוך 0.1 גרם באמצעות STE ו צנטריפוגה וכתוצאה מכך supernatant עבור 20 דקות ב 18,000 xg ו 4 מעלות צלזיוס. כדי לאזן את הצינורות, במקום כוס קטנה של קרח על האיזון, טרה את קנה המידה, ולשקול את הדגימות על הקרח כדי לשמור על הקור. לחלופין, להשתמש באיזון ואת ברדס קטר בחדר קר.
- מחק את supernatant. הוסף 1 מ"ל של ST כדי גלולה מחדש להשעות בעדינות אותנומכחול רך. הוסף 24 מ"ל של ST (נפח סופי של 25 מ"ל) ומערבבים / מערבולת ( כלומר, לחץ על מכחול בצד הצינור כדי להסיר את כל נוזל).
- לאזן את הצינורות בתוך 0.1 גרם באמצעות ST. צנטריפוגה במשך 20 דקות ב 18,000 XG ו 4 מעלות צלזיוס. בינתיים, להכין פתרון DNaseI (ראה טבלה 1 עבור מלאי פתרונות פתרון עבודה). עבור כל מדגם, לעשות אחד 200 aliquot μL בצינור 1.5 מ"ל.
- מחק supernatant, כתם את הצינור, מחדש להשעות את גלולה (עדיין צינור צנטריפוגה במהירות גבוהה) ב μL 300 של ST באמצעות מכחול רך. מניחים את מכחול בצינור מוכן 1.5 מ"ל לשעבר המכיל 200 μL של פתרון DNaseI ו מערבולת מכחול כדי להסיר כל גלולה שיורית תקועה במברשת. פיפטה הפתרון DNaseI בחזרה לתוך צינור צנטריפוגות במהירות גבוהה בעדינות מערבולת לערבב.
- דגירה על 37 מעלות צלזיוס למשך 30 דקות באמבט מים (לעטוף parafilm סביב החלק העליון של הצינור כדי למנוע עיבוי leakinG לתוך מכסה). מערבבים בעדינות על ידי סחרור 2 פעמים במהלך הדגירה.
- בעדינות פיפטה תערובת גלולה מתוך הצינור באמצעות קצה פיפטה עם פתח רחב ומניחים אותו צינור 1.5 מ"ל נמוך לאגד. הוסף 500 μL של 400 מ"מ EDTA, pH 8.0, כדי צינור צנטריפוגה במהירות גבוהה פיפטה בעדינות כדי לקבל את כל גלולה שיורית מתוך הצינור. מעבירים את EDTA לאותו 1.5 מ"ל, נמוך לאגד צינור כמו תערובת גלולה בעדינות לערבב על ידי היפוך.
- צנטריפוגה ב XG 18,000 במשך 20 דקות ב 4 מעלות צלזיוס. מחק את supernatant, כתם את הצינור, ולהשתמש בבת אחת עבור בידוד DNA. אם יש צורך, להקפיא pellets ב -20 מעלות צלזיוס, אבל זה עלול לגרום להפחתת התשואה, כמו DNaseI שיורית עלול להשפיל את הדנ"א מדגם אם זה לא מעובד מיד.
- קציר 5 גרם של רקמות טריות ולשטוף אותו במים קרים, סטרילי בכוס צונן על הקרח.
- מיצוי דנ"א מאורגנים מבודדים תוך שימוש בגישה מסחרית המבוססת על עמודות
הערה: עיין במדריך הערכה עבור הפרוטוקול המלא 25 , וראה להלן שינויים. יחסי ציבורOceeding ישירות מבידוד organellar כדי מיצוי ה- DNA הוא העדיף. הקפאה חוזרת ונשנית והפשרה תפחית את גודל הדגימה של ה- DNA ותוביל להשפלה של דנ"א על ידי DNaseI שיורי. הגבלת vortexing או pipetting נמרץ, כמו זה יכול לגזום את ה- DNA. השימוש צינורות microcentrifuge נמוכה לאגד מומלץ למקסם את התאוששות ה- DNA.- מיצוי דנ"א
הערה: קרא את הפרוטוקול המסחרי המפורט 25 לפני שתתחיל לוודא כי המאגרים נעשים כראוי / מאוחסנים וכי הליכי טור ספין מובנים.- הוסף 180 μL של ATL חיץ ישירות לתוך הצינור עם גלולה (מופשר אם בעבר קפוא equilibrated לטמפרטורת החדר על benchtop).
- המשך שלב 3 בפרוטוקול "טיהור דנ"א מ רקמות" בספר הדרכה Kit, עם השינויים הבאים: 30 דקות תמוגה בשלב 3, כולל עיכול אופציונלי RNase א, elute ב 3 x 200 μL של AE ( כל אחד לתוך seצינור parate ולאחר מכן לשלב את elutions).
- שמור aliquot (לפחות 20 μL) עבור qPCR (ראה שלב 4.1). כדי לכמת לפני ריכוז, לשמור עוד 1 μL עבור כימות רגישות גבוהה.
- אם תרצה, להמשיך עם ריכוז המדגם.
- מיצוי דנ"א
- ריכוז לדוגמה עם יחידות מסנן מסחרי
הערה: עיין בפרוטוקול המסחרי 26 לקבלת פרטים נוספים. בהתאם לשימוש במורד הזרם, ייתכן שלא יהיה צורך לבצע ריכוז מדגם ( למשל, עבור נקודות קצה PCR ויישומים qPCR). עם זאת, עבור NGS ספריית הבנייה, זה יהיה כנראה יהיה צורך לרכז דנ"א organellar שהושג לאחר החילוץ DNA.- תהליך ריכוז עמודה
- בזהירות שקול מראש (ראה טבלה 2 ) יחידת מסנן ריק (ללא צינור) על פיסת נייר שקילה על מאזן אנליטי דיגיטלי. רשום את המשקל.
- פאימקפלים את ה elutes המשולב לתוך יחידת המסנן ושוקלים בזהירות.
הערה: המדריך המסחרי 26 אומר כי נפח יחידת הסינון המקסימלי הוא 500 μL, אבל עד 575 μL ניתן להוסיף ליחידה בבת אחת ללא הצפת. - בזהירות במקום יחידת מילוי מלא לתוך צינור (מסופק עם העמודות). צנטריפוגה ב XG 500 עבור הזמן הרצוי כדי להשיג את נפח להתרכז הנדרש. עבור נפח מדגם של ~ 575 μL, ספין 20 דקות בדרך כלל התוצאה נפח להתרכז של μL 15 - 30.
- הסר את יחידת הסינון מהצינור ושקול שוב. השתמש בטבלה כדי לקבוע אם נפח הריכוז הרצוי הושג. אם לא, צנטריפוגה שוב ב 500 xg למשך זמן קצר יותר ולשקול שוב; חזור עד להופעת נפח הריכוז הרצוי.
- מניחים צינור חדש (מסופק עם העמודות) מעל החלק העליון של יחידת המסנן ולהפוך. צנטריפוגה במשך 3 דקות ב 1000 xg להעביר את שיתוףNcentrate אל הצינור.
- לקבוע את עוצמת הקול התאושש. זה יהיה בדרך כלל ~ 3 - 5 μL פחות נפח מחושב, עקב שימור מסנן. אם יתר מרוכז, לדלל עם מים סטריליים או TE להשיג את עוצמת הקול הרצויה.
- לכמת את ה- DNA באמצעות כימות רגישות גבוהה (לפי הוראות היצרן).
- תהליך ריכוז עמודה
3. שיטה מס '2: Methyl-Fractionation (MF) גישה להעשרת DNA של Organellar מתוך סך DNA גנומי
הערה: פרוטוקול זה שונה מן המשתמש פיתחה גנומי עצה קיט DNA פרוטוקול החילוץ עבור צמחים ופטריות 27 ו Microbiome DNA העשרה פרוטוקול Kit העשרה 28 . בתיאוריה, כל פרוטוקול בידוד דנ"א אשר מניב DNA גבוה משקל מולקולרית עשוי לשמש הנפתח. עבור רצף קריאה קצר, כל מיצוי החילוץ ברובו> 15 קילו שברי הוא מתאים לשימוש הנפתח. עבור loרצף קריאה ng, שברי גדול עשוי להיות רצוי. לכן, אנו אופטימיזציה זה פרוטוקול להניב דנ"א משקל מולקולרי גבוה.
- בידוד של ה- DNA הכולל
הערה: ראה רשימה של הציוד הנדרש ריאגנטים ולהכין את המאגר הנדרש מניות עבודה עבור שיטה מס '2 ( טבלה 1 ). הוסף lysing אנזימים מלאי מאגר תמוגה כדי להפוך את הפתרון תמוגה חיץ פתרון. הפעל את thermomixer ולהגדיר אותו 37 מעלות צלזיוס. הפעל את אמבט המים ל 50 מעלות צלזיוס במקום חיץ QF באמבטיה. מקום 70% EtOH במקפיא ולהגדיר את microcentrifuge ל 4 מעלות צלזיוס.- סה"כ מיצוי DNA באמצעות עמודות מסחריות של דנ"א
הערה: לפני שתתחיל, קרא את המדריך המסחרי 29 לקבלת מידע מפורט אודות השימוש בעמודות חילופי אניות זרימת הכבידה. העמודות ניתן להגדיר באמצעות מתלה מיוחד או להציב מעל צינורות באמצעות טבעות פלסטיק המסופקים. כל השלבים, כולל gטיפים אטומיים, יש לאפשר להמשיך על ידי זרימת הכבידה, נוזל שיורי לא צריך להיות כפוי דרך.- טוחנים 20 מ"ג של רקמה קפואה בחנקן נוזלי בצינור 2-mL נמוך לאגד באמצעות יד שחיקה טירות המיועד צינורות 2 מ"ל.
- הוסף 2 מ"ל של תמיסת תמיסה חיץ פתרון (צינורות יהיה מלא מאוד).
- דגירה thermomixer ב 37 מעלות צלזיוס למשך 1 שעות עם תסיסה עדינה בסל"ד 300. אם thermomixer אינו זמין, incubating על בלוק חום ערבוב ידי עדין מהבהב כל 15 דקות היא חלופה מתאימה.
- הוסף 4 μL של RNase A (100 מ"ג / מ"ל, ריכוז סופי של 200 מיקרוגרם / מ"ל). הפוך לערבב דגירה thermomixer במשך 30 דקות על 37 מעלות צלזיוס, עם תסיסה עדינה בסל"ד 300.
- הוסף 80 μL של proteinase K (20 מ"ג / מ"ל, ריכוז סופי של 0.8 מ"ג / מ"ל), להפוך לערבב, ו דגירה thermomixer עבור 2 שעות ב 50 מעלות צלזיוס, עם תסיסה עדינה ב 300 סל"ד.
- צנטריפוגה במשך 20 דקות ב 4 ° C ו 15,000 xg כדי גלולה פסולת מסיס.
- בעוד הדגימות צנטריפוגה, לאזן את העמודות עם 1 מ"ל של QBT הצפת ולאפשר את הטור לרוקן על ידי זרימת הכבידה.
- השתמש קצה פיפטה רחב נשא מיד ליישם את המדגם (למנוע את גלולה) אל עמוד equilibrated ולאפשר לו לזרום באופן מלא דרך העמודה. אם המדגם הופך מעונן, לסנן או צנטריפוגה שוב לפני היישום לעמודה (עיין במדריך מסחרי לפרטים 29 ).
- לאחר המדגם נכנס לחלוטין שרף, לשטוף את העמודה עם 4 x 1 מ"ל של QC חיץ.
- להשעות את הטור מעל נקי, 2 מ"ל, צינור נמוך microcentrifuge לאגד. Elute את הדנ"א הגנומי עם 0.8 מ"ל של prefmed QF חיץ ב 50 מעלות צלזיוס.
- לזרז את ה- DNA על ידי הוספת 0.56 מ"ל (0.7 כרכים של חיץ elution) של טמפרטורת החדר isopropanol כדי DNA eluted.
- מערבבים על ידי היפוך (10X) ו צנטריפוגות מיד 20 דקות ב 15,000 xg ו 4 ° C. לְטַפֵּלמלא להסיר את supernatant מבלי להפריע את גלולה, גלולה מצורף רופף.
- שטפו את צנטריפוגה דנ"א גלולה עם 1 מ"ל של אתנול 70% קר. צנטריפוגה במשך 10 דקות ב XG 15,000 ו 4 ° C.
- בזהירות להסיר את supernatant (להיות זהירים עם צעד זה גם כן) מבלי להפריע גלולה. אוויר יבש במשך 5-10 דקות ו resuspend את ה- DNA ב 0.1 מ"ל של חיץ elution (EB). ממיסים את הדנ"א בן לילה בטמפרטורת החדר. הימנע pipetting, אשר עשוי לגזום את ה- DNA.
- לכמת את דגימות באמצעות assay גבוהה רגישות כימות DNA (לפי הוראות היצרן).
- סה"כ מיצוי DNA באמצעות עמודות מסחריות של דנ"א
- פיצול מבוסס חרוז של דנ"א methylated ו unmethylated
הערה: פרסום שפורסם לאחרונה הוכיח את השימוש בערכה זמינה מסחרית 28 המנצלת את הגישה הנפתחת באמצעות CpG ספציפי חלבונים מתיל מחייב תחום התמזגו לחלק האדם IgG FC (חלבון MBD2-Fc) לשבראכלו צמח הגנום organellar (unmethylated) מ הגנום הגרעיני (מתילציה) תוכן 23 . יעילות הפיצול בדגימות חיטה לא נבדקה קודם לכן באמצעות ערכת MF המסחרית 28 .- דברים לעשות לפני שמתחילים
- טריים להכין 80% אתנול (לפחות 800 μL לכל תגובה). בחר 5x לאגד / לשטוף חיץ להפשיר על הקרח ולהכין 5 מ"ל של חיץ 1x לכל מדגם (לדלל 5X חיץ עם מים סטרילי, nuclease ללא ולשמור על הקרח במהלך הפרוטוקול).
- הכן MBD2-FC חרוזים מגנטיים מחויב
- הכינו את המספר הדרוש של ערכות חרוזים. קנה המידה את התגובות להשתמש בין 1 ו 2 מיקרוגרם של ה- DNA קלט הכולל, הדורש 160-1300 μL של חרוזים. שים לב כי התגובות המפורטות להלן הם עבור 1 מיקרוגרם של ה- DNA קלט הכולל, ולכן הם דורשים 160 μL של חרוזים. סולם את התגובות בהתאם לצרכים.
- שימוש רחב נשא טיפים, בעדינות פיפטה את חלבון מגנטי BEad slurry למעלה ולמטה כדי ליצור השעיה הומוגנית. כחלופה, בעדינות לסובב את הצינור של חרוזים במשך 15 דקות ב 4 מעלות צלזיוס.
הערה: אל מערבולת החרוזים. - המשך בהוראות לפי הוראות היצרן 28 .
- לכידת DNA גרעיני מתיל
- עבור כל מדגם בודדים, להוסיף 1 מיקרוגרם של ה- DNA קלט לצינור המכיל 160 μL של חרוזים מגנטיים MBD2-Fc- מגנטי.
- הוסף 5x לאגד / לשטוף חיץ לפי הצורך בהתחשב בנפח הדגימה קלט דנ"א לריכוז סופי של 1x (נפח של 5x לאגד / לשטוף חיץ להוסיף (μL) = נפח ה- DNA קלט (μL) / 4). פיפטה המדגם למעלה ולמטה כמה פעמים כדי לערבב באמצעות קצה פיפטה רחב נשא.
- סובב את הצינורות בטמפרטורת החדר למשך 15 דקות. בעדינות פיפטה דגימות עם קצה פיפטה נשא רחב להעיף את הדגימות 2 - 3 פעמים לאורך הדגירה כדי למנוע clumping חרוז.
הערה: pipetting ו flickiNg הוא קריטי כדי להבטיח הנפתח יעיל של ה- DNA methylated.
- איסוף דנ"א organellar מועשר, unmethylated
- בקצרה לסובב את הצינור המכיל את ה- DNA ו MBD2-Fc כבול תערובת חרוז מגנטי. מניחים את הצינור על מתלה מגנטי במשך לפחות 5 דקות לאסוף את החרוזים בצד הצינור. הפתרון צריך להיראות ברור.
- בעזרת טיפים רחבים, להסיר בזהירות supernatant לנקות ללא הפרעה חרוזים. מעבירים את supernatant (מכיל unmethylated, DNA מועשר organellar) כדי נקי, נמוך לאגד, 2 מ"ל microcentrifuge צינור. חנות מדגם זה -20 או -80 מעלות צלזיוס, או להמשיך ישירות לשלב 3.2.6 לטיהור.
- Elute שנתפסו DNA גרעיני מן MBD2-Fc חרוזים מגנטיים
- אם חלק הגרעין הוא גם הרצוי, בצע את הוראות היצרן 28 כדי elute DNA גרעיני מן חרוזים מגנטיים MBD2-Fc- מגנטי; לטהר כמתואר בשלב 3.2.7.
- חומצה המבוססת על טיהור חומצות גרעין
- ודא חרוזי טיהור הם בטמפרטורת החדר מעורבבים היטב. המשך בפרוטוקול בהתאם להוראות במדריך למשתמש MF 28 .
הערה: המדגם יכול כעת לשמש לבניית ספריית NGS או ניתוח נוסף במורד הזרם.
- ודא חרוזי טיהור הם בטמפרטורת החדר מעורבבים היטב. המשך בפרוטוקול בהתאם להוראות במדריך למשתמש MF 28 .
4. כימות לדוגמא בקרת איכות
- QPCR assay להעריך להעשיר organellar
הערה: התגובה qPCR ופרמטרים assay המפורטים כאן נועדו לשימוש על Roche LightCycler 480 וייתכן שיהיה צורך להתאים עבור ציוד שונים ריאגנטים. אם qPCR אינו זמין, נקודות קצה PCR ויזואליזציה על ג'ל agarose עשוי לשמש מדד איכותי של טוהר המדגם, תוך שימוש primers אותו התנאים המתוארים כאן. גדלי Amplicon יהיה ~ 150 BP עבור כל ערכות פריימר. ראה טבלה 3 עבור רצף פריימרCes ו זיווגים.- QPCR תגובה התגובה
- כדי להגדיר תגובה בודדת qPCR 20 μL, בזהירות פיפטה הבא לתוך באר אחת של צלחת qPCR 96 גם: 10 μL של 2x SYBR ירוק אני מאסטר; 2 μL של 10 מיקרומטר קדימה תערובת פריימר הפוך (עבור ריכוז סופי של 0.5 מיקרומטר); 2 μL של תבנית (בטווח של עקומה סטנדרטית); ו 6 μL של סטרילית, nuclease ללא H 2 O. כדי להפחית את השגיאות pipetting, עדיף לעשות תערובת הורים עם כל רכיבי התגובה למעט התבנית. הוסף את התמהיל הראשי לצלחת qPCR ולאחר מכן להוסיף את התבנית של עניין כל טוב. שלושה משכפל טכני עבור כל מדגם צריך להתבצע כדי למזער את ההשפעות של שגיאת pipetting.
הערה: בסופו של דבר, היחס בין מחזורי כימות לאורגנילאר משווים בין דגימות, ולכן הבדלים קלים בריכוז מקובלים. עם זאת, ריכוז ה- DNA צריך להיות בערך בטווח של eaCh אחר. - חותם את הצלחת עם איכות גבוהה qPCR איטום הסרט. בעדינות מערבולת דגימות, נזהר כדי למנוע את היצירה של כל הבועות. בקצרה לסובב את הצלחת למטה במשך 2 דקות ב 4 ° C כדי לאסוף את המדגם ולמנוע כל בועות קטנות.
- טען את הצלחת לתוך המכונה. הפעל את התוכנית qPCR לפי ההנחיות המפורטות להלן.
- כדי להגדיר תגובה בודדת qPCR 20 μL, בזהירות פיפטה הבא לתוך באר אחת של צלחת qPCR 96 גם: 10 μL של 2x SYBR ירוק אני מאסטר; 2 μL של 10 מיקרומטר קדימה תערובת פריימר הפוך (עבור ריכוז סופי של 0.5 מיקרומטר); 2 μL של תבנית (בטווח של עקומה סטנדרטית); ו 6 μL של סטרילית, nuclease ללא H 2 O. כדי להפחית את השגיאות pipetting, עדיף לעשות תערובת הורים עם כל רכיבי התגובה למעט התבנית. הוסף את התמהיל הראשי לצלחת qPCR ולאחר מכן להוסיף את התבנית של עניין כל טוב. שלושה משכפל טכני עבור כל מדגם צריך להתבצע כדי למזער את ההשפעות של שגיאת pipetting.
- Q פרמטרים התגובה
הערה: אלה הם פרמטרים המשמשים כברירת מחדל, למעט מחזור החישול של שלב ההגברה. התאם הגדרה זו כדי להתאים פריימרים ספציפיים אם אלה המשמשים שונים פריימרים המוצגים בפרוטוקול זה.- טרום דגירה על 95 מעלות צלזיוס למשך 5 דקות, עם שיעור הרמפה של 4.4 מעלות צלזיוס / s.
- ביצוע 45 מחזורים הגברה של (1) 95 מעלות צלזיוס במשך 10 s, עם שיעור הרמפה של 4.4 מעלות צלזיוס / s; (2) 60 מעלות צלזיוס במשך 20 s, עם שיעור הרמפה של 2.2 מעלות צלזיוס / s; ו (3) 72 מעלות צלזיוס במשך 10 s, עם שיעור הרמפה של 4.4 מעלות צלזיוס (נתונים שנרכשו במהלך (3)).
- השתמש באופטיוNal עקום מחזור עקומת של 95 מעלות צלזיוס במשך 5 s, עם שיעור הרמפה של 4.4 מעלות צלזיוס / s; 65 מעלות צלזיוס למשך 1 דקות, עם שיעור הרמפה של 2.2 מעלות צלזיוס / s; ו 97 ° C, עם מצב הרכישה מתמשך.
- השתמש במחזור קירור של 40 מעלות צלזיוס למשך 30 שניות, עם קצב הרמפה של 1.5 ° C / s.
- פרמטרים Assay
- בחר את תבנית ה- SYBR. בדוק את הפרמטרים של התוכנית בלחצן הניסוי. לאחר צלחת נטען, assay ניתן להתחיל, ואת ההגדרות עשוי להיות מותאם בזמן assay פועל.
- הקצה דוגמאות באמצעות עורך המדגם. בחר ABS כמות כמו זרימת העבודה ואת לייעד את דגימות כמו ידוע, סטנדרטים, או שולטת שלילית. לייעד משכפל ולמלא את שמות המדגם של הראשון של כל לשכפל. הוסף ריכוזים ויחידות לתקנים.
- הגדרת קבוצות משנה לניתוח; אלה מוקצים בעורך המשנה.
- לניתוח, בחר Abs Quant / 2nd נגזרת מקס מתוך "יצירת ניתוח חדש" רשימה.ייבוא עקומת תקן שנשמר חיצונית (אם רלוונטי) ולאחר מכן פגע לחשב; הדוח יכיל את המידע שנבחר.
- כדי לבצע כימות מוחלטת מדויקת לקביעת מספר העותק או הריכוז, יש להשתמש בעקומה סטנדרטית המייצגת את המדגם הנבדק ( לדוגמה, DNA של ה- organellar המבודד מהשיטות לעיל). מאחר שכמות הדנ"א המיטוכונדריאלי הנדרשת להכנת עקומה סטנדרטית גבוהה מדי להשגה עם כמות סבירה של רקמות, אל תשתמש בחישובי מספרי העותקים המסופקים על-ידי התוכנה, אלא נבחן את ערכי נקודת המעבר (Cp) כדי לקבוע את העשרה היחסית של organellar לעומת DNA גרעיני בדגימות. השווה אלה כמויות יחסית לאלה של הדנ"א הגנומי הכולל (ראה תוצאות נציג ). מבחן היעילות פריימר על חמש דילולים 1:10 של הדנ"א הגנומי הכולל מ מבוגר קל, שתילי חיטה בן שבועיים (יעילות נציג דיווחו בE האגדה של איור 2 ).
- QPCR תגובה התגובה
- ג'ל אלקטרופורזה פעימה בשדה (PFGE)
הערה: פרוטוקול זה מבוסס על הנחיות היצרן לבצע PFGE כדי לפתור גבוהה DNA משקל מולקולרי. ראה טבלת החומרים.- הכנת הג'ל ודגימות
- בצע את ההנחיות להכנת ג'ל והדוגמה ולהתאים אותם למערכת זמינה.
- הפעל את הפרמטרים
- בצע את ההנחיות להקמת מערכת אלקטרופורזה ולהשתמש בפרמטרים הבאים: זמן מתג התחלתי של 2 שניות, זמן מתג סופי של 13 שניות, זמן ריצה של 15 שעות ו- 16 דקות, V / cm של 6, וזווית כלולה של 120 ° .
- הכתם ואת התמונה ג'ל
- כתם ג'ל עם צבע של בחירה ( למשל, ethidium bromide או חלופה מתאימה) ואת התמונה עם מערכת תיעוד ג'ל מתאים.
- הכנת הג'ל ודגימות
- השתמש 1 ng של DNA כמו קלט עבור ערכת ה- DNA Prep Kit, לפי הוראות היצרן.
- ברקוד הבריכה דגימות עבור רצף בריצה אחת. בצע רצף בהתאם להנחיות היצרן.
הערה: ניתן לשנות את הפרמטרים של בריכה ורצף בהתאם למין האינטרס, רמת הכיסוי הרצויה והפלטפורמה המשמשת לרצף הספריות. לדוגמה, נתיב HiSeq יש תפוקה גדולה יותר מאשר נתיב MiSeq, כך דוגמאות רבות יותר ניתן multiplexed. רצף קבוצה קטנה יותר של דגימות כדי לקבוע אם רמות הכיסוי של genomes organellar נאותים עבור ניתוח במורד הזרם.- בדוק את איכות הקריאה באמצעות FastQC 31 כדי לקבוע את מידת הגזירה והסינון הדרושים לנתונים.
- חתוך ולסנן את הגלם קורא באמצעות Trimmomatic 32 או תוכנית דומה אחרת. השתמש בהגדרות הבאות: ILLUMINACLIP 2:30:10 (כדי להסיר מתאמים), מוביל 3, TRAILING 3, SLIDINGWINDOW 4:10, ו MINLEN 100.
- מפה את איכות מסוננים מתוחכם גזוז- end (PE) קורא את המינגוכונדריה הסינית האביבית (NCBI הפניה רצף NC_007579.1 33 ), chloroplast (NCBI הפניה רצף NC_002762.1 34 ), ואת הגרעין 35 התייחסות גנומים באמצעות Bowtie2 36 , עם ההגדרות הבאות: -I 0 -X 800 - רגיש.
- המרת קבצי יישור סם בפורמט bam (samtools) ומיין את קבצי bam. השתמש בקבצי בם כדי לחשב כיסוי גנום רחב כיסוי לכל בסיס עם bedtools. לדמיין את התוצאות עם פונקציה R- העלילה.
- דברים לעשות לפני שמתחילים
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
הפרוטוקולים המוצגים בכתב יד זה מתארים שתי שיטות שונות להעשרת דנ"א אורגני מהרקמה הצמחית. התנאים המוצגים כאן משקפים אופטימיזציה של רקמת החיטה. השוואה של שלבים עיקריים בפרוטוקולים, קלט רקמות נדרש, פלט ה- DNA מתוארים באיור 1 . השלבים של פרוטוקול DC בדקנו לעקוב אחר תנאים דומים לאלה שתוארו בעבר ( איור 1 א ). רקמות שנקטפו חייב להיות מעובד טרי ונתונה צנטריפוגה דיפרנציאלי ו / או gradients לבודד את האורגנים שלם. הדנ"א הגרעיני מסולק לפני שהאיברים הם lysed, ולבסוף, DNA מופק ומשמש עבור יישומים במורד הזרם. לעומת זאת, בפרוטוקול MF, רקמת הצמח ניתן לקצור ולאחסן לפני השימוש, ואת האורגנים שלם אינם נדרשים. במקום זאת, ה- DNA הגרעיני ו- organellar מופרדים מ- gDNA הכולל מעמד המתילציה של הדנ"א. שני הפרוטוקולים מניבים כמויות שוות בערך של DNA DNA ( איור 1C ). במונחים של תפוקת הדנ"א הכוללת ביחס לקלט רקמות, פרוטוקול MF הוא יתרון כאשר הרקמה מוגבלת, כמו מדגם קטן של צמח אחד עשוי לשמש, ואת הצמח עשוי להיות מותר לגדול לניתוח נוסף. בדרך כלל, בפרוטוקולי DC, כל הרקמות האוויריות של שתילים רבים נדרשים, צמחים אלה נמחקים. עם זאת, השיטה DC יכול להיות מותאם במיוחד להעשיר עבור סוג אחד organelle על פני השני, אשר אינו אפשרי עם הגישה MF. ראוי להזכיר כי הזמן הכולל עבור כל פרוטוקול הוא שווה ערך בערך, אם כי יש פחות הידיים על הזמן בגישה MF.
שתי שיטות להעשיר עבור DNA Organellar, אם כי עם פרופורציות שונות של מיטוכונדריה רצפים פלסטיד:
כמויות נמוכות מאוד של DNA אורגני מטוהרים מתקבלות משני השיטות (בסדר גודל של 50 - 100 ng, איור 1 ג ' ), על מנת להעריך את רמות העשרת הגנום של האורגן ואת זיהום הגנום הגרעיני בדנ"א המבודדים הן מן DC והן מ- MF שיטות, assay qPCR היה מועסק.ב assay זה, את השפע היחסי של שלושה amplicons ( כלומר, ספציפית גרעינית, ACTIN , המיטוכונדריה ספציפי, NAD3 , ו Chloroplast ספציפי, PSBB ) הוערכו סך DNA גנומי, ואת חלק ה- DNA organellar התקבל בשתי השיטות ( איור 2 ). ערכי כימות מחזור (C q) נבחנו עבור כל דגימה (איור 2 א), ומכיוון ש C מוגדר מחזור PCR שבו הקרינה מן עליות הגברת היעד מעל רמת קרינת הרקע, q ו- יעד C שפע יש מערכת יחסים הפוכה. בהמדגם DC, ה- C q של NAD3 ו PSBB הם, בהתאמה, ~ 17 ו ~ 15 מחזורים לפני אקטין (שבו יש q C של ~ 36) (ראה איור 2B עבור ערכי q C ורמות העשרה). זה משווה תיאורטית 167,181- ו 47,790 קפל העשרה עבור NAD3 ו PSBB , בהתאמה, יחסית ACTIN במדגם DC ( איור 2 ב , ראה את האגדה של איור 2 לחישוב). בשנת דגימת DNA הכולל גנומי, את העשרה לקפל עבור היחסי NAD3 ו PSBB כדי אקטין הם רק 158 ו 10,701, בהתאמה. אין זה מפתיע למצוא שפע גבוה יותר של amplicons organellar יחסית amplicon הגרעיני של הדנ"א הגנומי הכולל, בהתחשב בכך genomes organellar קיימים מספרי עותק גדול יותר לתא מאשר הגנום הגרעיני 37 וכי מספר האברונים peR עשויים להשתנות בהתאם לסוג הרקמה או לשלב ההתפתחותי 38 , 39 . בסך הכל, הנתונים מצביעים על כך ששיטת DC מעדיפה באופן מיטוכונדרי, כפי שניתן לצפות, כאשר מהירויות צנטריפוגה מיועדות לבידוד המיטוכונדריה באופן סלקטיבי ולהפחתת "זיהום גרעיני וכלורופלסט".חלק unmethylated של GDNA סך MF גם מראה העשרה משמעותית של שני amplicons organellar והוא צפוי לשמור על כמויות יחסית הילידים של מטרות אלה. Enrichments לקפל עבור היחסי NAD3 ו PSBB כדי אקטין בשבריר unmethylated הם 20,551 ו 1,703,253, בהתאמה (איור 2 א ו -2 ב). בשבריר מפוגל, את העשרה לקפל עבור היחסי NAD3 ו PSBB כדי אקטין הם 31 ו 823, בהתאמה, אינדיCating כי חלבון MBD2-Fc הוא יעיל מאוד ב הנפתח של DNA גרעיני מתיל. כמו amplicon chloroplast יש שפע גבוה יותר מאשר amplicon המיטוכונדריה ב DNA הגנומי הכולל (~ 6 C Q מוקדם יותר), חלק מתילאטד (~ 5 C Q מוקדם יותר), וחלק unmethylated (~ 6 ג Q מוקדם יותר) דגימות, זה מצביע על כך יליד שפע של amplicons אלה לא השתנה באופן משמעותי על ידי הנפתח MDB2. אנו מתמקדים כאן על חלק unmethylated (organellar) בשל העניין ברצף גנומים אלה במיוחד. עם זאת, אם הגנום הגרעיני הוא האינטרס העיקרי, MF ו רצף הבאים של חלק מתילציה יניב כיסוי גנום גרעיני הרבה יותר מאשר סך רצף גנומי DNA, בשל הפחתת זיהום DNA "organellar".
ראוי לציין כי אם qPCR אינו זמין, נקודת קצה PCR (באמצעות פריימרים אותו כמו qPCR) מספק את qualitaהערכה של טוהר organellar. במקרה זה, דגימות DNA אורגניות טהורות יראו הגברה עבור אמפליקונים מיטוכונדריאליים ופלסטידים, אך לא הגברה לזיהוי של amplicon הגרעיני על ג'ל agarose, בעוד סך DNA גנומי מראה הגברה עבור כל שלוש קבוצות פריימר, כפי שהוכח במחקרים קודמים 11 , 12 .
דנ"א Organellar מבודדת בשתי השיטות מתאים NGS:
Trimmed וניקוי PE רצף קורא (ראה שלב 4.3) היו ממופות שפורסמו בעבר genomes התייחסות חיטה organellar, ואת כמות הקריאות המשמש למיפוי כל דגימה נע בין ~ 800,000 ל 1,100,000 קורא ( איור 3I ). תוצאות מיפוי דה novo מסדרת Illumina קורא את כלורופלסט חיטה זמין הגנום המיטוכונדריה עולים בקנה אחד עם res QPCR, כאשר שיטת ה- DC מניבה דנ"א העשיר יותר בדנ"א המיטוכונדריאלי ( איור 3 א ' ו -3 ב' , ~ 80% ו -10% מהמפות הנקראות לגנום המיטוכונדריה (mt) ו- chloroplast (cp), בהתאמה) ושיטת MF דנ"א מניב אשר משקף ככל הנראה את השפע של שני הגנומים ( איור 3 א ' ו -3 ב' , 20 ~ 20% ו ~ 80% מהמפות הנקראות לגנום הר ו cp, בהתאמה). בשתי השיטות, הכיסוי התיאורטי (ראה את האגדה בתרשים 3 לחישוב) של שני הגנומים של אבקת החיטה עולה על 100X כיסוי (וטווחים עד כיסוי של 2,000X עבור הגנום של כלורופלסט בשבר unmethylated בשיטת MF), אפילו כאשר 12 ספריות מרובות ( איור 3 ג ו -3 , 6 הספריות שנכללו בניתוח זה נאספו עם 6 ספריות נוספות לניתוח נפרד, עבור סך של 12 ספריותנאספו בנתיב רצף יחיד). תצוגה מפורטת יותר של הכיסוי הושגה על ידי בחינת חלק הגנום המכוסה במעמקים ספציפיים, כמו גם על רמות כיסוי לכל בסיס ( איור 3E -3I ). בשיטת MF, הכיסוי הממוצע לכל בסיס היה ~ 300 - 450X עבור הגנום הר ו 4,000 - 5,000X עבור הגנום cp. עבור שיטת DC, הכיסוי הממוצע לכל בסיס היה ~ 900 - 1,300 ו ~ 500 - 700X עבור mt ו genomes cp, בהתאמה. עם זאת, היה חלק קטן של שני הר ו cp הגנום כי היה נמוך מאוד כיסוי גבוה, וזה היה לראות ב- DNA organellar נגזר משני השיטה ( איור 3I ). אזורים של כיסוי גבוה מהממוצע עשויים להתאים לאזורים של הומולוגיה בין הגנומים של האברונים, ואזורים עם כיסוי נמוך עשויים להצביע על SNP או על גרסאות קטנות אחרות בין הזנים שציירנו לבין הפניות שפורסמו. כדי לתמוך ברעיון זה, אלה דוקרניםשל כיסוי גבוה היו בולטים ביותר עבור דנ"א הר הנגזר בשיטת MF ( איורים 3E ו 3I ), ככל הנראה בשל הכיסוי הגבוה של גנום cp בשיטה זו. באופן בלתי מוסבר, הכיסוי של הגנום cp הוא לא אחיד יותר בשיטת MF מאשר בשיטה DC ( איור 3 ו -3 H ), אשר יכול להיות בגלל הטיות קלות ב- MBD2-Fc הנפתח לאורך ה- DNA cp. ניסויים נוספים יידרשו כדי לקבוע מדוע זה המקרה. עם זאת, הר ו גנומים cp היה כיסוי אפילו יחסית עם שתי השיטות ולא אזורים גדולים של כיסוי חסר, אשר ניתן להוכיח על ידי בחינה של חלק הגנום ברצף בעומק נתון ( איור 3E -3H ). בנוסף, רמות הכיסוי של שני הגנום נחשבות מספיקות לניתוח במורד הזרם, כגון ניתוח חלופי. אם נראה צורך ניתוח של גרסאות נדירות, הפחתת numbeR של דגימות מקובצים היה להשיג כיסוי גדול יותר. לחלופין, מספר גדול בהרבה של דגימות עשוי להיות מאוגדים על נתיב HiSeq, תוך השגת עומק רצף אפילו יותר, אם כי על הקורבן לרצף אורך, כמו ספריות HiSeq מוגבלים כרגע על אורך PE150 בניגוד לספריות PE300 MiSeq.
כדי לבדוק את רמות זיהום הגנום הגרעיני באמצעות גישת מיפוי, PE קרא קטגוריות מיפוי נבדקו. קריאה PE יכול למפות לגנום התייחסות במגוון תצורות. כאשר קורא 1 ו 2 ליישר את ההתייחסות ב ראש אל ראש אופנה, עם מרחק מסוים "צפוי" בין שני בני זוג (מבוסס על גודל ההכנסה הממוצע של הספרייה ו מוגדר בדרך כלל פרמטר קלט בתוכנת מיפוי ), אלה PE קורא נאמר למפות "concordantly". לעומת זאת, "מיצוב" מיפוי הוא המצב שבו בני זוג מפה עם פחות או יותר מהצפוי disכדי להצביע על גנום התייחסות או מפה בתצורות חלופיות (ראש אל זנב או זנב אל זנב). אם רק בן זוג אחד מיישר את הגנום הפניה, אז זה לקרוא PE הוא אמר למפות לא concordantly או discordantly לגנום התייחסות. בכל שלוש קטגוריות הקריאה-מיפוי, PE קורא יכול ליישר את הגנום התייחסות אחת או מספר פעמים.
עבור שניהם DNA ו- MF מבודדים DNA organellar, לקרוא מיפוי הגנום המיטוכונדריאלי היה בעיקר בקו אחד של פעם בקונקורדנט זמן ( איור 4 א ), בעוד קורא ממופה לגנום chloroplast בפרופורציות שוות יחסית של קונקורדנטי פעם אחת concordantly יותר פעם אחת ( איור 4 ב ' ), ככל הנראה בשל חוזר גדול הפוך הנוכחי בגנום chloroplast וגם לרמות כיסוי גבוהה מאוד. עם זאת, פחות קוראים PE ממופה הגנום הגרעיני ממופה בעיקר יותר מפעם אחתלא קונקורדית ולא מתוחכמת ( כלומר, רק בן זוג אחד מסוגל למפות). אלה הם כנראה מיפוי "מחוץ היעד" רצפים בגנום הגרעיני, אשר הומולוגיים לגנום organellar או אזורים misassembled. רק כמות קטנה של קריאות (<5%) ממופות לגנום הגרעיני באופן קונקורדנלי, דבר המצביע על רמות נמוכות של זיהום גנום גרעיני בדנ"א ב- organellar המבודד משיטת DC או MF ( איור 4 ג ' ), כפי שמשתקף גם בתוצאות qPCR ( איור 2 א ). החלק הגרעיני לאחר הנפתח MBD2-Fc הנפתח מ רקמות הסינית האביב הלא etiolated היה גם רצף כדי לקבוע כיצד יעיל הנפתח הוא בהסרת דנ"א unmethylated. פחות מ 1% של הקריאות בספריה חלק גרעיני הנגזר ממופה genomes הפניה organellar, ואילו ~ 45% מכלל הקריאות ממופה הגנום הגרעיני ( איור 4 ). עם זאת, רוב קורא ממופה באופן מתנגש, wסביר להניח כי משקף את רמות גבוהות של misassembly ופיצול בגנום התייחסות גרעינית חיטה. עם זאת, התוצאות מראות כי הנפתח MBD2-Fc יעיל מאוד בהסרת דנ"א organellar unmethylated מ DNA גרעיני מתיל. ראוי לציין כי, כי DNA מועשר organellar הנובע שיטות אלה מכילה תערובת של מיטוכונדריה רצפים chloroplast, ומכיוון הדמיון רצף הנובע העברת הגן העתיק בין האברונים האלה נשארים הגנום שלהם, את המשימה הנכונה של קורא ספציפיים יש לפתור את הגנום באופן ביואינפורמטיבי.
האטיולציה של רקמת העלה לא יכול לשנות את השפע Organell:
באופן מסורתי, רקמות etiolated הם העדיפו בידוד דנטלי צמח המיטוכונדריה על מנת להקטין את רמות הפנולים ועמילנים, אשר עלול להפריע extraction או יישומים במורד הזרם 13. כדי לקבוע אם ניתן לשנות או לשפר את רמות העשרת הגנום של organellar על ידי תנאי צמיחה, הן הרקמות etiolated ולא etiolated היו כפופים פרוטוקול MF ורצף. מעניין, etiolation לא במידה ניכרת לשנות את אחוז הקריאות הממופה גנומים התייחסות organellar ( איורים 3A ו 3B ) או כיסוי לכל בסיס ( איור 3I ) לעומת התנאים שאינם etiolated. אנחנו גם מבודדים DNA DNA באמצעות צנטריפוגה דיפרנציאלי, עם רקמות etiolated ולא etiolated, ואת ההבדל הקטן העשרה נמצא בין הרקמות השונות באמצעות qPCR (נתונים לא מוצג). זה מצביע על כך יותר פיזיולוגית רלוונטיות שאינם etiolated רקמות ניתן להשתמש עבור מחקרים רצף organellar, ללא שינוי ניכר של העשרה.
בקרת איכות מציע את זהMF DNA מתאים ביותר עבור רצף קריאה ארוכה:
כמו רצף קריאה ארוכה הופך נגיש יותר לחוקרים, בידוד של DNA מולקולרית גבוהה משקל הופך חשוב יותר ויותר. כדי להעריך דנ"א organellar מבודדת עם כל שיטה בשלמות ואיכות, PFGE הועסק. הדנ"א הגנומי הכללי נודד בדרך כלל כמרווח מפוזר ב- PFGE, והמשקל המולקולרי נקבע על פי הפרוטוקול וכיצד הדנ"א מאוחסן ומטופל לאחר החילוץ. סך DNA גנומי מבודדים עם טיפים גנומיים יעלה על 50 קילו, אשר אומת באמצעות PFGE ( איור 5 , נתיב 2). סך הדנ"א הגנומי מהטיפים הגנומיים משמש כקלט לתוך ערכת העשרה Microbiome כדי לחלק את הגרעין מן DNA organellar. החלק הגרעיני המתקבל לאחר החלוקה עושה ירידה בגודל, אבל נשאר מרוכז סביב 50 kb ( איור 5 , מסלול 4). זה לא סוRprising, בהתחשב בכך טיפול יחסית מחוספס של חלק הגרעין כמו elution מ MBD2-Fc מחרוזת חרוזים דורש חום ו proteinase K העיכול. בשל המסה מוגבלת, חלק organellar לא היה לרוץ על PFGE, אבל ניתוח לאחר מכן עם TapeStation ציין DNA> 50 kb (נתונים לא מוצג). ה- DNA organellar שהושג עם צנטריפוגה דיפרנציאלי יש מסה ממוצעת של ~ 20 kb, סביר שנגרם על ידי פרוטוקול בידוד organellar המורחבת ואת הבאים מבוססי עמודה דנ"א מיצוי וריכוז. הבידוד מבוססי הדרגתי, ושיטות חלופיות של דנ"א חלופיות עשויות לשמור על גדלים גדולים יותר של דנ"א. ללא קשר, DNA של גודל המתקבל בפרוטוקול זה עשוי לשמש ליצירת 10 או 15-kb רצף קורא אם הטיפול נלקח במהלך הכנת הספרייה.
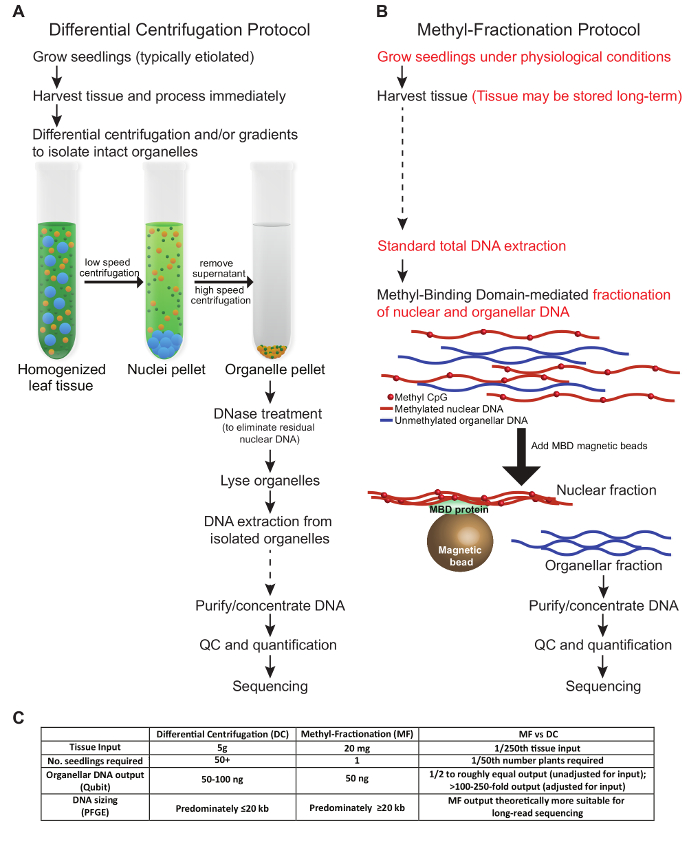
איור 1: מבט השוואתי על שני מת'וDs להעשיר לצמח DNA Organellar. פרוטוקול DC מסורתי ( A ) מנוגד לפרוטוקול MF ( B ). מומלץ להימנע מקפאה והפשרה של הדגימות; עם זאת, הצעדים שבהם הדגימות ניתן לאחסן לטווח ארוך מסומנים עם חצים מקווקווים ( A ו- B ). ההבדלים העיקריים בין הפרוטוקולים מודגשים באדום ( B ). ( ג ) השולחן משווה את השיטות במונחים של קלט רקמות, מספר הצמחים הדרושים, פלט ה- DNA, וכתוצאה מכך גודל ה- DNA. אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.

איור 2: הערכת זיהום DNA גרעיני ב- DNA Organellar מבודד באמצעות שתי שיטות. (
( ב ) הטבלה מציגה את ערכי C q , המוצגים בתרשים ב ( A ), ואת העשרה לקפל של amplicons organellar יחסית ACTIN . * לקפל העשרה = 2 (CQ ACTIN - Cq יעד) . הנוסחה מניחה יעילות מושלמת של 2 עבור כל קבוצה פריימר, מאז deviat קטיןיון של כל צבע יסוד להגדיר מ 2 זניחה תהיה השפעה מועטה על החישוב ואת המגמה הכללית (אקטין = 1.961, NAD3 = 1.95, ו PSBB = 1.989). היעילות פריימר הוערכו על ידי יצירת עקומה סטנדרטית עם סדרה של חמש דילולים 1:10 של הדנ"א הגנומי הכולל. אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.

איור 3: קריאה מיפוי ו סיקור תיאורטית של כלורופלסט ו מיטוכונדריאלי הגנום. אחוז הקריאות הממופות למיטוכונדריה ( A ) או chloroplast ( ב ) גנום התייחסות האביב הסיני. כיסוי תיאורי המקביל של המינגוכונדריה הסינית האביב ( C ) או chloroplast ( D ) התייחסות genoMes, בהנחה גודל הגנום של 450 ו 135 kb, בהתאמה, מחושב באמצעות המספר הכולל לקרוא את אחוז הקריאה מיפוי הגנומים השונים. הפצה רחבה של גנום עבור DNA DNA מ - MF בשיטת MF ( E ו- G ) או בשיטת DC ( F ו- H ). הנתונים לוחות E - H הוא מן המדגם האביב etiolated הסינית, אבל כל דגימות אחרות הראו מגמה דומה. ( I ) ממוצע, הנמוך ביותר, ואת הכיסוי לכל בסיס הבסיסי עבור כל דגימות לוחות A - D. תוויות לדוגמה כולל "E" מיועד דגימות etiolated, ו "NE" מייעד דגימות שאינם etiolated. DC מציין DNA מבודד עם שיטת צנטריפוגה ההפרש Unmethylated מציין DNA כי הוא חלק unmethylated לאחר הנפתח עם MBD2-Fc (פרוטוקול MF). דוגמאות שכותרתו "כריס" לייעד חיטה Triticum aestivum"כריס". CS מייצג דגימות של חיטה Triticum aestivum 'סינית האביב. הערה: בשל הומולוגיה בין רצפי הכלורופלסטים, המיטוכונדריה והגנום הגרעיני, הנובעים מהשינוי הגנטי העתיק בין הגנום האורגנילרי ובין הגנום הגרעיני לבין הגרעין הגרעיני, אחוז קטן של קוראי הגלם עשוי למפות לגנומים מרובים. בנוסף, קורא כי לא מפה או גנום התייחסות organellar אינם מיוצגים נתון זה. לפיכך, האחוזים המוצגים כאן ( A ו- B ) אינם כוללים 100%. אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.

איור 4: PE קרא מיפוי לגנום גרעיני חיטה. אחוז הקטגוריות של PE לקרוא מיפוי סוגי המיטוכונדריה (A) , chloroplast (B) , או גרעיני (ג) גנום התייחסות האביב הסיני. - E מייצג דגימות etiolated ו - NE מייעד דגימות לא etiolated. DC מציין דנ"א מבודד עם שיטת צנטריפוגה ההפרש, Unmethylated מציין DNA כי הוא חלק unmethylated לאחר הנפתח עם MBD2-Fc בפרוטוקול MF, ו Methylated מייעד את החלק הגרעיני לאחר הנפתח MBD2-Fc. דוגמאות שכותרתו "כריס" לייעד חיטה Triticum aestivum 'כריס'. CS מייצג דגימות של חיטה Triticum aestivum "האביב הסיני". קורא לא ממופה לא יוצגו. אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.
Oad / 55528 / 55528fig5.jpg "/>
איור 5: בחינת איכות ה- DNA באמצעות PFGE. חיטה מלאה DNA (נתיב 2), DNA DNA חיטה המתקבל צנטריפוגה ההפרש (מסלול 3), ואת חלק הגרעין לאחר MF עם הגישה הנפתחת MBD2-Fc הנתיב (מסלול 4) היו נתונים PFGE על 1% agarose ג'ל עם 1 kb המורחבת סולם משמש סמן (נתיבים 1 ו -5). אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.
| שם המאגר | מַתכּוֹן | הערות | שִׁיטָה |
| מאגר STE | 400 מ"ג סוכרוז, 50 מ"מ טריס pH 7.8, 20 מ"מ EDTA pH 8.0, 0.6% (w / v) polyvinylpyrrolidone (PVP), 0.2% (w / v) אלבומין בסרום שור (BSA), 00.1% (v / v) β-mercaptoethanol (BME) | תערובת חיץ המכיל רק סוכרוז, Tris, EDTA יכול להיעשות עד חודש מראש ושמרו על 4 מעלות צלזיוס. PVP, BSA, ו BME יש להוסיף טרי aliquot של הכמות הנדרשת של חיץ ממש לפני השימוש. | שיטה מס '1 |
| ST Buffer | 400 מ"ג סוכרוז, 50 מ"מ טריס pH 7.8, 0.6% (w / v) polyvinylpyrrolidone (PVP), 0.1% (w / v) אלבומין בסרום שור (BSA) | תערובת חיץ המכיל רק סוכרוז ו Tris יכול להיעשות עד חודש מראש ושמרו על 4 מעלות צלזיוס. שים לב כי מאגר ST אינו מכיל EDTA או BME, והוא מכיל ריכוז נמוך יותר של BSA. | שיטה מס '1 |
| מלאי DNase | 2 מ"ג / מ"ל DNase ב 0.15 M NaCl לריכוז מלאי של 2 מ"ג / מ"ל | חנות 200 aliquots ul ב -20 מעלות צלזיוס. כדי להכין פתרון עבודה DNase (200 μl של פתרון DNase לכל מדגם) לראותטבלה 1 להלן. עיין בפרוטוקול המלא להלן לקבלת פרטים מלאים על עיכול DNase. פתרון עבודה DNase צריך להיות מוכן טרי. כדי לעצור את התגובה DNase 400 מ"מ EDTA pH 8.0 פתרון נדרש (הריכוז הסופי הנדרש כדי לעצור את התגובה הוא 0.2 M EDTA, ראה פרוטוקול מלא לפרטים). | שיטה מס '1 |
| פתרון עבודה DNase | 0.25 מ"ג / מ"ל DNase ו 20 מ"מ MgCl 2 ב ST Buffer | הכן טרי, 200 ul לכל מדגם. ריכוז המוצגים הם עבור נפח התגובה הסופי, כך לערבב: 62.5 μl 2 מ"ג / מ"ל DNase (מבוסס על 500 נפח התגובה μl הסופי), 4 μl 1M MgCl 2 (מבוסס על 200 μl נפח פתרון DNase), ו 133.5 μl של חיץ ST עבור נפח סופי של 200 μl. | שיטה מס '1 |
| מאגר תמוגה | 20 מ"מ EDTA pH 8.0; 10 מ"מ טריס pH 7.9; 500 מ"מ Guanidine-HCl; 200 מ"מ NaCl; 1% טריטון X-100; 0.5 מ"ג / מ"ל אנזימים lysing מTrichoderma harzianum | מערבבים את כל החומרים למעט אנזימים lysing ולאחסן בטמפרטורת החדר. Lysing אנזימים יש להוסיף טרי aliquot קטן לשימוש מיידי. | שיטה מס '2 |
טבלה 1: מתכונים של מאגרים תוצרת בית ומלאי עבודה.
| גיליון עבודה ריכוז | |||||||
| SAMPLE NAME | משקל מכשיר ריק (g) | משקל של מכשיר מלא (g) | נפח מלא (ul, מלא משקולות ריקות) | משקל לאחר הספין הראשון (20 דקות *, g) | נפח לאחר הספין הראשון (ul, מלאללא משקולות ריקות) | משקל לאחר הספין השני (X דקות *, ז) | נפח לאחר הספין השני (ul, מלא מינוס משקולות ריקות) |
| שים לב כי נפח התאושש בפועל יהיה כמה ul פחות נפח מחושב. | |||||||
טבלה 2: גליון ריכוז.
| שֵׁם | גנום ספציפיות | מקור ג 'ין רצף | רצף (5 '- 3') |
| Ta_ACTIN - F | גַרעִינִי | Gramene Scaffold IWGSC_CSS_1AS_scaff_3272162: 10,663-12,557 | CAGGTATCGCTGACCGTATGA |
| Ta_ACTIN - R | גַרעִינִי | כנ"ל | GAAGGTAGGGCTGAACAAGAAAC |
| Ta_NAD3 - F | מיטוכונדריה | הצטרפות NCBI EU534409.1 | GGTGATGCCAGAAGTCGTTT |
| Ta_NAD3 - R | מיטוכונדריה | כנ"ל | CAGATCAATCTTGTTAGGAGGGACTGG |
| Ta_PSBB - F | כלורופלסט | הצטרפות NCBI | GCTACCTTTGCTTTGCTCTTCT |
| Ta_PSBB - R | כלורופלסט | כנ"ל | GCTGCCTGTTTCCTTGTAGTTTT |
טבלה 3: רשימת פריימרים qPCR.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
עד כה, רוב מחקרים ברצף organellar מרכז בשיטות DC המסורתית להעשיר עבור DNA ספציפיים. שיטות לבודד אורגנים ממגוון צמחים תוארו, כולל מוס 40 ; מונוקולטים כגון חיטה 15 ו שיבולת שועל 11 ; ו dicots כגון arabidopsis 11 , חמניות 17 , ו rapeseed 14 . רוב הפרוטוקולים מתמקדים רקמת העלה 13 , 14 , 15 , 16 , 17 , עם כמה כבר מותאם עבור מגוון רחב של סוגי רקמות, כולל זרעים 11 . בידוד של האברונים מן protoplasts הוכח גם 41 . עם זאת, זה לא מקובל לכל המערכות, וגם זה לא ריאלי כאשר הרקמה של עניין מוגבל. רבים מאלהשיטות בידוד nellar נועדו לשחזר organelles שלם עבור ניסויים ספציפיים, כגון מחקרים פיזיולוגיים. פרוטוקולים אלה הם מסורבלים בדרך כלל דורשים את השימוש של צפיפות gradients, כגון סוכרוז או Percoll gradients, אשר יעילים מאוד לבודד שברים ספציפיים organellar אבל דורשים קלט רקמות גדולות ( כלומר, מעל 5 גרם ומעלה של ק"ג, תלוי סוג הרקמה). עם זאת, השיטה DC יכול להיות מותאם להעשיר עבור שברים מסוימים הסלולר, כגון המיטוכונדריה או chloroplast, על ידי שינוי מהירויות ספין ו gradients צפיפות. לעומת זאת, גישת ה- MF דורשת הרבה פחות חומר התחלתי (20 מ"ג), אך דנ"א מיטוכונדריאלי ופלסטיד יהיה נוכח לכל השפע היחסי שלהם ברקמות המשמשות להפקת דנ"א. עם זאת, פרוטוקול MF מציע גישה חלופית לבידוד DNA DNA מעורבת והוא מועיל במיוחד עבור החל עם כמויות קטנות של רקמות.
T O להעריך טוהר מדגם בעקבות בידוד organelle, רוב המחקרים עד כה רק להשתמש בנקודת קצה PCR וג 'ל אלקטרופורזה 11 , 12 . זה נותן מדד איכותי הוגן של טוהר המדגם. עם זאת, רמות נמוכות של הגברה לא יכול להיות דמיינו על ג 'ל agarose. מעטים הדיווחים כוללים אמצעים כמותיים יותר של בקרת איכות, כגון QPCR 14 . עבור הערכה כמותית של טוהר דגימת DNA מבודדת בשתי השיטות, השתמשנו qPCR ורצף כדי לקבוע כמה DNA גרעיני נשאר במדגם, כמו גם את הפרופורציות היחסי של מיטוכונדריה לעומת DNA chloroplast. שתי השיטות המוערכות כאן יעילות בהוצאת DNA גרעיני. שתי השיטות מניבות שילוב של DNA מיטוכונדריאלי וצ'לורופלסט, אם כי בממדים שונים.
גידול צמחים בחושך (etiolation) הוא דיווח כדי לסייע להקל על בידוד organellar בשל הפחתת הפנוליםעם זאת, בהשוואה זו לא מצאנו יתרון ניכר לעבוד עם רקמות etiolated על דגימות האור גדל.למרות ששיעור chloroplasts המקצועית צפויה להיות גבוהה יותר כאשר קל, המספר הפלסטטי הכולל, כמו המשקף את שיעור הקריאה מיפוי לגנום chloroplast, הוא ללא שינוי בתנאי אור שונים.לכן, עבור ניתוח תפקודי במורד הזרם, כגון הערכה של heteroplasmy ברקמות שונות או תחת לחץ שונים או עבור ניתוחי ביטוי, אנו ממליצים לבצע רצף גנומי על צמחים הגדלים בתנאים פיזיולוגיים רלוונטיים.
עבור יישום עם טכנולוגיות קצוב לקרוא רצף, הן טכניקות לעומת כאן תשואה כמות DNA נאותה ואיכות. עם זאת, כדי להשיג קריאה ארוכה של> 20 kb עבור יישומים מולקולה אחת רצף, כמות גדולה יותר של DNA באיכות גבוהה יותר יש צורך. לדוגמה, באופן אידיאלי,> 1 מיקרוגרם של אורגא טהורNellar חיטה DNA עם משקל מולקולרי> 20 קילו נדרשת עבור בתוך הבית, קלט נמוך פרוטוקולים 20-kb להוסיף הספרייה ההכנות 42 . פרוטוקולים חדשים שפותחו על-ידי המשתמש, עשויים להפחית את דרישות ה- DNA ( כלומר ל -50 ng או אפילו פחות מ -20 ), אך האתגר נשאר להיות בעל איכות גבוהה, DNA מולקולרי במשקל גבוה, ההולך להכנות הספרייה. זה חיוני כי רוב הדנ"א הוא> 20 קילו, כמו שברים קטנים יותר יוכנסו מועדף לתוך SMRTbell ו לזרוק את הפצה גודל של הספריה 43 . ניסינו מספר פרוטוקולים להפקת דנ"א תוצרת בית ומספר פרוטוקולים מסחריים להפקת דנ"א (לא מוצג). עבור רקמות עלה החיטה, האיזון הטוב ביותר בין כמות DNA ואיכות, במיוחד אורך, הושג באמצעות ערכת מסחרי 27 , 29 . בהתאם הצמחים מינים ורקמות של עניין, alternatiפרוטוקולים מיצוי יש עשוי להיות מתאים או יותר פורה. עם זאת, אנו מסיקים כי סך החילוץ של משקל מולקולרי גבוה גנומי DNA> 50 kb בגודל, ואחריו חלוקה עם הגישה הנפתחת MBD2-Fc 28 , הוא מקובל רצף קריאה ארוכה מחומר המוצא מוגבל. עבודה עתידית צריך לבדוק את גבולות החומר ההתחלתי הנדרש לאחר שבירה עבור הכנה ארוכה הכנה ספריה רצף קריאה ארוכה ארוכה. באופן ביקורתי, גישה זו יכולה לספק שיטה חזקה לבודד את הדנ"א מדגם משנה של עלה בודד שמתאים לריצוף ארוך, ללא הגברה של הגנום כולו. אנו צופים כי גישה זו תהיה להתאמה בקלות סוגי רקמות נוספות החלים באופן נרחב על מיני צמחים אחרים. זה יהיה שימושי במיוחד במצבים שבהם כמויות רקמה מגבילים, כגון רצף בדורות בודדים במעבר או סוגי רקמות נדירים.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
המחברים מצהירים כי אין להם אינטרסים מתחרים.
אזכור שמות מסחריים או מוצרים מסחריים בפרסום זה נועד אך ורק לצורך מתן מידע ספציפי ואינו מרמז על המלצה או אישור על ידי משרד החקלאות האמריקאי. USDA היא הזדמנות שווה ספק ומעסיק.
Acknowledgments
ברצוננו להודות במימון של מחלקת החקלאות של ארצות הברית - החקלאות שירות המחקר ו מן הקרן הלאומית למדע (IOS 1025881 ו IOS 1361554). אנו מודים ר 'Caspers עבור תחזוקת החממה טיפול הצמח. אנו מודים גם על אוניברסיטת מינסוטה גנומיקה מרכז, שם ההכנות הספרייה Illumina ורצף בוצעו. אנו גם אסירי תודה על הערות עורכי היומן וארבעה מבקרים אנונימיים שחיזקו עוד יותר את כתב היד שלנו. אנו גם מודים ל- OECD על מלגה ל- SK לשלב פרוטוקולים אלה לפרויקטים משותפים עם עמיתים ביפן.
Materials
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol (beta-mercaptoethanol; BME) | Sigma Aldrich | M3148-100ml | |
| 2-propanol (Isopropyl alcohol/isopropanol), bioreagent | Sigma Aldrich | I9516 | |
| agarose, Bio-Rad Cetified Megabase agarose | Bio-Rad | 1613108 | |
| analytical balance | Mettler Toledo | AB54-S | |
| balance | Mettler Toledo | PB1502-S | |
| bovine serum albumin (BSA) | Sigma Aldrich | B4287-25G | |
| Ceramic grinding cylinders, 3/8in x 7/8in | SPEX SamplePrep | 2183 | |
| Cryogenic Blocks compatible with tissue homogenizer for holding 50 mL tubes | SPEX SamplePrep | 2664 | |
| DNaseI | Sigma | DN25 | |
| ethanol, absolute | Decon Laboratories | 2716 | |
| Ethylenediamine Tetraacetic Acid (EDTA), 0.5 M Solution, pH 8.0 | Fisher | BP2482-500 | |
| gel imaging system | |||
| gel stain | Such as GelRed or Ethidium Bromide | ||
| grinding pestle, wide tip for 2 mL conical tubes | |||
| Guanidine-HCl, 8 M solution | ThermoFisher | 24115 | |
| LightCycler 480 SYBR Green I Master | Roche | 4707516001 | |
| liquid nitrogen | |||
| Lysing enzymes from Trichoderma harzianum | Sigma | L1412 | |
| Magnesium Chloride | G Bioscience | 24115 | |
| magnetic rack | ThermoFisher | A13346 | |
| microcentrifuge tubes, LoBind 1.5 mL | Eppendorf | 22431021 | |
| microcentrifuge tubes, standard nuclease-free 1.5 mL | Eppendorf | ||
| microcentrifuge, refrigerated | Sorvall | Legend X1R | Or equivalent product, must be capable of reaching at least 18,000 x g with rotors for 50 mL tubes, Oak Ridge tubes, and 1.5 mL tubes |
| microcentrifuge, room temperature | Eppendorf | 5424 | Or equivalent product, must be capable of reaching at least 18,000 x g with rotor for 1.5 mL and 2 mL microcentrifuge tubes |
| Microcon DNA Fast Flow Centrifugal Filter Units | EMD Millipore | MRCFOR100 | |
| Miracloth, 1 square per sample cut to fit funnel | EMD Millipore | 475855 | |
| NEBNext Microbiome DNA Enrichment Kit | New England Biolabs | E2612L | |
| parafilm | Parafilm M | PM992 | |
| plastic pots and trays | |||
| polyvinylpyrrolidone (PVP) | Fisher | BP431-100 | |
| Proteinase K | Qiagen | 19131 | |
| Pulsed-Field Gel Electrophoresis rig (e.g. CHEF DR III) | Bio-Rad | 1703697 | |
| purification beads, Agencourt AMpureXP beads | Beckman Coulter | A63881 | |
| QIAamp DNA Mini Kit | Qiagen | 51304 | |
| Qiagen 20/g Genomic Tip DNA Extraction Kit | Qiagen | 10223 | |
| Qiagen Buffer EB (elution buffer) | Qiagen | 19086 | |
| Qiagen DNA Extraction Buffer Set | Qiagen | 19060 | |
| QiaRack | Qiagen | 19015 | |
| qPCR machine (e.g. Roche Light Cycler 480) | Roche | ||
| qPCR plate sealing film | Roche | 4729757001 | |
| qPCR plate, 96 well plate | Roche | 4729692001 | |
| Qubit assay tubes | Life Technologies | Q32856 | |
| Qubit Broad Spectrum assay kit | Life Technologies | Q32850 | |
| Qubit High Sensitivity assay kit | Life Technologies | Q32851 | |
| RNaseA | Qiagen | 19101 | |
| Serological pipettes (20 mL) and pipet-aid | Fisher | 13-678-11 | |
| Small funnels, 1 per sample | |||
| Sodium Chloride | Ambion | AM9759 | |
| Soft paintbrush, 2 per sample | |||
| SPEX SamplePrep 2010 Geno/Grinder or another type of tissue homogenizer | SPEX SamplePrep | Or another comparable tissue homogenizer. If you do not have access to a tissue homogenizer, then grinding in a pre-chilled mortar and pestle will suffice (see protocol for details). However, a homogenizer will give more consistent results and total homogenization time is reduced. | |
| Sucrose | Omnipure | 8550 | |
| TBE | |||
| thermomixer | |||
| Tris | Sigma | T2819-100ml | |
| Triton X-100 | Promega | H5142 | |
| tube rotater | |||
| tubes, 50 mL conical polypropylene | Corning | 352070 | |
| tubes, 50 mL high-speed polypropylene | ThermoScientific/Nalgene | 3119-0050 | e.g. Nalgene Oakridge tubes or equivalent |
| vermiculite | |||
| water bath | |||
| water, sterile and certified Nuclease-free | Fisher | 1481 | |
| water, sterile milliQ |
References
- Liberatore, K. L., Dukowic-Schulze, S., Miller, M. E., Chen, C., Kianian, S. F. The role of mitochondria in plant development and stress tolerance. Free Radic Biol Med. 100, 238-256 (2016).
- Samaniego Castruita, J. A., Zepeda Mendoza, M. L., Barnett, R., Wales, N., Gilbert, M. T. Odintifier--A computational method for identifying insertions of organellar origin from modern and ancient high-throughput sequencing data based on haplotype phasing. BMC Bioinformatics. 16 (232), 1-13 (2015).
- Zhang, T., Zhang, X., Hu, S., Yu, J. An efficient procedure for plant organellar genome assembly, based on whole genome data from the 454 GS FLX sequencing platform. Plant Methods. 7 (38), 1-8 (2011).
- Wambugu, P. W., Brozynska, M., Furtado, A., Waters, D. L., Henry, R. J. Relationships of wild and domesticated rices (Oryza AA genome species) based upon whole chloroplast genome sequences. Sci Rep. 5 (13957), 1-9 (2015).
- Iorizzo, M., et al. De novo assembly of the carrot mitochondrial genome using next generation sequencing of whole genomic DNA provides first evidence of DNA transfer into an angiosperm plastid genome. BMC Plant Biol. 12 (61), 1-17 (2012).
- Park, S., et al. Complete sequences of organelle genomes from the medicinal plant Rhazya stricta (Apocynaceae) and contrasting patterns of mitochondrial genome evolution across asterids. BMC Genomics. 15 (405), 1-18 (2014).
- Skippington, E., Barkman, T. J., Rice, D. W., Palmer, J. D. Miniaturized mitogenome of the parasitic plant Viscum scurruloideum is extremely divergent and dynamic and has lost all nad genes. Proc Natl Acad Sci U S A. 112 (27), E3515-E3524 (2015).
- Wicke, S., Schneeweiss, G. M. Chapter 1. Next Generation Sequencing in Plant Systematics. Hörandl, E., Appelhans, M. , Koeltz Scientific Books. (2015).
- Sloan, D. B. One ring to rule them all? Genome sequencing provides new insights into the 'master circle' model of plant mitochondrial DNA structure. New Phytol. 200 (4), 978-985 (2013).
- Woloszynska, M. Heteroplasmy and stoichiometric complexity of plant mitochondrial genomes--though this be madness, yet there's method in't. J Exp Bot. 61 (3), 657-671 (2010).
- Ahmed, Z., Fu, Y. B. An improved method with a wider applicability to isolate plant mitochondria for mtDNA extraction. Plant Methods. 11 (56), 1-11 (2015).
- Ejaz, M., et al. Comparison of small scale methods for the rapid and efficient extraction of mitochondrial DNA from wheat crop suitable for down-stream processes. Genet Mol Res. 13 (4), 10320-10331 (2014).
- Eubel, H., Heazlewood, J. L., Millar, A. H. Isolation and subfractionation of plant mitochondria for proteomic analysis. Methods Mol Biol. 355, 49-62 (2007).
- Hao, W., Fan, S., Hua, W., Wang, H. Effective extraction and assembly methods for simultaneously obtaining plastid and mitochondrial genomes. PLoS One. 9 (9), e108291 (2014).
- Pomeroy, M. K. Studies on the respiratory properties of mitochondria isolated from developing winter wheat seedlings. Plant Physiol. 53 (4), 653-657 (1974).
- Taylor, N. L., Stroher, E., Millar, A. H. Arabidopsis organelle isolation and characterization. Methods Mol Biol. 1062, 551-572 (2014).
- Triboush, S. O., Danilenko, N. G., Davydenko, O. G. A method for isolation of chloroplast DNA and mitochondrial DNA from Sunflower. Plant Mol Biol Rep. 16 (2), 183-189 (1998).
- Pinard, R., et al. Assessment of whole genome amplification-induced bias through high-throughput, massively parallel whole genome sequencing. BMC Genomics. 7 (216), 1-21 (2006).
- Lamble, S., et al. Improved workflows for high throughput library preparation using the transposome-based Nextera system. BMC Biotechnol. 13 (104), 1-10 (2013).
- Raley, C., et al. Preparation of next-generation DNA sequencing libraries from ultra-low amounts of input DNA: Application to single-molecule, real-time (SMRT) sequencing on the Pacific Biosciences RS II. bioRxiv. , (2014).
- Tsai, Y. C., et al. Resolving the Complexity of Human Skin Metagenomes Using Single-Molecule Sequencing. MBio. 7 (1), e01948 (2016).
- Feehery, G. R., et al. A method for selectively enriching microbial DNA from contaminating vertebrate host DNA. PLoS One. 8 (10), e76096 (2013).
- Yigit, E., Hernandez, D. I., Trujillo, J. T., Dimalanta, E., Bailey, C. D. Genome and metagenome sequencing: Using the human methyl-binding domain to partition genomic DNA derived from plant tissues. Appl Plant Sci. 2 (11), 1-6 (2014).
- Noyszewski, A. K., et al. Accelerated evolution of the mitochondrial genome in an alloplasmic line of durum wheat. BMC Genomics. 15 (67), 1-16 (2014).
- Qiagen. QIAamp DNA Mini and Blood Mini Handbook. , 5th ed, Available from: https://www.qiagen.com/ch/resources/ (2016).
- E.M. Corporation. User Guide: Microcon Centrifugal Filter Devices. , Available from: http://www.emdmillipore.com/US/en/product/Microcon-DNA-Fast-Flow-Centrifugal-Filter-Unit-with-Ultracel-membrane,MM_NF-MRCF0R100 (2013).
- Qiagen. User developed protocol: Isolation of genomic DNA from plants and filamentous fungi using the QIAGEN Genomic-tip - (EN). , Available from: https://www.qiagen.com/ch/resources/ (2001).
- New England BioLabs, Inc.. NEBNext Microbiome DNA Enrichment Kit: Instruction Manual Version 4.0. , Available from: http://www.neb.com/~/media/Catalog/All-Products/371BCB5A557C462D95D1E45E15BBFEA3/Datacards or Manuals/E2612Manual.pdf (2015).
- Qiagen. QIAGEN Genomic DNA Handbook. , Available from: https://www.qiagen.com/ch/resources/ (2012).
- PacificBiosciences. Guidelines for Using the BIO-RAD® CHEF Mapper® XA Pulsed Field Electrophoresis System. , Available from: http://www.pacb.com/wp-content/uploads/Unsupported-Guidelines-Using-BIO-RAD-CHEFMapper-XA-Pulsed-Field-Electrophoresis.pdf (2016).
- Andrews, S. FastQC: A quality control tool for high throughput sequence data. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Ogihara, Y., et al. Structural dynamics of cereal mitochondrial genomes as revealed by complete nucleotide sequencing of the wheat mitochondrial genome. Nucleic Acids Res. 33 (19), 6235-6250 (2005).
- Ogihara, Y., et al. Structural features of a wheat plastome as revealed by complete sequencing of chloroplast DNA. Mol Genet Genomics. 266 (5), 740-746 (2002).
- International Wheat Genome Sequencing Consortium (IWGSC). A chromosome-based draft sequence of the hexaploid bread wheat (Triticum aestivum) genome. Science. 345 (6194), (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nat Methods. 9 (4), 357-359 (2012).
- Bendich, A. J. Why do chloroplasts and mitochondria contain so many copies of their genome? Bioessays. 6 (6), 279-282 (1987).
- Kumar, R. A., Oldenburg, D. J., Bendich, A. J. Changes in DNA damage, molecular integrity, and copy number for plastid DNA and mitochondrial DNA during maize development. J Exp Bot. 65 (22), 6425-6439 (2014).
- Ma, J., Li, X. Q. Organellar genome copy number variation and integrity during moderate maturation of roots and leaves of maize seedlings. Curr Genet. 61 (4), 591-600 (2015).
- Lang, E. G., et al. Simultaneous isolation of pure and intact chloroplasts and mitochondria from moss as the basis for sub-cellular proteomics. Plant Cell Rep. 30 (2), 205-215 (2011).
- Tobin, A. K. Subcellular fractionation of plant tissues. Isolation of chloroplasts and mitochondria from leaves. Methods Mol Biol. 59, 57-68 (1996).
- PacificBiosciences. Procedure & Checklist - 10 kb to 20 kb Template Preparation and Sequencing with Low (100 ng) Input DNA. , Available from: http://www.pacb.com/wp-content/uploads/Procedure-Checklist-10-20kb-Template-Preparation-and-Sequencing-with-Low-Input-DNA.pdf (2015).
- PacificBiosciences. Template Preparation and Sequencing Guide. , Available from: http://www.pacb.com/wp-content/uploads/2015/09/Guide-Pacific-Biosciences-Template-Preparation-and-Sequencing.pdf (2014).



