Summary
여기에서 우리는 생활 C 에서 고해상도 현미경, 전산 도구 및 단일 세포 라벨링을 사용 하 여 조합 접근법을 제시 합니다. 신경 발달 중에 단일 세포 역학을 이해 하는.
Abstract
아닌, 캘리포니아 (C.엘 레 강 스)는 전체 신 경계의 세포 기원을 이해 하는 도전이 생체 내에서 단일 세포 분해능과 함께 관찰 될 수 있는 유일한 유기 체로 서 두드러집니다. 여기서 우리는 c. 엘 레 강 스 배아에서 신경 발달 검사를 위한 통합 프로토콜을 제시 합니다. 우리의 프로토콜은 개발 배아에서 단일 세포의 이미징, 선 노화 및 신경 해부학 적 추적을 결합 합니다. 살아있는 C. 이중 보기 반전 선택적인 평면 조명 현미경 검사 법 (diSPIM)을 사용 하 여 거의 등방성 공간 해상도를 가진 엘 레 건스 배아의 장기, 4 차원 (4d) 이미지를 얻습니다. 선 충 배아의 핵 및 뉴런 구조는 3 차원 모두에서 ~ 330 nm의 분해능으로 이미지를 산출 하 고이에이 트로피 칼 융합을 이미지화 한다. 이 분 별 고 분해능 4D 데이터 세트는 단일 셀 및 하위 셀룰러 수준의 세부 수준에서 유전자 발현 및 형태학 적 역학과 함께 확실 한 세포 계보 정체성을 연관 시키기 위해 분석 됩니다. 당사의 프로토콜은 설명 된 각 단계의 모듈식 구현을 가능 하 게 하 고 배아 발생, 유전자 발현 또는 신경 발달에 대 한 연구를 강화 하도록 구성 되어 있습니다.
Introduction
C. 엘 레 강 스는 배아의 모든 세포가 신경 발달을 통해 관찰 될 수 있는 유일한 유기 체로 서 두드러집니다. 전체 세포-계보 알려진 불변1, 그리고 배아에서 단일 세포의 라벨링 및 연속 이미징을 허용 하는 새로운 도구의 개발과 함께, 생물학자는 이제 신경 세포의 개발에 다른 단계를 검토 시작할 수 있습니다 긴장 모든 각도에서 시스템-세포 출생; 이주 및 분화; 신경 돌기 형성, 표적으로 한 성장 및 근 막; 시 냅 스 형성; 및 기능 회로의 튜닝. 안정적으로 발현 된 기자와 형광 현미경을 결합 하 여 c. 엘 레 강 스 배아에서 뉴런의 성장 역학을 포착 하는 것은 과학적 공동체에 게 가치가 있습니다.
C. 엘 레 강 스에서의 발달 연구는 무 손상 유기 체1내의 단일 세포 수준에서 문맥 이해를 증강 하기 위하여 수시로이 종의 불변 세포-혈통 그리고 세포 운명 지도를 활용 합니다. 자동 라인 에이징 분석-starrynite2,3,4 및 아 세 리5,6,7,8 소프트웨어 사용-고대비, 고해상도의 이점 형광 핵의 이미지. 최적으로 작동 하려면, 스타 린 트/아 세토 또한 개발 중에 이미지 배아의 예측 가능한 제한 방향에 따라 달라 집니다. 두 커버 슬립 사이에 압축 된 c. 엘 레 강 트 배아에서 수행 된 공초점 현미경 검사 법은 고 콘트라스트/고 분해능과 예측 가능한 제약을 모두 제공 하기 때문에 10 년 이상 표준 자동 라인 에이징 현미경 법입니다. 배아의 방향7,8. 우리는 이전에 신규 한 광 시트 기반의 이중 보기 거꾸로 선택적 평면 조명 현미경 (dispim)의 건설 및 사용을 설명 하는 등의 라이브 샘플 이미징 c. 엘 더 니스 배아 발생 발생9,10 , 11 , 12 , 13. 가벼운 시트 현미경은 일반적으로 라이브 3d 표본14,15의 낮은 광 독성, 고속 및 장기 이미징 기능을 제공 합니다. DiSPIM 방법은 특히 거의 등방성 공간 해상도가 약 330 nm9의 4 차원 (4d) 이미지를 생성 합니다.
DiSPIM은 공초점 현미경과 비교 하 여 더 높은 신호 대 잡음비와 속도, 등방성 공간 분해능을 제공 하며 장기 생체 이미징16에 더 적합 합니다. 따라서 우리는 StarryNite/아 세이 어에 입력을 위한 diSPIM 데이터를 조정 하 고 이것이 선 에이징 분석을 향상 시킬 수 있는지 여부를 조사 했습니다. 주요 장애물은 diSPIM 표본이 StarryNite/비구에 대 한 예상 방향을 채택 하기 위해 계란-압축에 의해 쉽게 구속 될 수 없다는 것입니다. 분석 중인 볼륨에서 셀 위치의 임의 방향은 자동 라인 에이징 분석의 정확도를 저하 시킵니다.
따라서 사용자가 diSPIM 이미지의 전처리 중에 배아의 정확한 3D 방향을 선택할 수 있는 뷰어 안내 사용자 인터페이스인 사이토 쇼를 채택 하 여 품질에 최적화 된 이미지 데이터를 산출 하 고 StarryNite에 입력을 위한 상황 인식 /비구. 이미지 배아의 사용자 선택에 따라, 사이토 쇼는 자동화 된 데이터 처리 파이프라인을 조율 합니다. 자른 및 배경 뺀 배아 이미지는 각 위치, 타임 포인트 및 보기에 대 한 TIFF 스택 파일 내에 저장 됩니다. 그런 다음, 사이토 쇼는 프로그램 spimfusion을 반복적으로 호출 하 여, 리차드 슨-루시17,18 알고리즘을 사용 하 여 등방성 고해상도 체적 이미지를 산출 하는 두 개의 사전 처리 된 뷰를 공동 등록 하 고 공동으로 해체 합니다. DiSPIM 특정 매개 변수 집합은 이미지 세분화 및이에이 트로피 칼 융합 이미지의 핵 추적 중에 동작을 제어 하기 위해 StarryNite에 최적화 되었습니다. 융합 된 이미지와 lineaging 결과는 사용자가 StarryNite에 의해 생성 된 자동 계보 추적의 오류를 식별 하 고 수정할 수 있는 아 세 트리를 사용 하 여 편집 됩니다. 아 세토 트리는 또한 배아에서 추적 된 핵의 계보 트리와 3D 모델링 렌더링을 제시할 수 있습니다. 우리는 자동 라인 에이징 속도와 정확도가 모두 SPIM 카메라의 raw 이미지와 비교할 때이 트로피 칼 융합 이미지를 사용 하 여 현저 하 게 향상 된 것을 발견 합니다. 여기에 설명 된 c. 엘 더 스 응용 프로그램에 최적화 된 우리의 프로토콜은 일반적으로 다른 종이나 표본에 대해 생성 된 diSPIM 데이터의 자동 라인 에이징에 적합 할 수 있습니다. 이 프로토콜이 의도 된 용도로 사용 되는 경우에는3,4와 같이 새 표본에 starrynite 매개 변수의 추가 조정이 필요할 수 있음을 유의 하십시오.
이 프로토콜의 성공적인 구현은 4D 등방성 해상도로 이미지를 구현 하 고 생물학자가 세포 라인을 추적 할 수 있게 하는 동시에 개발 C. 엘 레 강 스 배아에서 뉴런을 식별 하 고 분석 합니다. 또한, 몇 가지 후 처리 알고리즘을 병합 하 여 하드웨어 가속이 가장 많은 시간을 소비 하는-우리는 이제 본질적으로 실시간으로 미세 세포의 세부 사항과 살아있는 배아 세포의 운명 모두를 분석 할 수 있습니다. 이 새로운 프로토콜은 생체 내에서 분화 및 형태 형성을 연구 하는 동안 세포 거동을 정확 하 고 정보에 입각 한 조작 및 관찰을 가능 하 게 합니다. 이 원고에서 우리는 선 노화 및 세포 추적을 위해 개발 된 개선 된 프로토콜에 대 한 자세한 설명을 제시 합니다 c. 엘 레 강 스 배아를 개발, 배아 발생의 연구를 향상 시키기 위해, 유전자 발현 또는 신경 개발.
Protocol
1. diSPIM 스틸 이미징 챔버를 폴 리-L-라이 신 코팅 커버 슬립으로 조립
참고: 아래 단계는 모두 StarryNite/아 세토에의 한 네 마 충 배아의 연계 분석을 최적화 하 고 자동화 하는 데 필요 합니다. 여러 옵션 (이와 같이 표시 됨)은 C. 엘 더 스 세포의 추적을 필요로 하는 실험을 위해 생략 될 수 있다.
- 샤 프 파이 (또는 이와 유사한 펜)를 사용 하 여 깨끗 한 직사각형 커버 슬립 (1.5, 24mm×50 mm)의 중앙에 작은 직사각형 (2mm x 5mm)을 그립니다.
- 커버 슬립을 뒤집어 놓고 잠복 한 쪽으로 10 μ l의 폴 리 신 (고양이. P1524) 표시 된 사각형 위에 있습니다.
참고: 여과 된 물에 녹여 1 mg/mL 폴 리-L-라이 신의 작동 농도를 확인 하십시오 (또는이에 상응 하는). 장기 보관 시에는 5 ~ 10 mL의 알리 쿠오를 준비 하 고-20°c에 보관 하십시오. 해 동 한 후에는 3 ~ 4 주 동안 상 온 (23°c)에서 알 취 액을 보관할 수 있습니다. - 폴 리-L-라이 신이 커버 슬립을 5 분 동안 코팅 할 수 있도록 한다 (도 1a). 폴 리-L-라이 신은 배아가 장착 되는 유리 커버 전표를 코팅 하는 데 사용 되며, 목적을 이미징 매체에 침 지 하는 경우에도 배아 달걀 껍질이 커버 슬립에 단단히 고정 될 수 있도록 합니다.
- 폴 리-L-라이 신 코팅 커버 슬립을 스틸 이미징 챔버의 하단 절반에 놓습니다.
- 커버 슬립으로 강철 이미징 챔버의 상단 절반을 아래쪽 절반에 놓고 챔버와 연결 된 4 개의 나사로 조입니다. 측면에서 육안으로 확인 하 여 상단 반이 아래쪽 절반에 고르게 장착 되도록 합니다 (그림 1b).
- 초기 단계의 배아를 비정상적인 삼 투 압으로 굴복 예방 하는 데 도움이 되는 등 장성 완충 액 인 7 ~ 8 mL의 M9 버퍼19로 챔버를 채웁니다. 1-세포, 심지어 2 세포 배아는 삼 투 적 민감하고이 등장 완충 액에 있지 않다면 비정상적으로 발전할 수 있다. 또한 M9이 물로 치환 된 경우 배아는 3 배 단계에서 정지 하는 경향이 있습니다.
2. 준비 c. 엘 더 스 배아 설치를 위한 샘플
참고: 이미징 하기 약 18 시간 전, 5 명의 영 (최종 탈피 이후 1 일) 성인과 10 명의 라 발의 스테이지 4 (L4) c. 엘 레 강 스는 대장균 균 주 OP50로 시 딩 된 한 천 판으로 선별 됩니다 . 백 금 철사 후비는 동물을 해치지 않고 애벌레와 청년 성인 c. 엘 더를 이동 하는 데 사용 됩니다19.
- 1% 메 틸 셀 룰로 오 스를 제조 한다 (고양이. H7509) M9 버퍼에 있는 용액.
참고: 메 틸 셀 룰로 오 스는 용 해 될 때까지 따뜻한 M9에서 교 반 해야 합니다. 일단 준비 되 면,이 용액은 실 온에서 보관할 수 있다. - 오목 현미경 슬라이드의 우울증에 1% 메 틸 셀 룰로 오 스 인 M9 용액 500 µ L을 추가 합니다. 이 점성 완충 제는 2 가지 경우에 사용 됩니다: 1) 말기 배아를 NGM 플레이트에서 직접 고른 세척 할 때 성인 웜 및 2의 해 부에 의해 초기 배아를 수확 할 때.
참고: 메 틸 셀 룰로 오 스는 배아가 현미경 슬라이드에 들러붙는 것을 막기 위해 사용 됩니다. - 후반 단계 배아를 이미징 하는 경우에는 속눈썹을 선택 하 고, 배아를 1% 메 틸 셀 룰로 오 스를 오목 현미경 슬라이드에 이동 시켜 서 (바람직하게는 젊은 성인과 함께 NGM 플레이트에서 존재 하는), 선택 된 다이 요 로 배아를 선정 한다. 속눈썹 선택은 힘을 감소 시키고 따라서 취급 도중 배아에 대 한 스트레스 또는 손상을 최소화 하는 데 도움이 됩니다. 속눈썹을 선택 하는 절차는 Hart에 의해 덮여 있다20.
- 두 번째 속눈썹 선택으로 (반대 손에), 메 틸 셀 룰로 오 스에 배아를 일시 중단 하는 두 속눈썹을 함께 부드럽게 누릅니다.
- 선택 사항: StarryNite와 계보 배아 세포를 계획 하는 경우, 하나는 4 세포 배아에 1 세포를 탑재 해야 합니다. 이렇게 하려면 먼저 NGM 플레이트에서 젊은 성인을 선택 하 고 백 금 와이어 선택을 사용 하 여 오목한 현미경 슬라이드에 있는 M9 메 틸 셀 룰로 오 스 용액으로 옮깁니다.
- 선택 사항: 피하 주사 바늘 (no. 18G x 1 ½)의 날카로운 팁을 사용 하 여 중간 몸체에 있는 동물을 슬라이스 하 여 1 세포를 4 세포 배아에 방출 하십시오.
3. 입 피 펫: 미세 모 세관 피 펫으로 흡 기 튜브를 조립
참고: 우리는 튜브의 고무 가스 켓에 삽입 된 손으로 뽑아 마이크로 모 세관 파이 펫과 함께 흡 기 튜브를 사용 합니다. 이것은 우리가 버퍼 채워진 영상 챔버에 있는 폴 리-L-라이 신 코팅 표면에 해 부 슬라이드에서 배아를 전달 하는 것을 허용 한다.
- 열린 불꽃 위에 미세 모 세관 파이 펫을 수동으로 당겨 뻗어 있는 팁으로 두 반쪽을 만듭니다.
- 미세 모 세관 파이 펫의 1/2을 취하고 흡 인기 튜브의 고무 개스킷에 무딘 단 부를 삽입 합니다 (그림 1c). 나중에 사용 하기 위해 미세 모 세관 파이 펫의 나머지 절반을 따로 설정 하십시오 (필요한 경우).
- 미세 모 세관 파이 펫이 장착 된 조립 된 흡 기 튜브를 사용 하 여 미세 모 세관 팁을 부드럽게 분리 하 고 약 1 ~ 2 개의 배아를 넣을 수 있는 개 구를 만듭니다 (여기에서이 계측기는 "입 파이 펫" 이라고 함).
4. 폴 리 신 코팅 커버 슬립에 c. 엘 더 란 트 배아를 장착
- 흡 인기의 마우스 피스를 치아 사이에 부드럽게 고정 한 채 미세 모 세관에 10 ~ 15 µ L의 M9 완충 액을 미리 채우고 오목 슬라이드에서 모 세관으로 여러 배아를 부드럽게 빨 아 줍니다.
- 배아를 M9 버퍼로 채워진 스틸 이미징 챔버로 전달 하 고 모 세관 팁의 위치를 조정 하 여 배아가 커버 슬립의 중앙 직사각형에 들어갈 수 있도록 합니다.
- 배아에 대 한 부상을 방지 하 고, 속눈썹을 선택 하거나 구강 피 펫을 사용 하 여 배아를 수직으로 위치 시키거나, 배아의 긴 축이 커버 슬립의 장 축에 수직이 되도록 배아를 방향으로 이동 시킵니다 (도 1b 삽입 하단 패널).
참고: 배아를이 방향으로 배치 하면 이미지의 슬라이스 수가 최소화 되어 광 량 및 데이터 처리 시간이 줄어들어 획득 속도가 향상 됩니다. - 현미경 스테이지의 샘플 홀더에 스틸 이미징 챔버를 놓습니다 (그림 1d).
5. diSPIM을 사용 하 여 배아 이미징에 대 한 조립, 소프트웨어 설정 및 레이저 최적화
- 쿠마 르 외10,11 및 http://www.dispim.org에서 상업적으로 이용 가능한 부품에서 전체 섬유 결합 dispim을 조립 하는 방법에 대 한 단계별 지침을 참조 하십시오. DiSPIM을 조립 하는 방법의 비디오 프로토콜은 또한 ASI 웹 사이트 (http://www.asiimaging.com)에서 사용할 수 있습니다.
참고:이 프로토콜에 대 한 기기 설치는 이미징 용 40x 0.8 NA 물 침수 렌즈를 사용 하는 쿠마 르 외10,11 dispim과 동일 합니다. 이 프로토콜의 설정과 쿠마 르 외10,11 의 유일한 차이점은이 색 거울 (560 nm에서 분할) 및 적색 및 녹색의 영상 분할 장치 내부 필터 (모델 A12801)에 설치 된 모두 diSPIM 이미징 암. 이미지 분할 광학 장치를 추가 하면 두 개의 뚜렷한 형광 색 단 (561 nm 및 488 nm 레이저)에서 이미지를 동시에 캡처할 수 있으므로 방출 밴드를 동일한 카메라 칩의 두 반쪽으로 분리 합니다. - 계측기 조립 후 이미징 전에 diSPIM의 광학 정렬을 확인 하십시오.
참고: diSPIM을 제대로 정렬 하려면 https://youtu.be/qnOrg30NNuE를 참조 하 고 하드웨어 정보에 대해서는 http://dispim.org/hardware/objectives 및 http://www.asiimaging.com. - 고 처리량 셀룰러 이미징22를 위한 라이트 시트 현미경의 작동에 최적화 된 오픈 소스 플랫폼 마이크로 관리자 (https://micro-manager.org/)21을 사용 하십시오. 최대 30 개의 배아를 동시에 이미징 할 수 있는 다중 위치 수집을 위해 ASI diSPIM 플러그인을 사용 하는 것이 좋습니다 (23).
- 마이크로 관리자를 열고 488 nm 및 79 µW 0.25 (그림 2a, 빨간색 직사각형)에 대 한 179 µW 0.5)로 레이저 강도를 설정 합니다.
참고: 이들은 1 분 간격을 사용 하 여 c. 엘 더 스 배아의 장기 이미징에 대 한 권장 설정입니다. 장기 듀얼 컬러 이미징 동안, 561 nm 레이저는 배아가 빈 단계에 있을 때까지 핵 (Mcherry:: Histone)을 이미지 하는 데 사용 되며,이 시점에서 488 nm 레이저는 또한 GFP 표지 뉴런을 이미지화 합니다. 이러한 이미징 조건은 광 독성을 최소화 하 고 배아의 생존과 부 화를 보장 하는 동시에 신경 발달 및 선 노화 데이터를 장기간 (12 ~ 14 시간) 연속으로 획득할 수 있도록 최적화 되어 있습니다. - 마이크로 관리자에서 메뉴 플러그인 ≫ 장치 제어 ≫ asi dispim 을 선택 하 여 asi Dispim 창을 엽니다 (그림 2b). 획득 탭을 선택 합니다. 이 탭의 데이터 저장 설정 섹션 (녹색 직사각형), 볼륨 설정 섹션 (파란색 정사각형) 및 슬라이스 설정 섹션 (주황색 정사각형)에서 각 매개 변수가 그림 2b에 표시 된 대로 설정 되었는지 확인 합니다.
참고: 우리의 이미지 분석 소프트웨어 사이토 쇼는 마이크로 관리자에 내장 된 내보내기 기능의 사용을 통해 획득 후 대량 연결 오메-TIFF 파일 시리즈 및 TIFF 스택 파일 시리즈와 같은 다른 옵션 출력 데이터 형식으로 작동 하도록 구성 되어 있습니다. 일반적으로 원시 데이터를 획득 하는 즉시 이미지 볼륨을 실시간으로 보고 처리할 수 있기 때문에 단일 시간 포인트 스택 데이터 정렬 기능을 사용 합니다.
6. c. 강 장 배아의 장기 이미징에 최적화 된 자동 초점 매개 변수
- C. 엘 더 스 배아의 장기적인 계보 품질 diSPIM 이미징에 최적화 된 설정으로 마이크로 관리자 자동 초점 매개 변수를 설정 합니다. ASI diSPIM 창에서 자동 초점 탭을 클릭 합니다 (그림 2c). 일반 자동 초점 옵션 섹션 (검정색 정사각형)에서 표시 된 대로 정확 하 게 매개 변수를 지정 합니다. 자동 초점 채널 (빨간색 정사각형)은 lineaging 실험에서 핵 채널 형광 채널을 지정 해야 합니다.
참고: 최대 오프셋이 5 µm 보다 크면 이미지의 초점이 맞지 않는 경향이 있습니다. - 클릭, 플러그인 ≫ 수집 도구 > 패턴 오버레이.
- 패턴 오버레이 창에서 그리드 표시를 클릭 합니다.
- ASI diSPIM 창에서 탐색 탭을 클릭 합니다.
- 경로 A 또는 B의 빔 및 시트 에 대 한 확인란을 클릭 한 다음 라이브를 클릭 합니다. 이미지 수집이 시작 됩니다. 라이브 뷰 창이 열립니다. 6.1에서 선택 된 채널에 배아 주위에 상자를 그려 배아의 자동 초점 분석 영역을 선택 합니다.
참고: 일반적으로 이미징 세션 당 10 태아에 대 한 420 시간 포인트를 캡처합니다. 이미지 세션 당 원시 데이터는 일반적으로 1.7tb 이며, 해체 및 StarryNite 처리 된 데이터는 1.4tb(9 단계 및 10 참조)입니다. 이미지 수집을 위해 대용량 HDD (현재 시스템에 18TB)를 사용 하 고 이미지 저장을 위한 클라우드 플랫폼을 사용할 것을 권장 합니다. - "획득 탭에서 획득 시작 을 클릭 하 여 장기 다차원 이미지 캡처를 개시 합니다 (그림 2b).
7. 세포 쇼에서 원시 마이크로 관리자 이미지 열기
- Http://dispimlineage.wormguides.org에서 소프트웨어 번들을 다운로드 합니다.
참고: 소프트웨어 번들은 .zip 파일로 다운로드 되며 "c: \"로 추출 해야 합니다. 사용 하기 전에 디렉터리를 설치에 대 한 자세한 내용은 http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm에서 제공 됩니다. - 파일 C:\CytoSHOWextrasForC\CytoSHOW_APP.jnlp 를 두 번 클릭 하 여 사이토 쇼 실행을 시작 합니다.
- 새 ≫ Dispim 모니터 (마이크로 관리자)> 메뉴 파일 을 선택 합니다. 획득 timepoint 폴더가 저장 된 루트 데이터 세트 폴더를 찾습니다. Timepoint 폴더를 선택 하 고 열기를 클릭 합니다. Dispim 모니터 창 이라고 하는 다차원 탐색 창은 spima 및 SPIMB 모두에 대해 자동으로 열립니다 (그림 3a).
참고:이 창은 새로 저장 된 원시 timepoint 스택에 대 한 루트 데이터 폴더를 모니터링 합니다 (시 편이 여전히 기록 되 고 있는 경우). 각각의 새로운 timepoint가 획득 되 면 각각의 배아에 대 한 전체 멀티 채널 4D 데이터 셋을 표시 하기 위해 각각의 윈도우에서 뚜렷한 SPIM 암과 샘플 위치를 모니터링 하 게 됩니다.
8. 사이토 쇼로 최대 프로젝션 이미지 생성
심지어 해체 하기 전에 원시 데이터를 신속 하 게 처리 하 여 표본의 글로벌 기능을 칭찬 할 수 있습니다.
- 이미지 창의 왼쪽 패널에 있는 Z-MIP 버튼 (그림 3a, 빨간색 직사각형)을 클릭 하 여 주어진 위치 또는 spim 암의 전체 깊이 및 풀 타임 코스를 통해 최대 광도 투영을 만듭니다. Z 프로젝션 하이퍼스택 창이 표시 됩니다.
- Z 프로젝션 하이퍼스택 창에서 프로젝션 유형을 최대 강도로설정 합니다. 사용자 기본 설정에 따라 처리할 채널, 슬라이스 및 시간 포인트 프레임을 지정 합니다.
- 완료 되 면 확인을 클릭 합니다.
- 파일 대화 창에서 최대 광도 출력을 저장할 폴더 위치를 선택한 다음 확인을 클릭 합니다. 데이터 세트의 크기와 컴퓨터의 처리 능력에 따라 15 ~ 20 분 정도 시간을 두고 프로젝션 이미지를 생성 합니다.
9. 등방성 고 분해능 체적 데이터에서 셀 라인 분석
- 선택 사항: 세포 쇼에서 Dispim 모니터 를 통해 원시 데이터를 열면 다각형 선택 도구 (그림 3a, 검은 색 화살표)를 선택 하 고 배아의 전방, 후방, 등 쪽 및 아랫 배 가장자리의 바로 바깥을 클릭 하 여 배아 위에 "보우 타이" 패턴을 생성 합니다. 두 뷰 모두에 대해 수행 합니다 (SPIM 및 SPIM, 그림 3a).
주:이 선택은 배아가 중심에 있는 타원형 관심 영역 (ROI)을 지정 하 고 배아의 전방 후부 축을 기록 합니다. 보 타이 패턴 큐 토시 쇼는 사용자가 더 나아가 최종이 소부 트로피가 융합 된 체적의 정확한 회전을 StarryNite/비구에의 한 선 에이징 분석에 최적인 방향으로 지정할 계획입니다. StarryNite lineaging가 실험적 계획의 일부가 아닌 경우에는 이미지 처리를 위해 ROI를 설정 하기 위해 다른 선택 도구와 모양을 선택할 수 있습니다. - 다중 배아를 다 위치 획득 옵션을 이용 하 여 동시에 이미지화 한 경우, 모든 배아에 대해 9.1 단계를 열고 수행 한다. 이렇게 하면 한 세션에서 모든 배아에 대 한 향후 단계의 병렬 실행이 가능 합니다. 처리 하 고 싶지 않은 배아에 대해서는 SIMA 및 SPIMB 창을 닫으십시오.
- DiSPIM 모니터 창의 왼쪽 패널에 있는 dispim 버튼을 클릭 합니다 (그림 3a, 노란색으로 강조 표시 됨). 이는 diSPIM 처리와 관련 된 컨트롤의 하위 패널을 나타냅니다.
- 각 SPIM 암에 대해 녹색과 적색 채널을 정렬 합니다. 방출 채널 스플리터는 동일한 카메라에서 동시에 뚜렷한 적색 및 녹색 이미지를 캡처하는 데 사용 되기 때문에,이 두 개의 물리적으로 인접 한 이미지 필드의 정확한 픽셀 정합이 오버레이될 때 시각적으로 정렬 하는 것이 중요 합니다. 동일한 정렬 조정의 재사용은 일반적으로 많은 연속적인 이미징 세션에서 가능 하지만 9.4.1-9.4.5 단계에서와 같이 확인 해야 합니다.
- SPIMA 패널로 시작 하 여 CM 스크롤 막대를 왼쪽으로 이동 하 여 빨간색 채널을 선택 합니다 (그림 3a, 주황색 화살표, 왼쪽 패널).
- X, y 및 z 조정기 (그림 3a, 주황색 정사각형)를 사용 하 여 빨간색 채널을 녹색에 맞게 이동 합니다.
- Dispim 버튼 (그림 3a, 노란색으로 강조 표시 됨)을 클릭 하 여 하위 패널을 닫고 다른 모든 위치 창으로 동일한 교대조의 전파를 트리거합니다.
- "Z" 스크롤 막대를 이동 하 여 다른 프레임과 시간 지점으로 올바른 정렬이 전파 되었는지 확인 합니다(그림 3a, 파란색 화살표, 왼쪽 패널) 및/또는 "t" 스크롤 막대 (그림 3a, 녹색 화살표, 왼쪽 패널). 다중 위치 획득이 수행 되었고 여러 배아 들이 이미지화 된 경우 (단계 5.3), 정렬은 그 배아 들에도 전파 되어야 한다. X, y 및 z-스 터 드의 숫자를 검토 하 여 확인 합니다 (그림 3a, 주황색 정사각형은 모든 배아의 spima 패널에 대해 동일 해야 합니다).
- 9.4.1 단계를 반복 하 여 SPIMB diSPIM 모니터 창 (그림 3a, 오른쪽 패널)에 대해 9.4.4 합니다.
- "Dispim" 버튼을 클릭 한 다음 "퓨즈" 버튼 (그림 3a, 파란색 직사각형)을 눌러 "디스피브/퓨즈 dispim 원시 데이터 볼륨" 이라는 대화 상자를 엽니다. 그림 3b와 같이 매개 변수를 설정 합니다. 이러한 매개 변수는 다음 하위 단계에서 간략하게 다룹니다.
- 채널 1(488 nm 레이저) 또는 2(561 nm 레이저)에 키 등록 을 설정 합니다. 밀도가 크거나 더 많은 유비쿼터스 신호가 있는 채널을 선택 합니다. Lineaging 실험을 위해 항상 유비쿼터스 핵 히스톤 형광을 이미지 하는 데 사용 되는 채널을 선택 합니다.
- 입력 볼륨 상대 방향을 + 1 또는-1로 설정 합니다. 올바른 방향 인덱스는 특정 diSPIM의 카메라 배치에 따라 달라 집니다 (그림 4).
참고: 불확실 한 경우에는 9.1 – 9.12 단계에 따라 각 옵션을 SPIM A 및 B Dispim 모니터 창에서 복제 하 여 테스트에 대 한 상대 방향 입력 볼륨 을 임의로 선택 합니다. 방향이 잘못 된 경우 아티팩트를 사용 하 여 흐릿한 이미지가 생성 되지만 올바른 방향은 선명한 이미지를 생성 합니다. 그런 다음 명확한 이미지를 생성 하는 입력 볼륨 상대 방향 값을 지정 된 diSPIM 계측기의 모든 향후 데이터에 재사용할 수 있습니다. - 융합 볼륨을 사용자 기본 설정에 따라 A 또는 B 입력 볼륨과 동일한 방향으로 설정할지 여부를 선택 합니다.
- "모든 볼륨에 대해 새로 등록"을 선택 합니다. 이 옵션은 SpimFusion에서 각 timepoint의 각 볼륨 쌍에 대 한 등록 행렬을 계산 하는 방법을 제어 합니다. "Fresh" 옵션을 사용 하면 알고리즘이 각 타임 포인트에서 등록을 최적화할 수 있습니다.
- 1 번의 분해 반복 횟수를 10으로 설정 합니다. 이 숫자는 시간 효율적인 방식으로 원하는 높은 해상도를 안정적으로 산출 하는 경향이 있습니다.
- 선택 사항: 자동 라인 에이징 (적극 권장)이 필요한 경우 퓨즈 볼륨의 자동 발사 StarryNite 라인 에이징을 선택 하십시오. 이 옵션은 StarryNite를 자동으로 시작 하 여 SpimFusion에 의해 생성 된 이미지 볼륨의 셀을 세분화 하 고 추적 합니다.
- 선택 사항: 자동화 된 선 에이징에서 가장 높은 정확도를 위해이 소 트로피 칼 융합 배아 볼륨을 표준 "ADL" (nterior[x-y], [y-북쪽], 왼쪽에서 z 부근) 방향으로 재배치 하는 것이 가장 좋습니다. 이 선택 항목을 나타내려면 미리 보기에서 볼륨 출력 방향 정의 옵션을 선택 합니다. 사이토 쇼는 사용자가 밀접 하 게 관찰 하 고 ADL 등록을 달성 하는 데 필요한 회전을 지정할 수 있도록, 초기에이 트로픽 융합 볼륨의 쌍을 처리 하 여 응답 할 것 이다.
- 모든 매개 변수가 선택 되 면 예 를 클릭 합니다.
- 처리 된 파일을 저장할 출력 디렉터리 를 지정 합니다. 확인을클릭 합니다.
- 선택 사항: 미리 보기에서 볼륨 출력 방향 정의 옵션을 선택한 경우에는 ABa 및 ABp 셀이 비유에 도달한 초기 timepoint에 대 한 창 (그림 3a, 녹색 화살표, 왼쪽 패널)을 설정 합니다. SPIM-B 창의 t 스크롤 막대를 개발의 이후 쉼표 단계에 설정 합니다. 이는 ADL 방향을 지정 하는 데 도움이 됩니다.
- 선택 사항: 준비가 되 면 확인을 클릭 합니다. 위의 9.5.7 미리 보기 옵션을 선택한 경우 두 개의 미리 보기 볼륨만 SPIM A 및 SPIM 이미지 창의 t 슬라이더로 표시 되는 timepoints에 대해 동일 하 게 융합 됩니다. 이 두 미리 보기 timepoints는 아래에 설명 된 대로 ADL 방향에 대 한 출력 배아 볼륨의 정확한 재배치를 지정 하는 데 사용할 수 있습니다.
- 새로 표시 된 3DProjY_Decon-를 찾습니다. 창. T 스크롤 막대를이 미리 보기 창의 시간 지점 2로 이동 합니다. 배아의 긴 축이 바로 아래에 표시 될 때까지 Z 슬라이더를 이동 합니다.
- T 스크롤 막대를 3DProjY_Decon의 타임 포인트 1로 다시 이동 합니다. 창. 선 선택 도구를 선택 하 고 AB 셀은 유 세 플레이트의 평면을 통해 EMS 셀 (가장 둥근 핵)에서 선 선택을 그립니다.
- 3DProjY_Decon에서 주황색 diSPIM 미리 보기 버튼을 클릭 합니다. 창. 미리 보기 이미지 볼륨의 방향에 대 한 미세 조정은 전체 데이터 세트를 처리 하는 데 사용 하기 위해 저장 됩니다.
- 선택 사항: 위의 9.5 단계에서와 마찬가지로 디 스피브/퓨즈 diSPIM 원시 데이터 볼륨 대화 상자가 다시 나타납니다. 미리 보기에서 볼륨 출력 방향 정의 옵션을 선택 하지 않고 예를 클릭 합니다. 전체 데이터 처리 실행에 대 한 출력 폴더를 지정 합니다.
- DiSPIM 모니터 윈도우의 t 스크롤 막대 (그림 3a, 녹색 화살표, 왼쪽 패널)를 시작 타임 포인트 (spima)로 설정 하 고 전체 이미지 범위의 타임 포인트 (spimb)를 처리 합니다. 한 다음 확인을클릭 합니다.
- SpimFusion이 진행 됨에 따라, 세포 쇼는 각 배아에 대 한 슬라이스-4D 등방성 융합 체적을 나타내는 다차원 윈도우 뿐만 아니라 등방성 체적의 회전-4D 최대 강도 돌출부가 있는 두 개의 창을 새로 고칩니다. 이 시간 동안 등방성 융합 및 계보 추적이 완료 될 때까지 모든 사이토 쇼 창을 방해 하거나 닫지 마십시오 .
- 선택 사항: StarryNite 스플래시 화면이 나타난 후 나중에 사라진 경우 전체 데이터 처리 파이프라인이 완료 되었다는 점에 유의 하십시오. 이 창은 처리 중에 닫히지 않아야 하거나 StarryNite가 중단 됩니다.
10. 아 세 트리 (선택 사항)의 오프닝 StarryNite 계보 추적 시리즈
- 제공 된 "AceTree_16BitCompat"의 사용자 지정 버전을 엽니다.
- 메뉴 파일 > 파일 구성 열기를 선택 합니다. 이전에 사이토 쇼에 표시 된 출력 디렉토리 를 찾습니다. 배아 [n]에 대 한 Decon_Fuse_ _Pos [n] 하위 폴더를 엽니다. Aaa_edited 를 선택 하 고 엽니다.
- 비구 메뉴 편집 > 편집 도구 를 사용 하 여 트랙 편집 및 셀 조정 또는 삭제 창을 엽니다.
- 빨간색과 초록색 농도를 조정 하려면 반음영 원 그림 5b, 빨간색 정사각형 을 클릭 합니다.
- 계보 시각화 및 편집 앞에서 설명한 대로5,6,8 (원고도 우리의 다운로드 번들에 포함 되어 있습니다)로 진행 합니다.
Representative Results
우리는 먼저 diSPIM 취득을 위한 프로토콜의 매개 변수를 사용 하 여 이미지 된 배아의 생존 가능성을 검증 (섹션 1-6). 10 개의 배아는 2-세포 단계에서 2 배 단계 (7.5 시간, 451 부피/배아)로 20°c, 1 부피/배아에서 동시에 영상 촬영 하였다. 배아 발생 전반에 걸쳐 세포 분열을 모니터링 하기 위해 우리는 Mcherry를 표현 하는 UBIQUITOUSLY 스트레인 BV514를 사용 했습니다:: ujIs11324통합 된 이식 유전자 배열에서histone 리포터 구조. 도 6 은 이미지 된 배아 중 하나에 대 한 배아 개발의이 상반기의 타임 라인을 보여준다. 각 이미지는 이미지화 된 배아의 단일 뷰 최대 강도 투영 (7-8 단계에서 생성)을 나타냅니다. 우리는 최적화 된 프로토콜이 세포 분열 (도시 되지 않음)의 타이밍, 부 화 시간 및 발달 이정표와 관련 된 타이밍에 의해 평가 됨에 따라 배아에 대 한 검출 가능한 광 독성을 유발 하지 않는다는 것을 발견 했습니다 (그림 6 및 참조1 , 25 , 26).
그런 다음이 프로토콜을 적용 하 여 배아 발병에서 단일 뉴런의 성장 동력을 분석 했습니다. 우리는 DCR7692 (olaex4655), 정체 불명의 세포의 서브 세트에서의 neuropeptide flp-19 프로모터 떨어져 GFP를 표현 하는 형질 전환 된 네 마 충 균 주를 이미지화 하였다 (DACR2819, pflp- 199K(3.6KB)::: caax::p 10 3 ' UTR) . 여기에 설명 된 프로토콜의 단계에 따라, 우리는 알 수 없는 세포가 운동 뉴런 RMDDL 및 RMDDL, 배설물 운하 세포 및 2 개의 근육 세포에 상응 하는 것으로 판단 하였다 (도 7). 그런 다음 RMDDL 및 RMDDL 뉴런의 성장 동력을 검사 하 고 정량화 했습니다. 우리는 RMDDL 및 RMDDL 뉴런을 360 분 후에 경사지 게 형성 하는 것을 관찰 했 고,이는 신경 세포의 성장에 대 한 후속 축을 나타내는 더 긴 셀룰러 축 (그림 7 및 영화 S1)입니다. 피지의 "단순 신경 테 추적" 플러그인을 사용 하 여이 등 트로피 칼 융합 볼륨의 3d 재구성에 적용, 우리는 6 개의 배아에 대 한 rmddl 및 rmddl 신경의 입체 파생물을 정량화. 우리는 배아에 걸쳐 RMDDL과 RMDDL에 대 한 성장 역학이 입체 형식 이라고 결정 했습니다 (여기에 Rmddl 라고 함). 385-410 분부터 수정 후, RMDDs 신경은 6.0 ± 0.5 µm 확장 (평균 ± SEM; 세포 체의 전방 (도 7b, c1). 415-445 분부터 게시물 수정, 두 신경은도 마를 확장 하 고 추정 신경 링 ( 그림 7d에서 별표) 주위. 평균적으로 각각의 RMDD 신경은 확장 11.0 ± 0.6 µm (평균 ± SEM; n = 12 신경 세포)에서 링의 정점에서의 반대편 대응을 동기적으로 충족 시키기 전에 (그림 7i). 중요 한 것은, 우리의 대표적인 결과는 우리의 통합 프로토콜을 사용 하 여 단일 식별 가능한 세포에 대 한 신경 발달 기능을 검사, 비교 및 정량화 할 수 있다는 것을 입증 합니다 (그림 7 및 그림 8).
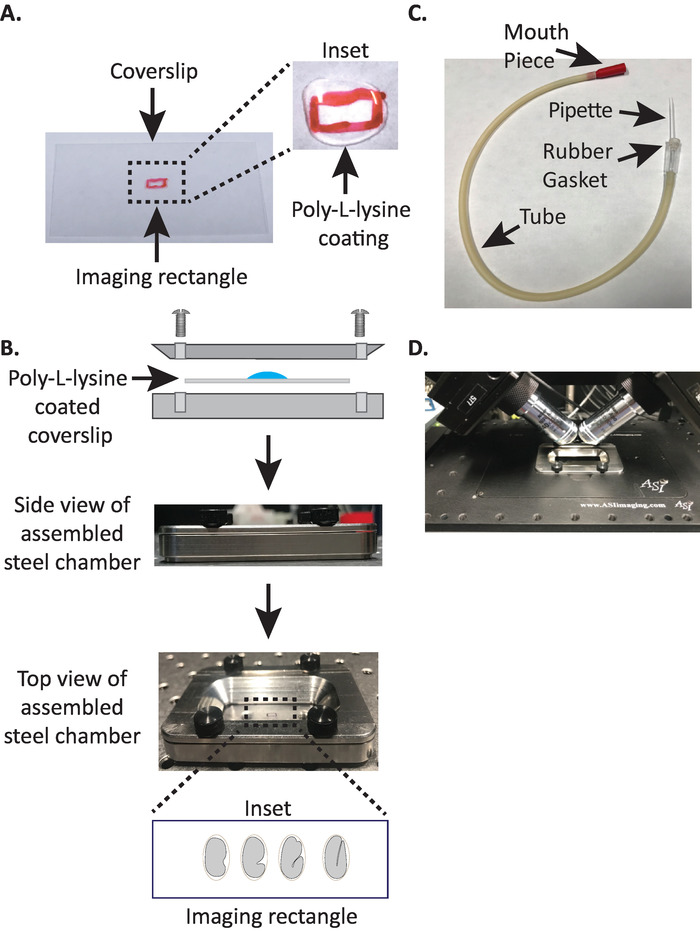
그림 1: diSPIM 샘플 장착 설정 (A) 폴 리 신을 사용한 커버 슬립의 제조. 인 셋에서, 10 µ L의 폴 리 라이 신은 커버 슬립을 5 분 동안 코팅 하는 데 사용 되었다. 폴 리-L-라이 신은 배아에 그 쉘이 직사각형의 커버 슬립에 단단히 붙어 있습니다. (B) 스틸 이미징 챔버와 조립 된 챔버의 개략도. 인 삽입에서, 대표적인 배아는 커버 슬립 상의 장 축에 수직인 전방 후방 축으로 지향 된다. (C) 미세 모 세관 파이 펫과 함께 조립 된 흡 기 튜브. (D) diSPIM 40x 대물 렌즈의 샘플 홀더에 장착 된 스틸 이미징 챔버. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 2: 마이크로 관리자의 장기 diSPIM 이미징 설정 (A) 광 독성을 줄이면서 장기간 이미징에 최적화 된 권장 diSPIM 레이저 전력 매개 변수 (적색 직사각형). 561 nm 레이저를 79 µW 0.25) 및 488 nm 레이저를 179 µW (0.5)로 설정 합니다. 레이저 전력에 대 한 소프트웨어 설정의 정확한 교정은 diSPIM 설치에 따라 다릅니다. 79 µW 561 nm) 및 179 488 µW 레이저 파워를 달성 하기 위해 사용자가 레이저 파워를 측정 하 고 교정 하는 것이 좋습니다. (B) 데이터 저장을 위한 dispim 매개 변수 (녹색 직사각형), 볼륨 설정 (파란색 정사각형) 및 슬라이스 설정 (주황색 정사각형) C.dispim af의 장기 이미징에 대 한 자동 초점 매개 변수 (6.1 ~ 6.6 단계 참조) 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 3: 사이토 쇼를 사용 하 여 이미지 시각화 및 데이터 처리 설정. (A) 사이토 쇼에 의해 열린 원시 diSPIM 이미지. 사이토 쇼는 두 카메라 경로 (SPIM A와 B)에 의해 캡처된 이미지를 열 수 있습니다. 이러한 raw 이미지는 Dispim 모니터라는 다차원 창에서 열립니다. Dispim 모니터에서 "보 타이 패턴"은 배아의 전방, 후방, 등 쪽 및 복 부 가장자리를 선택 하기 위해 생성 됩니다 (9.1 단계 참조). 리본 타이 선택은 해체 및 StarryNite 지원 lineaging 추적을 위한 배아 방향을 나타냅니다. (B) 등방성 이미지를 생성 하는 데 사용 되는 최적화 된 매개 변수입니다. 윈도우를 가져오는 동안 해체 에서 9.5.1-9.5.8 단계에 지정 된 매개 변수를 설정 하십시오. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 4: diSPIM 카메라 구성 (A) diSPIM 카메라 배치 및 방향에 대 한 사진. (B) 수집 된 spim 이미지와 일치 하도록 Spim A의 90도 회전을 묘사 합니다. (C) diSPIM의 카메라 구성에 따라 방향 인덱스 + 1에 상대적인 입력 볼륨 (9.5.2 단계 참조) 스 핌 B 이미지에 맞추기 위해, 등록 전에 스 핌 이미지 (들) + 90도를 Y 축 주위로 회전 시킵니다. 축척 막대 = 10 µm 이미지는 대표 단일 뷰, 최대 강도 프로젝션 및 1.5-표시 핵 (561-nm, 빨간색) 및 뉴런 (488-nm, 녹색)을 가진 배아의 해체 이미지입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 5: 큐 레이 션 및 편집 c. 아 세토에서의 배아 혈통. (A) 아세 트리를 사용 하 여 starrynite의 계보 흔적을 편집 합니다 (참고 문헌5,6,8참조). 원고는 또한 우리의 다운로드 번들에 포함 되어 있습니다. 그는 10.1-10.2 단계 완료 시 각 핵 (녹색 직사각형)에 대 한 체계적인 이름을 표시 합니다 . 이 창 (A)는 계보 추적을 추적 하 고 편집할 때 사용자에 게 도움이 되는 정보 (ABa, 파란색으로 강조 표시 됨)의 각 셀에 대 한 정보가 제공 됩니다. 사용자가 확인 하 고 선 세 셀과 그 위치를 비교 하는 것이 좋습니다 c. 엘 레 강 스 배아 세포 혈통은 이전에 술 스턴에 의해 보고 된.1 또한, 사용자가 특정 셀의 위치를 찾는 데 관심이 있는 경우 데이터 시리즈의 해체 (아래 참조, B)는 검색 바 (주황색 직사각형)에서 c. 엘 레 강 스 체계적인 이름을 입력 합니다. (B) 사용자의 자동 완성 된 데이터 계열은 10.1 ~ 10.2 단계를 완료 하는 즉시 열립니다. 빨간색으로 표시 된 핵이 있는 4 세포 단계 배아의이 트로피 칼 융합 이미지가 여기에 표시 됩니다. 핵을 추적 하는 동안 사용자는 이미지의 강도 (빨간색 정사각형)를 변경 하 고 키보드의 화살표 키 (왼쪽/오른쪽, z 위/아래)를 사용 하 여 시간과 z를 탐색 해야 합니다. (C) 회전 가능한 3d 시각화를 가능 하 게하는 특정 기능 (보라색 직사각형)의 timepoint의 3d 만화. 비구와 3d 기능에 대 한 개요는 참조5,6,8을 참조 하십시오. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 6: diSPIM에 대한 c. 엘 더 스 배아의 시간 개발 역학. 상부 패널은, 이미지 된 배아 중 하나에 대 한 배아 개발의 상반기를 보여주는 diSPIM 영상 (스트레인 BV514 ujIs11324). 배아는 7.5 시간 (20°c에서) 동안 매 분 마다 연속적으로 이미지화 되었다. 상단 패널의 처음 두 이미지는 핵 (빨간색)과 극성 체의 위치 (파란색 별표로 표시 된 짙은 빨간색 구형)와 4 및 8 셀 배아를 나타냅니다. 각 이미지는 이미지화 된 배아의 단일 뷰 최대 강도 프로젝션을 나타냅니다. 축척 막대 = 10 µm. 타임 라인 (가로 막대)은 c. 엘 더 란 스 배아의 개발의 포스트 시비 (m.p.f.) 분을 나타낸다. 우리는 diSPIM 취득에 대 한 우리의 프로토콜의 매개 변수는 생존, 세포 분열의 타이밍, 개발 이정표의 해칭 및 타이밍에 의해 평가 되는 이미지 배아에 대 한 검출 가능한 광 독성을 유도 하지 않았다 검증 (참조 1 , 25 , 26). 우리는 개발 이정표의 타이밍은 우리의 이미징 매개 변수와 배아에 걸쳐 재현할 수 있었다 (6.4 시간 긴 이미징 세션에 대 한 SEM ± 8.174 분; = 10). 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 7입니다. C. 엘 레 강 스 배아를 개발 하는 신경 세포의 성장 역학의 셀룰러 식별 및 단일 세포 특성화. BV514 ujIs113를 교차 하 여 만든 균 주의 이중 색 이미징 (선 노화 용) 및 DCR7692 (olaex4655), 알 수 없는 세포의 서브 세트에서 neuropeptide FLP-19 프로모터 떨어져 GFP를 표현 하는 형질 전환 네 마 충 균 주. (-H) 여기에 설명 된 프로토콜의 단계에 따라, 우리는 알 수 없는 세포가 배설물 운하 세포 (파란색 화살표)에, 그리고 두 개의 근육 세포 (백색 화살표)에 게 RMDDL 및 RMDDL (노란 화살)에 상응 하는 모터 뉴런에 대응 한다고 결정 했습니다. (I) 피지 플러그인을 사용 하 여 RMDDL 및 RMDDL 뉴런의 성장 역학의 정량화 "간단한 신경 테 추적" 및이 소부 트로피 칼 융합 볼륨의 3d 재구성에 적용. RMDDL과 RMDDL이 모두 전체 길이 11.0 ± 0.6 µm (평균 ± SEM) 동안 동기적으로 확장 되는 입체 형의 비 성장 역학을 보여 줍니다. 신경 링의 등 쪽 꼭대기에서 회합 하는 것이 가능 합니다 ( 영화 S1참조). 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

도 8: c. 엘 레 강 스 배아에서의 신경 형태에 대 한 등방성 디 스 핌 이미지의 검사. AVHL 및 AVHL 뉴런 (노란색 화살표)의 등방성 시각화. DiSPIM을 사용 하 여, 뉴런 형태는 약 330 nm의 등방성 공간 분해능을 갖는 4 차원 (4d) 이미지를 산출 할 수 있다. DiSPIM을 사용 하면 사용자가 모든 방향에서 동일한 해상도로 이미지 볼륨을 사실상 회전할 수 있습니다. A-D 의 이미지는 배아의 긴 축을 중심으로 별개의 회전에서 동일한이 소 트로피 칼 융합 dispim 이미지 볼륨의 최대 강도 투영입니다. 축척 막대 = 5 µm 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
보조 동영상 S1: 280에서 434 분 후에 개발 된 엘 더 스 배아. 등방성의 변형 DCR7692 (olaex4655)는 rmdd 신경에 DACR2819 희소 하 게 라벨링 된 ujIs113 ubiquitously를 발현 합니다 (그림 7a, 노란색 화살표). DACR2819는 또한 배아 개발 중에 두 개의 근육 세포 (그림7a-d, 흰색 화살표) 및 배설 운하 세포 (그림 7A-d, 파란색 화살표)를 레이블 합니다 (그림 7a-d). 축척 막대 = 10 µm 이 파일을 다운로드 하려면 여기를 클릭 하십시오.
Discussion
C. 엘 더는 각 성인 뉴런의 최종 위치 및 연결성을 가진 유일한 유기 체로 서27개의 알려진 것으로 눈에 띈다. 그러나, C 를 구성 하는 작업 회로 및 네트워크의 조직으로 이어지는 발달 역학은 커넥터는 알 수 없는 남아. 빛 현미경의 진보에서 떠오르는 기회에 기초 하 여, 우리는 지금 포착 하 고 C 에 걸쳐 세포 위치, 형태 형성 및 신경 형성을 분석할 수 있습니다.
우리가 설명 하 고 우리가 일상적으로 실험실에서 사용 하는 절차는 C 에서 세포 선 노화에 대 한 레이블이 있는 뉴런 및 핵의 4d 등방성 이미지를 산출 합니다. 더 중요 한 것은 분석의 속도와 정밀도를 향상 시키기 위해 diSPIM과 결합 된 반자동 라인 에이징 기능과 고해상도 이미지를 사용 하 여 장기적인 이미징 조건을 최적화 하 여 C. 엘 레 강 스 배아 발생을 개선 하는 것입니다. 이 통합 프로토콜은 사용자가 세포를 시각화 하 고 식별 하 고 초기 경련의 발병을 통해 신경 테 이주 및 축 삭 파스 트와 같은 3 차원 기능을 정량화 할 수 있게 합니다. 이 절차는 ASI diSPIM 시스템으로 모든 시설에 쉽게 적용 할 수 있으며이 프로토콜을 위해 특별히이 시스템을 권장 합니다. 상업적으로 제공 되는 다른 SPIM 제형은 샘플 챔버 및 광학 특성에서 ASI 구성과 다를 수 있다. 그러나 다른 플랫폼에서 내보낸 데이터는 데이터 파이프라인을 통해 넣을 수도 있습니다. 따라서 선 에이징에서의 가치 평가, 이미지 품질 및 기기 안정성에 대 한 까다로운 테스트가 가능 합니다. 우리가 적극적으로 다른 표본을 정기적으로 이미지 (예: 드로 소 필 라 및 제 브라 피쉬 배아)를 사용 하지만, 설명 된 배아의 포괄적 인 선 노화 분석은 여전히 현재 마 테 오드 종으로 제한 된다. 더 크거나 두꺼운 샘플의 경우, 우리는 고정 된 라이트 시트를 통해 샘플을 스캔 하는 단계 스캐닝 접근법을 사용 하도록 선택 합니다. 쿠마 르 외. 이전에는 diSPIM10에 추가 수정 없이 두꺼운 샘플에서 고품질 이미지를 생성 하기 위해 개선 된 dispim 단면을 시연 했습니다.
프로토콜 내의 중요 한 단계에는 폴 리-L-라이 신 코팅 커버 슬립, 데이터 수집 및 데이터 처리에 대 한 c. 엘 더 란 트 배아가 포함 됩니다. 유리 커버 슬립의 수확 및 장착 c. 엘 더 란 스 배아는 미숙한 사용자에 게 어려울 수 있지만 여기에서는 학습을 촉진 하기 위한 주요 단계의 상세한 프로토콜을 제공 합니다. 장기 화상 진 찰이 요구 되는 경우에, 우리는 8-10 청년 성인28에서 4 세포 또는 초기 배아를 수확 하는 최상의 결과를 얻습니다. 오래 된 성인은 자 궁과 수정 되지 않은 계란에 있는 이전 배아를 포함 하는 경향이 있기 때문에 초기 단계 배아를 수확 하는 것이 덜 바람직합니다. 배아를 장착 하는 것과 관련 하 여 조립 된 흡 인기 (입 피 펫)의 막힘 또는 미세 모 세관 파이 펫의 개 구 부의 너무 큰 등의 문제는 배아의 적절 한 장착 및 배 향을 방해할 수 있습니다. 최적의 이미징에 대비 하기 위해, 우리는 초기 및 후반 사전 경련 배아에 대 한 사전 획득 테스트를 수행 하 여 라이트 시트, 카메라, 목표 및 자동 초점의 성능을 확인 합니다. 우리는 이러한 모든 작업을 테스트 하 고 우리의 사전 획득 테스트 중에 높은 품질의 이미지를 얻을 때 최상의 결과를 얻을. 이는 등방성 공간 해상도로 이미지를 생성 하는 데 특히 적합 하며 두 뷰 (목표)에서 획득 한 raw 이미지는 고품질 이어야 합니다. 획득 후 각 뷰에 대해 획득 된 볼륨이 등방성 이미지를 생성 하도록 처리 됩니다. 이 프로토콜에 설명 된 대로 적절 한 GPU (그래픽 처리 장치) 카드를 사용 하는 것이 중요 합니다 (아래 참조). 이를 통해이 트로피 칼 융합 이미지가 생성 되는 처리 속도가 향상 되어 데이터 분석 시간이 단축 됩니다. 또한 사용자가 최신 버전의 사이토 쇼를 실행 하 고 StarryNite 자동 라인 에이징에 대 한 우리의 다운로드 번들과 함께 제공 되는 매개 변수를 사용 하는 것이 필수적 이다. 사용자가 다른 샘플에 대해 자동 라인 에이징 (예: 제 브라 피시, 초파리 등)을 사용 하려는 경우 starrynite에 사용 되는 매개 변수에 대 한 추가 최적화가 필요 합니다 (참조3,4참조).
우리의 통합 프로토콜은 사전 경련 배아에 이미지와 선 노화 결과를 제공 하지만, 사용자는 포스트 경련 배아의 자동화 된 선 노화가 현재 실현 가능 하지 않다는 것을 알고 있어야 합니다: 핵 위치는 초 단위로 변경 됩니다. 후 경련 배아, 너무 빠르게 계보 추적을 허용 합니다. 그러나, dispim은 실제로 신경 발달 사건을 포착 하 고 배아 발생23,29의 포스트 경련 단계에서 일부 세포 위치를 추적 할 수 있는 유망한 능력을 입증 했습니다. 사용자가 포스트 경련 배아를 검사 하는 데 관심이 있다면 diSPIM은 빠른 속도로 움직이는 배아에서 체적 스냅샷을 얻고 신경 근 성장 등의 미세 신경 발달 사건을 추적 하는 속도를 제공 합니다.
이 프로토콜은 WormGUIDES atlas30의 셀 바이 셀 완성을 위한 기초가 되며,이는 고해상도 등방성 이미지와 통합 된 접근법을 제공 하 여 표시 된 뉴런의 3d 형태학을 식별 하 고 포착 합니다. 첫 번째 430 분의 배아 발생. 그것은 스탠드, 프로토 타입 WormGUIDES atlas 개발 배아 세포의 핵 위치를 제공 하 고 배아 뉴런의 하위 집합의 발달 역학을 캡처하는 것을 목표로 합니다. 이 프로토콜은 추가 개발 뉴런을 WormGUIDES atlas30으로 통합 하기 위한 열쇠가 될 것입니다.
당사의 통합 프로토콜은 또한 c. 엘 레 강 스 배아에서 새로운 유전자 발현 프로필을 탐색 하는 것을 단순화 합니다. 형질 전환 c.엘 레 강 스에서, 많은 세포 특이 적 프로모터는 공간적으로 및 시간적으로 이식 유전자 발현을 조절 한다. 대부분의 유전자의 발현 패턴은 성인 동물 (31),32,33,34등을 광범위 하 게 특징으로 하는 반면에, 거의 모든 것이 아직 개발 (특히 후반 단계) 배아. C. 엘에이 칸의 상태 올리기형은 세포 특유의 이식 유전자 발현을 촉진 하 고, 유전자 기능이 세포 자치 또는 비 자율적 인지 여부를 결정할 수 있도록 웜 커뮤니티에 유용한 자원이 되었습니다. 유전자의 등방성 고 분해능 및 동적 발현 패턴을 포착 하 고, 선 노화를 통해 발현 세포를 정확 하 게 식별 하는 것은 과학계의 많은 사람들에 게 가치가 있습니다.
배아 발생은 두 가지 주요 과정, 세포 분화 및 조직 형태 형성을 포함 한다. 큰 거래는 메커니즘 및 C의 개발 하는 동안 고유 세포 유형을 정의 하는 분자에 대 한 알려져 있다. 그러나 c. 엘 레 강 스 배아에서 세포 이동, 세포 부착 및 세포 형태에 중요 한 메카니즘에 대해 거의 알려지지 않았다. 알려진 c. 엘 더 린 불변 세포 혈통으로, 우리의 프로토콜은 우리가 쉽게 새로운 세부 수준에서 형태학 상 동안 배아의 카탈로그 된 3d-미세 해부학을 분별 할 수 있습니다: 예를 들어, 축 삭 파스, synaptogenesis 및 신경 활동. 디스트 렐 외. 이전에는 C 에서 단일 뉴런의 수준에서 칼슘 과도 상태를 포착 하기 위해 diSPIM의 힘을 입증 했습니다. 엘 레 강 스 배아23. 발달 생리학의 그밖 많은 양상은이 방법에 의하여 조회를 위해 익은 다.
마지막으로,이 프로토콜은 주로 자동화 되 고 체계적으로 해체 이미지를 생성 하 고 StarryNite 및 비구를 통해 세포 라인 노화를 수행 하는 데 걸리는 시간을 줄입니다. 이 프로토콜에 사용 되는 소프트웨어 전략은 우리가 여기에서 보여준 매우 특정 분야에서 멀리 떨어진 생물학의 많은 질문에 적용 될 수 있습니다.
소프트웨어 호환성 및 다운로드 액세스에 대 한 세부 정보
DiSPIM 이미징 용 마이크로 관리자 및 플러그인에 대 한 정보는 http://dispim.org/software/micro-manager 및 https://micro-manager.org/wiki/ASIdiSPIM_Plugin에서 확인할 수 있습니다.
데이터 처리 파이프라인에는 현재 Windows 운영 체제가 필요 합니다. 우리는 모든 필요한 데이터 처리 프로그램 및 지원 파일의 설치를 단순화 하기 위해 단일 아카이브 파일을 번들로 제공 하 고 있습니다. 그것은 http://dispimlineage.wormguides.org에서 다운로드 할 수 있습니다.
사이토 쇼 (http://run.cytoshow.org/)는 널리 사용 되 고 오픈 소스 이미지 분석 플랫폼을 기반으로, ImageJ (v1). Java는 사이토 쇼를 사용 하는 컴퓨터에 최신 설치 되어 있어야 하며, 사이토 쇼에 대 한 업데이트는 자바 웹 시작을 통해 자동으로 배포 됩니다. 사이토 쇼의 많은 ImageJ 기반 기능은 https://imagej.nih.gov/ij/docs/examples/index.html에서 설명 되 고 도시 된 바와 같다. 사이토 쇼는 TIFF 출력을 생성 하는 다른 이미징 소프트웨어 뿐만 아니라 ASI diSPIM에서 다차원 원시 데이터를 표시 하도록 사용자 정의 되었습니다. 원칙적으로 다른 다중 뷰 SPIM 이미징 시스템은이 프로토콜이 다른 현미경 시스템에서 수행 될 수 있도록 사이토 쇼의 사소한 수정에 의해 지원 될 수 있습니다.
SpimFusion은 cuda 툴킷 v 7.5와 함께 Visual Studio 2013을 사용 하 여 CUDA/c + +로 작성 되었습니다. SpimFusion 실행에는 특정 컴퓨터 하드웨어가 필요 합니다: CUDA 컴퓨팅 기능이 1.0 이상인 NVIDIA 그래픽 처리 장치 (GPU) 카드 및 최소 2gb의 그래픽 카드 메모리. 우리의 프로토콜의 출판 시점에서 SpimFusion은 미 출간 (분 궈와 하 리 Shroff) 하지만 위에서 언급 한 소프트웨어 번들 아카이브에서 사용할 수 있습니다.
StarryNite의 특수 제작 된 명령줄 기반 버전을 사용 하려면 자유롭게 사용할 수 있는 MATLAB 컴파일러 런타임이 설치 되어 있어야 하지만 상업용 MATLAB 소프트웨어에 대 한 라이센스가 필요 하지 않습니다. MATLAB 컴파일러 런타임은 위에서 언급 한 소프트웨어 번들 아카이브에 포함 되어 있습니다. 이 프로토콜에서 사용 되는 StarryNite에 대 한 코드는 기본적으로 공초점 이미지6에 사용 되는 것과 변경 되지 않습니다. 그러나, StarryNite 처리 및 StarryNite 결과의 처리를 위한 입력 이미지의 생성에 몇 가지 운영 문제는 융합 등방성 diSPIM에 대 한 연속 데이터 처리 파이프 라인을 가능 하 게 하는 세포 쇼의 방법으로 여기에서 해결 되었습니다 볼륨. 이러한 변경 사항은 이러한 전처리 및 후 처리 단계를 처리 하는 사이토 쇼 코드에 의해 자동화 됩니다. 또한 사이토 쇼는 미리 최적화 된 diSPIM 특정 템플릿을 편집 하 여 이미지 데이터에서 핵의 형광 강도로 세그먼트 알고리즘을 자동으로 조정 하는 StarryNite 매개 변수를 설정 합니다. 각 diSPIM 데이터 세트에서 StarryNite가 사용 하는 고유한 매개 변수는 출력 이미지 및 lineaging 데이터와 함께 파일에 저장 됩니다.
16 비트 이미지와 함께 작동 하 고 Java3D 렌더링과의 호환성을 유지 하는 사용자 지정 버전의 비구는이 프로토콜에 가장 적합 합니다. 또한 위에서 언급 한 소프트웨어 번들 아카이브에 포함 되어 있습니다.
Disclosures
저자는 공개 할 것이 없습니다.
Acknowledgments
우리는 통합 된 스트레인에 대 한 존 머 레이 감사, ujIs113, 선 에이징 스트레인 BV514를 생성 하; 브 랜던 하 비 (NIBIB) 프로토콜 테스트에 대 한 도움; 존 다니엘 스와 게리 르노 (응용 과학 계측) 마이크로 관리자 및 diSPIM 악기에 대 한 지원을 위해; 그리고 앤드류 요크와 행 크 에덴은 diSPIM 시스템에 대 한 그들의 중요 한 피드백. 우리는 또한 회의 및 브레인스토밍 플랫폼을 제공 하기 위해 소수 기관 프로그램에 대 한 연구 센터와 Neurobiología 호세 델 카스 티 요 (Universidad 데 푸에르토 리코)에 게 감사를 드립니다. 이 작업의 대부분은 위트 먼 프로그램을 통해 우즈 홀에서 해양 생물 실험실에서 실시 되었다. 이 작업은 생물 의학 영상 및 생물 공학의 NIH 국립 연구소의 교내 연구 프로그램에 의해 지원 되었다 및 NIH 보조금 No. U01-HD075602 및 아니오. R24-OD016474 마크 w. Moyle는 F32-NS098616에 의해 지원 되었고, 레이 턴 h. 던컨는 R24 OD016474에 다양성 보충에 의해 지원 되었다.
Materials
| Name | Company | Catalog Number | Comments |
| Steps 1-4 | |||
| Concavity slides | ThermoFisher Scientific | 1519006 | 5-18mm diameter, 0.6-0.8mm deep, 1.2-1.5mm |
| Dissecting microscope with 10×–50× zoom range | Motic | SMZ-171 | |
| E. coli (OP50) | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Glass coverslips, no. 1.5, 24 × 50 mm | VWR International | 48393-241 | |
| M9 Buffer | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Methyl cellulose | Sigma-Aldrich | H7509-25G | |
| Microcapillary pipette aspirator tube | Sigma-Aldrich | A5177 | |
| Microcapillary pipettes, 0.4-mm i.d | Drummond Scientific | 1-000-800 | |
| Needle, no. 18G x 1 ½ (1.2mm x 40mm) | BD Precision Glide | 305196 | |
| NGM plates | prepared as described by Brenner (1974) | ||
| O-ring for imaging chamber | O-Rings West | M1.5X40 | |
| Pasteur pipette | Corning/Sigma-Aldrich | CLS7095D5X | |
| Platinum wire, 0.5-mm diameter | Sigma-Aldrich | 267201 | |
| Poly-L-lysine | Sigma-Aldrich | P1524 | |
| Stainless steel rectangular chamber (76.0 mm x 50.5 mm) | Applied Scientifics Instrumentations (ASI) | I2450 | |
| Worm Eyelash Pick | Hart, A. C. Behavior. WormBook. (2006). | ||
| Worm Pick | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Name | Company | Catalog Number | Comments |
| Steps 5-6 | |||
| 488 nm long-pass filter | Semrock | LP02-488 RU-2 | |
| 561-nm notch filter | Semrock | NF03-561E-25 | |
| BLP02-561R-25, quantity 2 | Semrock | 561 nm EdgeBasic best-value long-pass edge filter | |
| Control software for bottom camera | Jenoptik | ProgRes CapturePro | |
| diSIPM assembly video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/TAgbr6IrTqw ; http://www.asiimaging.com | |
| diSPIM alignment video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/qnOrg30NNuE | |
| diSPIM imaging PC | Intel | Intel Xeon CPU E5-2630 2.6GHz, 12 cores in total, 64 GB memory, Windows 7 | |
| FF01-525/45-25, quantity 2 | Semrock | 525/45 nm BrightLine single-band bandpass filter | |
| FF555-DI03-25X36, quantity 2 | Semrock | 555 nm edge BrightLine single-edge dichroic beamsplitter | |
| Imaging PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Kumar et al diSPIM Setup | Applied Scientifics Instrumentations (ASI) | Instrument setup for this protocol is identical to Kumar et al 10,11 diSPIM, which makes use of 40x 0.8NA water immersion lenses for imaging. (See steps 5.1 and note) | |
| Micro Manager | Micro-Manager | https://micro-manager.org/ | |
| Modifications to Kumar et al diSPIM Setup (see below) | |||
| Optical table with isolators, 4 feet × 6 feet × 12 inches | TMC | 784-651-02DR and 14-416-34 | |
| Name | Company | Catalog Number | Comments |
| Steps 7-10 | |||
| Analysis PC | Intel | Intel Core i7-8700K CPU 3.70GHz, 6 cores in total, 64 GB memory, Windows 10 | |
| Analysis PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Installation instructions | Software bundle | http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm | |
| Software bundle | Software bundle | http://dispimlineage.wormguides.org |
References
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Developmental Biology. 100 (1), 64-119 (1983).
- Bao, Z., et al. Automated cell lineage tracing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 103 (8), 2707-2712 (2006).
- Santella, A., Du, Z., Bao, Z. A semi-local neighborhood-based framework for probabilistic cell lineage tracing. BMC Bioinformatics. 15, 217 (2014).
- Santella, A., Du, Z., Nowotschin, S., Hadjantonakis, A. K., Bao, Z. A hybrid blob-slice model for accurate and efficient detection of fluorescence labeled nuclei in 3D. BMC Bioinformatics. 11, 580 (2010).
- Boyle, T. J., Bao, Z., Murray, J. I., Araya, C. L., Waterston, R. H. AceTree: a tool for visual analysis of Caenorhabditis elegans embryogenesis. BMC Bioinformatics. 7, 275 (2006).
- Katzman, B., Tang, D., Santella, A., Bao, Z. AceTree: a major update and case study in the long term maintenance of open-source scientific software. BMC Bioinformatics. 19 (1), 121 (2018).
- Murray, J. I., et al. Automated analysis of embryonic gene expression with cellular resolution in C. elegans. Nature Methods. 5 (8), 703-709 (2008).
- Murray, J. I., Bao, Z., Boyle, T. J., Waterston, R. H. The lineaging of fluorescently-labeled Caenorhabditis elegans embryos with StarryNite and AceTree. Nature Protocols. 1 (3), 1468-1476 (2006).
- Wu, Y., et al. Spatially isotropic four-dimensional imaging with dual-view plane illumination microscopy. Nature Biotechnology. 31 (11), 1032-1038 (2013).
- Kumar, A., et al. Using Stage- and Slit-Scanning to Improve Contrast and Optical Sectioning in Dual-View Inverted Light Sheet Microscopy (diSPIM). The Biological Bulletin. 231 (1), 26-39 (2016).
- Kumar, A., et al. Dual-view plane illumination microscopy for rapid and spatially isotropic imaging. Nature Protocols. 9 (11), 2555-2573 (2014).
- Wu, Y., Christensen, R., Colon-Ramos, D., Shroff, H. Advanced optical imaging techniques for neurodevelopment. Current Opinion in Neurobiology. 23 (6), 1090-1097 (2013).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17708-17713 (2011).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136 (12), 1963-1975 (2009).
- Stelzer, E. H. Light-sheet fluorescence microscopy for quantitative biology. Nature Methods. 12 (1), 23-26 (2015).
- Winter, P. W., Shroff, H. Faster fluorescence microscopy: advances in high speed biological imaging. Current Opinion in Chemical Biology. 20, 46-53 (2014).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. Astronomical Journal. 76 (6), 745-754 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. JOSA. 62 (1), 55-59 (1972).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Hart, A. C. Behavior. WormBook. , (2006).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. Chapter 14, Unit14 20 (2010).
- Gualda, E. J., et al. SPIM-fluid: open source light-sheet based platform for high-throughput imaging. Biomedical Optics Express. 6 (11), 4447-4456 (2015).
- Ardiel, E. L., et al. Visualizing Calcium Flux in Freely Moving Nematode Embryos. Biophysical Journal. 112 (9), 1975-1983 (2017).
- Walton, T., et al. The Bicoid class homeodomain factors ceh-36/OTX and unc-30/PITX cooperate in C. elegans embryonic progenitor cells to regulate robust development. PLoS Genetics. 11 (3), e1005003 (2015).
- Altun, Z. F. WormAtlas. , (2002).
- Wood, W. B. Embryology: In the nematode C. elegans. Cold Spring Harbor Laboratory Press. Chapter 8, 215-241 (1988).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314 (1165), (1986).
- Bao, Z., Murray, J. I. Mounting Caenorhabditis elegans embryos for live imaging of embryogenesis. Cold Spring Harb Protoc. (9), (2011).
- Christensen, R. P., et al. Untwisting the Caenorhabditis elegans embryo. eLife. 4, (2015).
- Santella, A., et al. WormGUIDES: an interactive single cell developmental atlas and tool for collaborative multidimensional data exploration. BMC Bioinformatics. 16, 189 (2015).
- Dupuy, D., et al. A first version of the Caenorhabditis elegans Promoterome. Genome Research. 14 (10B), 2169-2175 (2004).
- Reece-Hoyes, J. S., et al. Insight into transcription factor gene duplication from Caenorhabditis elegans Promoterome-driven expression patterns. BMC Genomics. 8 (27), (2007).
- WormBase. , Available from: https://www.wormbase.org (2019).
- Lee, R. Y. N., et al. WormBase 2017: molting into a new stage. Nucleic Acids Research. 46 (D1), D869-D874 (2018).



