Summary
Aquí, presentamos un enfoque combinatorio usando microscopía de alta resolución, herramientas computacionales y etiquetado de una sola célula en los embriones vivos de C. elegans para entender la dinámica de una sola célula durante el neurodesarrollo.
Abstract
Caenorhabditis elegans (C. elegans) se destaca como el único organismo en el que se puede observar el reto de entender los orígenes celulares de todo un sistema nervioso, con una resolución de célula única, in vivo. Aquí presentamos un protocolo integrado para el examen del neurodesarrollo en embriones C. elegans . Nuestro protocolo combina imágenes, lineaje y rastreo neuroanatómico de células individuales en el desarrollo de embriones. Logramos imágenes a largo plazo, de cuatro dimensiones (4D) de embriones vivos C. elegans con resolución espacial casi isotrópica mediante el uso de microscopía de iluminación de plano selectivo invertido de doble vista (dispim). Los núcleos y las estructuras neuronales de los embriones de nematodos son imágenes e isotrógicamente fusionados para producir imágenes con una resolución de ~ 330 nm en las tres dimensiones. A continuación, se analizan estos conjuntos de datos minuto a minuto de alta resolución 4D para correlacionar las identidades definitivas de linaje celular con la expresión génica y la dinámica morfológica en niveles de detalle de célula única y subcelular. Nuestro protocolo está estructurado para permitir la implementación modular de cada uno de los pasos descritos y mejorar los estudios sobre la embriogénesis, la expresión génica o el neurodesarrollo.
Introduction
C. elegans se destaca como el único organismo en el que cada célula en el embrión se puede observar a través del neurodesarrollo. Con todo el linaje celular conocido e invariable1, y con el desarrollo de nuevas herramientas que permiten el etiquetado y la imagen continua de células individuales en embriones, los biólogos pueden ahora comenzar a examinar diferentes pasos en el desarrollo de los nematodos nerviosos sistema de todos los ángulos-nacimiento de la célula; migración y diferenciación; formación de neurita, afloramiento y fasciculación dirigidos; formación de sinapsis; y la sintonización de circuitos funcionales. La captura de dinámicas de crecimiento neuronal en el embrión de C. elegans , mediante la combinación de periodistas expresablemente expresados y microscopía de fluorescencia, es valiosa para la comunidad científica.
Los estudios de desarrollo en C. elegans a menudo aprovechan los mapas invariantes de linaje celular y destino celular de esta especie para aumentar la comprensión contextual a nivel de célula única dentro del organismo intacto1. Análisis de lineaje automático-uso del software starrynite2,3,4 y acetree5,6,7,8 -beneficios de alto contraste, alta resolución imágenes de núcleos fluorescentes. Para trabajar de manera óptima, starrynite/acetree también depende de la orientación predecible y restringida de los embriones imágenes durante el desarrollo. La microscopía confocal, realizada en embriones C. elegans comprimidos entre dos cubreobjetos, ha sido el método de microscopía de lineaje automático estándar durante más de una década porque proporciona tanto contraste alto/alta resolución como un predecible orientación del embrión7,8. Anteriormente Describimos la construcción y el uso de un nuevo microscopio de iluminación de plano selectivo invertido de doble visión (dispim) basado en hojas ligeras para imágenes de muestras en vivo como la embriogénesis de C. elegans de9,10 , 11 , 12 , 13. la microscopía de láminas ligeras, en general, proporciona una fototoxicidad baja, alta velocidad y imágenes a largo plazo de especímenes 3D en vivo14,15. El método diSPIM, específicamente, produce imágenes de cuatro dimensiones (4D) con una resolución espacial casi isotrópica de aproximadamente 330 nm9.
En comparación con la microscopía confocal, diSPIM ofrece una mayor señal a ruido y velocidad, una resolución espacial más isotrópica y es más adecuada para imágenes in vivo de largo plazo16. Por lo tanto, trabajamos para adaptar los datos de diSPIM para su entrada en StarryNite/AceTree e investigamos si esto mejoraría los análisis de lineaje. Un obstáculo importante es que los especímenes de diSPIM no pueden ser fácilmente restringidos por la compresión de cáscara de huevo para adoptar las orientaciones esperadas para StarryNite/AceTree. La orientación aleatoria de las posiciones de celda en el volumen que se está analizando degrada la precisión del análisis de lineaje automático.
Por lo tanto, empleamos CytoSHOW, una interfaz de usuario guiada por el espectador que permite a los usuarios seleccionar la orientación 3D precisa de los embriones durante el preprocesamiento de imágenes de diSPIM, produciendo datos de imagen que están optimizados para la calidad y en el contexto para la entrada en StarryNite /AceTree. Tras la selección de los embriones con imagen, CytoSHOW orquesta una canalización automatizada de procesamiento de datos. Las imágenes de embriones recortadas y en segundo plano se guardan en archivos de pila TIFF para cada posición, punto de tiempo y vista. Cytoshow entonces iterativamente llama al programa spimfusion para co-registrarse y deconvolve conjuntamente las dos vistas pre-procesadas, utilizando el algoritmo Richardson-Lucy17,18 para producir imágenes volumétricas isotrópicas de alta resolución. Se ha optimizado un conjunto de parámetros específicos de diSPIM para que StarryNite gobierne su comportamiento durante la segmentación de imágenes y el seguimiento de núcleos en imagen fusionada isotrógicamente. Las imágenes fusionadas y los resultados de lineaging se editan con AceTree, lo que permite a los usuarios identificar y corregir cualquier error en la traza de linaje automático generada por StarryNite. El AceTree también puede presentar representaciones modeladas en árbol de linaje y modelado 3D de núcleos rastreados en el embrión. Encontramos que la velocidad y la precisión de los lineados automáticos se mejoran notablemente utilizando imágenes fusionadas isotrógicamente, en comparación con las imágenes RAW de cualquiera de las cámaras SPIM. Nuestro protocolo, aunque optimizado para la aplicación C. elegans descrita aquí, podría ser adaptado generalmente para el lineamiento automático de los datos de dispim producidos para otras especies o especímenes. Si este es el uso previsto del Protocolo, tenga en cuenta que es probable que el ajuste adicional de los parámetros de starrynite sea necesario para los nuevos especímenes, como se describe en3,4.
La implementación exitosa de este protocolo da como resultado imágenes con resolución 4D-isotrópica y permite a los biólogos rastrear linajes celulares, mientras que simultáneamente identifican y analizan neuronas en el desarrollo de embriones C. elegans . Por otra parte, mediante la fusión de varios algoritmos de post-procesamiento-con la aceleración de hardware es el más lento de estos-ahora podemos analizar tanto los detalles subcelulares finos y los linajes celulares y las células-Fates de embriones vivos en tiempo esencialmente real. Este nuevo protocolo permite la manipulación precisa, informada y la observación del comportamiento de las células durante los estudios probativos de diferenciación y morfogénesis in vivo. En este manuscrito, presentamos una explicación detallada de los protocolos mejorados que hemos desarrollado para el lineaje y el rastreo celular en el desarrollo de embriones C. elegans , para mejorar los estudios de embriogénesis, expresión génica o neurodesarrollo.
Protocol
1. montaje de la cámara de imagen de acero diSPIM con recubrimiento recubierto de poli-L-lisina
Nota: todos los pasos a continuación son necesarios para optimizar y automatizar el análisis de linaje de embriones de nematodo por StarryNite/acetree. Se pueden omitir varias opciones (indicadas por como tales) para experimentos que requieran el rastreo de linajes celulares C. elegans .
- Dibuje un pequeño rectángulo (2mm x 5 mm) en el centro de un recubrimiento rectangular limpio (no. 1,5, 24 x 50 mm) con un Sharpie (o bolígrafo similar).
- Voltear el cubreobjetos sobre el lado sin marcar y colocar 10 μl de poli-L-lisina (gato. no. P1524) sobre el rectángulo marcado.
Nota: hacer una concentración de trabajo de 1 mg/mL de poli-L-lisina disuelto en agua filtrada (o equivalente). Para el almacenamiento a largo plazo, prepare alícuas de 5 a 10 mL y guárdela a − 20 ° c. Una vez descongelada, las alícuas se pueden almacenar a temperatura ambiente (23 ° c) durante 3 – 4 semanas. - Permitir que la poli-L-lisina para cubrir el cubreobjetos durante 5 minutos (figura 1A). Poly-L-lisina se utiliza para el recubrimiento de los resbalones de cubierta de vidrio donde se montarán los embriones, permitiendo que la cáscara de huevo del embrión se adhiera firmemente al cubreobjetos, incluso cuando los objetivos se sumergen en el medio de imagen.
- Coloque el recubrimiento recubierto de poli-L-lisina en la mitad inferior de la cámara de imágenes de acero.
- Coloque la mitad superior de la cámara de imagen de acero en la mitad inferior con el resguardo y apriete con los cuatro tornillos asociados con la cámara. Compruebe visualmente desde el lado para asegurarse de que la mitad superior está colocada uniformemente en la mitad inferior (figura 1B).
- Llene la cámara con 7 – 8 mL de buffer M919, un tampón isotónico que ayuda a prevenir que los embriones de etapa temprana sucumba a una presión osmótica anormal. los embriones de 1 célula e incluso de 2 células son osmóticamente sensibles y pueden desarrollarse anormalmente si no en este tampón isotónico. Los embriones también tienden a arrestarlos en la etapa de 3 veces si el M9 se sustituye por agua.
2. preparar muestras de embriones de C. elegans para el montaje
Nota: aproximadamente 18 horas antes de la toma de imágenes, cinco jóvenes (1 día desde final molt) adultos y diez larval etapa 4 (L4) C. elegans se recogen en una placa de agar de medio de crecimiento nemátodo (NGM) sembrada con E. coli cepa OP50. Los picos de alambre de platino se utilizan para mover las larvas y los adultos jóvenes C. elegans sin dañar al animal19.
- Prepare un 1% de celulosa de metilo (gato. no. H7509-25G) solución en el buffer M9.
Nota: la celulosa de metilo debe agitarse en el M9 caliente hasta que se disuelva. Una vez preparada, esta solución puede almacenarse a temperatura ambiente. - Añadir 500 μL de 1% de metilcelulosa-en-M9 solución en la depresión de una diapositiva cóncava del microscopio. Este tampón viscoso se utilizará en dos ocasiones: 1) cuando se cosechen embriones tempranos por disección de gusanos adultos y 2) al lavar embriones de etapa tardía recogidos directamente de una placa NGM.
Nota: la celulosa de metilo se utiliza para evitar que los embriones se adhieran a la diapositiva del microscopio. - Para la toma de imágenes de embriones en etapa tardía, escoja los embriones de C. elegans (preferiblemente de una placa NGM con adultos jóvenes presentes) usando una escota de pestañas, y mueva los embriones al 1% de metilcelulosa en el portaobjetos cóncavo del microscopio. El Pick de pestañas ayuda a reducir la fuerza y así minimizar el estrés o daño a los embriones durante el manejo. El procedimiento para hacer un picking de pestañas está cubierto por Hart20.
- Con una segunda toma de pestañas (en la mano opuesta), toque suavemente ambas pestañas juntas para suspender los embriones en la celulosa de metilo.
- Opcional: Si planea el linaje de células embrionarias con StarryNite, uno debe montar embriones de 1 célula a 4 células. Para ello, primero seleccione adultos jóvenes de una placa NGM y muévase a la solución de celulosa M9-metilo en el portaobjetos cóncavo con un pick de platino.
- Opcional: con las puntas afiladas de agujas hipodérmicas (no. 18G x 1 1/2), corte el animal transversalmente en el cuerpo medio para liberar embriones de 1 célula a 4 células.
3. la Piera de la boca: montaje del tubo del aspirador con pipetas microcapilar
Nota: utilizamos un tubo aspirador con una Piera microcapilar extraída a mano insertada en la Junta de caucho del tubo. Esto nos permite transferir embriones de la diapositiva de disección a la superficie recubierta de poli-L-lisina en la cámara de imágenes llena de búfer.
- Tire manualmente de la picada microcapilar sobre una llama abierta para crear dos mitades con puntas estiradas.
- Tomar la mitad de la picada microcapilar e insertar el extremo romo en la Junta de caucho del tubo del aspirador (figura 1C). Poner a un lado la otra mitad de la pipetas microcapilar para su uso posterior (si es necesario).
- Con el tubo del aspirador montado equipado con pipetas microcapilar, rompa suavemente la punta de la picada microcapilar y cree una abertura que se ajuste a 1 – 2 dos embriones (de aquí en este instrumento se llama una "Piera de boca").
4. montaje de embriones C. elegans en el recubrimiento recubierto de poli-L-lisina
- Con la boquilla del aspirador que se mantiene suavemente entre los dientes, llene previamente la Piela microcapilar con 10 – 15 μL de buffer M9 y luego succiona suavemente varios embriones de la diapositiva cóncava en el capilar.
- Transfiera los embriones a la cámara de imagen de acero llena de buffer M9, colocando la punta capilar de manera que los embriones caigan en el rectángulo central del Coberto.
- Evitando lesiones en los embriones, muévelos suavemente con un pick de pestañas o utilice la Piera de la boca para posicionar los embriones verticalmente, para orientar los embriones de manera que el eje largo del embrión sea perpendicular al eje largo de la cubierta (figura 1B inserto , panel inferior).
Nota: posicionar el embrión en esta orientación minimiza el número de rebanadas a la imagen, reduciendo así la dosis de luz y el tiempo de procesamiento de datos mientras mejora la velocidad de adquisición. - Coloque la cámara de imagen de acero en el soporte de la muestra en la etapa del microscopio (Figura 1D).
5. montaje, configuración de software y optimización láser para imágenes embrionarias utilizando el diSPIM
- Consulte las instrucciones paso a paso sobre cómo ensamblar todo el diSPIM acoplado por fibra de las piezas disponibles comercialmente en Kumar et al.10,11 y en http://www.dispim.org. Un video-protocolo de Cómo ensamblar el diSPIM también está disponible en el sitio web de ASI (http://www.asiimaging.com).
Nota: la configuración de instrumentos para este protocolo es idéntica a Kumar et al.10,11 dispim, que hace uso de lentes de inmersión de agua 40x 0.8 na para la imagen. La única diferencia entre la configuración en este protocolo y Kumar et al.10,11 es la adición de un espejo dicroico (dividiendo a 560 nm) y filtros de paso de banda rojo y verde dentro de un dispositivo de división por imágenes (modelo A12801-01) instalado en los dos brazos de imagen de diSPIM. La adición de la imagen que separa la óptica permite la captura simultánea de imágenes de dos fluoróforos distintos – excitados por láseres de 561 nm y 488 nm – separando las bandas de emisión en dos mitades del mismo chip de la cámara. - Después del montaje del instrumento, Compruebe la alineación óptica del diSPIM antes de la toma de imágenes.
Nota: para alinear correctamente el diSPIM Vea https://youtu.be/qnOrg30NNuE, y para la información de hardware, http://dispim.org/hardware/objectives y http://www.asiimaging.com. - Utilice la plataforma de código abierto micro-Manager (https://micro-manager.org/)21, que se ha optimizado para el funcionamiento de microscopios de lámina de luz para imágenes celulares de alto rendimiento22. Recomendamos el uso del plugin ASI diSPIM para la adquisición de múltiples posiciones, que permite imágenes simultáneas de hasta 30 embriones según lo descrito23.
- Con el micro-administrador abierto, fije las intensidades del laser a ~ 179 μW (0,5) para 488 nm y ~ 79 μW (0,25) para 561 nm (figura 2A, rectángulo rojo).
Nota: Estos son los ajustes recomendados para la toma de imágenes a largo plazo de embriones C. elegans utilizando intervalos de 1 minuto. Durante las imágenes de doble color a largo plazo, el láser de 561 nm se utiliza para la imagen de núcleos (Mcherry:: Histone) hasta que los embriones están en la etapa de frijol, momento en el cual el láser de 488 Nm se enciende para también la imagen de las neuronas etiquetadas por GFP. Estas condiciones de imagen están optimizadas para minimizar la fototoxicidad y garantizar la supervivencia y la eclosión de los embriones al tiempo que permite la adquisición continua (12 – 14 horas) de datos de desarrollo neurológico y lineaging prolongados. - En micro-Manager, elija plugins de menú ≫ Device control > asi dispim para abrir la ventana de asi dispim (figura 2B). Elija la ficha adquisición . En la sección configuración de ahorro de datos de esta pestaña (rectángulo verde), sección configuración de volumen (cuadrado azul) y sección configuración del sector (cuadrado naranja), asegúrese de que cada parámetro se establece como se muestra en la figura 2B.
Nota: nuestro software de análisis de imágenes CytoSHOW está adaptado para trabajar con otros formatos de datos de salida opcionales, como la serie de archivos OME-TIFF concatenados a granel y la serie de archivos TIFF-Stack creados después de la adquisición mediante el uso de una función de exportación integrada en micro-Manager. Normalmente, el formato de datos de archivo OME-TIFF intercalado de pila de punto único se utiliza porque permite la visualización y el procesamiento en tiempo real del volumen de imagen tan pronto como se adquieren los datos sin procesar.
6. parámetros de enfoque automático optimizados para imágenes a largo plazo de embriones de C. elegans
- Establezca los parámetros de enfoque automático de micro-Manager en los ajustes optimizados para imágenes diSPIM de calidad de linaje a largo plazo de embriones C. elegans . En la ventana ASI diSPIM, haga clic en la pestaña autofocus (figura 2C). En la sección Opciones de enfoque automático general (cuadrado negro), especifique los parámetros con precisión como se muestra. Tenga en cuenta que el canal de enfoque automático (cuadrado rojo) debe especificar el canal de fluorescencia de canal nuclear en experimentos de lineaje.
Nota: Si el desfase máximo es superior a 5 μm, las imágenes tienden a desviarse del foco. - Clic, Plugins > adquisición de herramientas > superposición de patrones.
- En la ventana superposición de patrón , haga clic en Mostrar cuadrícula.
- En la ventana de asi diSPIM , haga clic en la pestaña de navegación .
- Haga clic en casillas de verificación para viga y hoja de ruta a o B y, a continuación, haga clic en Live. Comienza la adquisición de imágenes. Se abre una ventana de vista en vivo . Seleccione la región de análisis de enfoque automático del embrión dibujando una caja alrededor del embrión en el canal seleccionado de 6,1.
Nota: por lo general, capturamos 420 puntos de tiempo para 10 embriones por sesión de imágenes. Los datos sin procesar por sesión de imagen suelen ser de 1,7 TB, mientras que los datos procesados por StarryNite y desconalizados son 1.4 TB (consulte los pasos 9 y 10). Recomendamos el uso de HDD de gran capacidad (18TB en nuestro sistema actual) para la adquisición de imágenes y plataformas en la nube para el almacenamiento de imágenes. - Haga clic en iniciar adquisición en la pestaña "adquisición " para comenzar la captura de imágenes multidimensionales a largo plazo (figura 2B).
7. abrir imágenes de micro-Manager RAW en CytoSHOW
- Descargue el paquete de software de http://dispimlineage.wormguides.org.
Nota: el paquete de software se descargará como un archivo. zip y tendrá que ser extraído a la "C:\" directorio antes de su uso. Más detalles para la instalación se dan en http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm. - Haga doble clic en el archivo C:\citoshowextrasforc\cytoshow_app.jnlp para comenzar a ejecutar cytoshow.
- Elija el archivo de Menú > nuevo > monitor Dispim (micro-Manager). Localice la carpeta del conjunto de datos raíz donde se guardaron las carpetas de punto de tiempo de adquisición. Seleccione cualquier carpeta de punto de tiempo y haga clic en abrir. Las ventanas de navegación multidimensionales (denominadas ventanas de monitor Dispim ) se abren automáticamente tanto para spima como para SPIMB (figura 3a).
Nota: estas ventanas supervisarán la carpeta de datos raíz para las pilas de puntos de tiempo RAW recién guardadas (en el caso de que todavía se esté grabando una muestra). Después de adquirir cada nuevo punto de tiempo, cada una de las ventanas que supervisan los distintos brazos SPIM y las posiciones de la muestra se actualizarán para mostrar todo el DataSet 4D multicanal para cada embrión.
8. generación de imágenes de proyección máx con CytoSHOW
Incluso antes de la deconvolución, los datos sin procesar se pueden procesar rápidamente para evaluar las características globales de la muestra.
- Haga clic en el botón Z-MIP en el panel lateral izquierdo de la ventana de la imagen (Figura 3A, rectángulo rojo) para hacer proyecciones de máxima intensidad a través de la profundidad completa y el curso a tiempo completo de una posición determinada o del brazo spim. Aparecerá una ventana de hyperstack de proyección Z .
- En la ventana de hiperpila de proyección Z , establezca el tipo de proyección en intensidad máxima. Especifique qué canales, sectores y fotogramas de punto de tiempo se procesan según las preferencias del usuario.
- Haga clic en Aceptar cuando haya finalizado.
- Seleccione la ubicación de la carpeta para guardar las salidas de intensidad máxima desde la ventana de diálogo de archivo y, a continuación, haga clic en Aceptar. Permita un tiempo (15 – 20 minutos, dependiendo del tamaño del DataSet y de la potencia de procesamiento del equipo) para que CytoSHOW genere imágenes de proyección.
9. Análisis de linajes celulares en datos volumétricos isotrópicos de alta resolución
- Opcional: con los datos sin procesar abiertos a través del monitor Dispim en cytoshow, seleccione la herramienta de selección de polígonos (Figura 3A, flechas negras) y haga clic justo fuera de los bordes anterior, posterior, dorsal y ventral del embrión (en ese orden exacto) para generar un patrón de "pajarita" sobre el embrión. Hacer para ambas vistas (SPIM-A y SPIM-B, figura 3a).
Nota: esta selección especifica la región elíptica de interés (ROI) en la que se centra el embrión y registra el eje anterior-posterior del embrión. El patrón bowtie cues CytoSHOW que el usuario planea especificar una rotación precisa de los volúmenes finales fusionados isotrógicamente en una orientación que es óptima para los análisis de lineaje por StarryNite/AceTree. En los casos en que el lineaje de StarryNite no forma parte del plan experimental, se pueden elegir otras herramientas y formas de selección para establecer el ROI para el procesamiento de imágenes. - Si se han fotografiado varios embriones simultáneamente utilizando la opción de adquisición de varias posiciones, abra y realice el paso 9,1 para todos los embriones. Esto permitirá la ejecución paralela de futuros pasos para todos los embriones en una sola sesión. Cierre las ventanas SIMA y SPIMB para los embriones que no desee procesar.
- Haga clic en el botón dispim en el panel lateral izquierdo de la ventana del monitor dispim (figura 3a, resaltado en amarillo). Esto revela un subpanel de controles específicos para el procesamiento de diSPIM.
- Alinee los canales verde y rojo para cada brazo SPIM. Debido a que los divisores de canal de emisión se utilizan para capturar imágenes rojas y verdes distintas simultáneamente en la misma cámara, es importante alinear visualmente el registro exacto de píxeles de estos dos campos de imagen físicamente adyacentes cuando se superpone. La reutilización de los mismos ajustes de alineación suele ser factible en muchas sesiones de imágenes consecutivas, pero debe comprobarse (como en los pasos 9.4.1 – 9.4.5).
- Comenzando con el panel SPIMA, seleccione el canal rojo moviendo la barra de desplazamiento cm a la izquierda (figura 3a, flecha naranja, panel izquierdo).
- Con los Ajustadores x, y y z (figura 3a, cuadrado naranja), desplace el canal rojo para que coincida con el verde.
- Haga clic en el botón Dispim (figura 3a, resaltado en amarillo), para cerrar el subpanel y desencadenar la propagación de los mismos turnos a todas las demás ventanas de posición .
- Confirme que la alineación correcta se propague a otros fotogramas y puntos de tiempo moviendo la barra de desplazamiento "z" (figura 3a, flecha azul, panel izquierdo) y/o barra de desplazamiento "t" (figura 3a, flecha verde, panel izquierdo). Si se realizaba una adquisición de múltiples posiciones y se fotografiaban varios embriones (paso 5,3), la alineación también debería haberse propagado a esos embriones. Confirme examinando también los números para los Ajustadores x, y y z (figura 3a, cuadrado anaranjado, que debe ser el mismo para el panel spima de todos los embriones).
- Repita los pasos 9.4.1 – 9.4.4 para la ventana del monitor SPIMB diSPIM (figura 3a, panel derecho).
- Haga clic en el botón "Dispim" y luego en el botón "Fuse" (Figura 3A, rectángulo azul) para abrir un cuadro de diálogo llamado "Deconvolve/Fuse Dispim Raw Data Volumes" (figura 3B). Establezca los parámetros como se muestra en la figura 3B. Estos parámetros se abordan brevemente en los siguientes subpasos:
- Establezca el registro de claves en el canal 1 (láser de 488 nm) o 2 (láser de 561 nm). Seleccione el canal con una señal más densa o más ubicua. Para los experimentos de lineaje siempre Seleccione el canal utilizado para la imagen de la fluorescencia histona nuclear omnipresente.
- Establezca la orientación relativa de los volúmenes de entrada en + 1 o-1. El índice de orientación correcto depende de las colocaciones de cámara de diSPIM específicas (figura 4).
Nota: si es incierto, pruebe cada opción duplicando un solo punto de tiempo de la ventanadel monitor spim a y B dispim , siguiendo los pasos 9.1 – 9.12 y eligiendo arbitrariamente una orientación relativa de volúmenes de entrada para probar. Las orientaciones incorrectas generarán imágenes borrosas con artefactos, mientras que las orientaciones correctas generarán imágenes claras. El valor de orientación relativa de los volúmenes de entrada que produce la imagen nítida se puede reutilizar para todos los datos futuros del instrumento dispim especificado. - Elija si el volumen fusionado debe orientarse de la misma forma que el volumen de entrada A o B (según la preferencia del usuario).
- Seleccione "registro nuevo para cada volumen". Esta opción controla cómo SpimFusion calcula matrices de registro para cada par de volúmenes en cada punto de tiempo. La opción "Fresh" permite que el algoritmo optimice el registro de manera adaptable en cada punto de tiempo.
- Establezca el número de iteraciones de deconvolución en 10. Este número tiende a producir de forma fiable la alta resolución deseada de una manera eficiente en el tiempo.
- Opcional: si se desea el lineaje automático (muy recomendado), compruebe el Inicio automático de lineado de StarryNite de volúmenes fusionados. Esta opción iniciará starrynite automáticamente para segmentar y rastrear las celdas en los volúmenes imágenes producidos por spimfusion.
- Opcional: para una mayor precisión en el lineaje automatizado, es mejor reposicionar los volúmenes de embriones fusionados isotrógicamente en la orientación "ADL" (Anterior [x-West], Dorsal [y-norte], izquierda [z-Near]) canónica. Seleccione la opción definir orientación de salida de volumen en la vista preliminar para indicar esta opción. CytoSHOW responderá procesando un par inicial de volúmenes fusionados isotrógicamente, permitiendo al usuario observar de cerca y especificar las rotaciones necesarias para lograr el registro de ADL.
- Haga clic en sí una vez seleccionados todos los parámetros.
- Especifique el Directorio de salida en el que desea guardar los archivos procesados. Haga clic en Aceptar.
- Opcional: Si la opción definir orientación de salida de volumen en vista previa está seleccionada, establezca la barra de desplazamiento t (figura 3a, flecha verde, panel izquierdo) en la ventana spim-a en el punto de tiempo inicial en el que las celdas ABa y ABP han alcanzado la metafase. Establezca la barra de desplazamiento t en la ventana spim-B en la etapa de desarrollo posterior de la coma . Esto ayudará a especificar la orientación de ADL.
- Opcional: haga clic en Aceptar cuando esté listo. Si se seleccionó la opción de previsualización en 9.5.7 anterior, solo dos volúmenes de previsualización se fusionarán isotrótricamente para los puntos de tiempo indicados por los reguladores t de las ventanas de imagen SPIM-A y SPIM-B. Estos dos puntos de tiempo de previsualización se pueden utilizar para especificar la realineación precisa de los volúmenes de embriones de salida a la orientación de ADL, como se explica a continuación.
- Localice el recién visualizado 3DProjY_Decon-Fuse_.... Ventana. Mueva la barra de desplazamiento t al punto de tiempo 2 de esta ventana de vista previa. Mueva el regulador Z hasta que se muestre la vista directamente hacia abajo del eje largo del embrión.
- Mueva la barra de desplazamiento t de nuevo al punto de tiempo 1 de la 3DProjY_Decon-Fuse_.... Ventana. Elija la herramienta de selección de líneas y dibuje una selección de línea desde la celda EMS (núcleo más redondo ventral) a través del plano de las placas de metafase AB-Cell.
- Haga clic en el botón Orange diSPIM Preview en el 3DProjY_Decon-Fuse_.... Ventana. Los ajustes finos a la orientación del volumen imágenes previsualizados se guardarán para su uso en el procesamiento del conjunto de datos completo.
- Opcional: el diálogo Deconvolve/Fuse diSPIM Raw Data Volumes reaparecerá, al igual que en el paso 9,5 anterior. Haga clic en sí sin seleccionar la opción definir orientación de salida de volumen en vista previa . Especifique la carpeta de salida para la ejecución completa de procesamiento de datos.
- Establezca las barras de desplazamiento t (figura 3a, flecha verde, panel izquierdo) de las ventanas del monitor dispim en el punto de tiempo inicial (spima) y el punto de tiempo de finalización (SPIMB) del intervalo completo de imágenes que se van a procesar. A continuación, haga clic en Aceptar.
- A medida que avanza SpimFusion, CytoSHOW abre y refresca una ventana multidimensional que muestra el volumen fusionado isotrópico en rodajas-4D para cada embrión, así como dos ventanas con proyecciones de intensidad máxima de 4D rotatorias del volumen isotrópico. Durante este tiempo, no interrumpa ni cierre ninguna ventana de cytoshow hasta que se completen la fusión isotrópica y el rastreo de linaje.
- Opcional: tenga en cuenta que una vez que la pantalla de bienvenida de StarryNite ha aparecido y más tarde desapareció, se ha completado la canalización completa de procesamiento de datos. Esta ventana no se debe cerrar durante el procesamiento o StarryNite se interrumpirá.
10. abrir la serie de trazas de linaje StarryNite en AceTree (opcional)
- Abra la versión personalizada de "AceTree_16BitCompat. jar" proporcionada.
- Elija el menú archivo > Abrir archivo de configuración. Localice el Directorio de salida indicado previamente a cytoshow. Abra la subcarpeta Decon_Fuse_... _ pos [n] para el embrión [n]. Seleccione aaa_edited. XML y abra.
- Utilice el menú de AceTree editar > Editar herramientas para abrir la pista de edición y ajustar o eliminar ventanas de celdas.
- Haga clic en el círculo semisombreado figura 5b, cuadrado rojo para ajustar las intensidades rojo y verde.
- Proceda con la visualización y edición de linaje como se describió anteriormente como5,6,8 (los manuscritos también se incluyen en nuestro paquete de descarga).
Representative Results
Primero validamos la viabilidad de los embriones imágenes utilizando los parámetros del protocolo para la adquisición de dispim (secciones 1-6). Se fotografiaron diez embriones simultáneamente a 20 ° c, un volumen/embrión/minuto, desde la etapa de 2 células hasta la etapa de 2 veces (7,5 horas, 451 volúmenes/embrión). Para monitorear las divisiones celulares a lo largo de la embriogénesis, usamos la cepa BV514, la cual expresa ubicuamente las construcciones del reportero Mcherry::histone de la matriz transgénica integrada ujIs11324. La figura 6 muestra un cronograma de esta primera mitad de desarrollo embrionario para uno de los embriones con imagen. Cada imagen representa una proyección de una sola vista de máxima intensidad (producida por los pasos 7-8) del embrión con imagen. Descubrimos que los protocolos optimizados no indujeron ninguna fototoxicidad detectable a los embriones, como se evaluó por la temporización de las divisiones celulares (no mostradas), el tiempo de eclosión y la temporización relacionada con los hitos del desarrollo (figura 6 y referencias1 , 25 , 26).
Luego aplicamos el protocolo para analizar la dinámica de crecimiento de las neuronas individuales en el desarrollo de embriones. Imaginamos DCR7692 (olaex4655), una cepa de nematodo transgénico que expresa GFP en el promotor del neuropéptido FLP-19 en un subconjunto de células no identificadas (DACR2819, PFLP-19 (3.6 KB):: Syn21:: GFP:: caax::p 10 3 ' UTR) . Siguiendo los pasos del protocolo delineado aquí, determinamos que las células no identificadas corresponden a las neuronas motoras RMDDL y RMDDR, a la célula del canal excretor, y a dos células musculares (figura 7). A continuación, examinamos y cuantificamos la dinámica de consecuencia de las neuronas rmddl y rmddr. Observamos que las neuronas RMDDL y RMDDR tienen forma oblicua desde los 360 minutos posteriores a la fertilización, con el eje celular más largo que representa el eje subsiguiente para el crecimiento de la neurita (figura 7 y película S1). Usando el plugin "simple neurita Tracing" en Fiji y aplicándolo a reconstrucciones 3D de volúmenes fusionados isotrógicamente, luego cuantificamos el crecimiento estereotipado de las neuritas rmddl y rmddr para seis embriones. Determinamos que las dinámicas de consecuencia fueron estereotipadas para rmddl y rmddr a través de embriones (en la presente llamada rmdd). A partir de 385-410 minutos después de la fertilización, la neuritas RMDDs extendida 6.0 ± 0.5 μm (media ± SEM; n = 12 neuritas) anterior de los cuerpos celulares (figura 7b, C, I). A partir de 415-445 minutos después de la fertilización, ambas neuritas se extienden dorsalmente en y alrededor del anillo del nervio presuntuoso (asterisco en la figura 7D). En promedio, cada neurita RMDD extendió 11,0 ± 0,6 μm (media ± SEM; n = 12 neuritas) del cuerpo celular antes de reunirse sincrónicamente con su contraparte contralateral en el vértice del anillo (figura 7i). Es importante destacar que nuestros resultados representativos demuestran que somos capaces de examinar, comparar y cuantificar las características de desarrollo neuronal para celdas de identificación única mediante el uso de nuestro protocolo integrado (figura 7 y figura 8).
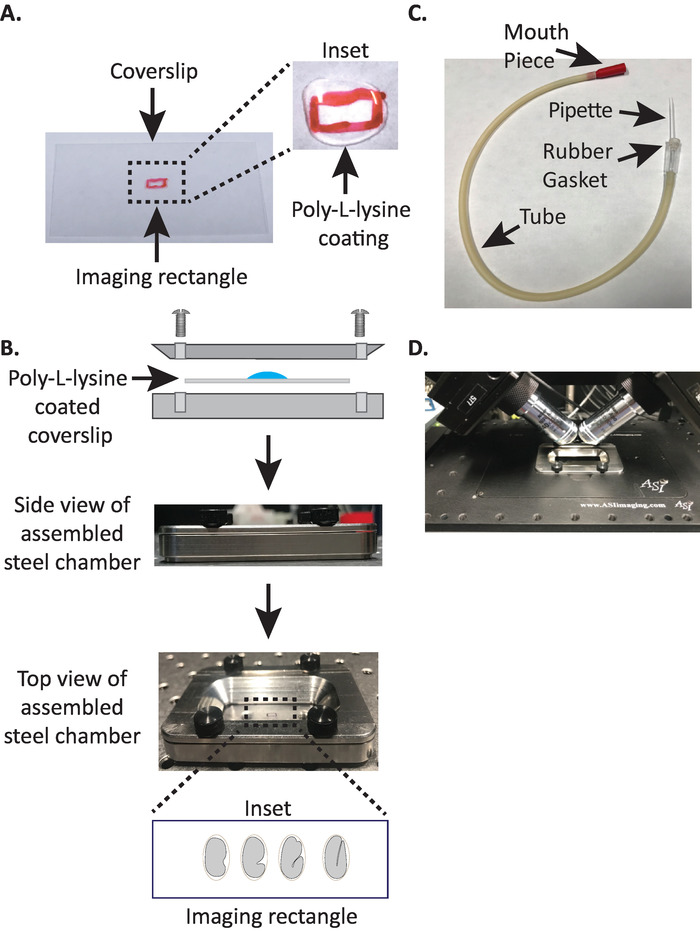
Figura 1: configuración de montaje de muestra de diSPIM. (A) preparación de cubreobjetos con poli-L-lisina. En el recuadro, se utilizaron 10 μL de lisina de poli-L para recubrir la cobertura durante 5 minutos. Poly-L-lisina permite que la cáscara de huevo del embrión se pegue firmemente a la cubierta en el rectángulo. (B) esquema de la cámara de imagen de acero y de la cámara montada. En el recuadro, los embriones representativos se orientan con el eje anterior-posterior perpendicular al eje largo en el Coberto. (C) tubo aspirador montado con pipetas microcapilar. (D) cámara de imagen de acero montada en el soporte de la muestra bajo objetivos dispim 40x. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 2: configuración de imágenes diSPIM a largo plazo en micro-Manager. (A) parámetros de potencia láser dispim recomendados (rectángulo rojo) optimizados para imágenes prolongadas y reduciendo la fototoxicidad (evaluado por una mayor tasa de eclosión de embriones de C. elegans ). Fije el laser 561 nm a 79 μW (0,25) y al laser 488 nm a 179 μW (0,5). Tenga en cuenta que la calibración exacta de la configuración de software a la potencia láser varía entre las instalaciones de diSPIM. Se recomienda que los usuarios miden y calibran la energía del laser para alcanzar 79 μW (561 nm) y 179 μW (488 nm) la energía del laser. (B) parámetros de dispim para guardar datos (rectángulo verde), ajustes de volumen (cuadrado azul) y ajustes de corte (cuadrado naranja). (C) parámetros de enfoque automático de dispim para imágenes a largo plazo de la embriogénesis C. elegans (ver los pasos 6.1-6.6). Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 3: visualización de imágenes y configuración de procesamiento de datos mediante CytoSHOW. (A) imágenes de dispim RAW abiertas por cytoshow. CytoSHOW es capaz de abrir imágenes capturadas por ambas trayectorias de la cámara (SPIM a y B). Estas imágenes RAW se abren en ventanas multidimensionales llamadas monitor Dispim. En el monitor Dispim, se genera un "patrón de pajarita" para seleccionar los bordes anterior, posterior, dorsal y ventral del embrión (ver paso 9,1). Las selecciones de lazo indican la orientación del embrión para la deconvolución y el rastreo de lineaje asistido por StarryNite. (B) parámetros optimizados utilizados para generar imágenes isotrópicas. En la ventana Deconrete mientras se adquiere , establezca los parámetros especificados en los pasos 9.5.1-9.5.8. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 4: configuración de la cámara diSPIM. (A) fotografía de las colocaciones y orientaciones de la cámara dispim. (B) representación de las rotaciones de + 90 grados de spim a para igualar las imágenes spim B recopiladas. (C) volúmenes de entrada relativos al índice de orientación + 1 en función de la configuración de la cámara de dispim (véase el paso 9.5.2). Rotamos SPIM una imagen (s) + 90 grados alrededor del eje Y antes del registro para que coincida con la imagen (s) de SPIM B. Barras de escala = 10 μm. las imágenes son representativas de una sola vista, proyecciones de máxima intensidad y imágenes de deconvolución de embriones 1,5-fold con núcleos etiquetados (561-nm, rojo) y neuronas (488-nm, verde). Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 5: curation y edición C. elegans linaje embrionario en acetree. (A) utilizamos acetree para editar las trazas de linaje de starrynite (véanse las referencias5,6,8; los manuscritos también se incluyen en nuestro paquete de descarga). AceTree mostrará C. elegans nombres sistemáticos para cada núcleo (rectángulo verde) al completar los pasos 10.1-10.2. Esta ventana (A) proporciona información (rectángulo negro) sobre cada celda en el trazado de linaje (ABa, resaltada en azul) que ayudan a guiar a los usuarios cuando rastrean y editan los seguimientos de linaje. Se recomienda que los usuarios verifiquen y comparen las células lineadas y sus posiciones con el linaje de células embrionarias C. elegans previamente reportado por Sulston et al.1 , además, si los usuarios están interesados en localizar celdas específicas en el serie de datos desconalizados (véase a continuación, B) Introduzca el nombre sistemático de C. elegans en la barra de búsqueda (rectángulo naranja). (B) la serie de datos desconalizados del usuario también se abre automáticamente al completar los pasos 10.1-10.2. Aquí se muestra una imagen fusionada isotrógicamente de un embrión de etapa de cuatro células con núcleos etiquetados en rojo. Durante el seguimiento de un núcleo, los usuarios deben cambiar la intensidad de la imagen (cuadrado rojo) y navegar a través del tiempo y z utilizando las teclas de flecha en su teclado (tiempo-izquierda/derecha, z-arriba/abajo). (C) dibujos animados 3D del punto de tiempo en (B) con ciertas funcionalidades (rectángulo púrpura) que permite la visualización 3D giratoria. Para obtener una descripción general del acetree y su funcionalidad 3D, consulte las referencias5,6,8. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 6: dinámicas de desarrollo cronometradas de embriones de C. elegans en el dispim. Panel superior, imágenes dispim que muestran la primera mitad del desarrollo embrionario de uno de los embriones imágenes (strain BV514 ujIs11324). Los embriones fueron imágenes continuamente, cada minuto durante 7,5 horas (a 20 ° c). Las dos primeras imágenes del panel superior representan embriones de 4 y 8 celdas con núcleos (rojos) y posiciones de cuerpos polares (esferas rojas densas, marcadas con asteriscos azules). Cada imagen representa una proyección de una sola vista de máxima intensidad del embrión con imagen. Barras de escala = 10 μm. La línea de tiempo (barra horizontal) representa los minutos posteriores a la fertilización (m.p.f.) del desarrollo de embriones de C. elegans . Validamos que los parámetros de nuestro protocolo para la adquisición de dispim no indujeron ninguna fototoxicidad detectable a los embriones imágenes como evaluados por la viabilidad, el momento de las divisiones celulares, el tiempo de eclosión y el momento de los hitos del desarrollo (ver referencias 1 , 25 , 26). observamos que el calendario de los hitos del desarrollo fue reproducible en los embriones con nuestros parámetros de imagen (SEM ± 8,174 minutos para sesiones de imágenes de 6,4 horas de duración; n = 10). Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 7. Identificación celular y caracterización de células individuales de la dinámica de crecimiento de neurita en el desarrollo de embriones C. elegans . Imágenes de doble color de una cepa hecha por el cruce de BV514 ujIs11324 (para lineaging) y DCR7692 (olaex4655), una cepa de nematodos transgénicos que expresa GFP del promotor del neuropéptido FLP-19 en un subconjunto de células no identificadas. (A-H) Siguiendo los pasos del protocolo delineado aquí, determinamos que las células no identificadas corresponden a las neuronas motoras RMDDL y RMDDR (flechas amarillas), a la célula del canal excretor (flechas azules), y a dos células musculares (flechas blancas). (I) cuantificación de la dinámica de consecuencia de las neuronas rmddl y rmddr mediante el uso del complemento de Fiji "simple neurita Tracing" y aplicarlo a reconstrucciones 3D de volúmenes fusionados isotrógicamente. Observe cómo tanto rmddl como rmddr muestran la dinámica de consecuencia estereotipica, cada una de las cuales se extiende sincrónicamente para una longitud total de 11.0 ± 0,6 μm (media ± SEM; n = 12 neuritas) y reunión en el vértice dorsal del anillo del nervio (ver también película S1). Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 8: examen de imágenes isotrópicas diSPIM de morfologías neuronales en embriones C. elegans . Visualización isotrópica de neuronas AVHL y AVHR (flechas amarillas). Utilizando el diSPIM, las morfologías neuronales pueden capturarse produciendo imágenes de cuatro dimensiones (4D) con una resolución espacial isotrópica de aproximadamente 330 nm. El diSPIM permite a los usuarios rotar virtualmente volúmenes de imágenes con resolución idéntica en todas direcciones. Las imágenes en la a-D son proyecciones de máxima intensidad del mismo volumen de imagen dispim fusionada isotrógicamente de rotaciones distintas alrededor del eje largo del embrión. Barras de escala = 5 μm. por favor, haga clic aquí para ver una versión más grande de esta figura.
Película complementaria S1: C. elegans embrión que se desarrolla de 280 a 434 minutos después de la fertilización. Película isotrópica de cepa DCR7692 (olaex4655) que expresa ujIs113 ubiquitemente con DACR2819 neuritas rmdd de etiquetado escasamente (Figura 7A-d, flechas amarillas). DACR2819 también etiqueta dos células musculares (Figura 7A-d, flechas blancas) y la célula del canal excretor (Figura 7A-d, flecha azul) durante el desarrollo embrionario (Figura 7A-d). Barras de escala = 10 μm. por favor, haga clic aquí para descargar este archivo.
Discussion
C. elegans se destaca como el único organismo con las posiciones finales y la conectividad de cada neurona adulta conocida27. Sin embargo, las dinámicas de desarrollo que conducen a la organización de los circuitos de trabajo y las redes que componen el connectome C. elegans permanecen desconocidas. Basándonos en las oportunidades que surgen de los avances en la microscopía de luz, ahora podemos capturar y analizar las posiciones celulares, la morfogénesis y la neurogénesis a lo largo del desarrollo embrionario de C. elegans .
El procedimiento que hemos descrito y que rutinariamente utilizamos en el laboratorio produce imágenes 4D-isotrópicas de neuronas etiquetadas y núcleos para el lineaje celular en embriones C. elegans . Lo que es más importante, hemos optimizado las condiciones de imagen a largo plazo con el diSPIM y las capacidades de lineado semi-automatizadas acopladas con imágenes de alta resolución para mejorar la velocidad y la precisión del análisis de la embriogénesis C. elegans . Este protocolo integrado permitirá a los usuarios visualizar e identificar las células y cuantificar las características tridimensionales, tales como la migración de la neurita y la fasciculación del axón a través del inicio de las contracciones tempranas. Este procedimiento se puede adaptar fácilmente a cualquier instalación con un sistema ASI diSPIM, y recomendamos este sistema específicamente para este protocolo. Otras formulaciones de SPIM ofrecidas comercialmente pueden diferir de la configuración de ASI en la cámara de muestreo y propiedades ópticas. Sin embargo, los datos exportados desde otras plataformas también se pueden poner a través de nuestra canalización de datos. Por lo tanto, es factible la evaluación de su valor en lineaging, una prueba exigente de calidad de imagen y estabilidad del instrumento. A pesar de que utilizamos activamente el diSPIM para la imagen periódica de otros especímenes (como los embriones de Drosophila y cebra), el análisis de lineaje descrito y exhaustivo de los embriones todavía se limita actualmente a las especies de nematodo. Para muestras más grandes o gruesas, optamos por utilizar enfoques de escaneo de etapa, que escanean las muestras a través de una lámina de luz estacionaria. Kumar et al. han demostrado previamente esta sección mejorada de diSPIM para producir imágenes de alta calidad de muestras gruesas sin modificaciones adicionales al diSPIM10.
Los pasos críticos dentro del protocolo incluyen el montaje de embriones C. elegans en el recubrimiento recubierto de poli-L-lisina, adquisición de datos y procesamiento de datos. La cosecha y el montaje de embriones C. elegans en el recubrimiento de vidrio puede ser difícil para los usuarios inexpertos, pero aquí proporcionamos un protocolo detallado de los pasos clave para facilitar el aprendizaje. Si se desea una toma de imágenes a largo plazo, obtenemos mejores resultados cosechando embriones de cuatro células o anteriores de 8-10 adultos jóvenes28. Tenga en cuenta que los adultos mayores son menos deseables para cosechar embriones de etapa temprana porque tienden a contener embriones más viejos en el útero y óvulos no fertilizados. En lo que respecta a los embriones de montaje, los problemas como el bloqueo en el aspirador montado (Piera bucal) o una abertura demasiado grande en la picada microcapilar pueden impedir el montaje y la orientación adecuados de los embriones. Para prepararse para obtener imágenes óptimas, realizamos pruebas previas a la adquisición en embriones pre-twitching tempranos y tardíos para comprobar el rendimiento de las láminas de luz, las cámaras, los objetivos y el enfoque automático. Obtenemos mejores resultados cuando todas estas operaciones se prueban y producen imágenes de alta calidad durante nuestras pruebas previas a la adquisición. Esto es especialmente relevante para generar imágenes con resolución espacial isotrópica, para las cuales las imágenes RAW adquiridas a partir de ambas vistas (objetivos) deben ser de alta calidad. Después de la adquisición, los volúmenes adquiridos para cada vista se procesan para producir imágenes isotrópicas. Es importante utilizar una tarjeta de unidad de procesamiento de gráficos (GPU) adecuada como se describe en este protocolo (ver más abajo). Esto mejora la velocidad de procesamiento a la que se generan las imágenes fusionadas isotrógicamente, acortando el tiempo a los análisis de datos. También es imperativo que los usuarios están ejecutando la última versión de CytoSHOW y están utilizando los parámetros proporcionados con nuestro paquete de descarga para StarryNite auto-lineaging. Si los usuarios están interesados en utilizar el lineamiento automático para otras muestras (p. ej., zebrafish, Drosophila, etc.), se requerirá una optimización adicional de los parámetros utilizados en starrynite (ver referencias3,4).
Aunque nuestro protocolo integrado proporciona imágenes y resultados de lineaje en el embrión pre-twitching, los usuarios deben ser conscientes de que el lineaje automatizado en el embrión post-twitching no es actualmente factible: las posiciones nucleares cambian en el orden de los segundos en el embrión post-twitching, demasiado rápido para permitir el rastreo de linaje. Sin embargo, el dispim ha demostrado de hecho una capacidad prometedora para capturar eventos de Neurodesarrollo y rastrear algunas posiciones celulares en las etapas post-twitching de la embriogénesis23,29. Si los usuarios están interesados en examinar el embrión post-twitching, el diSPIM proporciona la velocidad para obtener instantáneas volumétricas y realizar un seguimiento de los eventos de Neurodesarrollo finos, como el crecimiento de la neurita, en embriones que se mueven rápidamente.
Este Protocolo será fundamental para la terminación celda por célula del Atlas de WormGUIDES30, ya que proporcionará un enfoque integrado con imágenes isotrópicas de alta resolución para identificar y capturar morfologías 3D de neuronas etiquetadas durante los primeros 430 minutos de embriogénesis. Tal y como está, el prototipo del Atlas WormGUIDES proporciona posiciones nucleares de las células en el embrión en desarrollo y tiene como objetivo capturar la dinámica de desarrollo de un subconjunto de neuronas embrionarias. Este Protocolo será una clave para la integración de las neuronas en desarrollo adicionales en el Atlas30de wormguides.
Nuestro protocolo integrado también simplificará la exploración de nuevos perfiles de expresión génica en el embrión de C. elegans . En C. eleganstransgénicos, muchos promotores específicos de células controlan espacialmente y temporariamente la expresión transgénica. Mientras que los patrones de expresión de la mayoría de los genes se han caracterizado extensivamente en el animal adulto31,32,33,34, casi todos todavía tienen que ser caracterizados en el desarrollo (especialmente embrión en etapa tardía). El C. elegans promoterome ha sido un recurso útil para la comunidad de gusanos para impulsar la expresión transgénica específica de la célula, así como para determinar si la función génica es autónoma de células o no autónoma. La captura de los patrones de expresión dinámica y de alta resolución isotrópica de los genes, e identificar con precisión las células que expresan a través del lineaje será valiosa para muchos en la comunidad científica.
La embriogénesis comprende dos procesos principales entrelazados, la diferenciación celular y la morfogénesis de los tejidos. Se sabe mucho sobre los mecanismos y moléculas que definen distintos tipos de células durante el desarrollo de C. elegans. Sin embargo, se sabe poco sobre los mecanismos importantes para la migración celular, la adherencia celular y la forma celular en el embrión de C. elegans . Con el linaje de células invariantes de C. elegans conocido, nuestro protocolo nos permite discernir fácilmente la microanatomía 3D catalogada del embrión durante la morfogénesis a nuevos niveles de detalle: p. ej., fasciculación Axon, sinaptogénesis y actividad neuronal. Ardiel et al. han demostrado previamente el poder del diSPIM para capturar los transitorios de calcio a nivel de una sola neuronas en los embriones23de C. elegans . Muchos otros aspectos de la fisiología del desarrollo están maduros para la investigación por estos métodos.
Por último, este protocolo está en gran medida automatizado y reduce sistemáticamente el tiempo que se tarda en generar imágenes de deconvolución y realizar lineaje celular a través de StarryNite y Acetree. Las estrategias de software utilizadas en este protocolo se pueden aplicar a muchas cuestiones de la biología lejos de los campos muy específicos en los que los hemos demostrado aquí.
Detalles sobre compatibilidad de software y acceso de descarga
La información sobre micro-Manager y plugins para imágenes de diSPIM están disponibles en http://dispim.org/software/micro-manager y https://micro-manager.org/wiki/ASIdiSPIM_Plugin.
La canalización de procesamiento de datos requiere actualmente un sistema operativo Windows. Hemos incluido un único archivo de almacenamiento para simplificar la instalación de todos los programas de procesamiento de datos y archivos de soporte necesarios. Está disponible para su descarga en http://dispimlineage.wormguides.org.
CytoSHOW (http://run.cytoshow.org/) se basa en la plataforma de análisis de imágenes de código abierto y ampliamente utilizada, ImageJ (V1). Java debe estar instalado y actualizado en el equipo para utilizar CytoSHOW, y las actualizaciones de CytoSHOW se implementan automáticamente a través de Java Web Start. Muchas funciones basadas en ImageJ de CytoSHOW son como se describe e ilustra en https://imagej.nih.gov/ij/docs/examples/index.html. CytoSHOW se ha personalizado para Mostrar datos sin procesar multidimensionales de la ASI diSPIM, así como otro software de imágenes que crea salida TIFF. En principio, otros sistemas de imagen SPIM multivista podrían ser apoyados por pequeñas modificaciones de CytoSHOW para permitir que este protocolo se lleve a cabo en diferentes sistemas de microscopio.
SpimFusion se escribió en CUDA/C++ con Visual Studio 2013 con el kit de herramientas CUDA v 7.5. La ejecución de SpimFusion requiere hardware informático específico: una tarjeta de unidad de procesamiento de gráficos NVIDIA (GPU) con capacidad de cómputo CUDA 1,0 o superior y un mínimo de 2 GB de memoria de tarjeta gráfica. En el momento de la publicación de nuestro protocolo, SpimFusion está inédito (min Guo y Hari Shroff), pero está disponible en el archivo de paquete de software mencionado anteriormente.
Una versión de StarryNite basada en la línea de comandos especialmente diseñada requiere que MATLAB Compiler Runtime esté disponible de forma gratuita, pero no requiere una licencia para el software de MATLAB comercial. MATLAB Compiler Runtime se incluye en el archivo de paquete de software mencionado anteriormente. El código para StarryNite, tal como se utiliza en este protocolo, es esencialmente igual al utilizado para las imágenes confocales6. Sin embargo, varios aspectos operacionales en la creación de imágenes de entrada para el procesamiento de StarryNite y el manejo de los resultados de StarryNite han sido abordados aquí por métodos en CytoSHOW que permiten una canalización de procesamiento de datos continua para el isótropo diSPIM fusionado Volúmenes. Estos cambios se automatizan mediante el código CytoSHOW que gestiona estos pasos previos y posteriores al procesamiento. Cytoshow también edita una plantilla preoptimizada para dispim específica del parámetro starrynite configurada para ajustar automáticamente el algoritmo de segmentación a la intensidad de fluorescencia de los núcleos en los datos imágenes. Los parámetros únicos utilizados por StarryNite en cada conjunto de datos diSPIM se guardan en un archivo junto con la imagen de salida y los datos de lineaje.
Una versión personalizada de AceTree que funciona con imágenes de 16 bits y mantiene la compatibilidad con la representación de Java3D es la más adecuada para este protocolo. También se incluye en el archivo de paquete de software mencionado anteriormente.
Disclosures
Los autores no tienen nada que revelar.
Acknowledgments
Agradecemos a John Murray por la cepa integrada, ujIs113, para generar la cepa lineaging BV514; Brandon Harvey (NIBIB) para obtener ayuda para probar el protocolo; Jon Daniels y Gary Rondeau (instrumental científico aplicado) para asistencia con el instrumento micro-Manager y diSPIM; y Andrew York y Hank Eden por sus comentarios críticos sobre el sistema diSPIM. También agradecemos al programa del centro de investigación para instituciones minoritarias y al Instituto de Neurobiología Jose del castillo (Universidad de Puerto Rico) por proporcionar una plataforma de reunión y lluvia de ideas. Gran parte de este trabajo se llevó a cabo en el laboratorio biológico marino en Woods Hole a través del programa Whitman. Este trabajo fue apoyado por los programas de investigación intramuros del Instituto Nacional NIH de imágenes biomédicas y Bioingenierías y por el NIH Grant no. U01-HD075602 y no. R24-OD016474. Mark W. Moyle fue apoyado por F32-NS098616 y Leighton H. Duncan fue apoyado por un suplemento de diversidad a R24-OD016474.
Materials
| Name | Company | Catalog Number | Comments |
| Steps 1-4 | |||
| Concavity slides | ThermoFisher Scientific | 1519006 | 5-18mm diameter, 0.6-0.8mm deep, 1.2-1.5mm |
| Dissecting microscope with 10×–50× zoom range | Motic | SMZ-171 | |
| E. coli (OP50) | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Glass coverslips, no. 1.5, 24 × 50 mm | VWR International | 48393-241 | |
| M9 Buffer | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Methyl cellulose | Sigma-Aldrich | H7509-25G | |
| Microcapillary pipette aspirator tube | Sigma-Aldrich | A5177 | |
| Microcapillary pipettes, 0.4-mm i.d | Drummond Scientific | 1-000-800 | |
| Needle, no. 18G x 1 ½ (1.2mm x 40mm) | BD Precision Glide | 305196 | |
| NGM plates | prepared as described by Brenner (1974) | ||
| O-ring for imaging chamber | O-Rings West | M1.5X40 | |
| Pasteur pipette | Corning/Sigma-Aldrich | CLS7095D5X | |
| Platinum wire, 0.5-mm diameter | Sigma-Aldrich | 267201 | |
| Poly-L-lysine | Sigma-Aldrich | P1524 | |
| Stainless steel rectangular chamber (76.0 mm x 50.5 mm) | Applied Scientifics Instrumentations (ASI) | I2450 | |
| Worm Eyelash Pick | Hart, A. C. Behavior. WormBook. (2006). | ||
| Worm Pick | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Name | Company | Catalog Number | Comments |
| Steps 5-6 | |||
| 488 nm long-pass filter | Semrock | LP02-488 RU-2 | |
| 561-nm notch filter | Semrock | NF03-561E-25 | |
| BLP02-561R-25, quantity 2 | Semrock | 561 nm EdgeBasic best-value long-pass edge filter | |
| Control software for bottom camera | Jenoptik | ProgRes CapturePro | |
| diSIPM assembly video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/TAgbr6IrTqw ; http://www.asiimaging.com | |
| diSPIM alignment video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/qnOrg30NNuE | |
| diSPIM imaging PC | Intel | Intel Xeon CPU E5-2630 2.6GHz, 12 cores in total, 64 GB memory, Windows 7 | |
| FF01-525/45-25, quantity 2 | Semrock | 525/45 nm BrightLine single-band bandpass filter | |
| FF555-DI03-25X36, quantity 2 | Semrock | 555 nm edge BrightLine single-edge dichroic beamsplitter | |
| Imaging PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Kumar et al diSPIM Setup | Applied Scientifics Instrumentations (ASI) | Instrument setup for this protocol is identical to Kumar et al 10,11 diSPIM, which makes use of 40x 0.8NA water immersion lenses for imaging. (See steps 5.1 and note) | |
| Micro Manager | Micro-Manager | https://micro-manager.org/ | |
| Modifications to Kumar et al diSPIM Setup (see below) | |||
| Optical table with isolators, 4 feet × 6 feet × 12 inches | TMC | 784-651-02DR and 14-416-34 | |
| Name | Company | Catalog Number | Comments |
| Steps 7-10 | |||
| Analysis PC | Intel | Intel Core i7-8700K CPU 3.70GHz, 6 cores in total, 64 GB memory, Windows 10 | |
| Analysis PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Installation instructions | Software bundle | http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm | |
| Software bundle | Software bundle | http://dispimlineage.wormguides.org |
References
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Developmental Biology. 100 (1), 64-119 (1983).
- Bao, Z., et al. Automated cell lineage tracing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 103 (8), 2707-2712 (2006).
- Santella, A., Du, Z., Bao, Z. A semi-local neighborhood-based framework for probabilistic cell lineage tracing. BMC Bioinformatics. 15, 217 (2014).
- Santella, A., Du, Z., Nowotschin, S., Hadjantonakis, A. K., Bao, Z. A hybrid blob-slice model for accurate and efficient detection of fluorescence labeled nuclei in 3D. BMC Bioinformatics. 11, 580 (2010).
- Boyle, T. J., Bao, Z., Murray, J. I., Araya, C. L., Waterston, R. H. AceTree: a tool for visual analysis of Caenorhabditis elegans embryogenesis. BMC Bioinformatics. 7, 275 (2006).
- Katzman, B., Tang, D., Santella, A., Bao, Z. AceTree: a major update and case study in the long term maintenance of open-source scientific software. BMC Bioinformatics. 19 (1), 121 (2018).
- Murray, J. I., et al. Automated analysis of embryonic gene expression with cellular resolution in C. elegans. Nature Methods. 5 (8), 703-709 (2008).
- Murray, J. I., Bao, Z., Boyle, T. J., Waterston, R. H. The lineaging of fluorescently-labeled Caenorhabditis elegans embryos with StarryNite and AceTree. Nature Protocols. 1 (3), 1468-1476 (2006).
- Wu, Y., et al. Spatially isotropic four-dimensional imaging with dual-view plane illumination microscopy. Nature Biotechnology. 31 (11), 1032-1038 (2013).
- Kumar, A., et al. Using Stage- and Slit-Scanning to Improve Contrast and Optical Sectioning in Dual-View Inverted Light Sheet Microscopy (diSPIM). The Biological Bulletin. 231 (1), 26-39 (2016).
- Kumar, A., et al. Dual-view plane illumination microscopy for rapid and spatially isotropic imaging. Nature Protocols. 9 (11), 2555-2573 (2014).
- Wu, Y., Christensen, R., Colon-Ramos, D., Shroff, H. Advanced optical imaging techniques for neurodevelopment. Current Opinion in Neurobiology. 23 (6), 1090-1097 (2013).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17708-17713 (2011).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136 (12), 1963-1975 (2009).
- Stelzer, E. H. Light-sheet fluorescence microscopy for quantitative biology. Nature Methods. 12 (1), 23-26 (2015).
- Winter, P. W., Shroff, H. Faster fluorescence microscopy: advances in high speed biological imaging. Current Opinion in Chemical Biology. 20, 46-53 (2014).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. Astronomical Journal. 76 (6), 745-754 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. JOSA. 62 (1), 55-59 (1972).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Hart, A. C. Behavior. WormBook. , (2006).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. Chapter 14, Unit14 20 (2010).
- Gualda, E. J., et al. SPIM-fluid: open source light-sheet based platform for high-throughput imaging. Biomedical Optics Express. 6 (11), 4447-4456 (2015).
- Ardiel, E. L., et al. Visualizing Calcium Flux in Freely Moving Nematode Embryos. Biophysical Journal. 112 (9), 1975-1983 (2017).
- Walton, T., et al. The Bicoid class homeodomain factors ceh-36/OTX and unc-30/PITX cooperate in C. elegans embryonic progenitor cells to regulate robust development. PLoS Genetics. 11 (3), e1005003 (2015).
- Altun, Z. F. WormAtlas. , (2002).
- Wood, W. B. Embryology: In the nematode C. elegans. Cold Spring Harbor Laboratory Press. Chapter 8, 215-241 (1988).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314 (1165), (1986).
- Bao, Z., Murray, J. I. Mounting Caenorhabditis elegans embryos for live imaging of embryogenesis. Cold Spring Harb Protoc. (9), (2011).
- Christensen, R. P., et al. Untwisting the Caenorhabditis elegans embryo. eLife. 4, (2015).
- Santella, A., et al. WormGUIDES: an interactive single cell developmental atlas and tool for collaborative multidimensional data exploration. BMC Bioinformatics. 16, 189 (2015).
- Dupuy, D., et al. A first version of the Caenorhabditis elegans Promoterome. Genome Research. 14 (10B), 2169-2175 (2004).
- Reece-Hoyes, J. S., et al. Insight into transcription factor gene duplication from Caenorhabditis elegans Promoterome-driven expression patterns. BMC Genomics. 8 (27), (2007).
- WormBase. , Available from: https://www.wormbase.org (2019).
- Lee, R. Y. N., et al. WormBase 2017: molting into a new stage. Nucleic Acids Research. 46 (D1), D869-D874 (2018).



