Overview
Quelle: Alexander S. Gold1, Tonya M. Colpitts1
1 Department of Microbiology, Boston University School of Medicine, National Emerging Infections Diseases Laboratories, Boston, MA
Transduktion ist eine Form des genetischen Austauschs zwischen Bakterien, die Bakteriophagen oder Phagen verwenden, eine Klasse von Viren, die ausschließlich prokaryotische Organismen infiziert. Diese Form der DNA-Übertragung, von einem Bakterium zum anderen durch einen Phagen, wurde 1951 von Norton Zinder und Joshua Ledererg entdeckt, die den Prozess als "Transduktion" bezeichneten (1). Bakteriophagen wurden erstmals 1915 vom britischen Bakteriologen Frederick Twort entdeckt, 1917 vom französisch-kanadischen Mikrobiologen Felix d'Herelle (2) selbstständig wieder entdeckt. Seitdem sind die Struktur und Funktion dieser Phagen weithin charakterisiert (3), wodurch diese Phagen in zwei Klassen unterteilt wurden. Die erste dieser Klassen sind die lytischen Phagen, die sich bei einer Infektion innerhalb des Wirtsbakteriums vermehren, den bakteriellen Stoffwechsel stören, die Zelle lysieren und Nachkommenphagen freisetzen (4). Als Ergebnis dieser antibakteriellen Aktivität und der zunehmenden Prävalenz antibiotikaresistenter Bakterien haben sich diese lytischen Phagen in letzter Zeit als Ersatzbehandlung für Antibiotika erwiesen. Die zweite dieser Klassen sind die lysogene Phagen, die sich entweder innerhalb des Wirts über den lytischen Zyklus vermehren können oder in einen ruhestillenden Zustand eintreten, in dem ihr Genom in das des Wirts integriert ist (Abbildung 1), ein Prozess, der als Lysogenie bekannt ist, mit der Fähigkeit für Phagen Produktion in mehreren späteren Generationen induziert werden (4).
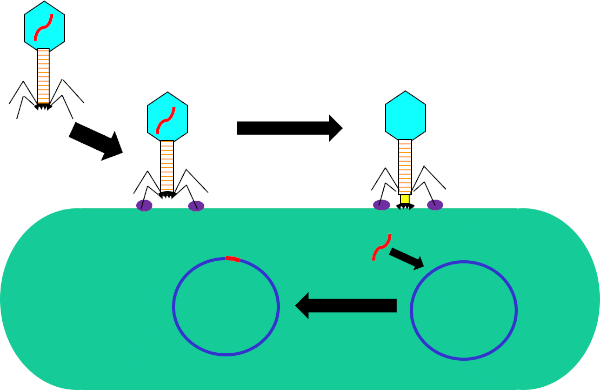
Abbildung 1: Infektion der Wirtszelle durch Bakteriophagen. Adsorption durch den Phagen an die bakterielle Zellwand über Wechselwirkungen zwischen den Schwanzfasern und dem Rezeptor (lila). Einmal auf der Zelloberfläche angekommen, wird der Phagen irreversibel mit der Grundplatte (schwarz) an der Bakterienzelle befestigt, die durch den kontraktilen Mantel (gelb) zur Zellwand bewegt wird. Das Phage-Genom (rot) gelangt dann in die Zelle und integriert sich in das Genom der Wirtszelle.
Während bakterielle Transduktion ein natürlich vorkommender Prozess ist, wurde sie mit moderner Technologie für den Transfer von Genen in Bakterien im Labor verfahren. Durch das Einfügen von Genen von Interesse in das Genom eines lysogenen Phagens, wie Phagen, ist man in der Lage, diese Gene in die Genome von Bakterien zu übertragen und folglich innerhalb dieser Zellen auszudrücken. Während andere Methoden des Gentransfers, wie die Transformation, ein Plasmid für Gentransfer und Expression verwenden, hat die Einfügung des Phagengenoms in das des Empfängerbakteriums nicht nur das Potenzial, diesem Bakterium neue Merkmale zu verleihen, sondern ermöglicht auch natürlich vorkommende Mutationen und andere Faktoren der zellulären Umgebung, um die Funktion des übertragenen Gens zu verändern.
Im Vergleich zu anderen Methoden des horizontalen Gentransfers, wie z. B. Konjugation, ist die Transduktion in den Kriterien, die für Spender- und Empfängerzellen erforderlich sind, ziemlich flexibel. Jedes genetische Element, das in das Genom des verwendeten Phagens passen kann, kann von jedem Stamm von Spenderbakterien auf jeden Stamm von Empfängerbakterien übertragen werden, solange beide für den Phagen freisind, was die Expression der notwendigen Phagenrezeptoren auf der Zelloberflächen. Sobald dieses Gen aus dem Spendergenom herausgebracht und in den Phagen verpackt ist, kann es an den Empfänger übertragen werden. Nach der Transduktion ist es notwendig, für EmpfängerBakterien auszuwählen, die das Gen von Interesse enthalten, für die ausgewählt werden muss. Dies könnte durch die Verwendung eines genetischen Markers, wie z. B. eines FLAG-Tags oder Polyhistidin-Tags, erfolgen, um das Gen von Interesse oder die intrinsische Funktion des Gens im Falle von Genen zu kennzeichnen, die für Antibiotikaresistenzen kodieren. Darüber hinaus könnte PCR verwendet werden, um eine erfolgreiche Transduktion weiter zu bestätigen. Durch die Verwendung von Primern für eine Region innerhalb des Gens von Interesse und den Vergleich von Signal enden mit einer positiven Kontrolle, Bakterien, die das Gen von Interesse haben, und einer negativen Kontrolle, Bakterien, die die gleichen Schritte wie die Transduktionsreaktion ohne Phagen durchliefen. Während die bakterielle Transduktion ein nützliches Werkzeug in der Molekularbiologie ist, spielt sie eine wichtige Rolle bei der Evolution von Bakterien, insbesondere im Hinblick auf den jüngsten Anstieg der Antibiotikaresistenz.
In diesem Experiment wurde die bakterielle Transduktion verwendet, um die Genkodierung für die Resistenz gegen das Antibiotikum Ampicillin vom W3110-Stamm von E.coli auf den J53-Stamm über das P1-Bakteriophagen (5) zu übertragen. Dieses Experiment bestand aus zwei Hauptschritten. Erstens die Herstellung von P1-Phagen, das das Ampicillin-Resistenzgen aus dem Spenderstamm enthält. Zweitens die Übertragung dieses Gens auf den Empfängerstamm durch Transduktion mit dem P1-Phagen (Abbildung 1). Nach der Durchführung konnte die erfolgreiche Übertragung des Ampicillinresistenzgens durch qPCR bestimmt werden (Abbildung 2). Wenn die Transduktion erfolgreich wäre, wäre der J53-Stamm von E. coli resistent gegen Ampicillin und das Gen, das diese Resistenz durch qPCR nachweisbar macht. Bei einem Erfolglosen würde es keinen Nachweis des Ampicillinresistenzgens geben und Ampicillin würde immer noch als wirksames Antibiotikum gegen den J53-Stamm wirken.
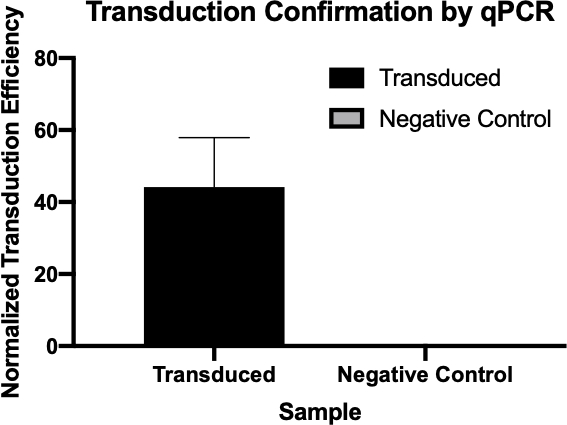
Abbildung 2: Bestätigung einer erfolgreichen Transduktion durch qPCR. Durch den Vergleich der C q-Werte, die für das Gen von Interesse aus der Transduktionsreaktion und der Negativkontrollreaktion nachgewiesen wurden, und die Normalisierung dieser Werte mit einem Housekeeping-Gen konnte man bestätigen, dass die bakterielle Transduktion erfolgreich war.
Procedure
1. Einrichtung
- Bevor Sie mit einer Arbeit mit Mikroben zu tun haben, sterilisieren Sie den Arbeitsbereich mit 70% Ethanol. Verwenden Sie immer die notwendige PSA (Labormantel und Handschuhe).
- Stellen Sie sicher, dass LB-Agarplatten mit 1x Ampicillin, handelsübliche P1-Phagenlysatlösung, Chloroform, 1 M Natriumcitrat, Glycerin und eine Schachtel vorsterilisierter Kunststoffpipettenspitzen und Zellstreuer in der Nähe sind.
- Bereiten Sie sterile LB durch Autoklavieren vor und verwenden Sie es, um drei 1 ml Aliquots von LB-Salzlösung herzustellen.
- mM MgCl2 (952.11-2.3803 'g), 5 mM CaCl2 (11.098 mg), 0.1-0.2% Glukose (100-200 g)
- mM MgSO4 (12.0366 mg), 5 mM CaCl2 (11.098 mg)
- mM Natriumcitrat (25.806 mg)
- Nach der Fertigstellung alle Oberflächen sowie Handschuhe mit 70% Ethanol sterilisieren und die Hände waschen.
2. Protokoll
- Spender Phage Lysate Vorbereitung
- Bereiten Sie eine 1 ml Kultur des Spenders W3110 Stamm E. coli in LB mit 1x Ampicillin über Nacht bei 37 °C mit Belüftung und Schütteln bei 220 U/min.
- Verdünnen Sie diese Nachtkultur 1:100 in 1 ml frischelb ergänzt mit 10-25 mM MgCl2, 5 mM CaCl2, und 0.1-0.2% Glukose.
- Wachsen Sie diese bakterielle Verdünnung bei 37 °C für 2 Stunden mit Belüftung und Schütteln bei 220 Rpm.
- Sobald diese Zellen eine frühe logarithmische Wachstumsphase erreicht haben (spürbares Wachstum und leichte Trübung), 40 l kommerziell erhältlichen P1-Phagen hinzufügen und bei 37 °C mit Belüftung und Schütteln bei 220 Umdrehungen pro Minute ablassen.
- Vor der Phagenzugabe sollte die gemessene optische Dichte bei 600 nm dieser Zellen etwa 0,4 (6) betragen.
- Überwachen Sie Zellen 1-3 Stunden lang, bis die Kultur lysiert ist.
- Lyse führt zu erhöhtem zellulären Schmutz sowie zu einer spürbaren Abnahme der Trübung (d.h. Zellen werden als lysiert betrachtet, sobald man in der Lage ist, durch die Kultur zu sehen).
- Fügen Sie dem Lysat mehrere Tropfen (50-100 l) Chloroform hinzu und mischen Sie sie durch Wirbel.
- Chloroform sterilisiert das Phagenlysat, indem es alle verbleibenden Spenderzellen tötet, nur noch Phagen hinterlässt und den Titer dieses Lysats erhöht.
- Zentrifuge lysiert bei 14.000 U/min für 2 Minuten, um Schmutz zu entfernen und Überstand in ein frisches Rohr zu übertragen.
- Ein paar Tropfen Chloroform dazugeben und bei 4 °C für nicht mehr als einen Tag lagern.
- Transduktion
- Bereiten Sie eine 1 ml Kultur des Empfängers J53-Stamm E. coli über Nacht in LB bei 37 °C mit Belüftung und Schütteln bei 220 U/min.
- Übertragen Sie 100 l Spenderphagenlysat (2,1) in ein 1,5 ml Mikrofugenrohr und inkubieren Sie mit einer Kappe, die bei 37 °C für 30 Minuten geöffnet ist.
- Diese Inkubation ermöglicht es, dass alle verbleibenden Chloroformen in der P1-Lysatlösung verdampfen, bevor sie den Empfängerzellen zugesetzt werden.
- Sanft Pellet-Empfänger-Stammzellen durch Zentrifugation bei 6.000 U/min für 5 Minuten.
- Setzen Sie diese Zellen in 300 l frischem LB mit 100 mM MgSO4 und 5 mM CaCl2wieder aus. (P1-Phagen erfordert Kalzium, um infektiös zu sein).
- Richten Sie zwei Reaktionen mit den Empfängerbakterienzellen und vorbereiteten Spenderphagenlysat ein: 1) Transduktionsreaktion, die 100-L-Empfänger-J53-Stamm E. coli und 100 L Spenderphagenlysat kombiniert, und 2) Negativkontrolle, die 100-L-Empfänger-J53-Stamm kombiniert E. coli und 100 l LB mit 100 mM MgSO4 und 5 mM CaCl2.
- Bei 37 °C 30 Minuten mit Schütteln bei 220 Rpm inkubieren.
- 1 ml LB und 200 l 1M Natriumcitrat (pH=5,5) hinzufügen und 1 Stunde bei 37 °C mit Schütteln bei 220 Rpm inkubieren.
- Citrat wird verwendet, um die Infektiosität von P1 durch Chelatmitat mit Kalzium zu reduzieren, die Lyse von Empfängerbakterien zu verhindern.
- Die Inkubation dieser Lösung ermöglicht die Expression des Ampicillin-Resistenzmarkers.
- Pelletzellen durch Zentrifugation bei 6.000 U/min für 5 Minuten.
- Zellpellets in 100 l LB mit 100 mM Natriumcitrat (pH 5,5) wieder aufsetzen. Vortex und Platte Gesamtlösung für beide Reaktionen auf zwei LB Agarplatten.
- LB-Platte sollte 1x Amp für die transduced Probe und keine Amp für die negative Kontrolle haben.
- P1-Phagenkontamination auf dieser Platte muss erneut gestreift werden, bevor Gefriervorräte vorbereitet werden können.
- Wenn phagen nicht entfernt wird, werden Kulturen, die aus diesen Kolonien wachsen, nur in Gegenwart eines Kalziumchelators wachsen.
- Wählen Sie etwa 3-4 Kolonien von beiden Platten und streifen Sie wieder auf zwei LB Agarplatten verteilt mit 100 l von 1 M Natriumcitrat (pH =5,5).
- LB-Platte sollte 1x Amp für die transduced Probe und keine Amp für die negative Kontrolle haben.
- Inkubieren Sie die Platten bei 37 °C über Nacht, damit Kolonien ohne Phagen wachsen können.
- Pflücken Sie Kolonien von beiden Platten und verwenden Sie sie, um Übernachtkulturen in 5 ml LB bei 37 °C mit Belüftung und Schütteln bei 220 Rpm anzubauen.
- Isolieren Sie DNA aus diesen Kulturen durch DNA Miniprep mit 4,5 ml des gesamten Kulturvolumens.
- Elute DNA mit 35 l nukleasefreiem Wasser.
- Messen Sie die resultierende Konzentration durch Nanodrop. Reine DNA erzeugt ein Absorptionsverhältnis (A260/280) von etwa 1,8.
- Verwenden Sie die restlichen 0,5 ml jeder Kultur, um 1 ml Glycerinvorräte zuzubereiten, indem Sie eine 1:1-Mischung aus 100% Glycerin und Bakterienkultur herstellen.
- Bakterielle Glycerinbestände bei -80 °C lagern.
3. Datenanalyse und Ergebnisse
- Bestätigung der Transduktion durch qPCR
- Bereiten Sie zwei qPCR-Master-Mischungen für sechs qPCR-Reaktionen vor, drei mit qPCR-Primern für das Ampicillin-Resistenzgen und die anderen drei qPCR-Primer für ein Housekeeping-Gen (14,5 l pro Reaktion): 12,5 l qPCR-Puffermischung + 1 L Vorwärtsprimer + 1 L-Reverse-Primer.
- In diesem Experiment haben wir SYBR Green Master Mix verwendet.
- Housekeeping-Genprimer wurden entwickelt, um ein DNA-Segment innerhalb des bakteriellen Gens zu verstärken, das für DNA-Gyrase B (7) kodiert.
- Kombinieren Sie für jede qPCR-Reaktion 100 g DNA aus jeder Reaktion (10,5 l) mit 14,5 l qPCR-Master-Mix.
- Mit Hilfe einer qPCR-Maschine und des in Tabelle 1 aufgeführten Thermocycling-Protokolls wurde die Amplifikation für die Ampicillinresistenz- und Haushaltsgene für alle sechs Reaktionen gemessen.
- Cq-Werte, die von qPCR generiert wurden, wurden verwendet, um die normalisierte Transduktionseffizienz des Ampicillinresistenzgens zu berechnen (Abbildung 3), was die erfolgreiche Transduktion des Ampicillinresistenzgens bestätigt.
- Der Cq-Wert oder Zyklusquantifizierungswert einer Stichprobe ist die früheste PCR-Zyklusnummer, bei der ein Signal, das den Hintergrundschwellenwert überschritten hat, erkannt wird. Niedrige Cq-Werte entsprechen mehr Zielsequenz, umgekehrt.
- Die normalisierte Transduktionseffizienz eines Gens innerhalb einer Probe kann anhand dieser Cq-Werte berechnet werden, indem der Wert des Housekeeping-Gens von dem des Zielgens subtrahiert wird, wodurch ein Cq-Wert generiert wird, der zur Berechnung normalisierter Transduktion verwendet werden kann. Effizienz um 2(-Cq).
- Bereiten Sie zwei qPCR-Master-Mischungen für sechs qPCR-Reaktionen vor, drei mit qPCR-Primern für das Ampicillin-Resistenzgen und die anderen drei qPCR-Primer für ein Housekeeping-Gen (14,5 l pro Reaktion): 12,5 l qPCR-Puffermischung + 1 L Vorwärtsprimer + 1 L-Reverse-Primer.
| temperatur | zeit | |
| Denaturierung | 94 °C | 2 Min. |
| 40 Zyklen: | ||
| Denaturierung | 94 °C | 15 Sek. |
| Glühen, Verlängerung und Fluoreszenz lesen | 60 °C oder 5 °C unter niedrigster Grundierung Tm | 1 Min. |
Tabelle 1: qPCR Thermocycling Protocol
Bakterien können sich schnell an eine sich schnell verändernde Umgebung anpassen, indem sie genetisches Material austauschen, und eine Möglichkeit, dies zu tun, ist die Transduktion, der Austausch von genetischem Material, das durch bakterielle Viren vermittelt wird. Ein Bakteriophagen, oft abgekürzt als Phagen, ist eine Art von Virus, das Bakterien infiziert, indem es zuerst an der Oberfläche des Wirts befestigt und dann seine DNA in die Bakterienzelle injiziert. Es degradiert dann die eigene DNA der Wirtszelle und repliziert ihr virales Genom, während die Maschinerie der Zelle entführt wird, um viele Kopien ihrer Proteine zu synthetisieren. Diese Phagenproteine bauen sich dann selbst zusammen und verpacken die Phagengenome zu mehreren Nachkommen. Aufgrund der geringen Genauigkeit des DNA-Verpackungsmechanismus verpackt der Phagen jedoch gelegentlich Fragmente bakterieller DNA in das Phagenkapsid. Nach der Induktion der Lyse des Wirts werden die Phagennachkommen freigesetzt und, sobald ein solcher Phagen eine andere Wirtszelle infiziert, überträgt es das DNA-Fragment seines vorherigen Wirts. Diese kann sich dann rekombinieren und dauerhaft in das Chromosom des neuen Wirts einfließen lassen, wodurch der Gentransfer zwischen den beiden Bakterien vermittelt wird.
Um die Phagentransduktion im Labor durchzuführen, bedarf es eines Spenderstamms, der ein Gen von Interesse enthält, einen Empfängerstamm, dem es fehlt, einen Phagen, der sowohl die Stämme infizieren kann, als auch eine Methode zur Auswahl der transduzierten Bakterien. In den meisten Fällen wird dies ein selektives festes Wachstumsmedium sein, das das Wachstum von transduzierten Bakterien unterstützt, aber das Wachstum von nicht-transduced hemmt. Zunächst wird der Spenderstamm, der das Gen von Interesse enthält, in einem flüssigen Wachstumsmedium kultiviert. Wenn sich alle Bakterien aktiv in der Logphase ihres Wachstums aufteilen, wird die Kultur mit dem Zielphagen geimpft. Nach drei bis vier Stunden Inkubation, wenn fast alle Bakterien die Phagenpartikel gelysiert und freigesetzt haben, wird das Spenderphagenlysat in eine frisch gewachsene Kultur des Empfänger-Bakterienstamms geimpft. Nach einer kurzen Inkubation von einer Stunde sollte die Kultur nun eine Mischung aus transduzierten und nicht transduzierten Bakterienzellen enthalten, die auf die transduzierten Zellen abgeschirmt wird, indem ein Bruchteil der Suspension auf ein geeignetes selektives, festes Wachstumsmedium verteilt wird. Nach einer weiteren Inkubation sollten die transduzierten Zellen wachsen und sich vermehren, um sichtbare Kolonien zu ergeben. Diese Kolonien können dann für eine weitere Analyse mit einer Vielzahl von Methoden ausgewählt werden, um eine erfolgreiche Transduktion weiter zu bestätigen, wie Kolonie-PCR, DNA-Sequenzierung oder quantitative PCR.
Bevor Sie mit dem Eingriff beginnen, ziehen Sie geeignete persönliche Schutzausrüstungen an, einschließlich laborierter Kleidung und Handschuhe. Als nächstes sterilisieren Sie den Arbeitsbereich mit 70% Ethanol und wischen Sie die Oberfläche ab.
Danach drei Ein-Milliliter-Aliquots LB-Salzlösung vorbereiten. Bereiten Sie nun eine Spenderstammkultur vor, indem Sie 100 Mikroliter E. coli zu einer 15 Milliliter konischen Durchstechflasche hinzufügen, die fünf Milliliter LB-Wachstumsmedium mit 500 Mikrogramm Ampicillin enthält. Dann wachsen die Kultur über Nacht bei 37 Grad Celsius mit Belüftung und Schütteln bei 220 Rpm. Wischen Sie am nächsten Tag die Bankspitze mit 70% Ethanol ab, bevor Sie die Kultur aus dem zitternden Inkubator entfernen. Als nächstes verdünnen Sie die Nachtkultur eins bis 100, indem Sie 10 Mikroliter Spenderstamm zu 990 Mikrolitern frischem LB hinzufügen, die mit Salzlösung ergänzt werden.
Lassen Sie die bakterielle Verdünnung bei 37 Grad Celsius für zwei Stunden mit Belüftung und Schütteln bei 220 Rpm wachsen. Sobald die Zellen die frühe Log-Phase erreicht haben, entfernen Sie die Kultur aus dem Inkubator, fügen Sie 40 Mikroliter P1-Phagen in die Kultur ein und brüten sie erneut an. Überwachen Sie die Zellen ein bis drei Stunden lang, bis die Kultur lysiert ist. Als nächstes 50 bis 100 Mikroliter Chloroform in das Lysat geben und durch Wirbeln mischen. Zentrifugieren Sie dann das Lysat, um Schmutz zu entfernen und den Überstand in ein frisches Rohr zu übertragen. Fügen Sie dem Überstand ein paar Tropfen Chloroform hinzu und lagern Sie es bei vier Grad Celsius für nicht mehr als einen Tag.
Um das Transduktionsverfahren zu beginnen, erhalten Sie eine Ein-Milliliter-Kultur der Empfänger-Sorte. Als nächstes übertragen Sie 100 Mikroliter Spenderphagen lysiert in ein 1,5 Milliliter Mikrozentrifugenrohr und inkubieren Sie es bei 37 Grad Celsius, wobei die Kappe 30 Minuten geöffnet ist, damit die verbleibende Chloroform verdampfen kann. Während der Spender phagen lysiert, pellet der Empfänger die Zellen durch sanfte Zentrifugation. Entsorgen Sie den Überstand und setzen Sie das Zellpellet in 300 Mikroliter frischem LB mit 100 Millimolaren Magnesiumsulfat und fünf Millimolar Calciumchlorid wieder auf.
Als nächstes richten Sie die Transduktionsreaktion ein, indem Sie 100 Mikroliter des Empfängerstamms und 100 Mikroliter des Spenderphagenlysats in einem Mikrozentrifugenrohr kombinieren. Richten Sie dann die Negativkontrolle ein, indem Sie 100 Mikroliter des Empfängerstamms und 100 Mikroliter des LB mit Magnesiumsulfat und Calciumchlorid kombinieren. Nach der Inkubation 200 Mikroliter eines molaren Natriumcitrats und einen Milliliter LB in beide Schläuche geben und durch sanftes Pipetieren nach oben und unten mischen. Nachdem die Schläuche eine Stunde lang inkubiert wurden, pellet enden die Zellen vorsichtig per Zentrifugation.
Nach der Zentrifugierung den Überstand entsorgen und die pelletierten Zellen in 100 MikroliterLB mit 100 Millimolar-Natriumcitrat wieder aufsetzen. Wirbel die Lösungen und Pipette die gesamte transduced Probe auf eine LB Agarplatte mit 1X Ampicillin. Schließlich pipette das gesamte Volumen der negativen Kontrollzellmischung auf eine LB-Agarplatte ohne Ampicillin. Nachdem Sie die Platten über Nacht bei 37 Grad Celsius inkubiert haben, verwenden Sie eine sterile Pipettenspitze, um drei bis vier Kolonien von der Transduktionsplatte zu pflücken und auf eine neue LB-Agarplatte mit 1X Ampicillin und 100 Mikrolitern eines molaren Natriumcitrats zu streifen. Wiederholen Sie diese Beschichtungsmethode für die Negativkontrolle auf einer anderen LB-Agarplatte, die nur 100 Mikroliter eines molaren Natriumcitrats enthält. Dann bebrüten die Teller bei 37 Grad Celsius über Nacht, damit Kolonien ohne Phagen wachsen können.
Wischen Sie am nächsten Tag die Bankspitze mit 70% Ethanol ab, bevor Sie Ihre Platten aus dem Inkubator entfernen. Mit einer sterilen Pipettenspitze drei Kolonien von der Transduktionsplatte pflücken und jeweils in ein separates Rohr mit fünf MilliliterN LB-Medien geben. Wählen Sie dann drei Kolonien aus der negativen Kontrollplatte aus und fügen Sie sie einer anderen Röhre hinzu, die fünf Milliliter LB-Medien enthält. Wachsen Sie die Kulturen über Nacht bei 37 Grad Celsius mit Belüftung und Schütteln bei 220 Rpm. Nach der Sterilisation der Tischplatte, wie zuvor gezeigt, verwenden Sie ein DNA-Miniprep-Kit, um DNA von 4,5 Milliliter jeder Kultur gemäß den Anweisungen des Herstellers zu isolieren. Dann die DNA mit 35 Mikroliter nukleasefreiem Wasser zu deuten und die resultierende Konzentration per Laborspektrophotometer zu messen. Schließlich bereiten Sie Glycerin-Bestände vor, indem Sie die verbleibenden 0,5 Milliliter beider bakteriellen Lösungen auf 0,5 Milliliter 100% Glycerin aufstocken.
Um die Transduktion zu bestätigen, bereiten Sie zunächst zwei qPCR-Mastermischungen für 24 qPCR-Reaktionen vor. Für den ersten Master-Mix fügen Sie 150 Mikroliter qPCR-Puffermischung in ein Mikrozentrifugenrohr und 12 Mikroliter einer Vorwärts- und Reverse-Primer hinzu, die entwickelt wurden, um das Ampicillin-Resistenzgen zu verstärken. Als nächstes bereiten Sie einen zweiten qPCR-Master-Mix vor, indem Sie 150 Mikroliter qPCR-Master-Mix zu einem Mikrozentrifugenrohr hinzufügen und dann jeweils 12 Mikroliter einer Vorwärtsgrundierung und einer Reverse Primer hinzufügen, die entwickelt wurden, um ein Housekeeping-Gen zu verstärken.
Kombinieren Sie für jede qPCR-Reaktion 100 Mikrogramm experimentelle DNA aus jeder Reaktion mit 14,5 MikroliterqPCR-Master-Mix. Bereiten Sie nun die verbleibenden Reaktionen vor, wie zuvor gezeigt. Übertragen Sie die Reaktionen auf einen thermocycler vorgewärmt auf 94 Grad Celsius und dann das Programm initiieren. Verwenden Sie schließlich die von qPCR erzeugten Zyklusquantifizierungswerte oder Cq, um die normalisierte Transduktionseffizienz des Ampicillinresistenzgens zu berechnen.
Die Zyklusquantitation oder Cq-Werte für die Gene von Interesse wurden für jede der negativen Kontrollen und transduzierten Proben tabellarisch dargestellt. Niedrige Cq-Werte, in der Regel unter 29 Zyklen, wie die transduced Samples in diesem Beispiel zeigen hohe Mengen der Zielsequenz.
Ein Housekeeping-Gen, das auch hier tabellarisch dargestellt wird, wird als Ladekontrolle verwendet, um die Menge der DNA in jeder Reaktion zu normalisieren und als positive Kontrolle, um sicherzustellen, dass die qPCR funktioniert. Sofern die gleichen Mengen des Haushaltsgens geladen werden, wird es in jeder Probe relativ gleich schnell gefunden.
Um den Delta-Cq-Wert für jede Probe zu berechnen, subtrahieren Sie den Cq-Wert des Housekeeping-Gens für jede Probe vom Cq-Wert des entsprechenden Zielgens. Der Delta-Cq der ersten Negativkontrolle ist beispielsweise 13.54. Verwenden Sie dann diesen Wert, um die normalisierte Transduktionseffizienz jeder Probe mit der hier gezeigten Formel zu berechnen. Schließlich kann die durchschnittliche normalisierte Transduktionseffizienz für jede Stichprobengruppe berechnet werden.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
Die Übertragung von Genen zu und von Bakterien durch Bakteriophagen, während ein natürlicher Prozess, hat sich als äußerst nützlich für eine Vielzahl von Forschungszwecken. Während andere Methoden des Gentransfers wie Transformation und Konjugation möglich sind, verwendet transduktionsweise ausschließlich Bakteriophagen; nicht nur die Genintegration in das Wirtsgenom, sondern auch die Genabgabe an mehrere Bakterien, die nicht anfällig für andere Methoden sind. Dieser Prozess ist zwar besonders im Labor nützlich, wurde aber auch im neu entstehenden Bereich der Gentherapie eingesetzt, genauer gesagt in der alternativen Gentherapie, einer therapeutischen Strategie, die Bakterien nutzt, um Therapeutika für das Zielgewebe zu liefern. viele davon sind nicht anfällig für andere Verabreichungsmethoden und haben viel klinische Relevanz (8,9).
Subscription Required. Please recommend JoVE to your librarian.
References
- Lederberg J, Lederberg E.M., Zinder, N.D., et al. Recombination analysis of bacterial heredity. Cold Spring Harbor symposia Quantitative Biol. 1951;16:413-43.
- Duckworth DH. "Who Discovered Bacteriophage?". Bacteriology Reviews. 1976;40:793-802.
- Yap ML, Rossman, M.G. Structure and Function of Bacteriophage T4. Future Microbiol. 2014;9:1319-27.
- Sulakvelidze A, Alavidze, Z., Morris, J. G. Bacteriophage Therapy Antimicrobial Agents and Chemotherapy 2001;45(3):649-59.
- Moore S. Sauer:P1vir phage transduction 2010 [Available from: https://openwetware.org/wiki/Sauer:P1vir_phage_transduction].
- Kobayashi A, et al. Growth Phase-Dependent Expression of Drug Exporters in
- Escherichia coli and Its Contribution to Drug Tolerance. Journal of Bacteriology. 2006;188(16):5693-703.
- Rocha D, Santos, CS, Pacheco LG. Bacterial reference genes for gene expression studies by RT-qPCR: survey and analysis. Antonie Van Leeuwenhoek. 2015;108:685-93.
- Pálffy R. et al. Bacteria in gene therapy: bactofection versus alternative gene therapy. Gene Ther. 2006 13:101-5.
- O'Neill JM, et al. Intestinal delivery of non-viral gene therapeutics: physiological barriers and preclinical models. Drug Discovery Today. 2011;16:203-2018.




