Overview
Fonte: Alexander S. Gold1, Tonya M. Colpitts1
1 Departamento de Microbiologia, Escola de Medicina da Universidade de Boston, Laboratórios Nacionais de Doenças de Infecções Emergentes, Boston, MA
Transdução é uma forma de troca genética entre bactérias que utilizam bacteriófagos, ou phages, uma classe de vírus que infecta exclusivamente organismos procarióticos. Esta forma de transferência de DNA, de uma bactéria para outra por meio de um phage, foi descoberta em 1951 por Norton Zinder e Joshua Ledererg, que chamou o processo de "transdução" (1). Bacteriphages foram descobertos pela primeira vez em 1915 pelo bacteriologista britânico Frederick Twort, então descoberto independentemente novamente em 1917 pelo microbiologista franco-canadense Felix d'Herelle (2). Desde então, a estrutura e a função desses phages têm sido amplamente caracterizadas (3), dividindo essas pragas em duas classes. A primeira dessas classes são as fábulas líticas que, após a infecção, se multiplicam dentro da bactéria hospedeira, interrompendo o metabolismo bacteriano, lisendo a célula e liberando phage progênero (4). Como resultado dessa atividade antibacteriana e da crescente prevalência de bactérias resistentes a antibióticos, esses frascos líticos têm se mostrado recentemente úteis como um tratamento substituto para antibióticos. A segunda dessas classes são as fálvias lisogênicas que podem se multiplicar dentro do hospedeiro através do ciclo lítico ou entrar em um estado quiescente no qual seu genoma é integrado ao hospedeiro (Figura 1), um processo conhecido como lisogenia, com a capacidade de a produção de phage ser induzida em múltiplas gerações posteriores (4).
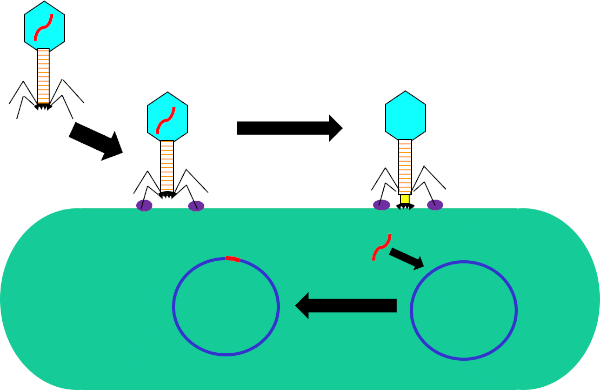
Figura 1: Infecção de célula hospedeira por bacteriófago. Adsorção pela phage para a parede celular bacteriana através de interações entre as fibras traseiras e receptor (roxo). Uma vez na superfície celular, o phage é irreversivelmente ligado à célula bacteriana usando a placa base (preta) que é movida para a parede celular pela baia contraltil (amarelo). O genoma phage (vermelho) então entra na célula e se integra ao genoma das células hospedeiras.
Embora a transdução bacteriana seja um processo natural, usando tecnologia moderna foi manipulado para a transferência de genes para bactérias no ambiente laboratorial. Ao inserir genes de interesse no genoma de uma praga lisogênica, como a praga, é capaz de transferir esses genes para os genomas das bactérias e, consequentemente, expressá-los dentro dessas células. Enquanto outros métodos de transferência genética, como a transformação, usam um plasmídeo para transferência e expressão genética, a inserção do genoma phage na da bactéria receptora não só tem o potencial de conferir novos traços a esta bactéria, mas também permite que mutações que ocorram naturalmente e outros fatores do ambiente celular alterem a função do gene transferido.
Em comparação com outros métodos de transferência de genes horizontais, como a conjugação, a transdução é bastante flexível nos critérios necessários para as células doadoras e receptoras. Qualquer elemento genético que possa caber dentro do genoma da praga que está sendo usada pode ser transferido de qualquer cepa de bactérias doadoras para qualquer cepa de bactérias receptoras, desde que ambas sejam permissivas ao phage, exigindo a expressão de receptores phage necessários nas superfícies celulares. Uma vez que este gene é removido do genoma do doador e embalado para o phage, ele pode ser transferido para o receptor. Após a transdução, é necessário selecionar para o receptor bactérias que contenham o gene de interesse devem ser selecionados. Isso poderia ser feito pelo uso de um marcador genético, como uma marca BANDEIRA ou polyhistidina-tag, para marcar o gene de interesse, ou a função intrínseca do gene, no caso de genes que codificam para resistência a antibióticos. Além disso, o PCR poderia ser usado para confirmar ainda mais a transdução bem sucedida. Usando primers para uma região dentro do gene de interesse e comparando sinal a um controle positivo, bactérias que têm o gene de interesse, e um controle negativo, bactérias que passaram pelos mesmos passos da reação de transdução sem phage. Embora a transdução bacteriana seja uma ferramenta útil na biologia molecular, ela tem e continua a desempenhar um papel importante na evolução das bactérias, particularmente no que diz respeito ao recente aumento da resistência a antibióticos.
Neste experimento, a transdução bacteriana foi utilizada para transferir a codificação genética para resistência à ampicilina antibiótico da cepa W3110 de E.coli para a cepa J53 através do bacteriófago P1 (5). Este experimento consistia em dois passos principais. Primeiro, a preparação do phage P1 contendo o gene de resistência à ampicilina da cepa do doador. Em segundo lugar, a transferência deste gene para a cepa receptora por transdução com a phage P1 (Figura 1). Uma vez realizada, a transferência bem sucedida do gene de resistência à ampicilina poderia ser determinada por qPCR (Figura 2). Se a transdução fosse bem sucedida, a cepa J53 de E. coli seria resistente à ampicilina, e o gene conferenciando essa resistência detectável por qPCR. Se não tivesse sucesso, não haveria detecção do gene de resistência à ampicilina e a ampicillina ainda funcionaria como um antibiótico eficaz contra a cepa J53.
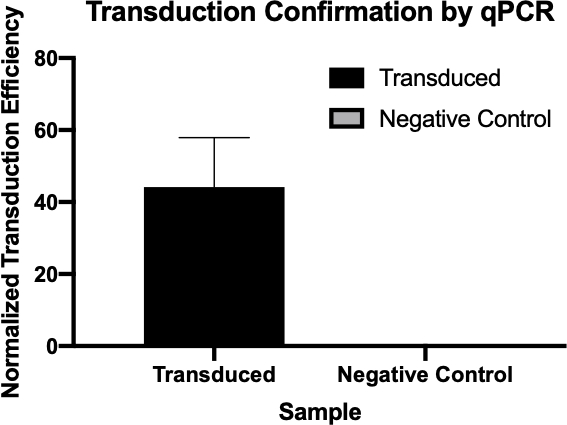
Figura 2: A confirmação da transdução bem sucedida por qPCR. Comparando os valores Cq detectados para o gene de interesse da reação de transdução e a reação de controle negativo, e normalizando esses valores em relação a um gene de limpeza, foi possível confirmar que a transdução bacteriana foi bem sucedida.
Procedure
1. Configuração
- Antes de iniciar qualquer trabalho envolvendo micróbios, esterilize o espaço de trabalho usando 70% de etanol. Use sempre os EPI necessários (jaleco e luvas).
- Certifique-se de que as placas de ágar LB com 1x ampicillina, solução de lise de phage P1 disponíveis comercialmente, clorofórmio, citrato de sódio de 1 M, glicerol e uma caixa de pontas de pipeta plástica pré-esterilizada e espalhadores de células estão próximas.
- Prepare LB estéril autoclaving e use-o para fazer três alíquotas de 1 mL de solução de sal LB.
- mM MgCl2 (952,11-2,3803 μg), 5 mM CaCl2 (11.098 mgs), 0,1-0,2% de glicose (100-200 μg)
- mM MgSO4 (12,0366 mgs), 5 mM CaCl2 (11.098 mgs)
- citrato de sódio mM (25.806 mgs)
- Uma vez terminado, esterilize todas as superfícies, bem como luvas com 70% de etanol e lave as mãos.
2. Protocolo
- Preparação de doadores phage lysate
- Prepare uma cultura de 1 mL do doador W3110 espeto E. coli em LB com 1x ampicilina cultivada durante a noite a 37 °C com aeração e tremendo a 220 rpm.
- Diluir esta cultura durante a noite 1:100 em 1 mL de LB fresco suplementado com 10-25 mM MgCl2, 5 mM CaCl2e 0,1-0,2% de glicose.
- Cresça esta diluição bacteriana a 37 °C durante 2 horas com aeração e tremendo a 220 rpm.
- Uma vez que essas células tenham atingido a fase de crescimento logarítmico precoce (crescimento perceptível e leve turbidez), adicione 40 μL de phage P1 comercialmente disponível e deixe a 37 °C com aeração e tremendo a 220 rpm.
- Antes da adição de phage, a densidade óptica medida a 600 nm dessas células deve ser de aproximadamente 0,4 (6).
- Monitore as células por 1-3 horas até que a cultura tenha sido lise.
- A lise resultará em aumento de detritos celulares, bem como uma notável diminuição da turbidez (ou seja, as células serão consideradas lísedas uma vez que se possa ver através da cultura).
- Adicione várias gotas (50-100 μL) de clorofórmio ao lise e misture por vórtice.
- O clorofórmio esteriliza o flesato de praga matando todas as células doadoras restantes, deixando apenas a praga e aumentando o título deste lysate.
- Centrifugar lysate a 14.000 rpm por 2 minutos para remover detritos e transferir supernante para um tubo fresco.
- Adicione algumas gotas de clorofórmio e armazene a 4 °C por não mais do que um dia.
- Transdução
- Prepare uma cultura de 1 mL da cepa J53 receptorA E. coli cultivada durante a noite em LB a 37 °C com aeração e tremendo a 220 rpm.
- Transfira 100 μL de flesato de doador (2.1) em um tubo de microfuça de 1,5 mL e incubar com tampa aberta a 37 °C por 30 minutos.
- Esta incubação permite que qualquer clorofórmio restante na solução de lise P1 evapore antes de ser adicionado às células receptoras.
- Células de cepa suavemente receptoras de pelotização por centrifugação a 6.000 rpm por 5 minutos.
- Resuspense essas células em 300 μL de LB fresco contendo 100 mM MgSO4 e 5 mM CaCl2. (A praga P1 requer que o cálcio seja infeccioso).
- Configure duas reações usando as células de bactérias receptoras e o teor de phage de doadores preparados: 1) reação de transdução combinando 100 μL receptor J53 cepa E. coli e 100 μL doador phage lysate, e 2) controle negativo combinando 100 μL receptor J53 cepA E. coli e 100 μL de LB contendo 100 mM MgSO4 e 5 mM CaCl2.
- Incubar a 37 °C por 30 minutos com agitação a 220 rpm.
- Adicione 1 mL LB e 200 μL 1M citrato de sódio (pH=5,5) e incubar por 1 hora a 37 °C com agitação a 220 rpm.
- O citrato é usado para reduzir a infectividade de P1 por quechelating com cálcio, prevenindo a lise das bactérias receptoras.
- A incubação desta solução permite a expressão do marcador de resistência à ampicilina.
- Células de pelotas por centrifugação a 6.000 rpm por 5 minutos.
- Pelotas de células resuspendas em 100 μL de LB com citrato de sódio de 100 mM (pH 5.5). Vórtice e placa inteira solução para ambas as reações em duas placas de ágar LB.
- A placa LB deve ter 1x Amp para a amostra transduzida e nenhum Amp para o controle negativo.
- A contaminação por phage P1 nesta placa requer re-streaking antes que os estoques do congelador possam ser preparados.
- Se a praga não for removida, as culturas cultivadas a partir dessas colônias não crescerão a menos que na presença de um quequeador de cálcio.
- Escolha cerca de 3-4 colônias de ambas as placas e listra novamente em duas placas de ágar LB espalhadas com 100 μL de 1 M de citrato de sódio (pH=5,5).
- A placa LB deve ter 1x Amp para a amostra transduzida e nenhum Amp para o controle negativo.
- Incubar as placas a 37 °C durante a noite para permitir que colônias livres de phage cresçam.
- Escolha colônias de ambas as placas e use-as para cultivar culturas durante a noite em 5 mL de LB a 37 °C com aeração e tremendo a 220 rpm.
- Isole o DNA dessas culturas por miniprep de DNA usando 4,5 mL do volume total da cultura.
- DNA eluto usando 35 μL de água sem nuclease.
- Meça a concentração resultante por Nanodrop. O DNA puro gerará uma razão de absorção (A260/280) de aproximadamente 1,8.
- Use os 0,5 mL restantes de cada cultura para preparar 1 mL de glicerol, fazendo uma mistura de 1:1 de 100% glicerol e cultura bacteriana.
- Armazene estoques de glicerol bacteriano a -80 °C.
3. Análise e Resultados de Dados
- Confirmação da transdução por qPCR
- Prepare duas misturas mestres qPCR para seis reações qPCR, três usando primers qPCR para o gene de resistência à ampicilina, e as outras três usando primers qPCR para um gene de limpeza (14,5 μL por reação): 12,5 μL qPCR buffer mix + 1 μL primer para a frente + 1 μL primer reverso.
- Neste experimento, usamos a mistura mestre SYBR Green.
- Os primers genéticos de limpeza foram projetados para amplificar um segmento de DNA dentro da codificação genética bacteriana para o DNA gyrase B (7).
- Para cada reação qPCR, combine 100 μg de DNA de cada reação (10,5 μL) com 14,5 μL de mix mestre qPCR.
- Utilizando uma máquina qPCR e o protocolo termociclizador listado na Tabela 1, a amplificação foi medida para a resistência à ampicilina e genes de limpeza para todas as seis reações.
- Os valores de CQ gerados pelo qPCR foram utilizados para calcular a eficiência de transdução normalizada do gene de resistência à ampicilina (Figura 3), confirmando a transdução bem sucedida do gene de resistência à ampicilina.
- O valor do Cq, ou valor de quantificação do ciclo, de uma amostra é o número de ciclo PCR mais antigo no qual um sinal excedeu o limiar de fundo é detectado. Os valores baixos do Cq correspondem a mais sequência de alvos, vice-versa.
- A eficiência de transdução normalizada de um gene dentro de uma amostra pode ser calculada usando esses valores de Cq subtraindo o valor do gene de limpeza do gene alvo, gerando um valor ΔCq, que pode ser usado para calcular a eficiência de transdução normalizada em 2(-ΔCq).
- Prepare duas misturas mestres qPCR para seis reações qPCR, três usando primers qPCR para o gene de resistência à ampicilina, e as outras três usando primers qPCR para um gene de limpeza (14,5 μL por reação): 12,5 μL qPCR buffer mix + 1 μL primer para a frente + 1 μL primer reverso.
| Temperatura | Hora | |
| Desnaturação | 94 °C | 2 min. |
| 40 ciclos: | ||
| Desnaturação | 94 °C | 15 seg |
| Annealing, extensão e fluorescência ler | 60 °C ou 5 °C abaixo do primer mais baixo Tm | 1 min. |
Tabela 1: protocolo de termociclismo qPCR
As bactérias podem se adaptar rapidamente a um ambiente em rápida mudança, trocando material genético e uma maneira de fazer isso é através da transdução, a troca de material genético mediado por vírus bacterianos. Um bacteriófago, muitas vezes abreviado para phage, é um tipo de vírus que infecta bactérias primeiro anexando-se à superfície do hospedeiro e, em seguida, injetando seu DNA na célula bacteriana. Em seguida, degrada o próprio DNA da célula hospedeira e replica seu genoma viral, enquanto sequestra as máquinas da célula para sintetizar muitas cópias de suas proteínas. Essas proteínas de phage então se auto-montam e embalam os genomas phage para formar múltiplas progêneres. No entanto, devido à baixa fidelidade do mecanismo de embalagem de DNA, ocasionalmente, o phage embala fragmentos de DNA bacteriano no capsídeo phage. Depois de induzir a lise do hospedeiro, a prole phage é liberada e, uma vez que tal phage infecta outra célula hospedeira, transfere o fragmento de DNA de seu hospedeiro anterior. Isso pode então recombinar e se incorporar permanentemente ao cromossomo do novo hospedeiro, mediando assim a transferência genética entre as duas bactérias.
Para realizar a transdução de phage em laboratório é necessário uma cepa de doador que contenha um gene de interesse, uma cepa receptora que não o possui, um phage que pode infectar ambas as cepas, e um método para selecionar as bactérias transduzidas. Na maioria dos casos, esta será uma mídia de crescimento sólido seletiva que apoia o crescimento de bactérias transduzidas, mas inibe o crescimento de não transdutos. Para começar, a cepa de doadores que contém o gene de interesse é cultivada em um meio de crescimento líquido. Quando todas as bactérias estão se dividindo ativamente na fase de registro de seu crescimento, a cultura é inoculada com o phage alvo. Após três a quatro horas de incubação, quando quase todas as bactérias têm lise e liberado as partículas de phage, o lísato de phage do doador é inoculado em uma cultura recém-cultivada da cepa bacteriana receptora. Após uma breve incubação de uma hora, a cultura deve agora conter uma mistura de células bacterianas transduzidas e não transduzidas e isso é rastreado para as células transduzidas espalhando uma fração da suspensão em uma mídia de crescimento sólido seletiva apropriada. Após uma maior incubação, as células transduzidas devem crescer e se multiplicar para produzir colônias visíveis. Essas colônias podem então ser selecionadas para análise suplementar usando uma variedade de métodos para confirmar ainda mais a transdução bem sucedida, como pcr colônia, sequenciamento de DNA ou PCR quantitativo.
Antes de iniciar o procedimento, coloque qualquer equipamento de proteção individual adequado, incluindo um jaleco e luvas. Em seguida, esterilize o espaço de trabalho com 70% de etanol e limpe a superfície.
Depois disso, prepare três alíquotas de um mililitro da solução de sal LB. Agora, prepare uma cultura de variedade de doadores adicionando 100 microliters de E. coli a um frasco cônico de 15 mililitros contendo cinco mililitros de meio de crescimento LB com 500 microgramas de ampicillina. Então, cresça a cultura durante a noite a 37 graus Celsius com aeração e tremendo a 220 rpm. No dia seguinte, limpe a bancada com 70% de etanol antes de remover a cultura da incubadora de agitação. Em seguida, diluir a cultura da noite para o dia de 100 adicionando 10 microliters de cepa de doadores a 990 microliters de LB fresco suplementado com solução de sal.
Permita que a diluição bacteriana cresça a 37 graus Celsius por duas horas com aeração e tremendo a 220 rpm. Uma vez que as células tenham atingido a fase de tronco precoce, remova a cultura da incubadora, adicione 40 microliters de P1 phage à cultura e incuba novamente. Continue monitorando as células por uma a três horas até que a cultura tenha sido lise. Em seguida, adicione 50 a 100 microlitadores de clorofórmio ao lise e misture por vórtice. Em seguida, centrifugar o lise para remover detritos e transferir o sobrenatante para um tubo fresco. Adicione algumas gotas de clorofórmio ao supernascente e armazene-o a quatro graus Celsius por não mais do que um dia.
Para iniciar o procedimento de transdução, obtenha uma cultura de um mililitro de cepa receptora. Em seguida, transfira 100 microliters de phage lysate doador em um tubo de microcentrífuga de 1,5 mililitro e incuba-lo a 37 graus Celsius com a tampa aberta por 30 minutos para permitir que qualquer clorofórmio restante evaporar. Enquanto o doador incuba o lysato de phage, pelota as células de cepa receptoras através de centrifugação suave. Descarte o supernatante e resuspenja a pelota celular em 300 microliters de LB fresco contendo 100 milimônios de sulfato de magnésio e cinco cloreto de cálcio milialar.
Em seguida, configure a reação de transdução combinando 100 microlitres da cepa receptora e 100 microliters do lysato de phage do doador em um tubo de microcentrifuuagem. Em seguida, configure o controle negativo combinando 100 microliters da cepa receptora e 100 microliters do LB com sulfato de magnésio e cloreto de cálcio. Após a incubação, adicione 200 microlitres de um citrato de sódio molar e um mililitro de LB em ambos os tubos, e misture suavemente tubos para cima e para baixo. Então, depois que os tubos foram incubados por uma hora, pelota suavemente as células através de centrifugação.
Após a centrifugação, descarte o supernascer e resuspenque as células pelotas em 100 microlitrais de LB com citrato de sódio de 100 milimônios. Vórtice as soluções e pipeta toda a amostra transduzida em uma placa de ágar LB com ampicillina 1X. Finalmente, pipeta todo o volume da mistura de célula de controle negativo em uma placa de ágar LB sem ampicillina. Depois de incubar as placas durante a noite a 37 graus Celsius, use uma ponta de pipeta estéril para colher de três a quatro colônias da placa de transdução e estrá-las em uma nova placa de ágar LB contendo 1X ampicillin e 100 microlitres de uma citrato de sódio molar. Repita este método de chapeamento para o controle negativo em outra placa de ágar LB contendo apenas 100 microlitres de uma citrato de sódio molar. Em seguida, incubar as placas a 37 graus Celsius durante a noite para permitir que colônias livres de phage cresçam.
No dia seguinte, limpe a bancada com 70% de etanol antes de retirar as placas da incubadora. Usando uma ponta de pipeta estéril, escolha três colônias da placa de transdução e adicione-as cada uma a um tubo separado contendo cinco mililitros de mídia LB. Em seguida, selecione três colônias da placa de controle negativo e adicione-as a outro tubo contendo cinco mililitros de mídia LB. Cresça as culturas durante a noite a 37 graus Celsius com aeração e tremendo a 220 rpm. Depois de esterilizar a parte superior do banco como demonstrado anteriormente, use um kit de miniprep de DNA para isolar o DNA de 4,5 mililitros de cada cultura de acordo com as instruções do fabricante. Em seguida, elute o DNA com 35 microliters de água sem nuclease e meça a concentração resultante por espectógrafo de laboratório. Por fim, prepare os estoques de glicerol adicionando os 0,5 mililitros restantes de ambas as soluções bacterianas a 0,5 mililitros de 100% de glicerol.
Para confirmar a transdução, primeiro prepare duas misturas mestres qPCR para reações de 24 qPCR. Para a primeira mistura mestre, adicione 150 microliters de mistura de tampão qPCR a um tubo de microcentrifuuge e 12 microliters cada um de um primer para frente e reverso projetado para amplificar o gene de resistência à ampicilina. Em seguida, prepare uma segunda mistura mestre qPCR adicionando 150 microliters de mix mestre qPCR a um tubo de microcentrifuuge e, em seguida, adicionando 12 microliters cada um de um primer dianteiro e primer reverso projetado para amplificar um gene de limpeza.
Para cada reação qPCR, combine 100 microgramas de DNA experimental de cada reação com 14,5 microliters de mix mestre qPCR. Agora, prepare as reações restantes como demonstrado anteriormente. Transfira as reações para um termociclador pré-aquecido a 94 graus Celsius e, em seguida, inicie o programa. Por fim, utilize a quantificação do ciclo, ou Cq, valores gerados pelo qPCR para calcular a eficiência de transdução normalizada do gene de resistência à ampicilina.
A quantitação cicloviária, ou Cq, valores para os genes de interesse foram tabulados para cada um dos controles negativos e amostras transduzidas. Valores baixos de Cq, tipicamente abaixo de 29 ciclos, como as amostras transduzidas neste exemplo indicam altas quantidades da sequência de destino.
Um gene de limpeza, também tabulado aqui, é usado como um controle de carga para normalizar a quantidade de DNA em cada reação e como um controle positivo para garantir que o qPCR esteja funcionando. Desde que as mesmas quantidades do gene de limpeza sejam carregadas, ele é encontrado a uma taxa relativamente a mesma em cada amostra.
Em seguida, para calcular o valor delta Cq para cada amostra, subtraia o valor de Cq do gene de limpeza para cada amostra do valor Cq de seu gene alvo correspondente. Por exemplo, o delta Cq do primeiro controle negativo é de 13,54. Em seguida, use esse valor para calcular a eficiência de transdução normalizada de cada amostra usando a fórmula mostrada aqui. Finalmente, pode ser calculada a eficiência média normalizada de transdução para cada grupo amostral.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
A transferência de genes para e de bactérias por bacteriófago, enquanto um processo natural, tem se mostrado extremamente útil para uma infinidade de propósitos de pesquisa. Enquanto outros métodos de transferência genética, como transformação e conjugação, a transdução usa exclusivamente bacteriófagos; não apenas permitindo a integração genética no genoma hospedeiro, mas também para a entrega de genes a múltiplas bactérias que não são suscetíveis a outros métodos. Esse processo, embora especialmente útil em laboratório, também tem sido utilizado no campo recentemente emergente da terapia genética, mais especificamente na terapia genética alternativa, uma estratégia terapêutica que utiliza bactérias para fornecer terapêuticas a tecidos-alvo, muitos dos quais não são suscetíveis a outros métodos de parto e têm muita relevância clínica (8,9).
Subscription Required. Please recommend JoVE to your librarian.
References
- Lederberg J, Lederberg E.M., Zinder, N.D., et al. Recombination analysis of bacterial heredity. Cold Spring Harbor symposia Quantitative Biol. 1951;16:413-43.
- Duckworth DH. "Who Discovered Bacteriophage?". Bacteriology Reviews. 1976;40:793-802.
- Yap ML, Rossman, M.G. Structure and Function of Bacteriophage T4. Future Microbiol. 2014;9:1319-27.
- Sulakvelidze A, Alavidze, Z., Morris, J. G. Bacteriophage Therapy Antimicrobial Agents and Chemotherapy 2001;45(3):649-59.
- Moore S. Sauer:P1vir phage transduction 2010 [Available from: https://openwetware.org/wiki/Sauer:P1vir_phage_transduction].
- Kobayashi A, et al. Growth Phase-Dependent Expression of Drug Exporters in
- Escherichia coli and Its Contribution to Drug Tolerance. Journal of Bacteriology. 2006;188(16):5693-703.
- Rocha D, Santos, CS, Pacheco LG. Bacterial reference genes for gene expression studies by RT-qPCR: survey and analysis. Antonie Van Leeuwenhoek. 2015;108:685-93.
- Pálffy R. et al. Bacteria in gene therapy: bactofection versus alternative gene therapy. Gene Ther. 2006 13:101-5.
- O'Neill JM, et al. Intestinal delivery of non-viral gene therapeutics: physiological barriers and preclinical models. Drug Discovery Today. 2011;16:203-2018.




