Summary
Kombinasjonen av kromatin immunoprecipitation og ultra-high-throughput sekvensering (ChIP-seq) kan identifisere og kartlegge protein-DNA vekselvirkninger i et gitt vev eller cellelinje. Skissert er hvordan å generere en høy kvalitet ChIP mal for etterfølgende sekvensering, ved hjelp av erfaring med transkripsjonsfaktor TCF7L2 som et eksempel.
Abstract
ChIP-sekvensering (chip-seq) metoder direkte gir hel-genom dekning, hvor kombinere kromatin immunoutfelling (chip) og massivt parallelle sekvensering kan benyttes til å identifisere den repertoaret av pattedyr-DNA-sekvenser som er bundet av transkripsjonsfaktorer in vivo. "Neste generasjon" genom sekvensering teknologi gir 1-2 størrelsesordener økning i mengden av sekvensen som kan være kostnadseffektivt generert over eldre teknologier og dermed gir for ChIP-seq metoder for å direkte gi hel-genom dekning for effektiv profilering av pattedyr protein-DNA vekselvirkninger.
For vellykkede ChIP-seq nærmer seg, må man generere høy kvalitet ChIP DNA mal for å oppnå de beste sekvensering utfall. Beskrivelsen er basert rundt erfaring med proteinproduktet av genet mest sterkt implisert i patogenesen av type 2 diabetes, nemlig transkripsjonsfaktor transkripsjonsfaktor 7-lignende 2 (TCF7L2). Denne faktor har også blitt innblandet i ulike kreftformer.
Skissert er hvordan å generere høy kvalitet ChIP DNA mal avledet fra kolorektal karsinom cellelinje, HCT116, for å bygge en høy oppløsning kartet gjennom sekvensering finne de genene som er bundet av TCF7L2, noe som gir ytterligere innsikt i sin nøkkelrolle i patogenesen av komplekse egenskaper.
Introduction
I mange år har det vært et udekket behov for å identifisere sett av gener bundet og regulert av et gitt protein genom bred, særlig de i transkripsjonsfaktor klassen.
Odom et al. En brukt kromatin immunoprecipitation (chip) kombinert med promoter mikromatriser å systematisk identifisere genene okkupert av pre-spesifiserte transkripsjonelle regulatorer i human lever og bukspyttkjertelen holmer. Deretter utviklet Johnson et al. 2 en storstilt kromatin immunoprecipitation analysen basert på direkte ultra high-throughput DNA-sekvensering (ChIP-seq) for å grundig kartlegge protein-DNA interaksjoner på tvers av hele pattedyr genomer. Som en test, kartlagt de in vivo binding av nervecellen-restriktiv lyddemper faktor (NRSF) til 1946 steder i det menneskelige genom. Dataene vises skarp oppløsning av forpliktende stilling (+ 50 basepar), som tilrettelagt både isolation av motiver og identifisering av NRSF-bindende motiver. Disse ChIP-seq data også hadde høy sensitivitet og spesifisitet og statistisk sikkerhet (P <10 -4), egenskaper som er viktige for dedusere nye kandidat interaksjoner.
Robertson et al. 3 også brukt ChIP-seq for å kartlegge STAT1 mål i interferon-γ (IFN-γ)-stimulert og unstimulated humane HeLa S3 celler in vivo. Av Chip-seq, bruker 15,1 og 12,9 millioner unikt kartlagt sekvens leser, og en estimert falske funnrate på mindre enn 0.001, identifiserte de 41 582 og 11 004 antatte STAT1-bindende regioner i stimulert og unstimulated celler, henholdsvis. Av de 34 loci kjent for å inneholde STAT1 interferonresponsive bindingssteder 4-8, funnet brikkespoler seq 24 (71%). ChIP-seq målene ble beriket i sekvenser som ligner på kjente STAT1 bindende motiver. Sammenligninger med to eksisterende ChIP-PCR datasett foreslåttDen brikken-seq følsomhet var mellom 70% og 92% og spesifisiteten var minst 95%. I tillegg var det klart at ChIP-seq tilbyr både lav analytisk kompleksitet og følsomhet som øker med sekvensering dybde.
Som sådan, "neste-generasjons" genom sekvensering teknologi gir 1-2 størrelsesordener økning i mengden av sekvensen som kan være kostnadseffektivt generert over eldre teknologier ni. ChIP-seq metoder derfor direkte gi hel-genom dekning for effektiv profilering av pattedyr protein-DNA vekselvirkninger tre.
I 2006 ble en sterk sammenslutning av varianter i transkripsjonsfaktor 7-aktig 2 (TCF7L2) genet med type 2 diabetes oppdaget 10. Andre forskere har allerede uavhengig kopiert dette funnet i ulike etnisiteter og interessant, fra de første genomet wide forening studier av type 2 diabetes publisert i Nature 11,12 Science 13-15 og ellers 16,17, var den sterkeste foreningen faktisk med TCF7L2, og dette er nå ansett som den mest betydningsfulle genetiske funn i type 2 diabetes hittil 18-20. I tillegg har TCF7L2 blitt knyttet til kreft risiko 21,22, ja, denne forbindelsen ble mer tydelig når 8q24 locus avslørte av genomet wide forening studier av en rekke kreftformer, inkludert kolorektale karsinomer, ble vist å være på grunn av en ekstrem oppstrøms TCF7L2-bindende element kjøring transkripsjon av MYC 23,24. Som sådan, det er stor interesse i å bestemme nedstrøms gener som reguleres av denne tasten transkripsjonsfaktor.
Basert på erfaring med TCF7L2 som et eksempel på metodikken, skisserer denne artikkelen hvordan å generere høy kvalitet ChIP DNA mal. Brikken ble utført i kolorektal karsinom-cellelinje, HCT116, for etterfølgende sekvensering for å bygge en høy resolution kart over genene bundet av TCF7L2 25 i en forsøke å gi ytterligere innsikt i sin nøkkelrolle i patogenesen av komplekse egenskaper.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Cross-link Chromatin
- Vokse celler i 100x20mm cellekultur retter. Mengden av celler kan variere fra 1 til 10 millioner celler pr tallerken avhengig av celletype. Ca 2 millioner celler er tilstrekkelig for en immunoutfelling.
- Cross-lenke celler i 1% Formaldehyd i 10 min ved romtemperatur med sporadisk rocking.
- Quench tverrbinding ved tilsetning av en endelig konsentrasjon på 125 mM glysin og inkuber i 5 minutter ved romtemperatur.
- Vask cellene med 1X Fosfatbufret saltvann (PBS) to ganger, dekanter PBS, og deretter legge 0,2 ml PBS.
- Høste celler med en plast celle skraper inn et mikrosentrifugerør.
- Spinn ned cellene ved 2000 rpm i 5 min ved 4 ° C.
- Aspirer supernatanten. Resuspender celler i SDS Lysis buffer (1% SDS, 10 mM EDTA, 50 mM Tris-HCl pH 8,1) for hele cellelysat eller beholde dem som en pellet for kjernefysisk utvinning.
- Celler kan lagres ved -80 ° C, eller man kan fortsette immediatEly med lydbehandling.
2. Forbered Nuclei (Gå til trinn 3.5 for Whole Cell Lysate)
- Supplement Cell Lysis Buffer (5 mM PIPES pH 8,0, 85 mM KCl, 0,5% NP-40) med 1X Proteinase Inhibitor hvert forsøk.
- Resuspender tint cellen pellet på ca 10 ganger pellet volum med Cell lysebuffer.
- Douce-homogenisere 10 ganger med pistill og deretter inkuberes på is i 10 min.
- Sentrifuger prøven ved 4000 rpm i 5 min ved 4 ° C, kast supernatanten, og lagre kjernefysisk pellet.
3. Sonikering *
- Varm opp SDS lysebuffer og supplere mengden av buffer som skal brukes med Proteinase Inhibitor.
- Gjensuspender kjernefysiske pellet i SDS lysebuffer (ca. 0,5 ml buffer per 1-10 millioner celler)
- Inkuber på is i 10 min.
- Tilsett 0,5 ml porsjoner av prøver til mikrosentrifugerør.
- Sonicate på våt is ved hjelp Misonix sonicator med 30 sek på og 45 sekav på en amplitude innstilling av to. Antall sykluser for ideell fragment størrelse kan bestemmes ved først å prøve ut ulike syklus tall (ex. 2, 4, 8, 12, 16, og 20 eller flere sykluser). Et annet merke sonicator kan anvendes, vil imidlertid variere med forholdene. Eksperimentering med antall sykluser og mengden av tid og på må utføres for å bestemme den ideelle forhold.
- Samle 20 ul av hver prøve for å kontrollere sonikering resultater og for å gjøre kvantifisering,. Resten av prøven kan lagres ved -80 ° C.
- Fortynn 20 prøve ved å tilsette 30 pl av 0,1 x TE-buffer.
- Behandle prøven med 1 mL av RNase A ved 37 ° C i 1 time og deretter tilsett 1 pl proteinase K og inkuber ved 62 ° C i 2 timer.
- Kjør 20 ul av prøven på en 2% agarosegel.
- Rense resterende beløpet av prøven med QIAquick PCR Rensing kit deretter kvantifisere hjelp NanoDrop spektrofotometer.
* For innfødte chiP, kan microccocal nuklease fordøyelse alternativt brukes til å skjære DNA.
4. Block agarosekuler *
- Hvis perler er allerede blokkert, fortsett til trinn 5.1.
- Bruk Protein A eller Protein G agarose. For fem immunoprecipitations (IP-adresser), bruker 600 mL 50% perle slurry (300 ul perle pellet)
- For å vaske perlene, spinne dem ned ved 800 opm i 1 min ved 4 ° C og supernatanten kastes. Legg litt mer enn 2 ml ChIP Dilution buffer (0,01% SDS, 1,2 mM EDTA, 167 mM NaCl, 1,1% Triton X-100, 16,7 mM Tris-HCl, pH 8,1) og blandes ved å snu røret langsomt 10X. Spinn ned igjen ved 800 opm i 1 min ved 4 ° C og supernatanten kastes. Gjenta vask to ganger til.
- Blokker perler ved å rotere ved 4 ° C over natten i blokkering løsning. Se tabell 1 for oppskriften til blokkering løsning.
5. Pre-klar Chromatin
- Tine sonicated kromatin på is.
- Spinn ned ved 12.000 rpm feller 10 min ved 4 ° C og så satt på is med en gang for å fjerne SDS (hvit pellet).
- Samle supernatanten, Pelleten kastes, og kombinere prøver hvis nødvendig.
- Ta ut de beløp som trengs for forsøket basert på beregninger (1-10 ug av kromatin per IP).
- Fortynne kromatin 10X i ChIP fortynning Buffer supplert med proteinase inhibitor.
- Tilsett 100 ul av blokkerte perler per IP.
- Roter ved 4 ° C i 1 time.
6. Immunoprecipitation
- Spinn ned prøvene ved 800 opm i 1 min og Overfør supernatanten til et nytt rør.
- Spinn ned supernatanten ved 800 opm i 1 min og overfør til en annen rent rør.
- Lagre 20 pl av supernatanten, for å tjene som inn-kontroll, ved -20 ° C.
- Delmengde kromatin til antall IP-adresser som skal gjøres i forsøket.
- Tilsett 2 ug av antistoff pr 1-10 ug av kromatin til hver prøve.
- Inkuber over natten 4 ° C med rotasjon.
- Tilsett 100 ul av blokkerte perler til hver IP-prøven.
- Inkuber i 1 time ved 4 ° C med rotasjon.
- Pellet perlene ved å spinne ned ved 800 opm i 1 min og kast så mye av supernatanten som mulig.
- Vask perler gang med Low Salt Immune Complex vaskebufferen. Tilsett 1 ml buffer til hvert rør; rotere ved romtemperatur i 5-8 minutter; spinne ned ved 800 opm i 1 min, og deretter kaste supernatanten. Gjenta vaskes en gang med høyt salt immunkompleks vaskebuffer og LiCl immunkompleks vaskebuffer og to ganger med TE-buffer for en total av 5 vaskinger (tabell 2).
7. Eluering
- Tine inn prøver fra gårsdagen på å bli behandlet med elueringsmidler.
- Gjør Elution Buffer frisk (Tabell 3).
- Lag en mester blanding av nok Elution Buffer nødvendig for IP-adresser og innspill kontrollprøver pluss 1-2 ekstra prøver.
- Tilsett 100 ul Elution Buffer til hver IP-prøven og ruge på rommet temp for 15 mpå med rotasjon.
- Spinn ned ved 800 opm i 1 min og tilsett supernatanten til et nytt rør.
- Legg til 100 mL av elueringsbuffer til hvert rør av perler og inkuber ved romtemperatur i 15 min med rotasjon.
- Vortex i 15 sek etter inkubering; spinne ned ved 5000 opm i 1 min og deretter kombinere supernatant med supernatanten fra den første eluering. (Pass på at det ikke er noen til overs perler i supernatantene. Hvis du er usikker, spinner ned supernatanten igjen ved 5000 rpm i 1 min og samle supernatanten i et nytt rør.
- Legg 180 mL av Elution Buffer til 20 ul av input kontrollprøver.
8. Omvendt Cross-link
- Til 200 ul elueringsmidler og innspill kontroller, tilsett 8 mL av 5 M NaCl.
- Seal rør med parafilm og inkuberes på vannbad ved 65 ° C over natten.
9. DNA Rensing
- Behandle hver prøve med en pl av RNase A i 1 time ved 37 ° C.
- Tilsett 4 μl av 0,5 M EDTA, 8 ul 1M tris-HCl, bland, tilsett 1 pl proteinase K til hver prøve og inkuber ved 45 ° C i 2 timer.
- Rense prøvene ved hjelp QIAquick PCR Rensing kit. Prøvene kan lagres ved -20 ° C og PCR sjekk kan gjøres på et senere tidspunkt.
* Eventuelt kan chip-grade magnetiske perler bli brukt i stedet for agarose for immunoutfelling porsjon.
10. PCR sjekk
- For PCR sjekk, bruke primere for regionene kjent for å være bundet av protein av interesse. Også bruke primere for ikke-bindende regioner som negative kontroller.
- Blanding av reagensene for reaksjonen. Fortynn Input prøven ved 1:100 (tabell 3).
- Kjør reaksjon. PCR Program:
Trinn 1: 94 ° C 3 min
Trinn 2: 94 ° C 20 sek
59 ° C 30 sek
72 ° C 30 sek
(Gjenta trinn 2 for minst 30 CYCLes)
Trinn 3: 72 ° C 2 min
- Løpe prøver på 1% agarosegel.
- Anrikning kan også bestemmes kvantitativt med sanntids-PCR.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Når kromatin ble sonikert og har blitt behandlet med RNase og Proteinase, bør prøvene kjørt på 2% agarosegel presentere en smøre og mesteparten av DNA i den ønskede størrelse. Hvis flere forskjellige sykluser testes, bør en gradvis reduksjon i størrelse betraktes som antall sykluser øker (figur 2).
Etter endt immunoprecipitation del av protokollen berikelse kan enten kontrolleres ved PCR eller real-time PCR. For PCR prøvene kjøres på en agarose gel bør det være band i Input og Chip (ved hjelp av antistoff for protein av interesse, som er TCF7L2 i dette tilfellet) sample baner og ingenting, eller på de fleste, en veldig svak bandet (bakgrunnsstøy) i IgG (negative) kontroll kjørefelt for den positive bindende regionen. For den negative bindende regionen bør det være veldig svak eller ingen band for IgG kontroll og Chip baner. Det bør være et band i Input kjørefelt (figur 3).
Figur 4 viser de samme prøvene undersøkt av real-time PCR. Som med den foregående figur, bør det være en betydelig fold anrikning av den positive bindende region for chip Eksempel over IgG-kontroll. I tillegg bør det være liten anrikning, om noen, sett i den negative bindende region.

Figur 1. Flow diagram av ChIP prosess. Klikk her for å se større figur .
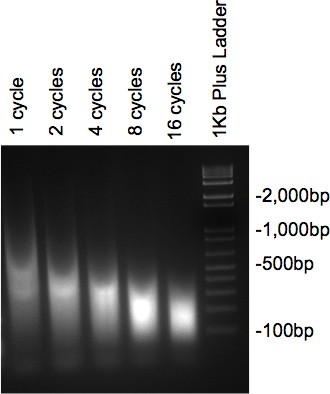
Figur 2. Gel sjekk av DNA lydbehandling.

Figur 3. PCR Sjekk av chip.
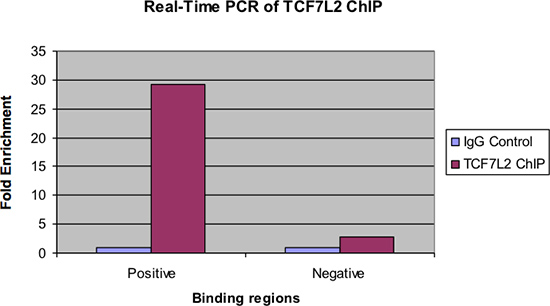
Figur 4. Real-time PCR av TCF7L2 ChIP.
| Reagens | Volum |
| Bead pellet | 300 mL |
| BSA (50 mg / ml) | 30 ul |
| 100X Proteinase Inhibitor | 10 pl |
| ChIP fortynning Buffer | 660 ul |
| Total | 1000 ul |
Tabell 1. Oppskrift for å blokkere agarose.
| Buffer | Komponenter |
| Lav Salt Immune Complex Wash Buffer | 0,1% SDS 1% Triton X-100 2 mM EDTA 20 mM Tris-HCl pH 8.1 150 mM NaCl |
| Høy Salt Immune Complex Wash Buffer | 0,1% SDS 1% Triton X-100 2 mM EDTA 20 mM Tris-HCl pH 8.1 500 mM NaCl |
| LiCl Immune Complex Wash Buffer | 0,25 M LiCl 1% NP-40 1% deoxycholate 1 mM EDTA 10 mM Tris-HCl pH 8.1 |
| TE Buffer | 10 mM Tris-HCl pH 8.1 1 mM EDTA pH 8,0 |
Tabell 2. Chip vask buffere.
| Reagens | Volum |
| 10 pl | |
| 1 M NaHCO3 | 20 pl |
| H 2 O | 170 ul |
Tabell 3. Elueringsbuffer for én IP.
| Reagens | 50 pl Reaction | 20 ul Reaction |
| Vann | 27 ul | 10.8 ul |
| 5X PCR reaksjon buffer | 10 pl | 4 pl |
| MgCl2 | 4 pl | 1.6 ul |
| dNTP (10 mM) | 1 ul | 0.4 ul |
| Primerblanding (5 UM hver) | 2 ul | 0.8 ul |
| Taq (Promega Hotstart) | En ul </ Td> | 0.4 ul |
| ChIP DNA | 5 ul | 2 ul |
Tabell 4. PCR reaksjon volumer.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Det er nå mulig å gjennomføre et stort genom-profil av protein-DNA-interaksjoner forening ved hjelp brikkespoler seq, som har nylig blitt demonstrert med andre transkripsjonsfaktorer 2,3. Nøkkelen til en vellykket sekvensering utfallet er den generasjonen av høy kvalitet kromatin immunoprecipitation DNA mal.
Når DNA-malen har blitt generert og forsikret å være tilstrekkelig anriket, kan man deretter ta det i bibliotek forberedelse for etterfølgende sekvensering. For eksempel kan man bruke sekvensering bibliotek protokollen gitt av leverandøren, Illumina. Størrelsen utvalg av dette biblioteket kan bli utført ved gelelektroforese og påfølgende fjerning og rensing av DNA i ~ 200 - til 700-bp-serien. Redusere størrelsen og innsnevring størrelsen spekter av DNA hentet fra gel rensing er ment å bedre posisjonell oppløsning på ChIP-seq. Ved berikende for mindre biter av innspill DNA bundet til den faktoren av interest, vil man forvente at nettstedet plasseringen vil få oppløsning. Strammere størrelse valg forbedrer også størrelsen ensartethet av molekylære kolonier produsert på Illumina plattformen. Slike kolonistørrelse ensartethet øker også den effektive leste nummer erholdt. Kortere innspill DNA-størrelse gir også mer robuste kolonier på Illumina plattform, og dette kan bety at kortere DNA stykker innenfor et gitt innspill prøven distribusjon vil være representert mer effektivt i den endelige rekkefølgen utgang enn er lengre innspill stykker fra samme fordeling.
De bioinformatiske tilnærminger til "neste-generasjons" sekvens analyse fortsetter å utvikle seg, med mange leverandører gjør deres programvare med åpen kildekode for videre foredling. Man kan forvandle leser det kartet til unike genomisk steder inn i en DNA fragment overlapping profil. Vesentlige topper kan identifiseres ved threshholding profiler i en høyde som tilsvarer en beregnet falske funnrate. Stillingen specific frekvens matriser avledet fra dette arbeidet kan benyttes til å identifisere og lokalisere DNA-bindende områder over det humane genom for en gitt faktor.
Men man må være forsiktig med hensyn til hvilke faktorer man ønsker å studere med Chip-seq. Før du tar fatt på en slik studie bør man vurdere om et antistoff er tilgjengelig på markedet som er brukbare i ChIP innstillingen, som en fattig antistoff kan ha svært uheldige effekter på ens eksperimentelle resultater. I tillegg bør man vurdere om det er spleise isoformer av det protein som studeres; riktignok blir TCF7L2 kjent for å ha mange isoformer slik at vi var ekstra forsiktig ved valg av et antistoff som bindes til aminosyre gjennomgående stede i alle de viktigste isoformer av denne transkripsjonsfaktor 25.
Oppsummert kan kombinasjonen av kromatin immunoprecipitation og ultra-high-throughput sekvensering (ChIP-seq) identifisere og kartlegge protein-DNA vekselvirkninger i et gitt vev eller cell linje. Vi har skissert hvordan å generere en høy kvalitet ChIP mal for etterfølgende sekvensering.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Forfatterne erklærer at de har ingen konkurrerende finansielle interesser.
Acknowledgments
Arbeidet er støttet av en Institute Development Award fra The Children Hospital of Philadelphia.
Materials
| Name | Company | Catalog Number | Comments |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| EZ-ChIP Kit | Millipore | 17-371 | |
| GoTaq Hot Start Polymerase | Promega | M5001 | |
| Misonix Sonicator | Qsonica | XL-2000 | |
| NanoDrop 1000 Spectrophotometer | Thermo-Scientific | ||
| Positive control primer sequences (TCF7L2-1) Forward- 5'-TCGCCCTGTCAATAATCTCC-3' Reverse- 5'-GCTCACCTCCTGTATCTTCG-3' Negative control primer sequences (CTRL-1) Forward-5'-ATGTGGTGTGGCTGTGATGGGAAC-3' Reverse- 5'-CGAGCAATCGGTAAATAGGTCTGG-3' |
|||
References
- Odom, D. T., et al. Control of pancreas and liver gene expression by HNF transcription factors. Science. 303, 1378-1381 (2004).
- Johnson, D. S., Mortazavi, A., Myers, R. M., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316, 1497-1502 (2007).
- Robertson, G., et al. Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing. Nature Methods. 4, 651-657 (2007).
- Reich, N. C., Liu, L. Tracking STAT nuclear traffic. Nat. Rev. Immunol. 6, 602-612 (2006).
- Lodige, I., et al. Nuclear export determines the cytokine sensitivity of STAT transcription factors. The Journal of Biological Chemistry. 280, 43087-43099 (2005).
- Schroder, K., Sweet, M. J., Hume, D. A. Signal integration between IFNgamma and TLR signalling pathways in macrophages. Immunobiology. 211, 511-524 (2006).
- Vinkemeier, U. Getting the message across, STAT! Design principles of a molecular signaling circuit. The Journal of Cell Biology. 167, 197-201 (2004).
- Brierley, M. M., Fish, E. N. Stats: multifaceted regulators of transcription. J. Interferon Cytokine Res. 25, 733-744 (2005).
- Bentley, D. R. Whole-genome re-sequencing. Current Opinion in Genetics & Development. 16, 545-552 (2006).
- Grant, S. F., et al. Variant of transcription factor 7-like 2 (TCF7L2) gene confers risk of type 2 diabetes. Nature Genetics. 38, 320-323 (2006).
- Sladek, R., et al. A genome-wide association study identifies novel risk loci for type 2 diabetes. Nature. 445, 881-885 (2007).
- Wellcome Trust Case Control Consortium. Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls. Nature. 447, 661-678 (2007).
- Saxena, R., et al. Genome-wide association analysis identifies loci for type 2 diabetes and triglyceride levels. Science. 316, 1331-1336 (2007).
- Zeggini, E., et al. Replication of genome-wide association signals in UK samples reveals risk loci for type 2 diabetes. Science. 316, 1336-1341 (2007).
- Scott, L. J., et al. A genome-wide association study of type 2 diabetes in Finns detects multiple susceptibility variants. Science. 316, 1341-1345 (2007).
- Steinthorsdottir, V., et al. A variant in CDKAL1 influences insulin response and risk of type 2 diabetes. Nature Genetics. 39, 770-775 (2007).
- Salonen, J. T., et al. Type 2 Diabetes Whole-Genome Association Study in Four Populations: The DiaGen Consortium. American Journal of Human Genetics. 81, 338-345 (2007).
- Zeggini, E., McCarthy, M. I. TCF7L2: the biggest story in diabetes genetics since HLA. Diabetologia. 50, 1-4 (2007).
- Weedon, M. N. The importance of TCF7L2. Diabet. Med. 24, 1062-1066 (2007).
- Hattersley, A. T. Prime suspect: the TCF7L2 gene and type 2 diabetes risk. The Journal of Clinical Investigation. 117, 2077-2079 (2007).
- Yochum, G. S., et al. Serial analysis of chromatin occupancy identifies beta-catenin target genes in colorectal carcinoma cells. Proceedings of the National Academy of Sciences of the United States of America. 104, 3324-3329 (2007).
- Duval, A., Busson-Leconiat, M., Berger, R., Hamelin, R. Assignment of the TCF-4 gene (TCF7L2) to human chromosome band 10q25.3. Cytogenet. Cell Genet. 88, 264-265 (2000).
- Pomerantz, M. M., et al. The 8q24 cancer risk variant rs6983267 shows long-range interaction with MYC in colorectal cancer. Nature Genetics. 41, 882-884 (2009).
- Tuupanen, S., et al. The common colorectal cancer predisposition SNP rs6983267 at chromosome 8q24 confers potential to enhanced Wnt signaling. Nature Genetics. 41, 885-890 (2009).
- Zhao, J., Schug, J., Li, M., Kaestner, K. H., Grant, S. F. Disease-associated loci are significantly over-represented among genes bound by transcription factor 7-like 2 (TCF7L2) in vivo. Diabetologia. 53, 2340-2346 (2010).
- Benjamini, Y., Yekutieli, D. Quantitative trait Loci analysis using the false discovery rate. Genetics. 171, 783-790 (2005).



