Protocolo para HER2 de los pescados usando un fijador de tejido no enlazan a la Cruz, libre de formol para combinar las ventajas de la criopreservación y la fijación de formalina
Summary
Hibridación in situ de la fluorescencia (pescado) se requiere a menudo en combinación con la histopatología y el diagnóstico molecular para la selección del tratamiento en la medicina personalizada. Fijador de tejido no enlazan a la Cruz, libre de formol novedoso que permite alta calidad morfológica, molecular y análisis de peces de la misma muestra por adición de un paso de la fijación antes de pescado se presenta.
Abstract
Evaluación morfológica de muestras de tejidos formalina-fijos, parafina-encajado (FFPE) ha sido el estándar de oro para el diagnóstico de cáncer durante décadas debido a su excelente conservación de la morfología. Medicina personalizada proporciona cada vez más terapias individualmente adaptadas y dirigidas para caracterizadas enfermedades individuales habilitadas por tecnologías combinadas de análisis morfológicas y moleculares y diagnóstico. Realización de análisis morfológicos y moleculares de la misma muestra FFPE es difícil debido al impacto negativo de formalina por modificación química y cross-linking de proteínas y ácidos nucleicos. Un fijador de tejido no enlazan a la Cruz, libre de formol se ha desarrollado recientemente para cumplir con ambos requisitos, es decir, para preservar la morfología como FFPE y biomoléculas como criopreservación. Puesto que los peces se requieren a menudo en combinación con la histopatología y diagnóstico molecular, probamos la aplicabilidad de los protocolos de pescado sobre los tejidos tratados con este nuevo fijador. Se encontró la fijación de formalina de secciones histológicas de no-cross-linking, libre de formol y embebido en parafina (NCFPE) mama cáncer tejido generado resultados equivalentes a los tejido FFPE en receptor de factor de crecimiento epidérmico humano 2 (HER2) Análisis de los pescados. Este protocolo describe cómo se puede utilizar un ensayo FISH originalmente desarrollado y validado para tejido FFPE para tejidos NCFPE por un simple paso de posteriores a la fijación de las secciones histológicas.
Introduction
Medicina personalizada depende cada vez más pruebas de múltiples parámetros que involucran análisis morfológicos y moleculares de los tejidos. Fijación con formalina de los tejidos como el estándar de oro proporciona excelente calidad morfológica1,2. Sin embargo, modificación química inducida de formol y cross-linking de proteínas y ácidos nucleicos3,4,5 negativamente interfiere con el análisis molecular6. Estas modificaciones moleculares limitar la calidad de los ácidos nucleicos y proteínas y puede resultar en gene secuencia artefactos7 o disminución de la sensibilidad de la reacción en cadena de polimerasa (polimerización en cadena)-basado en análisis8. Aunque se obtuvieron grandes esfuerzos para optimizar las pruebas moleculares para tejido FFPE, crio-preservación de los tejidos es en general superior a la fijación de formalina, lo que es necesario dividir las muestras de tejido para los procedimientos de conservación diferentes. Para evitar la necesidad de criopreservación para análisis moleculares, un no-cross-linking, fijador libre de formol, PAXgene tejido fue desarrollado. Este sistema comercialmente disponible consiste en una fijación y una solución de estabilización que contiene diversos alcoholes, ácido acético y un compuesto orgánico soluble. Adecuada preservación de los ácidos nucleicos, las proteínas (phospo) y la morfología fue demostrado en varios estudios6,9,10,11,12,13.
Una particular aplicación en el diagnóstico de cáncer es detectar una amplia gama de alteraciones cromosómicas, translocaciones, deleciones submicroscópicas y amplificaciones 14peces. Por ejemplo, 20% de los tumores de cáncer de mama invasivo muestran amplificación del receptor del factor de crecimiento epidérmico humano 2 (HER2) gene15,16,17, que se asocia a pronóstico pobre18 ,19. Determinación del estado de la sobreexpresión y amplificación de HER2 en cáncer de mama por los peces es necesario para seleccionar a pacientes para la terapia anti-HER2-dirigida y es un complemento diagnóstico enumerado por la Asociación Federal de drogas (FDA) (http://www.fda.gov/ MedicalDevices/ProductsandMedicalProcedures/InVitroDiagnostics/ucm301431.htm)17. Para permitir una amplia aplicación de diagnósticos de compañero a base de pescado en la atención sanitaria, ensayos fueron desarrollados y aprobados para los tejidos FFPE.
En un estudio previo, demostró que los tejidos NCFPE no pueden utilizarse para ensayos de peces que han sido aprobados para el tejido FFPE. Sin embargo, las pruebas de tiempo serie de post diferentes métodos de fijación de los tejidos NCFPE con 4% tamponada formaldehído demostrada que publicar tiempos de fijación de h 18 o más de las secciones de tejido NCFPE lograr resultados equivalentes a los de los tejidos FFPE14.
En este estudio, proporcionar un protocolo detallado y demostrar el impacto de fijación y 24 h el tejido no enlazan a la Cruz, libre de formol 4% tamponada con posteriores a la fijación formaldehído en secciones de calidad FISH HER2 y el ARN de la misma muestra de tejido primario.
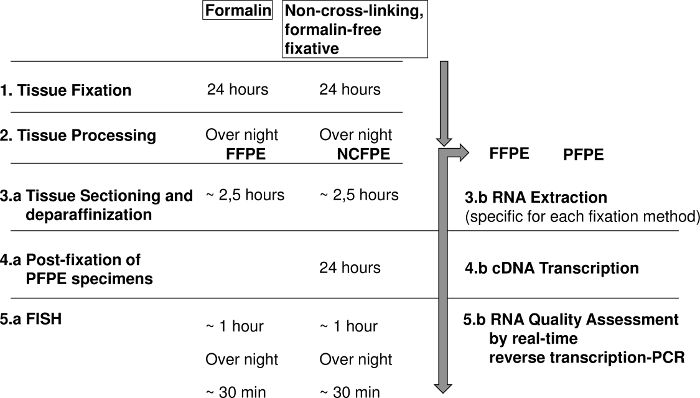
Figura 1: diagrama que muestra los pasos para en situ hibridación de la fluorescencia (pescado) y análisis de ARN de formalina fijada parafina incrustado (FFPE) y la vinculación de cruz no, libre de formol fija parafina encajados los tejidos (NCFPE). Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
Los resultados muestran dos resultados claves. En primer lugar, un paso de la fijación simple 24 h SBFS de secciones cortadas de los tejidos NCFPE es suficiente para obtener resultados equivalentes a los FFPE en análisis de peces utilizando pescado ensayos aprobados para los tejidos FFPE (véase también la referencia14). Este protocolo tiene la ventaja de utilizar ensayos de peces originalmente desarrollado y aprobado para FFPE sin necesidad de revalidación global (por ejemplo, mediante la optimización de las condiciones de la hibridación de tejidos no-cruz-ligado). El protocolo de pescado puede usarse exactamente como se describe en las instrucciones del fabricante.
Pequeñas modificaciones de las instrucciones de manufacturer´s que se describe en este resultado de protocolo (por ejemplo tejido sección diámetro incubación períodos y en los pasos de parafina) de anteriores adaptaciones, explícitamente recomendado por el fabricante debido al uso de tejidos diferentes tipos y métodos de fijación.
El paso de la fijación es crítico porque requiere un tiempo de reacción química de 18-24 h, que se extiende el tiempo de análisis por un día pero que permite el mismo flujo de trabajo diario, desarrollado para los tejidos FFPE (figura 1). SBFS se divulga para penetrar el tejido a una tasa promedio de 1 mm por hora22 a 5 mm en 2 h según el tipo de tejido23,24. La observación de que sólo la fijación prolongada de más de 18 h puede cambiar las propiedades de NCFPE secciones FFPE, indica que no la penetración de este fijador pero el tiempo de reacción química es fundamental para lograr los resultados deseados1 ,14.
En segundo lugar, el tejido restante de NCFPE que no se utiliza para la fijación puede utilizarse para otros análisis moleculares como las biomoléculas están bien conservadas. Esto fue demostrado por transcripción reversa en tiempo real PCR (figura 3). El ensayo es más sensible y específico que el electroferograma deriva valor RIN (número de integridad del RNA) con respecto a la calidad del RNA (es decir, modificación química y fragmentación) para mRNA aislado de tejidos parafina-encajado8. Control de calidad se limita en este estudio a la PCR en tiempo real ya que los grupos independientes han demostrado que además de RNA, la calidad del ADN y las proteínas aisladas de NCFPE es mejor que la de FFPE tejidos 8,9, 13.
El protocolo presentado sigue, en su caso (por ejemplo, receta SBFS, las condiciones de fijación, control de calidad de RNA, validación), los requisitos de las especificaciones técnicas del CEN para procedimientos pre-analíticos para el diagnóstico in vitro (IVD), que han sido liberados recientemente por el Comité Europeo de normalización (CEN) (e.g. CEN/TS 16827-1:2015 para el aislamiento del RNA de tejidos FFPE que se refiere a la norma ISO 15189).
El método se limita a este fijador específico. Aunque la buena preservación de las biomoléculas y morfología mediante el fijador no enlazan a la Cruz, libre de formol se ha presentado en varios estudios, se utiliza principalmente en la investigación pero no en aplicaciones médicas rutinarias. Una de las razones es que cambiar el estándar de oro SBFS fijación por otro fijador requeriría una variedad de estudios de validación como no todos los protocolos optimizados para material FFPE pueden ser utilizados para materiales con fijadores no enlazan a la Cruz. No se probaron otros métodos de fijación de tejido para este protocolo de pesca.
El paso de posteriores a la fijación simple que se describe en este manuscrito tiene la ventaja de utilizar ensayos de IVD aprobado tanto para tejidos FFPE y NCFPE.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Agradecemos al equipo del laboratorio de patología molecular en el Instituto de patología de la Universidad médica de Graz por su experiencia y apoyo. Además, agradecemos a Iris Kufferath y Daniela Pabst para asistencia técnica, Bernadette Rieger y Sylvia Eidenhammer para el procesamiento de los casos HER2 como Kinga Szurian (patólogo) y Tamas Regényi (3DHISTEC). Agradecemos a Penelope Kungl para corregir el manuscrito. Este trabajo ha sido apoyado financieramente por el fondo de investigación Doppler cristiano, del Ministerio Federal austriaco de ciencia, investigación y economía y la Fundación Nacional para la investigación, tecnología y desarrollo.
Materials
| SAV LP GmbH, Flintsbach a. I., Germany | www.sav-lp.de | FN-200L-4-1 | Tissue fixative |
| Simport Plastic Ltd., Beloeil, Canada | www.simport.com | M-498 | Safekeeping device providing labelling, fixation and paraffin embedding for tissue samples. |
| Qiagen GmbH, Hilden Germany | www.qiagen.com | 765112 | For collection, fixation, and stabilization of tissue samples: 10 Prefilled Reagent Containers, containing PAXgene Tissue FIX and PAXgene Tissue STABILIZER. |
| ThermoFisher Scientific | www.thermofisher.com | 813150 | Processing of biological specimens from chemical fixation to paraffin infiltration |
| Merck Millipore, Munich, Germany | www.analytics-shop.com/de/hersteller/millipore.html | MC1009834000 | Dehydration of tissue samples |
| VWR Chemicals, Darmstadt, Germany | https://at.vwr.com | 10293EP | Dehydration of tissue samples |
| ACM Herba Chemosan Apotheker AG, Vienna, Austria | www.herba-chemosan.at | 2549662 32 | Paraffin used in a tissue processing device |
| Leica Mikrosysteme Handels GmbH, Vienna, Austria | www.LeicaBiosystems.com | 39602004 | Paraffin embedding medium |
| Sanova Diagnostik, Vienna, Austria | www.sanova.at | 5229 | Paraffin embedding instrument for tissues for histology |
| Histocom Medizintechnik Vertriebs GmbH, Wiener Neudorf, Austria | www.histocom.info | M 910010 | This device is used for especially sophisticated paraffin sectioning techniques in biology and medicine. Only skilled or specially trained personnel must operate the microtome, i.e. clamping the specimen, trimming, sectioning and taking off the sections from the instrument. |
| Dako Denmark A/S, Glostrup, Denmark | www.chem.agilent.com | K802021-2 | Coated glass slides, intended for mounting formalin-fixed, paraffin-embedded tissue sections. |
| Qiagen GmbH, Hilden Germany | www.qiagen.com | 73504 | For purification of total RNA from formalin-fixed, paraffin-embedded tissue sections |
| Qiagen GmbH, Hilden Germany | www.qiagen.com | 765134 | For purification of total RNA from PAXgene-fixed, paraffin-embedded tissue sections |
| ZytoVision GmbH, Bremerhaven, Germany | www.zytovision.com | Z-2015-200 | For the detection of ERBB2 (a.k.a HER2) gene amplification frequently observed in solid malignant neoplasms e.g. breast cancer samples. |
| 3DHISTEC, Budapest, Hungary | www.3dhistech.com | Digital device for scanning tissue slides equipped with a 40x/1.2 NA objective, Quad band filters (DAPI/FITC/TRITC/Cy5) filters and a 5.5Mpx, 16 bit, cooled scientific CMOS (Complementary Metal Oxide Semiconductor) camera. | |
| ThermoFisher Scientific | www.thermofisher.com | 4368814 | The High Capacity cDNA Reverse Transcription Kit contains all components necessary for the quantitative conversion of up to 2 µg of total RNA to single-stranded cDNA in a single 20 µL reaction. |
| ThermoFisher Scientific | www.thermofisher.com | 4367659 | PCR Master Mix containing polymerase, nucleotides and SYBR Green for PCR and quanitification of amplicons. |
| ThermoFisher Scientific, Wilmington, DE | www.thermofisher.com | Ser. Nr. F239 | Spectrophotometer for nucleic acid quantification. |
| QuantStudio 7 Flex Real-time PCR System | www.thermofisher.com | 4485701 | PCR machine, |
| Vysis/Abbott Laboratories. Abbott Park, Illinois, U.S.A | www.molecular.abbott/us/en/products/instrumentation/thermobrite#order | e.g. (ThermoBrite) 07J91-020 | Temperature controlled slide processing system for in-situ denaturation/hybridization procedures. |
References
- Fox, C. H., Johnson, F. B., Whiting, J., Roller, R. P. Formaldehyde fixation. J. Histochem.Cytochem. 33 (8), 845-853 (1985).
- Oosterhuis, J. W., Coebergh, J. W., van Veen, E. B. Tumour banks: well-guarded treasures in the interest of patients. Nat. Rev. Cancer. 3 (1), 73-77 (2003).
- Masuda, N., Ohnishi, T., Kawamoto, S., Monden, M., Okubo, K. Analysis of chemical modification of RNA from formalin-fixed samples and optimization of molecular biology applications for such samples. Nucleic Acids Res. 27 (22), 4436-4443 (1999).
- Metz, B., Kersten, G. F., Hoogerhout, P., Brugghe, H. F., Timmermans, H. A., de Jong, A., et al. Identification of formaldehyde-induced modifications in proteins: reactionswith model peptides. J. Biol. Chem. 279 (8), 6235-6243 (2003).
- Evers, D. L., Fowler, C. B., Cunningham, B. R., Mason, J. T., O’Leary, T. J. The effect of formaldehyde fixation on RNA: optimization of formaldehyde adduct removal. J.Mol. Diagn. 13 (3), 282-288 (2011).
- Groelz, D., Sobin, L., Branton, P., Compton, C., Wyrich, R., Rainen, L. Non-formalin fixative versus formalin-fixed tissue: A comparison of histology and RNA quality. Exp. Mol. Pathol. 94 (1), 188-194 (2013).
- Do, H., Dobrovic, A. Sequence artifacts in DNA from formalin-fixed tissues: causes and strategies for minimization. Clin. Chem. 61, 64-71 (2015).
- Kashofer, K., Viertler, C., Pichler, M., Zatloukal, K. Quality Control of RNA Preservation and Extraction from Paraffin-Embedded Tissue: Implications for RT-PCR and Microarray Analysis. PloS One. 8 (7), e70714 (2013).
- Viertler, C., Groelz, D., Gündisch, S., Kashofer, K., Reischauer, B., Riegman, P. H., et al. A new technology for stabilization of biomolecules in tissues for combined histological and molecular analyses. J. Mol. Diagn. 14 (5), 458-466 (2012).
- Gundisch, S., Slotta-Huspenina, J., Verderio, P., Maura, C., Ciniselli, S. P., Schott, S., et al. Evaluation of colon cancer histomorphology: A comparison between formalin and PAXgene tissue fixation by an international ring trial. Virchows Arch. 465 (5), 509-519 (2014).
- Ergin, B., Meding, S., Langer, R., Kap, M., Viertler, C., Schott, C., et al. Proteomic analysis of PAXgene-fixed tissues. J. Proteome Res. 9 (10), 5188-5196 (2010).
- Kap, M., Smedts, F., Oosterhuis, W., Winther, R., Christensen, N., Reischauer, B., et al. Histological Assessment of PAXgene Tissue Fixation and Stabilization Reagents. PLoS ONE. 6 (11), e27704 (2011).
- Mathieson, W., Marcon, N., Antunes, L., Ashford, D. A., Betsou, F., Frasquilho, S. G., et al. A Critical Evaluation of the PAXgene Tissue Fixation System: Morphology, Immunohistochemistry, Molecular Biology, and Proteomics. Am J Clin Pathol. 146, 25-40 (2016).
- Oberauner-Wappis, L., Loibner, M., Viertler, C., Groelz, D., Wyrich, R., Zatloukal, K. Protocol for HER2 FISH determination on PAXgene-fixed and paraffin-embedded tissue in breast cancer. Int J. Exp. Path. 97 (2), 202-206 (2016).
- Pauletti, G., Godolphin, W., Press, M. F., Slamon, D. J. Detection and quantitation ofHER-2/neu gene amplification in human breast cancer archival material using fluorescence in situ hybridization. Oncogene. 13 (1), 63-72 (1996).
- Owens, M. A., Horten, B. C., Da Silva, M. M. HER2 amplification ratios by fluorescence in situ hybridization and correlation with immunohistochemistry in a cohort of 6556 breast cancer tissues. Clin.Breast Cancer. 5 (1), 63-69 (2004).
- Wolff, A. C., Hammond, M. E., Schwartz, J. N., Hagerty, K. L., Allred, D. C., Cote, R. J., et al. American Society of Clinical Oncology/College of American Pathologists guideline recommendations for human epidermal growth factor receptor 2 testing in breast cancer. Arch Pathol Lab Med. 131 (1), 18-43 (2007).
- Press, M. F., Bernstein, L., Thomas, P. A., Meisner, L. F., Zhou, J. Y., Ma, Y., et al. HER-2/neu gene amplification characterized by fluorescence in situ hybridization: poor prognosis in node-negative breast carcinomas. J. Clin. Oncol. 15 (8), 2894-2904 (1997).
- Yamauchi, H., Stearns, V., Hayes, D. F. When is a tumor marker ready for prime time? A case study of c-erbB-2 as a predictive factor in breast cancer. J. Clin. Oncol. 19 (8), 2334-2356 (2001).
- Paulik, R., Micsik, T., Kiszler, G., Kaszál, P., Székely, J., Paulik, N., Várhalmi, E., Prémusz, V., Krenács, T., Molnár, B. An optimized image analysis algorithm for detecting nuclear signals in digital whole slides for histopathology. Cytometry A. , (2017).
- Applied Biosystems. . QuantStudio 6 and 7 Flex Real-Time PCR System Software. , (2013).
- NCCLS. . Quality Assurance for Immunocytochemistry. , (1999).
- Baker, J. R. . Principles of Biological Microtechnique. , (1958).
- Hewitt, S. M., Lewis, F., Cao, Y., Conrad, R. C., Cronin, M., Danenberg, K. D., et al. Tissue handling and specimen preparation in surgical pathology: issues concerning the recovery of nucleic acids from formalin-fixed, paraffin-embedded tissue. Arch Pathol. Lab Med. 132 (12), 1929-1935 (2008).

