Protokoll für HER2 Fischen mit nicht-Cross-linking, Formalin-freies Gewebe Fixiermittel, Vorteile der Kryokonservierung und Formalin Fixierung zu kombinieren
Summary
Fluoreszenz in Situ Hybridisierung (FISH) wird häufig in Kombination mit Histopathologie und molekulare Diagnostik für die Auswahl der Therapie in der personalisierten Medizin benötigt. Eine neuartige nicht-Cross-linking, Formalin-freies Gewebe Fixiermittel, mit dem hochwertigen morphologische, molekulare und Fisch Analysen aus der gleichen Probe durch Zugabe von einem Post-Fixierung Schritt bevor Fisch dargestellt wird.
Abstract
Morphologische Beurteilung von Formalin fixiert, Paraffin-eingebetteten (FFPE) Gewebeproben ist seit Jahrzehnten durch seine ausgezeichnete Erhaltungszustand der Morphologie der Goldstandard für die Krebsdiagnostik. Personalisierter Medizin bietet zunehmend individuell angepasste und gezielte Therapien für zeichnet sich einzelne Krankheiten, die durch kombinierte morphologische und molekulare analytische Technologien und Diagnostik ermöglicht. Leistung der morphologischen und molekularen Tests aus der gleichen FFPE-Probe ist schwierig wegen der negativen Auswirkungen von Formalin durch chemische Modifikation und Vernetzung von Nukleinsäuren und Proteine. Ein nicht-Cross-linking, Formalin-freies Gewebe Fixiermittel wurde vor kurzem entwickelt, d. h. beide Voraussetzungen zu erfüllen, Morphologie wie Proteinkinase und Biomoleküle wie Kryokonservierung zu bewahren. Da Fische oft in Kombination mit Histopathologie und molekulare Diagnostik erforderlich ist, testeten wir die Anwendbarkeit des Fisch-Protokolle auf Gewebe, die mit dieser neuen Fixativ behandelt. Wir fanden, dass Formalin Post-Fixierung der histologischen Abschnitte von nicht-Cross-linking, Formalin-frei und Paraffin-eingebetteten (NCFPE) Brust Krebs Gewebe erzeugt gleichwertige Ergebnisse mit FFPE Gewebe im menschlichen epidermalen Wachstumsfaktor-Rezeptor 2 (HER2) FISH-Analyse. Dieses Protokoll beschreibt, wie ein Fisch-Assay, ursprünglich entwickelt und validiert für FFPE Gewebe durch einen einfachen Post-Fixierung Schritt der histologischen Abschnitte für NCFPE Gewebe verwendet werden kann.
Introduction
Personalisierter Medizin setzt zunehmend auf Multiparameter-Tests mit morphologischen und molekularen Gewebe Analysen. Formalin Fixierung des Gewebes als Goldstandard bietet ausgezeichnete morphologische Qualität1,2. Molekulare Analyse6stört jedoch Formalin-induzierte chemische Modifikation und Vernetzung von Proteinen und Nukleinsäuren3,4,5 negativ. Diese molekularen Änderungen die Qualität von Nukleinsäuren und Proteinen zu begrenzen und gen-Sequenz Artefakte7 oder verringerte Empfindlichkeit der Polymerase-Kettenreaktion (PCR) unter Umständen-basierte Assays8. Obwohl große Anstrengungen unternommen wurden, um molekulare Tests für FFPE Gewebe zu optimieren, ist Kryokonservierung von Gewebe in Generaloberin, Formalin Fixierung, machen es notwendig, Gewebeproben für verschiedene Erhaltung Verfahren aufgeteilt. Zur Vermeidung der Notwendigkeit einer Kryokonservierung für molekulare Analysen, eine nicht-Cross-linking, entwickelte Formalin-freie Fixativ PAXgene Gewebe. Diese im Handel erhältliche System besteht aus einer Fixierung und einer Stabilisierung-Lösung mit verschiedenen Alkoholen, Essigsäure und eine lösliche organische Verbindung. Pflege und Bewahrung von Nukleinsäuren, Proteine (Phospo) und Morphologie wurde in mehreren Studien6,9,10,11,12,13gezeigt.
Eine besondere Anwendung in der Krebs-Diagnose ist Fisch erkennen eine umfassende Palette von chromosomalen Veränderungen, wie z. B. Translokationen, submikroskopische Deletionen und Vergrößerungen 14. Zwanzig Prozent der invasiven Brustkrebstumoren zeigen beispielsweise Verstärkung des menschlichen epidermalen Wachstumsfaktor-Rezeptor 2 (HER2) gen15,16,17, die im Zusammenhang mit einer schlechten Prognose18 ,19. Bestimmung des HER2-Überexpression und Verstärkung Status bei Brustkrebs von Fischen ist erforderlich, um Patienten für Anti-HER2-gerichtete Therapie auswählen und ist ein Begleiter diagnostische aufgeführt von der Federal Drug Association (FDA) (http://www.fda.gov/ MedicalDevices/ProductsandMedicalProcedures/InVitroDiagnostics/ucm301431.htm)17. Um eine breite Anwendung der Fisch basierende Begleiter Diagnostik in der Gesundheitsversorgung zu ermöglichen, wurden Tests entwickelt und zugelassen für FFPE Gewebe.
In einer früheren Studie zeigten wir, dass NCFPE Gewebe für Fisch-Assays verwendet werden können, die für FFPE Gewebe genehmigt worden sind. Prüfungen der Reihe von anderen Dienstposten Fixierung Verfahren von NCFPE Gewebe mit 4 %, gepuffert Formaldehyd hat gezeigt, dass Fixierung Zeiten von 18 h oder länger von NCFPE Gewebeschnitte buchen erreichte aber gleichwertige Ergebnisse mit FFPE Gewebe14.
In dieser Studie wir bieten ein detailliertes Protokoll und zeigen, dass die Auswirkungen von nicht-Cross-linking, Formalin-freies Gewebe Fixierung und 24 h 4 % Formaldehyd Post-Fixierung auf Abschnitten verwendet für die HER2-Fisch und RNA aus der gleichen primären Gewebeprobe gepuffert.
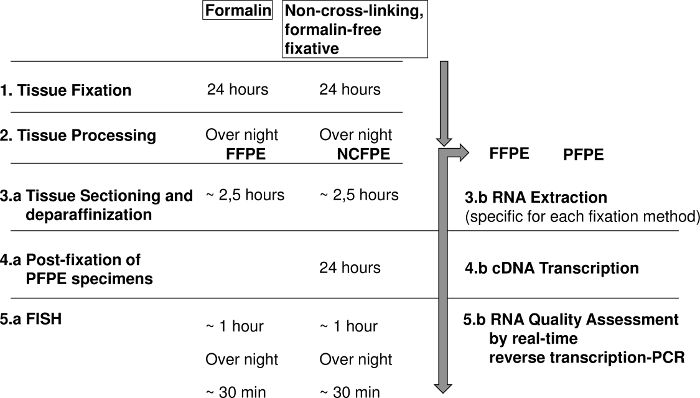
Abbildung 1: das Diagramm zeigt die Schritte zur Fluoreszenz in Situ Hybridisierung (FISH) und RNA Analyse von Formalin fixiert Paraffin eingebettet (FFPE) und nicht-Cross-linking, Formalin-freie feste Paraffin eingebetteten Gewebe (NCFPE). Bitte klicken Sie hier für eine größere Version dieser Figur.
Protocol
Representative Results
Discussion
Die Ergebnisse zeigen zwei wichtige Erkenntnisse. Erstens ist ein einfache 24 h SBFS Post-Fixierung Schritt Abschnitte geschnitten aus NCFPE Gewebe ausreichend zu gleichwertigen Ergebnissen mit Proteinkinase in Fisch Analysen mit Fisch-Assays für FFPE Gewebe zugelassen (siehe auch Hinweis14). Dieses Protokoll hat den Vorteil der Verwendung von Fisch-Assays ursprünglich entwickelt und zugelassen für Proteinkinase ohne die Notwendigkeit einer umfassenden Revalidierung (z. B. durch Optimierung der Pre-Hybridisierung Bedingungen für nicht-Cross-linked Gewebe). Die Fisch-Protokoll kann genau wie beschrieben in den Anweisungen des Herstellers verwendet werden.
Geringfügige Änderungen von den letztgültigen Anweisungen in diesem Protokoll (z. B. Gewebe Abschnitt Durchmesser und Inkubation Perioden 4.wenn di Spagna) Ergebnis aus vorherigen Bearbeitungen, die ausdrücklich vom Hersteller empfohlen wird durch den Einsatz von verschiedenen Gewebetypen und Fixierung Methoden.
Der Post-Fixierung Schritt ist entscheidend, da es eine chemische Reaktion Zeit von 18-24 h,, die die Analysezeit um einen Tag verlängert, sondern ermöglicht den gleichen täglichen Workflow erfordert wie für FFPE Gewebe (Abbildung 1) entwickelt. SBFS wird berichtet, dass Gewebe mit einer durchschnittlichen Rate von 1 mm pro Stunde22 , 5 mm in 2 h, je nach Gewebe Typ23,24eindringen. Die Beobachtung, dass nur Post-Fixierung längere mehr als 18 h FFPE-Abschnitte, die Eigenschaften des NCFPE ändern könnte weist darauf hin, dass nicht das Eindringen von das Fixiermittel, aber die chemische Reaktion Zeit entscheidend für die Erreichung der angestrebten1 ,14.
Zweitens kann das restliche NCFPE Gewebe, das nicht für Post-Fixierung dient für weitere molekulare Analysen verwendet werden, wie die Biomoleküle gut erhalten sind. Dies zeigte sich durch Echtzeit-reverse Transkription PCR (Abbildung 3). Der Test ist empfindlicher und spezifischer als die Electropherogram abgeleitet RIN Wert (RNA-Integrität-Nummer) im Hinblick auf RNS-Qualität (z. B. chemische Modifikation und Fragmentierung) für die mRNA isoliert von Paraffin-eingebetteten Gewebe8. Qualitätskontrolle war in dieser Studie auf Real-Time PCR beschränkt, da unabhängige Gruppen gezeigt haben, dass neben der RNA, die Qualität von DNA und Proteinen aus NCFPE isoliert besser als die von FFPE Gewebe 8,9, 13.
Das Protokoll präsentiert folgt, soweit anwendbar (z. B. SBFS Rezept, Fixierung Bedingungen, RNA-Qualitätssicherung, Validierung), die Anforderungen der CEN technische Spezifikationen für pre-analytische Verfahren zur in-vitro- Diagnostika (IVD), die wurden vor kurzem vom Europäischen Ausschuss der Normung (CEN) veröffentlicht (z. B. CEN/TS 16827-1:2015 für RNA-Isolation aus FFPE Gewebe bezieht sich auf die Norm ISO 15189).
Die Methode beschränkt sich auf diese spezifische Fixiermittel. Obwohl die gute Erhaltung der Biomoleküle und Morphologie mit nicht-Cross-linking, Formalin-freie Fixiermittel in mehreren Studien vorgestellt wurde, wird es hauptsächlich in der Forschung aber nicht in Routine medizinische Anwendungen verwendet. Ein Grund ist, dass anstelle der Goldstandard SBFS Fixierung durch ein weiteres Fixiermittel eine Vielzahl von Validierungsstudien erfordern würde, da nicht alle Protokolle für Proteinkinase Material optimiert für Materialien mit nicht-Cross-linking Fixiermittel fixiert verwendbar sind. Andere Gewebe Fixierung Methoden wurden für dieses Fisch-Protokoll nicht getestet.
Der einfachen Post-Fixierung Schritt beschrieben in dieser Handschrift hat den Vorteil der Verwendung genehmigt IVD-Assays für Proteinkinase und NCFPE Gewebe.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wir danken dem Team des Labors für Molekulare Pathologie am Institut für Pathologie der medizinischen Universität Graz für ihre Expertise und Unterstützung. Darüber hinaus danken wir Iris Kufferath und Daniela Pabst für technische Hilfe, Bernadette Rieger und Sylvia Eidenhammer für die Verarbeitung der HER2-Fällen sowie Kinga Szurian (Pathologe) und Tamas Regényi (3DHISTEC). Wir danken für das Manuskript Korrektur Penelope Kungl. Diese Arbeit hat von Christian Doppler Forschungsfonds, des österreichischen Bundesministeriums für Wissenschaft, Forschung und Wirtschaft und der Nationalstiftung für Forschung, Technologie und Entwicklung finanziell unterstützt.
Materials
| SAV LP GmbH, Flintsbach a. I., Germany | www.sav-lp.de | FN-200L-4-1 | Tissue fixative |
| Simport Plastic Ltd., Beloeil, Canada | www.simport.com | M-498 | Safekeeping device providing labelling, fixation and paraffin embedding for tissue samples. |
| Qiagen GmbH, Hilden Germany | www.qiagen.com | 765112 | For collection, fixation, and stabilization of tissue samples: 10 Prefilled Reagent Containers, containing PAXgene Tissue FIX and PAXgene Tissue STABILIZER. |
| ThermoFisher Scientific | www.thermofisher.com | 813150 | Processing of biological specimens from chemical fixation to paraffin infiltration |
| Merck Millipore, Munich, Germany | www.analytics-shop.com/de/hersteller/millipore.html | MC1009834000 | Dehydration of tissue samples |
| VWR Chemicals, Darmstadt, Germany | https://at.vwr.com | 10293EP | Dehydration of tissue samples |
| ACM Herba Chemosan Apotheker AG, Vienna, Austria | www.herba-chemosan.at | 2549662 32 | Paraffin used in a tissue processing device |
| Leica Mikrosysteme Handels GmbH, Vienna, Austria | www.LeicaBiosystems.com | 39602004 | Paraffin embedding medium |
| Sanova Diagnostik, Vienna, Austria | www.sanova.at | 5229 | Paraffin embedding instrument for tissues for histology |
| Histocom Medizintechnik Vertriebs GmbH, Wiener Neudorf, Austria | www.histocom.info | M 910010 | This device is used for especially sophisticated paraffin sectioning techniques in biology and medicine. Only skilled or specially trained personnel must operate the microtome, i.e. clamping the specimen, trimming, sectioning and taking off the sections from the instrument. |
| Dako Denmark A/S, Glostrup, Denmark | www.chem.agilent.com | K802021-2 | Coated glass slides, intended for mounting formalin-fixed, paraffin-embedded tissue sections. |
| Qiagen GmbH, Hilden Germany | www.qiagen.com | 73504 | For purification of total RNA from formalin-fixed, paraffin-embedded tissue sections |
| Qiagen GmbH, Hilden Germany | www.qiagen.com | 765134 | For purification of total RNA from PAXgene-fixed, paraffin-embedded tissue sections |
| ZytoVision GmbH, Bremerhaven, Germany | www.zytovision.com | Z-2015-200 | For the detection of ERBB2 (a.k.a HER2) gene amplification frequently observed in solid malignant neoplasms e.g. breast cancer samples. |
| 3DHISTEC, Budapest, Hungary | www.3dhistech.com | Digital device for scanning tissue slides equipped with a 40x/1.2 NA objective, Quad band filters (DAPI/FITC/TRITC/Cy5) filters and a 5.5Mpx, 16 bit, cooled scientific CMOS (Complementary Metal Oxide Semiconductor) camera. | |
| ThermoFisher Scientific | www.thermofisher.com | 4368814 | The High Capacity cDNA Reverse Transcription Kit contains all components necessary for the quantitative conversion of up to 2 µg of total RNA to single-stranded cDNA in a single 20 µL reaction. |
| ThermoFisher Scientific | www.thermofisher.com | 4367659 | PCR Master Mix containing polymerase, nucleotides and SYBR Green for PCR and quanitification of amplicons. |
| ThermoFisher Scientific, Wilmington, DE | www.thermofisher.com | Ser. Nr. F239 | Spectrophotometer for nucleic acid quantification. |
| QuantStudio 7 Flex Real-time PCR System | www.thermofisher.com | 4485701 | PCR machine, |
| Vysis/Abbott Laboratories. Abbott Park, Illinois, U.S.A | www.molecular.abbott/us/en/products/instrumentation/thermobrite#order | e.g. (ThermoBrite) 07J91-020 | Temperature controlled slide processing system for in-situ denaturation/hybridization procedures. |
References
- Fox, C. H., Johnson, F. B., Whiting, J., Roller, R. P. Formaldehyde fixation. J. Histochem.Cytochem. 33 (8), 845-853 (1985).
- Oosterhuis, J. W., Coebergh, J. W., van Veen, E. B. Tumour banks: well-guarded treasures in the interest of patients. Nat. Rev. Cancer. 3 (1), 73-77 (2003).
- Masuda, N., Ohnishi, T., Kawamoto, S., Monden, M., Okubo, K. Analysis of chemical modification of RNA from formalin-fixed samples and optimization of molecular biology applications for such samples. Nucleic Acids Res. 27 (22), 4436-4443 (1999).
- Metz, B., Kersten, G. F., Hoogerhout, P., Brugghe, H. F., Timmermans, H. A., de Jong, A., et al. Identification of formaldehyde-induced modifications in proteins: reactionswith model peptides. J. Biol. Chem. 279 (8), 6235-6243 (2003).
- Evers, D. L., Fowler, C. B., Cunningham, B. R., Mason, J. T., O’Leary, T. J. The effect of formaldehyde fixation on RNA: optimization of formaldehyde adduct removal. J.Mol. Diagn. 13 (3), 282-288 (2011).
- Groelz, D., Sobin, L., Branton, P., Compton, C., Wyrich, R., Rainen, L. Non-formalin fixative versus formalin-fixed tissue: A comparison of histology and RNA quality. Exp. Mol. Pathol. 94 (1), 188-194 (2013).
- Do, H., Dobrovic, A. Sequence artifacts in DNA from formalin-fixed tissues: causes and strategies for minimization. Clin. Chem. 61, 64-71 (2015).
- Kashofer, K., Viertler, C., Pichler, M., Zatloukal, K. Quality Control of RNA Preservation and Extraction from Paraffin-Embedded Tissue: Implications for RT-PCR and Microarray Analysis. PloS One. 8 (7), e70714 (2013).
- Viertler, C., Groelz, D., Gündisch, S., Kashofer, K., Reischauer, B., Riegman, P. H., et al. A new technology for stabilization of biomolecules in tissues for combined histological and molecular analyses. J. Mol. Diagn. 14 (5), 458-466 (2012).
- Gundisch, S., Slotta-Huspenina, J., Verderio, P., Maura, C., Ciniselli, S. P., Schott, S., et al. Evaluation of colon cancer histomorphology: A comparison between formalin and PAXgene tissue fixation by an international ring trial. Virchows Arch. 465 (5), 509-519 (2014).
- Ergin, B., Meding, S., Langer, R., Kap, M., Viertler, C., Schott, C., et al. Proteomic analysis of PAXgene-fixed tissues. J. Proteome Res. 9 (10), 5188-5196 (2010).
- Kap, M., Smedts, F., Oosterhuis, W., Winther, R., Christensen, N., Reischauer, B., et al. Histological Assessment of PAXgene Tissue Fixation and Stabilization Reagents. PLoS ONE. 6 (11), e27704 (2011).
- Mathieson, W., Marcon, N., Antunes, L., Ashford, D. A., Betsou, F., Frasquilho, S. G., et al. A Critical Evaluation of the PAXgene Tissue Fixation System: Morphology, Immunohistochemistry, Molecular Biology, and Proteomics. Am J Clin Pathol. 146, 25-40 (2016).
- Oberauner-Wappis, L., Loibner, M., Viertler, C., Groelz, D., Wyrich, R., Zatloukal, K. Protocol for HER2 FISH determination on PAXgene-fixed and paraffin-embedded tissue in breast cancer. Int J. Exp. Path. 97 (2), 202-206 (2016).
- Pauletti, G., Godolphin, W., Press, M. F., Slamon, D. J. Detection and quantitation ofHER-2/neu gene amplification in human breast cancer archival material using fluorescence in situ hybridization. Oncogene. 13 (1), 63-72 (1996).
- Owens, M. A., Horten, B. C., Da Silva, M. M. HER2 amplification ratios by fluorescence in situ hybridization and correlation with immunohistochemistry in a cohort of 6556 breast cancer tissues. Clin.Breast Cancer. 5 (1), 63-69 (2004).
- Wolff, A. C., Hammond, M. E., Schwartz, J. N., Hagerty, K. L., Allred, D. C., Cote, R. J., et al. American Society of Clinical Oncology/College of American Pathologists guideline recommendations for human epidermal growth factor receptor 2 testing in breast cancer. Arch Pathol Lab Med. 131 (1), 18-43 (2007).
- Press, M. F., Bernstein, L., Thomas, P. A., Meisner, L. F., Zhou, J. Y., Ma, Y., et al. HER-2/neu gene amplification characterized by fluorescence in situ hybridization: poor prognosis in node-negative breast carcinomas. J. Clin. Oncol. 15 (8), 2894-2904 (1997).
- Yamauchi, H., Stearns, V., Hayes, D. F. When is a tumor marker ready for prime time? A case study of c-erbB-2 as a predictive factor in breast cancer. J. Clin. Oncol. 19 (8), 2334-2356 (2001).
- Paulik, R., Micsik, T., Kiszler, G., Kaszál, P., Székely, J., Paulik, N., Várhalmi, E., Prémusz, V., Krenács, T., Molnár, B. An optimized image analysis algorithm for detecting nuclear signals in digital whole slides for histopathology. Cytometry A. , (2017).
- Applied Biosystems. . QuantStudio 6 and 7 Flex Real-Time PCR System Software. , (2013).
- NCCLS. . Quality Assurance for Immunocytochemistry. , (1999).
- Baker, J. R. . Principles of Biological Microtechnique. , (1958).
- Hewitt, S. M., Lewis, F., Cao, Y., Conrad, R. C., Cronin, M., Danenberg, K. D., et al. Tissue handling and specimen preparation in surgical pathology: issues concerning the recovery of nucleic acids from formalin-fixed, paraffin-embedded tissue. Arch Pathol. Lab Med. 132 (12), 1929-1935 (2008).

