Summary
यहां, हम फॉर्मेलिन-फिक्स्ड पैराफिन-एम्बेडेड ट्यूमर ऊतक वर्गों में प्रतिरक्षा कोशिकाओं की कल्पना, मात्रा और मानचित्रण के लिए एक सरल और सुलभ रणनीति का वर्णन करते हैं। यह पद्धति मौजूदा इमेजिंग और डिजिटल विश्लेषण तकनीकों को मल्टीप्लेक्सिंग क्षमता और इमेजिंग परखों के बहुमापदंडीय विश्लेषण के विस्तार के उद्देश्य से जोड़ती है।
Abstract
ट्यूमर माइक्रोएनवायरमेंट (TME) की प्रतिरक्षा परिदृश्य कैंसर की प्रगति और चिकित्सा के प्रति प्रतिक्रिया में एक निर्धारक कारक है। विशेष रूप से, टीएमई में प्रतिरक्षा कोशिकाओं के घनत्व और स्थान में महत्वपूर्ण नैदानिक और शकुन मूल्य हैं। TME की मल्टीोमिक प्रोफाइलिंग ने ट्यूमर दीक्षा और प्रगति को विनियमित करने वाले कई सेलुलर और आणविक नेटवर्क की हमारी समझ में तेजी से वृद्धि की है। हालांकि, ये तकनीक कोशिकाओं या सेल-सेल इंटरैक्शन के स्थानिक संगठन के बारे में जानकारी प्रदान नहीं करती है। एकल सेल-आधारित उच्च-थ्रूपुट प्रौद्योगिकियों के पूरक के लिए ऊतक वर्गों में प्रतिरक्षा कोशिकाओं के स्थानिक समाधान की अनुमति देने वाली मल्टीप्लेक्सिंग तकनीकों को निष्पादित करने में सस्ती, सुलभ और आसान। यहां, हम एक रणनीति का वर्णन करते हैं जो पूरे ऊतक वर्गों की आभासी मल्टीपैरामीटर स्लाइड उत्पन्न करने के लिए सीरियल इमेजिंग, अनुक्रमिक लेबलिंग और छवि संरेखण को एकीकृत करती है। आभासी स्लाइड बाद में उपयोगकर्ता-परिभाषित प्रोटोकॉल का उपयोग करके स्वचालित फैशन में विश्लेषण किया जाता है जो ब्याज की कोशिका आबादी की पहचान, मात्रा और मानचित्रण को सक्षम करता है। छवि विश्लेषण किया जाता है, इस मामले में विश्लेषण मॉड्यूल टिश्यूसंरेखित, लेखक और हिस्ताओमैप का उपयोग करके। हम एक उदाहरण प्रस्तुत करते हैं जहां हमने इस रणनीति को सफलतापूर्वक एक नैदानिक नमूने पर लागू किया, जो सीमित ऊतक नमूनों से प्राप्त की जा सकने वाली जानकारी को अधिकतम करता है और पूरे ऊतक अनुभाग में टीएमई का निष्पक्ष दृश्य प्रदान करता है।
Introduction
कैंसर विकास एक मल्टीस्टेप प्रक्रिया का परिणाम है जिसमें घातक कोशिकाओं और TME के बीच पारस्परिक बातचीत शामिल है। ट्यूमर कोशिकाओं के अलावा, TME गुटनिरपेक्ष कोशिकाओं, स्ट्रोमल कोशिकाओं, प्रतिरक्षा कोशिका आबादी, और एक्स्ट्रासेलुलर मैट्रिक्स (ईसीएम)1से बना है । ट्यूमर ऊतक के विभिन्न सेलुलर और संरचनात्मक घटकों का स्थानिक संगठन और कैंसर और पड़ोसी गैर - कैंसर कोशिकाओं के बीच गतिशील आदान - प्रदान अंततः ट्यूमर की प्रगति और चिकित्सा2,3,,4के प्रति प्रतिक्रिया को मिलाना । यह दर्शाया गया है कि कैंसर में प्रतिरक्षा प्रतिक्रिया 5,6कोविनियमित कर रही है । नियोप्लास्टिक घाव और आसन्न ऊतक में घुसपैठ करने वाली विभिन्न प्रतिरक्षा कोशिका आबादी विशिष्ट स्थानिक वितरण पैटर्न और विभिन्न सक्रियण और भेदभाव राज्यों को विभिन्न कार्यों (जैसे, समर्थक बनाम एंटीट्यूमर) से जुड़ी प्रदर्शित करती है। ये अलग-अलग प्रतिरक्षा आबादी और उनके पैरामीटर ट्यूमर और स्ट्रोमल डिब्बों के साथ ओवरटाइम को विकसित करते हैं।
एकल सेल मल्टीोमिक्स प्रोफाइलिंग की अनुमति देने वाली प्रौद्योगिकियों के उद्भव ने कैंसरजन्यऔर ट्यूमर प्रगति को विनियमित करने वाले कई सेलुलर और आणविक नेटवर्क ों की हमारी समझ को तेजी से बढ़ाया है। हालांकि, अधिकांश एकल सेल-आधारित उच्च-थ्रूपुट विश्लेषणात्मक उपकरणों को ऊतक व्यवधान और एकल सेल अलगाव की आवश्यकता होती है, जिसके परिणामस्वरूप कोशिकाओं के स्थानिक संगठन और सेल-सेल इंटरैक्शन7के बारे में जानकारी की हानि होती है। क्योंकि TME में विशिष्ट प्रतिरक्षा कोशिकाओं के स्थान और व्यवस्था में नैदानिक और शकुन मूल्य है, स्थानिक संकल्प की अनुमति देने वाली प्रौद्योगिकियां एकल कोशिका-आधारित प्रतिरक्षा प्रोफाइलिंग तकनीकों का एक अनिवार्य पूरक हैं।
परंपरागत रूप से, इम्यूनोहिस्टोकेमिस्ट्री (आईएचसी) और मल्टीप्लेक्स इम्यूनोफ्लोरेसेंस (एमआईएफ) जैसी इमेजिंग तकनीकों को कम संख्या में बायोमार्कर तक सीमित किया गया है जिन्हें एक साथ कल्पना की जा सकती है। इस सीमा ने ट्यूमर-घुसपैठ प्रतिरक्षा कोशिकाओं की स्थानिक गतिशीलता के अध्ययन में बाधा डाली है, जो आमतौर पर कई फेनोटाइपिक मार्कर द्वारा परिभाषित किए जाते हैं। इमेजिंग और एनालिटिकल टूल्स में हाल ही में हुई प्रगति ने मल्टीप्लेक्सिंग की संभावनाओं का विस्तार किया है। हिस्टो-साइटोमेट्री और इमेजिंग मास साइटोमेट्री जैसी नई एंटीबॉडी आधारित लेबलिंग प्रौद्योगिकियों का उपयोगक्रमशः8,9तक 12 और 32 बायोमार्कर को अलग करने के लिए किया गया है। मास स्पेक्ट्रोमेट्री इमेजिंग, लेबलिंग की आवश्यकता नहीं तकनीक, एक ऊतक अनुभाग10,,11में एक साथ हजारों बायोमार्कर छवि की क्षमता रखता है। हालांकि इन तकनीकों को पहले से ही कैंसर में ऊतक प्रतिरक्षा परिदृश्य विच्छेदन के लिए महान क्षमता दिखाई है, वे अत्यधिक परिष्कृत और महंगे उपकरण और सॉफ्टवेयर का उपयोग करें और शोधकर्ताओं के बहुमत के लिए आसानी से सुलभ नहीं हैं ।
वैकल्पिक रूप से, पारंपरिक आईएचसी और mIF की मल्टीप्लेक्सिंग क्षमता का विस्तार धारावाहिक इमेजिंग, लेबलिंग के अनुक्रमिक दौर और स्पेक्ट्रल इमेजिंग7,,12,,13,,14,,15,,16के उपयोग के माध्यम से किया गया है। ये तकनीकें उसी से या धारावाहिक ऊतक वर्गों से कई छवियां उत्पन्न करती हैं जिन्हें छवि विश्लेषण सॉफ्टवेयर का उपयोग करके आभासी मल्टीपैरामीटर स्लाइड में समेकित किया जा सकता है। नतीजतन, मार्कर की संख्या है कि कल्पना की जा सकती है और एक साथ विश्लेषण बढ़ जाती है ।
यहां, हम व्यावसायिक रूप से उपलब्ध रिएजेंट्स, किफायती माइक्रोस्कोपी उपकरण, और उपयोगकर्ता के अनुकूल सॉफ्टवेयर(चित्रा 1)का उपयोग करके ऊतक मल्टीप्लेक्स परखों के तर्कसंगत डिजाइन के लिए एक रणनीति का प्रस्ताव करते हैं। यह पद्धति वर्चुअल मल्टीपैरामीटर स्लाइड उत्पन्न करने के लिए सीरियल इमेजिंग, अनुक्रमिक मल्टीप्लेक्स लेबलिंग, पूरे टिश्यू इमेजिंग और ऊतक संरेखण को एकीकृत करती है जिसका उपयोग ऊतक वर्गों में प्रतिरक्षा कोशिकाओं के स्वचालित परिमाणीकरण और मानचित्रण के लिए किया जा सकता है। इस रणनीति का उपयोग करते हुए, हमने एक वर्चुअल स्लाइड बनाई जिसमें 11 बायोमार्कर प्लस दो अक्सर इस्तेमाल किए जाने वाले हिस्टोलॉजिकल दाग: हेमैटोक्सिलिन और इओसिन (एच एंड ई) और पिप्रोसिरियस रेड (पीएसआर) शामिल हैं। कई प्रतिरक्षा कोशिका आबादी की पहचान की गई, स्थित है, और विभिन्न ऊतक डिब्बों में मात्रा निर्धारित और उनके स्थानिक वितरण ऊतक हीटमैप ्स का उपयोग करहल किया। यह रणनीति उन जानकारी को अधिकतम करती है जिन्हें सीमित नैदानिक नमूनों से प्राप्त किया जा सकता है और पूरे ऊतक, कोर सुई बायोप्सी और ऊतक माइक्रोरेस सहित फॉर्मेलिन-फिक्स्ड पैराफिन-एम्बेडेड (एफएफपीई) संग्रहीत ऊतक नमूनों पर लागू होता है। हम TME में प्रतिरक्षा कोशिका आबादी की पहचान, मात्राकरण और मानचित्रण के लिए कस्टम परख डिजाइन करने के लिए एक उपयोगी गाइड के रूप में इस पद्धति का प्रस्ताव करते हैं।
Protocol
रीक्रिएट्री हेपेटाइटिस बी वायरस (एचबीवी) से जुड़े मानव हेपेटोसेलुलर कार्सिनोमा से तीन धारावाहिक एफएफपीई खंड केंद्र अस्पताल में भर्ती डी एल 'यूनीवर्सिट डी मॉन्ट्रियल (CHUM) हेपाटोपैनेओबिओबिलेरी कैंसर नैदानिक डेटाबेस और जैविक नमूना से प्राप्त किए गए थे भंडार (एचबीपी बायोबैंक) । इस टिश्यू बैंक में भाग लेने वाले रोगियों ने सूचित सहमति प्रदान की । इस अध्ययन को संस्थागत आचार समिति (प्रोटोकॉल संख्या 09.237) द्वारा अनुमोदित किया गया था और हेलसिंकी की घोषणा के अनुसार प्रदर्शन किया गया था।
1. हेमातक्सीलिन और इओसिन (एच एंड ई) धुंधला प्रोटोकॉल
नोट: एच एंड ई धुंधला केंद्र डी रेचेचेस डु सेंटर अस्पताल डी एल 'यूनीवर्सिट डी मॉन्ट्रियल (सीआरक्यूयूएम) की आणविक पैथोलॉजी कोर सुविधा द्वारा निम्नलिखित कार्यक्रम का उपयोग करके शंडन मल्टीप्रोग्राम रोबोटिक स्लाइड स्टेनर का उपयोग करके किया गया था।
- deparaffinization के लिए, जाइलीन विकल्प में प्रत्येक 2.5 मिन के लिए स्लाइड 3x विसर्जित कर देता है।
सावधानी: जाइलीन विकल्प ज्वलनशील, त्वचा की परेशानी, और हानिकारक अगर साँस रहे हैं । - रिहाइड्रेशन के लिए, 2.5 मिन प्रत्येक के लिए 100% इथेनॉल 3x में स्लाइड विसर्जित करें। डबल आसुत पानी (डीडीएच2ओ) में 1 मिन के लिए धोकर फिर से हाइड्रेट करें।
- हेमैटोक्सिलिन में 1 मिन के लिए इनक्यूबेट। डीडीएच2ओ में प्रत्येक में 1 मिन के लिए 3x धोएं।
- eosin के साथ 5 एस के लिए इनक्यूबेट। 95% इथेनॉल के साथ 30 एस धोएं। 100% इथेनॉल के साथ 1 मिन के लिए 2x धोएं।
सावधानी: इथेनॉल ज्वलनशील और एक आंख अड़चन है। Eosin एक आंख अड़चन है। - निर्जलीकरण के लिए, जाइलीन विकल्प में प्रत्येक 1.5 मिन के लिए 3x विसर्जित करें। मैन्युअल रूप से स्लाइड माउंट।
नोट: प्रोटोकॉल के इस हिस्से को निष्पादित करने के लिए अनुमानित समय 30 min है।
2. एफएफपीई वर्गों के लिए मल्टीप्लेक्स इम्यूनोफ्लोरेसेंस धुंधला प्रोटोकॉल
नोट: इस प्रोटोकॉल रॉबर्टसन एट अल17से अनुकूलित किया गया था ।
- डिपरफिनाइजेशन और रिहाइड्रेशन
नोट: आईएचसी या mIF द्वारा एफएफपीई अनुभागों की एंटीबॉडी-मध्यस्थता लेबलिंग से पहले, पैराफिन को हटा दिया जाना चाहिए। पैराफिन को कुशलतापूर्वक हटाने में विफलता के परिणामस्वरूप सबऑप्टिमल धुंधला हो जाता है।- ग्लास स्लाइड धारकों में 4 μm FFPE ऊतक अनुभाग स्लाइड रखें। धूम हुड के तहत, 10 मिन के लिए 37 डिग्री सेल्सियस प्रीवार्म्ड जाइलीन युक्त एक कोप्लिन जार में स्लाइड विसर्जित करें।
सावधानी: जाइलीन ज्वलनशील है, एक त्वचा अड़चन है, और हानिकारक अगर साँस । - 10 एस हर 2 मिन के लिए मैन्युअल रूप से आंदोलन। 5 मिन के लिए ताजा जाइलीन में 1x दोहराएं।
- रासायनिक हुड में, निम्नलिखित समाधानों में से प्रत्येक में 5 मिन के लिए क्रमिक रूप से स्लाइड को विसर्जित करें: 1) जाइलीन: इथेनॉल (1:1 v/v); 2) 100% इथेनॉल; 3) 70% इथेनॉल; 4) 50% इथेनॉल; 5) 30% इथेनॉल; 6) फॉस्फेट-बफरेड लवकुश (पीबीएस)।
नोट: पीबीएस में स्लाइड्स रखें जब तक एंटीजन रिट्रीवल करने के लिए तैयार न हो जाए। डिलक ्ड सेक्शन को हर समय हाइड्रेटेड रखें। बाहर सुखाने गैर विशिष्ट एंटीबॉडी बाध्यकारी और इसलिए उच्च पृष्ठभूमि धुंधला कारण होगा ।
- ग्लास स्लाइड धारकों में 4 μm FFPE ऊतक अनुभाग स्लाइड रखें। धूम हुड के तहत, 10 मिन के लिए 37 डिग्री सेल्सियस प्रीवार्म्ड जाइलीन युक्त एक कोप्लिन जार में स्लाइड विसर्जित करें।
- गर्मी से प्रेरित एंटीजन रिट्रीवल
नोट: एंटीजन को फॉर्मेलिन-फिक्सेशन पर नकाबपोश किया जा सकता है, एंटीबॉडी बाइंडिंग को रोकता है और फलस्वरूप दृश्य। एंटीजन अनमास्किंग बफ़र्स और प्रक्रियाओं का उपयोग आंशिक रूप से एपिटोप के मूल संरचना को फिर से स्थापित करता है और इस तरह एंटीबॉडी मान्यता को पुनर्स्थापित करता है। एंटीजन पुनर्प्राप्ति बफर और अवधि के प्रकार विशिष्ट परख शर्तों (जैसे, लक्ष्य, एंटीबॉडी, ऊतक, आदि) के लिए अनुकूलित किया जाना चाहिए।- एंटीजन रिट्रीवल सॉल्यूशन (सामग्री की तालिकामें नुस्खा) युक्त एक कोप्लिन जार में डिलक्टेड स्लाइड विसर्जित करें।
- नल के पानी के साथ एक बिजली के प्रेशर कुकर में बंद Coplin जार जगह है। जल स्तर जार की आधी ऊंचाई से अधिक नहीं होना चाहिए ताकि पानी एंटीजन पुनर्प्राप्ति समाधान के साथ मिश्रण न हो।
- कुकर के ढक्कन और प्रेशर वाल्व को बंद करें। 10 मिन के लिए उच्च दबाव का चयन करें और शुरू करें। जब किया जाता है, तो कुकर को अनप्लग करें, दबाव छोड़ें, ढक्कन खोलें, और जार को 30 min के लिए कुकर के अंदर रखें, जिससे स्लाइड को ठंडा होने की अनुमति हो।
- गैर विशिष्ट बाध्यकारी को अवरुद्ध करना
- स्लाइड्स के साथ रैक को पीबीएस से भरे कोप्लिन जार में ट्रांसफर करें। प्रत्येक 5 मिन के लिए पीबीएस 2x के साथ एंटीजन पुनर्प्राप्ति बफर को कुल्ला करें।
- हाइड्रोफोबिक बैरियर बनाने के लिए ओपीपी पेन के साथ ऊतक वर्गों को घेरना। स्लाइड्स में विसर्जित करें पीबीएस में 0.1 एम ग्लाइसिन वाले कोप्लिन जार। कमरे के तापमान (आरटी) पर 15 न्यूनतम के लिए इनक्यूबेट।
नोट: ग्लाइसिन एंटीजन रिट्रीवल के दौरान उत्पन्न एल्डिहाइड समूहों को संतृप्त करता है। ये समूह प्राथमिक और माध्यमिक एंटीबॉडी को अविशेष रूप से बांध सकते थे । - 5 मिन के लिए पीबीएस के साथ 2x धोकर ग्लाइसिन समाधान को कुल्ला करें। स्लाइडको आर्द्रता कक्ष में रखें और सभी ऊतक वर्गों को कवर करने के लिए पर्याप्त अवरुद्ध समाधान जोड़ें। हाइड्रोफोबिक बैरियर को बहने से बचें। आरटी में 30 मिन के लिए इनक्यूबेट ।
नोट: अवरुद्ध समाधान के लिए नुस्खा सामग्री की मेजमें पाया जा सकता है । अवरुद्ध समाधान में गैर-विशिष्ट बाध्यकारी साइटों को अवरुद्ध करने के लिए एक प्रोटीन (जैसे, बीएसए) होना चाहिए। यह ट्राइटन एक्स-१०० या ट्वीन 20 जैसे डिटर्जेंट को भी शामिल कर सकता है जो एंटीबॉडी और टिश्यू टारगेट के बीच हाइड्रोफोबिक इंटरैक्शन को कम करते हैं, जिससे एंटीजन रिकग्निशन को ज्यादा चयनात्मक बनाया जा सकता है । प्रजातियों से 10% कुल सीरम के अलावा जहां ऊतक से आता है एफसी रिसेप्टर्स ब्लॉक होगा, और इस तरह गैर विशिष्ट एंटीबॉडी बाध्यकारी कम । अंत में, प्रजातियों से सीरम के 10% के अलावा माध्यमिक एंटीबॉडी में उठाया गया था ऊतक अनुभाग के लिए माध्यमिक एंटीबॉडी के प्रत्यक्ष गैर विशिष्ट लगाव को कम करेगा ।
- इम्यूनोफ्लोरेसेंस लेबलिंग
- 5 मिन के लिए पीबीएस-ट्वीन (0.1% v/v) 2x के साथ कुल्ला करें और स्लाइडको वापस आर्द्रता कक्ष में रखें।
- समाधान को अवरुद्ध करने में पुनर्निलंबित प्राथमिक एंटीबॉडी के कॉकटेल जोड़ें। रात भर 4 डिग्री सेल्सियस पर इनक्यूबेट। इस अध्ययन के लिए उपयोग किए जाने वाले प्राथमिक और माध्यमिक एंटीबॉडी सामग्री तालिकामें सूचीबद्ध हैं।
नोट: प्राथमिक एंटीबॉडी के कॉकटेल में या तो विभिन्न प्रजातियों में उठाए गए एंटीबॉडी, या एक ही प्रजाति से बल्कि विभिन्न आइसोटाइप शामिल होने चाहिए। इस अध्ययन परामर्श तालिका 2में उपयोग किए जाने वाले प्राथमिक-माध्यमिक एंटीबॉडी जोड़े की सूची के लिए। उपयोग किए जाने वाले सभी एंटीबॉडी का विवरण सामग्री और तालिका 2की तालिका में है। - 5 मिन के लिए पीबीएस-ट्वीन (0.1% v/v) 3x के साथ कुल्ला करें और स्लाइडको वापस आर्द्रता कक्ष में रखें। अंधेरे में, माध्यमिक एंटीबॉडी का कॉकटेल जोड़ें और आरटी में 1 घंटे के लिए इनक्यूबेट करें।
नोट: जब प्राथमिक एंटीबॉडी विभिन्न प्रजातियों से हैं, तो माध्यमिक एंटीबॉडी का चयन किया जाना चाहिए ताकि उनमें से प्रत्येक केवल प्राथमिक एंटीबॉडी में से एक को बांधे और एक-दूसरे के लिए नहीं। यह आमतौर पर माध्यमिक एंटीबॉडी का उपयोग करके एक ही प्रजाति में उठाया जाता है जब तक कि यह प्रजाति उन प्रजातियों से अलग होती है जहां प्राथमिक एंटीबॉडी उत्पन्न होते थे। ऐसे मामलों में जहां प्राथमिक एंटीबॉडी एक ही प्रजाति में उठाए गए थे लेकिन अलग-अलग आइसोटाइप होते हैं, आइसोटाइप-विशिष्ट माध्यमिक एंटीबॉडी का उपयोग किया जाना चाहिए। - प्रत्येक 5 मिन के लिए पीबीएस-ट्वीन (0.1% v/v) 3x के साथ कुल्ला करें। DDH2O. अतिरिक्त तरल निकालें और DAPI के साथ बढ़ते मीडिया में माउंट के साथ कुल्ला। उपयोग की गई मात्रा अनुभाग के आकार पर निर्भर करती है। आमतौर पर 40 μL एक नियमित माइक्रोस्कोपी स्लाइड की सतह को कवर करने के लिए पर्याप्त है।
- अनुभाग पर कवर स्लाइड रखें और धीरे से बुलबुला गठन से बचने के अतिरिक्त बढ़ते मीडिया निचोड़। स्लाइड्स में 20 मिन के लिए अंधेरे में आरटी में स्टोर करें और अधिग्रहण के लिए तैयार होने तक 4 डिग्री सेल्सियस पर स्टोर करें ।
- पूरी स्लाइड स्कैनर का उपयोग कर सभी चैनलों के लिए छवियां प्राप्त (सामग्री की तालिकादेखें) ।
नोट: एंटीबॉडी को सकारात्मक नियंत्रण के रूप में मानव हेपेटोसेलुलर कार्सिनोमा ऊतक का उपयोग करके मान्य किया गया था। प्रत्येक प्राथमिक एंटीबॉडी के लिए, तीन धारावाहिक वर्गों को या तो प्राथमिक एंटीबॉडी, आइसोटाइप नियंत्रण, या केवल अवरुद्ध समाधान के साथ क्रमशः दाग दिया गया था जिसमें बाकी धुंधला प्रोटोकॉल में कोई भिन्नता नहीं थी। अधिग्रहीत छवियों की तुलना धुंधला की विशिष्टता स्थापित करने के लिए की गई थी । धुंधला विशिष्ट माना जाता था जब प्राथमिक एंटीबॉडी के साथ इनक्यूबेटेड अनुभाग में संकेत की उम्मीद पैटर्न था और पृष्ठभूमि से आसानी से अलग था। आइसोटाइप में उच्च पृष्ठभूमि संकेत या लेबलिंग ऊतक घटक देने वाले प्राथमिक एंटीबॉडी और कोई प्राथमिक एंटीबॉडी वर्गों को गैर-विशिष्ट नहीं माना जाता था। प्रोटोकॉल के इस हिस्से को पूरा करने का अनुमानित समय 2 दिन है। आवश्यक नियंत्रणों में शामिल हैं: (1) पृष्ठभूमि संकेत के लिए प्राथमिक एंटीबॉडी के गैर-विशिष्ट बाध्यकारी के योगदान को स्थापित करने के लिए आइसोटाइप नियंत्रण। एक खंड को उसी तरह दाग दिया जाता है जैसे अन्य नमूना ऊतकों को छोड़कर कि यह एक ही आइसोटाइप और प्राथमिक एंटीबॉडी की उत्पत्ति के साथ एक एंटीबॉडी के साथ इनक्यूबेटेड है लेकिन ऊतक अनुभाग में अनुपस्थित लक्ष्य के लिए विशिष्ट है। यदि उपयुक्त आइसोटाइप नियंत्रण एंटीबॉडी उपलब्ध नहीं है, तो इसे उसी प्रजाति से कुल आईजीजी द्वारा प्रतिस्थापित किया जा सकता है जहां प्राथमिक एंटीबॉडी को उठाया गया था; (2) धुंधला की विशिष्टता स्थापित करने और पृष्ठभूमि संकेत के लिए माध्यमिक एंटीबॉडी के गैर-विशिष्ट बाध्यकारी के योगदान का अनुमान लगाने के लिए कोई प्राथमिक एंटीबॉडी नियंत्रण (यानी नकारात्मक नियंत्रण) नहीं । इस मामले में, नियंत्रण अनुभाग को उसी तरह दाग दिया जाता है जैसे अन्य अनुभागों को छोड़कर कोई प्राथमिक एंटीबॉडी नहीं जोड़ा जाता है; (3) यह स्थापित करने के लिए सकारात्मक नियंत्रण कि धुंधला काम करता है। इस मामले में, धुंधला एक ऊतक अनुभाग है कि प्राथमिक एंटीबॉडी द्वारा मांयता प्राप्त मार्कर व्यक्त करने के लिए जाना जाता है पर किया जाता है ।
3. पिरो-सीरियस रेड (पीएसआर)/फास्ट ग्रीन धुंधला प्रोटोकॉल
नोट: इस धुंधला का लक्ष्य एफएफपीई ऊतक वर्गों में फिब्रिलर कोलेजन I और III की कल्पना करना है। इस प्रोटोकॉल को सेग्नानी एट अल18से अनुकूलित किया गया था। सभी कदम एक रासायनिक हुड में किया जाता है।
- एफएफपीई वर्गों (धारा 2.1) के लिए मल्टीप्लेक्स इम्यूनोफ्लोरेसेंस धुंधला प्रोटोकॉल के समान ऊतक वर्गों के अपपैरीकरण और रिहाइड्रेशन का प्रदर्शन करें।
नोट: यदि दाग होने वाले अनुभाग का उपयोग पहले इम्यूनोफ्लोरेसेंस लेबलिंग के लिए किया गया है और पैराफिन को पहले ही हटा दिया गया है, तो बढ़ते मीडिया को हटाने के लिए अपाफिनीकरण-रिहाइड्रेशन कदम उपयोगी हैं। DAPI इस प्रक्रिया का उपयोग कर हटा नहीं है, लेकिन यह कथित रूप से पीएसआर धुंधला के साथ हस्तक्षेप नहीं करता है । - स्लाइड्स में विसर्जित करें जिसमें पिट्रो-सीरियस रेड/फास्ट ग्रीन सॉल्यूशन (टेबल ऑफ मैटेरियल)युक्त जार में और आरटी में 30 मिन के लिए इनक्यूबेट (हेपेटोसाइट्स के नाभिक के गैर-विशिष्ट धुंधला होने के परिणामस्वरूप 30 से अधिक) ।
- डीडीएच2ओ (5 डिप्स) में स्लाइड्स जल्दी धोएं। फिर, इथेनॉल 100% (5 डिप्स) में जल्दी धो लें। जाइलीन-100% इथेनॉल (1:1 v/v) में 30 एस के लिए धोएं। जाइलीन में 30 एस के लिए धोलें। बढ़ते मीडिया के साथ माउंट (सामग्री की तालिकादेखें) इससे पहले कि जाइलीन पूरी तरह से सुखाया गया है (यह बढ़ते के साथ मदद करता है) ।
नोट: प्रोटोकॉल के इस हिस्से को निष्पादित करने के लिए अनुमानित समय 1 घंटे है।
4. ऊतक वर्गों से एंटीबॉडी का एल्यूशन
नोट: अनुक्रमिक लेबलिंग परखों में ऊतक वर्गों का पुन: उपयोग करने के लिए, प्राथमिक और माध्यमिक एंटीबॉडी को पूरी तरह से हटाने की आवश्यकता है। बाउंड एंटीबॉडी को पहले13वर्णित के रूप में छीन लिया गया था ।
पानी के स्नान को 56 डिग्री सेल्सियस तक पहले से गरम करें। एक जार के अंदर वर्गों को अलग करना बफर (सामग्री की मेजमें नुस्खा), ढक्कन बंद करो, और मिलाते हुए के दौरान लीक को रोकने के लिए पैराफिन फिल्म टेप के साथ सील ।
- जार को पानी के स्नान के अंदर रखो और आंदोलन के साथ 30 मिन के लिए इनक्यूबेट करें।
- आरटी में डीडीएच2ओ में 15 मिन के लिए 4x धोएं । PBS-ट्वीन (०.१% v/v) के साथ कुल्ला ।
- प्राथमिक एंटीबॉडी के दूसरे दौर के साथ अनुभाग की फिर से जांच करने के लिए तैयार होने तक पीबीएस-ट्वीन या पानी में हाइड्रेटेड अनुभागों को रखें।
नोट: प्रोटोकॉल के इस हिस्से को निष्पादित करने के लिए अनुमानित समय 2 घंटे है। - एंटीबॉडी एल्यूशन प्रक्रिया की दक्षता सत्यापित करें।
नोट: एक अनुक्रमिक लेबलिंग परख में एंटीबॉडी एल्यूशन के लिए प्रोटोकॉल का उपयोग करने से पहले, प्राथमिक और माध्यमिक एंटीबॉडी को हटाने की दक्षता सत्यापित की जानी चाहिए।- एफएफपीई वर्गों (वर्ग 2.1-2.4.6) के लिए मल्टीप्लेक्स इम्यूनोफ्लोरेसेंस धुंधला प्रोटोकॉल में इंगित ब्याज की एक दी गई प्राथमिक-माध्यमिक एंटीबॉडी जोड़ी के साथ एक अनुभाग के धुंधला और छवि अधिग्रहण करें।
- छवि अधिग्रहण पर, ऊतक-बाध्य प्राथमिक-माध्यमिक एंटीबॉडी परिसरों का एल्यूशन करें जैसा कि धाराओं 4.1-4.3 में इंगित किया गया है।
- एक ही माध्यमिक एंटीबॉडी और एक ही चरण 2.4.3 में इस्तेमाल शर्तों के साथ अनुभाग इनक्यूबेट।
- 2.4.4-2.4.6 में संकेत के रूप में धोने, बढ़ते, और छवि अधिग्रहण कदम प्रदर्शन करते हैं।
- अलग करने से पहले और बाद में अधिग्रहीत साइड इमेज से तुलना करें ताकि यह स्थापित किया जा सके कि विशिष्ट संकेत गायब हो गया है या नहीं।
नोट: एंटीबॉडी हटाने से पहले और बाद में छवियों की तुलना एल्यूशन प्रक्रिया की दक्षता को मान्य करेगी। हालांकि, सभी चैनलों में बैकग्राउंड सिग्नल में वृद्धि देखने के साथ-साथ डीपीआई का प्रसार देखना सामान्य बात है। यह अलग करना है कि एक ही ऊतक अनुभाग पर निष्पादित किया जा सकता है के दौर की संख्या सीमा । स्ट्रिपिंग के तीन राउंड अधिकतम लगते हैं ।
5. छवि अधिग्रहण
- एक पूरी स्लाइड स्कैनर का उपयोग कर छवियां उत्पन्न करें।
- 20x 0.75 NA ऑब्जेक्टिव लेंस और 0.3225 माइक्रोन/पिक्सल का रेजोल्यूशन का इस्तेमाल करें ।
6. छवि विश्लेषण
नोट: यहां उल्लिखित विधि वर्तमान उदाहरण को संदर्भित करती है। कृपया टेबल 1 और पाठ को अन्य विशिष्ट नमूनों के अनुकूल करने के लिए देखें।
- छवि विश्लेषण सॉफ्टवेयर के टिसुसंरेखित मॉड्यूल का उपयोग करके ऊतक संरेखण करें (इस प्रोटोकॉल में विज़, सामग्री की तालिकादेखें)।
- इमेज एनालिसिस सॉफ्टवेयर खोलें और टिश्यूऑर्ड टैब पर क्लिक करें।
- फाइल करने के लिए जा रहा द्वारा स्लाइड ट्रे में गठबंधन किया जा करने के लिए छवियों का आयात करें । डेटाबेस और गठबंधन करने के लिए पहली छवि का चयन करें। टिश्यूऑर्ड टैब पर वापस जाएं और स्लाइड ट्रेमें लोड बटन पर क्लिक करके इमेज लोड करें । छवि स्लाइड ट्रे में और कार्यक्षेत्र में दिखाई देगा।
नोट: केवल ब्याज के ढेर स्लाइड ट्रे में लोड किया जाना चाहिए । - गठबंधन करने के क्रम में सभी छवियों के लिए चरण 6.1.2 दोहराएं, उन्हें एक-एक करके लोड करें। एक बार ब्याज की सभी छवियों को स्लाइड ट्रे पर लोड कर रहे हैं रिबन में वर्कफ्लो कदम में अगले दबाने के द्वारा छवियों को जोड़ने के लिए आगे बढ़ना।
- इसके बाद, पहली छवि के शीर्ष पर दूसरी छवि खींचें और छोड़ दें। पहली और दूसरी छवियां अब जुड़े हुए हैं । अन्य छवियों को व्यवस्थित तरीके से एक-एक करके गठबंधन करने के लिए इस चरण को दोहराएं। पहली छवि का नाम बदल जाएगा, यह दर्शाता है कि इसे अन्य छवियों से जोड़ा गया है। इसके साथ ही, लिंक किए गए चित्र स्लाइड ट्रे के दाईं ओर कार्यक्षेत्र में प्रदर्शित किए जाएंगे।
- इस बिंदु पर, छवियों को या तो स्वचालित संरेखण, अर्धस्वचालित संरेखण, या मैनुअल संरेखण का उपयोग करके संरेखित करें। पहले स्वचालित संरेखण की कोशिश करना हमेशा बेहतर होता है। स्वचालित संरेखण के लिए रिबन में वर्कफ़्लो चरण (चरण 3) में अगला बटन दबाएं।
- ऊतक के विभिन्न स्थानों को नेविगेट करके स्वचालित संरेखण की समीक्षा करें और नेत्रहीन यह सत्यापित करें कि छवि के दो आयामों में विभिन्न छवियों में संबंधित संरचनाओं को उसी तरह व्यवस्थित किया जाता है।
- यदि स्वचालित संरेखण का परिणाम संतोषजनक नहीं है, तो लिंक किए गए चित्रों में निकटतम ऊतक सुविधाओं का संकेत देते हुए पिन (प्रति छवि कम से कम तीन पिन का उपयोग करें) का उपयोग करके इसे बेहतर करें। लिंक्ड छवियों में निकटतम स्थानों पर पिन रखे जाने के बाद, उपयोगकर्ता के पास दो विकल्प होते हैं: अर्धस्वचालित संरेखण या मैनुअल संरेखण। अर्धस्वचालित संरेखण के लिए रिबन में वर्तमान पिनपॉइंट्स के आधार पर बटन ऑटो-संरेखित पर क्लिक करें। मैन्युअल अलाइनमेंट के लिए बटन पर क्लिक करें रिबन पर पिन लगाएं।
- जब संरेखण से संतुष्ट होकर कार्यप्रवाह चरणों में अगले बटन पर क्लिक करें और डेटाबेस में समग्र छवि को सहेजें।
नोट: 11 मार्कर प्लस एच एंड ई और पीएसआर छवियों में फैले छह स्लाइड संरेखित प्रस्तुत विश्लेषण में 15 min लिया ।
- उपयोगकर्ता-परिभाषित प्रोटोकॉल विश्लेषण प्रोटोकॉल पैकेज 1 (एपीपी 1, तालिका 1)का उपयोग करके ऊतक का पता लगाने का प्रदर्शन करें।
- रिबन में इमेज एनालिसिस टैब पर क्लिक करके सॉफ्टवेयर का इमेज एनालिसिस मॉड्यूल खोलें।
- फाइल करने के लिए जा रहा द्वारा समग्र (गठबंधन) छवि आयात करें । डेटाबेस और रुचि की छवि का चयन और छवि विश्लेषण टैब वापस क्लिक।
- ओपन ऐप आइकन पर क्लिक करके ऐप चयन संवाद खोलें और उपयोग करने के लिए कौन सा विश्लेषण प्रोटोकॉल पैकेज (एपीपी) चुनें। इस मामले में ऊतक का पता लगाने के लिए एपीपी 1 का चयन करें।
- एक बार APP 1 खोल दिया जाता है, पुष्टि करें कि APP1 एक चयनित ऊतक स्थान पर जाकर और पूर्वावलोकन बटन पर क्लिक करके ठीक से काम कर रहा है । यदि परिणाम संतोषजनक हैं, तो अगले चरण में जाएं।
- APP 1 चलाने के लिए क्लिक करें और चयनित APP का उपयोग कर छवि प्रक्रिया ।
- जब विश्लेषण फ़ाइल/निर्यात पर क्लिक करके किया जाता है, तो डेटा (जैसे, छवियां, माप, आदि) का निर्यात करें।
नोट: APP 1 ऊतक (आरओआई ऊतक) को डिलिनिंग करने वाला ब्याज (आरओआई) बनाता है और ऊतक के क्षेत्र की गणना करता है। - फ़ाइल करने के लिए जा रहा द्वारा नव निर्मित आरओआई के साथ संशोधित छवि को बचाओ । बचाओ।
नोट: ऊतक का पता लगाने और प्रदान किए गए उदाहरण में APP 1 के साथ एक आरओआई बनाने के लिए वर्णित छवि विश्लेषण स्टेशन में 5 मिन लिया। ऊतक संसाधित का क्षेत्र 3.2 सेमी2था।
- एपीपी 2(टेबल 1)का उपयोग करके स्ट्रोमा और परेंचिमा में ऊतक विभाजन करें।
नोट: एपीपी 2 पूर्वनिर्धारित आरओआई ऊतक पर काम करता है। एपीपी 2 ऊतकों को आरओआई स्ट्रोमा और परेंचिमा में खंडित करता है।- रिबन में इमेज एनालिसिस टैब पर क्लिक करके इमेज एनालिसिस मॉड्यूल खोलें।
- फाइल करने के लिए जा रहा द्वारा आरओआई ऊतक युक्त छवि आयात करें । डेटाबेस और चरण 6.2.7 में सहेजी गई छवि का चयन। इमेज एनालिसिस टैब पर वापस जाएं और स्लाइड ट्रेमें लोड बटन पर क्लिक करके इमेज लोड करें । छवि स्लाइड ट्रे में और कार्यक्षेत्र में दिखाई देगा।
- 6.2.3 में ऐप चयन संवाद का उपयोग करके ऐप 2 खोलें।
- व्यू के एक चयनित क्षेत्र में प्रसंस्करण द्वारा एपीपी 2 का पूर्वावलोकन करें। यदि परिणाम संतोषजनक हैं, तो रन बटन पर क्लिक करके पूरी छवि पर ऐप 2 चलाएं। एपीपी 2 के आउटपुट के रूप में, आरओआई ऊतक को आरओआई स्ट्रोमा और परेंचिमा और उनके संबंधित क्षेत्रों में विभाजित किया गया है। निर्यात परिणाम 6.2.6 में। संशोधित छवि को 6.2.7 में सहेजें।
नोट: एपीपी 2 का उपयोग करस्ट्रोमा और परेंचिमा में ऊतक को विभाजित करने से प्रस्तुत विश्लेषण स्टेशन में 4 घंटे लग गए। ऊतक संसाधित का क्षेत्र 3.2 सेमी2था।
- उपयोगकर्ता-परिभाषित प्रोटोकॉल ऐप 3(तालिका 1)का उपयोग करके FoxP3हायCD4 + कोशिकाओं की पहचान करें और मात्रा निर्धारित करें।
नोट: एपीपी 3 पूर्वनिर्धारित आरओआई स्ट्रोमा और परेंचिमा पर काम करता है।- इमेज एनालिसिस मॉड्यूल खोलें और 6.3.1 और 6.3.2 में आरओआई स्ट्रोमा और परेंचिमा वाली छवि का आयात करते हैं। 6.2.3 में ऐप चयन संवाद का उपयोग करके ऐप 3 खोलें।
- पूर्वावलोकन APP 3 संसाधन FoxP3हायCD4 + कोशिकाओं में समृद्ध दृश्य के एक चयनित क्षेत्र में। यदि परिणाम संतोषजनक हैं, तो पूरी छवि पर ऐप 3 चलाएं। एपीपी 3 के आउटपुट के रूप में, सभी व्यक्तिगत FoxP3हायCD4 + वस्तुओं को लेबल किया जाएगा और उनके ऊतक संग्रहीत निर्देशांक। आरओआई स्ट्रोमा और परेंचिमा में FoxP3हायसीडी4 + वस्तुओं के घनत्व का निर्धारण किया जाएगा। परिणाम 6.2.6 में निर्यात करें।
- FoxP3हायCD4 + लेबल वस्तुओं के ऊतक हीटमैपिंग प्रदर्शन करें।
- 6.2.3 में ऐप चयन संवाद का उपयोग करके उपयोगकर्ता-परिभाषित प्रोटोकॉल FoxP3hiCD4 + मानचित्र खोलें।
नोट: FoxP3हायCD4 + MAP घनत्व हीटमैप पैदा करने के लिए FoxP3हायCD4+ लेबल वाली वस्तुओं के निर्देशांक का उपयोग करता है। APP 3 का उपयोग कर FoxP3हायCD4 + लेबल वस्तुओं की पहचान करने और गिनती छवि विश्लेषण स्टेशन में 25 min लिया वर्णित है । ऊतक संसाधित का क्षेत्र 3.2 सेमी2था। - रन बटन दबाकर FoxP3हायCD4+ MAP चलाएं। फ़ाइल पर क्लिक करके ऊतक हीटमैप निर्यात करें । निर्यात । कार्य क्षेत्र।
नोट: FoxP3हायCD4 + लेबल वस्तुओं का मानचित्रण FoxP3हायCD4 + मानचित्र का उपयोग कर के छवि विश्लेषण स्टेशन में 5 मिन लिया वर्णित है ।
- 6.2.3 में ऐप चयन संवाद का उपयोग करके उपयोगकर्ता-परिभाषित प्रोटोकॉल FoxP3hiCD4 + मानचित्र खोलें।
- उपयोगकर्ता-परिभाषित प्रोटोकॉल ऐप 4, एपी5, APP6, APP7 और APP 8 का उपयोग करके सीडी 8 +, सीडी68 +, एमपीओ +, αSMA और CD34 + वस्तुओं की पहचान और मात्रा निर्धारित करें, जैसा कि धारा 6.4 से 6.4.3.2 में किया गया है, प्रत्येक मामले में रुचि का ऐप लोड कर रहा है।Table 1
नोट: एपीपी 4 से 8 पूर्वनिर्धारित आरओआई स्ट्रोमा और परेंचिमा पर काम करते हैं।
Representative Results
TME में रुचि की कल्पना, मात्रा और मानचित्रण सेल आबादी के लिए रणनीति का अवलोकन
विभिन्न ऊतक डिब्बों (टीसी) में ब्याज (COIs) की सेल आबादी की मात्रा निर्धारित करने और उनके स्थानिक संगठन की विशेषता के लिए, हमने एक कार्यप्रवाह तैयार किया जो सस्ती और तकनीकों का उपयोग करने में आसान है और स्थितीय जानकारी को अधिकतम करता है जिसे कीमती एफएफपीई नैदानिक नमूनों(चित्रा 1)से प्राप्त किया जा सकता है। सबसे पहले, धारावाहिक पूरे ऊतक FFPE वर्गों COIs (जैसे, प्रतिरक्षा कोशिकाओं) और टीसी (जैसे, स्ट्रोमा बनाम parenchyma)(चित्रा 1,कदम 1) के दृश्य के लिए दाग थे । लगातार वर्गों की संख्या को दाग दिया जाना चाहिए न्यूनतम रखा जाना चाहिए जो अनुसंधान प्रश्न को संबोधित करने के लिए आवश्यक ब्याज या ऊतक सुविधाओं की कोशिकाओं के दृश्य की अनुमति देता है। धारावाहिक वर्गों की संख्या जितनी छोटी होगी, ऊतक वास्तुकला समानता और समीपस्थ वर्गों में सामंजस्य उतना ही अधिक होगा। इसके अलावा, मल्टीप्लेक्सिंग क्षमता का विस्तार19तकनीकों को अलग करने और फिर से जांच के माध्यम से फ्लोरोसेंटी दाग वर्गों के पुन: उपयोग के माध्यम से किया जा सकता है ।
एक बार धुंधला कदम किया गया, एक पूरी स्लाइड स्कैनर छवियों को डिजिटाइज करने के लिए इस्तेमाल किया गया था । सीरियल सेक्शन से हासिल की गई छवियों को स्वचालित फैशन(चित्र 1,धारा 2) में वर्चुअल मल्टीप्लेक्स स्लाइड में गठबंधन और समेकित किया गया था। इसके बाद, ऊतक के लिए एक आरओआई को उपयोगकर्ता-परिभाषित प्रोटोकॉल के साथ चित्रित किया गया था जिसने ऊतक से जुड़े पिक्सल (टीएपीएस)(चित्रा 1,चरण 3) की पहचान की थी। बाद में, आरओआई ऊतक को अतिरिक्त आरओआई के रूप में परिभाषित टीसी में खंडित किया गया था। (चित्रा 1,चरण 4)। इसके बाद, उपयोगकर्ता-परिभाषित प्रोटोकॉल का पता लगाया जाता है और विभिन्न टीसी(चित्रा 1,चरण 5) में COIs का पता लगाया जाता है और निर्धारित किया जाता है। अंत में, सीओआई के ऊतक हीटमैप उनके घनत्व और उनके ऊतक निर्देशांक(चित्र 1,चरण 6) के आधार पर उत्पन्न किए गए थे।
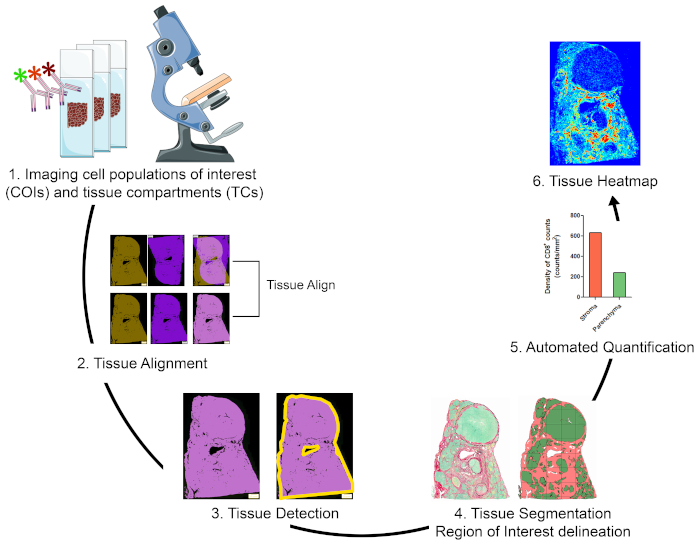
चित्रा 1: TME में प्रतिरक्षा कोशिकाओं की कल्पना, मात्रा और मानचित्रण के लिए रणनीति का योजनाबद्ध प्रतिनिधित्व। (1)सीओआई और टीसी लेबलिंग के लिए सीरियल पूरे टिश्यू सेक्शन दाग दिए गए। पूरी स्लाइड स्कैनर का इस्तेमाल करते हुए पूरे टिश्यू सेक्शन को डिजिटाइज्ड किया गया। (2)सीरियल सेक्शन से प्राप्त छवियों को टिश्यूसंरेखित विश्लेषण मॉड्यूल का उपयोग करके स्वचालित फैशन में जोड़ा, गठबंधन और सहपंजीकृत किया गया था। व्यक्तिगत छवियों के उच्च सटीक संरेखण से एक समग्र छवि उत्पन्न की गई थी। (3)समग्र छवि में ऊतक से जुड़े पिक्सल (टीएपीएस) का स्वचालित पता लगाने के लिए उपयोगकर्ता-परिभाषित प्रोटोकॉल का उपयोग किया गया था। (4)ऊतक को आरओआई के रूप में परिभाषित टीसी (जैसे, स्ट्रोमा और परेंचिमा) में खंडित किया गया था। (5)विभिन्न टीसी में सीओआई के स्वचालित पता लगाने और क्वांटिफिकेशन के लिए उपयोगकर्ता परिभाषित प्रोटोकॉल का उपयोग किया गया था।(6)सीओआई के ऊतक हीटमैप उत्पन्न किए गए थे। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
इमेजिंग COIs और TCs
एचबीवी से जुड़े हेपेटोसेलुलर कार्सिनोमा के साथ एक विषय से पुन: क्रम में कटौती ट्यूमर के तीन धारावाहिक FFPE पूरे ऊतक वर्गों के रूप में चित्रा 2एमें धुंधला के एक या एक से अधिक दौर में दाग थे । सेक्शन मुझे ऊतक वास्तुकला, सेल आकृति विज्ञान दिखाने और घातक रूप से प्रासंगिक मापदंडों जैसे द्रोह, ट्यूमर ग्रेड और प्रतिरक्षा घुसपैठ के समग्र मूल्यांकन(चित्रा 2सी)को दिखाने के लिए एच एंड ई के साथ दाग दिया गया था। समीपस्थ खंड II में, जिगर परेंचिमल और गैर-पैरान्चिमल कोशिकाओं(चित्रा 2ए)लेबलिंग के लिए MIF के दो दौर का उपयोग किया गया था। पहले दौर में, सामान्य और ट्यूमर जहाजों को एंडोथेलियल कोशिकाओं के CD34 धुंधला का उपयोग करके कल्पना की गई थी। इसके अतिरिक्त, साइटोकेराटिन 8/18 का उपयोग करके एपिथेलियल कोशिकाओं (हेपेटोसाइट्स और कोलंगियोसाइट्स) की पहचान की गई, और फाइब्रोजेनिक सक्रिय हेपेटिक स्टेलेट कोशिकाओं को अल्फा चिकनी मांसपेशी ऐक्टिन सकारात्मक (αSMA +) कोशिकाओं(चित्रा 2सी)के रूप में पहचाना गया। छवि अधिग्रहण के बाद, ऊतक वर्गों को निर्वस्त्र कर मैक्रोफेज (सीडी68) और मायोफिब्रोब्लास्ट (डेसमिन) के खिलाफ एंटीबॉडी के साथ फिर से जांच की गई। बेहतर ट्यूमर प्रतिरक्षा घुसपैठ की विशेषता के लिए, आसन्न धारावाहिक अनुभाग III सेलुलर मार्कर CD3, CD4, CD8, फोर्कहेड बॉक्स P3 (FoxP3), और myeloperoxidase (MPO) के लिए MIF के दो दौर का उपयोग कर दाग था । सभी मामलों में DAPI एक परमाणु प्रतिदाग के रूप में इस्तेमाल किया गया था । अंत में, सेक्शन III को पीएसआर दाग के साथ दाग दिया गया और फिब्रिलर कोलेजन की कल्पना करने के लिए तेजी से हरे रंग के साथ प्रतिदाग किया गया और ऊतक को स्ट्रोमा और परेंचिमा(चित्रा 2सी)में खंडित किया गया।
एक 20X उद्देश्य लेंस से लैस एक पूरी स्लाइड स्कैनर दाग वर्गों डिजिटाइज करने के लिए और आभासी स्लाइड बनाने के लिए इस्तेमाल किया गया था । तीन धारावाहिक वर्गों(चित्रा 2बी)से छह छवियों का अधिग्रहण किया गया था और बाद में चित्रा 1में योजनाबद्ध प्रतिनिधित्व के अनुसार विस सॉफ्टवेयर का उपयोग करके आभासी स्लाइड का विश्लेषण किया गया ।
छवि विश्लेषण
छवि विश्लेषण में पांच चरण शामिल थे: 1) ऊतक संरेखण; 2) ऊतक का पता लगाना; 3) ऊतक विभाजन; 4) COIs के स्वचालित मात्राकरण; और 5) ऊतक हीट मैपिंग। छवि विश्लेषण के लिए सभी प्रोटोकॉल छवि विश्लेषण सॉफ्टवेयर के लेखक मॉड्यूल का उपयोग करके विकसित किए गए थे और पाठ में ऐप के रूप में संदर्भित किए जाते हैं।
ऊतक संरेखण
तीन धारावाहिक वर्गों से छह आभासी स्लाइड, 11 मार्कर प्लस एच एंड ई और पीएसआर दाग फैले, छवि विश्लेषण सॉफ्टवेयर के Tissualign मॉड्यूल में लोड किया गया । इसके बाद, छवियों को स्वचालित फैशन में जोड़ा, गठबंधन और सहपंजीकृत किया गया था, जिसमें 11-प्लेक्स प्लस एच एंड ई और पीएसआर वर्चुअल कंपोजिट छवि उत्पन्न की गई थी, जिसमें व्यक्तिगत छवियों(आंकड़े 2A-C)की सभी परतें शामिल थीं। संरेखण आसन्न धारावाहिक वर्गों से उत्पन्न छवियों के मामले में सटीक था, जिसमें अनुरूप ऊतक संरचनाओं को संरेखण पर एक समरूप फैशन में तैनात और व्यवस्थित दिखाया गया था(चित्रा 2सी और चित्रा S1A)। इसके अलावा, संरेखण एक ही खंड(चित्रा S1B)से निकलने वाली छवियों के लिए व्यक्तिगत सेल स्तर पर सटीक था। स्वचालित संरेखण का समय छवियों की संख्या, आकार, जटिलता और समानता पर निर्भर करता है। उपरोक्त छह आभासी स्लाइड के संरेखण हमारे विस स्टेशन में 15 मिन लिया ।

चित्रा 2: धारावाहिक ऊतक वर्गों और छवि संरेखण का धुंधला । (A)COIs और TCs के दृश्य के लिए तीन धारावाहिक वर्गों पर किए गए दाग का सारांश । कोष्ठक में संख्या छवि पदनाम का संकेत मिलता है । धाराओं द्वितीय और III के लिए, ऊतकों छीन लिया और एंटीबॉडी के एक दूसरे कॉकटेल के साथ फिर से जांच की गई । (ख)ऊतक संरेखण से पहले और बाद में छह व्यक्तिगत पूरे ऊतक छवियों का अवलोकन (बाएं और दाएं, क्रमशः) । स्केल बार = 3,500 माइक्रोन.(सी)गठबंधन छवियों का दृश्य ज़ूम किया। स्केल बार = 80 माइक्रोन. कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
ऊतक का पता लगाना
एक बार छवियों को जोड़ा और गठबंधन किया गया, हम TAPs(चित्रा 3ए)की पहचान करने की मांग की । TAPs (APP 1, तालिका 1)के स्वचालित पता लगाने के लिए एक ऐप डिजाइन करने के लिए, हमने दो गुणों का लाभ उठाया जो ऊतक से जुड़े पिक्सेल से टैप्स को अलग करते हैं। सबसे पहले, डीपीआई सिग्नल (ब्लू बैंड) नाभिक तक सीमित है, जो विशेष रूप से ऊतक में स्थित है, जिसका अर्थ है कि सभी डीपीआई + पिक्सल टैप्स का सबसेट हैं। दूसरा, टीएपीएस में ऊतक से जुड़े पिक्सेल की तुलना में हरे और पीले रंग के बैंड में उच्च ऑटोफ्लोरेसेंस सिग्नल होता है। नतीजतन, हमने ऊतक का पता लगाने(तालिका 1)के लिए एपीपी 1 विकसित किया, जो सरल थ्रेसहोल्ड तकनीकों का उपयोग करके इन चैनलों में बेसलाइन सिग्नल के आधार पर टीएपीएस का पता लगाता है। नीले, हरे और पीले बैंड के लिए थ्रेसहोल्ड सेट किए गए थे ताकि TAPs के पास थ्रेसहोल्ड के ऊपर पृष्ठभूमि तीव्रता मूल्य हों, जबकि ऊतक से जुड़े नहीं पिक्सेल में नीचे मूल्य थे। ऊतक का पता लगाने के लिए ऐप 1 को इमेज आईआईए पर लागू किया गया था, जिसमें नीले, हरे और पीले चैनलों(चित्रा 3ए)में परतें शामिल हैं। एपीपी 1 के आउटपुट के रूप में, टीएपीएस के शीर्ष पर एक उज्ज्वल हरे रंग का मुखौटा निर्धारित किया गया था, और "ऊतक" नामक एक आरओआई को चित्रित किया गया था (आउटपुट, चित्रा 3ए)। इसके अलावा, ऊतक के क्षेत्र को मात्रात्मक आउटपुट चर के रूप में निर्धारित किया गया था। क्योंकि ऐप 1 आरओआई ऊतक में ऊतक से जुड़े पिक्सेल को शामिल नहीं करता है, इसलिए उन्हें इस आरओआई(चित्रा 3ए)के आधार पर बाद के विश्लेषण से बाहर रखा गया था। TAPs की पहचान करने में एपीपी 1 की सटीकता चित्रा 3एमें दिखाई गई है।
ऊतकों के लिए आरओआई का ऊतक विभाजन और चित्रण
इसके बाद, हमने ऊतक को स्ट्रोमा बनाम परेंचिमा में विभाजित करके आरओआई ऊतक के अंदर विभिन्न डिब्बों को परिभाषित करने के लिए आगे बढ़ाया। हमने पीएसआर दाग छवि (आईआईआईसी, फिगर 2सी)का उपयोग किया, जहां स्ट्रोमा को फिब्रिलर कोलेजन (रेड बैंड) के जमाव से जुड़े क्षेत्र के रूप में परिभाषित किया जा सकता है, जिस क्षेत्र के रूप में पैरानिमा अनुपस्थित हैं, और तेजी से हरे रंग के काउंटरदाग (ग्रीन बैंड)(चित्रा 3बी)की तस है। हमने टीसी स्ट्रोमा और परेंचिमा को डिजिटल रूप से परिसीमित करने के लिए एपीपी 2(टेबल 1)बनाया। यह ऐप पूर्वनिर्धारित आरओआई ऊतक (आउटपुट, चित्रा 3ए)पर काम करता है और छवि विश्लेषण मॉड्यूल में एकीकृत क्लासिफायर टूल को प्रशिक्षित करने के लिए प्रतिनिधि स्ट्रोमा और पैरान्चिमा क्षेत्रों का उपयोग करता है। प्रशिक्षित Classifier या तो एक स्ट्रोमा या एक parenchyma लेबल (सामन और हरे रंग, क्रमशः, चित्रा 3बी)के लिए पिक्सल असाइन करता है । पिक्सल के वर्गीकरण पर, एपीपी 2 ने आरओआई स्ट्रोमा और परेंचिमा(चित्रा 3बी और तालिका 1)को परिभाषित करने के उद्देश्य से रूपात्मक संचालन निष्पादित किया। पिक्सल के वर्गीकरण और संबंधित आरओआई को जेनरेट करने पर एपीपी 2 का प्रदर्शन चित्रा 3बीमें दिखाया गया है । इसके अतिरिक्त, एपीपी 2 स्ट्रोमा और पैरान्चिमा के क्षेत्र की मात्रा निर्धारित करता है। अंत में, भले ही विभाजन पीएसआर दाग अनुभाग का उपयोग करके किया जाता है, उल्लिखित स्ट्रोमा और पैरान्चिमा क्षेत्रों को पीएसआर छवि के अनुरूप किसी भी छवि में स्थानांतरित किया जा सकता है।

चित्रा 3: स्वचालित ऊतक का पता लगाने/विभाजन और संबंधित ROIs की पीढ़ी । (A)इमेज आईआईए का उपयोग टीएपीएस (बाएं छवि, स्केल बार = 6,000 माइक्रोन) की पहचान करने के लिए किया गया था। एक उज्ज्वल हरे रंग का मुखौटा एपीपी 1(तालिका 1)का उपयोग करके ऊतक (आउटपुट 1) नामक आरओआई उत्पन्न करने के लिए सौंपा गया था। ठीक है, इनसेट से पता चलता है जूम दृश्य TAPs का पता लगाने में APP 1 की सटीकता का प्रदर्शन । स्केल बार = 350 माइक्रोन।(बी)आरओआई टिश्यू (आउटपुट 1) को एपीपी 2 का उपयोग करके स्ट्रोमा और पैरान्चिमा में खंडित किया गया है। बाईं ओर छवि आरओआई स्ट्रोमा (सामन) और आरओआई पैरान्चिमा (हरे) में खंडित आरओआई ऊतक के एक दृश्य को दर्शाती है। स्केल बार = 4,500 माइक्रोन। दाईं ओर, आरओआई ऊतक, मूल पीएसआर धुंधला (छवि आईआईओस), और आरओआई स्ट्रोमा और पैरान्चिमा के लिए इनसेट के दृश्यों को ज़ूम किया गया। स्केल बार = 250 माइक्रोन। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
COIs का स्वचालित मात्राीकरण
इसके बाद, हम ROIs स्ट्रोमा और Parenchyma में COIs की पहचान करने, पता लगाने और मात्रा निर्धारित करने के लिए रवाना हुए। निम्नलिखित सीओआई का पता लगाने और गिनने के लिए एपीपी 3 से 8(टेबल 1)बनाए गए थे: सीडी4 +FoxP3+, CD8+, CD68+, MPO+, αSMA+, और CD34 + कोशिकाओं, क्रमशः। APP 3 को नियामक टी कोशिकाओं (ट्रेग्स) के सरोगेट मार्कर के रूप में CD4 + FoxP3 + कोशिकाओं (छवि IIIA, चित्रा 2C)का पता लगाने और गिनने के लिए डिज़ाइन किया गया था। यह प्रोटोकॉल परमाणु प्रतिलेखन कारक FoxP3 (लाल बैंड) और डीएनए लेबलिंग dye DAPI (ब्लू बैंड) से संकेत के colocalization का पता लगाता है । यह देखते हुए कि हाल ही में सक्रिय टी कोशिकाएं FoxP3 को अपरेच करती हैं, ट्रेग्स के लिए समृद्ध करने के लिए हमने केवल उज्ज्वल FoxP3 + कोशिकाओं (FoxP3हाय)का चयन करने के लिए थ्रेसहोल्ड निर्धारित किए हैं। इसके बाद, सभी पूर्वचयनित DAPI + FoxP3हाय कोशिकाओं में से, केवल उन है कि उज्ज्वल अंगूठी के आकार का CD4 संकेतों (ग्रीन बैंड) से घिरे थे लेबल और FoxP3हायCD4 + कोशिकाओं (गुलाबी लेबल, चित्रा 4ए)के रूप में गिना जाता था । आरओआई स्ट्रोमा और परेंचिमा में FoxP3हायसीडी4 + कोशिकाओं का घनत्व एपीपी 3(चित्रा 4ए)के मात्रात्मक आउटपुट चर के रूप में निर्धारित किया गया था।
इसी तरह, एपीपी 4 से 6 को सीडी8 +, सीडी68 +और एमएसपी + कोशिकाओं का पता लगाने के लिए डिजाइन किया गया था। ये एपीपी सीओआई का पता लगाने और मात्रा निर्धारित करने के लिए एक ही बेसलाइन डिजाइन साझा करते हैं। विशेष रूप से, सीओआई की पहचान विशिष्ट सेल जनसंख्या बायोमार्कर से संकेत तीव्रता के आधार पर की जाती है, और फिर व्यक्तिगत कोशिकाओं(तालिका 1)को रेखांकित करने के लिए कई पोस्टप्रोसेसिंग रूपात्मक चरणनिष्पादित किए जाते हैं। व्यक्तिगत कोशिकाओं या COIs लेबल, गिना जाता है, और उनके ऊतक निर्देशांक पंजीकृत कर रहे हैं । एपीपी 4 से 6 आरओआई स्ट्रोमा और परेंचिमा(चित्रा 4बी-डी)में सीओआई के घनत्व का भी निर्धारण करते हैं।
हमारे DAPI धुंधला की गुणवत्ता APPs 3 से 6 में नाभिक विभाजन को एकीकृत करने के लिए काफी अच्छा नहीं था, इसलिए हम यह सुनिश्चित नहीं कर सकते कि सभी व्यक्तिगत रूप से लेबल वाली वस्तुएं व्यक्तिगत कोशिकाएं हैं। इस कारण से, हमने लेबल वाली वस्तुओं/मिमी2 (चित्रा 4)की गिनती में कोशिकाओं का घनत्व व्यक्त किया। हालांकि, एएपी 3 से 6 में निर्मित पोस्टप्रोसेसिंग चरणों में सेल समुचेश को सफलतापूर्वक अलग किया गया था, और व्यापक दृश्य निरीक्षण से पता चला कि अधिकांश लेबल वाली वस्तुएं एकल कोशिकाओं से मेल खाती थीं।
αSMA + और CD34 + क्षेत्र का पता लगाने के लिए, हमने क्रमशः एपीपी 7 और 8(तालिका 1)विकसित किया। दोनों एपीपी थ्रेसहोल्ड के आधार पर विशिष्ट संकेत का पता लगाते हैं और आरओआई स्ट्रोमा और परेंचिमा(चित्रा 4ई-एफ)में सकारात्मक क्षेत्र का प्रतिशत निर्धारित करते हैं।
आभासी मल्टीप्लेक्स स्लाइड पैदा करने की सबसे दिलचस्प संभावनाओं में से एक सहस्थानीयकरण अभिव्यक्ति का विश्लेषण है। हमने αSMA और desmin के बीच colocalization का पता लगाने के लिए APP 10 उत्पन्न किया, दो मार्कर जिगर में myofibroblasts द्वारा सह-व्यक्त किए गए। APP 10 αSMA, desmin, और αSMA प्लस desmin(तालिका 1)के लिए सकारात्मक पिक्सल खोजने के लिए थ्रेसहोल्ड का उपयोग करता है । मात्रात्मक आउटपुट चर के रूप में, ऐप 10 αSMA + क्षेत्र, डेसमिन + क्षेत्र और इन दो मार्कर(चित्रा S3)की सहस्थानीय अभिव्यक्ति का क्षेत्र निर्धारित करता है।

चित्रा 4: टीसी स्ट्रोमा और पैरान्चिमा में सीओआई की पहचान और मात्राकरण। (A-एफ) सीडी4 +FoxP3+, CD8+, CD68+, MPO+, αSMA +, और CD34+ COIs में प्रोटोकॉल 3, 4, 5, 6, 7 और 8, क्रमशः(तालिका 1)का उपयोग करके स्वचालित पता लगाने और मात्रा का पता लगाना। बाईं ओर दिखाए गए मूल छवियां हैं, बीच में प्रसंस्कृत छवियां, और सही मात्रा पर। आंकड़े 4A-D,स्केल बार = 40 माइक्रोन के लिए। आंकड़े 4E और एफके लिए, स्केल बार = 350 माइक्रोन. कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
टीसी स्ट्रोमा और परेंचिमा में सीओआई को मात्रा देने के विकल्प के रूप में, हमने 1 से 4(चित्र5ए, एचऔर I)नाम के विभिन्न घातक पिंडों में प्रतिरक्षा कोशिकाओं के घनत्व का निर्धारण किया। प्रत्येक नोड्यूल के लिए आरओआई को मैन्युअल रूप से चित्रित किया गया था जैसा कि चित्र5एमें इंगित किया गया था। विशिष्ट ऊतक प्रतिरक्षा हस्ताक्षर प्रत्येक नोड्यूल की विशेषता है, आगे TME के आंतरिक विषमता का खुलासा ।
टिश्यू हीटमैप्स
जैसा कि ऊपर बताया गया है, एपीपी 3 से 8 हर व्यक्तिगत रूप से लेबल वाली वस्तु के ऊतक निर्देशांक स्टोर करते हैं। यह सुविधा ऊतक मानचित्रों की स्वचालित पीढ़ी की अनुमति देती है जहां किसी दिए गए सेल आबादी के उच्च घनत्व के क्षेत्रों को हॉट स्पॉट (लाल) के रूप में प्रदर्शित किया जाता है, और ठंडे धब्बे (गहरे नीले) के रूप में अपेक्षाकृत कम घनत्व वाले क्षेत्र। इंटरमीडिएट घनत्व मूल्यों को चित्रा 5में दिखाए गए रंग पैमाने के अनुसार रंग सौंपे जाते हैं। ऊतक हीटमैपएपी द्वारा उत्पन्न किए गए थे जो छवियों को 50 माइक्रोन व्यास के सर्कल में विभाजित करते थे और सर्कल के अंदर दिए गए सीओआई के सापेक्ष घनत्व के अनुसार एक रंग सौंपा जाता था। जैसा कि चित्रा 5बी-जीमें प्रदर्शित किया गया है, TME में विभिन्न COIs के पोजिशनिंग पैटर्न और तीव्रता वितरण काफी भिन्न थे। इसके अलावा, व्यक्तिगत नोड्यूल के स्तर पर, ऊतक क्षेत्र में विभिन्न आबादी की व्यवस्था अद्वितीय थी(चित्रा S2A-C)। इस तकनीक की शक्ति का एक उदाहरण प्रदान करने और एक ही नोड्यूल में विभिन्न आबादी से गर्म स्थानों के स्थानिक संगठन की कल्पना करने के लिए, व्यक्तिगत सेल प्रकारों से हॉट स्पॉट मैन्युअल रूप से निकाले गए और नोड्यूल 2(चित्रा S2, Figure D,और चित्रा ई)की रूपरेखा पर एक साथ मैप किए गए थे।

चित्रा 5: TME में COIs के ऊतक हीटमैप्स। (A)पिक्रोसिरियस रेड धुंधला नाद का स्थान दिखाना 1, 2, 3 और 4। (B-जी) क्रमशः CD4+FoxP3+, CD8+, CD68+, MPO+, CD34+, और αSMA + COIs के लिए ऊतक हीटमैप्स। गहरा नीला सापेक्ष कम घनत्व को इंगित करता है, और लाल सापेक्ष उच्च घनत्व को इंगित करता है। मध्यवर्ती घनत्व मूल्यों को दिखाए गए रंग पैमाने के अनुसार रंग सौंपे जाते हैं। (एच और I)क्रमशः नोड्यूल 1, 2 और 3 + 4 प्रति सेल प्रकार और प्रति नोड्यूल में सीओआई का परिमाणीकरण। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।

अनुपूरक चित्रS1: ऊतक संरेखण का सत्यापन। (A)सीडी 34 धुंधला (लाल रंग में) धारा द्वितीय (इनपुट 1) पर किया जाता है हरे रंग में एक सीडी 34 मुखौटा पैदा करने के लिए प्रयोग किया जाता है (उत्पादन 1) । ग्रीन मास्क (आउटपुट 1) गठबंधन धारावाहिक अनुभाग I (इनपुट 2) से एच एंड ई छवि पर मढ़ा जाता है। विलय छवि संवहनी संरचनाओं का सही पत्राचार दिखाती है। स्केल बार = 50 माइक्रोन। (ख)इमेज IIIA का उपयोग डीपीआई, सीडी 4 और फॉक्सपी 3 (इनपुट 1) के विलय को दिखाने के लिए सीडी4 + FoxP3+ कोशिकाओं (मजेंटा में आउटपुट 1) के लिए लेबल उत्पन्न करने के लिए किया गया था। आउटपुट 1 लेबल गठबंधन छवि IIIB (इनपुट 2) पर स्थानांतरित किया गया था और जोड़े FoxP3/DAPI, और CD4/CD3 के बीच विलय छवि में सही पत्राचार दिखाता है । स्केल बार = 15 माइक्रोन। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।

पूरक चित्रा S2: ऊतक हीटमैप्स का तेजी से देखा गया। (A-C) सीडी4 +FoxP3+, CD8 +, CD68+, और MPO + कोशिकाओं के लिए ओड्यूल 1-4 में ऊतक हीटमैप्स। नोड्यूल 1, 2 और 3 +4 में स्केल बार क्रमशः 1,500 माइक्रोन, 700 माइक्रोन और 500 माइक्रोन का प्रतिनिधित्व करते हैं। (D)काले ठोस लाइन के साथ नोड्यूल 2 की रूपरेखा। (ई)सीडी4 +FoxP3 +, CD8 +, CD68 +, और नोवो + कोशिकाओं के लिए हॉट स्पॉट नोड्यूल 2 में निकाले गए और डीमें परिभाषित नोड्यूल 2 रूपरेखा पर एक साथ मैप किए गए । कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।

अनुपूरक चित्रS 3: Colocalization विश्लेषण । (क)बाईं और मध्य पर क्रमशः लाल रंग में हरे और डेसमिन लेबल में αSMA लेबल की छवियां हैं । दाईं ओर पीले रंग में एक αSMA/desmin डबल सकारात्मक क्षेत्र है । (ख)αSMA + क्षेत्र, desmin + क्षेत्र, और αSMA/desmin डबल सकारात्मक क्षेत्र के परिमाणीकरण । स्केल बार = 150 माइक्रोन। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
| App | उद्देश्य | वर्गीकरण | वर्गीकरण | प्रोसेसिंग के बाद के कदम | आउटपुट वेरिएबल्स |
| विधि | सुविधाऐं | ||||
| (पिक्सेल मूल्य) | |||||
| 1 | ऊतक का पता लगाना | शुरूआत | चैनल डीपीआई (150) | ओ 3 चैनलों के लिए सहस्थानीयकृत उपरोक्त सीमा मूल्यों के साथ लेबल ऑब्जेक्ट्स | ओ आरओआई टिश्यू |
| चैनल FITC/A488 (१२०) | ओ क्लोज पॉजिटिव ऑब्जेक्ट 5 पिक्सल | ओ टिश्यू एरिया | |||
| चैनल TRITC/A568 (४०) | ओ आरओआई टिश्यू बनाएं | ||||
| 2 | ऊतक विभाजन | निर्णय वन | आरजीबी-आर मीडियन | ओ भरें छेद | ओ आरओआई स्ट्रोमा |
| आरजीबी-जी मीडियन | ओ आरओआई स्ट्रोमा बनाएं | ओ स्ट्रोमा एरिया | |||
| आरजीबी-बी मीडियन | ओ आरओआई परेंचिमा बनाएं | ओ आरओआई परेंचिमा | |||
| आईएचएस-एस मीडियन | ओ परेंचिमा क्षेत्र | ||||
| एच एंड ई इओसिन मीडियन | |||||
| 3 | CD4+ FoxP3 + कोशिकाओं का पता लगाने और मात्रा निर्धारित करने के लिए | शुरूआत | चैनल डीपीआई (>600) | डीएपीआई और Cy5/A647 के सहस्थानीयकरण के साथ ओ लेबल ऑब्जेक्ट्स, FITC/A488 सिग्नल से घिरा हुआ है | आरओआई स्ट्रोमा और परेंचिमा में सीडी 4 +FoxP3 + कोशिकाओं की गिनती और घनत्व |
| चैनल FITC/A488 पॉली चौरसाई (>850) | ओ 7 माइक्रोन2 से छोटी स्पष्ट वस्तुएं | ओ व्यक्तिगत CD4 + FoxP3 + कोशिकाओं का निर्देशांक | |||
| चैनल Cy5/A647 (>800) | |||||
| 4 | CD8 + कोशिकाओं का पता लगाने और मात्रा निर्धारित करने के लिए | शुरूआत | चैनल डीपीआई (<1200) | ओ स्पष्ट सकारात्मक वस्तुओं 15 μm2 से छोटी | आरओआई स्ट्रोमा और परेंचिमा में सीडी 8 + कोशिकाओं की गिनती और घनत्व |
| चैनल Cy5/A647 मीडियन (>80) | ओ क्लोज पॉजिटिव ऑब्जेक्ट्स 2 पिक्सल | ओ अलग-अलग कोशिकाओं का निर्देशांक | |||
| ओ अलग-अलग वस्तुएं | |||||
| 5 | CD68 + कोशिकाओं का पता लगाने और मात्रा निर्धारित करने के लिए | शुरूआत | चैनल FITC/A488 (>200) | ओ स्पष्ट सकारात्मक वस्तुओं 20 μm2 से छोटी | आरओआई स्ट्रोमा और परेंचिमा में सीडी 68 + कोशिकाओं की गिनती और घनत्व |
| ओ डिलेट पॉजिटिव ऑब्जेक्ट्स 3 पिक्सल | ओ व्यक्तिगत CD68 + कोशिकाओं का निर्देशांक | ||||
| ओ अलग-अलग वस्तुएं | |||||
| 6 | डीपीओ + कोशिकाओं का पता लगाने और निर्धारित करने के लिए | शुरूआत | चैनल डीपीआई (>400) | ओ स्पष्ट वस्तुओं 5 μm2 से छोटी | आरओआई स्ट्रोमा और परेंचिमा में एमपीओ + कोशिकाओं की गिनती और घनत्व। |
| चैनल TRITC/A568 (900-4000) | ओ डिलेट 3 पिक्सल पॉजिटिव ऑब्जेक्ट्स | ओ व्यक्तिगत डीपीओ + कोशिकाओं का समन्वय। | |||
| ओ अलग-अलग वस्तुएं | |||||
| 7 | αSMA + क्षेत्र का पता लगाने और मात्रा निर्धारित करने के लिए | शुरूआत | चैनल TRITC/CF568 (>1050) | ओ स्पष्ट सकारात्मक वस्तुओं 25 μm2 से छोटी | आरओआई स्ट्रोमा और परेंचिमा में αSMA + क्षेत्र की गिनती और घनत्व |
| ओ डिलेट 3 पिक्सल पॉजिटिव ऑब्जेक्ट्स | αSMA + पिक्सल के निर्देशांक | ||||
| 8 | CD34 + क्षेत्र का पता लगाने और मात्रा निर्धारित करने के लिए | शुरूआत | चैनल DAPI (<5000) | ओ स्पष्ट सकारात्मक वस्तुओं 25 μm2 से छोटी | आरओआई स्ट्रोमा और परेंचिमा में सीडी 34 + क्षेत्र की गिनती और घनत्व |
| चैनल Cy5/A647 मीडियन (>120) | ओ डिलेट 3 पिक्सल पॉजिटिव ऑब्जेक्ट्स | सीडी 34+ पिक्सल का समन्वय | |||
| 9 | किसी दिए गए सेल आबादी के लिए ऊतक हीटमैप बनाएं | ऑब्जेक्ट हीटमैप | ऑब्जेक्ट हीटमैप | ओ हीटमैप | |
| ड्राइंग त्रिज्या 50 माइक्रोन | --- | ||||
| 10 | αSMA और Desmin के बीच की मात्रा सहस्थानीयकरण | शुरूआत | चैनल TRITC (CF568) (>1050) | TRITC (CF568) के लिए उपरोक्त सीमा मूल्यों के साथ ओ लेबल ऑब्जेक्ट्स | αSMA और Desmin की o Quantify colocalized अभिव्यक्ति |
| चैनल Cy5 (A647) (>1000) | Cy5 (A647) के लिए उपरोक्त सीमा मूल्यों के साथ लेबल ऑब्जेक्ट्स | ||||
| TRITC (CF568) और Cy5 (A647) के लिए उपरोक्त सीमा मूल्यों के सहस्थानीयकरण के साथ ओ लेबल ऑब्जेक्ट्स | |||||
| ओ स्पष्ट सकारात्मक वस्तुओं 25 μm2 से छोटी |
तालिका 1: छवि विश्लेषण के लिए नियोजित एपीपी के डिजाइन के लिए उपयोग किए जाने वाले सामान्य पैरामीटर। इस तालिका में निर्दिष्ट मापदंडों को इस विश्लेषण (जैसे, पृष्ठभूमि, कलाकृतियों, आदि) में उपयोग की जाने वाली छवियों की अनूठी विशेषताओं में समायोजित किया जाता है और अन्य छवियों पर लागू नहीं हो सकता है। क्योंकि इस अध्ययन में विश्लेषण की गई विशिष्ट छवियों के लिए उल्लिखित पोस्ट-प्रोसेसिंग चरणों को परिभाषित किया गया था, इसलिए वे जानबूझकर विस्तृत नहीं हैं। उपयोगकर्ता को विश्लेषण किए जाने वाले छवियों के लिए एपीपी को अनुकूलित करना चाहिए।
| धारा/धुंधला | प्राथमिक एंटीबॉडी | सेकेंडरी एंटीबॉडी |
| धारा II/1सेंट धुंधला | माउस IgG2a विरोधी मानव αSMA माउस IgG1 विरोधी मानव CD34 खरगोश विरोधी मानव साइटोकेराटिन 8/18 |
बकरी विरोधी माउस IgG2a CF568 चूहा विरोधी माउस IgG1 A647 गधा विरोधी खरगोश A488 |
| धारा II/2धुंधला | खरगोश विरोधी मानव डेसमिन माउस एंटी-ह्यूमन सीडी68 |
गधा विरोधी खरगोश A647 गधा विरोधी माउस DyLight 755 |
| धारा III/1सेंट धुंधला | माउस एंटी-ह्यूमन सीडी4 खरगोश विरोधी मानव FoxP3 बकरी विरोधी मानव एमएसपीओ |
गधा विरोधी माउस A488 गधा विरोधी खरगोश A647 गधा विरोधी बकरी A568 |
| धारा III/2धुंधला | खरगोश विरोधी मानव CD3 माउस एंटी-ह्यूमन सीडी8 |
गधा विरोधी माउस DyLight 755 गधा विरोधी खरगोश A647 |
तालिका 2: MIF के लिए प्राथमिक-माध्यमिक एंटीबॉडी जोड़े।
Discussion
कैंसर और अन्य इम्यूनोलॉजिकल विकारों में प्रतिरक्षा परिदृश्य को मैप करने के लिए ऊतक वर्गों में प्रतिरक्षा कोशिकाओं के स्थानिक समाधान की अनुमति देने वाली मल्टीप्लेक्सिंग तकनीकों को निष्पादित करने के लिए सरल, सुलभ और आसान। यहां, हम एक ऐसी रणनीति का वर्णन करते,हैं जो12,,13,17,19के मल्टीप्लेक्सिंग क्षमता और बहुआयामी मूल्यांकन का विस्तार करने के लिए व्यापक रूप से उपलब्ध लेबलिंग और डिजिटल विश्लेषण तकनीकों को एकीकृत करती है।, विभिन्न मार्कर के लिए तीन धारावाहिक वर्गों का धुंधला, और अलग-अलग तकनीकों के माध्यम से वर्गों का पुन: उपयोग, हमें एच एंड ई और पीएसआर दाग के अलावा 11 मापदंडों की कल्पना करने में सक्षम बनाया। इन वर्गों से छह छवियों को ऊतक संरेखण मॉड्यूल का उपयोग करके स्वचालित फैशन में गठबंधन किया गया था। संरेखण एक ही खंड से उत्पन्न छवियों के लिए व्यक्तिगत सेल स्तर पर सटीक था और पड़ोसी वर्गों से उत्पन्न छवियों के लिए अत्यधिक सामंजस्य था। वर्चुअल मल्टीप्लेक्सिंग ने हमें यह निर्धारित करने में सक्षम बनाया कि एक अनुभाग में कल्पना किए गए मार्कर किसी अन्य समीपस्थ अनुभाग में कल्पना किए गए मार्कर से कैसे संबंधित हैं। जबकि कुछ स्टेनिंग्स ने COIs का लेबल लगाया, अन्य ने टीसी का लेबल लगाया, जिससे हमें विभिन्न टीसी में COIs की मात्रा निर्धारित करने की अनुमति मिली । COIs के स्वचालित मात्राकरण के लिए सॉफ्टवेयर उपकरणों के उपयोग ने छवियों के प्रसंस्करण को बहुत सरल और त्वरित किया। इसके अलावा, डिजिटल विश्लेषण को चयनित क्षेत्रों के बजाय पूरे ऊतक वर्गों पर लागू किया गया था, जिसके परिणामस्वरूप TME का निष्पक्ष प्रतिनिधित्व होता है। इसके अलावा, क्योंकि सीओआई के ऊतक निर्देशांक पंजीकृत थे, ऊतक हीटमैप उत्पन्न करना संभव था।
इस प्रोटोकॉल में कई क्षेत्र हैं जहां समस्या निवारण की आवश्यकता हो सकती है। सबसे पहले, गरीब एंटीजन पुनर्प्राप्ति mIF की गुणवत्ता को प्रभावित कर सकते हैं, इसलिए एंटीजन पुनर्प्राप्ति बफर और अवधि के प्रकार विशिष्ट परख के लिए अनुकूलित किया जाना चाहिए/ दूसरा, उपयोग किए जाने वाले अवरुद्ध समाधान के प्रकार को प्राथमिक और द्वितीयक एंटीबॉडी के ऊतकों/एंटीजन/प्रजातियों के अनुकूल किया जाना चाहिए । हमारे हाथों में, प्रजातियों से 10% कुल सीरम के अलावा जहां ऊतक अवरुद्ध एफसी रिसेप्टर्स से आता है, और इस प्रकार गैर विशिष्ट एंटीबॉडी बाध्यकारी कम। प्रजातियों से सीरम के 10% के अलावा माध्यमिक एंटीबॉडी में उठाया गया था ऊतक अनुभाग के लिए माध्यमिक एंटीबॉडी के प्रत्यक्ष गैर विशिष्ट लगाव को कम करेगा । तीसरा, उचित सकारात्मक और नकारात्मक नियंत्रणों का उपयोग करके प्राथमिक और माध्यमिक एंटीबॉडी की विशिष्टता का सत्यापन आवश्यक है। चौथा, कुछ चैनलों में ऑटोफ्लोरेसेंस में वृद्धि और प्राथमिक एंटीबॉडी विपठ्ठन पर डीपीआई का प्रसार भी आम है। बढ़ी हुई ऑटोफ्लोरेसेंस को संबोधित करने के लिए, हमने प्राथमिक/माध्यमिक एंटीबॉडी जोड़े का उपयोग किया जहां विशिष्ट संकेत में पृष्ठभूमि के कम से कम 5x तीव्रता मूल्य थे । अंत में, कुछ उच्च आत्मीयता एंटीबॉडी नियमित रूप से अलग करना प्रक्रियाओं के साथ नहीं किया जा सकता है । इस मामले में, हम लेबलिंग के अंतिम दौर में इस तरह के एंटीबॉडी का उपयोग करने की सलाह देते हैं। उपयोगकर्ता को ब्याज के एंटीबॉडी के लिए इष्टतम विन्यास खोजने के लिए अलग-अलग धुंधला दृश्यों की कोशिश करनी पड़ सकती है। लेबलिंग के दूसरे या तीसरे दौर में आगे बढ़ने से पहले अलग करने की दक्षता की पुष्टि की जानी चाहिए ।
इस रणनीति की मुख्य सीमा और चुनौती ब्याज के मार्कर के लिए प्राथमिक और माध्यमिक फ्लोरोसेंट एंटीबॉडी के सही संयोजन खोज रही है। विभिन्न प्रजातियों में या विभिन्न आइसोटाइप के साथ उठाए गए प्राथमिक एंटीबॉडी ढूंढना जो एक साथ उपयोग किया जा सकता है, वाणिज्यिक रूप से उपलब्ध है द्वारा सीमित है। अधिकांश पूरी स्लाइड स्कैनर लैंप और फिल्टर से लैस हैं जो इमेजिंग को अधिकतम पांच चैनलों की अनुमति देते हैं, और सही प्रजातियों और सही फ्लोरोफोर में माध्यमिक एंटीबॉडी हमेशा उपलब्ध नहीं होते हैं। हम आंशिक रूप से धारावाहिक धुंधला और अनुक्रमिक लेबलिंग का उपयोग कर इन सीमाओं पर काबू पा लिया । ब्याज के मार्कर के लिए सबसे अच्छा संयोजन पर पहुंचने के लिए कई एंटीबॉडी संयोजनों का परीक्षण करने की आवश्यकता हो सकती है। एक और सीमा डीपीआई धुंधला की गुणवत्ता है, क्योंकि अलग करना और पुनर्जांच हमेशा नाभिक विभाजन प्रदर्शन की अनुमति नहीं दे सकता है।
ऊतक संरेखित मॉड्यूल के लिए उपयोगकर्ताओं से न्यूनतम प्रशिक्षण और कोई प्रोग्रामिंग कौशल की आवश्यकता होती है। सॉफ्टवेयर सैद्धांतिक रूप से असीमित संख्या में छवियों के संरेखण की अनुमति देता है। हालांकि, सटीक संरेखण वर्गों की संबंधितता पर निर्भर करता है, जहां करीब वर्ग जो अधिक हिस्टोलॉजिकल रूप से सामंजस्यपूर्ण हैं, अधिक सटीक रूप से गठबंधन कर रहे हैं। हमने एपीपी उत्पन्न करने के लिए विज़ के लेखक मॉड्यूल का उपयोग किया। एपीपी बनाने के लिए छवि विश्लेषण के बुनियादी ज्ञान की आवश्यकता होती है, लेकिन किसी अन्य छवि विश्लेषण सॉफ्टवेयर का उपयोग करते समय यह समान रूप से मामला है। अन्य छवि विश्लेषण सॉफ्टवेयर की तुलना में विज़ के अद्वितीय फायदों में विभिन्न तरीकों (जैसे, आईएफ, हिस्टोकेमिस्ट्री, आईएचसी) का उपयोग करके तैयार किए गए वर्गों से छवियों का स्वचालित संरेखण शामिल है। यह वर्चुअल मल्टीप्लेक्सिंग का उपयोग करके ब्याज के कई मार्कर के सहस्थानीयकरण अध्ययन की अनुमति देता है। इसके अलावा, एपीपी का लचीला और उपयोगकर्ता के अनुकूल डिजाइन उपयोगकर्ता-विशिष्ट अनुकूलन की अनुमति देता है। स्वचालित मात्राीकरण और मानचित्रण, और पूरे ऊतक वर्गों को संसाधित करने की संभावना, समय बचाता है और दृश्य निरीक्षण द्वारा मैनुअल गिनती की तुलना में पूर्वाग्रह को कम करता है।
यह रणनीति कैंसर और ऑटोइम्युनिटी के संदर्भ में ऊतक इम्यूनोलॉजी के लिए एक बहुत ही उपयोगी अनुसंधान उपकरण है लेकिन नैदानिक उपयोग के लिए अमान्य बनी हुई है। अतिरिक्त मानकीकरण और सत्यापन के साथ, इसका उपयोग भविष्य में कई अनुप्रयोगों (उदाहरण के लिए, कैंसर में प्रतिरक्षा परिदृश्य को भविष्यवाणी करने और इम्यूनोथेरप्यूटिक एजेंटों की प्रतिक्रिया की निगरानी करने के लिए मैप करने के लिए किया जा सकता है)। शकुन बायोमार्कर के साथ रोग मूल्यांकन गठबंधन करने के लिए इसे विभिन्न भड़काऊ स्थितियों (उदाहरण के लिए, भड़काऊ आंत्र रोग) के अनुकूल भी बनाया जा सकता है।
इस प्रोटोकॉल में मुख्य महत्वपूर्ण कदम लेबलिंग की दक्षता/विशिष्टता और इच्छित उपयोग या बायोमार्कर के लिए डिजाइन किए गए एपीपी की मजबूती है । इसलिए, दृश्य निरीक्षण द्वारा नियमित सत्यापन, विशेष रूप से एक नया ऐप डिजाइन करने पर, आवश्यक है। एक ही खंड पर अलग करने और फिर से जांच या विभिन्न प्रकार के दाग के कई दौरों का कुशल उपयोग महत्वपूर्ण घटक हैं और ऊतक या अनुभाग विशिष्ट हो सकते हैं। बड़े बैच विश्लेषण के साथ आगे बढ़ने से पहले ऐसी प्रक्रियाओं की दक्षता की पुष्टि करना महत्वपूर्ण है।
संक्षेप में, हम एक रणनीति प्रदान करते हैं जो मात्रात्मक और स्थानिक जानकारी को अधिकतम करती है जिसे मूल्यवान नैदानिक ऊतक नमूनों से प्राप्त किया जा सकता है। इस पद्धति को लागू करने के लिए आवश्यक संसाधन, उपकरण और ज्ञान व्यापक रूप से सुलभ हैं। हम TME में प्रतिरक्षा सेल आबादी की पहचान करने, मात्रा निर्धारित करने और मानचित्रण करने के उद्देश्य से परखों की योजना बनाने के लिए एक उपयोगी गाइड के रूप में इस पद्धति का प्रस्ताव करते हैं।
Disclosures
लेखक हितों के टकराव की घोषणा नहीं करते ।
Acknowledgments
हम अध्ययन प्रतिभागी को धन्यवाद देते हैं। हम ऊतक के नमूनों और सभी संबद्ध नैदानिक जानकारी की वसूली के लिए एचबीपी बायोबैंक के समन्वयक लुईस रूसो को धन्यवाद देते हैं। हम उत्कृष्ट तकनीकी सहायता के लिए विसिओपंसे से सीआरएचयूएम और माइकल पेर्श में आणविक विकृति और सेल इमेजिंग कोर सुविधाओं को स्वीकार करते हैं। वित्तपोषण: इस अध्ययन को कनाडाई लिवर फाउंडेशन, फोड्स डी रेचेचे डु क्यूबेक-सैंटे (एफआरक्यूएस) एड्स एंड संक्रामक रोग नेटवर्क (रेसेउ सिडा-एमआई) और हेपेटाइटिस सी (CanHepC) पर कनाडाई नेटवर्क से अनुदान द्वारा समर्थित किया गया था। CanHepC कनाडा के स्वास्थ्य अनुसंधान संस्थानों (CIHR) (एनएचसी-142832) और कनाडा की सार्वजनिक स्वास्थ्य एजेंसी से एक संयुक्त पहल द्वारा वित्त पोषित है । एम.एफ.एम. को यूनीवर्सिट डी मॉन्ट्रियल, बोर्स गेब्रियल मार्क्विस और एफआरक्यूएस से फैलोशिप मिली। टीएफ को CIHR और CanHepC से डॉक्टरेट फैलोशिप प्राप्त हुई । एससी हेपेटोबिलियरी और अग्नाशय ऑन्कोलॉजिकल सर्जरी, यूनीवर्सिट डी मॉन्ट्रियल में रोजर-डेस-ग्रोसिलर्स चेयर रखती है।
लेखक योगदान: M.F.M. डिजाइन, प्रयोग किया, और डेटा का विश्लेषण किया । टीएफ डिजाइन प्रयोग। ए.सी.बी. तकनीकी मार्गदर्शन प्रदान किया। जी.एस. अध्ययन विषय के सभी रोग मूल्यांकन किया और सभी रोग पहलुओं पर इनपुट प्रदान की है। एलएम ने एच एंड ई धुंधला, अनुकूलित और छवि अधिग्रहण किया। एमएने ने पीएसआर दाग का प्रदर्शन किया और मूल्यवान तकनीकी इनपुट प्रदान किया । एनबी छवि विश्लेषण में योगदान दिया । एससी एचबीपी बायोबैंक के लिए प्रमुख अन्वेषक है और बायोबैंक के समग्र संचालन की देखरेख के लिए जिम्मेदार है। उन्होंने परियोजना के सभी पहलुओं और इसके नैदानिक प्रभावों पर अमूल्य इनपुट भी प्रदान किया । M.F.M., T.F., और N.H.S. संकल्पनात्मक और अध्ययन डिजाइन किया । N.H.S. काम की निगरानी की और धन प्राप्त किया । M.F.M., T.F., A.C.B, और N.H.S. पांडुलिपि लिखा था । सभी लेखकों ने पांडुलिपि की समीक्षा की और उसे मंजूरी दी ।
Materials
| Name | Company | Catalog Number | Comments |
| Antigen Retrieval Solution: Sodium Citrate Buffer (10 mM Sodium Citrate, 0.05% v/v Tween 20, pH 6.0) | |||
| Blocking Solution: 1 % BSA, 10 % filtered human serum, 10 % filtered donkey serum, 0.1 % Tween 20, and 0.3% Triton in PBS | |||
| Bovine serum albumin (BSA) | Multicell | 800-095-EG | |
| Coplin jars (EASYDIP SLIDE STAINING SYSTEM) | Newcomersupply | 5300KIT | |
| Cover slides | Fisherbrand | 12-545E 22*50 | |
| Direct Red 80 | Sigma Aldrich | 365548 | |
| Donkey Serum | Sigma Aldrich | D9663 | |
| Ethanol 100% | |||
| Electric pressure cooker | Salton | ||
| Eosin | Leica Biosystems | 3801600 | CAUTION, eye irritation |
| Fast Green FCF | Sigma Aldrich | F7252 | CAUTION, harmful by inhalation, ingestion and skin absortion |
| FFPE section (4μm) slides | |||
| Glycine 0,1 M in PBS | |||
| Hematoxylin Stain Solution, Gil 1. Formulation, Regular Strength | Ricca Chemical Company | 3535-32 | |
| Holder (EasyDip Staining Jar Holder) | Newcomersupply | 5300RK | |
| Human Serum | Gemini | 22210 | |
| Humidity chamber | Millipore Sigma | Z670138-1EA | |
| Pap pen | abcam | ab2601 | |
| PBS | |||
| PBS-Tween 20 (0.1% v/v) | |||
| Permount Mounting Media | Fisher Chemical | SP15-500 | |
| Picric Acid 1.3 % | Sigma Aldrich | P6744 | CAUTION, skin and eye irritation |
| Picro-Sirius Red/Fast Green solution: Fast Green 0.1 % w/v + Sirius Red 0.2 % w/v in 1,3 % picric acid solution | |||
| Primary Antibody Anti-αSMA | Mouse IgG2a 1A4 | Sigma A2547 | Dilution 1/100 |
| Primary Antibody Anti-CD34 | Mouse IgG1 HPCA1/763 | Novus Biologicals NBP2-44568 | Dilution 1/250 |
| Primary Antibody Anti-Cytokeratin 8/18 | Rabbit EP17/EP30 | Agilent IR09461-2 | Ready to use |
| Primary Antibody Anti-CD68 | Mouse KP1 | Abcam ab955 | Dilution 1/200 |
| Primary Antibody Anti-Desmin | Rabbit Polyclonal | Invitrogen PA5-16705 | Dilution 1/200 |
| Primary Antibody Anti-CD4 | Mouse N1UG0 | Affymetrix 14-2444 | Dilution 1/250 |
| Primary Antibody Anti-FoxP3 | Rabbit 1054C | R & D MAB8214 | Dilution 1/100 |
| Primary Antibody Anti-MPO | Goat Polyclonal | R & D Systems AF3667 | Dilution 1/250 |
| Primary Antibody Anti-CD3 | Rabbit SP7 | Abcam ab16669 | Dilution 1/200 |
| Primary Antibody Anti-CD8 | Mouse C8/144B | Invitrogen 14-0085-80 | Dilution 1/200 |
| Secondary Antibody Donkey anti-mouse A488 | Polyclonal | Invitrogen A-21202 | Dilution 1/500 |
| Secondary Antibody Donkey anti-Rabbit A488 | Polyclonal | Invitrogen A-21206 | Dilution 1/500 |
| Secondary Antibody Donkey anti-goat A568 | Polyclonal | Invitrogen A-11057 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit A647 | Polyclonal | Invitrogen A-31573 | Dilution 1/500 |
| Secondary Antibody Rat anti-mouse IgG1 A647 | RMG1-1 | Biolegend 406618 | Dilution 1/500 |
| Secondary Antibody Goat anti-mouse IgG2a CF568 | Polyclonal | Sigma Aldrich SAB4600315 | Dilution 1/500 |
| Secondary Antibody Donkey anti-mouse DyLight 755 | Polyclonal | Invitrogen SA5-10171 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit DyLight 755 | Polyclonal | Invitrogen SA5-10043 | Dilution 1/500 |
| SDS | BioShop | SDS001,500 | CAUTION, oral skin and eye toxicity |
| Shandon multi-program robotic slide stainer | LabX | 11384903 | |
| Shandon Xylene Substitute, | Thermo Fisher Scientific | CA89413-336 | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Shaking water bath | |||
| SlowFade Gold antifade reagent with DAPI | Invitrogen | S36938 | |
| Sodium Citrate Dihydrate | Millipore Sigma | 1545801 | CAUTION, eye irritation |
| Stripping Buffer: mix 20 ml 10% w/v SDS with 12.5 ml 0.5 M Tris-HCl (pH 6.8), and 67.5 ml ultra-pure water. Under a fume hood, add 800 uL of 2-mercapto ethanol (114,4 mM final concentration) | |||
| Triton X-100 | Sigma Aldrich | T8787-50ML | |
| Tris-HCl | BioShop | 77-86-1 | |
| Tween 20 | Fisher Scientific | BP337-500 | |
| VIS Software | Visiopharm | ||
| Whole slide scanner Olympus BX61VS | Olympus | Microscope: Olympus Slide Scanner BX61VS, 5 slides scanner, motorized stage, autofocus. Camera: Lightsource: Xcite-120. Filters: BrightLine® Sedat filter set (# LED-DA/FI/TR/Cy5-4X4M-B-000, Semrock) | |
| Xylene | Sigma Aldrich | 214736-4L | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Xylene : Ethanol solution (1:1 v/v) | |||
| 2-mercaptoethanol | Sigma | M6250 | CAUTION, harmful by ingestion, inhalation, fatal if sking absortion. Eye irritation. Use fume hood |
References
- Greten, F. R., Grivennikov, S. I. Inflammation and Cancer:Triggers, Mechanisms, and Consequences. Immunity. 51 (1), 27-41 (2019).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Binnewies, M., et al. Understanding the tumor immune microenvironment (TIME) for effective therapy. Nature Medicine. 24 (5), 541-550 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathology: an official journal of the United States and Canadian Academy of Pathology, Inc. 31 (2), 214-234 (2018).
- Bindea, G., et al. Spatiotemporal dynamics of intratumoral immune cells reveal the immune landscape in human cancer. Immunity. 39 (4), 782-795 (2013).
- Galon, J., et al. Towards the introduction of the 'Immunoscore' in the classification of malignant tumours. The Journal of Pathology. 232 (2), 199-209 (2014).
- Finotello, F., Eduati, F. Multi-Omics Profiling of the Tumor Microenvironment: Paving the Way to Precision Immuno-Oncology. Frontiers in Oncology. 8, 430 (2018).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: a method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Porta Siegel, T., et al. Mass Spectrometry Imaging and Integration with Other Imaging Modalities for Greater Molecular Understanding of Biological Tissues. Molecular Imaging and Biology : MIB: the official publication of the Academy of Molecular Imaging. 20 (6), 888-901 (2018).
- Buchberger, A. R., DeLaney, K., Johnson, J., Li, L. Mass Spectrometry Imaging: A Review of Emerging Advancements and Future Insights. Analytical Chemistry. 90 (1), 240-265 (2018).
- Pirici, D., et al. Antibody elution method for multiple immunohistochemistry on primary antibodies raised in the same species and of the same subtype. Journal of Histochemistry and Cytochemistry. 57 (6), 567-575 (2009).
- Gendusa, R., Scalia, C. R., Buscone, S., Cattoretti, G. Elution of High-affinity (>10-9 KD) Antibodies from Tissue Sections: Clues to the Molecular Mechanism and Use in Sequential Immunostaining. Journal of Histochemistry and Cytochemistry. 62 (7), 519-531 (2014).
- van der Loos, C. M. Multiple immunoenzyme staining: methods and visualizations for the observation with spectral imaging. Journal of Histochemistry and Cytochemistry. 56 (4), 313-328 (2008).
- Stack, E. C., Wang, C., Roman, K. A., Hoyt, C. C. Multiplexed immunohistochemistry, imaging, and quantitation: a review, with an assessment of Tyramide signal amplification, multispectral imaging and multiplex analysis. Methods. 70 (1), 46-58 (2014).
- Toth, Z. E., Mezey, E. Simultaneous visualization of multiple antigens with tyramide signal amplification using antibodies from the same species. Journal of Histochemistry and Cytochemistry. 55 (6), 545-554 (2007).
- Robertson, D., Savage, K., Reis-Filho, J. S., Isacke, C. M. Multiple immunofluorescence labeling of formalin-fixed paraffin-embedded (FFPE) tissue. BMC Cell Biology. 9, 13 (2008).
- Segnani, C., et al. Histochemical Detection of Collagen Fibers by Sirius Red/Fast Green Is More Sensitive than van Gieson or Sirius Red Alone in Normal and Inflamed Rat Colon. PloS One. 10 (12), 0144630 (2015).
- Bolognesi, M. M., et al. Multiplex Staining by Sequential Immunostaining and Antibody Removal on Routine Tissue Sections. Journal of Histochemistry and Cytochemistry. 65 (8), 431-444 (2017).



