- 00:01Concepts
- 03:13gDNA Isolation and Quality Check
- 03:43Isolation of gDNA and gDNA Quality Check
- 05:52Amplification and Purification of 16S rRNA Gene by PCR
- 07:20Analysis of the DNA Sequences
- 09:02Sequence Assembly and Database Search
16S rRNA 시퀀싱: 박테리아 종 식별을위한 PCR 기반 기술
English
Share
Overview
출처: 에와 부코스카-패니반드1,틸드 앤더슨1,롤프 루드1
1 임상 과학 룬드학과, 감염 의학 학과, 생물 의학 센터, 룬드 대학, 221 00 룬드, 스웨덴
행성 지구는 세균 종의 수백만에 대한 서식지입니다, 각각의 특정 특성을 가지고. 세균성 종의 식별은 감염된 환자를 진단하기 위해 환경 샘플 및 의료 미생물학의 생물 다양성을 결정하기 위해 미생물 생태학에서 널리 사용됩니다. 박테리아는 현미경 검사법과 같은 기존의 미생물학 방법, 특정 매체에 대한 성장, 생화학 및 혈청 술 검사 및 항생제 민감성 분석을 사용하여 분류될 수 있습니다. 최근 수십 년 동안 분자 미생물학 방법은 세균 식별에 혁명을 일으켰습니다. 16S 리보소말 RNA(rRNA) 유전자 염기서열분석이 인기 있는 방법이다. 이 방법은 기존의 방법보다 빠르고 정확할 뿐만 아니라 실험실 조건에서 성장하기 어려운 균주를 식별할 수 있습니다. 더욱이, 분자 수준에서 균주의 분화는 표현형 동일 박테리아 (1-4)의 차별을 가능하게합니다.
16S rRNA는 세균성 리보솜(5)의 30S 하위 단위를 형성하기 위해 19개의 단백질의 복합체와 결합한다. 그것은 16S rRNA 유전자에 의해 인코딩된다, 이는 존재하고 리보솜 조립에 그것의 필수적인 기능때문에 모든 박테리아에 높게 보존; 그러나 특정 종에 대한 지문 역할을 할 수있는 가변 영역도 포함되어 있습니다. 이러한 특징은 16S rRNA 유전자를 박테리아의 식별, 비교 및 계통유전학 적 분류에 사용되는 이상적인 유전 적 단편으로 만들었습니다 (6).
16S rRNA 유전자 염기서열분석은 중합효소 연쇄 반응(PCR) (7-8)를 기반으로 DNA 염기서열 분석(9)을 선행한다. PCR은 다음과 같은 일련의 주기를 통해 DNA의 특정 단편을 증폭시키는 데 사용되는 분자 생물학 방법입니다.
i) 이중 좌초 된 DNA 템플릿의 거부
ii) 템플릿에 보완프라이머 (짧은 올리고 뉴클레오티드)의 안다골
iii) 새로운 DNA 가닥을 합성하는 DNA 폴리머라제 효소에 의한 프라이머 확장
메서드의 회로도 개요는 도 1에표시됩니다.
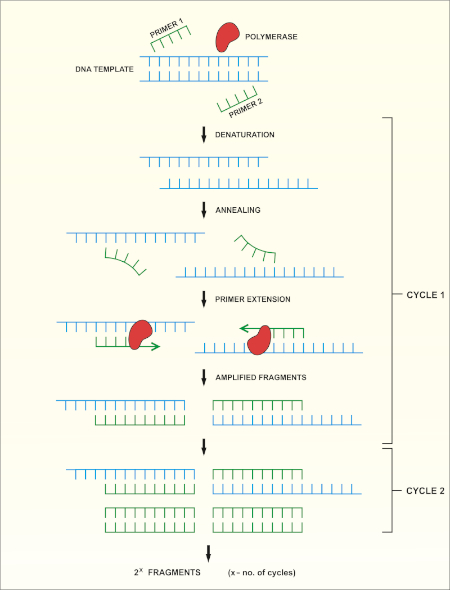
그림 1: PCR 반응의 회로도 개요. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
성공적인 PCR 반응에 중요한 몇 가지 요인이 있으며, 그 중 하나는 DNA 템플릿의 품질입니다. 박테리아로부터 염색체 DNA의 격리는 표준 프로토콜 또는 상업적 키트를 사용하여 수행될 수 있다. PCR 반응을 억제할 수 있는 오염물질이 없는 DNA를 얻기 위해 특별한 주의를 기울여야 한다.
16S rRNA 유전자의 보존된 영역은 임의의 세균종에서 표적 부위에 결합하고 증폭될 수 있는 범용 프라이머 쌍(1개의 전진 및 1개의 역)의 설계를 허용한다. 대상 영역의 크기가 다를 수 있습니다. 몇몇 프라이머 쌍은 16S rRNA 유전자의 대부분을 증폭할 수 있는 동안, 그 외는 그것의 단지 부분을 증폭합니다. 일반적으로 사용되는 프라이머의 예는 표 1에 표시되며 바인딩 사이트는 그림 2에표시됩니다.
| 프라이머 이름 | 시퀀스(5’→3′) | 전진/역방향 | 참조 |
| 8F b) | 아가그트가트CCTGGCTCAG | 전달 | -1 |
| 27F | AGAGTGATCMTGGCTCAG | 전달 | -10 |
| 515F | GTGCCAGCMGCCGCGGGTAA | 전달 | -11 |
| 911R | GCCCCCGTCAATTCMTTTGA | 후진 | -12 |
| 1391R | 가CGGGCGGTGTGTRCA | 후진 | -11 |
| 1492R | GGTTGTTACGACTT | 후진 | -11 |
표 1: 16S rRNA 유전자a)의증폭에 사용되는 표준 올리고뉴클레오티드의 예.
a) 상이한 프라이머 조합을 사용하여 생성된 PCR 제품의 예상 길이는 전방 및 역프라이머(그림 2 참조)에 대한 결합 부위 사이의 거리를 계산하여 추정할 수 있으며, 예를 들어 프라이머 쌍 8F-1492R을 사용하여 PCR 제품의 크기는 ~1500bp, 프라이머 쌍 27F-91R~90bp.90 bp.
b) fD1이라고도 함

그림 2: 16S rRNA 서열 및 프라이머 결합 부위의 대표적인 수치. 보존된 영역은 회색으로 표시되고 가변 영역은 대각선으로 채워져 있습니다. 가장 높은 해상도를 허용하기 위해, 프라이머 8F 및 1492R(rRNA 서열에 근거한 이름)는 전체 서열을 증폭시키는 데 사용되어 유전자의 여러 가변 영역의 시퀀싱을 허용한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
PCR에 대한 사이클링조건(즉, DNA가 변성되고, 프라이머로 어닐링되고, 합성되는 데 필요한 온도 및 시간)은 사용되는 폴리머라제의 종류와 프라이머의 특성에 의존한다. 특정 폴리머라제에 대한 제조업체의 지침을 따르는 것이 좋습니다.
PCR 프로그램이 완료되면, 제품은 아가로즈 젤 전기포광에 의해 분석된다. 성공적인 PCR은 예상 크기의 단일 밴드를 생성합니다. 제품은 PCR 반응에 존재했던 잔류 프라이머, 데옥시리보뉴클레오티드, 폴리머라제 및 완충물을 제거하기 위해 시퀀싱 전에 정제되어야 한다. 정제 된 DNA 단편은 일반적으로 상업적 시퀀싱 서비스에 시퀀싱을 위해 전송됩니다. 그러나 일부 기관은 자신의 핵심 시설에서 DNA 시퀀싱을 수행합니다.
DNA 서열은 컴퓨터에 의해 DNA 크로마토그램에서 자동으로 생성되며 수동 편집이 때로는 필요하기 때문에 품질을 주의 깊게 검사해야합니다. 이 단계에 이어, 유전자 서열은 16S rRNA 데이터베이스에 증착된 서열과 비교된다. 유사성 영역이 식별되고 가장 유사한 시퀀스가 전달됩니다.
Procedure
Applications and Summary
Identifying bacterial species is important for different researchers, as well as for those in healthcare. 16S rRNA sequencing was initially used by researchers to determine phylogenetic relationships between bacteria. In time, it has been implemented in metagenomic studies to determine biodiversity of environmental samples and in clinical laboratories as a method to identify potential pathogens. It enables a quick and accurate identification of bacteria present in clinical samples, facilitating earlier diagnosis and faster treatment of patients.
References
- Weisburg, W.G., Barns, S.M., Pelletier, D.A. and Lane D.J. 16S ribosomal DNA amplification for phylogenetic study. J Bacteriol. 173 (2): 697-703. (1991)
- Drancourt, M., Bollet, C., Carlioz, A., Martelin, R., Gayral, J.P., Raoult D. 16S ribosomal DNA sequence analysis of a large collection of environmental and clinical unidentifiable bacterial isolates. J Clin Microbiol. 38 (10):3623-3630. (2000)
- Woo, P.C., Lau, S.K., Teng, J.L., Tse, H., Yuen, K.Y. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories. Clin Microbiol Infect. 14 (10):908-934. (2008)
- Tang, Y.W., Ellis, N.M., Hopkins, M.K., Smith, D.H., Dodge, D.E., Persing, D.H. Comparison of phenotypic and genotypic techniques for identification of unusual aerobic pathogenic gram-negative bacilli. J Clin Microbiol. 36 (12):3674-3679. (1998)
- Tsiboli, P., Herfurth, E., Choli, T. Purification and characterization of the 30S ribosomal proteins from the bacterium Thermus thermophilus. Eur J Biochem. 226 (1):169-177. (1994)
- Woese, C.R. Bacterial evolution. Microbiol Rev. 51 (2):221-271. (1987)
- Bartlett, J.M., Stirling, D. A short history of the polymerase chain reaction. Methods Mol Biol. 226:3-6. (2003)
- Wilson, K.H., Blitchington, R.B., Greene, R.C. Amplification of bacterial 16S ribosomal DNA with polymerase chain reaction. J Clin Microbiol. 28 (9):1942-1946. (1990)
- Shendure, J., Balasubramanian, S., Church, G.M., Gilbert, W., Rogers, J., Schloss, J.A., Waterston, R.H. (2017) DNA sequencing at 40: past, present and future. Nature. 550:345-353.
- Lane, D.J. 16S/23S rRNA sequencing. (1991) In Nucleic acid techniques in bacterial systematics. (Goodfellow, M. and Stackebrandt, E., eds.) p.115-175. Wiley and Sons, Chichester, United Kingdom.
- Turner, S., Pryer, K.M., Miao, V.P., Palmer, J.D. (1999) Investigating deep phylogenetic relationships among cyanobacteria and plastids by small subunit rRNA sequence analysis. J Eukaryot Microbiol. 46:327-338.
- Fredricks, D.N., Relman, D.A. (1998) Improved amplification of microbial DNA from blood cultures by removal of the PCR inhibitor sodium polyanetholesulfonate. J Clin Microbiol. 36:2810-2816.
- Wilson, K. Preparation of genomic DNA from bacteria. (2001) Curr Protoc Mol Biol. Chapter 2:Unit 2.4.
- Wright, M. H., Adelskov, J., Greene, A.C. (2017) Bacterial DNA extraction using individual enzymes and phenol/chloroform separation. J Microbiol Biol Educ. 18:18.2.48.
- Huang, X., Madan, A. (1999). CAP3: A DNA sequence assembly program. Genome Res. 9:868-877.
Transcript
Earth is home to millions of bacterial species, each with unique characteristics. Identifying these species is critical in evaluating environmental samples. Doctors also need to distinguish different bacterial species to diagnose infected patients.
To identify bacteria, a variety of techniques can be employed, including microscopic observation of morphology or growth on a specific media to observe colony morphology. Genetic analysis, another technique for identifying bacteria has grown in popularity in recent years, due in part to 16S ribosomal RNA gene sequencing.
The bacterial ribosome is a protein RNA complex consisting of two subunits. The 30S subunit, the smaller of these two subunits, contains 16S rRNA, which is encoded by the 16S rRNA gene contained within the genomic DNA. Specific regions of 16S rRNA are highly conserved, due to their essential function in ribosome assembly. While other regions, less critical to function, may vary among bacterial species. The variable regions in 16S rRNA, can serve as unique molecular fingerprints for bacterial species, allowing us to distinguish phenotypically identical strains.
After obtaining a quality sample of gDNA, PCR of the 16S rRNA encoding gene can begin. PCR is a commonly used molecular biology method, consisting of cycles of denaturation of the double-stranded DNA template, annealing of universal primer pairs, which amplify highly conserved regions of the gene, and the extension of primers by DNA polymerase. While some primers amplify most of the 16S rRNA encoding gene, others only amplify fragments of it. After PCR, the products can be analyzed via agarose gel electrophoresis. If amplification was successful, the gel should contain a single band of an expected size, depending upon the primer pair used, up to 1500bp, the approximate length of the 16S rRNA gene.
After purification and sequencing, the obtained sequences can then be entered into the BLAST database, where they can be compared with reference 16S rRNA sequences. As this database returns matches based on the highest similarity, this allows confirmation of the identity of the bacteria of interest. In this video, you will observe 16S rRNA gene sequencing, including PCR, DNA sequence analysis and editing, sequence assembly and database searching.
When handling microorganisms, it is essential to follow good microbiological practice, including using aseptic technique and wearing appropriate personal protective equipment. After performing an appropriate risk assessment for the microorganism or environmental sample of interest, obtain a test culture. In this example, a pure culture of Bacillus subtilis is used.
To begin, grow your microorganism on a suitable medium in the appropriate conditions. In this example, Bacillus subtilis 168 is grown in LB broth overnight in a shaking incubator set to 200 rpm at 37 degrees Celsius. Next, use a commercially available kit to isolate genomic DNA or gDNA from 1.5 milliliters of the B. subtilis overnight culture.
To check the quality of the isolated DNA, first mix five microliters of the isolated gDNA with one microliter of DNA gel loading dye. Then, load the sample onto a 0.8% agarose gel, containing DNA staining reagent, such as SYBR safe or EtBr. After this, load a one kilobase molecular mass standard onto the gel, and run the electrophoresis until the front dye is approximately 0.5 centimeters from the bottom of the gel. Once the gel electrophoresis is complete, visualize the gel on a blue light transilluminator. The gDNA should appear as a thick band, above 10 kilobase in size and have minimal smearing.
After this, to create serial dilutions of the gDNA, label three microcentrifuge tubes as 10X, 100X, and 1000X. Then, use a pipette to dispense 90 microliters of sterile distilled water into each of the tubes. Next, add 10 microliters of the gDNA solution to the 10X tube. Pipette the whole volume up and down to ensure the solution is mixed thoroughly. Then, remove 10 microliters of the solution from the 10X tube and transfer this to the 100X tube. Mix the solution as previously described. Finally, transfer 10 microliters of the solution in the 100X tube, to the 1000X tube.
To begin the PCR protocol, thaw the necessary reagents on ice. Then, prepare the PCR master mix. Since the DNA polymerase is active at room temperature, the reaction set up must occur on ice. Aliquot 49 microliters of the master mix into each of the PCR tubes. Then, add one microliter of template to each of the experimental tubes and one microliter of sterile water to the negative control tube, pipetting up and down to mix. After this, set the PCR machine according to the program described in the table. Place the tubes into the thermocycler and start the program.
Once the program is complete, examine the quality of your product via agarose gel electrophoresis, as previously demonstrated. A successful reaction using the described protocol should yield a single band of approximately 1.5 kilobase. In this example, the sample containing 100X diluted gDNA yielded the highest quality product. Next, purify the best PCR product, in this case, the 100X gDNA, with a commercially available kit. Now the PCR product can be sent for sequencing.
In this example, the PCR product is sequenced using forward and reverse primers. Thus, two data sets, each containing a DNA sequence and a DNA chromatogram, are generated: one for the forward primer and the other for the reverse primer. First, examine the chromatograms generated from each primer. An ideal chromatogram should have evenly spaced peaks with little to no background signals.
If the chromatograms display double peaks, multiple DNA templates may have been present in the PCR products and the sequence should be discarded. If the chromatograms contained peaks of different colors in the same location, the sequencing software likely miscalled nucleotides. This error can be manually identified and corrected in the text file. The presence of broad peaks in the chromatogram indicates a loss of resolution, which causes miscounting of the nucleotides in the associated regions. This error is difficult to correct and mismatches in any of the subsequent steps should be treated as unreliable. Poor chromatogram reading quality, indicated by the presence of multiple peaks, usually occurs at the five prime and three prime ends of the sequence. Some sequencing programs remove these low quality sections automatically. If your sequence was not truncated automatically, identify the low quality fragments and remove their respective bases from the text file.
Use a DNA assembly program to assemble the two primer sequences into one continuous sequence. Remember, sequences obtained using forward and reverse primers should partially overlap. In the DNA assembly program, insert the two sequences in FASTA format into the appropriate box. Then, click the submit button and wait for the program to return the results.
To view the assembled sequence, click on Contigs in the results tab. Then, to view the details of the alignment, select assembly details. Navigate to the website for the basic local alignment search tool, or BLAST, and select the nucleotide BLAST tool to compare your sequence to the database. Enter your sequence into the query sequence text box and select the appropriate database in the scroll down menu. Finally, click the BLAST button on the bottom of the page, and wait for the tool to return the most similar sequences from the database.
In this example, the top hit is B. subtilis strain 168, showing 100% identity with the sequence in the BLAST database. If the top hit does not show 100% identity to your expected species or strain, click on the sequence which most closely matches your query to see the details of the alignment. Aligned nucleotides will be joined by short vertical lines and mismatched nucleotides will have gaps between them. Focusing on the identified mismatched regions, revise the sequence and repeat the BLAST search if desired.
