- 00:01Concepts
- 03:13gDNA Isolation and Quality Check
- 03:43Isolation of gDNA and gDNA Quality Check
- 05:52Amplification and Purification of 16S rRNA Gene by PCR
- 07:20Analysis of the DNA Sequences
- 09:02Sequence Assembly and Database Search
16S rRNA-Sequenzierung: Eine PCR-basierte Technik zur Identifizierung bakterieller Arten
English
Share
Overview
Quelle: Ewa Bukowska-Faniband1, Tilde Andersson1, Rolf Lood1
1 Institut für Klinische Wissenschaften Lund, Abteilung für Infektionsmedizin, Biomedical Center, Universität Lund, 221 00 Lund, Schweden
Planet Erde ist ein Lebensraum für Millionen von Bakterienarten, von denen jede spezifische Eigenschaften hat. Die Identifizierung von Bakterienarten wird in der mikrobiellen Ökologie häufig verwendet, um die Artenvielfalt von Umweltproben und medizinische Mikrobiologie zur Diagnose infizierter Patienten zu bestimmen. Bakterien können mit konventionellen mikrobiologischen Methoden wie Mikroskopie, Wachstum auf bestimmten Medien, biochemischen und serologischen Tests und Antibiotika-Empfindlichkeitstests klassifiziert werden. In den letzten Jahrzehnten haben molekulare mikrobiologische Methoden die bakterielle Identifizierung revolutioniert. Eine beliebte Methode ist die 16S ribosomale RNA (rRNA) Gensequenzierung. Diese Methode ist nicht nur schneller und genauer als herkömmliche Methoden, sondern ermöglicht auch die Identifizierung von Stämmen, die unter Laborbedingungen schwer zu züchten sind. Darüber hinaus ermöglicht die Differenzierung von Stämmen auf molekularer Ebene eine Diskriminierung von phänotypisch identischen Bakterien (1-4).
16S rRNA verbindet sich mit einem Komplex von 19 Proteinen zu einer 30S-Untereinheit des bakteriellen Ribosoms (5). Es wird durch das 16S rRNA-Gen kodiert, das in allen Bakterien aufgrund seiner wesentlichen Funktion in der Ribosom-Montage vorhanden und hochkonserviert ist; Es enthält jedoch auch variable Regionen, die als Fingerabdrücke für bestimmte Arten dienen können. Diese Eigenschaften haben das 16S rRNA-Gen zu einem idealen genetischen Fragment gemacht, das bei der Identifizierung, dem Vergleich und der phylogenetischen Klassifizierung von Bakterien verwendet werden kann (6).
16S rRNA Gensequenzierung basiert auf der Polymerase-Kettenreaktion (PCR) (7-8) gefolgt von DNA-Sequenzierung (9). PCR ist eine molekularbiologische Methode, die verwendet wird, um bestimmte DNA-Fragmente durch eine Reihe von Zyklen zu verstärken, die Folgendes umfassen:
i) Denaturierung einer doppelsträngigen DNA-Vorlage
ii) Glühen von Primern (kurze Oligonukleotide), die die Vorlage ergänzen
iii) Erweiterung der Primer durch das DNA-Polymerase-Enzym, das einen neuen DNA-Strang synthetisiert
Eine schematische Übersicht über die Methode ist in Abbildung 1dargestellt.
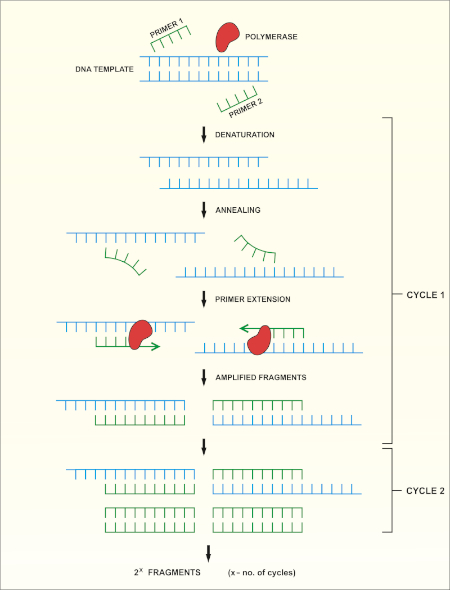
Abbildung 1: Schematische Übersicht über die PCR-Reaktion. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Es gibt mehrere Faktoren, die für eine erfolgreiche PCR-Reaktion wichtig sind, einer davon ist die Qualität der DNA-Vorlage. Die Isolierung chromosomaler DNA von Bakterien kann mit Standardprotokollen oder kommerziellen Kits durchgeführt werden. Besondere Vorsicht sollte darauf geachtet werden, DNA zu erhalten, die frei von Verunreinigungen ist, die die PCR-Reaktion hemmen können.
Konservierte Regionen des 16S rRNA-Gens ermöglichen das Design universeller Primerpaare (ein Vorwärts- und ein Rückwärtsgang), die die Zielregion in jeder Bakterienart binden und verstärken können. Die Zielregion kann in ihrer Größe variieren. Während einige Primerpaare den größten Teil des 16S rRNA-Gens verstärken können, verstärken andere nur Teile davon. Beispiele für häufig verwendete Primer sind in Tabelle 1 dargestellt und ihre Bindungsstellen sind in Abbildung 2dargestellt.
| Primername | Sequenz (5”3′) | Vorwärts/ Rückwärts | verweis |
| 8F b) | AGAGTGTGATCCTGGCTCAG | vorwärts | -1 |
| 27F | AGAGTGTGATCMTGGCTCAG | vorwärts | -10 |
| 515F | GTGCCAGCMGCCGCGGTAA | vorwärts | -11 |
| 911R | GCCCCCGTCAATTCMTGA | Rückwärts | -12 |
| 1391R | GACGGGCGGTGTGTRCA | Rückwärts | -11 |
| 1492R | GGTTTTTTTACGACTT | Rückwärts | -11 |
Tabelle 1: Beispiele für Standardoligonukleotide, die bei der Amplifikation von 16S rRNA-Genen verwendet werden a).
a) Die erwarteten Längen des PCR-Produkts, das mit den verschiedenen Primerkombinationen erzeugt wird, können durch Berechnung des Abstands zwischen den Bindungsstellen für den Vorwärts- und den Reverse-Primer geschätzt werden (siehe Abbildung 2), z. B. die Größe der PCR Produkt mit Primer-Paar 8F-1492R ist 1500 bp, und für Primer-Paar 27F-911R 900 bp.
b) auch bekannt als fD1

Abbildung 2: Repräsentative Abbildung der 16S rRNA-Sequenz und der Primer-Bindungsstellen. Konservierte Bereiche sind grau gefärbt und variable Bereiche sind mit diagonalen Linien gefüllt. Um die höchste Auflösung zu ermöglichen, werden Primer 8F und 1492R (Name basierend auf der Position auf der rRNA-Sequenz) verwendet, um die gesamte Sequenz zu verstärken, was die Sequenzierung mehrerer variabler Bereiche des Gens ermöglicht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die Fahrradbedingungen für PCR(d. h. die Temperatur und Die Zeit, die für die Denaturierung, das Geglüht mit Primern und synthetisiert erforderlich sind) hängen von der Verwendeten-Polymerase-Art und den Eigenschaften der Primer ab. Es wird empfohlen, die Herstellerrichtlinien für eine bestimmte Polymerase zu befolgen.
Nach Abschluss des PCR-Programms werden die Produkte durch Agarose-Gel-Elektrophorese analysiert. Eine erfolgreiche PCR ergibt ein einzelnes Band in der erwarteten Größe. Das Produkt muss vor der Sequenzierung gereinigt werden, um Restgrundierungen, Desoxyribonukleotide, Polymerase und Puffer zu entfernen, die in der PCR-Reaktion vorhanden waren. Die gereinigten DNA-Fragmente werden in der Regel zur Sequenzierung an kommerzielle Sequenzierungsdienste gesendet; Einige Institutionen führen jedoch DNA-Sequenzierungen in ihren eigenen Kerneinrichtungen durch.
Die DNA-Sequenz wird automatisch aus einem DNA-Chromatogramm von einem Computer erzeugt und muss sorgfältig auf Qualität überprüft werden, da manchmal eine manuelle Bearbeitung erforderlich ist. Nach diesem Schritt wird die Gensequenz mit Sequenzen verglichen, die in der 16S rRNA-Datenbank abgelagert sind. Die Regionen mit Ähnlichkeit werden identifiziert, und die ähnlichsten Sequenzen werden geliefert.
Procedure
Applications and Summary
Identifying bacterial species is important for different researchers, as well as for those in healthcare. 16S rRNA sequencing was initially used by researchers to determine phylogenetic relationships between bacteria. In time, it has been implemented in metagenomic studies to determine biodiversity of environmental samples and in clinical laboratories as a method to identify potential pathogens. It enables a quick and accurate identification of bacteria present in clinical samples, facilitating earlier diagnosis and faster treatment of patients.
References
- Weisburg, W.G., Barns, S.M., Pelletier, D.A. and Lane D.J. 16S ribosomal DNA amplification for phylogenetic study. J Bacteriol. 173 (2): 697-703. (1991)
- Drancourt, M., Bollet, C., Carlioz, A., Martelin, R., Gayral, J.P., Raoult D. 16S ribosomal DNA sequence analysis of a large collection of environmental and clinical unidentifiable bacterial isolates. J Clin Microbiol. 38 (10):3623-3630. (2000)
- Woo, P.C., Lau, S.K., Teng, J.L., Tse, H., Yuen, K.Y. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories. Clin Microbiol Infect. 14 (10):908-934. (2008)
- Tang, Y.W., Ellis, N.M., Hopkins, M.K., Smith, D.H., Dodge, D.E., Persing, D.H. Comparison of phenotypic and genotypic techniques for identification of unusual aerobic pathogenic gram-negative bacilli. J Clin Microbiol. 36 (12):3674-3679. (1998)
- Tsiboli, P., Herfurth, E., Choli, T. Purification and characterization of the 30S ribosomal proteins from the bacterium Thermus thermophilus. Eur J Biochem. 226 (1):169-177. (1994)
- Woese, C.R. Bacterial evolution. Microbiol Rev. 51 (2):221-271. (1987)
- Bartlett, J.M., Stirling, D. A short history of the polymerase chain reaction. Methods Mol Biol. 226:3-6. (2003)
- Wilson, K.H., Blitchington, R.B., Greene, R.C. Amplification of bacterial 16S ribosomal DNA with polymerase chain reaction. J Clin Microbiol. 28 (9):1942-1946. (1990)
- Shendure, J., Balasubramanian, S., Church, G.M., Gilbert, W., Rogers, J., Schloss, J.A., Waterston, R.H. (2017) DNA sequencing at 40: past, present and future. Nature. 550:345-353.
- Lane, D.J. 16S/23S rRNA sequencing. (1991) In Nucleic acid techniques in bacterial systematics. (Goodfellow, M. and Stackebrandt, E., eds.) p.115-175. Wiley and Sons, Chichester, United Kingdom.
- Turner, S., Pryer, K.M., Miao, V.P., Palmer, J.D. (1999) Investigating deep phylogenetic relationships among cyanobacteria and plastids by small subunit rRNA sequence analysis. J Eukaryot Microbiol. 46:327-338.
- Fredricks, D.N., Relman, D.A. (1998) Improved amplification of microbial DNA from blood cultures by removal of the PCR inhibitor sodium polyanetholesulfonate. J Clin Microbiol. 36:2810-2816.
- Wilson, K. Preparation of genomic DNA from bacteria. (2001) Curr Protoc Mol Biol. Chapter 2:Unit 2.4.
- Wright, M. H., Adelskov, J., Greene, A.C. (2017) Bacterial DNA extraction using individual enzymes and phenol/chloroform separation. J Microbiol Biol Educ. 18:18.2.48.
- Huang, X., Madan, A. (1999). CAP3: A DNA sequence assembly program. Genome Res. 9:868-877.
Transcript
Earth is home to millions of bacterial species, each with unique characteristics. Identifying these species is critical in evaluating environmental samples. Doctors also need to distinguish different bacterial species to diagnose infected patients.
To identify bacteria, a variety of techniques can be employed, including microscopic observation of morphology or growth on a specific media to observe colony morphology. Genetic analysis, another technique for identifying bacteria has grown in popularity in recent years, due in part to 16S ribosomal RNA gene sequencing.
The bacterial ribosome is a protein RNA complex consisting of two subunits. The 30S subunit, the smaller of these two subunits, contains 16S rRNA, which is encoded by the 16S rRNA gene contained within the genomic DNA. Specific regions of 16S rRNA are highly conserved, due to their essential function in ribosome assembly. While other regions, less critical to function, may vary among bacterial species. The variable regions in 16S rRNA, can serve as unique molecular fingerprints for bacterial species, allowing us to distinguish phenotypically identical strains.
After obtaining a quality sample of gDNA, PCR of the 16S rRNA encoding gene can begin. PCR is a commonly used molecular biology method, consisting of cycles of denaturation of the double-stranded DNA template, annealing of universal primer pairs, which amplify highly conserved regions of the gene, and the extension of primers by DNA polymerase. While some primers amplify most of the 16S rRNA encoding gene, others only amplify fragments of it. After PCR, the products can be analyzed via agarose gel electrophoresis. If amplification was successful, the gel should contain a single band of an expected size, depending upon the primer pair used, up to 1500bp, the approximate length of the 16S rRNA gene.
After purification and sequencing, the obtained sequences can then be entered into the BLAST database, where they can be compared with reference 16S rRNA sequences. As this database returns matches based on the highest similarity, this allows confirmation of the identity of the bacteria of interest. In this video, you will observe 16S rRNA gene sequencing, including PCR, DNA sequence analysis and editing, sequence assembly and database searching.
When handling microorganisms, it is essential to follow good microbiological practice, including using aseptic technique and wearing appropriate personal protective equipment. After performing an appropriate risk assessment for the microorganism or environmental sample of interest, obtain a test culture. In this example, a pure culture of Bacillus subtilis is used.
To begin, grow your microorganism on a suitable medium in the appropriate conditions. In this example, Bacillus subtilis 168 is grown in LB broth overnight in a shaking incubator set to 200 rpm at 37 degrees Celsius. Next, use a commercially available kit to isolate genomic DNA or gDNA from 1.5 milliliters of the B. subtilis overnight culture.
To check the quality of the isolated DNA, first mix five microliters of the isolated gDNA with one microliter of DNA gel loading dye. Then, load the sample onto a 0.8% agarose gel, containing DNA staining reagent, such as SYBR safe or EtBr. After this, load a one kilobase molecular mass standard onto the gel, and run the electrophoresis until the front dye is approximately 0.5 centimeters from the bottom of the gel. Once the gel electrophoresis is complete, visualize the gel on a blue light transilluminator. The gDNA should appear as a thick band, above 10 kilobase in size and have minimal smearing.
After this, to create serial dilutions of the gDNA, label three microcentrifuge tubes as 10X, 100X, and 1000X. Then, use a pipette to dispense 90 microliters of sterile distilled water into each of the tubes. Next, add 10 microliters of the gDNA solution to the 10X tube. Pipette the whole volume up and down to ensure the solution is mixed thoroughly. Then, remove 10 microliters of the solution from the 10X tube and transfer this to the 100X tube. Mix the solution as previously described. Finally, transfer 10 microliters of the solution in the 100X tube, to the 1000X tube.
To begin the PCR protocol, thaw the necessary reagents on ice. Then, prepare the PCR master mix. Since the DNA polymerase is active at room temperature, the reaction set up must occur on ice. Aliquot 49 microliters of the master mix into each of the PCR tubes. Then, add one microliter of template to each of the experimental tubes and one microliter of sterile water to the negative control tube, pipetting up and down to mix. After this, set the PCR machine according to the program described in the table. Place the tubes into the thermocycler and start the program.
Once the program is complete, examine the quality of your product via agarose gel electrophoresis, as previously demonstrated. A successful reaction using the described protocol should yield a single band of approximately 1.5 kilobase. In this example, the sample containing 100X diluted gDNA yielded the highest quality product. Next, purify the best PCR product, in this case, the 100X gDNA, with a commercially available kit. Now the PCR product can be sent for sequencing.
In this example, the PCR product is sequenced using forward and reverse primers. Thus, two data sets, each containing a DNA sequence and a DNA chromatogram, are generated: one for the forward primer and the other for the reverse primer. First, examine the chromatograms generated from each primer. An ideal chromatogram should have evenly spaced peaks with little to no background signals.
If the chromatograms display double peaks, multiple DNA templates may have been present in the PCR products and the sequence should be discarded. If the chromatograms contained peaks of different colors in the same location, the sequencing software likely miscalled nucleotides. This error can be manually identified and corrected in the text file. The presence of broad peaks in the chromatogram indicates a loss of resolution, which causes miscounting of the nucleotides in the associated regions. This error is difficult to correct and mismatches in any of the subsequent steps should be treated as unreliable. Poor chromatogram reading quality, indicated by the presence of multiple peaks, usually occurs at the five prime and three prime ends of the sequence. Some sequencing programs remove these low quality sections automatically. If your sequence was not truncated automatically, identify the low quality fragments and remove their respective bases from the text file.
Use a DNA assembly program to assemble the two primer sequences into one continuous sequence. Remember, sequences obtained using forward and reverse primers should partially overlap. In the DNA assembly program, insert the two sequences in FASTA format into the appropriate box. Then, click the submit button and wait for the program to return the results.
To view the assembled sequence, click on Contigs in the results tab. Then, to view the details of the alignment, select assembly details. Navigate to the website for the basic local alignment search tool, or BLAST, and select the nucleotide BLAST tool to compare your sequence to the database. Enter your sequence into the query sequence text box and select the appropriate database in the scroll down menu. Finally, click the BLAST button on the bottom of the page, and wait for the tool to return the most similar sequences from the database.
In this example, the top hit is B. subtilis strain 168, showing 100% identity with the sequence in the BLAST database. If the top hit does not show 100% identity to your expected species or strain, click on the sequence which most closely matches your query to see the details of the alignment. Aligned nucleotides will be joined by short vertical lines and mismatched nucleotides will have gaps between them. Focusing on the identified mismatched regions, revise the sequence and repeat the BLAST search if desired.
