Overview
Fonte: Andrew J. Van Alst1, Rhiannon M. LeVeque1, Natalia Martin1e Victor J. DiRita1
1 Dipartimento di Microbiologia e Genetica Molecolare, Michigan State University, East Lansing, Michigan, Stati Uniti d'America
Le curve di crescita forniscono preziose informazioni sulla cinetica di crescita batterica e sulla fisiologia cellulare. Ci permettono di determinare come i batteri rispondono in condizioni di crescita variabili e di definire parametri di crescita ottimali per un determinato batterio. Una curva di crescita archetipica progredisce attraverso quattro fasi di crescita: ritardo, esponenziale, stazionaria e morte (1).
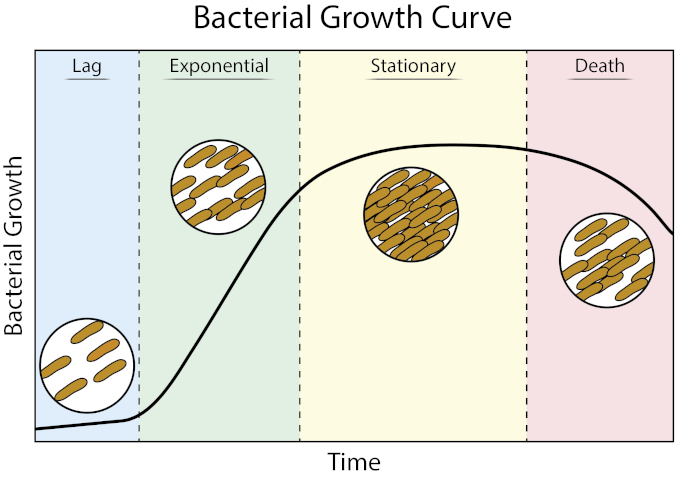
Figura 1: Curva di crescita batterica. I batteri cresciuti in coltura batch progrediscono attraverso quattro fasi di crescita: ritardo, esponenziale, stazionario e morte. La fase di ritardo è il periodo di tempo necessario ai batteri per raggiungere uno stato fisiologico in grado di una rapida crescita e divisione cellulare. La fase esponenziale è lo stadio di crescita e divisione cellulare più veloce durante il quale la replicazione del DNA, la trascrizione dell'RNA e la produzione di proteine avvengono tutte a un ritmo costante e rapido. La fase stazionaria è caratterizzata da un rallentamento e plateau della crescita batterica dovuto alla limitazione dei nutrienti e/o all'accumulo intermedio tossico. La fase di morte è la fase durante la quale si verifica la lisi cellulare a causa di una grave limitazione dei nutrienti.
La fase di ritardo è il periodo di tempo necessario ai batteri per raggiungere uno stato fisiologico in grado di una rapida crescita e divisione cellulare. Questo ritardo si verifica perché ci vuole tempo perché i batteri si adattino al loro nuovo ambiente. Una volta che i componenti cellulari necessari sono generati in fase di ritardo, i batteri entrano nella fase esponenziale di crescita in cui la replicazione del DNA, la trascrizione dell'RNA e la produzione di proteine avvengono tutte a un ritmo costante e rapido (2). Il tasso di rapida crescita e divisione cellulare durante la fase esponenziale è calcolato come il tempo di generazione, o tempo di raddoppio, ed è il tasso più veloce al quale i batteri possono replicarsi nelle condizioni date (1). Il tempo di raddoppio può essere utilizzato per confrontare diverse condizioni di crescita per determinare quale è più favorevole per la crescita batterica. La fase di crescita esponenziale è la condizione di crescita più riproducibile in quanto la fisiologia delle cellule batteriche è coerente in tutta la popolazione (3). La fase stazionaria segue la fase esponenziale in cui la crescita cellulare si stabilizze. La fase stazionaria è causata dall'esaurimento dei nutrienti e / o dall'accumulo di intermedi tossici. Le cellule batteriche continuano a sopravvivere in questa fase, anche se il tasso di replicazione e divisione cellulare è drasticamente ridotto. La fase finale è la morte, dove un grave esaurimento dei nutrienti porta alla lisi delle cellule. Le caratteristiche della curva di crescita che forniscono la maggior parte delle informazioni includono la durata della fase di ritardo, il tempo di raddoppio e la densità massima della cella raggiunta.
La quantificazione dei batteri in coltura batch può essere determinata utilizzando sia le unità formanti colonie che le misurazioni ottiche della densità. L'enumerazione per unità formanti colonie (CFU) fornisce una misurazione diretta della conta delle cellule batteriche. L'unità di misura standard per i CFU è il numero di batteri coltivabili presenti per 1 mL di coltura (CFU/mL) determinato mediante tecniche di diluizione seriale e placcatura diffusa. Per ogni punto temporale, viene eseguita una serie di diluizione 1:10 della coltura batch e 100 μl di ciascuna diluizione vengono distribuiti placcati utilizzando uno spandiconci cella.
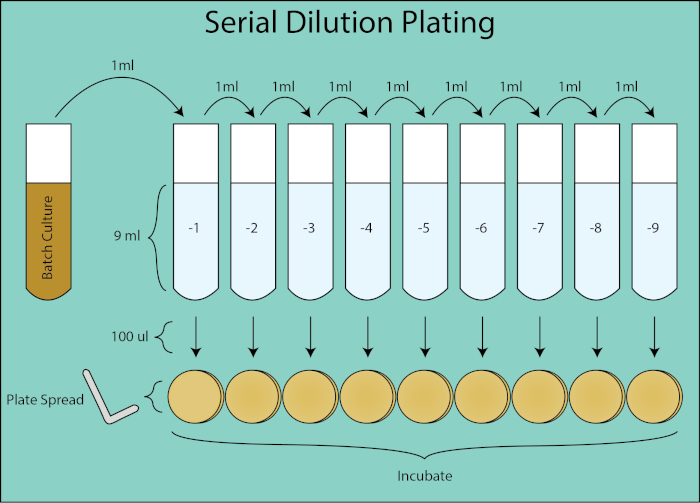
Figura 2. Schema di placcatura a diluizione seriale. Flusso generale per la placcatura di diluizione da coltura batch. La coltura del lotto viene diluita in serie 1:10 trasferendo 1 mL della diluizione precedente nel tubo successivo contenente 9 ml di PBS. Da ogni tubo di diluizione, 100 μl vengono distribuiti placcati utilizzando uno spandipiatti che è una diluizione aggiuntiva di 1:10 in quanto è 1/10 del volume di 1 mL di volume nel calcolo di CFU / mL. Le piastre vengono incubate ed enumerate una volta che le colonie clonali crescono sulle piastre.
Le placche vengono quindi incubate durante la notte e le colonie clonali enumerate. La piastra di diluizione che ha coltivato 30-300 colonie viene utilizzata per calcolare il CFU/ mL per il punto temporale dato (4, 5). La variazione stocastica nel conteggio delle colonie inferiore a 30 è soggetta a un errore maggiore nel calcolo dei CFU/mL e il conteggio delle colonie superiori a 300 può essere sottovalutato a causa dell'affollamento e della sovrapposizione delle colonie. Utilizzando il fattore di diluizione per la piastra data, è possibile calcolare il CFU della coltura batch per ciascun punto temporale.
La densità ottica fornisce un'approssimazione istantanea della conta cellulare batterica misurata utilizzando uno spettrofotometro. La densità ottica è una misura dell'assorbanza delle particelle di luce che passano attraverso 1 cm di coltura e rilevate da un sensore di fotodiodi (6). La densità ottica di una coltura viene misurata in relazione a un mezzo vuoto e aumenta all'aumentare della densità batterica. Per le cellule batteriche, una lunghezza d'onda di 600 nm (OD600) viene in genere utilizzata quando si misura la densità ottica (4). Generando una curva standard relativa alle unità formanti colonie e alla densità ottica, la misurazione della densità ottica può essere utilizzata per approssimare facilmente il conteggio delle cellule batteriche di una coltura batch. Tuttavia, questa relazione inizia a deteriorarsi già a 0,3 OD600 quando le cellule iniziano a cambiare forma e ad accumulare prodotti extracellulari nel mezzo, influenzando la lettura della densità ottica in relazione ai CFU (7). Questo errore diventa più pronunciato durante le fasi stazionarie e di morte.
Qui, L'Escherichia coli viene coltivata in brodo Luria-Bertani (LB) a 37°C nel corso di 30 ore (7). Sono state generate sia curve di crescita CFU/mL che curve di crescita della densità ottica, nonché la curva standard che correla la densità ottica con cFU.
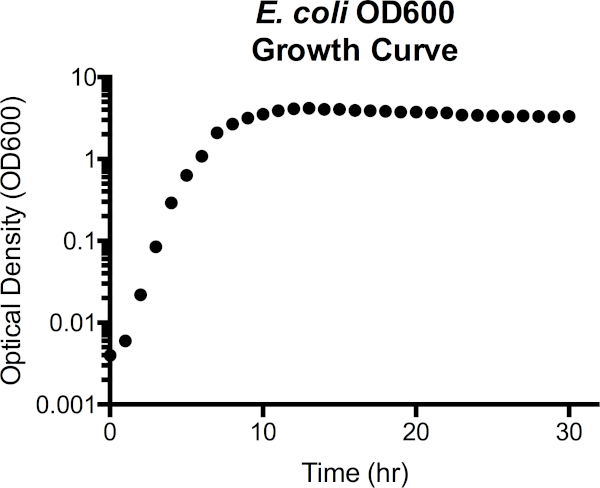
Figura 3. Densità ottica di Escherichia coli alla curva di crescita della lunghezza d'onda di 600 nm (OD600). I valori di densità ottica sono stati prelevati direttamente dallo spettrofotometro dopo l'oscuramento con supporti LB sterili. I valori di OD600 superiori a 1,0 sono stati diluiti 1:10 combinando la coltura di 100 μl con LB fresco di 900 μl, nuovamente misurato, e quindi moltiplicato per 10 per ottenere il valore OD600. Questo passo viene fatto quando la precisione nella misurazione dello spettrofotometro viene ridotta ad alta densità di cellule. Dalla curva, la fase di ritardo si estende a circa 1 ora di crescita, passa alla fase esponenziale da 2h a 7h, quindi inizia a stabilizzarsi, entrando in fase stazionaria. La fase di morte non è una transizione netta, tuttavia, poiché la densità ottica inizia gradualmente a diminuire dopo 15 ore.

Figura 4. Unità formante colonia di Escherichia coli per millilitro (CFU/mL) curva di crescita. I valori di CFU/mL per ogni timepoint sono stati calcolati dalla piastra di diluizione che conteneva 30-300 colonie. Dalla curva, la fase di ritardo si estende fino a circa 2 ore di crescita, passa alla fase esponenziale da 2h a 7h, quindi inizia a stabilizzarsi, entrando in fase stazionaria. La fase di morte non è una transizione netta, tuttavia, poiché CFU / mL inizia gradualmente a diminuire dopo 15 ore da un picco di 2 x 109 a circa 5 x 108 a 30 ore.
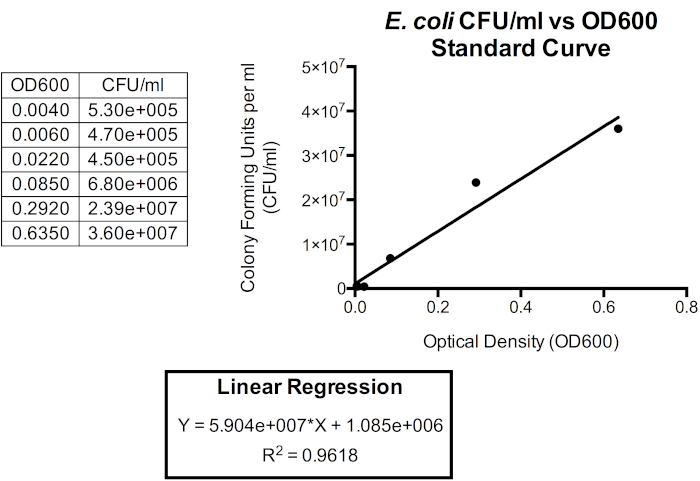
Figura 5. Curva di standardizzazione per CFU/mL rispetto a OD600. Una regressione lineare può essere utilizzata per correlare queste unità in modo che la densità ottica possa essere utilizzata per approssimare la densità cellulare batterica. La densità ottica può essere utilizzata per fornire un'approssimazione istantanea del CFU/mL della coltura batch. Qui, vengono tracciati solo i primi sei punti temporali poiché la relazione tra OD600 e CFU / mL è meno accurata oltre 1,0 OD600 poiché la forma cellulare e i prodotti extracellulari iniziano ad accumularsi quando i batteri entrano in fase stazionaria, che si verifica poco dopo aver raggiunto 1,0 OD600. I cambiamenti nella forma cellulare e nei prodotti extracellulari nei media influenzano la lettura della densità ottica e quindi anche la relazione tra densità ottica e numero di batteri nella coltura è influenzata.
Anche il tempo di raddoppio è stato determinato in 15 minuti e 19 secondi. Da questi dati, la capacità di crescita in LB per E. coli può essere visualizzata ed essere utilizzata per il confronto tra diversi mezzi o batteri.
Procedure
1. Configurazione
- Materiali di laboratorio richiesti: mezzi liquidi, mezzi di agar solidificati, palloni Erlenmeyer, provette da 15 ml, soluzione salina tamponata con fosfato (PBS), diffusore di cellule batteriche, etanolo al 70% e uno spettrofotometro. Tutte le soluzioni e i bicchieri devono essere sterilizzati prima dell'uso.
- Preparare la postazione di lavoro sterilizzando con etanolo al 70%. Lavora vicino a un bruciatore Bunsen per prevenire la contaminazione dei supporti.
- Quando si lavora con i batteri, devono essere utilizzati adeguati dispositivi di protezione individuale e tecnica asettica. Un cappotto da laboratorio e guanti sono necessari quando si lavora con colture batteriche.
- Ricette per tamponi, soluzioni e reagenti
- Soluzione salina tamponata con fosfato (PBS) (8).
- Brodo Luria-Bertani (LB) (9).
2. Protocollo
- Preparazione dei media
- Identificare il mezzo di crescita con cui far crescere i batteri e preparare sia il brodo liquido che il mezzo di agar solido (1,5% p/v agar) in flaconi autoclavabili separati. Qui, il brodo LB e l'agar LB sono stati preparati per la crescita di Escherichia coli.
- Sterilizzare il materiale con un tappo semisertato in autoclave impostato a 121 °C per 35 min.
- Per i mezzi di agar, dopo l'autoclave, porre a bagnomaria impostato a 50 °C per 30 minuti per raffreddare. Una volta raffreddato, versare 20-25 mL di agar media in piastre di Petri circolari 100x15mm. Lasciare impostare le piastre 24 ore a temperatura ambiente prima dell'uso.
- Preparazione iniziale dei batteri
- Da stock congelati, strisciare batteri per l'isolamento su agar media selezionati per ottenere isolati di singole colonie. Incubare in condizioni di crescita consentite per i batteri scelti. Qui, E. coli viene striato su LB agar e viene incubato a 37 ° C durante la notte (16-18h).
- Utilizzando un anello di inoculazione sterile, selezionare una singola colonia dalla piastra striata e inoculare 4 mL di liquido in una provetta da 15 ml e crescere in condizioni consentite per i batteri scelti. Qui, E. coli viene coltivato a 37 °C con agitazione a 210 giri / min durante la notte (16-18h).
- Impostazione della curva di crescita
- Preparazione del pallone di crescita
- Palloni Erlenmeyer di dimensioni adeguate per autoclave. In genere, viene utilizzato un rapporto 1:5 tra il supporto e il volume totale del pallone. Qui, 100 mL LB media viene utilizzato in un pallone da 500 ml.
- Utilizzando una pipetta sierologica, trasferire il mezzo sterile al pallone Erlenmeyer.
- Preparazione della serie di diluizione
- Etichetta 15 mL di provette: -1, -2, -3, -4, -5, -6, -7, -8 e -9, distribuendo 9 ml di PBS in ciascuna. Questi numeri corrispondono al fattore di diluizione utilizzato per calcolare CFU/mL. È necessario un nuovo set di tubi per ogni timepoint di raccolta. (Figura 2)
- Preparazione del piatto di agar
- Targhette etichettanti con tempo di raccolta e fattore di diluizione. Per ogni timepoint ci sarà una piastra per ogni diluizione.
- Preparazione del pallone di crescita
- Protocollo della curva di crescita
- Inoculazione dei media
- Utilizzando la coltura liquida durante la notte preparata come parte del punto 2.2.2, inoculare il mezzo del pallone con un volume di coltura di 1:1000. Qui, 100 μL di coltura liquida durante la notte vengono aggiunti a 100 mL di liquido LB.
- Ruotare i media per distribuire uniformemente i batteri.
- Raccolta Timepoint
- Impostazione delle condizioni di crescita
- Posizionare il pallone in condizioni di crescita sperimentali scelte per i batteri dati. I timepoint dovrebbero essere presi frequentemente per i batteri a crescita rapida e possono essere presi a intervalli più lunghi per i batteri a crescita lenta. Qui, E. coli viene coltivato a 37 ° C con agitazione a 210 giri al minuto (rpm) e timepoint presi ogni 1 ora.
- Misurazione della densità ottica (OD600)
- In ogni punto temporale, incluso il timepoint di partenza (t = 0), prelevare 1 mL di coltura batterica ed erogare in una cuvetta spettrofotometrica.
- Pulire la cuvetta e registrare la densità ottica a una lunghezza d'onda di 600 nm. Se la lettura della densità ottica è maggiore di 1,0, diluire 100 μL di coltura 1:10 con 900 μL di materiale fresco, registrare la densità ottica e moltiplicare questo valore per 10 per la misurazione OD600.
- Misurazione dell'unità formante colonie (CFU/mL)
- In ogni momento, prelevare 1 mL di coltura batterica ed erogare nella provetta di vetro -1 contenente 9 mL di PBS.
- Per la serie di diluizione, trasferire in serie 1 mL dal tubo -1 lungo tutti i tubi di diluizione al -9, vorticosamente dopo ogni trasferimento. (Figura 2)
- Per ogni diluizione, erogare 100 μL di sospensione cellulare alla piastra di agar del supporto solido marcata in modo corrispondente. (Figura 2)
- Utilizzando uno spandiconcime che è stato sterilizzato in etanolo, passato attraverso una fiamma del bruciatore Bunsen e raffreddato toccando la superficie dell'agar, distribuire i 100 μL di sospensione cellulare fino a quando la superficie della piastra di agar diventa asciutta.
- Incubare le piastre di diffusione capovolte a una temperatura che supporti la crescita dei batteri. Qui, E. coli viene incubato a 37°C.
- Dopo l'incubazione, una volta che sorgono colonie visibili, contare il numero di colonie batteriche su ciascuna piastra e registrare questi valori insieme al fattore di diluizione associato per tutte le piastre in ogni punto temporale.
- Impostazione delle condizioni di crescita
- Inoculazione dei media
3. Analisi dei dati e risultati
- Grafico della curva di crescita della densità ottica (OD600)
- Traccia la densità ottica (OD600) rispetto al tempo su una scala semi-logaritmo. (Figura 3)
- Grafico della curva di crescita dell'unità formante colonia (CFU/mL)
- Per ogni punto temporale, scegli la piastra di diluizione in cui i conteggi delle colonie rientravano nell'intervallo di 30-300 batteri. Moltiplicare il numero di conteggi delle colonie per il fattore di diluizione e quindi per 10 poiché la diffusione di 100 μL è considerata un'ulteriore diluizione 1:10 nel calcolo di CFU/ mL.

- Traccia le unità che formano la colonia rispetto al tempo su una scala semi-logaritmo. (Figura 4)
- Per ogni punto temporale, scegli la piastra di diluizione in cui i conteggi delle colonie rientravano nell'intervallo di 30-300 batteri. Moltiplicare il numero di conteggi delle colonie per il fattore di diluizione e quindi per 10 poiché la diffusione di 100 μL è considerata un'ulteriore diluizione 1:10 nel calcolo di CFU/ mL.
- Correlazione tra densità ottica e unità formanti colonie
- Tracciare le unità formanti la colonia rispetto alla densità ottica su una scala lineare per letture OD600 inferiori o uguali a 1,0 OD600 poiché la relazione tra OD600 e CFU / mL è meno accurata oltre 1,0 OD600. Qui vengono tracciati i primi sei punti temporali. (Figura 5)
- Generare una linea di tendenza di regressione lineare che visualizza l'equazione e il valore R2.
- Determinazione del tempo di raddoppio dei batteri
- Utilizzando il grafico della curva di crescita dell'unità di formazione della colonia, durante la fase esponenziale, identificare due punti sul grafico con la pendenza più ripida tra loro per calcolare il tempo di raddoppio.
- Calcolo del tempo di raddoppio

- ΔTime = t2 - t1, dove t1 = Timepoint 1 e t2 = Timepoint 2
-
 , dove b = numero di batteri a t2, B = numero di batteri a t1e n = numero di generazioni. Derivato da:
, dove b = numero di batteri a t2, B = numero di batteri a t1e n = numero di generazioni. Derivato da: .
- Calcola il tempo di raddoppio utilizzando:

I batteri si riproducono attraverso un processo chiamato divisione cellulare, che si traduce in due cellule figlie identiche. Se le condizioni di crescita sono favorevoli, le popolazioni batteriche cresceranno in modo esponenziale.
Le curve di crescita batterica tracciano la quantità di batteri in una coltura in funzione del tempo. Una tipica curva di crescita progredisce attraverso quattro fasi: fase di ritardo, fase esponenziale, fase stazionaria e fase di morte. La fase di ritardo è il tempo necessario ai batteri per raggiungere uno stato in cui possono crescere e dividersi rapidamente. Dopo questo, i batteri passano alla fase esponenziale, caratterizzata da una rapida crescita e divisione cellulare. Il tasso di crescita esponenziale della coltura batterica durante questa fase può essere espresso come il tempo di raddoppio, il tasso più veloce al quale i batteri possono riprodursi in condizioni specifiche. La fase stazionaria viene dopo, dove la crescita delle cellule batteriche si stabilizzano e i tassi di crescita e mortalità si uniformano a causa dell'esaurimento dei nutrienti ambientali. Infine, i batteri entrano nella fase di morte. È qui che la crescita batterica diminuisce bruscamente e un grave esaurimento dei nutrienti porta alla lisi delle cellule.
Due tecniche possono essere utilizzate per quantificare la quantità di batteri presenti in una coltura e tracciare una curva di crescita. Il primo di questi è attraverso unità formanti colonie, o CFU. Per ottenere CFU viene eseguita una serie da una a dieci di nove diluizioni in punti temporali regolari. La prima di queste diluizioni, negativa in questo esempio, contiene 9mL di PBS e 1mL di coltura batterica. Risultante in un fattore di diluizione 1:10. Quindi, 1 mL di questa soluzione viene trasferito al tubo successivo, negativo due, con conseguente fattore di diluizione 1:100. Questo processo continua attraverso l'ultimo tubo, negativo nove, con conseguente fattore di diluizione finale di 1:1 miliardi. Dopo questo, 100 microlitri di ogni diluizione sono placcati. Le piastre vengono quindi incubate e le colonie clonali vengono contate. La piastra di diluizione per un dato punto temporale che cresce tra 30 e 300 colonie viene utilizzata per calcolare i CFU per millilitro per quel punto temporale.
Il secondo metodo comune per misurare la concentrazione batterica è la densità ottica. La densità ottica di una coltura può essere misurata istantaneamente, in relazione a un media vuoto, con uno spettrofotometro. In genere una lunghezza d'onda di 600 nanometri, nota anche come OD600, viene utilizzata per queste misurazioni, che aumentano all'aumentare della densità cellulare. Mentre la densità ottica è meno precisa delle CFU, è conveniente perché può essere ottenuta istantaneamente e richiede relativamente pochi reagenti. Entrambe le tecniche possono essere utilizzate insieme per creare una curva standard che approssima più accuratamente il conteggio delle cellule batteriche di una coltura. In questo video, imparerai come ottenere CFU e misurazioni OD600 da diluizioni seriali temporizzare di E. coli. Quindi, due curve di crescita che utilizzano le misurazioni CFU e OD600, rispettivamente, verranno tracciate prima di essere correlate da una curva standard.
Quando si lavora con i batteri, è importante utilizzare i dispositivi di protezione individuale appropriati come un camice da laboratorio e guanti e osservare una corretta tecnica asettica.
Successivamente, sterilizzare la stazione di lavoro con etanolo al 70%. In primo luogo, preparare il brodo LB e il supporto agar solido LB in flaconi autoclavabili separati. Dopo aver parzialmente chiuso i tappi delle bottiglie, sterilizzare il supporto in un'autoclave impostata a 121 gradi Celsius per 35 minuti. Quindi, lasciare raffreddare il mezzo agar a bagnomaria impostato a 50 gradi Celsius per 30 minuti. Una volta raffreddato, versare da 20 a 25 ml in ogni capsula di Petri. Successivamente, lasciare che le piastre si impostino per 24 ore a temperatura ambiente.
Per preparare gli isolati della singola colonia che verranno successivamente utilizzati per produrre una coltura batterica liquida, utilizzare stock precedentemente congelati e una tecnica di placcatura a strisce adeguata per strisciare E. coli per l'isolamento su LB agar. Incubare il piatto a 37 gradi Celsius durante la notte. Successivamente, raffreddare un anello di inoculazione sterilizzato a fiamma sull'agar prima di selezionare una singola colonia dalla piastra striata. Inoculare 4 mL di liquido in una provetta da 15 mL. Quindi, coltivare l'E. coli a 37 gradi Celsius durante la notte con agitazione a 210 giri / min.
Per impostare il volume 1:1000 di coltura batterica che verrà utilizzato nella curva di crescita, ottenere prima un pallone Erlenmeyer autoclavato da 500 ml. Quindi, utilizzare una pipetta sierologica da 50 ml per trasferire 100 mL di mezzo sterile al pallone. Quindi, etichettare nove provette da 15 ml consecutivamente da una a nove. Questi numeri corrispondono al fattore di diluizione che verrà utilizzato per calcolare l'unità formante la colonia, o CFU. Quindi, aggiungere 9 ml di 1X PBS a ciascun tubo. Successivamente, etichettare le piastre di agar preparate con i corrispondenti punti temporali e fattori di diluizione che verranno coltivati. In questo esempio con E. coli, dopo il punto di tempo di inizio, i punti temporali vengono presi una volta ogni ora. Utilizzando la coltura liquida di E. coli preparata in precedenza, inoculare il mezzo nell'autoclave 500 mL di pallone Erlenmeyer con 1:1000 volume di coltura. Ruotare i media per distribuire uniformemente i batteri.
Dopo aver spento uno spettrofotometro, pulire la cuvetta con una salvietta priva di lanugine. Quindi, erogare 1 mL della coltura nella cuvetta e posizionarla nello spettrofotometro per ottenere la densità ottica della coltura nel punto temporale zero. Quindi, coltivare l'E. coli a 37 gradi Celsius con agitazione a 210 giri / min. In ogni punto temporale dopo il punto temporale zero, prelevare un altro 1 mL di coltura batterica dal pallone e ripetere la misurazione della densità ottica. Se la lettura della densità ottica è maggiore di 1,0, diluire 100 microlitri di coltura batterica con 900 microlitri di mezzi freschi e quindi misurare nuovamente la densità ottica. Questo valore può essere moltiplicato per 10 per la misurazione OD 600.
Per ottenere la misura dell'unità di formazione della colonia per ogni punto temporale, prelevare un ulteriore 1 mL di coltura batterica dal pallone in ogni punto temporale. Erogare la coltura batterica in una provetta negativa e vortice per mescolare. Quindi, eseguire la serie di diluizione trasferendo prima 1 mL dal tubo negativo a due tubi e vortice per mescolare. Trasferire 1 mL dal tubo negativo due al tubo negativo tre e vortice per mescolare. Continuare questo trasferimento seriale lungo tutti i tubi di diluizione fino al tubo a nove negativi. Erogare 100 microlitri di sospensione cellulare sulla piastra etichettata corrispondente per ogni diluizione. Per ogni diluizione, sterilizzare uno spandicelle in etanolo, passarlo attraverso una fiamma del bruciatore Bunsen e raffreddarlo toccando la superficie dell'agar lontano dall'inoculato. Quindi, utilizzare lo spandicelle per diffondere la sospensione cellulare fino a quando la superficie della piastra di agar diventa asciutta. Incubare le piastre a testa in giù a 37 gradi Celsius. Una volta che sorgono colonie visibili, conta il numero di colonie batteriche su ciascuna piastra. Registrare questi valori e i fattori di diluizione associati per ogni piastra in ogni punto temporale.
Per creare una curva di crescita OD 600, dopo aver assicurato che tutti i punti dati siano inseriti correttamente in una tabella, selezionare tutti i punti temporali e i dati corrispondenti. Per generare un grafico della curva di crescita dell'unità di formazione della colonia, scegliere la piastra di diluizione in cui i conteggi delle colonie rientravano nell'intervallo da 30 a 300 batteri per ogni punto temporale. Moltiplicare il numero di conteggio delle colonie per il fattore di diluizione e quindi per dieci. Questo perché la diffusione di 100 microlitri è considerata un'ulteriore diluizione 1:10 quando si calcolano le unità formanti colonie per millilitro. Dopo questo, traccia le unità che formano la colonia rispetto al tempo su una scala semi-logaritmo.
Questi appezzamenti prodotti con misurazioni OD 600 e CFU, rispettivamente, possono fornire preziose informazioni sulla cinetica di crescita di E. coli. La densità ottica e le unità formanti colonie possono essere correlate, in modo che le CFU per millilitro possano essere stimate da misurazioni OD 600, risparmiando tempo e materiali in esperimenti futuri.
Per fare ciò, tracciare le unità formanti la colonia contro la densità ottica su una scala lineare per letture OD 600 inferiori o uguali a 1. 0. Successivamente, genera una linea di tendenza di regressione lineare in formato Y = MX + B, dove M è la pendenza e B è l'intercetta y. Fare clic con il pulsante destro del mouse sui punti dati e selezionare Aggiungi linea di tendenza e lineare. Quindi, selezionare la casella per visualizzare l'equazione sul grafico e visualizzare il valore quadrato R sul grafico. Il valore al quadrato R è la misura statistica di quanto strettamente i dati corrispondevano alla linea di regressione adattata. In questo esempio, i primi 6 punti temporali vengono tracciati con OD 600 sull'asse x e CFU per millilitro sull'asse y. In esperimenti futuri con le stesse condizioni di crescita, questi valori di pendenza e intercetta y possono essere inseriti in questa equazione per stimare le CFU dalle letture OD 600. Quindi, guarda il grafico della curva di crescita dell'unità che forma la colonia. Durante la fase esponenziale, identifica due punti temporali con la pendenza più ripida tra di loro. Per calcolare il tempo di raddoppio, calcolare prima la variazione di tempo tra i punti temporali selezionati. Quindi, calcola il cambiamento di generazioni usando l'equazione mostrata qui. Qui, la b minuscola è il numero di batteri al punto temporale tre e la B maiuscola è il numero di batteri al punto temporale due. Infine, dividi il cambiamento nel tempo per il cambiamento delle generazioni. In questo esempio, il tempo di raddoppio è 0. 26 ore o 15 minuti e 19 secondi. Confrontare i tempi di raddoppio tra diversi trattamenti sperimentali ci consente di identificare le migliori condizioni di crescita per una determinata specie batterica. Pertanto, il trattamento con il tempo di raddoppio più basso sarà il più ottimale delle condizioni testate.
Subscription Required. Please recommend JoVE to your librarian.
Results
I grafici delle unità formanti colonie e la densità ottica sono due modi per visualizzare la cinetica di crescita. Determinando la relazione tra CFU/mL e OD600, il grafico a densità ottica fornisce anche una stima di CFU/mL nel tempo. Le condizioni che comportano il più breve tempo di raddoppio sono considerate ottimali per la crescita dei batteri dati.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
Le curve di crescita sono preziose per comprendere la cinetica di crescita e la fisiologia dei batteri. Ci permettono di determinare come i batteri rispondono in condizioni di crescita variabili e di definire i parametri di crescita ottimali per un determinato batterio. I grafici di unità formante colonie e densità ottica contengono entrambi informazioni preziose che descrivono la durata della fase di ritardo, la densità massima cellulare raggiunta e consentono il calcolo del tempo di raddoppio batterico. Le curve di crescita consentono anche il confronto tra diversi batteri nelle stesse condizioni di crescita. Inoltre, la densità ottica fornisce un mezzo per standardizzare gli inoculi iniziali, migliorando la coerenza in altri esperimenti.
Determinare quale approccio utilizzare quando si progetta un esperimento di curva di crescita richiede considerazione. Come metodo preferito per generare curve di crescita, i grafici unitari di formazione delle colonie riflettono più accuratamente i conteggi delle cellule vitali nella coltura batch. I grafici unitari di formazione di colonie consentono anche di misurare la crescita batterica in condizioni che altrimenti interferirebbero con una misurazione della densità ottica. Tuttavia, è un processo più dispendioso in termini di tempo, che richiede un uso estensivo di reagenti e deve essere eseguito manualmente. I grafici di densità ottica sono meno accurati e forniscono solo una stima delle unità formanti la colonia, richiedendo la generazione di una curva standard per ogni batterio unico. La densità ottica viene utilizzata principalmente per la sua praticità in quanto richiede molto meno tempo e non richiede molti reagenti per funzionare. Ciò che è più interessante per la densità ottica è che gli incubatori spettrofotometrici possono generare automaticamente curve di crescita, aumentando notevolmente il numero di condizioni di coltura che possono essere testate contemporaneamente ed eliminando la necessità di frequentare costantemente la cultura.
Subscription Required. Please recommend JoVE to your librarian.
References
- R. E. Buchanan. 1918. Life Phases in a Bacterial Culture. J Infect Dis 23:109-125.
- CAMPBELL A. 1957. Synchronization of cell division. Bacteriol Rev 21:263-72.
- Wang P, Robert L, Pelletier J, Dang WL, Taddei F, Wright A, Jun S. 2010. Robust growth of Escherichia coli. Curr Biol 20:1099-103.
- Goldman E, Green LH. 2015. Practical Handbook of Microbiology, Third Edition. CRC Press.
- Ben-David A, Davidson CE. 2014. Estimation method for serial dilution experiments. J Microbiol Methods 107:214-221.
- Koch AL. 1968. Theory of the angular dependence of light scattered by bacteria and similar-sized biological objects. J Theor Biol 18:133-156.
- Sezonov G, Joseleau-Petit D, D'Ari R. 2007. Escherichia coli physiology in Luria-Bertani broth. J Bacteriol 189:8746-9.







 , dove b = numero di batteri a t2, B = numero di batteri a t1e n = numero di generazioni. Derivato da:
, dove b = numero di batteri a t2, B = numero di batteri a t1e n = numero di generazioni. Derivato da: