- 00:01Concepts
- 04:08Preparation of Plates and Bacteria
- 06:04Determining MIC Using E-Test
- 07:01Synergy Testing: Cross Approach
- 07:47Synergy Testing: Non-Cross Approach
- 08:56MIC Determination Using Broth Dilution
- 10:33Data Analysis: Broth Microdilution
- 11:10Data Analysis and Results: Synergy Testing
Test di suscettibilità agli antibiotici: test dell'epsilometro per determinare i valori MIC di due antibiotici e valutare la sinergia antibiotica
English
Share
Overview
Fonte: Anna Bläckberg1, Rolf Lood1
1 Dipartimento di Scienze Cliniche Lund, Divisione di Medicina delle Infezioni, Centro Biomedico, Università di Lund, 221 00 Lund Svezia
La conoscenza delle interazioni tra antibiotici e batteri è importante per capire come i microbi evolvono la resistenza agli antibiotici. Nel 1928, Alexander Fleming scoprì la penicillina, un antibiotico che esercita la sua funzione antibatterica interferendo con la rigenerazione della parete cellulare (1). Successivamente sono stati scoperti altri antibiotici con diversi meccanismi d’azione, tra cui farmaci che inibiscono la replicazione del DNA e la traduzione proteica nei batteri; tuttavia, negli ultimi anni non sono stati sviluppati nuovi antibiotici. La resistenza agli antibiotici attuali è in aumento, con conseguenti gravi malattie infettive che non possono essere trattate efficacemente (2). Qui, descriviamo diversi metodi per valutare la resistenza agli antibiotici nelle popolazioni batteriche. Ognuno di questi metodi funziona, indipendentemente dal meccanismo d’azione degli antibiotici utilizzati, perché la morte batterica è il risultato misurato. La resistenza agli antibiotici non è solo rapidamente diffusa in modo specifico attraverso le impostazioni ospedaliere, ma anche in tutta la società. Al fine di studiare tali mezzi di resistenza, sono stati sviluppati diversi metodi, tra cui il test Epsilometro (E-test) e il test di diluizione del brodo (3).
L’E-test è un metodo consolidato ed è uno strumento economico che quantifica i dati della concentrazione minima inibitoria (MIC), la concentrazione più bassa di un antimicrobico che inibisce la crescita visibile di un microrganismo. A seconda del ceppo batterico e degli antibiotici utilizzati, il valore del MIC può variare tra sub μg/mL e >1000 μg/mL (4). L’E-test viene eseguito utilizzando una striscia di plastica contenente un gradiente antibiotico predefinito, che è impresso con la scala di lettura MIC in μg / mL. Questa striscia viene trasferita direttamente sulla matrice di agar quando viene applicata alla piastra di agar inoculata. Dopo l’incubazione, una zona di inibizione ellittica simmetrica è visibile lungo la striscia poiché viene impedita la crescita batterica. MIC è definito dall’area di inibizione, che è il punto finale in cui l’ellisse interseca la striscia. Un altro metodo comune per determinare la MIC è il metodo di diluizione delle microbroti. La diluizione dei microbroti incorpora diverse concentrazioni dell’agente antimicrobico aggiunto a un mezzo di brodo contenente batteri inoculati. Dopo l’incubazione, la MIC è definita come la più bassa concentrazione di antibiotico che impedisce la crescita visibile (5). È anche un metodo quantitativo e può essere applicato a diversi batteri. Gli svantaggi di questo metodo includono la possibilità di errori durante la preparazione delle concentrazioni dei reagenti e il gran numero di reagenti necessari per l’esperimento. Misurare la resistenza agli antibiotici è imperativo sia dal punto di vista clinico che di ricerca, e questi metodi in vitro per studiare la resistenza sono discussi e mostrati di seguito.
Il profilo di resistenza per un batterio specifico può essere applicato al fine di ottimizzare il trattamento antibiotico per determinare se un paziente trarrebbe beneficio dal trattamento combinato rispetto alla singola terapia. Per l’uso di più di un antibiotico alla volta, è imperativo conoscere le loro interazioni tra loro e se hanno un effetto additivo, sinergico o antagonista. Un effetto additivo può essere visto quando l’effetto congiunto degli antibiotici è uguale alla potenza dei singoli antibiotici somministrati a una dose uguale. La sinergia tra antibiotici, d’altra parte, è presente quando l’effetto congiunto degli antibiotici è più potente che se il farmaco fosse somministrato da solo (6). L’applicazione di combinazioni di trattamento antimicrobico viene utilizzata per evitare il verificarsi di resistenza antimicrobica, migliorando così l’effetto del trattamento antibiotico individuale (7). La conoscenza dell’antagonismo è anche altrettanto importante per prevenire l’uso non necessario di combinazioni antimicrobiche. La metodologia E-test offre modi semplici e diversi per determinare possibili sinergie e antagonismi tra diversi agenti antimicrobici. Per far fronte alla proliferazione di patogeni resistenti agli antibiotici, è importante conoscere i possibili meccanismi sinergici e antagonisti di alcuni antibiotici con conseguente efficacia clinica e lotta contro la multifarmaco-resistenza.
La determinazione della sinergia utilizzando gli E-test può essere suddivisa in due approcci generali: cross e non cross testing. Mentre entrambi i test sinergici si basano sulla precedente conoscenza dei singoli valori MIC, i due approcci sono leggermente diversi nella metodologia e nell’approccio concettuale. In un test di sinergia non incrociata, il primo antibiotico nella coppia da testare viene posto su una piastra di agar inoculata con batteri. Dopo aver permesso agli antibiotici della prima striscia di infondere la piastra (ad esempio dopo 1 ora), la striscia viene rimossa e una nuova striscia contenente il secondo antibiotico viene posizionata nello stesso punto esatto del primo, assicurandosi di posizionare i due singoli valori MIC uno sopra l’altro. La zona di inibizione risultante può quindi essere analizzata come descritto sopra e la sinergia calcolata in base all’equazione 1.
Equazione 1 – Concentrazioni inibitorie frazionarie (FIC)
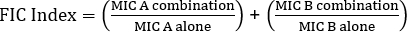
Valori >0,5 dimostra sinergia.
Mentre premia l’esaminatore con piastre facili da analizzare, il metodo è un po ‘laborioso e richiede tempo a causa del cambio di strisce, nonché della necessità di utilizzare due piastre per esperimento. Invece, viene spesso utilizzato un test incrociato. Invece di aggiungere le due diverse strisce E-test successivamente una sopra l’altra (dopo la rimozione della prima), entrambe vengono posizionate contemporaneamente ma sotto forma di una croce  (angolo di 90 °), con i due valori MIC precedentemente determinati che formano l’angolo di 90 °. Con questo approccio è necessaria una sola piastra per test sinergico, oltre a meno lavoro, rendendola una scelta preferita nonostante sia leggermente più difficile da analizzare. I nuovi valori MIC nell’approccio combinato agli antibiotici possono essere visualizzati come zone di inibizione modificate, dopo di che la sinergia può essere determinata dall’equazione 1.
(angolo di 90 °), con i due valori MIC precedentemente determinati che formano l’angolo di 90 °. Con questo approccio è necessaria una sola piastra per test sinergico, oltre a meno lavoro, rendendola una scelta preferita nonostante sia leggermente più difficile da analizzare. I nuovi valori MIC nell’approccio combinato agli antibiotici possono essere visualizzati come zone di inibizione modificate, dopo di che la sinergia può essere determinata dall’equazione 1.
Invece di utilizzare un approccio a piastra di agar, un approccio a microbroto può spesso essere preferenziale a causa della sua maggiore flessibilità (ad esempio la capacità di scegliere concentrazioni specifiche di antibiotici al di fuori dei limiti di una striscia reattiva E). Inoltre, si suggerisce che i test delle microbrotole siano più sensibili a causa della loro distribuzione uniforme degli antibiotici in una soluzione liquida, non dipendenti dalla dissociazione all’interno di una fase solida (piastra di agar). I pozzetti in una micropiastra a 96 pozzetti saranno inoculati con un determinato numero di batteri (106 cfu / mL: la concentrazione batterica può essere stimata da misurazioni OD600 nm, standard di torbidità o diffondendo campioni di placcatura da 10x diluizioni seriali batteriche) e antibiotici in diverse diluizioni saranno aggiunti ai pozzetti. Allo stesso modo, per le strisce reattive E MIC è determinato come l’intersezione (pozzo / spot) con la più bassa concentrazione di antibiotici che inibiscono la crescita visibile dei batteri.
Obiettivo sperimentale
- Il progetto seguente descrive le strategie per determinare i valori MIC della penicillina G e della gentamicina dello streptococco gruppo G con due diversi metodi, E-test e diluizione dei microbroti. Per l’E-test, le placche di agar Mueller-Hinton inoculate con Streptococcus gruppo G sono state utilizzate in combinazione con strisce sfumate di penicillina G e/o gentamicina; mentre il brodo MH con il 50% di sangue di cavallo llisi e 20 mg/mL di β-NAD è stato utilizzato con antibiotici solubili insieme allo Streptococco di gruppo G in un approccio a microbroto.
Materiali
- Colonie batteriche su una piastra di agar nel sangue, conservate <7 giorni in 4°C
- Piastre di agar di sangue
- 0.5 Standard McFarland
- 1% BaCl2
- 1% H2SO4
- Tubo salino (2 mL)
- Applicatore con punta in cotone
- Piastre di agar Mueller-Hinton (piastre MHA)
- Brodo MH con sangue di cavallo lisato al 50% e 20 mg/mL β-NAD (MH-F)
- E-test penicillina/gentamicina (o antibiotici di interesse) (BioMerieux, Marcy l’Etoile, Francia, Svezia)
- Antibiotici penicillina/gentamicina (o antibiotici di interesse (polvere/soluzione))
Nota: i mezzi specifici utilizzati per la crescita batterica possono variare a seconda delle specie.
Procedure
Results
MIC values in E-test
Individual MIC values were identified in Figure 1 as 0.094 μg/mL for penicillin G and 8 μg/mLfor gentamicin. For synergy tests, both demonstrated an MIC value for penicillin G of 0.064 μg/mL (Figures 2, 3), while gentamicin had an MIC 4 μg/mL for cross and non-cross tests. Note a slight discrepancy between the cross and non-cross tests may occur due to the different incubation times of the strips in the two settings.
Calculation of synergy
The equation for FIC is:
 = 1.18 >0.5 (no synergy)
= 1.18 >0.5 (no synergy)
MIC determination in broth
Cloudiness of the wells indicated bacterial growth, and thus no inhibition occurred. The first clear well with penicillin G (Figure 4) contained 0.12 μg/mL penicillin G, and hence this was the MIC value. For gentamicin the first clear well was present at 8 μg/mL gentamicin. The penicillin G value was slightly higher than when using an E-test, due to the higher resolution of the strip (e.g. based on a 1.5x factor serial dilution, not a 2x factor).
Inoculum size
To determine the inoculum size, an approach as outlined in Figure 5 and 6 was used. Colonies were counted in the D-row (1000x dilution), adding up to 7, 8, and 8 in the triplicate series with a mean value of 7.67 cfu. The number of colonies neeed to be multiplied with the dilution factor (e.g. 1000x), as well as with 100 to obtain cfu/mL, giving an inoculum size of approximately 8 x 105, well within the targeted inoculum size of 105-6 cfu/mL.
Applications and Summary
Antibiotic resistance is a worldwide health problem. In order to determine resistance mechanisms of microbes, methods testing for synergy and antagonism with different antibiotics is crucial. The E-test method is rapid, easy to replicate, and can be used to investigate any synergistic potential of combination therapies. The broth dilution method can also be assessed to predict bactericidal activity. In order to investigate the resistance mechanisms of different microbes, knowledge of synergistic and antagonistic antibiotic interactions is crucial. Combining antibiotics may be a strategy to increase treatment efficacy and face antibiotic resistance. In the tests performed here, we were able to determine the MIC values of penicillin G and gentamicin for group G Streptococcus. We also demonstrated that the two antibiotics do not display synergistic effects, thus would not be a preferred treatment option for such infections.
References
- Tan SY, Tatsumura Y. Alexander Fleming (1881-1955): Discoverer of penicillin. Singapore Medical Journal. 56 (7):366-7. (2015)
- Aminov RI. A brief history of the antibiotic era: lessons learned and challenges for the future. Frontiers in Microbiology. 1:134. (2010)
- Pankey GA, Ashcraft DS, Dornelles A. Comparison of 3 E-test (®) methods and time-kill assay for determination of antimicrobial synergy against carbapenemase-producing Klebsiella species. Diagnostic Microbiology and Infectious Disease. 77 (3):220-6. (2013)
- EUCAST: European Committee On Antimicrobial Susceptibility Testing (www.eucast.org).
- Wiegand I, Hilpert K, Hancock RE. Agar and broth dilution methods to determine the minimal inhibitory concentration (MIC) of antimicrobial substances. Nature Protocols. 3 (2):163-75. (2008)
- Doern CD, When does 2 plus 2 equal 5? A review of antimicrobial synergy testing. Journal of Clinical Microbiology. 52 (12):4124-28. (2014)
- Worthington RJ, Melander C. Combination approaches to combat multi-drug resistant bacteria. Trends in Biotechnology. 31 (3):177-84. (2013)
Transcript
Antibiotic susceptibility is defined as the sensitivity of a bacteria to antibiotics and can be measured using a broth dilution test or an Epsilometer test, also called an E-test.
In the broth dilution method, a standardized number of bacteria are added to a growth media containing serial antibiotic dilutions. If susceptible, the bacteria cannot grow at the higher antibiotic concentrations but continue to multiply at the lower antibiotic concentrations, causing media to turn turbid. The lowest antibiotic concentration at which the bacteria can no longer survive or multiply is referred to as the minimum inhibitory concentration, or MIC, value of the antibiotic for the given bacteria.
In an E-test, a plastic strip impregnated with a predefined gradient of antibiotic is applied over a freshly spread lawn of bacteria on a Mueller-Hinton agar, or MH-A, Petri plate. The antibiotic diffuses out into the agar media, where it is taken up by the bacteria. If susceptible, the bacteria cannot multiply and will die off, forming a clear zone around the E-strip, which is referred to as the growth inhibition zone. At the point where the growth intersects with the E-strip, the corresponding value on the scale gives the MIC value of the antibiotic.
Often antibiotics are used in combination to prevent the emergence of antibiotic resistant strains of bacteria. This often results in a synergistic, rather than additive, effect. Synergistic means that the combined effect of the two antibiotics is greater than the sum of their individual activities. However, the effect is considered significant only when the MIC value of the antibiotic combination decreases by at least two-fold. This criterion is evaluated by calculating the fractional inhibitory concentration, or FIC, index. By summing the ratio of the MIC of each antibiotic in combination with the MIC of each antibiotic individually, an FIC index less than 0.5 indicates synergy.
Antibiotic synergy can be measured using two E-test based methods: a non-cross test or a cross test. In a non-cross test, first, the E-strips for two different antibiotics with predetermined MIC values are applied to two separate plates. After the antibiotics have diffused into the medium, the original E-strips are removed and the E-strips for the alternate antibiotics are placed such that their MIC scales lay exactly over the MIC scales of the previous strips. In a cross test, which is a faster version of the non-cross test, the E-strips of the two antibiotics are placed together in a cross formation, such that the scales of their MIC marks form a 90 degree angle at the intersection. Following incubation in both techniques, the MIC value of each antibiotic in combination with the other antibiotic is read at the point where the growth inhibition zone intersects with the edge of the E-strip. Then, the FIC index is calculated.
This video will demonstrate how to determine the MIC value of a given antibiotic for a given bacteria using an E-test and a micro broth dilution test. You will also learn how to determine synergy between two antibiotics using a cross test and a non-cross test.
To begin, put on any appropriate personal protective equipment, including laboratory gloves and a lab coat. Next, sterilize the work space using 70% ethanol. Next, collect 15 milliliters of sterile Mueller-Hinton broth with 50% lysed horse blood and 20 milligrams per milliliter beta-nicotinamide. And five to eight Mueller-Hinton agar plates. Now, to prepare a McFarland turbidity standard number 0.5, measure out 9.95 milliliters of 1% sulfuric acid solution. Then, add 50 microliters of 1% barium chloride solution to the sulfuric acid solution. Vortex the solution well to obtain a turbid suspension. Cover the tube with aluminum foil and set it aside. Next, dispense one milliliter of saline solution into a 15 milliliter tube.
Use a sterile loop to scrape up a sample of the bacterial growth from your bacterial test plate, here, Streptococcus group G. Then, place the bacteria-laden loop into the saline solution, stir gently, and then vortex the tube well. Now, place the bacterial suspension and McFarland turbidity standards side by side and compare them for turbidity equivalence. Add either additional saline or bacterial colonies until the bacterial suspension’s turbidity matches that of the standard. Once the desired turbidity is obtained, dip a sterile cotton tip applicator into the bacterial suspension. To inoculate the MH-A plate, swab the entire surface of the plate gently with a zigzag motion. Next, label the bottom sides of the plates with the name of the bacteria and the date.
To begin, take out a penicillin G E-test strip, holding it by the edge with forceps. Gently place strip into the center of the freshly swabbed MH-A plate and replace the lid. In this example, a second antibiotic, gentamicin, is also tested. Thus, the strip placement process is repeated with the second plate and a gentamicin E-test strip. To determine the results of the E-test, collect the first plate that contains the penicillin G E-test strip. Now, determine the point where the inhibition zone intersects with the antibiotic strip. Read the corresponding numerical value on the scale. This value represents the MIC value of penicillin G. Determine the MIC value for gentamicin in the same manner.
To begin, inoculate an MH-A plate with Streptococcus group G strain bacteria. Label the bottom of the plate with the name of the bacteria, antibiotics to be used, and the date. Now, place an E-test strip for the antibiotic of interest in the center of the plate. Then, hold the second test strip at a 90 degree angle to the first strip and locate its MIC mark. Gently lay the second E-strip over the first at the point where the two MIC values intersect. Once the strips are placed, do not move them. Next, incubate the plates at 37 degrees celsius for 18 to 20 hours.
After inoculating two MH-A plates, with Streptococcus group G strain bacteria, place an E-test strip for one antibiotic on the surface of one plate. Then, place an E-test strip for the other antibiotic on the second plate as demonstrated. Using a plastic inoculation loop, mark the MIC value of each antibiotic on the surface of its respective plate. Next, cover the plates and incubate them at room temperature for one hour. After this, use forceps to remove the E strips. Next, collect one of the plates and an E-test strip for the other antibiotic. Hold the E-test strip over the imprint left by the first strip and locate the point where the MIC value on the E strip aligns with the marked line. Gently place the strip at this intersecting point. Repeat this process for the second plate and incubate both plates at 37 degrees celsius for 18 to 20 hours.
First, obtain a bacterial suspension with an established bacterial concentration and dilute the culture in MHF broth to achieve an OD600 of 0.003. Next, weigh out 16 milligrams of penicillin G and 128 milligrams of gentamicin. Transfer each weighed dry antibiotic into 215 milliliter conical tubes. Add 10 milliliters of distilled water to each of the conical tubes and mix well by vortexing. Label the tubes with the antibiotic name and concentration.
Performing the assay in triplicate, add 400 microliters of the working bacterial solution into the first wells of three rows of a 96-well microtiter plate. Next, add 200 microliters of the working bacterial solution in MHF broth to the wells of the three rows. Now, to generate a two-fold serial antibiotic dilution, first add four microliters of antibiotic stock to the first well, generating a 100 fold dilution. Sequentially, transfer 200 microliters of bacteria-antibiotic solution to each well, beginning from the first well through the second to last well in each row, ensure proper mixing by pipetting two to three times after every transfer. Discard the final 200 microliters of bacteria-antibiotic solution.
To determine the results of the broth micro dilution test for penicillin G, first locate the wells that exhibit no visible bacterial growth, indicated by a lack of turbidity. From these wells, identify the well with the lowest antibiotic concentration. This represents the MIC value of penicillin G for the tested bacteria. The MIC value of gentamicin can be determined using the same assay and technique.
To determine the results of the non-cross test, collect the first plate, which contains a penicillin G E strip. Then, determine the point where the growth inhibition zone intersects with the antibiotic strip. The corresponding value on the scale represents the MIC value for penicillin G in combination with gentamicin. In this example, the MIC value in combination is 0.064 micrograms per milliliter.
Now, collect the second plate, which contains the gentamicin E strip, and determine the MIC value in combination as previously demonstrated. To evaluate the effect of combination, first calculate the fractional inhibitory concentration or FIC for penicillin G by dividing the MIC in combination by the MIC of the antibiotic alone. Repeat this process for gentamicin. Then, calculate the FIC index using the equation shown here. A two-fold reduction in the MIC value in combination yields an FIC index value that is less than or equal to 0.5 and demonstrates synergy between penicillin G and gentamicin. In this case, the calculated FIC value is 1.18 which is greater than 0.5. Thus, the results do not demonstrate synergy between penicillin G and gentamicin against the Streptococcus group G strain.
To determine the results of the cross test, first determine the point where the growth inhibition zones intersect with their respective E strips. Read the numerical value on each E-test strip that corresponds to this intersection point. These values represent the MIC value in combination for penicillin G and gentamicin. Next, to evaluate the effect of the combination, calculate the FIC index using the equation shown here. In this example, the calculated FIC value is 1.18, which is greater than 0.5. This means that penicillin G and gentamicin do not act synergistically against the Streptococcus group G strain.
