ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Die vorliegende Studie berichtet über ein Protokoll für das Chromosomen-Screening menschlicher Embryonen, das verbrauchtes Kulturmedium verwendet, das eine Embryonenbiopsie vermeidet und die Identifizierung der Chromosomenploidie mittels NGS ermöglicht. Der vorliegende Artikel stellt das detaillierte Verfahren vor, einschließlich der Herstellung des Nährmediums, der Amplifikation des gesamten Genoms (WGA), der Vorbereitung der Next-Generation-Sequencing-Bibliothek (NGS) und der Datenanalyse.
Abstract
Bei der klinischen In-vitro-Fertilisation (IVF) erfordert die vorherrschende Methode für PGT-A die Biopsie einiger Zellen aus dem Trophektoderm (TE). Dies ist die Abstammungslinie, die die Plazenta bildet. Diese Methode erfordert jedoch spezielle Fähigkeiten, ist invasiv und leidet unter falsch-positiven und negativen Ergebnissen, da die Chromosomenzahlen im TE und die innere Zellmasse (ICM), die sich zum Fötus entwickelt, nicht immer gleich sind. NICS, eine Technologie, die die Sequenzierung von DNA erfordert, die sowohl von TE als auch von ICM in das Nährmedium freigesetzt wird, könnte einen Ausweg aus diesen Problemen bieten, hat sich aber bisher als begrenzt wirksam erwiesen. Die vorliegende Studie berichtet über das vollständige NICS-Protokoll, das Methoden zur Probenahme von Nährmedien, zur Amplifikation des gesamten Genoms (WGA) und zur Bibliotheksvorbereitung sowie zur NGS-Datenanalyse durch Analysesoftware umfasst. In Anbetracht der unterschiedlichen Kryokonservierungszeiten in verschiedenen Embryonenlabors stehen den Embryologen zwei Methoden zur Entnahme des Embryokulturmediums zur Verfügung, die je nach den tatsächlichen Bedingungen des IVF-Labors ausgewählt werden können.
Introduction
Assistierte Reproduktionstechnologien (ARTs) werden zunehmend zur Behandlung von Unfruchtbarkeit eingesetzt. Die Erfolgsrate von ART, wie z. B. IVF, ist jedoch begrenzt, und die Rate der Fehlgeburten ist deutlich höher als die der Normalbevölkerung1. Die Hauptursache für diese Probleme sind Chromosomenanomalien, die häufig bei menschlichen Präimplantationsembryonen auftreten2. PGT-A ist eine wirksame Methode zum Screening von Embryonen auf Chromosomengleichgewicht vor der Implantation 3,4. Einige Studien haben bewiesen, dass PGT-A die Abtreibungsrate senken und die Schwangerschaftsrate verbessern kann 5,6,7,8. PGT-A erfordert jedoch komplexes technisches Fachwissen, das eine spezifische Ausbildung und Erfahrung erfordert. Die invasive Embryonenbiopsie kann auch zu einer Schädigung der Embryonen führen9. Studien haben gezeigt, dass die Blastomerbiopsie die spätere Entwicklung behindern kann und die Anzahl der biopsierten TEs die Implantationsraten beeinflussen kann10. Obwohl das Problem der langfristigen biologischen Sicherheit der Embryonenbiopsie beim Menschen noch nicht gründlich untersucht wurde, haben Tierstudien gezeigt, dass sie negative Auswirkungen auf die Embryonalentwicklung hat11,12,13.
Frühere Berichte deuteten darauf hin, dass während der Embryonalentwicklung Spuren von DNA-Material in das Kulturmedium abgesondert wurden, und es wurden Anstrengungen unternommen, ein umfassendes Chromosomen-Screening (CCS) mit verbrauchtem Embryokulturmedium durchzuführen 14,15,16,17,18. Die Nachweisraten und die Genauigkeit der Tests haben jedoch nicht die Anforderungen für einen umfassenden klinischen Einsatz erfüllt. Die vorliegende Studie berichtete über eine Verbesserung des NICS-Assays zur Erhöhung der Detektionsraten sowie der Genauigkeit des NICS-Tests19. In den letzten Jahren wurde Blastocoele-Flüssigkeit (BF) als analytische Probe der minimalinvasiven PGT-A untersucht. Der Anteil der erfolgreichen genomweiten Amplifikation und der nachweisbaren DNA in Blastozystenflüssigkeitsproben liegt jedoch zwischen 34,8 % und 82 %20,21,22. Das in verschiedenen Studien berichtete BF-Volumen reicht von 0,3 nL bis 1 μL. Angesichts der geringen Menge an DNA in BF ist es möglich, die Menge an zellfreier DNA durch Mischen von Blastozystenflüssigkeit und Kulturmedium zu erhöhen, um die Erfolgsrate und Konsistenz des Nachweises zu verbessern. Kuznyetsov et al.23 und Li et al.24 behandelten die Zona pellucida mit einem Laser und setzten Blastozystenflüssigkeit in das Kulturmedium frei, um die Gesamtmenge der embryonalen DNA zu verbessern, und die Amplifikationsrate der kombinierten Medium/BF-Proben nach WGA betrug 100 % bzw. 97,5 %. Jiao et al.25 erhielten mit der gleichen Methode ebenfalls eine 100%ige Amplifikationserfolgsrate.
Die vorliegende Studie berichtet über ein detailliertes Protokoll, das die Probenvorbereitung für verbrauchte Medien, die NGS-Vorbereitung und die Datenanalyse umfasst. Durch die sorgfältige Entfernung von Kumuluszellen aus den Eizellen wurden in der vorliegenden Studie intrazytoplasmatische Einzelspermieninjektionen (ICSI) und Blastozystenkulturen durchgeführt. Das verbrauchte Medium an Tag 4 Tag 5/Tag 6 wurde für die Vorbereitung der WGA- und NGS-Bibliothek gesammelt. Durch den Einsatz der NICS-Technologie konnte die vorliegende Studie die Vorbereitungsschritte der WGA- und NGS-Bibliothek in ca. 3 h rationalisieren und die nicht-invasiven CCS-Ergebnisse in ca. 9 h erhalten.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Die ethische Erlaubnis wurde von der Ethikkommission des Dritten Krankenhauses der Universität Peking eingeholt.
1. Vorbereitung
HINWEIS: Die erforderlichen Materialien und Ausrüstungen sind in der Materialtabelle aufgeführt.
- Reagenzien
- 20-30 μl Gametenmedium/Befruchtungsmedium und Spalt-/Blastozystenstadium Kulturmedium (mit Mineralöl bedeckt) und Hyaluronidase (in einem dicht verschlossenen Röhrchen) bei 37 °C, 5 % CO2 und 5 %O2 in einem Trigas-Inkubator über Nacht vor der Verwendung vorwärmen und ausbalancieren (balanciert).
- Hyaluronidase auf 37 °C auf einer Arbeitsfläche in einem Abzug vorwärmen.
- Vitrifikationspuffer und Reagenzien für die Probenentnahme gemäß den Anweisungen des Herstellers vorbereiten.
- Werkzeuge
- Bereiten Sie Probenentnahme- und Transferpipetten (Innendurchmesser von ~200 bis 250 μm), Denudations-/Stripperpipetten (Innendurchmesser von ≥150 μm, ~130-140 μm und ~120 μm) und Pipetten zum Waschen (Innendurchmesser von ~150 μm) vor, indem Sie Pasteurpipetten aus Glas ziehen, um feuerpolierte, offene feine Spitzen zu erzeugen.
HINWEIS: Die Pipetten, die für die Probenentnahme/-übertragung, Denudation und das Waschen verwendet werden, können direkt erworben werden. Die Haltenadeln und Injektionsnadeln können auch direkt erworben werden.
- Bereiten Sie Probenentnahme- und Transferpipetten (Innendurchmesser von ~200 bis 250 μm), Denudations-/Stripperpipetten (Innendurchmesser von ≥150 μm, ~130-140 μm und ~120 μm) und Pipetten zum Waschen (Innendurchmesser von ~150 μm) vor, indem Sie Pasteurpipetten aus Glas ziehen, um feuerpolierte, offene feine Spitzen zu erzeugen.
2. Protokoll 1: Probenentnahme
- Vorbehandlung des Oozyten-Corona-Cumulus-Komplexes (OCCCs) vor der Verdauung mit Hyaluronidase
- Erreichen Sie eine Stimulation der Eierstöcke sowohl mit follikelstimulierendem Hormon (FSH) als auch mit humanen menopausalen Gonadotropinpräparaten (hMG). Wenn der Leitfollikel >18 mm groß ist, verwenden Sie 10.000 IE Choriongonadotropin (hCG) für die endgültige Eizellreifung.
- Führen Sie die Eizellentnahme 36 Stunden nach dem Triggerschuss durch. Die Eizellen werden mit 2,5 ml vorgewärmtem m-HTF, die mit Mineralöl bedeckt sind, entnommen und in Gewebekulturschalen übertragen.
- Die OCCCs werden mit einer Transferpipette schnell in die zentrale Vertiefung einer Organkulturschale mit 1 ml Befruchtungsmedium überführt und dann mit den Eizellen bei 37 °C in einem 5%igen CO2- und 5%O2-Inkubator für 2-4 h inkubiert.
- OCCCs mit Hyaluronidase werden verdaut, indem 1 ml 37 °C vorgewärmte Hyaluronidase (80 I.E./ml) in die zentrale Vertiefung einer Organkulturschale mit OCCCs gegeben wird (Schritt 2.1.3). Halten Sie die Endkonzentration der Hyaluronidase bei 40 IE/ml und mischen Sie sie gründlich.
- Inkubieren Sie die OCCCs 2 Minuten lang auf einer 37 °C heißen thermischen Plattform. Beobachten Sie die Veränderungen unter dem Mikroskop alle 30 s, bis nur noch 1-2 Schichten Granulosazellen übrig sind.
- Denudation von Granulosazellen
- Überführen Sie die verdauten OCCCs schnell in die Kulturschale für die Handhabung der Eizellen und bedecken Sie sie in jeder Vertiefung mit Mineralöl.
- Betrachten Sie die abgetrennten Granulosazellen unter dem Mikroskop. Saugen Sie die Eizellen vorsichtig ab und lassen Sie sie 5 Mal frei, um die restlichen Granulosazellen um die Eizellen herum zu entfernen.
- Wiederholen Sie den vorherigen Schritt in den verbleibenden 3 Vertiefungen, um die Granulosazellen vollständig zu entfernen.
HINWEIS: Die oben genannten Schritte (2.1-2.3) können entsprechend dem Routinebetrieb jedes Labors durchgeführt werden.
- Auswertung der Eizelle
- Beurteilen Sie die Vollständigkeit der Entfernung von Granulosazellen mit einem Mikroskop. Wenn die Zellen nicht vollständig entfernt werden konnten, ist die Retention von 5 oder weniger Granulosazellen zu diesem Zeitpunkt akzeptabel.
HINWEIS: Wenn noch Cumuluszellen an die Eizelle gebunden sind, kann der Rest später am Tag 3 entfernt werden, bevor der Embryo vom Kulturmedium im Spaltstadium in das Kulturmedium im Blastozystenstadium überführt wird.
- Beurteilen Sie die Vollständigkeit der Entfernung von Granulosazellen mit einem Mikroskop. Wenn die Zellen nicht vollständig entfernt werden konnten, ist die Retention von 5 oder weniger Granulosazellen zu diesem Zeitpunkt akzeptabel.
- Nach der intrazytoplasmatischen Spermieninjektion (ICSI)26 werden die Eizellen mit Hilfe von Transferpipetten in Mikrotröpfchen mit einem Spaltmedium für Embryonenkulturen (eine Eizelle entspricht einem Mikrotröpfchen) in 20-30 μl Embryonenkulturmedium überführt und in einem 37 °C heißen Inkubator mit 5 % CO2 und 5 %O2 inkubiert.
- Notieren Sie den Tag der ICSI als Tag 0. Kontrollieren Sie die Embryonen und bewerten Sie sie gemäß dem Istanbuler Konsens-Workshop zur Beurteilung der Embryonen von Tag 1 für die Befruchtung (ca. 18 Stunden), Tag 2 (ca. 45 Stunden) und Tag 3 (ca. 68 Stunden) für die Embryonenspaltung27.
- Waschung von Embryonen
- Bereiten Sie 20-30 μl Mikrotröpfchen des Blastozystenkulturmediums für jeden mit Mineralöl bedeckten Embryo in Gewebekulturschalen an Tag 2 in einem Inkubator mit 5 % CO2 und 5 %O2 vor.
- Bereiten Sie weitere drei mit Mineralöl bedeckte Mikrotröpfchen vor und beschriften Sie das neue Gewebekulturgeschirr zum Spülen Nr. 1-3.
- Übertragen Sie die Embryonen von Tag 3 in die Waschmikrotröpfchen. Die Embryonen werden vorsichtig abgesaugt und 3 Mal in jedem Tröpfchen mit Denudationspipetten freigesetzt.
HINWEIS: Dieses Verfahren kann auch dazu beitragen, die verbleibenden körnigen Zellen zu entfernen, die am Embryo haften. - Beobachten und beurteilen Sie die Embryonen unter dem Mikroskop am Tag 3, bevor das Medium für die morphologische Bewertung von einem Kulturmedium im Spaltstadium in ein Blastozystenkulturmedium gewechselt wurde. Wenn noch Cumuluszellen am Embryo haften, pipettieren Sie das mit Mineralöl bedeckte Tröpfchen in einem anderen vorgewärmten und äquilibrierten Blastozystenkulturmedium mit einer Stripperpipette auf und ab, bis die Cumuluszellen vollständig entfernt sind.
HINWEIS: Alle anhaftenden Cumuluszellen mussten am Tag 3 vollständig entfernt werden, bevor der Embryo von der Kulturmediumplatte im Spaltstadium auf die Kulturmediumplatte im Blastozystenstadium übertragen wurde. Alle verbleibenden Kumuluszellen stören die endgültige Analyse und liefern falsch negative Ergebnisse.
- Zwei Optionen für die Sammlung von Nährmedien
HINWEIS: Das IVF-Zentrum kann je nach den Ressourcen, Anforderungen und Präferenzen des Zentrums eine von zwei Methoden für die Entnahme von Nährmedien wählen.- Option 1: Embryonenwäsche und -kultur
HINWEIS: Diese Option gilt für IVF-Labore, die die Vitrifikation am Morgen des 5. Tages durchführen.- Der Embryo wird in vorgewärmte (37 °C) Mikrotröpfchen des Kulturmediums überführt und jeder Embryo am Nachmittag des 4. Tages nacheinander vorsichtig in 3 Mikrotröpfchen durch Pipettieren gewaschen.
- Jeder Embryo wird zur Probenentnahme in ein einzigartiges, vorgewärmtes (37 °C) einzelnes Mikrotröpfchen Kulturmedium übertragen. Das Volumen eines einzelnen Tropfens Nährmedium darf 25 μl nicht überschreiten.
- Die Blastozystenembryonenkultur wird an Tag 5/Tag 6 bei 37 °C, 5 % CO2 und 5 %O2 durchgeführt.
- Option 2: Embryonenwäsche und -kultur
HINWEIS: Diese Option ist für IVF-Labore gedacht, die die Vitrifikation am 5. Tag nachmittags oder am 6. Tag durchführen.- Übertragen Sie den Embryo in vorgewärmte (37 °C) Mikrotröpfchen von 10-15 μl Kulturmedium und waschen Sie jeden Embryo am 5. Tag sanft in 3 Mikrotröpfchen durch Pipettieren.
- Jeder Embryo wird zur Probenentnahme in ein einzigartiges, vorgewärmtes (37 °C) einzelnes Mikrotröpfchen Kulturmedium übertragen. Das Volumen eines einzelnen Tropfens Nährmedium darf 15 μl nicht überschreiten.
- Blastozystenembryonenkultur am Tag 5/Tag 6 bei 37 °C und 5 % CO2 durchführen.
- Option 1: Embryonenwäsche und -kultur
- Musterkollektion
- Stellen Sie das ICM vorsichtig in einem beträchtlichen Abstand vom Zielpunkt des Laserstrahls ein, der sich auf die Zellverbindung des Trophektoderms konzentriert, um ein kleines Loch im Trophektoderm zu erzeugen, um die Flüssigkeit aus der Blastocoelhöhle freizusetzen. Dann werden die Embryonen zur Kryokonservierung nach dem herkömmlichen Verfahren in eine Gefrierlösung gebracht.
- Transfer des Kulturmediums von jedem kultivierten Embryo in ein RNase/DNase-freies PCR-Röhrchen, das 5 μl Zelllysepuffer enthält.
- Sammeln Sie die gleiche Menge an Nährmedium, ohne für die Embryonenkultur als Negativkontrolle verwendet zu werden. Alle gesammelten Proben werden sofort in flüssigem Stickstoff eingefroren und nach der Entnahme bei −80 °C gelagert, bis sie dem NICS-Assay unterzogen werden.
- Führen Sie die Vitrifikation wie im Protokoll beschrieben durch.
3. Protokoll 2: Bibliotheksaufbau
- Lyse des Nährmediums
- Verdünnen Sie 1 μl der Positivkontrolle (10 ng humane gDNA) mit 199 μl frischem Kulturmedium. Gründlich mischen und das Röhrchen kurz zentrifugieren (200 x g für 5 s).
- 10 μl Blastozystenkulturmedium von Tag 5 bis Tag 6, verdünnte Positivkontrolle und frisches Nährmedium in neue 0,2-ml-PCR-Röhrchen überführen.
- Geben Sie 1 μl MT Enzymmischung in jedes PCR-Röhrchen und mischen Sie es gründlich durch Pipettieren und zentrifugieren Sie sofort 2-3 s lang bei 200 x g.
- Das/die PCR-Röhrchen(s) aus Schritt 3.1.3 in eine vorgeheizte NICS-Probenvorbereitungsstation geben und das Lyseprogramm wie folgt ausführen: 10 min bei 75 °C; 4 min bei 95 °C; bei 22 °C halten.
HINWEIS: Die Probenvorbereitungsstation ist vergleichbar mit einem Standard-PCR-Gerät.- Klicken Sie auf das Lysis-Symbol , um den Setup-Bildschirm aufzurufen.
- Wählen Sie Tube für den Steuerungsmodus; Eingang 10 μl für Probenvolumen; Wählen Sie Ein für Hotlid-Steuerung und geben Sie 105 °C als Temperatur ein. Wählen Sie Nein für Pause beim ersten Seg. Klicken Sie auf OK , um fortzufahren.
- Warten Sie, bis unter Verbleibenszeit -- :--:-- angezeigt wird, was das Ende des Programms anzeigt, und klicken Sie dann auf Beenden , um das Programm zu beenden.
- Beenden Sie das Programm, nachdem der Vorgang abgeschlossen ist. Fahren Sie sofort mit dem nächsten Schritt fort.
- Vorbereitung auf die Bibliothek
- Tauen Sie den Pre-Lib-Puffer auf RT auf. Durch Pipettieren gründlich mischen und sofort 2-3 s bei 200 x g zentrifugieren.
- Bereiten Sie eine Mastermischung für die Vorbibliotheksreaktion wie folgt vor: 2 μl Pre-Lib Enzymmischung zu 60 μl Pre-Lib Puffer geben, die Reaktion gründlich mischen und kurz zentrifugieren.
- Geben Sie 60 μl der Reaktionsmischung aus der Vorbibliothek in jede vorbehandelte Medienprobe aus dem vorherigen Schritt. Durch Pipettieren gründlich mischen und sofort 2-3 s bei 200 x g zentrifugieren.
- Legen Sie das/die PCR-Röhrchen(s) aus Schritt 3.2.3 in die Probenvorbereitungsstation und führen Sie das Vorbibliotheksprogramm wie folgt aus: 95 °C für 2 Minuten; 12 Zyklen mit 15 °C für 40 s, 22 °C für 40 s, 33 °C für 30 s, 65 °C für 30 s, 72 °C für 40 s, 95 °C für 10 s und 63 °C für 10 s; und bei 4 °C halten.
- Klicken Sie auf das Pre_Lib Symbol, um den Einrichtungsbildschirm aufzurufen.
- Wählen Sie Tube für den Steuerungsmodus; Eingang 70 μL für Probenvolumen; Wählen Sie Ein für Heißdeckelsteuerung und geben Sie 105 °C als Temperatur ein. Wählen Sie Nein für Pause beim ersten Seg. Klicken Sie auf OK , um fortzufahren.
- Warten Sie, bis unter Verbleibenszeit --:--:-- angezeigt wird, was das Ende des Programms anzeigt, und klicken Sie auf Beenden, um das Programm zu beenden.
- Beenden Sie das Programm, wenn der Vorgang abgeschlossen ist. Fahren Sie sofort mit dem nächsten Schritt fort.
- Vorbereitung der Bibliothek
- Den Bibliothekspuffer auf RT auftauen. Durch Pipettieren gründlich mischen und sofort 2-3 s bei 200 x g zentrifugieren.
- Bereiten Sie eine Mastermischung für die Bibliotheksreaktion wie folgt vor: 1,6 μl Library Enzyme Mix zu 60 μl Library Buffer geben, die Reaktion gründlich mischen und kurz zentrifugieren.
- Geben Sie 60 μl Bibliotheksreaktionsmischung und 2 μl Barcode-Primer zu jedem Vorbibliotheksprodukt aus Schritt 3.2.3. Die Reaktion gründlich mischen und kurz zentrifugieren.
- Legen Sie das/die PCR-Röhrchen(s) aus Schritt 3.2.3 in den Thermocycler und führen Sie das Bibliotheksvorbereitungsprogramm wie folgt durch: 94 °C für 30 s; 17 Zyklen mit 94 °C für 25 s, 62 °C für 30 s und 72 °C für 45 s); und dann bei 4 °C halten.
- Klicken Sie auf das Lib_Prep Symbol, um den Einrichtungsbildschirm aufzurufen.
- Wählen Sie Tube für den Steuerungsmodus; Eingang 130 μL für Probenvolumen; Wählen Sie Ein für Hotlid-Steuerung und geben Sie 105 °C für die entsprechende Temperatur ein. Wählen Sie Nein für Pause beim ersten Seg. Klicken Sie auf OK , um fortzufahren.
- Warten Sie, bis unter Verbleibenszeit --:--:-- angezeigt wird, was das Ende des Programms anzeigt, und klicken Sie auf Beenden, um das Programm zu beenden.
- Bibliotheksreinigung
- Nehmen Sie Magbeads vor dem Reinigungsschritt mindestens 20 Minuten lang bei 2-8 °C aus dem Lager. Vortex und Mischen Sie die Magbeads 20 s lang. Geben Sie genügend Kügelchen für den Reinigungsschritt in ein neues 1,5-ml-Mikrozentrifugenröhrchen und erwärmen Sie die Kügelchen für RT.
- Fügen Sie 1x Magbeads zu jeder Bibliothek hinzu. Durch Auf- und Abpipettieren ≥10 Mal mischen und 5 Minuten bei RT inkubieren.
HINWEIS: Geben Sie z. B. 100 μl Magbeads zu 100 μl Bibliotheksprobe. - Nach der Inkubation das Röhrchen kurz zentrifugieren und auf einen magnetischen Ständer stellen.
- Warten Sie ca. 5 Minuten, bis die Lösung klar ist. Während Sie das Röhrchen auf dem magnetischen Ständer lassen, saugen Sie die Lösung vorsichtig an und entsorgen Sie sie.
- 200 μl frisch zubereitetes 80%iges Ethanol in das Röhrchen geben. 30 s bei RT inkubieren und den Überstand vorsichtig entfernen. Wiederholen Sie den Vorgang noch einmal.
- Entfernen Sie das Ethanol so vollständig wie möglich. Lassen Sie die Perlen auf dem magnetischen Ständer ca. 5-10 Minuten bei RT an der Luft trocknen.
- Nehmen Sie das Röhrchen vom magnetischen Ständer, fügen Sie 17,5 μl Elutionspuffer hinzu und wirbeln Sie das Röhrchen auf, um die Kügelchen wieder zu suspendieren. Das Röhrchen kurz zentrifugieren und 5 min bei RT inkubieren.
- Legen Sie das Röhrchen auf den magnetischen Ständer und warten Sie, bis die Lösung klar ist. 15 μl Überstand vorsichtig in ein neues Röhrchen überführen.
- Quantifizierung von Bibliotheken
- Quantifizieren Sie gereinigte Bibliotheken unter Verwendung des Fluorometers gemäß der Bedienungsanleitung der qubit dsDNA HS Assay-Kits28. Die Ausbeute der Bibliotheken reicht von ~15 bis 300 ng.
- Bibliotheks-Pooling
- Verwenden Sie 10 Nanogramm jeder Bibliotheksprobe für das Pooling.
- Sequenzierung
- Weitere Informationen finden Sie im Benutzerhandbuch für die Sequenzierung (15027617 v01)29.
- Gereinigte Bibliothekssequenzen von 50 bp an einem einzigen Ende auf der Plattform ergaben etwa 2 Millionen Reads für jede Probe, und es wurde eine Sequenzierungstiefe von 0,03 × empfohlen.
- Datenanalyse
- Geben Sie den Namen und das Kennwort des Benutzers auf der Anmeldeseite ein
- Nachdem Sie sich beim System angemeldet haben, klicken Sie auf Analyse und eine neue Seite wird angezeigt. Klicken Sie auf der Registerkarte NICS-A auf Übermittlung erstellen . Wählen Sie dann NGS für die Plattform aus, wählen Sie Unternehmen aus, wählen Sie NICSInst für das Reagenz, geben Sie die Projektinformationen in das Feld unter Projekt-ID ein, legen Sie die Analyseeinstellungen fest und laden Sie die Dateien hoch. Nachdem alle Sequenzierungsdateien erfolgreich hochgeladen wurden, klicken Sie auf Senden , um die Analyse zu starten (Abbildung 3A).
- Klicken Sie auf Einreichungen anzeigen , um die Liste der eingereichten Projekte anzuzeigen. Sobald die Analyse abgeschlossen ist, ändert sich der Status eines Projekts in "Abgeschlossen" und im Berichtsfeld wird die Schaltfläche "Anzeigen" angezeigt. Klicken Sie auf die Schaltfläche Anzeigen , um die Übersichtstabelle der NICS-Analyse anzuzeigen (Abbildung 3B).
- Klicken Sie auf die Schaltfläche Bericht exportieren , um die Berichte zu speichern (Abbildung 3C).
HINWEIS: Für jede Analyse werden drei Dateitypen exportiert. Eine Grafikdatei, die alle CNV-Diagramme (Copy Number Variation) für jedes Chromosom und das gesamte Genom enthält, die im Ordner "graph" gespeichert werden. eine Tabelle, die die Beispiel-QC-Details dieses Analyselaufs enthält; eine Dokumentdatei, die die vom Benutzer angepassten NICS-Berichte enthält; und eine Tabelle, die die Beispielzusammenfassungsinformationen dieses Analyselaufs enthält.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
In der vorliegenden Studie wurde die vorgeschlagene Methode auf einen Patienten angewendet. Die IRB-Genehmigung und die Einwilligung nach Aufklärung wurden vor der Anwendung der NICS-Analyse eingeholt. In der vorliegenden Studie wurden 6 Blastozysten von Patientinnen entnommen und NICS an allen 6 Embryonen an Tag 4 bis Tag 5 durchgeführt. Chromosomenanomalien, die durch die balancierte Translokation der Eltern verursacht wurden, wurden in fünf Chromosomen mit dem NICS-Assay nachgewiesen; daher konnten sie nicht für die Übertragung verwendet werden (Abbildung 4A-E). Die NICS-Ergebnisse der beiden Embryonen zeigten den gleichen Karyotyp 45, und XN und -18 (×1) waren beide Deletionen des Chromosoms 18 (Abbildung 4A, B). Der Karyotyp 46, XN, -1p (pter→p21.1, ×1) ist nur der kurze Arm der Chromosom-1-Pter→p21.1-Regionendeletion (Abbildung 4D).
Die NICS-Ergebnisse zeigten die Karyotypen 46, XN, +1p (pter→p21.2, ×3) und -18(q21.32→qter, ×1), was darauf hindeutete, dass sowohl der lange Arm der Chromosom-18-q21.32→qter-Region als auch der kurze Arm der Chromosom-1-Pter→p21.2-Region dupliziert waren (Abbildung 4E). Obwohl die Karyotypen 46, XN, +5q (×4) und -8 (×1, mos) Chromosomen-5-Duplikationen sind und 8 Mosaikunterschiede aufweisen, kann der NICS-Assay alle 24 Chromosomen auf Aneuploidie untersuchen. Dieses Verfahren bietet eine neue Methode zur Übertragung einzelner Blastozysten des normalen Karyotyps.

Abbildung 1. Die vollständige Entfernung von Cumulus-Zellen. (A) Die Eizellen mit Cumuluszellen. (B) Die Eizellen ohne angehängte Kumuluszellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2. Cumuluszellen werden bei D3 aus einem Embryo entnommen, bevor sie in den BM übertragen werden. Alle anhaftenden Kumuluszellen müssen entfernt werden, bevor das Medium von der anfänglichen Spaltmittelplatte des Embryos in die Blastozystenkulturmediumplatte übergeht, was am Tag 3 nach Erreichen des 8-Zell-Stadiums der Embryonen geschieht. Alle Kumuluszellen, die nicht entfernt werden, stören die endgültige Analyse und führen zu falsch negativen Ergebnissen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3. Datenanalyse. (A) Für die Benutzeranwendung gibt es verschiedene Optionen. Für die Sequenzierungsplattform können Benutzer Illumina, Ion Torrent oder MGI wählen. Die Benutzer können wählen, ob die Geschlechtsinformationen gemeldet werden. Nachdem Sie die obige Parametereinstellung abgeschlossen haben, klicken Sie auf das Kästchen unter Datei-Upload und wählen Sie die entsprechenden Sequenzdateien zum Hochladen aus. Wählen Sie für Illumina die Dateien mit der Erweiterung fastq.gz aus. Klicken Sie auf Senden, um die Analyse nach erfolgreichem Hochladen zu starten. (B) Die Ansicht der Übersichtstabelle. Die Übersichtstabelle besteht aus den folgenden Informationen: Beispielname: Der Name jedes NICS-Beispiels wird aufgelistet. Daten-QC: Gibt an, ob die Sequenzierungsdatei die QC für die NICS-Analyse besteht. AI-Bewertung: Die Bewertung (A, B oder C) für jede NICS-Probe; AI_Rating Interpretation: Bewertung des Einnistungspotenzials des Embryos; AI-Benotung: Die Punktzahl für jede NICS-Stichprobe; CNV-Diagramm (Whole Genome): Zeigen Sie die CNV-Profile aller Chromosomen an; (c) Die Seite "Bericht speichern". Klicken Sie auf die Schaltfläche Bericht exportieren neben der Zusammenfassung der Ergebnisse. Wählen Sie die Informationen aus, die im Abschlussbericht angezeigt werden sollen, und klicken Sie auf Exportieren. Die Berichte werden im Download-Ordner Ihres Computers gespeichert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4. Embryonen-Screening und -Selektion mittels NICS bei einer Patientin. Insgesamt sechs Embryonen entwickelten sich erfolgreich bis zum Blastozystenstadium, und von jedem Embryo wurde Tag-4-Tag5-Kulturmedium für den NICS-Assay entnommen. (A) und (B) sind die NICS-Ergebnisse der beiden Blastozysten-Embryonen, die den gleichen Karyotyp 45, XN, -18(×1) aufweisen und beide Chromosom-18-Deletionen aufweisen. (C) zeigte den Karyotyp 46, XN, +5q (×4), -8(×1, mos) ist Chromosom-5-Duplikation und 8-Mosaik. (D) zeigte, dass der Karyotyp 46, XN, -1p (pter→p21.1, ×1) nur der kurze Arm der Chromosom-1-Pter→p21.1-Regionendeletion ist, während (E) zeigte, dass der Karyotyp 46, XN, +1p (pter→p21.2, ×3), -18(q21.32→qter, ×1) ein kurzer Arm der Chromosom-1-Pter→P21.2-Region und ein langer Arm des Chromosoms 18 q21.32 → qter-Region (F) eine ausgewogene chromosomale Zusammensetzung zeigte. Die x-Achse bedeutet 22 Autosomen in Rot und Blau, die y-Achse gibt die Kopiennummer jedes Autosoms an. Die grauen Punkte sind die Linealskala der Kopiennummernantwort, jedes Abschnittsfensters und der normale Karyotyp der Kopienzahl muss 2 sein. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle S1. Die Erfolgsraten des DNA-Nachweises Option 1 und Option 2. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle S2. Die Konkordanz zwischen NICS und PGT-A in verschiedenen Optionen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Modifikationen und Fehlerbehebung
Wenn die NICS-Ergebnisse mit elterlichem genetischen Material verunreinigt sind, stellen Sie sicher, dass alle Cumulus-Corona-Radiata-Zellen entfernt werden und stellen Sie sicher, dass eine ICSI zur Befruchtung durchgeführt wird. Unsachgemäße Medienlagerung oder Template-Vorbereitungsprozesse werden vermieden, die die DNA abbauen können. Der Arbeitsraum wurde gründlich mit DNase- und RNase-Dekontaminationsreagenzien gereinigt. Um eine Kontamination durch andere Embryonen zu vermeiden, wurde ab Tag 4 immer ein Embryo in einem einzigen Mediumtröpfchen kultiviert, um eine Kreuzkontamination zu vermeiden. Das Phänomen der Kontamination wird minimiert, wenn die Platzierung der Embryonen in den endgültigen Kulturtropfenverzögert wird 30,31,32,33. Um die mütterliche Kontamination zu minimieren, modifizierte Kuznyetsov34 die Embryokulturverfahren von Tag 0 auf Tag 4, einschließlich der sorgfältigen Entfernung der verbleibenden Koronazellen durch Pipettieren und Spülen.
Lane et al.30 zeigen, dass bei der Einnahme des Embryokulturmediums von Tag 4 bis Tag 5 die Genauigkeit des Euploidie-Nachweises verbessert wird, die Konsistenz der Embryoploidie mehr als 95% beträgt und die Konsistenz des Geschlechtschromosoms 100% erreicht. Lledo et al.33 fanden heraus, dass die Koinzidenzrate zwischen dem Tag-3-Tag-5-Kulturmedium und TE-Proben 74,6 % und 92,0 % betrug, wenn die Embryonen von Tag 4 bis Tag 6 kultiviert wurden.
Auch unsere internen Daten stützen diese Schlussfolgerung , wie aus Tabelle S1 hervorgeht. Im Vergleich zur herkömmlichen Tag-3-Tag-5-Kulturmethode wurden die Granulosazellen aufgrund eines weiteren Wechsels des Nährmediums an Tag 4 oder Tag 5 weiter entfernt. Wir stellen interne Daten (Tabelle S1) zur Verfügung, die zeigen, dass unsere beiden Methoden (Option 1 und Option 2) im Vergleich zu PGT-A eine gute Konsistenz aufweisen, was besser ist als die Probenahmemethode ohne die gründliche Entfernung von CC.
IF-amplifizierte Produkte traten in der Negativkontrolle auf, und externe DNA-Materialien könnten das Reagenz oder den Arbeitsraum kontaminiert haben. Der Arbeitsbereich sollte durch DNA/RNA-entfernende Reagenzien gereinigt werden, es sollten nukleasefreie Materialien verwendet werden und die Reagenzien sollten nach dem ersten Gebrauch aliquotiert werden.
Unterschiede in den Erfolgsquoten zwischen Option 1 und Option 2 werden in Tabelle S1 und Tabelle S2 erörtert.
Einschränkungen des NICS-Assays
Es gibt zwei Haupteinschränkungen von NICS. 1) Vor der ICSI müssen alle Kumuluszellen (i.d.R. mütterlichen Ursprungs, meist normale Chromosomenzusammensetzung) entfernt werden. Wenn die Entfernung unvollständig ist, können die Kumuluszellen während der Embryonalentwicklung DNA freisetzen und die externe DNA wird vervielfältigt, was die Ursache für einen falsch negativen Nachweis sein kann. 2) Es ist schwierig, die an der Zona pellucida befestigten Spermien zu entfernen, und es wird dringend empfohlen, das NICS-Verfahren mit ICSI durchzuführen. Obwohl der regelmäßige Austausch von Spaltmedien an Tag 3 die Möglichkeit einer Kontamination durch Kumuluszellen und überflüssige Spermien verringern kann, muss diese Kontamination minimiert werden, wenn NICS in der klinischen IVF verwendet wird. Es wurde jedoch eine Methode zum Nachweis von NICS in IVF-Embryonen entwickelt, einschließlich der Funktion der Erkennung von exogener DNA, die in naher Zukunft demonstriert werden soll.
In dieser Studie wurden die Unterschiede zwischen verschiedenen Medien nicht verglichen, da in groß angelegten klinischen Studien Nährmedien verglichen wurden. Acht Zentren verwendeten 4 verschiedene Nährmedien, sequentiell und kontinuierlich, und 2 verschiedene Prozentsätze der Albumin-Supplementierung (5 % und 10 %), und diese Unterschiede hatten keine signifikanten Auswirkungen auf die Genauigkeit der embryonalen cfDNA-Ergebnisse31. Diese Ergebnisse unterstützen die potenzielle Anwendbarkeit der embryonalen cfDNA-Analyse auf jedes IVF-Labor, wenn es unter dem spezifischen Protokoll arbeitet.
Bedeutung in Bezug auf bestehende Methoden
Die NICS-Methode vermeidet eine Embryonenbiopsie und verbessert so die Anwendungssicherheit erheblich. Im Vergleich zu Blastozysten ist NICS eine einfache, zeitsparende, sensitive und reproduzierbare Präimplantations-Screening-Technik, die für assistierte reproduktive Populationen mit einer hohen Wahrscheinlichkeit von Aneuploidie geeignet ist. Im Gegensatz zur invasiven Biopsie, die für das Blastozystenbiopsieverfahren beträchtliche und professionelle Kenntnisse erfordert, kann NICS umfassend angewendet werden, da die einfache Entnahme des verbrauchten Mediums nur dem regulären Ablauf der IVF19 folgt und in einigen Ländern keine PGS/PID-Qualifikation erfordert.
Zukünftige Anwendungen
NICS hat das Potenzial für eine breite Anwendbarkeit für das Chromosomen-Screening in der klinischen IVF, nicht nur für ICSI, sondern auch für IVF-Embryonen. Obwohl die ICSI sehr empfohlen wird, sind Methoden zur Entfernung der an der Zona pellucida befestigten Spermien erforderlich, um den Einfluss der Spermien zu verhindern.
Die morphologische Beurteilung ist eine traditionelle Methode zur Beurteilung von Embryonen, aber in den meisten Fällen können chromosomal abnorme Embryonen morphologisch ähnlich aussehen wie chromosomal normale (euploide) Embryonen. Die Kombination der morphologischen Beurteilung mit dem NICS-Assay beim Transfer von ploiden Embryonen mit guter Morphologie in die Gebärmutter könnte die laufenden Schwangerschaftsraten und die Lebendgeburtenrate verbessern. Eine randomisierte klinische Studie wird durchgeführt, um die klinische Wirksamkeit des Einzelembryotransfers mit der NICS-Technologie zu bewerten.
Kritische Schritte im Protokoll
Alle Cumulus-Corona radiata-Zellen müssen vor der Befruchtung aus den Eizellen entfernt werden. Die Eizellen wurden durch intrazytoplasmatische Spermieninjektion (ICSI) befruchtet. Die Zugabe von humanen Proteinen/Nahrungsergänzungsmitteln zum Nährmedium wurde vermieden. Das Nährmedium wurde an Tag 4 gewechselt und an Tag 5 bis Tag 6 gesammelt, wenn sich die Blastozysten vollständig ausgebreitet hatten. Die Embryonen wurden ab Tag 4 in einzelnen Tröpfchen des Nährmediums kultiviert. Bei der Entnahme des Nährmediums wurden die Transferpipetten zwischen den Proben gewechselt, um eine Kontamination zu vermeiden.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yaxin Yao, Jieliang Ma, Jing Wang und Sijia Lu sind Mitarbeiter von Yikon Genomics Co., Ltd.
Acknowledgments
Die Autoren danken Shiping Bo und Shujie Ma für ihre Unterstützung bei der NGS-Datenanalyse. Finanzierung: Diese Arbeit wurde durch das National Key Research and Development Program unterstützt (Grant No. 2018YFC1003100).
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
References
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Jr Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T. Jr, Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid? Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- Thermo Fisher Scientific. Qubit dsDNA HS Assay Kit. , Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015).
- Miseq system use guide. , Available from: https://support.illumina.com/downloads/miseq_system user _ guide 15027617.html (2016).
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Tags
Chromosomen-Screening menschliche Präimplantationsembryonen verbrauchtes Kulturmedium Probenentnahme Chromosomen-Ploidie-Analyse In-vitro-Fertilisation (IVF) Präimplantations-Gentest auf Aneuploidie (PGT-A) Trophektoderm (TE) innere Zellmasse (ICM) falsch positive und negative Ergebnisse NICS-Technologie DNA-Sequenzierung Probenahme von Kulturmedien Amplifikation des gesamten Genoms (WGA) Bibliotheksvorbereitung NGS-Datenanalyse Kryokonservierung IVF-LaborErratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021.
Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
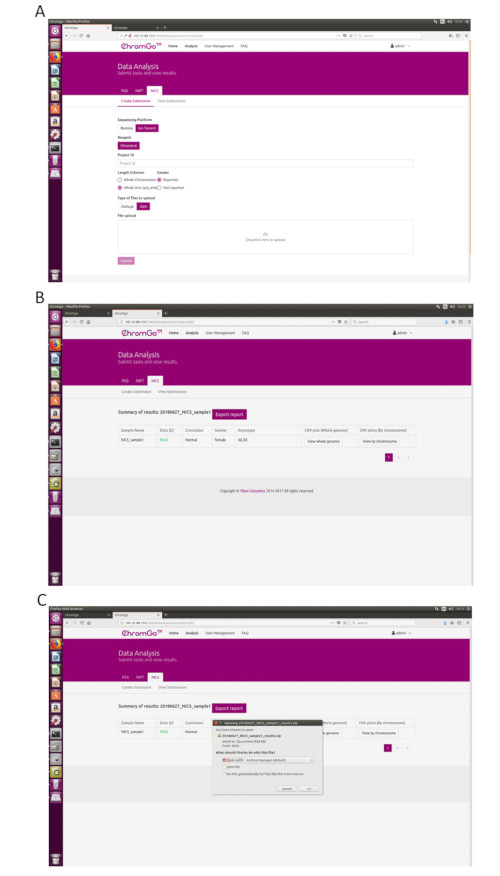
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
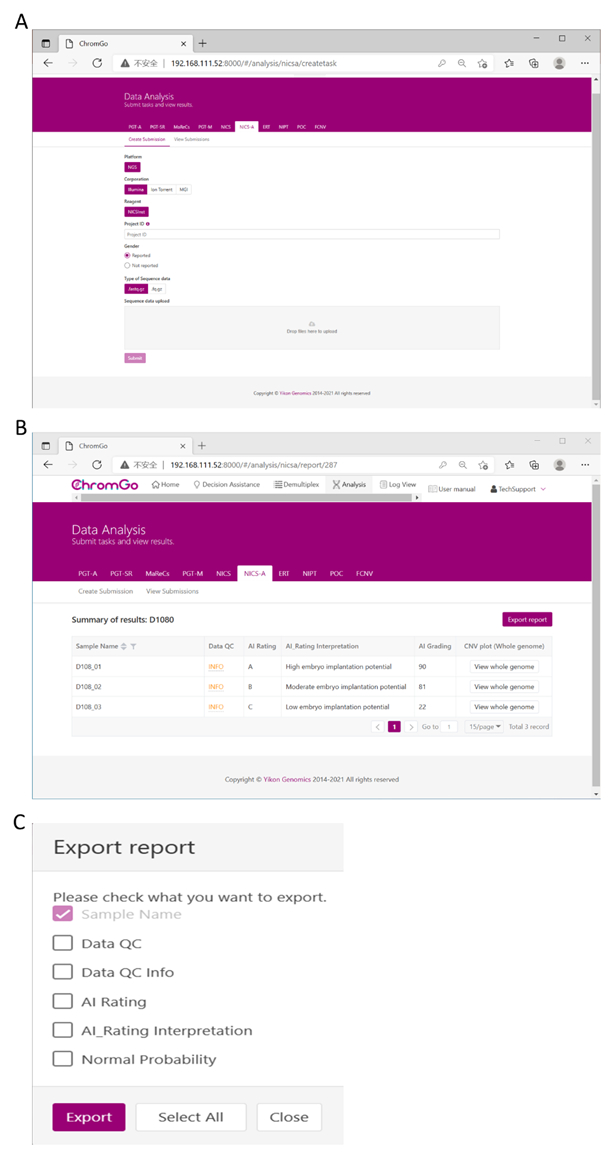
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.



