ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
El presente estudio reporta un protocolo para el tamizaje cromosómico de embriones humanos que utiliza medio de cultivo gastado, lo que evita la biopsia embrionaria y permite la identificación de ploidía cromosómica mediante NGS. El presente artículo presenta el procedimiento detallado, que incluye la preparación del medio de cultivo, la amplificación del genoma completo (WGA), la preparación de la biblioteca de secuenciación de nueva generación (NGS) y el análisis de datos.
Abstract
En la fertilización clínica in vitro (FIV), el método predominante para el PGT-A requiere la biopsia de unas pocas células del trofoectodermo (TE). Este es el linaje que forma la placenta. Este método, sin embargo, requiere habilidades especializadas, es invasivo y sufre de falsos positivos y negativos porque el número de cromosomas en el TE y la masa celular interna (MCI), que se desarrolla en el feto, no siempre son los mismos. La NICS, una tecnología que requiere la secuenciación del ADN que se libera en el medio de cultivo tanto desde TE como desde ICM, puede ofrecer una salida a estos problemas, pero se ha demostrado previamente que tiene una eficacia limitada. El presente estudio informa el protocolo completo de NICS, que incluye métodos de muestreo en medio de cultivo, amplificación del genoma completo (WGA) y preparación de bibliotecas, y análisis de datos de NGS mediante software de análisis. Teniendo en cuenta los diferentes tiempos de criopreservación en los diferentes laboratorios de embriones, los embriólogos tienen dos métodos para recolectar el medio de cultivo de embriones que se pueden seleccionar de acuerdo con las condiciones reales del laboratorio de FIV.
Introduction
Las tecnologías de reproducción asistida (TRA) se han utilizado cada vez más para el tratamiento de la infertilidad. Sin embargo, la tasa de éxito de las TRV, como la FIV, ha sido limitada, y la tasa de pérdida de embarazos es significativamente mayor que la de la población normal1. La principal causa de estos problemas son las anomalías cromosómicas, que existen comúnmente en los embriones humanos preimplantacionales2. El PGT-A es un método eficaz para evaluar el equilibrio cromosómico de los embriones antes de la implantación 3,4. Algunos estudios han demostrado que el PGT-A puede reducir la tasa de aborto y mejorar la tasa de embarazo 5,6,7,8. Sin embargo, el PGT-A requiere conocimientos técnicos complejos que requieren formación y experiencia específicas. El procedimiento invasivo de biopsia embrionaria también podría causar daño a los embriones9. Los estudios han demostrado que la biopsia de blastómeros puede dificultar el desarrollo posterior, y el número de TE biopsiadas puede afectar las tasas de implantación10. A pesar de que el problema de la biopsia embrionaria a largo plazo aún no ha sido evaluado a fondo en humanos, los estudios en animales han demostrado sus influencias negativas en el desarrollo embrionario11,12,13.
Informes anteriores indicaron que se secretaron trazas de materiales de ADN en el medio de cultivo durante el desarrollo embrionario, y se han realizado esfuerzos para realizar un cribado cromosómico completo (CCS) utilizando el medio de cultivo de embriones gastados 14,15,16,17,18. Sin embargo, las tasas de detección y la precisión de las pruebas no han cumplido con los requisitos para un uso clínico extensivo. El presente estudio reportó una mejoría en el ensayo NICS para aumentar las tasas de detección, así como la precisión de la prueba NICS19. En los últimos años, el líquido blastocelele (BF) se ha estudiado como una muestra analítica de PGT-A mínimamente invasiva. Sin embargo, la proporción de amplificación exitosa de todo el genoma y ADN detectable en muestras de líquido blastocisto oscila entre el 34,8% y el 82%20,21,22. El volumen de LM reportado en varios estudios oscila entre 0,3 nL y 1 μL. En vista de la baja cantidad de ADN en BF, es posible aumentar la cantidad de ADN libre de células mezclando líquido de blastocisto y medio de cultivo para mejorar la tasa de éxito y la consistencia de la detección. Kuznyetsov y cols.23 y Li et al.24 trataron la zona pelúcida con un láser y liberaron líquido blastocisto en el medio de cultivo para mejorar la cantidad total de ADN embrionario, y la tasa de amplificación de las muestras combinadas de medio/BF después de WGA fue del 100% y del 97,5%, respectivamente. Jiao et al.25 también obtuvieron una tasa de éxito de amplificación del 100% utilizando el mismo método.
El presente estudio reporta un protocolo detallado que incluye la preparación de la muestra de medios gastados, la preparación de NGS y el análisis de datos. Mediante la extracción cuidadosa de células cúmulos de ovocitos, el presente estudio realizó una inyección intracitoplasmática de espermatozoides únicos (ICSI) y un cultivo de blastocisto. El medio gastado del día 4 al día 5 y al día 6 se recolectó para la preparación de la biblioteca WGA y NGS. Mediante el uso de la tecnología NICS, el presente estudio agilizó los pasos de preparación de la biblioteca WGA y NGS en aproximadamente 3 h y obtuvo resultados de CCS de forma no invasiva en aproximadamente 9 h.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Se obtuvo el permiso ético del Comité de Ética del Tercer Hospital de la Universidad de Pekín.
1. Preparación
NOTA: Los materiales y equipos necesarios se enumeran en la Tabla de materiales.
- Reactivos
- Precalentar y equilibrar (equilibrado) 20-30 μL de medio de gametos/medio de fertilización y medio de cultivo de escisión/blastocisto (cubierto con aceite mineral) e hialuronidasa (en un tubo bien tapado) a 37 °C, 5% de CO2 y 5% deO2 en una incubadora Tri-gas durante la noche antes de su uso.
- Precalentar hialuronidasa a 37 °C sobre una superficie de trabajo en una campana extractora.
- Prepare el tampón de vitrificación y los reactivos de recolección de muestras de acuerdo con las instrucciones del fabricante.
- Herramientas
- Prepare pipetas de recogida y transferencia de muestras (diámetro interior de ~200 a 250 μm), pipetas de denudación/extracción (diámetro interior de ≥150 μm, ~130-140 μm y ~120 μm) y pipetas para lavado (diámetro interior de ~150 μm) tirando de pipetas Pasteur de vidrio para generar puntas finas abiertas pulidas al fuego.
NOTA: Las pipetas utilizadas para la recolección/transferencia de muestras, la denudación y el lavado se pueden comprar directamente. Las agujas de sujeción y las agujas de inyección también se pueden comprar directamente.
- Prepare pipetas de recogida y transferencia de muestras (diámetro interior de ~200 a 250 μm), pipetas de denudación/extracción (diámetro interior de ≥150 μm, ~130-140 μm y ~120 μm) y pipetas para lavado (diámetro interior de ~150 μm) tirando de pipetas Pasteur de vidrio para generar puntas finas abiertas pulidas al fuego.
2. Protocolo 1: Recogida de muestras
- Pretratamiento del complejo ovocito-corona-cúmulo (OCCCs) antes de la digestión con hialuronidasa
- Logre la estimulación ovárica con preparaciones de hormona estimulante del folículo (FSH) y gonadotropina menopáusica humana (hMG). Cuando el folículo principal esté >18 mm, use 10,000 UI de gonadotropina coriónica (hCG) para la maduración final del ovocito.
- Realizar la extracción de ovocitos 36 h después del disparo gatillo. Recoja y transfiera los ovocitos a placas de cultivo de tejidos con 2,5 ml de m-HTF precalentado cubierto por aceite mineral.
- Transferir rápidamente los OCCC al pocillo central de una placa de cultivo de órganos que contenga 1 mL de medio de fertilización utilizando una pipeta de transferencia y luego incubar con los ovocitos a 37 °C en una incubadora de 5% de CO 2 y 5% de O 2 durante2-4 h.
- Digiera los OCCC con hialuronidasa añadiendo 1 ml de hialuronidasa precalentada a 37 °C (80 UI/ml) al pocillo central de una placa de cultivo de órganos que contenga OCCC (paso 2.1.3). Mantenga la concentración final de hialuronidasa en 40 UI/ml y mezcle bien.
- Incubar los OCCC en una plataforma térmica a 37 °C durante 2 min. Observe los cambios bajo un microscopio cada 30 s hasta que solo queden 1-2 capas de células de la granulosa.
- Denudación de las células de la granulosa
- Transfiera rápidamente los OCCC digeridos en la placa de cultivo para su manipulación de ovocitos y cúbralos con aceite mineral en cada pocillo.
- Observe las células de la granulosa separadas bajo un microscopio. Aspire suavemente y libere los ovocitos 5 veces para eliminar las células residuales de la granulosa alrededor de los ovocitos.
- Repita el paso anterior en los 3 pocillos restantes para eliminar completamente las células de la granulosa.
NOTA: Los pasos anteriores (2.1-2.3) se pueden realizar de acuerdo con la operación rutinaria de cada laboratorio.
- Evaluación del ovocito
- Evalúe la integridad de la extracción de células de la granulosa con un microscopio. Si las células no se pudieron eliminar por completo, entonces la retención de 5 o menos células de la granulosa es aceptable en este momento.
NOTA: Si las células del cúmulo todavía están adheridas al ovocito, el remanente se puede eliminar más tarde en el día 3 antes de que el embrión se transfiera del medio de cultivo en etapa de escisión al medio de cultivo en etapa de blastocisto.
- Evalúe la integridad de la extracción de células de la granulosa con un microscopio. Si las células no se pudieron eliminar por completo, entonces la retención de 5 o menos células de la granulosa es aceptable en este momento.
- Después de realizar la inyección intracitoplasmática de espermatozoides (ICSI)26, transferir los ovocitos a microgotas de medio de cultivo embrionario de escisión de 20-30 μL (un ovocito corresponde a una microgota) utilizando pipetas de transferencia e incubar en una incubadora de 37 °C, 5% CO2 y 5%O2 .
- Registre el día de ICSI como día 0. Comprobar los embriones y puntuar según el taller de consenso de Estambul sobre evaluación embrionaria del día 1 para la fecundación (aproximadamente 18 h), el día 2 (aproximadamente 45 h) y el día 3 (aproximadamente 68 h) para la escisión embrionaria27.
- Lavado de embriones
- Preparar 20-30 μL de microgotas de medio de cultivo de blastocisto para cada embrión cubierto con aceite mineral en placas de cultivo de tejidos en el día 2 en una incubadora a 37 °C, 5% CO 2 y 5% O2.
- Prepare otras tres microgotas cubiertas con aceite mineral y etiquete las nuevas placas de cultivo de tejidos para el lavado No. 1-3.
- Transfiera los embriones del día 3 a las microgotas de lavado. Aspirar suavemente y liberar los embriones 3 veces en cada gota utilizando pipetas de denudación.
NOTA: Este procedimiento también puede ayudar a eliminar las células granulares residuales adheridas al embrión. - Observar y evaluar los embriones bajo un microscopio el día 3 antes de que el medio se cambiara de medio de cultivo en etapa de escisión a medio de cultivo de blastocisto para la puntuación morfológica. Si las células del cúmulo todavía estaban unidas al embrión, pipetear apropiadamente hacia arriba y hacia abajo en otra gota de medio de cultivo de blastocisto precalentada y equilibrada cubierta con aceite mineral con una pipeta separadora hasta que las células del cúmulo se eliminaron por completo.
NOTA: Todas las células del cúmulo adheridas tuvieron que ser extraídas por completo el día 3 antes de que el embrión se transfiriera de la placa de medio de cultivo en etapa de escisión a la placa de medio de cultivo en etapa de blastocisto. Cualquier cúmulo restante interferirá en el análisis final y dará resultados falsos negativos.
- Dos opciones para la recolección del medio de cultivo
NOTA: El centro de FIV puede elegir uno de los dos métodos para la recolección del medio de cultivo en función de los recursos, demandas y preferencias del centro.- Opción 1: Lavado y cultivo de embriones
NOTA: Esta opción es para los laboratorios de FIV que realizan vitrificación en la mañana del día 5.- Transfiera el embrión a microgotas precalentadas (37 °C) de medio de cultivo y lave suavemente cada embrión en serie en 3 microgotas pipeteando el día 4 por la tarde.
- Transfiera cada embrión a una única microgota precalentada (37 °C) de medio de cultivo para la recogida de muestras. El volumen de una sola gota de medio de cultivo no puede exceder de 25 μL.
- Realizar cultivo embrionario de blastocisto el día 5/día 6 a 37 °C, 5% CO 2 y 5% O2.
- Opción 2: Lavado y cultivo de embriones
NOTA: Esta opción es para laboratorios de FIV que realizan vitrificación el día 5 por la tarde o el día 6.- Transfiera el embrión a microgotas precalentadas (37 °C) de medio de cultivo de 10-15 μL y lave suavemente cada embrión en serie en 3 microgotas pipeteando el día 5.
- Transfiera cada embrión a una única microgota precalentada (37 °C) de medio de cultivo para la recogida de muestras. El volumen de una sola gota de medio de cultivo no puede exceder de 15 μL.
- Realizar cultivo embrionario de blastocisto el día 5/día 6 a 37 °C y 5% de CO2.
- Opción 1: Lavado y cultivo de embriones
- Recogida de muestras
- Ajuste suavemente el ICM a una distancia considerable del punto objetivo del rayo láser, que se enfoca en la unión celular del trofoectodermo para generar un pequeño orificio en el trofoectodermo para liberar el líquido de la cavidad del blastocelo. A continuación, los embriones se trasladan a una solución de congelación para su criopreservación según el proceso convencional.
- Transfiera el medio de cultivo de cada embrión cultivado a un tubo de PCR libre de RNasa/DNasa que contenga 5 μL de tampón de lisis celular.
- Recolectar la misma cantidad de medio de cultivo sin ser utilizado para el cultivo de embriones como control negativo. Congelar todas las muestras recogidas inmediatamente en nitrógeno líquido y luego almacenarlas a -80 °C después de recogerlas hasta que se sometan al ensayo NICS.
- Realice la vitrificación como se describe en el protocolo.
3. Protocolo 2: Construcción de la biblioteca
- Lisis del medio de cultivo
- Diluir 1 μL de control positivo (10 ng de ADNg humano) con 199 μL de medio de cultivo fresco. Mezclar bien y centrifugar el tubo brevemente (200 x g durante 5 s).
- Transfiera 10 μL de medio de cultivo de blastocistos de 5 a 6 días, control positivo diluido y medio de cultivo fresco a tubos de PCR nuevos de 0,2 ml.
- Añadir 1 μL de MT Enzyme Mix a cada tubo de PCR y mezclar bien pipeteando y centrifugando inmediatamente durante 2-3 s a 200 x g.
- Coloque los tubos de PCR del paso 3.1.3 en una estación de preparación de muestras NICS precalentada y ejecute el programa de lisis de la siguiente manera: 10 min a 75 °C; 4 min a 95 °C; mantener a 22 °C.
NOTA: La estación de preparación de muestras es comparable a una máquina de PCR estándar.- Haga clic en el icono Lysis para ingresar a la pantalla de configuración.
- Seleccione Tubo para el modo de control; entrada 10 μL para el volumen de la muestra; seleccione Activado para el control de tapa caliente e introduzca 105 °C para la temperatura. Seleccione No para Pausa en el primer intervalo. Haga clic en Aceptar para continuar.
- Espere hasta que el tiempo restante muestre --:-- :--, que indica el final del programa y, a continuación, haga clic en Detener para finalizar el programa.
- Detenga el programa una vez finalizado el proceso. Continúe con el siguiente paso inmediatamente.
- Preparación previa a la biblioteca
- Descongele el tampón Pre-Lib a RT. Mezcle bien pipeteando y centrifugue inmediatamente durante 2-3 s a 200 x g.
- Prepare una mezcla maestra para la reacción de prebiblioteca de la siguiente manera: agregue 2 μL de mezcla de enzimas Pre-Lib a 60 μL de tampón Pre-Lib, mezcle bien la reacción y centrifugue brevemente.
- Agregue 60 μL de mezcla de reacción de prebiblioteca en cada muestra de medio pretratada del paso anterior. Mezclar bien pipeteando y centrifugar inmediatamente durante 2-3 s a 200 x g.
- Coloque los tubos de PCR del paso 3.2.3 en la estación de preparación de muestras y ejecute el programa de prebiblioteca de la siguiente manera: 95 °C durante 2 min; 12 ciclos de 15 °C durante 40 s, 22 °C durante 40 s, 33 °C durante 30 s, 65 °C durante 30 s, 72 °C durante 40 s, 95 °C durante 10 s y 63 °C durante 10 s; y mantener a 4 °C.
- Haga clic en el icono Pre_Lib para entrar en la pantalla de configuración.
- Seleccione Tubo para el modo de control; entrada 70 μL para el volumen de la muestra; seleccione Activado para Control de tapa caliente e introduzca 105 °C para la temperatura. Seleccione No para Pausa en el primer intervalo. Haga clic en Aceptar para continuar.
- Espere hasta que el tiempo restante muestre --:-- :--, que indica el final del programa, y haga clic en Detener para finalizar el programa.
- Detenga el programa cuando se complete el proceso. Continúe con el siguiente paso inmediatamente.
- Preparación de la biblioteca
- Descongele el tampón de biblioteca a RT. Mezcle bien pipeteando y centrifugue inmediatamente durante 2-3 s a 200 x g.
- Prepare una mezcla maestra para la reacción de biblioteca de la siguiente manera: agregue 1,6 μL de mezcla de enzimas de biblioteca a 60 μL de tampón de biblioteca, mezcle bien la reacción y centrifugue brevemente.
- Agregue 60 μL de mezcla de reacción de biblioteca y 2 μL de cebador de código de barras a cada producto de prebiblioteca del paso 3.2.3. Mezcle bien la reacción y centrifugue brevemente.
- Coloque los tubos de PCR del paso 3.2.3 en el termociclador y ejecute el programa de preparación de la biblioteca de la siguiente manera: 94 °C durante 30 s; 17 ciclos de 94 °C durante 25 s, 62 °C durante 30 s y 72 °C durante 45 s); y luego mantener a 4 °C.
- Haga clic en el icono Lib_Prep para entrar en la pantalla de configuración.
- Seleccione Tubo para el modo de control; entrada 130 μL para el volumen de la muestra; seleccione Activado para el control de la tapa caliente e introduzca 105 °C para la temperatura correspondiente. Seleccione No para Pausa en el primer intervalo. Haga clic en Aceptar para continuar.
- Espere hasta que el tiempo restante muestre --:-- :--, que indica el final del programa, y haga clic en Detener para finalizar el programa.
- Purificación de la biblioteca
- Retire las Magbeads del almacenamiento a 2-8 °C durante al menos 20 minutos antes de la etapa de purificación. Vórtice y mezcle las Magbeads durante 20 s. Dispense suficientes perlas para el paso de purificación en un nuevo tubo de microcentrífuga de 1,5 ml y caliente las perlas a RT.
- Añade 1x Magbeads a cada biblioteca. Mezclar pipeteando hacia arriba y hacia abajo ≥10 veces e incubar a RT durante 5 min.
NOTA: Por ejemplo, agregue 100 μL de Magbeads a 100 μL de muestra de biblioteca. - Después de la incubación, centrifugue el tubo brevemente y colóquelo en un soporte magnético.
- Espere aproximadamente 5 minutos hasta que la solución se aclare. Mientras mantiene el tubo en el soporte magnético, aspire con cuidado la solución y deséchela.
- Añadir 200 μL de etanol al 80% recién preparado al tubo. Incubar a RT durante 30 s y retirar con cuidado el sobrenadante. Repite una vez más.
- Retire el etanol lo más completamente posible. Secar al aire las cuentas en el soporte magnético durante aproximadamente 5-10 minutos a RT.
- Retire el tubo del soporte magnético, agregue 17,5 μL de tampón de elución y haga un vórtice en el tubo para volver a suspender las perlas. Centrifugar el tubo brevemente e incubar a RT durante 5 min.
- Coloque el tubo en el soporte magnético y espere hasta que la solución se aclare. Transfiera con cuidado 15 μL de sobrenadante a un tubo nuevo.
- Cuantificación de bibliotecas
- Cuantifique las bibliotecas purificadas utilizando el fluorómetro de acuerdo con la guía del usuario de los kits de ensayo qubit dsDNA HS28. El rendimiento de las bibliotecas oscila entre ~15 y 300 ng.
- Agrupación de bibliotecas
- Utilice 10 nanogramos de cada muestra de biblioteca para la agrupación.
- Secuenciación
- Consulte la guía del usuario de secuenciación (15027617 v01)29.
- Las secuencias de biblioteca purificadas de 50 pb en un solo extremo de la plataforma produjeron aproximadamente 2 millones de lecturas para cada muestra, y se recomendó una profundidad de secuenciación de 0,03 ×.
- Análisis de datos
- Introduzca el nombre y la contraseña de los usuarios en la página de inicio de sesión
- Después de iniciar sesión en el sistema, haga clic en Análisis y aparecerá una nueva página. Haga clic en Crear envío en la pestaña NICS-A. A continuación, elija NGS para la plataforma, seleccione corporación, elija NICSInst para el reactivo, introduzca la información del proyecto en el cuadro de ID de proyecto, establezca las preferencias de análisis y cargue los archivos. Una vez que todos los archivos de secuenciación se hayan cargado correctamente, haga clic en Enviar para iniciar el análisis (Figura 3A).
- Haga clic en Ver envíos para mostrar la lista de proyectos enviados. Una vez finalizado el análisis, el estado de un proyecto pasará a ser Completado y aparecerá un botón Mostrar en el campo del informe. Haga clic en el botón Mostrar para ver la tabla de resumen del análisis de NICS (Figura 3B).
- Haga clic en el botón Exportar informe para guardar los informes (Figura 3C).
NOTA: Se exportarán tres tipos de archivos para cada análisis. Un archivo gráfico que incluye todos los gráficos de variación del número de copias (CNV) para cada cromosoma y genoma completo, que se almacenará en la carpeta "graph"; una hoja de cálculo que contiene los detalles de control de calidad de muestra de esta ejecución de análisis; un archivo de documento que contiene los informes NICS personalizados por el usuario; y una hoja de cálculo que contiene la información de resumen de ejemplo de esta ejecución de análisis.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
En el presente estudio se aplicó el método propuesto a un paciente. Se obtuvo la aprobación del IRB y el consentimiento informado antes de la aplicación del análisis NICS. En el presente estudio se obtuvieron 6 blastocistos de pacientes y se realizó NICS en los 6 embriones en el medio día 4 a día 5. Las anomalías cromosómicas causadas por la translocación equilibrada de los padres se detectaron en cinco de los cromosomas con el ensayo NICS; por lo tanto, no se podían utilizar para la transferencia (Figura 4A-E). Los resultados del NICS de los dos embriones mostraron el mismo cariotipo 45, y XN y -18 (×1) fueron deleciones del cromosoma 18 (Figura 4A, B). El cariotipo 46, XN, -1p (pter→p21.1, ×1) es solo el brazo corto de la deleción de la región pter→p21.1 del cromosoma 1 (Figura 4D).
Los resultados del NICS mostraron cariotipo 46, XN, +1p (pter→p21.2, ×3) y -18(q21.32→qter, ×1), lo que indicó que tanto el brazo largo de la región q21.32→qter del cromosoma 18 como el brazo corto de la región pter→p21.2 del cromosoma 1 estaban duplicados (Figura 4E). Aunque los cariotipos 46, XN, +5q (×4) y -8 (×1, mos) son duplicaciones del cromosoma 5 y muestran 8 diferencias en mosaico, el ensayo NICS puede detectar aneuploidías en los 24 cromosomas. Este proceso proporciona un nuevo método para transferir blastocistos de cariotipo normal único.

Figura 1. La integridad de la eliminación de las células del cúmulo. (A) Los ovocitos con células cúmulos. (B) Los ovocitos sin células cúmulos adheridas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2. Las células del cúmulo se extraen de un embrión en D3 antes de transferirse al BM. Todas las células del cúmulo adheridas deben eliminarse antes de que el medio cambie de la placa inicial del medio de cultivo de embriones de escisión a la placa del medio de cultivo de blastocisto, que es el día 3 después de que los embriones alcancen la etapa de 8 células. Cualquier cúmulo de células que no se elimine interferirá en el análisis final dando resultados falsos negativos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Análisis de datos. (A) Hay diferentes opciones para la aplicación de usuario. Para la corporación de plataformas de secuenciación, los usuarios pueden elegir Illumina, Ion Torrent o MGI. Los usuarios pueden elegir si se informa la información de género. Finalizada la configuración del parámetro anterior, haga clic en el cuadro debajo de Carga de archivos y elija los archivos de secuenciación apropiados para cargar. Para Illumina, elija los archivos con una extensión de fastq.gz. Haga clic en Enviar para iniciar el análisis después de cargarlo correctamente. (B) La vista del cuadro sinóptico. La tabla de resumen consta de la siguiente información: Nombre de la muestra: se muestra el nombre de cada muestra de NICS; Control de calidad de datos: indica si el archivo de secuenciación pasa el control de calidad para el análisis NICS; Clasificación AI: La calificación (A, B o C) para cada muestra de NICS; AI_Rating Interpretación: Evaluación del potencial de implantación embrionaria; Calificación AI: la puntuación de cada muestra de NICS; Gráfico CNV (Genoma completo): vea los perfiles de CNV de todos los cromosomas; (c) La página Guardar informe. Haga clic en el botón Exportar informe junto al Resumen de resultados. Seleccione la información que desea mostrar en el informe final y haga clic en Exportar. Los informes se guardarán en la carpeta de descargas de su computadora. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4. Cribado y selección de embriones mediante NICS de una paciente. Un total de seis embriones se desarrollaron con éxito hasta la etapa de blastocisto, y se recolectó el medio de cultivo Day4-Day5 de cada embrión para el ensayo NICS. (A) y (B) son los resultados de la NICS de los dos embriones de blastocisto que mostraron el mismo cariotipo 45, XN, -18(×1) son ambos deleción del cromosoma 18. (C) mostró que el cariotipo 46, XN, +5q (×4), -8(×1, mos) es duplicación del cromosoma 5 y mosaico 8. (D) mostró que el cariotipo 46, XN, -1p (pter→p21.1, ×1) es solo el brazo corto de la deleción de la región pter→p21.1 del cromosoma 1, mientras que (E) mostró cariotipo 46, XN, +1p (pter→p21.2, ×3), -18(q21.32→qter, ×1) es el brazo corto de la duplicación de la región pter→p21.2 del cromosoma 1 y el brazo largo del cromosoma 18 q21.32 → región qter (F) mostró una composición cromosómica equilibrada. El eje x significa 22 autosomas en rojo y azul, el eje y indica el número de copias de cada autosoma. Los puntos grises son la escala de la regla de la respuesta del número de copia en cada ventana de la bandeja y el cariotipo normal del número de copia debe ser 2. Haga clic aquí para ver una versión más grande de esta figura.
Tabla S1. Las tasas de éxito de la detección de ADN Opción 1 y Opción 2. Haga clic aquí para descargar esta tabla.
Tabla S2. La concordancia entre NICS y PGT-A en diferentes opciones. Haga clic aquí para descargar esta tabla.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Modificaciones y solución de problemas
Si los resultados de la NICS están contaminados con materiales genéticos parentales, asegúrese de que se eliminen todas las células del cúmulo radiata de la corona y de que se realice la ICSI para la fertilización. Se evitan los procesos inadecuados de almacenamiento en el medio o de preparación de plantillas, que pueden degradar el ADN. El espacio de trabajo se purificó a fondo con reactivos de descontaminación de DNasa y RNasa. Para evitar la contaminación de otros embriones, siempre se cultivó un embrión en una sola gota de medio para evitar la contaminación cruzada a partir del día 4. El fenómeno de contaminación se minimiza al retrasar la colocación de los embriones en el cultivo final 30,31,32,33. Para minimizar la contaminación materna, Kuznyetsov34 modificó los procedimientos de cultivo embrionario desde el día 0 hasta el día 4, incluyendo la eliminación cuidadosa de las células corona residuales mediante pipeteo y lavado.
Lane et al.30 muestran que cuando se toma el medio de cultivo embrionario del día 4 al día 5, se mejora la precisión de la detección de euploidía, la consistencia de la ploidía embrionaria es superior al 95% y la consistencia del cromosoma sexual alcanza el 100%. Lledó et al.33 encontraron que la tasa de coincidencia entre el medio de cultivo del día 3 al día 5 y las muestras de TE fue del 74,6% y del 92,0% cuando los embriones se cultivaron desde el día 4 hasta el día 6.
Nuestros datos internos también respaldan esta conclusión, como se muestra en la Tabla S1. En comparación con el método convencional de cultivo de 3 días a 5 días, las células de la granulosa se eliminaron aún más debido a un cambio más en el medio de cultivo en el día 4 o el día 5. Proporcionamos datos internos (Tabla S1) que muestran que nuestros dos métodos (opción 1 y opción 2) tienen buena consistencia en comparación con PGT-A, que es mejor que el método de muestreo sin la eliminación completa de CC.
Los productos amplificados con FI aparecieron en el control negativo, y los materiales externos de ADN pueden haber contaminado el reactivo o el espacio de trabajo. El espacio de trabajo debe limpiarse con reactivos que eliminen el ADN/ARN, se deben utilizar materiales libres de nucleasa y los reactivos deben alícuota después del primer uso.
Las diferencias en las tasas de éxito entre la Opción 1 y la Opción 2 se analizan en la Tabla S1 y la Tabla S2.
Limitaciones del ensayo NICS
Hay dos limitaciones principales de NICS. 1) Antes de la ICSI, se deben extirpar todas las células del cúmulo (generalmente de origen materno, generalmente de composición cromosómica normal). Si la eliminación es incompleta, las células del cúmulo pueden liberar ADN durante el desarrollo embrionario y el ADN externo se amplifica, lo que puede ser la causa de la detección de falsos negativos. 2) Es difícil eliminar los espermatozoides adheridos a la zona pelúcida, y se recomienda encarecidamente realizar el procedimiento NICS con ICSI. Aunque el reemplazo regular de los medios de escisión en el día 3 puede reducir la posibilidad de contaminación debido a las células del cúmulo y los espermatozoides redundantes, esta contaminación debe minimizarse si se utiliza NICS en la FIV clínica. Sin embargo, se ha desarrollado un método para detectar NICS en embriones de FIV, que incluye la función de reconocimiento de ADN exógeno, que se demostrará en un futuro próximo.
Este estudio no comparó las diferencias entre los diferentes medios, ya que los ensayos clínicos a gran escala han comparado los medios de cultivo. Ocho centros utilizaron 4 medios de cultivo diferentes, secuencial y continuo, y 2 porcentajes diferentes de suplementación con albúmina (5% y 10%), y estas diferencias no tuvieron efectos significativos en la precisión de los resultados embrionarios de ADNcf31. Estos hallazgos respaldan la posible aplicabilidad del análisis de ADNcf embrionario a todos los laboratorios de FIV cuando se trabaja bajo el protocolo específico.
Importancia con respecto a los métodos existentes
El método NICS evita la biopsia embrionaria y, por lo tanto, mejora en gran medida la seguridad de uso. En comparación con los blastocistos, la NICS es una técnica de cribado preimplantacional sencilla, que ahorra tiempo, sensible y reproducible, que es adecuada para poblaciones de reproducción asistida con una alta probabilidad de aneuploidía. A diferencia de la biopsia invasiva, que requiere un conocimiento considerable y profesional para el procedimiento de biopsia de blastocisto, la NICS se puede aplicar ampliamente, ya que su simple recolección del medio gastado solo sigue la operación regular de FIV19 y no requiere calificación PGS/PGD en algunos países.
Aplicaciones futuras
La NICS tiene el potencial de una amplia aplicabilidad para el cribado cromosómico en la FIV clínica, no sólo para la ICSI sino también para los embriones de FIV. Aunque la ICSI es muy recomendable, se requieren métodos para eliminar los espermatozoides adheridos a la zona pelúcida para evitar la influencia de los espermatozoides.
La evaluación morfológica es un método tradicional para la evaluación embrionaria, pero en la mayoría de los casos, los embriones cromosómicamente anormales pueden parecer morfológicamente similares a los embriones cromosómicamente normales (euploides). La combinación de la evaluación morfológica con el ensayo NICS cuando se transfieren embriones ploides con buena morfología al útero podría mejorar las tasas de embarazo en curso y la tasa de nacidos vivos. Se llevará a cabo un ensayo clínico aleatorizado para evaluar la eficacia clínica de la transferencia de un solo embrión utilizando la tecnología NICS.
Pasos críticos en el protocolo
Todas las células del cúmulo radiado de la corona radiata deben eliminarse de los ovocitos antes de la fecundación. Los ovocitos fueron fecundados mediante inyección intracitoplasmática de espermatozoides (ICSI). Se evitó la adición de proteínas/suplementos de origen humano al medio de cultivo. El medio de cultivo se cambió el día 4 y se colectó el día 5-día 6 cuando los blastocistos se expandieron por completo. Los embriones se cultivaron en gotas individuales de medio de cultivo a partir del día 4. Al recolectar el medio de cultivo, se cambiaron las pipetas de transferencia entre muestras para evitar la contaminación.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yaxin Yao, Jieliang Ma, Jing Wang y Sijia Lu son empleados de Yikon Genomics Co., Ltd.
Acknowledgments
Los autores desean agradecer a Shiping Bo y Shujie Ma por su ayuda en el análisis de datos de NGS. Financiamiento: este trabajo fue apoyado por el Programa Nacional de Investigación y Desarrollo Clave (Subvención No. 2018YFC1003100).
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
References
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Jr Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T. Jr, Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid? Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- Thermo Fisher Scientific. Qubit dsDNA HS Assay Kit. , Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015).
- Miseq system use guide. , Available from: https://support.illumina.com/downloads/miseq_system user _ guide 15027617.html (2016).
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Tags
Cribado Cromosómico Embriones Preimplantacionales Humanos Medio de Cultivo Gastado Recolección de Muestras Análisis de Ploidía Cromosómica Fertilización In Vitro (FIV) Pruebas Genéticas Preimplantacionales para Aneuploidía (PGT-A) Trophectoderm (TE) Masa Celular Interna (ICM) Falsos Positivos y Negativos Tecnología NICS Secuenciación de ADN Muestreo de Medio de Cultivo Amplificación del Genoma Completo (WGA) Preparación de Bibliotecas Análisis de Datos NGS Criopreservación Laboratorio de FIVErratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021.
Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
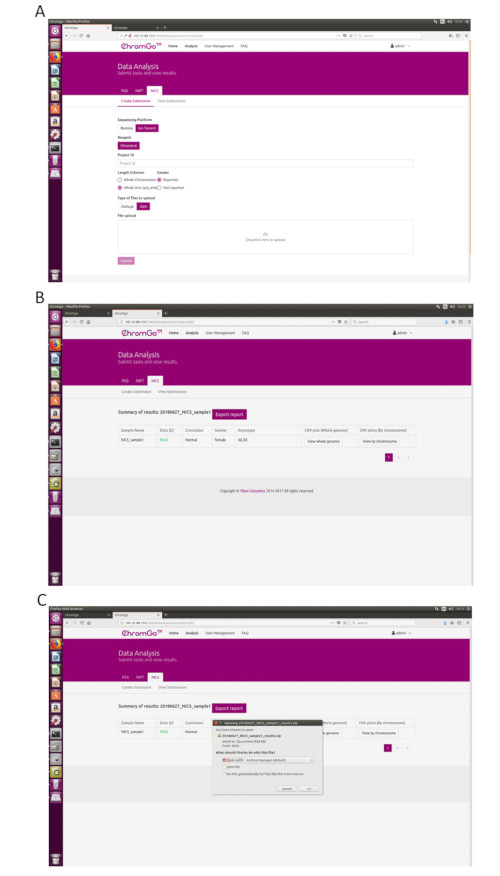
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
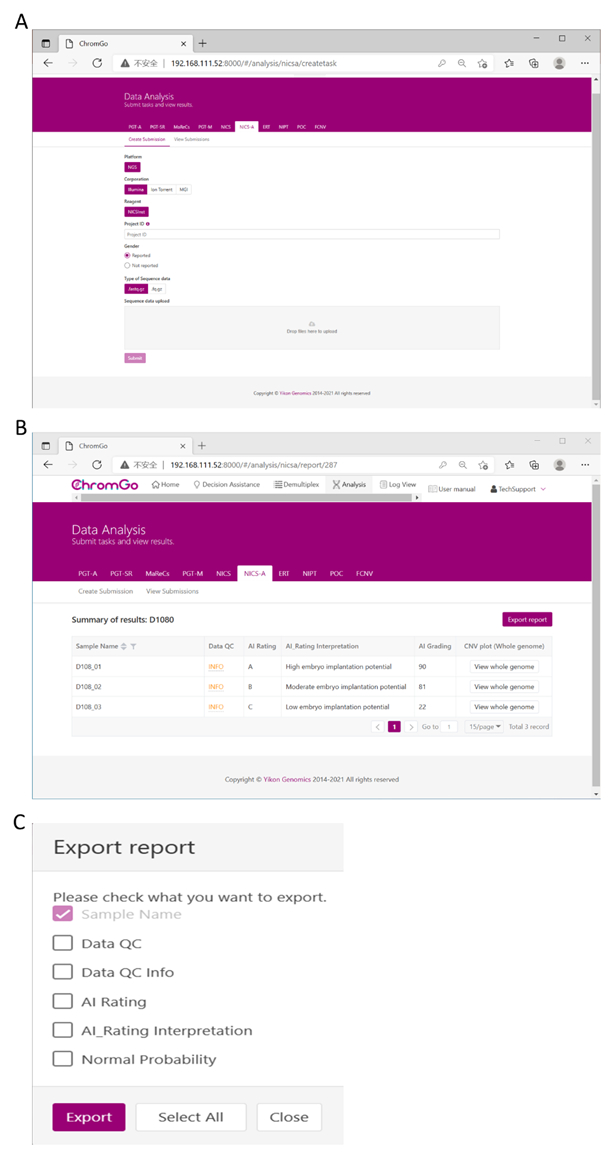
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.



