ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
La présente étude fait état d’un protocole de criblage chromosomique d’embryons humains qui utilise un milieu de culture épuisé, ce qui évite la biopsie embryonnaire et permet l’identification de la ploïdie chromosomique à l’aide du NGS. Le présent article présente la procédure détaillée, y compris la préparation du milieu de culture, l’amplification du génome entier (WGA), la préparation de la bibliothèque de séquençage de nouvelle génération (NGS) et l’analyse des données.
Abstract
Dans la fécondation clinique in vitro (FIV), la méthode prédominante pour le PGT-A nécessite la biopsie de quelques cellules du trophectoderme (TE). C’est la lignée qui forme le placenta. Cette méthode, cependant, nécessite des compétences spécialisées, est invasive et souffre de faux positifs et de faux négatifs, car le nombre de chromosomes dans le TE et la masse cellulaire interne (ICM), qui se développe dans le fœtus, ne sont pas toujours les mêmes. Le NICS, une technologie nécessitant le séquençage de l’ADN libéré dans le milieu de culture à la fois par TE et par ICM, peut offrir une solution à ces problèmes, mais il a déjà été démontré qu’il avait une efficacité limitée. La présente étude rend compte du protocole complet du NICS, qui comprend les méthodes d’échantillonnage des milieux de culture, l’amplification du génome entier (WGA) et la préparation de la bibliothèque, ainsi que l’analyse des données NGS par un logiciel d’analyse. Compte tenu des différents temps de cryoconservation dans les différents laboratoires d’embryons, les embryologistes disposent de deux méthodes de collecte du milieu de culture embryonnaire qui peuvent être sélectionnées en fonction des conditions réelles du laboratoire de FIV.
Introduction
Les techniques de procréation assistée (ART) sont de plus en plus utilisées pour le traitement de l’infertilité. Cependant, le taux de réussite des traitements antirétroviraux, tels que la FIV, a été limité et le taux de fausse couche est significativement plus élevé que celui de la population normale1. La principale cause de ces problèmes est les anomalies chromosomiques, qui existent couramment dans les embryons humains préimplantatoires2. Le PGT-A est une méthode efficace de dépistage de l’équilibre chromosomique des embryons avant l’implantation 3,4. Certaines études ont prouvé que le PGT-A peut réduire le taux d’avortement et améliorer le taux de grossesse 5,6,7,8. Cependant, le PGT-A nécessite une expertise technique complexe qui nécessite une formation et une expérience spécifiques. La procédure invasive de biopsie embryonnaire pourrait également causer des dommages aux embryons9. Des études ont montré que la biopsie du blastomère peut entraver le développement ultérieur et que le nombre de TE biopsiées peut affecter les taux d’implantation10. Bien que la question de la biosécurité à long terme de la biopsie embryonnaire n’ait pas encore été évaluée de manière approfondie chez l’homme, des études animales ont montré ses influences négatives sur le développement de l’embryon11,12,13.
Des rapports antérieurs indiquaient que des traces d’ADN étaient sécrétées dans le milieu de culture au cours du développement de l’embryon, et des efforts ont été faits pour effectuer un criblage chromosomique complet (CCS) à l’aide de milieux de culture embryonnaires épuisés 14,15,16,17,18. Cependant, les taux de détection et la précision des tests n’ont pas répondu aux exigences d’une utilisation clinique étendue. La présente étude a fait état d’une amélioration du test NICS pour augmenter les taux de détection ainsi que la précision du test NICS19. Au cours des dernières années, le liquide blastocoele (BF) a été étudié en tant qu’échantillon analytique de PGT-A mini-invasif. Cependant, la proportion d’amplification réussie à l’échelle du génome et d’ADN détectable dans les échantillons de liquide de blastocyste varie de 34,8 % à 82 %20,21,22. Le volume de BF rapporté dans diverses études varie de 0,3 nL à 1 μL. Compte tenu de la faible quantité d’ADN dans BF, il est possible d’augmenter la quantité d’ADN acellulaire en mélangeant le liquide du blastocyste et le milieu de culture pour améliorer le taux de réussite et la cohérence de la détection. Kuznyetsov et al.23 et Li et al.24 ont traité la zone pellucide avec un laser et ont libéré du liquide de blastocyste dans le milieu de culture pour améliorer la quantité totale d’ADN embryonnaire, et le taux d’amplification des échantillons combinés milieu/BF après WGA était de 100 % et 97,5 %, respectivement. Jiao et al.25 ont également obtenu un taux de réussite d’amplification de 100% en utilisant la même méthode.
La présente étude fait état d’un protocole détaillé qui comprend la préparation des échantillons de milieux usés, la préparation du NGS et l’analyse des données. En retirant soigneusement les cellules de cumulus des ovocytes, la présente étude a effectué une injection intracytoplasmique de spermatozoïdes uniques (ICSI) et une culture de blastocystes. Le support dépensé du jour 4, du jour 5 et du jour 6 a été collecté pour la préparation de la bibliothèque WGA et NGS. En utilisant la technologie NICS, la présente étude a rationalisé les étapes de préparation des bibliothèques WGA et NGS en environ 3 h et a obtenu des résultats CCS non invasifs en environ 9 h.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
L’autorisation éthique a été obtenue auprès du Comité d’éthique du Troisième Hôpital de l’Université de Pékin.
1. Préparation
REMARQUE : Le matériel et l’équipement requis sont énumérés dans le tableau des matériaux.
- Réactifs
- Préchauffer et équilibrer (équilibré) 20 à 30 μL de milieu de gamètes/milieu de fertilisation et milieu de culture de clivage/blastocyste (recouvert d’huile minérale) et de hyaluronidase (dans un tube hermétiquement fermé) à 37 °C, 5 % de CO 2 et 5 % d’O2 dans un incubateur tri-gaz pendant la nuit avant utilisation.
- Préchauffer la hyaluronidase à 37 °C sur un plan de travail dans une hotte.
- Préparez le tampon de vitrification et les réactifs de prélèvement d’échantillons conformément aux instructions du fabricant.
- Outils
- Préparer des pipettes de prélèvement et de transfert d’échantillons (diamètre intérieur de ~200 à 250 μm), des pipettes de dénudation/dénudage (diamètre intérieur de ≥150 μm, ~130-140 μm et ~120 μm) et des pipettes de lavage (diamètre intérieur de ~150 μm) en tirant des pipettes Pasteur en verre pour générer des pointes fines ouvertes polies au feu.
REMARQUE : Les pipettes utilisées pour le prélèvement/transfert d’échantillons, la dénudation et le lavage peuvent être achetées directement. Les aiguilles de maintien et les aiguilles d’injection peuvent également être achetées directement.
- Préparer des pipettes de prélèvement et de transfert d’échantillons (diamètre intérieur de ~200 à 250 μm), des pipettes de dénudation/dénudage (diamètre intérieur de ≥150 μm, ~130-140 μm et ~120 μm) et des pipettes de lavage (diamètre intérieur de ~150 μm) en tirant des pipettes Pasteur en verre pour générer des pointes fines ouvertes polies au feu.
2. Protocole 1 : Prélèvement d’échantillons
- Prétraitement du complexe ovocyte-corona-cumulus (OCCC) avant digestion avec la hyaluronidase
- Obtenir une stimulation ovarienne avec des préparations d’hormone folliculo-stimulante (FSH) et de gonadotrophine de la ménopause humaine (hMG). Lorsque le follicule principal mesure >18 mm, utilisez 10 000 UI de gonadotrophine chorionique (hCG) pour la maturation finale de l’ovocyte.
- Effectuer le prélèvement des ovocytes 36 h après la piqûre de déclenchement. Prélever et transférer les ovocytes dans des boîtes de culture tissulaire avec 2,5 mL de m-HTF préchauffé recouvert d’huile minérale.
- Transférer rapidement les OCCC dans le puits central d’une boîte de culture d’organes contenant 1 mL de milieu fertilisant à l’aide d’une pipette de transfert, puis incuber avec les ovocytes à 37 °C dans un incubateur à 5 % de CO 2 et 5 % d’O2 pendant2 à 4 h.
- Digérer les CCCO contenant de la hyaluronidase en ajoutant 1 mL de hyaluronidase préchauffée à 37 °C (80 UI/mL) dans le puits central d’une boîte de culture d’organes contenant des CCC (étape 2.1.3). Maintenir la concentration finale de hyaluronidase à 40 UI/mL et bien mélanger.
- Incuber les OCCC sur une plateforme thermique à 37 °C pendant 2 min. Observez les changements au microscope toutes les 30 s jusqu’à ce qu’il ne reste plus que 1 à 2 couches de cellules de la granulosa.
- Dénudation des cellules de la granulosa
- Transférez rapidement les OCCC digérés dans la boîte de culture pour la manipulation des ovocytes et recouvrez d’huile minérale dans chaque puits.
- Observez les cellules de la granulosa séparées au microscope. Aspirez doucement et libérez les ovocytes 5 fois pour éliminer les cellules résiduelles de la granulosa autour des ovocytes.
- Répétez l’étape précédente dans les 3 puits restants pour éliminer complètement les cellules de la granulosa.
REMARQUE : Les étapes ci-dessus (2.1-2.3) peuvent être effectuées en fonction du fonctionnement de routine de chaque laboratoire.
- Évaluation de l’ovocyte
- Évaluer l’exhaustivité de l’élimination des cellules de la granulosa à l’aide d’un microscope. Si les cellules n’ont pas pu être complètement éliminées, la rétention de 5 cellules de la granulosa ou moins est acceptable à ce stade.
REMARQUE : Si les cellules du cumulus sont toujours attachées à l’ovocyte, le reste peut être retiré plus tard le jour 3 avant que l’embryon ne soit transféré du milieu de culture au stade de clivage au milieu de culture au stade de blastocyste.
- Évaluer l’exhaustivité de l’élimination des cellules de la granulosa à l’aide d’un microscope. Si les cellules n’ont pas pu être complètement éliminées, la rétention de 5 cellules de la granulosa ou moins est acceptable à ce stade.
- Après avoir effectué l’injection intracytoplasmique de spermatozoïdes (ICSI)26, transférer les ovocytes dans des microgouttelettes de milieu de culture embryonnaire de 20 à 30 μL (un ovocyte correspond à une microgouttelette) à l’aide de pipettes de transfert et incuber dans un incubateur à 37 °C, 5 % de CO 2 et 5 % d’O2.
- Enregistrez le jour de l’ICSI comme jour 0. Vérifier les embryons et obtenir un score selon l’atelier de consensus d’Istanbul sur l’évaluation embryonnaire du jour 1 pour la fécondation (environ 18 h), du jour 2 (environ 45 h) et du jour 3 (environ 68 h) pour le clivage embryonnaire27.
- Lavage d’embryons
- Préparer 20 à 30 μL de microgouttelettes de milieu de culture de blastocystes pour chaque embryon recouvert d’huile minérale dans des boîtes de culture tissulaire au jour 2 dans un incubateur à 37 °C, 5 % de CO 2 et 5 % d’O2.
- Préparez trois autres microgouttelettes recouvertes d’huile minérale et étiquetez les nouvelles boîtes de culture tissulaire pour le lavage n° 1-3.
- Transférez les embryons du jour 3 dans les microgouttelettes de lavage. Aspirez doucement et libérez les embryons 3 fois dans chaque gouttelette à l’aide de pipettes à dénudation.
REMARQUE : Cette procédure peut également aider à éliminer les cellules granulaires résiduelles attachées à l’embryon. - Observer et évaluer les embryons au microscope le jour 3 avant que le milieu ne soit changé de milieu de culture au stade de clivage à milieu de culture de blastocyste pour le marquage morphologique. Si les cellules de cumulus étaient encore attachées à l’embryon, pipeter de haut en bas de manière appropriée dans une autre gouttelette de milieu de culture de blastocyste préchauffée et équilibrée recouverte d’huile minérale à l’aide d’une pipette à stripper jusqu’à ce que les cellules de cumulus soient complètement éliminées.
NOTA : Toutes les cellules de cumulus attachées devaient être complètement retirées le jour 3 avant que l’embryon ne soit transféré de la plaque de milieu de culture au stade de clivage à la plaque de milieu de culture au stade de blastocyste. Toutes les cellules de cumulus restantes interféreront dans l’analyse finale et donneront des résultats faussement négatifs.
- Deux options pour la collecte des milieux de culture
REMARQUE : Le centre de FIV peut choisir l’une des deux méthodes de collecte des milieux de culture en fonction des ressources, des demandes et des préférences du centre.- Option 1 : Lavage et culture d’embryons
REMARQUE : Cette option est destinée aux laboratoires de FIV qui effectuent la vitrification le matin du jour 5.- Transférer l’embryon dans des microgouttelettes préchauffées (37 °C) de milieu de culture et laver délicatement chaque embryon en série dans 3 microgouttelettes par pipetage au jour 4 après-midi.
- Transférer chaque embryon dans une microgouttelette unique préchauffée (37 °C) de milieu de culture pour le prélèvement d’échantillons. Le volume d’une seule gouttelette de milieu de culture ne peut pas dépasser 25 μL.
- Réaliser une culture d’embryons de blastocyste le jour 5/jour 6 à 37 °C, 5 % de CO 2 et 5 % d’O2.
- Option 2 : Lavage et culture d’embryons
REMARQUE : Cette option est destinée aux laboratoires de FIV qui effectuent la vitrification le jour 5 après-midi ou le jour 6.- Transférer l’embryon dans des microgouttelettes préchauffées (37 °C) de milieu de culture de 10 à 15 μL et laver délicatement chaque embryon en série dans 3 microgouttelettes par pipetage au jour 5.
- Transférer chaque embryon dans une microgouttelette unique préchauffée (37 °C) de milieu de culture pour le prélèvement d’échantillons. Le volume d’une seule gouttelette de milieu de culture ne peut pas dépasser 15 μL.
- Réaliser une culture d’embryons de blastocyste le jour 5/jour 6 à 37 °C et 5 % de CO2.
- Option 1 : Lavage et culture d’embryons
- Prélèvement d’échantillons
- Ajustez doucement l’ICM à une distance considérable du point ciblé du faisceau laser, qui se concentre sur la jonction cellulaire du trophectoderme pour générer un petit trou dans le trophectoderme afin de libérer le liquide de la cavité du blastocèle. Ensuite, les embryons sont déplacés vers une solution de congélation pour la cryoconservation selon le processus conventionnel.
- Transférer le milieu de culture de chaque embryon en culture dans un tube PCR sans RNase/DNase contenant 5 μL de tampon de lyse cellulaire.
- Recueillir la même quantité de milieu de culture sans être utilisé pour la culture d’embryons comme témoin négatif. Congeler immédiatement tous les échantillons prélevés dans de l’azote liquide, puis les conserver à −80 °C après avoir été prélevés jusqu’à ce qu’ils soient soumis au test NICS.
- Effectuez la vitrification comme décrit dans le protocole.
3. Protocole 2 : Construction d’une bibliothèque
- Lyse du milieu de culture
- Diluer 1 μL de témoin positif (10 ng d’ADNg humain) avec 199 μL de milieu de culture frais. Bien mélanger et centrifuger brièvement le tube (200 x g pendant 5 s).
- Transférer 10 μL de milieu de culture de blastocyste de 5 jours à 6 jours, de témoin positif dilué et de milieu de culture frais dans de nouveaux tubes PCR de 0,2 mL.
- Ajouter 1 μL de MT Enzyme Mix dans chaque tube PCR et bien mélanger par pipetage et centrifugation immédiatement pendant 2-3 s à 200 x g.
- Placez le(s) tube(s) PCR de l’étape 3.1.3 dans une station de préparation d’échantillons NICS préchauffée et exécutez le programme de lyse comme suit : 10 min à 75 °C ; 4 min à 95 °C ; à 22 °C.
REMARQUE : La station de préparation des échantillons est comparable à une machine PCR standard.- Cliquez sur l’icône Lysis pour accéder à l’écran de configuration.
- Sélectionnez Tube pour le mode de contrôle ; entrée 10 μL pour le volume de l’échantillon ; sélectionnez Activé pour le contrôle du couvercle chauffant et entrez 105 °C pour la température. Sélectionnez Non pour Pause lors de la première séquence. Cliquez sur OK pour continuer.
- Patientez jusqu’à ce que l’heure de repos affiche -- :-- :--, qui indique la fin du programme, puis cliquez sur Arrêter pour arrêter le programme.
- Arrêtez le programme une fois le processus terminé. Passez immédiatement à l’étape suivante.
- Préparation préalable à la bibliothèque
- Décongeler le tampon pré-lib jusqu’à ce qu’il soit RT. Bien mélanger par pipetage et centrifuger immédiatement pendant 2 à 3 s à 200 x g.
- Préparez un mélange maître pour la réaction de pré-bibliothèque comme suit : ajoutez 2 μL de mélange enzymatique Pre-Lib à 60 μL de tampon Pre-Lib, mélangez bien la réaction et centrifugez brièvement.
- Ajouter 60 μL de mélange réactionnel de pré-bibliothèque dans chaque échantillon de milieu prétraité de l’étape précédente. Bien mélanger par pipetage et centrifuger immédiatement pendant 2-3 s à 200 x g.
- Placez le(s) tube(s) PCR de l’étape 3.2.3 dans la station de préparation des échantillons et exécutez le programme de prébibliothèque comme suit : 95 °C pendant 2 min ; 12 cycles de 15 °C pendant 40 s, 22 °C pendant 40 s, 33 °C pendant 30 s, 65 °C pendant 30 s, 72 °C pendant 40 s, 95 °C pendant 10 s et 63 °C pendant 10 s ; et maintenir à 4 °C.
- Cliquez sur l’icône Pre_Lib pour accéder à l’écran de configuration.
- Sélectionnez Tube pour le mode de contrôle ; entrée 70 μL pour le volume de l’échantillon ; sélectionnez Activé pour le contrôle du couvercle chaud et entrez 105 °C pour la température. Sélectionnez Non pour Pause lors de la première séquence. Cliquez sur OK pour continuer.
- Patientez jusqu’à ce que l’heure de repos affiche -- :-- :--, qui indique la fin du programme, puis cliquez sur Arrêter pour mettre fin au programme.
- Arrêtez le programme lorsque le processus est terminé. Passez immédiatement à l’étape suivante.
- Préparation de la bibliothèque
- Décongeler le tampon de la bibliothèque jusqu’à ce qu’il soit RT. Bien mélanger par pipetage et centrifuger immédiatement pendant 2 à 3 s à 200 x g.
- Préparez un mélange maître pour la réaction de la bibliothèque comme suit : ajoutez 1,6 μL de mélange enzymatique de bibliothèque à 60 μL de tampon de bibliothèque, mélangez bien la réaction et centrifugez brièvement.
- Ajouter 60 μL de mélange réactionnel de bibliothèque et 2 μL d’apprêt pour codes-barres à chaque produit de prébibliothèque à partir de l’étape 3.2.3. Mélangez bien la réaction et centrifugez brièvement.
- Placez le(s) tube(s) PCR de l’étape 3.2.3 dans le thermocycleur et exécutez le programme de préparation de la bibliothèque comme suit : 94 °C pendant 30 s ; 17 cycles de 94 °C pendant 25 s, 62 °C pendant 30 s et 72 °C pendant 45 s) ; puis maintenir à 4 °C.
- Cliquez sur l’icône Lib_Prep pour accéder à l’écran de configuration.
- Sélectionnez Tube pour le mode de contrôle ; entrée 130 μL pour le volume de l’échantillon ; sélectionnez Activé pour le contrôle du couvercle et entrez 105 °C pour la température correspondante. Sélectionnez Non pour Pause lors de la première séquence. Cliquez sur OK pour continuer.
- Patientez jusqu’à ce que l’heure de repos affiche -- :-- :--, qui indique la fin du programme, puis cliquez sur Arrêter pour mettre fin au programme.
- Purification de la bibliothèque
- Retirer Magbeads du stockage à une température de 2 à 8 °C pendant au moins 20 minutes avant l’étape de purification. Vortex et mélanger les Magbeads pendant 20 s. Distribuez suffisamment de billes pour l’étape de purification dans un nouveau tube de microcentrifugation de 1,5 ml et réchauffez les billes jusqu’à ce qu’elles soient RT.
- Ajoutez 1x Magbeads dans chaque bibliothèque. Mélanger en pipetant de haut en bas ≥10 fois et incuber à RT pendant 5 min.
REMARQUE : Par exemple, ajoutez 100 μL de Magbeads à 100 μL d’échantillon de bibliothèque. - Après l’incubation, centrifugez brièvement le tube et placez-le sur un support magnétique.
- Attendez environ 5 minutes jusqu’à ce que la solution devienne claire. Tout en gardant le tube sur le support magnétique, aspirez soigneusement la solution et jetez-la.
- Ajoutez 200 μL d’éthanol à 80 % fraîchement préparé dans le tube. Incuber à RT pendant 30 s et retirer délicatement le surnageant. Répétez l’opération une fois de plus.
- Retirez l’éthanol aussi complètement que possible. Séchez les perles à l’air libre sur le support magnétique pendant environ 5 à 10 minutes à RT.
- Retirez le tube du support magnétique, ajoutez 17,5 μL de tampon d’élution et faites vortex le tube pour remettre les billes en suspension. Centrifuger brièvement le tube et incuber à RT pendant 5 min.
- Placez le tube sur le support magnétique et attendez que la solution devienne claire. Transférez délicatement 15 μL de surnageant dans un nouveau tube.
- Quantification de la bibliothèque
- Quantifier les banques purifiées à l’aide du fluorimètre conformément au guide d’utilisation des kits de test qubit dsDNA HS28. Le rendement des bibliothèques varie de ~15 à 300 ng.
- Mise en commun des bibliothèques
- Utilisez 10 nanogrammes de chaque échantillon de bibliothèque pour la mise en commun.
- Séquençage
- Reportez-vous au guide d’utilisation du séquençage (15027617 v01)29.
- Des séquences de bibliothèque purifiées de 50 pb à une seule extrémité de la plate-forme ont donné environ 2 millions de lectures pour chaque échantillon, et une profondeur de séquençage de 0,03 × a été recommandée.
- Analyse des données
- Entrez le nom et le mot de passe de l’utilisateur sur la page de connexion
- Après vous être connecté au système, cliquez sur Analyse et une nouvelle page apparaîtra. Cliquez sur Créer une soumission sous l’onglet NICS-A. Ensuite, choisissez NGS pour la plate-forme, sélectionnez corporation, choisissez NICSInst pour le réactif, entrez les informations du projet dans la zone sous ID de projet, définissez les préférences d’analyse et téléchargez les fichiers. Une fois que tous les fichiers de séquençage ont été téléchargés, cliquez sur Envoyer pour lancer l’analyse (Figure 3A).
- Cliquez sur Afficher les soumissions pour afficher la liste des projets soumis. Une fois l’analyse terminée, le statut d’un projet devient Terminé et un bouton Afficher apparaît dans le champ du rapport. Cliquez sur le bouton Afficher pour afficher le tableau récapitulatif de l’analyse NICS (Figure 3B).
- Cliquez sur le bouton Export report (Exporter le rapport ) pour enregistrer les rapports (Figure 3C).
REMARQUE : Trois types de fichiers seront exportés pour chaque analyse. Un fichier graphique qui comprend tous les tracés de variation du nombre de copies (CNV) pour chaque chromosome et génome entier, qui sera stocké dans le dossier « graph » ; une feuille de calcul qui contient les détails de l’exemple de contrôle qualité de cette exécution d’analyse ; un fichier document qui contient les rapports NICS personnalisés par l’utilisateur ; et une feuille de calcul qui contient l’exemple d’informations récapitulatives de cette exécution d’analyse.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
La présente étude a appliqué la méthode proposée à un patient. L’approbation de la CISR et le consentement éclairé ont été obtenus avant l’application de l’analyse NICS. La présente étude a permis d’obtenir 6 blastocystes de patients et d’effectuer un NICS sur les 6 embryons du milieu du jour 4 au jour 5. Des anomalies chromosomiques causées par la translocation équilibrée des parents ont été détectées dans cinq des chromosomes avec le test NICS ; par conséquent, ils ne pouvaient pas être utilisés pour le transfert (figure 4A-E). Les résultats NICS des deux embryons ont montré le même caryotype 45, et XN et -18 (×1) étaient tous deux des délétions du chromosome 18 (Figure 4A, B). Le caryotype 46, XN, -1p (pter→p21.1, ×1) n’est que le bras court de la délétion de la région pter→p21.1 du chromosome 1 (Figure 4D).
Les résultats du NICS ont montré que le caryotype 46, XN, +1p (pter→p21.2, ×3) et -18 (q21.32→qter, ×1), ce qui indiquait que le bras long de la région q21.32→qter du chromosome 18 et le bras court de la région pter→p21.2 du chromosome 1 étaient dupliqués (Figure 4E). Bien que les caryotypes 46, XN, +5q (×4) et -8 (×1, mos) soient des duplications du chromosome 5 et présentent 8 différences de mosaïque, le test NICS peut dépister l’aneuploïdie des 24 chromosomes. Ce procédé fournit une nouvelle méthode de transfert de blastocystes de caryotype normal unique.

Graphique 1. L’exhaustivité de l’élimination des cellules de cumulus. (A) Les ovocytes avec des cellules de cumulus. (B) Les ovocytes sans cumulus attachés. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 2. Les cellules cumulus sont prélevées d’un embryon à J3 avant d’être transférées dans la BM. Toutes les cellules de cumulus attachées doivent être retirées avant que le milieu ne passe de la plaque de milieu de culture embryonnaire de clivage initial à la plaque de milieu de culture de blastocyste, qui se trouve au jour 3 après que les embryons ont atteint le stade de 8 cellules. Toutes les cellules de cumulus qui ne sont pas éliminées interféreront dans l’analyse finale et donneront des résultats faussement négatifs. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 3. Analyse des données. (A) Il existe différentes options pour l’application utilisateur. Pour la société de plate-forme de séquençage, les utilisateurs peuvent choisir Illumina, Ion Torrent ou MGI. Les utilisateurs peuvent choisir si les informations sur le sexe sont rapportées. Une fois le réglage des paramètres ci-dessus terminé, cliquez sur la case sous Téléchargement de fichier et choisissez les fichiers de séquençage appropriés à télécharger. Pour Illumina, choisissez les fichiers avec l’extension fastq.gz. Cliquez sur Envoyer pour démarrer l’analyse une fois le téléchargement réussi. (B) La vue du tableau récapitulatif. Le tableau récapitulatif contient les informations suivantes : Nom de l’échantillon : le nom de chaque échantillon NICS est répertorié ; Contrôle qualité des données : indique si le fichier de séquençage réussit le contrôle qualité pour l’analyse NICS ; Évaluation de l’IA : la cote (A, B ou C) pour chaque échantillon NICS ; AI_Rating Interprétation : Évaluation du potentiel d’implantation embryonnaire ; Notation de l’IA : le score de chaque échantillon NICS ; Graphique CNV (génome entier) : Visualisez les profils CNV de tous les chromosomes ; (C) La page Enregistrer le rapport. Cliquez sur le bouton Exporter le rapport à côté du résumé des résultats. Sélectionnez les informations que vous souhaitez afficher dans le rapport final et cliquez sur Exporter. Les rapports seront enregistrés dans le dossier de téléchargement de votre ordinateur. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 4. Dépistage et sélection d’embryons à l’aide du NICS chez un patient. Au total, six embryons se sont développés avec succès jusqu’au stade de blastocyste, et le milieu de culture Day4-Day5 de chaque embryon a été prélevé pour le test NICS. (A) et (B) sont les résultats NICS des deux embryons de blastocyste ont montré le même caryotype 45, XN, -18 (×1) sont tous deux une délétion du chromosome 18. (C) a montré que le caryotype 46, XN, +5q (×4), -8(×1, mos) est la duplication du chromosome 5 et la mosaïque 8. (D) a montré que le caryotype 46, XN, -1p (pter→p21.1, ×1) n’est que le bras court de la délétion de la région pter→p21.1 du chromosome 1, tandis que (E) a montré que le caryotype 46, XN, +1p (pter→p21.2, ×3), -18(q21.32→qter, ×1) est le bras court de la duplication de la région pter→p21.2 du chromosome 1 et le bras long du chromosome 18 q21.32 → la région qter (F) ont montré une composition chromosomique équilibrée. L’axe des x signifie 22 autosomes en rouge et bleu, l’axe des y indique le nombre de copies de chaque autosome. Les points gris sont l’échelle de la règle de réponse du nombre de copies dans chaque fenêtre de bac et le caryotype normal du nombre de copies doit être 2. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau S1. Les taux de réussite de la détection de l’ADN Option 1 et Option 2. Veuillez cliquer ici pour télécharger ce tableau.
Tableau S2. La concordance entre NICS et PGT-A dans différentes options. Veuillez cliquer ici pour télécharger ce tableau.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Modifications et dépannage
Si les résultats du NICS sont contaminés par du matériel génétique parental, assurez-vous que toutes les cellules cumulus-corona radiata sont retirées et assurez-vous que l’ICSI est effectuée pour la fécondation. Les processus inadéquats de stockage du milieu ou de préparation des modèles sont évités, ce qui peut dégrader l’ADN. L’espace de travail a été purifié en profondeur avec des réactifs de décontamination DNase et RNase. Pour éviter la contamination par d’autres embryons, un embryon a toujours été cultivé dans une seule gouttelette de milieu afin d’éviter la contamination croisée à partir du jour 4. Le phénomène de contamination est minimisé lorsque l’on retarde la mise en place des embryons dans la couche finale de culture30,31,32,33. Pour minimiser la contamination maternelle, Kuznyetsov34 a modifié les procédures de culture d’embryons du jour 0 au jour 4, y compris l’élimination soigneuse des cellules corona résiduelles par pipetage et rinçage.
Lane et al.30 montrent que lors de la prise du milieu de culture embryonnaire du jour 4 au jour 5, la précision de la détection de l’euploïdie est améliorée, la consistance de la ploïdie embryonnaire est supérieure à 95% et la consistance du chromosome sexuel atteint 100%. Lledo et al.33 ont constaté que le taux de coïncidence entre le milieu de culture du jour 3 au jour 5 et les échantillons TE était de 74,6 % et de 92,0 % lorsque les embryons étaient cultivés du jour 4 au jour 6.
Nos données internes appuient également cette conclusion, comme le montre le tableau S1. Par rapport à la méthode de culture conventionnelle du jour 3 au jour 5, les cellules de la granulosa ont été retirées en raison d’un changement supplémentaire de milieu de culture le jour 4 ou le jour 5. Nous fournissons des données internes (tableau S1) montrant que nos deux méthodes (option 1 et option 2) ont une bonne cohérence par rapport au PGT-A, ce qui est meilleur que la méthode d’échantillonnage sans élimination complète du CC.
Des produits amplifiés par l’IF sont apparus dans le témoin négatif, et des matériaux d’ADN externes peuvent avoir contaminé le réactif ou l’espace de travail. L’espace de travail doit être nettoyé par des réactifs d’élimination de l’ADN/ARN, des matériaux exempts de nucléase doivent être utilisés et les réactifs doivent être alicités après la première utilisation.
Les différences dans les taux de réussite entre l’option 1 et l’option 2 sont décrites dans les tableaux S1 et S2.
Limites du test NICS
Il y a deux limitations principales de la carte réseau. 1) Avant l’ICSI, toutes les cellules du cumulus (généralement d’origine maternelle, généralement de composition chromosomique normale) doivent être retirées. Si l’élimination est incomplète, les cellules du cumulus peuvent libérer de l’ADN pendant le développement de l’embryon et l’ADN externe est amplifié, ce qui peut être la cause d’une détection de faux négatifs. 2) Il est difficile d’éliminer les spermatozoïdes attachés à la zone pellucide, et il est fortement recommandé d’effectuer la procédure NICS avec ICSI. Bien que le remplacement régulier du milieu de clivage au jour 3 puisse réduire la possibilité de contamination due aux cellules cumulus et aux spermatozoïdes redondants, cette contamination doit être minimisée si le NICS est utilisé dans la FIV clinique. Cependant, une méthode de détection des NICS dans les embryons de FIV a été développée, y compris la fonction de reconnaissance de l’ADN exogène, qui sera démontrée dans un proche avenir.
Cette étude n’a pas comparé les différences entre les différents milieux puisque des essais cliniques à grande échelle ont comparé les milieux de culture. Huit centres ont utilisé 4 milieux de culture différents, séquentiels et continus, et 2 pourcentages différents de supplémentation en albumine (5 % et 10 %), et ces différences n’ont pas eu d’effets significatifs sur la précision des résultats de l’ADNcf embryonnaire31. Ces résultats soutiennent l’applicabilité potentielle de l’analyse de l’ADNcf embryonnaire à tous les laboratoires de FIV lorsqu’ils travaillent dans le cadre du protocole spécifique.
Importance par rapport aux méthodes existantes
La méthode NICS permet d’éviter la biopsie embryonnaire et améliore ainsi considérablement la sécurité d’utilisation. Par rapport aux blastocystes, le NICS est une technique de dépistage préimplantatoire simple, rapide, sensible et reproductible, qui convient aux populations de procréation assistée présentant une forte probabilité d’aneuploïdie. Contrairement à la biopsie invasive, qui nécessite des connaissances considérables et professionnelles pour la procédure de biopsie du blastocyste, le NICS peut être largement appliqué car sa simple collecte de milieu épuisé ne suit que le fonctionnement régulier de la FIV19 et ne nécessite pas de qualification PGS/DPI dans certains pays.
Applications futures
Le NICS a le potentiel d’une large applicabilité pour le criblage chromosomique dans la FIV clinique, non seulement pour l’ICSI mais aussi pour les embryons de FIV. Bien que l’ICSI soit fortement recommandée, des méthodes d’élimination des spermatozoïdes attachés à la zone pellucide sont nécessaires pour prévenir l’influence des spermatozoïdes.
L’évaluation morphologique est une méthode traditionnelle d’évaluation embryonnaire, mais dans la plupart des cas, les embryons chromosomiquement anormaux peuvent sembler morphologiquement similaires aux embryons chromosomiquement normaux (euploïdes). La combinaison de l’évaluation morphologique avec le test NICS lors du transfert d’embryons ploïdes avec une bonne morphologie dans l’utérus pourrait améliorer les taux de grossesse en cours et le taux de naissances vivantes. Un essai clinique randomisé sera mené pour évaluer l’efficacité clinique du transfert d’embryon unique à l’aide de la technologie NICS.
Étapes critiques du protocole
Toutes les cellules de cumulus-corona radiata doivent être retirées des ovocytes avant la fécondation. Les ovocytes ont été fécondés par injection intracytoplasmique de spermatozoïdes (ICSI). L’ajout de protéines/suppléments d’origine humaine au milieu de culture a été évité. Le milieu de culture a été changé le jour 4 et prélevé le jour 5 au jour 6, lorsque les blastocystes se sont complètement développés. Les embryons ont été cultivés dans des gouttelettes individuelles de milieu de culture à partir du jour 4. Lors de la collecte du milieu de culture, les pipettes de transfert entre les échantillons ont été changées pour éviter toute contamination.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yaxin Yao, Jieliang Ma, Jing Wang et Sijia Lu sont des employés de Yikon Genomics Co., Ltd.
Acknowledgments
Les auteurs tiennent à remercier Shiping Bo et Shujie Ma pour leur aide dans l’analyse des données NGS. Financement : ces travaux ont été financés par le Programme national de recherche et de développement clé (subvention n° 2018YFC1003100).
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
References
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Jr Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T. Jr, Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid? Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- Thermo Fisher Scientific. Qubit dsDNA HS Assay Kit. , Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015).
- Miseq system use guide. , Available from: https://support.illumina.com/downloads/miseq_system user _ guide 15027617.html (2016).
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Tags
Dépistage chromosomique embryons préimplantatoires humains milieu de culture épuisé collecte d’échantillons analyse de la ploïdie chromosomique fécondation in vitro (FIV) tests génétiques préimplantatoires pour l’aneuploïdie (PGT-A) trophectoderme (TE) masse cellulaire interne (ICM) faux positifs et négatifs technologie NICS séquençage de l’ADN échantillonnage du milieu de culture amplification du génome entier (WGA) préparation de la bibliothèque analyse des données NGS cryoconservation laboratoire de FIVErratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021.
Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
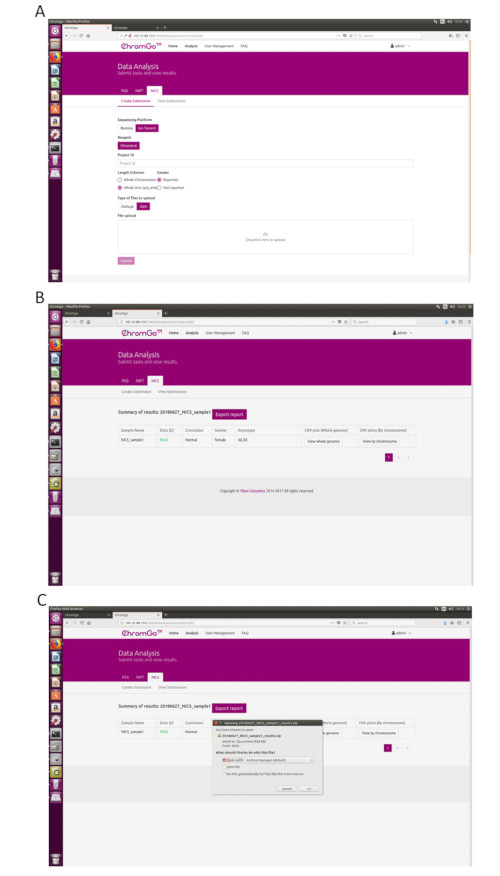
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
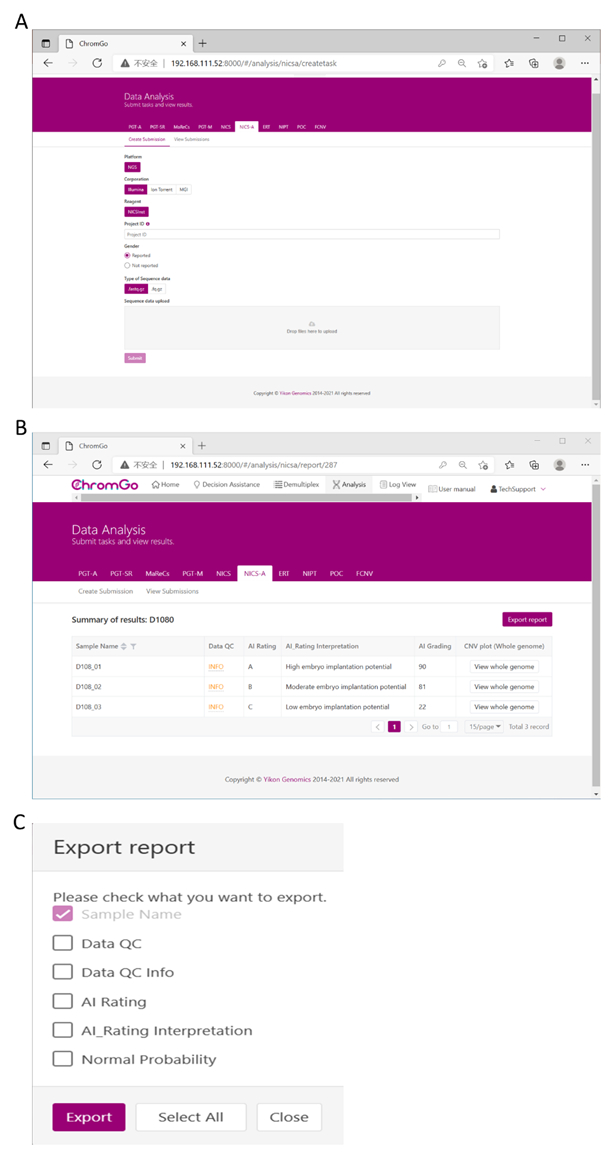
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.



