Dilutions en série et ensemencement des géloses : numération des micro-organismes
English
Share
Overview
Source: Jonathan F. Blaize1, Elizabeth Suter1, et Christopher P. Corbo1
1 Département des sciences biologiques, Wagner College, 1 Campus Road, Staten Island NY, 10301
L’évaluation quantitative des procaryotes peut être onéreuse compte tenu de leur abondance, de leur propension à une prolifération exponentielle, de la diversité des espèces au sein d’une population et de leurs besoins physiologiques spécifiques. À ce défi s’ajoute la nature en quatre phases dans laquelle les bactéries se reproduisent (lag, journal, stationnaire et mort). La capacité d’estimer avec précision la concentration de micro-organismes est nécessaire pour réussir l’identification, l’isolement, la culture et la caractérisation (6). À ce titre, les microbiologistes ont utilisé la dilution en série et diverses techniques de placage depuis plus d’un siècle pour quantifier de façon fiable la charge bactérienne et virale dans les environnements cliniques, industriels, pharmaceutiques et universitaires de laboratoire (2,4,6). Les descriptions de cette méthodologie sont apparues pour la première fois en 1883 lorsque le scientifique et médecin allemand Robert Koch a publié ses travaux sur les agents pathogènes (2). Souvent appelées le père de la bactériologie moderne, les techniques prénommées de Koch sont devenues l’étalon-or de l’énumération des micro-organismes, culturables ou autres, dans le monde entier.
La dilution en série est une réduction systématique d’une entité connue ou inconnue (un soluté, un organisme, etc.) par une re-suspension successive d’une solution initiale (solution0) en volumes fixes d’un diluant liquide (blancs). Ces blancs se composent généralement de 0,45% salin, bien que la composition peut être variée (7). Bien qu’un expérimentateur puisse choisir n’importe quel volume pour chaque diluant, il s’agit le plus souvent d’un multiple de 10, ce qui facilite la réduction logarithmique de l’échantillon. Par exemple, la solution0 contient un total de 100 cellules E. coli suspendues dans 10 ml de bouillon nutritif. Si 1 ml de solution0 est enlevé et ajouté à 9 ml de solution saline (diluant1),la nouvelle solution (solution1) contiendrait 1/10e de la concentration initiale d’E. coli. Dans cet exemple, la nouvelle solution (solution1) contiendrait 10 cellules E. coli. Répéter ce processus en supprimant 1 ml de la solution1 et en l’ajoutant à 9 ml de salin (diluant2) donnerait la solution2, contenant seulement une seule cellule E. coli. Étant donné que chaque nouvelle solution (9 mL de diluant et 1 ml de solution) contient un total de 10 mL, nous pouvons conclure que le facteur de dilution pour cette réduction est de 10 ou qu’il s’agissait d’une dilution en série 10 fois(figure 1). Puisque nous avons seulement commencé avec 100 cellules dans cet exemple et nous diluons par un facteur de 10, seulement deux étapes sont nécessaires pour atteindre la concentration minimale absolue de 1 cellule.
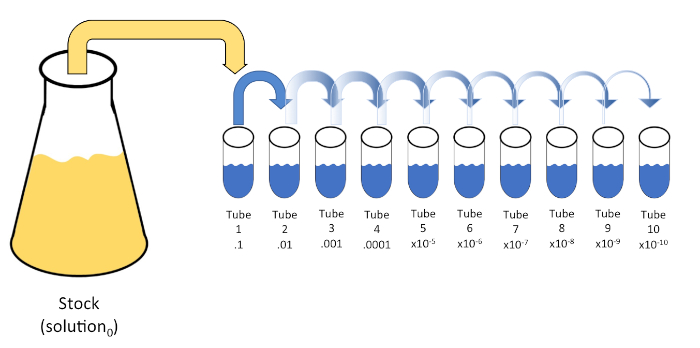
Figure 1 : Dilution en série d’une solution de stock. Un aliquot de 1 ml de la solution de stock (solution0) est ajouté au tube 1 qui contient 9 ml de 0,45% salin (dilent1); le produit de ce mélange est la solution1. Répétez en ajoutant 1 mL de la solution1 nouvellement créée et en l’ajoutant au tube 2. Aliquoting et la résuspension se poursuit de cette façon jusqu’à ce que le tube final est atteint, diluant la concentration du stock par un facteur de 10 chacun à chaque étape. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
La dilution en série est la technique la plus simple pour obtenir des concentrations gérables d’un organisme désiré et elle est complétée par des stries et des diffusions de boîtes de Pétri, seulement deux des nombreuses techniques de placage utilisées par les microbiologistes. Cet avantage de cette approche est que l’expérimentateur peut récolter des souches pures d’une seule espèce ou des souches séparées d’une population mixte (7). Le strage est accompli en introduisant un organisme à un milieu solide (généralement composé d’agarose) qu’il se développera sur si les éléments nutritifs appropriés sont disponibles. Balayer doucement une boucle d’inoculation stérile à travers le milieu (de sorte qu’une strie subtile reste) dans un modèle sinusoïdal rigide distribuera l’organisme proportionnellement à la fréquence de la forme d’onde de l’expérimentateur. Diviser le plat Petri en tiers ou en quarts (quadrant strie) et diminuer la fréquence de chaque strie à mesure qu’une nouvelle région du plat est entrée réduira graduellement le nombre de micro-organismes qui peuvent occuper cette région, produisant des colonies uniques au lieu d’un pelouse bactérienne non quantifiable. Le placage de propagation ne dilue pas en outre les échantillons; un épandeur de verre stérile est utilisé pour distribuer un aliquot de support de suspension sur un plat de pétri entier (figure 2). Les colonies qui poussent sur la plaque de propagation proviennent d’une seule cellule et chaque colonie sur le plat peut être comptée pour estimer le nombre d’unités de formation de colonies par millilitre (CFU) dans une suspension donnée, représentée comme CFU/mL (6) (Figure 3) Agar molle et réplique le placage sont des variations des techniques susmentionnées et permettent l’isolement du bactériophage et du criblage mutant, respectivement (1,7).
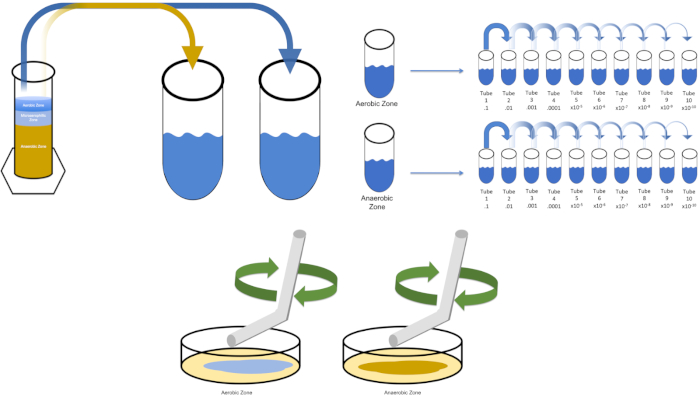
Figure 2 : Stries de plaques pour l’énumération bactérienne et l’isolement des souches. Étiquetez le fond d’un plat de pétri avec des informations d’identification (nom de la bactérie, date, média) et divisez-les en tiers. Après avoir sélectionné une dilution appropriée de l’échantillon de stock, prendre une boucle d’inoculation stérile (jetable ou enflammée) et la submerger le tube à essai (ici, T3). Soulevez légèrement le couvercle de la boîte de Pétri d’un côté de sorte que seule la boucle inoculation puisse accéder à l’agar. Glissez la boucle inoculée sur le dessus des médias d’une manière zig-zag en prenant soin de ne pas compromettre l’agar. Faites pivoter la plaque d’environ 1/3rd (118 degrés) et réduisez la fréquence des mouvements en zigzag. Tournez une dernière fois et réduisez une fois de plus la fréquence des zigzags. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
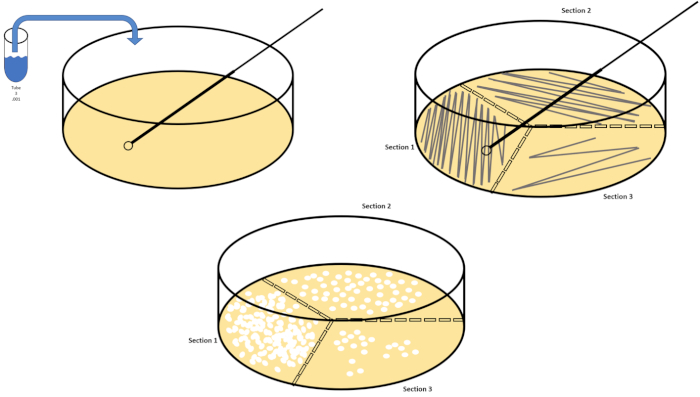
Figure 3 : Placage de propagation. 1 g de la zone aérobie a été resuspendu en T1, puis dilué en série. Un verre stérile ou une tige d’épandage jetable en plastique est utilisé pour distribuer de l’inoculum dans chaque plat. Ceci a été répété avec 1 g de la zone anaérobie. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Comme pour les dilutions en série, une échelle logarithmique est utilisée pour exprimer la concentration de l’organisation. Le nombre de colonies cultivées dans des boîtes de Pétri standard mesurant 100 mm x15 mm peut être énuméré manuellement (ou automatisé à l’aide du traitement computationnel) en identifiant des grappes isolées de croissance. Les comptes qui totalisent moins de 30 ou plus de 300 devraient être définis comme trop peu nombreux à compter (TFTC) ou trop nombreux pour être comptés (TNTC), respectivement. Dans le cas de ce dernier, une dilution en série doit être effectuée pour réduire la concentration avant de restreaking un nouveau plat de pétri. La moyenne du nombre de colonies autonomes identifiées à partir de trois boîtes de Pétri distinctes et la multiplication de la moyenne par le facteur de dilution donneront cFU/mL; tracer le journal10 de CFU/mL contre le temps révélera le temps de génération moyen de l’organisme (7).
Procedure
Applications and Summary
Bacterial enumeration and strain isolation by plating requires manageable concentrations of target organisms. Successful plating is therefore contingent upon serial dilution. As such, the aforementioned techniques remain the cornerstone of microbiological examination and experimentation. Though simple by design, dilution factors and plating technique can be modified to by the experimenter to bolster outcomes without compromising the integrity of each method. Plotting the four phases of bacterial growth can be helpful when characterizing desired microbes. These phases, lag, log, stationary, and death, are marked by changes in bacterial replication. The lag phase features slow growth due to physiological adaptation, the log phase is the period of maximum proliferation featuring an exponential rise in viable cells, stationary phase is then reached due to environmental limitations and accumulations of toxins, before the death phase where cell counts begin to fall. This can be accomplished by serially diluting (or 1-step diluting to avoid confusion) Solution0 every hour for a total of 8 hours, beginning at Time0 (Solution0 should be returned to a shaking incubator after each dilution). Calculate the log10 of CFU/ml for a single diluent of Time0 and plot on the Y-axis. Repeat this calculation for the sample Time1 (make sure calculate CFU/mL using the same dilution factor as Time0). Repeat until each time (Time1-Time8) are plotted on the X-axis.
References
- Allen, M.E., Gyure, R.A. (2013) An Undergraduate Laboratory Activity Demonstrating Bacteriophage Specificity. Journal of Microbial Biological Education 14: 84-92.
- Ben-David, A., Davidson, C.E. (2014) Estimation Method for Serial Dilution Experiments. Journal of Microbiological Methods 107:214-221.
- Goldman, E., Green, L.H. (2008) Practical Handbook of Microbiology.
- Koch, R. (1883) New Research Methods for Detection of Microcosms in Soil, Air and Water.
- Lederberg, J., Lederberg, E.M. (1952) Replica Plating and Indirect Selection of Bacterial Mutants. Journal of Bacteriology 63:399-406
- Pepper, I., Gerba, C., Ikner, L. (2019) Bacterial Growth Curve Analysis and its Environmental Changes. JoVE Science Education Database. Environmental Microbiology.
- Sanders., E.R. (2012) Aseptic Laboratory Technique: Plating Methods. JoVE 63:e3063.
Transcript
Sometimes, in order to identify and study bacteria we first need to isolate and enrich them from a sample. For example, samples obtained from a Winogradsky Column are mixed, meaning they contain multiple species or strains of bacteria, so studying an individual bacterium or enumerating the different kinds present can be challenging. To this end, serial dilution and plating techniques are typically employed to reliably quantify bacterial load and isolate individual colonies.
Serial dilution is a process through which the concentration of an organism, bacteria in this example, is systematically reduced through successive resuspension in fixed volumes of liquid diluent. Usually the volume of the diluent is a multiple of 10 to facilitate logarithmic reduction of the sample organism. For example, one gram of sediment is first removed from the Winogradsky zone of interest and added to 10 milliliters of an appropriate liquid medium. Then, one milliliter of this first dilution is added to another tube containing nine milliliters of medium. The process can be repeated until several different concentrations of bacteria have been prepared. Serial dilution is the key to enumeration of bacteria in this example, since mixed samples from a Winogradsky Column contain an unknown, often large, number of bacteria.
Next, streak plating and spread plating enable the isolation and enumeration of bacteria within a sample, respectively. Streaking is accomplished by introducing a diluted sample to one section of the solid medium supplemented with nutrient, which is divided into thirds. This inoculum is then spread over each third of the plate in a zig-zag pattern. As different sections of the plate are streaked, crossing from the previous sample only once, the sample is spread more thinly. This means that you may only need to streak from one dilution to achieve individual colonies in the later sections. After incubation, the streaked plates allow for observations of colony morphology, information that can help differentiate between different bacterial species.
Alternatively, if the main goal is the enumeration of the bacteria in the sample spread plating may be used. In spread plating, an aliquot of a single sample is spread evenly over the entire surface of solid medium. Typically, because we don’t know the bacterial numbers in the mixed sample, a spread plate is made for each of the dilutions or a representative sample of them. After incubation, enumeration can be performed using these spread plates. Any plates with colony counts fewer than 30 should be discarded, since small counts are subject to greater error. Similarly, any counts over 300 should be discarded because colony crowding and overlapping can lead to underestimation of colony count. If the colony counts of each of these remaining dishes is recorded and multiplied by the dilution factor, and then divided by the volume plated, this yields the colony forming units, or CFUs, per milliliter of suspension. In this video, you will learn how to qualitatively and quantitatively evaluate a sample containing a known bacterium, and the microbial communities contained in various regions of a Winogradsky Column via serial dilution, spread plating, and streak plating.
First, put on any appropriate personal protective equipment including a lab coat, gloves, and goggles. Next, sterilize the workspace with 70% ethanol and wipe down the surface. Next, gather two 500 milliliter Erlenmeyer flasks and label one broth and the other agar. To prepare LB agar solution, mix approximately 6.25 grams of LB agar, three grams of technical agar, and 250 milliliters of distilled water in the flask labeled agar.
Then, prepare LB broth by combining 2. 5 grams of LB media and 100 milliliters of distilled water in the flask labeled broth. After autoclaving the flasks, use a heat resistant glove to remove the flasks from the autoclave and place them in a 40 to 50 degree Celsius water bath. Once the flasks are 50 degrees Celsius, carefully prepare three 100 milliliter aliquots of the broth solution and label each aliquot solution zero. Next, gather 10 sterile petri dishes and label them with the date, name, type of media used, and the Winogradsky Column zone that the organisms will be harvested from. Pipette 15 milliliters of agar from the agar flask into each petri dish. Then, use the pipette tip to remove any bubbles, replace the plate lids, and allow them to solidify on the bench top overnight.
The next day, wipe down the bench top with 70% ethanol. Next, label 10 20 milliliter test tubes T1 through T10 and place them in a rack. Pipette nine milliliters of .45% saline into each tube. Now, cover each of the 10 test tubes loosely with their caps and transfer them to an autoclave-compatible test tube rack. After the cycle is complete, remove the saline blanks using heat resistant gloves and allow them to cool. Store the tubes at room temperature until they have reached approximately 22 degrees Celsius.
To cultivate a known target organism, E. coli in this example, inoculate 100 milliliters of solution zero with a single colony from a previously streaked plate. Then, cover the tube and incubate it over night at 37 degrees Celsius. To evaluate the regions of a Winogradsky Column, add approximately one gram of material from the aerobic zone to T1 and resuspend by vortexing. Then, repeat this process with one gram of material from the anaerobic zone.
Remove the tube containing solution zero inoculated with E. coli from the incubator and shake it. Then, pipette one milliliter of the solution into a T1 test tube and vortex to mix. Remove one milliliter of solution from T1 and transfer it to T2, vortexing to mix. Repeat this process through tube T10. To evaluate the aerobic and anaerobic zones of the Winogradsky Column, remove one milliliter of solution from each of the previously prepared T1 tubes and transfer it to the appropriate T2 tubes. Then, continue the serial dilutions through the T10 tubes as previously demonstrated.
To spread plate, pipette 100 microliters of the diluted sample from each T3 tube on to the corresponding petri dish. Then, use a sterile spreading rod to gently distribute the sample on to the petri dish and replace the plate lid. Repeat this process for the T6 and T9 dilutions, as previously demonstrated. Incubate the plates containing aerobic organisms in a 37 degree Celsius incubator for 24 hours. Incubate the plates containing anaerobic organisms in an anaerobic chamber set to 37 degrees Celsius for 24 hours. The next day remove the T3, T6, and T9 dilution plates from the incubator and the anaerobic chamber and transfer them to the bench top. Working with one plate at a time, glide a sterile inoculating loop across the top of the media in a zig-zag pattern. Then, replace the petri dish lid. Next, rotate the plate by 1/3 and sterilize the loop to reduce the frequency of the previously made zig-zag pattern. Again, after sterilizing the loop, rotate the plate by 1/3, reduce the frequency of the zig-zag pattern one last time, and replace the lid. Repeat this streaking method for the remaining plates, as previously shown. Then, place the streaked plates containing aerobic organisms in a 37 degree Celsius incubator overnight and the streaked plates containing anaerobic organisms in an anaerobic chamber set to 37 degrees Celsius overnight.
Cultures were harvested from the aerobic and anaerobic zones of a seven day Winogradsky Column. Then, the cultures were serially diluted prior to streaking and spreading on LB agar plates. Streaking revealed a mixed population from each of the evaluated Winogradsky zones, and the spread plates produced similar results. A plate streaked from a mixed population will result in bacterial colonies of different shapes, sizes, textures, and colors. In contrast, the streaked and spread plates containing the known organism, E. coli, demonstrated a homologous population. Generally, it is best to calculate CFUs per milliliter using the average colony count of three plates spread with the same sample and dilution factor. Multiply the average number of colonies by the dilution factor and divide by the amount aliquoted. Finally, isolated colonies chosen from each plate can be used in further enrichment assays to determine species identity.
