Universelles und effizientes Elektroporationsprotokoll für die Gentechnik von gastrointestinalen Organoiden
Summary
Dieses Protokoll beschreibt eine effiziente Elektroporationsmethode zur Transfektion von vier verschiedenen gastrointestinalen organoiden Entitäten mit größeren Plasmiden (bis zu 10 kB). Es kann innerhalb eines Tages durchgeführt werden und benötigt keine umfangreiche Vorbereitung oder spezielle, kostenintensive Elektroporationspuffer.
Abstract
Elektroporation ist eine gängige Methode zur Transfektion mit verschiedenen Arten von Molekülen durch elektrische Permeabilisierung der Plasmamembran. Mit dem zunehmenden Einsatz von Organoiden als Kultivierungsmethode für Primärpatientenmaterial in den letzten Jahren sind effiziente Übertragungsmethoden von Komponenten für die Gentechnik in diesem 3D-Kultursystem notwendig. Besonders bei Organoiden hängt die Effizienz genetischer Manipulationen von einer erfolgreichen Transfektion ab. So wurde dieses Protokoll entwickelt, um die Elektroporation von Organoiden zu erleichtern und seine universelle Funktionalität in verschiedenen Einheiten zu beweisen. Humane Dickdarm-, Bauchspeicheldrüsen-, Leber- und Magenkrebs-Organoide wurden im Vergleich mit kleinen und großen Plasmiden erfolgreich elektropoiert. Basierend auf GFP-Codierungsvektoren wurde die Transfektionseffizienz von FACS bestimmt. Eine umfangreiche Vorbereitung der Zellen oder spezielle, kostenintensive Elektroporationspuffer sind nicht erforderlich, und das Protokoll kann innerhalb eines Tages durchgeführt werden.
Introduction
In den letzten Jahren wurde ein neuartiges 3D-Zellkultursystem, die so genannten Organoide, für verschiedene normale und krebserende Gewebe entwickelt. Organoide sind funktionell und morphologisch sehr nah an ihrem Ursprungsgewebe. Sie können aus verschiedenen Arten erzeugt werden, sind leicht erweiterbar, genomstabil und genetisch veränderbar, was sie zu einem idealen Modellsystem für genetische Untersuchungen1,2,3macht. Gentechnik-Techniken wie das CRISPR -System (Clustered Regularly Interspaced Short Palindromic Repeats)/Cas9-System ermöglichen vielfältige Manipulationen. Die Auswahl der Klone kann durch definierte Medienbedingungen realisiert werden, z.B. durch WNT-Ligandentzug für APC (Adenomatosis Polyposis Coli) Knockout-Klone4,5. Alternativ müssen Selektionsmarker durch homologe gezielte Reparatur eines Targeting-Vektors6,7eingeführt werden. Aufgrund der Tatsache, dass oft mehr als ein Plasmid eingeführt werden muss, wird eine effiziente Transfektion zu einem entscheidenden Parameter. Um unspezifische Off-Target-Effekte zu reduzieren, ist eine vorübergehende Expression der Cas9-Endonuklease wünschenswert8.
Elektroporation ist eine vergleichsweise einfache Methode, um Zellen mit DNA, RNA, Proteinen oder anderen Makromolekülen zu transfekieren. Durch elektrische Impulse wird die Zellmembran durchlässiger und verursacht eine erhöhte Aufnahme9. In einem zuvor veröffentlichten Elektroporationsprotokoll von Kolonorganoiden wurde in einem viertägigen Verfahren10ein 30 %iger Wirkungsgrad mit einem Piggy-Bac GFP (grünes fluoreszierendes Protein) Exzessvektor (7,4 kB) erreicht. Das folgende Protokoll wurde entwickelt, um eine effiziente Transfektion von Krebs oder gesunden Organoiden mit großen Plasmiden zu ermöglichen, die für Single Guide RNA (sgRNA) und die Cas9 Endonuklease-Sequenz (z.B. px458 als Vektor mit 9,3 kb) kodieren. Der gesamte Elektroporationsprozess kann innerhalb eines Tages ohne spezielle Elektroporationspuffer und mit mindestens vergleichbaren Wirkungsgraden zwischen verschiedenen gastrointestinalen Organoiden, nämlich pankreastischem duktogalem Adenokarzinom (PDAC), Dickdarmkrebs (CRC), Cholangiokarzinom (CCC) und Magenkrebs (GC) Organoiden durchgeführt werden.
Protocol
Representative Results
Discussion
Dieses Protokoll enthält detaillierte Anweisungen für eine effiziente, schnelle und einfach durchzuführende Elektroporation verschiedener organoider Einheiten. Zusätzlich zu den vorgestellten Tumororganoiden von PDAC, CRC, CCC und GC, funktioniert es erfolgreich für Organoide aus gesundem Gewebe sowie. Das Protokoll kann innerhalb eines Tages ausgeführt werden. In veröffentlichten Organoid-Transfektionsprotokollen dauerte das gesamte Verfahren vier Tage, einschließlich zwei Tage Vorbereitungen mit verschiedenen Arten von Anbaumedien10,21. In unserem Protokoll ist keine spezielle Vorbehandlung erforderlich. Durch Waschen mit Elektroporationspuffer vor der Elektroporation wurden die antibiotischen Komponenten der Medien ausgewaschen und eine Einstellung der Saline-Konzentrationen für optimale Impedanzwerte erreicht. Dennoch sollten einige kritische Aspekte für eine erfolgreiche Elektroporation in Betracht gezogen werden:
Zellen
Im Elektroporationsprotokoll von Fujii et al.10 wird empfohlen, Organoide mit Einzelzellen zu dissoziieren und durch ein 20-m-Zell-Sieb zu filtern. In unseren Händen verringert die Verdauung einzelner Zellen die Überlebensfähigkeit der Zellen stark. Wie in Merenda et al.21vorgeschlagen, dissoziierten wir auch Organoide zu Clustern von 10-15 Zellen und konnten keine verminderte Effizienz im Vergleich zur Einzelzelldissoziation bestimmen. Nach der Elektroporation ist es ein sehr wichtiger Schritt, den weißen Schaum zu dissoziieren, so dass keine angeschlossenen Zellen verloren gehen.
Für die 2D-Zellkultur hat sich gezeigt, dass eine Regenerationszeit nach Elektroporation von mehr als 10 min bis zu 40 min die Überlebensfähigkeit und Transfektionseffizienz insbesondere bei großen Plasmiden erhöht22. In Testexperimenten konnte dasselbe für Organoide dokumentiert werden, was zu einem Inkubationsschritt von 40 min nach Elektroporation in diesem Protokoll führte. Um die Erholung von der Elektroporation zu erhöhen, kultivierten wir sie mit Rho-assoziiertem Proteinkinase (ROCK) Inhibitor Y-27632 für fünf bis sieben Tage23. In ähnlicher Weise soll die zusätzliche Supplementierung von Glykogen-Synthase-Kinase-3-Hemmer CHIR99021 einzelnen Zellen helfen,10zurückzugewinnen.
Einstellungen
Einer der Vorteile des verwendeten Elektroporators ist, dass die Impedanz vor der Elektroporation für optimale Bedingungen gemessen werden kann. Nach Angaben des Herstellers sollten die Impedanzwerte 30-55 ° betragen. In unseren Händen haben Impedanzwerte von 30-40 ° eine optimale Effizienz gezeigt. In einem vorderen Experiment wurden unterschiedliche Spannungen und Pulslängenwerte des Porenimpulses variiert, um den optimalen Anteil der Effizienz an der Überlebensfähigkeit zu finden. Zusammenfassend konnten wir die beschriebenen Werte von Fujii et al.10 in den verschiedenen hier beschriebenen Entitäten bestätigen.
Dna
Die Wirkung unterschiedlicher DNA-Mengen wurde in Vorversuchen bis zu 45 g DNA pro Probe getestet. Es konnten keine zytotoxischen Wirkungen festgestellt werden. Die Transfektionseffizienz wurde in einer dosisabhängigen Weise mit Sättigung > 30 g erhöht. Wir haben also im Endprotokoll 30 g pro Probe verwendet, aber natürlich kann sie erhöht werden (z.B. für die Parallelelektroporation von mehr Plasmiden). Darüber hinaus scheint die Reinheit und Konzentration der DNA sehr wichtig zu sein. Eine Konzentration von mehr als 5 g/l hat eine optimale Transfektionseffizienz gezeigt.
Wie erwartet konnte das 9,3 kB-Plasmid mit einem geringeren Wirkungsgrad transfiziert werden als das kleinere 4,2 kB-Plasmid (siehe Abbildung 4). Die Verwendung von noch größeren Plasmiden als 10 kB wird die Effizienz weiter verringern. Für zukünftige Anwendungen könnte es interessant sein, Minikreis-DNA als Vektor zu testen, da diesen Genträgern das bakterielle Rückgrat eines Plasmids fehlt, was sie kleiner macht24. Dies sollte zu einer verbesserten Transfektionseffizienz führen. Darüber hinaus könnte für CRISPR-basierte Manipulationen von Organoiden eine direkte Elektroporation von sgRNAs, die an Cas9 als Ribonukleoprotein (RNP) Komplex gebunden sind, eine Alternative oder Ergänzung sein25.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wir danken Juliane Fohgrub, Ann-Christin Meinecke und Max Heiduk für die hervorragende technische Unterstützung. Gefördert wurden die Deutsche Krebshilfe (Nr. 111350 und 70112925), die Sander Stiftung (Nr. 2014.104.1), die Hector Stiftung (Nr. M65.2) und die Europäische Union (ERC-Nr. 639050).
Materials
| For establishment and culture medium | |||
| [Leu15] Gastrin | Sigma-Aldrich | G9145 | |
| A83-01 | Tocris Bioscience | 2939 | |
| Advanced DMEM/F-12 | Invitrogen | 12634010 | |
| B27 | Invitrogen | 17504044 | |
| B27 Supplement, minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| CHIR99021 | Stemgent | 04-0004 | |
| Collagenase II | Life Technologies | 17101-015 | |
| Collagenase XI | Sigma-Aldrich | C9407-100MG | |
| Collagenase D | Roche | 11088866001 | |
| Dispase II | Roche | 4942078001 | |
| Dnase I | Sigma-Aldrich | D5319 | |
| D-sorbitol | Roth | 6213.1 | |
| Dithiothreitol | Thermo Scientific | 1859330 | |
| EDTA | Roth | 8040 | |
| Forskolin | Tocris Bioscience | 1099 | |
| Glutamax | Life Technologies | 35050061 | |
| Hepes | Thermo Fisher Scientific | 15630106 | |
| hFGF-10 | Preprotech | 100-26 | |
| KCl | Sigma-Aldrich | P9541 | |
| KH2PO4 | Roth | 3904.2 | |
| Matrigel | Corning | 356231 | basement matrix |
| mEGF | Invitrogen | PMG8043 | |
| N2 | Invitrogen | 17502048 | |
| NaCl | Roth | 3957.1 | |
| Na2HPO4 | Roth | K300.2 | |
| N-Acetyl-L-Cystein | Sigma-Aldrich | A9165 | |
| Nicotinamid | Sigma-Aldrich | N0636 | |
| Noggin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (Hek293-mNoggin-Fc) |
| PBS | Gibco | 14190169 | |
| Penicillin Streptomycin | Life Technologies | 15140122 | |
| Primocin | InvivoGen | ant-pm-1 | |
| Prostaglandin E2 | Tocris Bioscience | 2296 | |
| Recombinant Human HGF | Preprotech | 100-39H | |
| Rspondin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (HA-Rspo1-Fc-293T) |
| SB202190 | Sigma-Aldrich | S7067 | |
| Sucrose | VWR | 27,480,294 | |
| TrypLE Express | Gibco | 12604021 | Dissociation reagent |
| Wnt3A | n.a. | n.a. | Conditioned medium produced from L-Wnt3a cells (from Sylvia Boj) |
| Y-27632 | Sigma-Aldrich | Y0503 | |
| Consumables | |||
| Cell Strainer 100 µm | Falcon | 352360 | |
| 48-well plate | Corning | 3548 | |
| Nepa Electroporation Cuvettes 2mm gap w/pipettes | Nepa Gene Co., Ltd. | EC-002S | |
| Tubes 15 ml | Greiner | 188271 | |
| Tubes 15 ml low binding | Eppendorf | 30122208 | Tubes for FACS preparing |
| Equipment | |||
| Electroporator Nepa21 | Nepa Gene Co., Ltd. | n.a. | |
| EVOS FL Auto | Invitrogen | AMAFD1000 | Fluorescence microscope |
| LSRFortessa | BD Bioscience | 647800E6 | FACS |
| Reagents and plasmids | |||
| Live/dead fixable blue dead cell stain kit | Invitrogen | L34962 | Includes antibody for live/dead staining for FACS analysis |
| EndoFree Plasmid Maxi Kit | Qiagen | 12362 | |
| Nutlin-3 | Selleckchem | S1061/07 | |
| Opti-MEM | Gibco | 31985047 | Electroporation buffer |
| 2 gRNA concatemer vector | AddGene | 84879 | |
| pCMV-EGFP | Nepa Gene Co., Ltd. | n.a. | |
| px458 plasmid | AddGene | 48138 | coding for sgRNA and Cas9 |
| px458_Conc2 plasmid | AddGene | 134449 | px458 plasmid containing 2x U6 promotors for two different sgRNAs |
| sgRNA_hTP53_1a | Eurofins Genomics | n.a. | 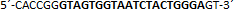 |
| sgRNA_hTP53_1b | Eurofins Genomics | n.a. | 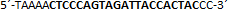 |
| sgRNA_hTP53_2a | Eurofins Genomics | n.a. | 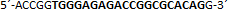 |
| sgRNA_hTP53_2b | Eurofins Genomics | n.a. | 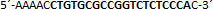 |
References
- Huch, M., et al. Long-term culture of genome-stable bipotent stem cells from adult human liver. Cell. 160 (1-2), 299-312 (2015).
- Werner, K., Weitz, J., Stange, D. E. Organoids as Model Systems for Gastrointestinal Diseases: Tissue Engineering Meets Genetic Engineering. Current Pathobiology Reports. 4 (1), 1-9 (2016).
- Olayanju, A., Jones, L., Greco, K., Goldring, C. E., Ansari, T. Application of porcine gastrointestinal organoid units as a potential in vitro tool for drug discovery and development. Journal of applied toxicology. 39 (1), 4-15 (2019).
- Drost, J., et al. Sequential cancer mutations in cultured human intestinal stem cells. Nature. 521 (7550), 43-47 (2015).
- Matano, M., et al. Modeling colorectal cancer using CRISPR-Cas9-mediated engineering of human intestinal organoids. Nature Medicine. 21 (3), 256-262 (2015).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Drost, J., et al. Use of CRISPR-modified human stem cell organoids to study the origin of mutational signatures in cancer. Science. 358 (6360), 234-238 (2017).
- Zhang, X. H., Tee, L. Y., Wang, X. G., Huang, Q. S., Yang, S. H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy Nucleic Acids. 4, e264 (2015).
- Tsong, T. Y. Electroporation of cell membranes. Biophysical journal. 60 (2), 297-306 (1991).
- Fujii, M., Matano, M., Nanki, K., Sato, T. Efficient genetic engineering of human intestinal organoids using electroporation. Nature Protocols. 10 (10), 1474-1485 (2015).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett’s epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Bartfeld, S., et al. In vitro expansion of human gastric epithelial stem cells and their responses to bacterial infection. Gastroenterology. 148 (1), 126-136 (2015).
- Boj, S. F., et al. Organoid models of human and mouse ductal pancreatic cancer. Cell. 160 (1-2), 324-338 (2015).
- Broutier, L., et al. Culture and establishment of self-renewing human and mouse adult liver and pancreas 3D organoids and their genetic manipulation. Nature Protocols. 11 (9), 1724-1743 (2016).
- Broutier, L., et al. Human primary liver cancer-derived organoid cultures for disease modeling and drug screening. Nature Medicine. 23 (12), 1424-1435 (2017).
- Hennig, A., et al. CFTR Expression Analysis for Subtyping of Human Pancreatic Cancer Organoids. Stem Cells Int. 2019, 1024614 (2019).
- Seidlitz, T., et al. Human gastric cancer modelling using organoids. Gut. 68 (2), 207-217 (2019).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Current protein & peptide science. 9 (4), 338-369 (2008).
- Andersson-Rolf, A., et al. Simultaneous paralogue knockout using a CRISPR-concatemer in mouse small intestinal organoids. Dev Biol. 420 (2), 271-277 (2016).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8, 2281 (2013).
- Merenda, A., et al. A Protocol for Multiple Gene Knockout in Mouse Small Intestinal Organoids Using a CRISPR-concatemer. Journal of Visualized Experiments. (125), (2017).
- Lesueur, L. L., Mir, L. M., Andre, F. M. Overcoming the Specific Toxicity of Large Plasmids Electrotransfer in Primary Cells In Vitro. Molecular Therapy Nucleic Acids. 5, e291 (2016).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
- Darquet, A. M., Cameron, B., Wils, P., Scherman, D., Crouzet, J. A new DNA vehicle for nonviral gene delivery: supercoiled minicircle. Gene Therapy. 4 (12), 1341-1349 (1997).
- Jacobi, A. M., et al. Simplified CRISPR tools for efficient genome editing and streamlined protocols for their delivery into mammalian cells and mouse zygotes. Methods. 121-122, 16-28 (2017).

