ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Il presente studio riporta un protocollo per lo screening cromosomico di embrioni umani che utilizza un terreno di coltura esausto, che evita la biopsia embrionale e consente l'identificazione della ploidia cromosomica utilizzando NGS. Il presente articolo presenta la procedura dettagliata, compresa la preparazione del terreno di coltura, l'amplificazione dell'intero genoma (WGA), la preparazione della libreria di sequenziamento di nuova generazione (NGS) e l'analisi dei dati.
Abstract
Nella fecondazione clinica in vitro (FIV), il metodo prevalente per la PGT-A richiede la biopsia di alcune cellule del trofoectoderma (TE). Questo è il lignaggio che forma la placenta. Questo metodo, tuttavia, richiede competenze specialistiche, è invasivo e soffre di falsi positivi e negativi perché il numero di cromosomi nella TE e nella massa cellulare interna (ICM), che si sviluppa nel feto, non sono sempre gli stessi. La NICS, una tecnologia che richiede il sequenziamento del DNA che viene rilasciato nel terreno di coltura sia da TE che da ICM, può offrire una via d'uscita a questi problemi, ma in precedenza ha dimostrato di avere un'efficacia limitata. Il presente studio riporta il protocollo completo del NICS, che include i metodi di campionamento del terreno di coltura, l'amplificazione dell'intero genoma (WGA) e la preparazione della libreria, e l'analisi dei dati NGS tramite software di analisi. Considerando i diversi tempi di crioconservazione nei diversi laboratori di embrioni, gli embriologi hanno due metodi per raccogliere il terreno di coltura embrionale che possono essere selezionati in base alle condizioni effettive del laboratorio di fecondazione in vitro.
Introduction
Le tecnologie di riproduzione assistita (PMA) sono sempre più utilizzate per il trattamento dell'infertilità. Tuttavia, il tasso di successo della fecondazione assistita, come la fecondazione in vitro, è stato limitato e il tasso di aborto è significativamente superiore a quellodella popolazione normale. La causa principale di questi problemi sono le anomalie cromosomiche, che esistono comunemente negli embrioni umani preimpianto2. La PGT-A è un metodo efficace per lo screening dell'equilibrio cromosomico degli embrioni prima dell'impianto 3,4. Alcuni studi hanno dimostrato che la PGT-A può ridurre il tasso di aborto e migliorare il tasso di gravidanza 5,6,7,8. Tuttavia, il PGT-A richiede competenze tecniche complesse che richiedono una formazione e un'esperienza specifiche. Anche la procedura invasiva di biopsia embrionale potrebbe potenzialmente causare danni agli embrioni9. Gli studi hanno dimostrato che la biopsia dei blastomeri può ostacolare lo sviluppo successivo e il numero di TE sottoposti a biopsia può influenzare i tassi di impianto10. Sebbene il problema della biosicurezza a lungo termine della biopsia embrionale non sia ancora stato valutato a fondo nell'uomo, gli studi sugli animali hanno dimostrato le sue influenze negative sullo sviluppo embrionale11,12,13.
Rapporti precedenti hanno indicato che tracce di materiali di DNA sono state secrete nel terreno di coltura durante lo sviluppo embrionale e sono stati compiuti sforzi per eseguire uno screening cromosomico completo (CCS) utilizzando il terreno di coltura embrionaleesausto 14,15,16,17,18. Tuttavia, i tassi di rilevamento e l'accuratezza dei test non hanno soddisfatto i requisiti per un uso clinico estensivo. Il presente studio ha riportato un miglioramento nel test NICS per aumentare i tassi di rilevamento e l'accuratezza del test NICS19. Negli ultimi anni, il liquido blastocele (BF) è stato studiato come campione analitico di PGT-A minimamente invasivo. Tuttavia, la percentuale di amplificazione dell'intero genoma e di DNA rilevabile nei campioni di liquido di blastocisti varia dal 34,8% all'82%20,21,22. Il volume di BF riportato in vari studi varia da 0,3 nL a 1 μL. In considerazione della bassa quantità di DNA nel BF, è possibile aumentare la quantità di DNA libero da cellule mescolando il liquido della blastocisti e il terreno di coltura per migliorare il tasso di successo e la coerenza del rilevamento. Kuznyetsov et al.23 e Li et al.24 hanno trattato la zona pellucida con un laser e hanno rilasciato il liquido della blastocisti nel terreno di coltura per migliorare la quantità totale di DNA embrionale e il tasso di amplificazione dei campioni combinati di terreno / BF dopo WGA era rispettivamente del 100% e del 97,5%. Anche Jiao et al.25 hanno ottenuto un tasso di successo dell'amplificazione del 100% utilizzando lo stesso metodo.
Il presente studio riporta un protocollo dettagliato che include la preparazione del campione di terreno esausto, la preparazione NGS e l'analisi dei dati. Rimuovendo con cura le cellule del cumulo dagli ovociti, il presente studio ha eseguito l'iniezione intracitoplasmatica di singoli spermatozoi (ICSI) e la coltura di blastocisti. Il giorno 4-giorno 5/giorno 6 è stato raccolto per la preparazione della libreria WGA e NGS. Utilizzando la tecnologia NICS, il presente studio ha semplificato le fasi di preparazione delle librerie WGA e NGS in circa 3 ore e ha ottenuto risultati CCS in modo non invasivo in circa 9 ore.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Il permesso etico è stato ottenuto dal Comitato Etico del Terzo Ospedale dell'Università di Pechino.
1. Preparazione
NOTA: I materiali e le attrezzature richiesti sono elencati nella Tabella dei materiali.
- Reagenti
- Preriscaldare ed equilibrare (bilanciato) 20-30 μL di terreno di gameti/terreno di fertilizzazione e terreno di coltura allo stadio di scissione/blastocisti (ricoperto di olio minerale) e ialuronidasi (in una provetta ben tappata) a 37 °C, 5% CO2 e 5 % O2 in un incubatore Tri-gas per una notte prima dell'uso.
- Preriscaldare la ialuronidasi a 37 °C su una superficie di lavoro in una cappa aspirante.
- Preparare il tampone di vitrificazione e i reagenti per la raccolta dei campioni secondo le istruzioni del produttore.
- Utensileria
- Preparare le pipette per la raccolta e il trasferimento dei campioni (diametro interno da ~200 a 250 μm), le pipette per denudazione/stripper (diametro interno di ≥150 μm, ~130-140 μm e ~120 μm) e le pipette per il lavaggio (diametro interno di ~150 μm) tirando le pipette Pasteur in vetro per generare puntali fini aperti lucidati a fuoco.
NOTA: Le pipette utilizzate per la raccolta/trasferimento dei campioni, la denudazione e il lavaggio possono essere acquistate direttamente. Gli aghi di tenuta e gli aghi per iniezione possono anche essere acquistati direttamente.
- Preparare le pipette per la raccolta e il trasferimento dei campioni (diametro interno da ~200 a 250 μm), le pipette per denudazione/stripper (diametro interno di ≥150 μm, ~130-140 μm e ~120 μm) e le pipette per il lavaggio (diametro interno di ~150 μm) tirando le pipette Pasteur in vetro per generare puntali fini aperti lucidati a fuoco.
2. Protocollo 1: Raccolta del campione
- Pretrattamento del complesso ovocita-corona-cumulo (OCCC) prima della digestione con ialuronidasi
- Ottieni la stimolazione ovarica sia con l'ormone follicolo-stimolante (FSH) che con i preparati a base di gonadotropina umana della menopausa (hMG). Quando il follicolo principale è >18 mm, utilizzare 10.000 UI di gonadotropina corionica (hCG) per la maturazione finale dell'ovocita.
- Eseguire il prelievo degli ovociti 36 ore dopo lo scatto del grilletto. Prelevare e trasferire gli ovociti in piastre di coltura tissutale con 2,5 mL di m-HTF preriscaldato ricoperto di olio minerale.
- Trasferire rapidamente gli OCCC nel pozzetto centrale di una capsula di coltura d'organo contenente 1 mL di terreno di fertilizzazione utilizzando una pipetta di trasferimento e quindi incubare con gli ovociti a 37 °C in un'incubatrice al 5% di CO 2 e al 5% di O 2 per2-4 ore.
- Digerire gli OCCC con ialuronidasi aggiungendo 1 mL di ialuronidasi preriscaldata a 37 °C (80 UI/mL) al pozzetto centrale di una piastra di coltura d'organo contenente OCCC (passaggio 2.1.3). Mantenere la concentrazione finale di ialuronidasi a 40 UI/mL e mescolare accuratamente.
- Incubare gli OCCC su una piattaforma termica a 37 °C per 2 min. Osservare i cambiamenti al microscopio ogni 30 secondi fino a quando non sono rimasti solo 1-2 strati di cellule della granulosa.
- Dedenudazione delle cellule della granulosa
- Trasferire rapidamente gli OCCC digeriti nel piatto di coltura per la manipolazione degli ovociti e coprire con olio minerale in ogni pozzetto.
- Osservare le cellule della granulosa separate al microscopio. Aspirare delicatamente e rilasciare gli ovociti 5 volte per rimuovere le cellule residue della granulosa intorno agli ovociti.
- Ripetere il passaggio precedente nei restanti 3 pozzetti per rimuovere completamente le cellule della granulosa.
NOTA: I passaggi precedenti (2.1-2.3) possono essere eseguiti in base alle operazioni di routine di ciascun laboratorio.
- Valutazione dell'ovocita
- Valutare la completezza della rimozione delle cellule della granulosa utilizzando un microscopio. Se le cellule non possono essere rimosse completamente, la ritenzione di 5 o meno cellule della granulosa è accettabile in questo momento.
NOTA: Se le cellule del cumulo sono ancora attaccate all'ovocita, il resto può essere rimosso più tardi il giorno 3 prima che l'embrione venga trasferito dal terreno di coltura allo stadio di clivaggio al terreno di coltura allo stadio di blastocisti.
- Valutare la completezza della rimozione delle cellule della granulosa utilizzando un microscopio. Se le cellule non possono essere rimosse completamente, la ritenzione di 5 o meno cellule della granulosa è accettabile in questo momento.
- Dopo aver eseguito l'iniezione intracitoplasmatica di spermatozoi (ICSI)26, trasferire gli ovociti in microgoccioline di terreno di coltura embrionale da 20-30 μL (un ovocita corrisponde a una microgoccia) utilizzando pipette di trasferimento e incubare in un'incubatrice a 37 °C, 5% CO2 e 5% O2 .
- Registrare il giorno dell'ICSI come giorno 0. Controllare gli embrioni e il punteggio secondo il workshop di consenso di Istanbul sulla valutazione embrionale del giorno 1 per la fecondazione (circa 18 ore), il giorno 2 (circa 45 ore) e il giorno 3 (circa 68 ore) per la scissione dell'embrione27.
- Lavaggio embrionale
- Preparare 20-30 μL di microgoccioline di terreno di coltura di blastocisti per ciascun embrione ricoperto di olio minerale in piastre di coltura tissutale al giorno 2 in un'incubatrice a 37 °C, 5% CO 2 e 5% O2 .
- Preparare altre tre microgoccioline ricoperte di olio minerale ed etichettare le nuove piastre di coltura tissutale per il lavaggio n. 1-3.
- Trasferire gli embrioni del giorno 3 nelle microgoccioline di lavaggio. Aspirare delicatamente e rilasciare gli embrioni 3 volte in ogni gocciolina utilizzando pipette per la denudazione.
NOTA: Questa procedura può anche aiutare a rimuovere le cellule granulari residue attaccate all'embrione. - Osservare e valutare gli embrioni al microscopio il giorno 3 prima che il terreno di coltura fosse cambiato da terreno di coltura in fase di clivaggio a terreno di coltura a blastocisti per il punteggio morfologico. Se le cellule del cumulo erano ancora attaccate all'embrione, pipettare opportunamente su e giù in un'altra gocciolina di terreno di coltura di blastocisti preriscaldata ed equilibrata coperta di olio minerale con una pipetta stripper fino a quando le cellule del cumulo non sono state completamente rimosse.
NOTA: Tutte le cellule del cumulo attaccate dovevano essere rimosse completamente il giorno 3 prima che l'embrione fosse trasferito dalla piastra del terreno di coltura allo stadio di clivaggio alla piastra del terreno di coltura allo stadio di blastocisti. Eventuali cellule cumulose rimanenti interferiranno nell'analisi finale e daranno risultati falsi negativi.
- Due opzioni per la raccolta del terreno di coltura
NOTA: Il centro di fecondazione in vitro può scegliere tra uno dei due metodi per la raccolta del terreno di coltura in base alle risorse, alle richieste e alle preferenze del centro.- Opzione 1: lavaggio e coltura dell'embrione
NOTA: Questa opzione è per i laboratori di fecondazione in vitro che eseguono la vitrificazione la mattina del giorno 5.- Trasferire l'embrione in microgoccioline preriscaldate (37 °C) di terreno di coltura e lavare delicatamente ogni embrione in serie in 3 microgoccioline mediante pipettaggio al 4° giorno pomeriggio.
- Trasferire ogni embrione in un'unica microgoccia singola preriscaldata (37 °C) di terreno di coltura per la raccolta del campione. Il volume di una singola goccia di terreno di coltura non può superare i 25 μL.
- Eseguire la coltura embrionale di blastocisti il giorno 5/giorno 6 a 37 °C, 5% CO2 e 5% O2 .
- Opzione 2: lavaggio e coltura dell'embrione
NOTA: Questa opzione è per i laboratori di fecondazione in vitro che eseguono la vitrificazione il giorno 5, il pomeriggio o il giorno 6.- Trasferire l'embrione in microgoccioline preriscaldate (37 °C) di terreno di coltura da 10-15 μL e lavare delicatamente ogni embrione in serie in 3 microgoccioline mediante pipettaggio il 5° giorno.
- Trasferire ogni embrione in un'unica microgoccia singola preriscaldata (37 °C) di terreno di coltura per la raccolta del campione. Il volume di una singola goccia di terreno di coltura non può superare i 15 μL.
- Eseguire la coltura embrionale di blastocisti il giorno 5/giorno 6 a 37 °C e 5% di CO2 .
- Opzione 1: lavaggio e coltura dell'embrione
- Raccolta dei campioni
- Regolare delicatamente l'ICM a una distanza considerevole dal punto mirato del raggio laser, che si concentra sulla giunzione cellulare del trofoectoderma per generare un piccolo foro nel trofoectoderma per rilasciare il fluido dalla cavità del blastocele. Quindi gli embrioni vengono spostati nella soluzione di congelamento per la crioconservazione secondo il processo convenzionale.
- Trasferire il terreno di coltura da ciascun embrione in coltura in una provetta PCR priva di RNasi/DNasi contenente 5 μL di tampone di lisi cellulare.
- Raccogliere la stessa quantità di terreno di coltura senza essere utilizzato per la coltura embrionale come controllo negativo. Congelare immediatamente tutti i campioni raccolti in azoto liquido e quindi conservarli a -80 °C dopo essere stati raccolti fino a quando non vengono sottoposti al test NICS.
- Eseguire la vetrificazione come descritto nel protocollo.
3. Protocollo 2: Costruzione della Biblioteca
- Lisi del terreno di coltura
- Diluire 1 μL di controllo positivo (10 ng di gDNA umano) con 199 μL di terreno di coltura fresco. Mescolare accuratamente e centrifugare brevemente la provetta (200 x g per 5 s).
- Trasferire 10 μL di terreno di coltura per blastocisti giorno 5-giorno 6, controllo positivo diluito e terreno di coltura fresco in nuove provette PCR da 0,2 mL.
- Aggiungere 1 μL di MT Enzyme Mix a ciascuna provetta PCR e mescolare accuratamente mediante pipettaggio e centrifugare immediatamente per 2-3 s a 200 x g.
- Mettere la/e provetta/e PCR/e del punto 3.1.3 in una stazione di preparazione del campione NICS preriscaldata ed eseguire il programma di lisi come segue: 10 min a 75 °C; 4 min a 95 °C; tenere a 22 °C.
NOTA: La stazione di preparazione del campione è paragonabile a una macchina PCR standard.- Fare clic sull'icona Lysis per accedere alla schermata di configurazione.
- Selezionare Tube per la modalità di controllo; ingresso 10 μL per il volume del campione; selezionare On per il controllo Hotlid e immettere 105 °C per la temperatura. Selezionare No per Pausa alla prima seg. Fare clic su OK per procedere.
- Attendere fino a quando l'ora di permanenza non viene visualizzata --: --:--, che indica la fine del programma, quindi fare clic su Interrompi per terminare il programma.
- Arrestare il programma al termine del processo. Procedere immediatamente al passaggio successivo.
- Preparazione della pre-libreria
- Scongelare il tampone Pre-Lib a RT. Mescolare accuratamente mediante pipettaggio e centrifugare immediatamente per 2-3 s a 200 x g.
- Preparare una miscela master per la reazione di prelibreria come segue: aggiungere 2 μL di Pre-Lib Enzyme Mix a 60 μL di Pre-Lib Buffer, mescolare accuratamente la reazione e centrifugare brevemente.
- Aggiungere 60 μL di miscela di reazione pre-libreria in ciascun campione di terreno pretrattato della fase precedente. Mescolare accuratamente mediante pipettaggio e centrifugare immediatamente per 2-3 s a 200 x g.
- Posizionare la/e provetta/e PCR/e del punto 3.2.3 nella stazione di preparazione del campione ed eseguire il programma di prelibreria come segue: 95 °C per 2 min; 12 cicli di 15 °C per 40 s, 22 °C per 40 s, 33 °C per 30 s, 65 °C per 30 s, 72 °C per 40 s, 95 °C per 10 s e 63 °C per 10 s; e tenere a 4 °C.
- Fare clic sull'icona Pre_Lib per accedere alla schermata di configurazione.
- Selezionare Tube per la modalità di controllo; ingresso 70 μL per il volume del campione; selezionare On per Controllo coperchio caldo e immettere 105 °C per la temperatura. Selezionare No per Pausa alla prima seg. Fare clic su OK per procedere.
- Attendere fino a quando il tempo rimanente non viene visualizzato --: --:--, che indica la fine del programma, quindi fare clic su Interrompi per terminare il programma.
- Arrestare il programma al termine del processo. Procedere immediatamente al passaggio successivo.
- Preparazione della libreria
- Scongelare il Library Buffer a RT. Miscelare accuratamente mediante pipettaggio e centrifugare immediatamente per 2-3 s a 200 x g.
- Preparare una miscela master per la reazione di libreria come segue: aggiungere 1,6 μL di miscela di enzimi di libreria a 60 μL di tampone di libreria, mescolare accuratamente la reazione e centrifugare brevemente.
- Aggiungere 60 μL di miscela di reazione della libreria e 2 μL di Barcode Primer a ciascun prodotto della prelibreria dal punto 3.2.3. Mescolare accuratamente la reazione e centrifugare brevemente.
- Posizionare la/e provetta/e PCR/e del passaggio 3.2.3 nel termociclatore ed eseguire il programma di preparazione della libreria come segue: 94 °C per 30 s; 17 cicli di 94 °C per 25 s, 62 °C per 30 s e 72 °C per 45 s); e poi tenere a 4 °C.
- Fare clic sull'icona Lib_Prep per accedere alla schermata di configurazione.
- Selezionare Tube per la modalità di controllo; ingresso 130 μL per il volume del campione; selezionare On per il controllo Hotlid e immettere 105 °C per la temperatura corrispondente. Selezionare No per Pausa alla prima seg. Fare clic su OK per procedere.
- Attendere fino a quando il tempo rimanente non viene visualizzato --: --:--, che indica la fine del programma, quindi fare clic su Interrompi per terminare il programma.
- Purificazione della libreria
- Rimuovere Magbeads dalla conservazione a 2-8 °C per almeno 20 minuti prima della fase di purificazione. Vorticare e mescolare i Magbeads per 20 s. Erogare un numero sufficiente di microsfere per la fase di purificazione in una nuova provetta per microcentrifuga da 1,5 mL e riscaldare le microsfere a RT.
- Aggiungi 1x Magbeads in ogni libreria. Mescolare pipettando su e giù ≥10 volte e incubare a RT per 5 minuti.
NOTA: Ad esempio, aggiungere 100 μL di Magbeads a 100 μL di campione di libreria. - Dopo l'incubazione, centrifugare brevemente la provetta e posizionarla su un supporto magnetico.
- Attendere circa 5 minuti fino a quando la soluzione diventa limpida. Mantenendo il tubo sul supporto magnetico, aspirare con cautela la soluzione e gettarla.
- Aggiungere 200 μL di etanolo all'80% appena preparato nella provetta. Incubare a RT per 30 s e rimuovere con cautela il surnatante. Ripeti ancora una volta.
- Rimuovere l'etanolo nel modo più completo possibile. Asciugare all'aria le perline sul supporto magnetico per circa 5-10 minuti a RT.
- Rimuovere il tubo dal supporto magnetico, aggiungere 17,5 μL di tampone di eluizione e vorticare il tubo per risospendere le perle. Centrifugare brevemente la provetta e incubare a RT per 5 min.
- Posizionare il tubo sul supporto magnetico e attendere che la soluzione diventi limpida. Trasferire con cautela 15 μL di surnatante in una nuova provetta.
- Quantificazione della libreria
- Quantificare le librerie purificate utilizzando il fluorimetro secondo la guida per l'utente dei kit di analisi dsDNA HSqubit 28. La resa delle librerie varia da ~15 a 300 ng.
- Pooling delle librerie
- Utilizzare 10 nanogrammi di ciascun campione di libreria per il pooling.
- Sequenziamento
- Fare riferimento alla guida per l'utente del sequenziamento (15027617 v01)29.
- Le sequenze di libreria purificate di 50 bp a una singola estremità sulla piattaforma hanno prodotto circa 2 milioni di letture per ciascun campione ed è stata raccomandata una profondità di sequenziamento di 0,03 ×.
- Analisi dei dati
- Inserisci il nome e la password degli utenti nella pagina di accesso
- Dopo aver effettuato l'accesso al sistema, fare clic su Analisi e verrà visualizzata una nuova pagina. Fare clic su Crea invio nella scheda NICS-A. Quindi, scegli NGS per la piattaforma, seleziona corporation, scegli NICSInst per il reagente, inserisci le informazioni sul progetto nella casella sotto ID progetto, imposta le preferenze di analisi e carica i file. Una volta caricati correttamente tutti i file di sequenziamento, fare clic su Invia per avviare l'analisi (Figura 3A).
- Fare clic su Visualizza invii per visualizzare l'elenco dei progetti inviati. Una volta completata l'analisi, lo stato di un progetto diventerà Completato e nel campo del report verrà visualizzato un pulsante Mostra. Fare clic sul pulsante Mostra per visualizzare la tabella riepilogativa dell'analisi NICS (Figura 3B).
- Fare clic sul pulsante Esporta report per salvare i report (Figura 3C).
NOTA: Per ogni analisi verranno esportati tre tipi di file. Un file grafico che include tutti i grafici di variazione del numero di copie (CNV) per ogni cromosoma e l'intero genoma, che verrà memorizzato nella cartella "graph"; un foglio di calcolo che contiene i dettagli del CQ di esempio di questa esecuzione dell'analisi; un file di documento che contiene i report NICS personalizzati dall'utente; e un foglio di calcolo che contiene le informazioni di riepilogo di esempio di questa esecuzione dell'analisi.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Il presente studio ha applicato il metodo proposto ad un paziente. L'approvazione dell'IRB e il consenso informato sono stati ottenuti prima dell'applicazione dell'analisi NICS. Il presente studio ha ottenuto 6 blastocisti da pazienti e ha eseguito NICS su tutti e 6 gli embrioni dal giorno 4 al giorno 5 medio. Le anomalie cromosomiche causate dalla traslocazione bilanciata dei genitori sono state rilevate in cinque dei cromosomi con il test NICS; pertanto, non potevano essere utilizzati per il trasferimento (Figura 4A-E). I risultati NICS dei due embrioni hanno mostrato lo stesso cariotipo 45 e XN e -18 (×1) erano entrambe delezioni del cromosoma 18 (Figura 4A, B). Il cariotipo 46, XN, -1p (pter→p21.1, ×1) è solo il braccio corto della delezione della regione pter→p21.1 del cromosoma 1 (Figura 4D).
I risultati del NICS hanno mostrato cariotipo 46, XN, +1p (pter→p21.2, ×3) e -18 (q21.32→qter, ×1), il che ha indicato che sia il braccio lungo della delezione della regione del cromosoma 18 q21.32→qter che il braccio corto della regione pter→p21.2 del cromosoma 1 erano duplicati (Figura 4E). Sebbene i cariotipi 46, XN, +5q (×4) e -8 (×1, mos) siano duplicazioni del cromosoma 5 e mostrino 8 differenze di mosaico, il test NICS può esaminare tutti i 24 cromosomi per aneuploidia. Questo processo fornisce un nuovo metodo per il trasferimento di singole blastocisti cariotipiche normali.

Figura 1. La completezza della rimozione delle cellule del cumulo. (A) Gli ovociti con cellule cumuliformi. (B) Gli ovociti senza cellule cumuliformi attaccate. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2. Le cellule cumuliformi vengono rimosse da un embrione in D3 prima di essere trasferite nel midollo osseo. Tutte le cellule del cumulo attaccate devono essere rimosse prima che il terreno di coltura dell'embrione cambi dalla piastra del terreno di coltura embrionale di clivaggio iniziale alla piastra del terreno di coltura della blastocisti, che è il giorno 3 dopo che gli embrioni hanno raggiunto lo stadio di 8 cellule. Eventuali cellule del cumulo che non vengono rimosse interferiranno nell'analisi finale dando risultati falsi negativi. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3. Analisi dei dati. (A) Sono disponibili diverse opzioni per l'applicazione utente. Per la società di sequenziamento della piattaforma, gli utenti possono scegliere Illumina, Ion Torrent o MGI. Gli utenti possono scegliere se riportare o meno le informazioni sul sesso. Terminata l'impostazione del parametro di cui sopra, fare clic sulla casella sotto Caricamento file e scegliere i file di sequenziamento appropriati da caricare. Per Illumina, scegli i file con estensione fastq.gz. Fare clic su Invia per avviare l'analisi dopo il caricamento. (B) La vista della tabella riepilogativa. La tabella riepilogativa è costituita dalle seguenti informazioni: Nome campione: viene elencato il nome di ogni campione NICS; QC dati: indica se il file di sequenziamento supera il QC per l'analisi NICS; Valutazione AI: la valutazione (A, B o C) per ciascun campione NICS; AI_Rating Interpretazione: Valutazione del potenziale di impianto dell'embrione; AI Grading: il punteggio per ogni campione NICS; Grafico CNV (Whole Genome): Visualizza i profili CNV di tutti i cromosomi; (c) La pagina Salva report. Fare clic sul pulsante Esporta report accanto al Riepilogo dei risultati. Selezionare le informazioni che si desidera visualizzare nel report finale e fare clic su Esporta. I report verranno salvati nella cartella Download del computer. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4. Screening e selezione embrionale mediante NICS da un paziente. Un totale di sei embrioni si sono sviluppati con successo fino allo stadio di blastocisti e il terreno di coltura Day4-Day5 da ciascun embrione è stato raccolto per il test NICS. (A) e (B) sono i risultati NICS dei due embrioni di blastocisti che hanno mostrato lo stesso cariotipo 45, XN, -18(×1) sono entrambi delezione del cromosoma 18. (C) ha mostrato che il cariotipo 46, XN, +5q (×4), -8(×1, mos) è la duplicazione del cromosoma 5 e il mosaico 8. (D) ha mostrato che il cariotipo 46, XN, -1p (pter→p21.1, ×1) è solo il braccio corto della delezione della regione pter→p21.1 del cromosoma 1, mentre (E) ha mostrato il cariotipo 46, XN, +1p (pter→p21.2, ×3), -18(q21.32→qter, ×1) è il braccio corto della duplicazione della regione pter→p21.2 del cromosoma 1 e il braccio lungo del cromosoma 18 q21.32 → regione qter (F) ha mostrato una composizione cromosomica bilanciata. L'asse x indica 22 autosomi in rosso e blu, l'asse y indica il numero di copie di ciascun autosoma. I punti grigi sono la scala del righello della risposta del numero di copie, ogni finestra del contenitore e il cariotipo normale del numero di copia deve essere 2. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella S1. Le percentuali di successo del rilevamento del DNA Opzione 1 e Opzione 2. Clicca qui per scaricare questa tabella.
Tabella S2. La concordanza tra NICS e PGT-A in diverse opzioni. Clicca qui per scaricare questa tabella.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Modifiche e risoluzione dei problemi
Se i risultati del NICS sono contaminati da materiale genetico parentale, assicurarsi che tutte le cellule del cumulo-corona radiata siano state rimosse e assicurarsi che l'ICSI venga eseguita per la fecondazione. Si evitano processi impropri di conservazione del terreno o di preparazione del modello, che possono degradare il DNA. Lo spazio di lavoro è stato accuratamente purificato con reagenti di decontaminazione DNasi e RNasi. Per evitare la contaminazione da parte di altri embrioni, un embrione è stato sempre coltivato in una singola gocciolina di terreno per evitare la contaminazione incrociata a partire dal 4° giorno. Il fenomeno della contaminazione è ridotto al minimo quando si ritarda l'inserimento degli embrioni nella coltura finale goccia30,31,32,33. Per ridurre al minimo la contaminazione materna, Kuznyetsov34 ha modificato le procedure di coltura embrionale dal giorno 0 al giorno 4, compresa un'attenta rimozione delle cellule corona residue mediante pipettaggio e lavaggio.
Lane et al.30 mostrano che quando si prende il terreno di coltura embrionale dal giorno 4 al giorno 5, l'accuratezza del rilevamento dell'euploidia è migliorata, la consistenza della ploidia embrionale è superiore al 95% e la consistenza del cromosoma sessuale raggiunge il 100%. Lledo et al.33 hanno scoperto che il tasso di coincidenza tra il terreno di coltura giorno 3-giorno 5 e i campioni di TE era del 74,6% e del 92,0% quando gli embrioni sono stati coltivati dal giorno 4 al giorno 6.
Anche i dati interni della Corte supportano questa conclusione, come illustrato nella Tabella S1. Rispetto al metodo di coltura convenzionale giorno 3-giorno 5, le cellule della granulosa sono state ulteriormente rimosse a causa di un ulteriore cambiamento nel terreno di coltura il giorno 4 o il giorno 5. Forniamo dati interni (Tabella S1) che dimostrano che i nostri due metodi (opzione 1 e opzione 2) hanno una buona coerenza rispetto al PGT-A, che è migliore del metodo di campionamento senza la rimozione completa di CC.
Nel controllo negativo sono apparsi prodotti amplificati dal IF e i materiali esterni del DNA possono aver contaminato il reagente o lo spazio di lavoro. L'area di lavoro deve essere pulita con reagenti per la rimozione del DNA/RNA, devono essere utilizzati materiali privi di nucleasi e i reagenti devono essere aliquotati dopo il primo utilizzo.
Le differenze nelle percentuali di successo tra l'opzione 1 e l'opzione 2 sono discusse nelle tabelle S1 e S2.
Limiti del test NICS
Esistono due limitazioni principali di NICS. 1) Prima dell'ICSI, tutte le cellule del cumulo (di solito di origine materna, di solito la composizione cromosomica normale) devono essere rimosse. Se la rimozione è incompleta, le cellule del cumulo possono rilasciare DNA durante lo sviluppo dell'embrione e il DNA esterno viene amplificato, il che può essere la causa del rilevamento di falsi negativi. 2) È difficile rimuovere gli spermatozoi attaccati alla zona pellucida e si consiglia vivamente di eseguire la procedura NICS con ICSI. Sebbene la sostituzione regolare dei mezzi di scissione il giorno 3 possa ridurre la possibilità di contaminazione dovuta alle cellule del cumulo e agli spermatozoi ridondanti, questa contaminazione deve essere ridotta al minimo se il NICS viene utilizzato nella fecondazione in vitro clinica. Tuttavia, è stato sviluppato un metodo per rilevare la NICS negli embrioni di fecondazione in vitro, compresa la funzione di riconoscimento del DNA esogeno, che sarà dimostrata nel prossimo futuro.
Questo studio non ha confrontato le differenze tra i diversi terreni poiché studi clinici su larga scala hanno confrontato i terreni di coltura. Otto centri hanno utilizzato 4 diversi terreni di coltura, sequenziali e continui, e 2 diverse percentuali di supplementazione di albumina (5% e 10%), e queste differenze non hanno avuto effetti significativi sull'accuratezza dei risultati del cfDNA embrionale31. Questi risultati supportano la potenziale applicabilità dell'analisi del cfDNA embrionale a ogni laboratorio di fecondazione in vitro quando si lavora nell'ambito del protocollo specifico.
Significatività rispetto ai metodi esistenti
Il metodo NICS evita la biopsia embrionale e quindi migliora notevolmente la sicurezza d'uso. Rispetto alle blastocisti, la NICS è una tecnica di screening preimpianto semplice, che consente di risparmiare tempo, sensibile e riproducibile, adatta alle popolazioni di riproduzione assistita con un'alta probabilità di aneuploidia. A differenza della biopsia invasiva, che richiede una notevole conoscenza professionale per la procedura di biopsia della blastocisti, la NICS può essere applicata in modo estensivo poiché la sua semplice raccolta del terreno esausto segue solo il normale funzionamento della FIVET19 e non richiede la qualifica PGS/PGD in alcuni paesi.
Applicazioni future
Il NICS ha il potenziale per un'ampia applicabilità per lo screening cromosomico nella fecondazione in vitro clinica, non solo per l'ICSI ma anche per gli embrioni di fecondazione in vitro. Sebbene l'ICSI sia altamente raccomandata, sono necessari metodi di rimozione degli spermatozoi attaccati alla zona pellucida per prevenire l'influenza degli spermatozoi.
La valutazione morfologica è un metodo tradizionale per la valutazione embrionale, ma nella maggior parte dei casi, gli embrioni cromosomicamente anomali possono apparire morfologicamente simili agli embrioni cromosomicamente normali (euploidi). La combinazione della valutazione morfologica con il test NICS durante il trasferimento di embrioni ploidi con una buona morfologia nell'utero potrebbe migliorare i tassi di gravidanza in corso e il tasso di nascite vive. Verrà condotto uno studio clinico randomizzato per valutare l'efficacia clinica del trasferimento di un singolo embrione utilizzando la tecnologia NICS.
Passaggi critici del protocollo
Tutte le cellule del cumulo-corona radiata devono essere rimosse dagli ovociti prima della fecondazione. Gli ovociti sono stati fecondati mediante iniezione intracitoplasmatica di spermatozoi (ICSI). È stata evitata l'aggiunta di proteine/integratori di derivazione umana al terreno di coltura. Il terreno di coltura è stato cambiato il giorno 4 e raccolto il giorno 5-giorno 6 quando le blastocisti si sono completamente espanse. Gli embrioni sono stati coltivati in singole goccioline di terreno di coltura a partire dal 4° giorno. Durante la raccolta del terreno di coltura, le pipette di trasferimento tra i campioni sono state cambiate per evitare la contaminazione.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yaxin Yao, Jieliang Ma, Jing Wang e Sijia Lu sono dipendenti di Yikon Genomics Co., Ltd.
Acknowledgments
Gli autori desiderano ringraziare Shiping Bo e Shujie Ma per la loro assistenza nell'analisi dei dati NGS. Finanziamento: questo lavoro è stato sostenuto dal National Key Research and Development Program (Grant No. 2018YFC1003100).
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
References
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Jr Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T. Jr, Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid? Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- Thermo Fisher Scientific. Qubit dsDNA HS Assay Kit. , Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015).
- Miseq system use guide. , Available from: https://support.illumina.com/downloads/miseq_system user _ guide 15027617.html (2016).
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Tags
Screening cromosomico embrioni umani preimpianto terreno di coltura esausto raccolta di campioni analisi della ploidia cromosomica fecondazione in vitro (FIV) test genetici preimpianto per aneuploidia (PGT-A) trofectoderma (TE) massa cellulare interna (ICM) falsi positivi e negativi tecnologia NICS sequenziamento del DNA campionamento del terreno di coltura amplificazione dell'intero genoma (WGA) preparazione della libreria analisi dei dati NGS crioconservazione laboratorio di fecondazione in vitroErratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021.
Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
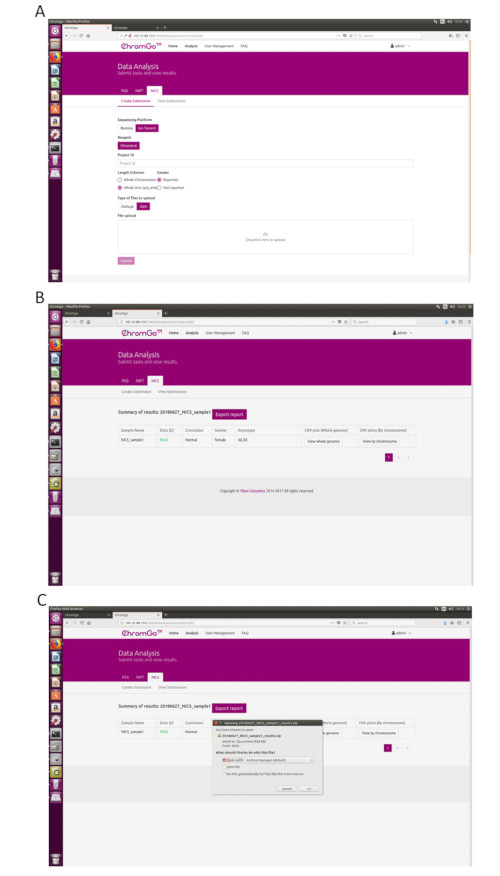
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
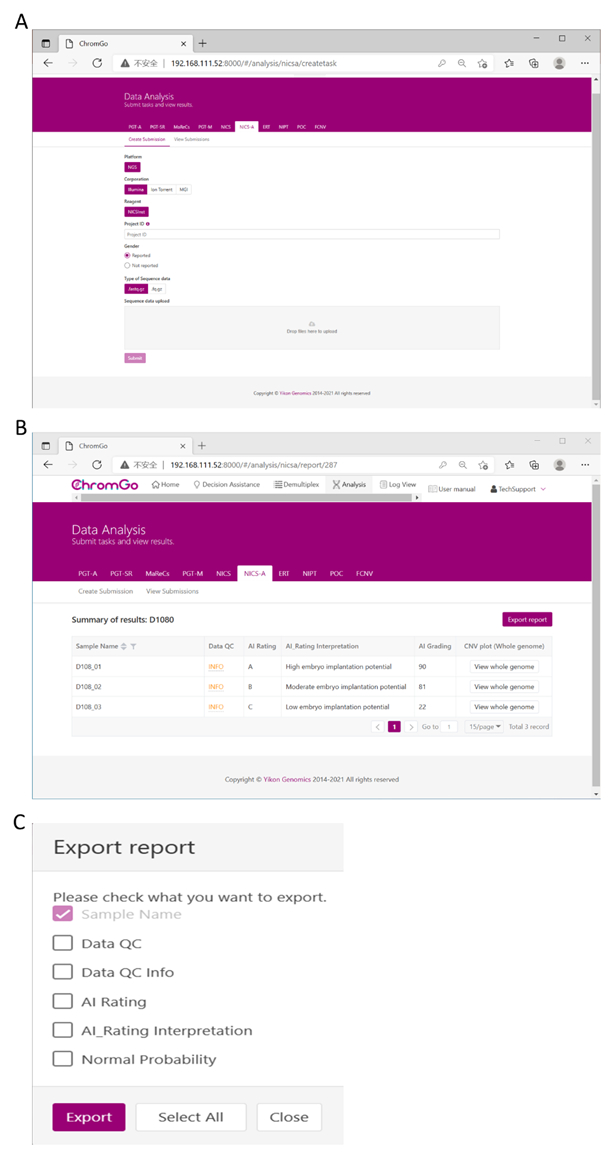
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.



