Transformação de células de E. coli usando um protocolo adaptado de cloreto de cálcio
English
Share
Overview
Fonte: Natalia Martin1, Andrew J. Van Alst1, Rhiannon M. LeVeque1, e Victor J. DiRita1
1 Departamento de Microbiologia e Genética Molecular, Universidade Estadual de Michigan
As bactérias têm a capacidade de trocar material genético (Ácido Desoxiribonucleico, DNA) em um processo conhecido como transferência de genes horizontais. A incorporação do DNA exógeno fornece um mecanismo pelo qual as bactérias podem adquirir novos traços genéticos que lhes permitem adaptar-se às mudanças nas condições ambientais, como a presença de antibióticos ou anticorpos (1) ou moléculas encontradas em habitats naturais (2). Existem três mecanismos de transferência genética horizontal: transformação, transdução e conjugação (3). Aqui vamos focar na transformação, na capacidade das bactérias de tirar DNA livre do meio ambiente. Em laboratório, o processo de transformação tem quatro etapas gerais: 1) Preparação de células competentes, 2) Incubação de células competentes com DNA, 3) Recuperação de células e 4) Revestimento das células para crescimento dos transformadores (Figura 1).
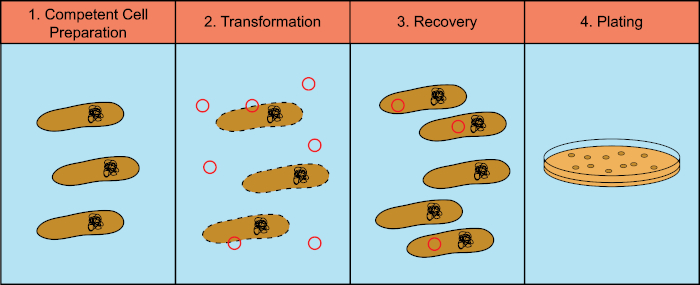
Figura 1: Etapas gerais do processo de transformação. O processo de transformação tem quatro etapas gerais: 1) Preparação de células competentes, 2) Incubação com DNA, 3) Recuperação das células e 4) Células de chapeamento para crescimento dos transformadores.
Para que a transformação ocorra, a bactéria receptora deve estar em um estado conhecido como competência. Algumas bactérias têm a capacidade de se tornarem naturalmente competentes em resposta a certas condições ambientais. No entanto, muitas outras bactérias não se tornam competentes naturalmente, ou as condições para esse processo ainda são desconhecidas. A capacidade de introduzir DNA em bactérias tem uma gama de aplicações de pesquisa: gerar múltiplas cópias de uma molécula de DNA de interesse, expressar grande quantidade de proteínas, como componente em procedimentos de clonagem, entre outros. Devido ao valor da transformação para a biologia molecular, existem vários protocolos que visam tornar as células artificialmente competentes quando as condições de competência natural são desconhecidas. Dois métodos principais são utilizados para preparar células artificialmente competentes: 1) através do tratamento químico das células e 2) expondo as células a pulsos elétricos (eletroporação). O primeiro usa diferentes produtos químicos dependendo do procedimento para criar atração entre o DNA e a superfície celular, enquanto o segundo usa campos elétricos para gerar poros na membrana celular bacteriana através da qual moléculas de DNA podem entrar. A abordagem mais eficiente para a competência química é a incubação com cátions divalent, mais notavelmente cálcio (Ca2+) (4,5) Competência induzida por cálcio é o procedimento que será descrito aqui (6). Este método é usado principalmente para a transformação de bactérias Gram-negativas, e esse será o foco deste protocolo.
O procedimento de transformação química envolve uma série de etapas em que as células são expostas a ceações para induzir a competência química. Essas etapas são posteriormente seguidas por uma mudança de temperatura – choque térmico – que favorece a absorção de DNA estranho pela célula competente (7). Os envelopes de células bacterianas são carregados negativamente. Em bactérias gram-negativas como escherichia coli,a membrana externa é carregada negativamente devido à presença de lipopólise (LPS) (8). Isso resulta em repulsa das moléculas de DNA igualmente carregadas negativamente. Na indução de competência química, íons de cálcio carregados positivamente neutralizam essa repulsão de carga permitindo a absorção de DNA na superfície celular (9). O tratamento de cálcio e a incubação com DNA são feitos no gelo. Posteriormente, é realizada uma incubação a temperaturas mais altas (42°C), o choque térmico. Esse desequilíbrio de temperatura favorece ainda mais a absorção de DNA. As células bacterianas precisam estar na fase de crescimento exponencial médio para suportar o tratamento de choque térmico; em outros estágios de crescimento, as células bacterianas são muito sensíveis ao calor, resultando em perda de viabilidade, o que diminui significativamente a eficiência da transformação.
Diferentes fontes de DNA podem ser usadas para transformação. Tipicamente, plasmídeos, pequenas moléculas circulares de DNA de dupla laga, são usados para transformação na maioria dos procedimentos laboratoriais em E. coli. Para que os plasmídeos sejam mantidos na célula bacteriana após a transformação, eles precisam conter uma origem da replicação. Isso permite que eles sejam replicados na célula bacteriana independentemente do cromossomo bacteriano. Nem todas as células bacterianas são transformadas durante o procedimento de transformação. Assim, a transformação produz uma mistura de células transformadas e células não transformadas. Para distinguir entre essas duas populações, é utilizado um método de seleção para identificar as células que adquiriram o plasmídeo. Plasmídeos geralmente contêm marcadores selecionáveis, que são genes codificando um traço que confere uma vantagem para o crescimento (ou seja, resistência a um antibiótico ou químico ou resgate de uma auxotrofia de crescimento). Após a transformação, as células bacterianas são banhadas em mídias seletivas, o que só permite o crescimento das células transformadas. No caso de células transformadas com uma resistência plasmid conferenciando a um dado antibiótico, a mídia seletiva será a mídia de crescimento contendo esse antibiótico. Vários métodos diferentes podem ser usados para confirmar que as colônias cultivadas nas mídias seletivas são transformadores (ou seja, incorporaram o plasmídeo). Por exemplo, plasmídeos podem ser recuperados dessas células usando métodos de preparação plasmídeos (10) e digeridos para confirmar o tamanho plasmídeo. Alternativamente, a COLÔNIA PCR pode ser usada para confirmar a presença do plasmídeo de interesse (11).
O objetivo deste experimento é preparar células quimicamente competentes E. coli DH5α, utilizando uma adaptação do procedimento de cloreto de cálcio (12), e transformá-las com o pUC19 plasmídeo para determinar a eficiência da transformação. A cepa E. coli DH5α é uma cepa comumente usada em aplicações de biologia molecular. Devido ao seu genótipo, especificamente o recA1 e o endA1,esta cepa permite maior estabilidade da inserção e melhorar a qualidade do DNA plasmídeo nas preparações subsequentes. Uma vez que a eficiência de transformação diminui com o aumento do tamanho do DNA, o plasmídeo pUC19 foi usado neste protocolo por causa de seu pequeno tamanho (2686 bp) (ver https://www.mobitec.com/cms/products/bio/04_vector_sys/standard_cloning_vectors.html para um mapa vetorial). pUC19 confere resistência à ampicilina e, portanto, este foi o antibiótico utilizado para seleção.
Procedure
Results
Although TE is dependent on many factors, non-commercial competent cell preparations, like this one, normally yield 106 to 107 transformants per microgram of plasmid. Therefore, this preparation, with a TE = 2.46 x 108 cfu/µg, yielded a TE well beyond the expected range. Additional protocols are available for making supercompetent cells when higher transformation efficiencies are required for a given application (13).
Analysis of the digestion of the plasmid DNA recovered from the transformed cells indicated that this plasmid has the expected size of pUC19 DNA (2686 bp).
Applications and Summary
Transformation is a powerful method for introducing exogenous DNA into bacterial cells that is key to many molecular biology applications in the laboratory. Additionally, it plays a major role in nature by allowing bacterial cells to exchange genetic material that could result in increased genetic variation and allow for acquisition of different beneficial traits for survival under a wide range of conditions. Many bacterial strains encode the genes required for natural competence. However, the conditions in which these genes are induced are still unknown. Further research is required to determine these conditions.
References
- Croucher, N. J. et al. Rapid pneumococcal evolution in response to clinical interventions. Science. 331 (6016):430-434. (2011)
- Borgeaud, S. et al. The type VI secretion system of Vibrio cholerae fosters horizontal gene transfer. Science. 347(6217):63-67. (2015)
- Burmeister, A. R. Horizontal Gene Transfer. Evol Med Public Health. 2015 (1):193-194. (2015)
- Weston A, Brown MG, Perkins HR, Saunders JR, Humphreys GO. Transformation of Escherichia coli with plasmid deoxyribonucleic acid: calcium-induced binding of deoxyribonucleic acid to whole cells and to isolated membrane fractions. J Bacteriol. 145 (2):780-7. (1981)
- Dagert M, Ehrlich SD. Prolonged incubation in calcium chloride improves the competence of Escherichia coli cells. Gene. 6 (1):23-8. (1979)
- Asif A, Mohsin H, Tanvir R, and Rehman Y. Revisiting the Mechanisms Involved in Calcium Chloride Induced Bacterial Transformation. Front Microbiol. 8:2169. (2017)
- Panja S, Aich P, Jana B, Basu T. How does plasmid DNA penetrate cell membranes in artificial transformation process of Escherichia coli? Mol Membr Biol. 25 (5):411-22. (2008)
- Silhavy, TJ, Kahne D, Walker S. The Bacterial Cell Envelope. Cold Spring Harb Perspect Biol. 2 (5): a000414. (2010)
- Panja S, Aich P, Jana B, Basu T. (2008) Plasmid DNA binds to the core oligosaccharide domain of LPS molecules of E. coli cell surface in the CaCl2-mediated transformation process. Biomacromolecules. 9 (9):2501-9.
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Plasmid Purification. JoVE, Cambridge, MA. (2018)
- Bergkessel M and Guthrie C. Colony PCR. Methods in Enzymology. 529: 299-309. (2013)
- Sambrook J and Russell DW. Molecular Cloning A Laboratory Manual. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY.Protocol 25 (1.116-118). (2001)
- Wirth R, Friesenegger A, Fiedler S. Transformation of various species of gram-negative bacteria belonging to 11 different genera by electroporation. Molecular & General Genetics. 216 (1): 175-7. (1989)
Transcript
Bacteria are remarkably adaptable and one mechanism which facilitates this adaptation is their ability to take in external DNA molecules. One type of DNA that bacteria can uptake is called a plasmid, a circular piece of DNA that frequently contains useful information, such as antibiotic resistance genes. The process of bacteria being modified by new genetic information incorporated from an external source is referred to as transformation. Transformation can easily be performed in the laboratory using Escherichia coli, or E. coli.
In order to be transformed, E. coli cells must first be made competent, which means capable of taking in DNA molecules from their environment. The protocol for accomplishing this is surprisingly simple, a short incubation of the cells in a calcium chloride solution. This incubation causes the cells to become permeable to DNA molecules. After the cells are pelleted by centrifugation, the supernatant is removed. The plasmid DNA is now added to the competent cells. After incubating the cells with DNA, the mix is briefly heated to 42 degrees Celsius, followed by rapid cooling on ice. This heat shock causes the DNA to be transferred across the cell’s wall and membranes. The cells are then incubated in fresh media. Then, the bacteria are placed at 37 degrees to allow them to reseal their membranes and express resistant proteins.
Those cells which have taken in the plasmids will faithfully copy the DNA and pass it to their progeny and express any proteins that might be encoded by it, including antibiotic resistance mediators. Those resistance genes can be used as selectable markers to identify bacteria which have been successfully transformed because cells that have not taken up the plasmid will not express the resistance gene product. This means that when the cells are plated on a solid medium which contains the appropriate antibiotic, only cells that have taken up the plasmid will grow. Transformation of the cells in a growing colony can be further confirmed by culturing those cells in liquid media overnight to increase the yield before extracting the DNA from the sample. Once the DNA is isolated, a diagnostic restriction enzyme digest can be carried out. Because restriction enzymes cut DNA in predictable locations, running these digests on a gel should show a predictable pattern if the desired plasmid was successfully transformed. For example, if pUC19 is prepared and cut with the restriction enzyme HindIII, a single band of 2686 nucleotides should be seen on the gel.
In this lab, you will transform E. coli strain DH-5 Alpha with pUC19, and then confirm the successful transformation by DNA gel electrophoresis.
Before starting the procedure, put on the appropriate personal protective equipment, including a lab coat and gloves. Next, sterilize the workspace with 70% ethanol.
Now, prepare chemically competent cells by depositing a loopfull of bacteria onto a sterile LB agar plate and streaking the bacteria with a new loop. Then, incubate the plate at 37 degrees Celsius overnight. The next day, sterilize the bench top with 70% ethanol again, and remove the plate from the incubator.
Inoculate a single, well-isolated colony into 3 milliliters of LB broth in a tube with a sterile loop. Then, grow the culture at 37 degrees Celsius overnight, with shaking at 210 RPM. The next day, measure the optical density of the overnight culture with a spectrophotometer. Then, add 100 milliliters of LB broth to a one-liter flask, and inoculate it with the overnight culture at an optical density of 0. 01. Now, incubate the culture at 37 degrees Celsius with shaking, and check the OD600 every 15 to 20 minutes until the culture reaches mid-exponential growth phase.
After approximately three hours, transfer 50 milliliters of the culture to two ice-cold polypropylene bottles. Then, place the bottles back on ice for 20 minutes to cool. Next, recover the cells via centrifugation. Discard the supernatants and place the bottles upside down on a paper towel. Next, resuspend the bacterial pellet in five milliliters of ice-cold calcium chloride magnesium chloride solution and swirl carefully until the pellet has dissolved completely. Then, add another 25 milliliters of the solution to the dissolved bacterial pellet. Resuspend the other bacterial pellet as previously demonstrated. After this, repeat the centrifugation, and remove the supernatants.
If the competent cells are going to be directly transformed, resuspend each bacterial pellet in two milliliters of an ice-cold 0.1 molar calcium chloride solution by swirling the tubes carefully. To begin the transformation procedure, transfer 50 microliters of competent cells to two labeled 1.5 milliliter polypropylene tubes. Then, add one microliter of pUC19 plasmid DNA to one of the tubes. Mix gently, avoiding bubble formation, and incubate both tubes for 30 minutes on ice. After incubation, transfer the tubes to a heat block and incubate at 42 degrees Celsius for 45 seconds. Immediately transfer the tubes to ice, and incubate for two minutes. Now, add 950 microliters of SOC media to each tube and incubate them for one hour at 37 degrees Celsius to allow the bacteria to recover, and express the antibiotic resistant marker encoded in the plasmid.
To make a 1 to 100 dilution, add 990 microliters of SOC media and 10 microliters of cell suspension to a 1.5 milliliter tube. Then, make a 1 to 10 dilution by adding 900 microliters of SOC media and 100 microliters of cell suspension to a 1.5 milliliter tube. Next, plate 100 microliters of the diluted cell suspensions and 100 microliters of the negative control, onto separate selective plates containing ampicillin using a spreader and incubate the plates at 37 degrees Celsius for 12 to 16 hours. After incubation, count the colony-forming units, or CFUs, per plate, obtained through transformation, and record these data. To verify that the transformants have the pUC19 plasmid, pick a single, well-isolated colony from a plate with a sterile loop, and introduce it to a tube containing 3 milliliters of LB broth. Then, incubate the culture at 37 degrees Celsius with shaking, overnight. The next day, use a DNA mini prep kit to isolate DNA from 3 milliliters of the culture, according to the manufacturer’s instructions. After completing the DNA mini prep, digest the 1 microgram of purified pUC19 with a restriction enzyme at 37 degrees Celsius for 1 hour. Now, load 20 microliters of a molecular weight ladder, 1 microgram of digested plasmid DNA, and 1 microgram of undigested plasmid DNA into consecutive wells of a 1% agarose gel containing 1 microgram per milliliter ethidium bromide. Then, run the gel for 1 hour at 95 volts. Finally, visualize the gel with a UV illuminator.
In this experiment, E. coli DH5 Alpha chemically competent cells were prepared using an adaptation of the calcium chloride procedure, and then transformed with the plasmid pUC19 to determine transformation efficiency. To calculate the transformation efficiency, use the recorded CFU counts for the 1 in 100 and 1 in 10 dilutions, and any other dilutions with CFU counts between 30 and 300. First, the recorded CFU count, 246 in this example, is divided by the amount of DNA, .0001 micrograms here, that was plated. Then, this number is divided by the dilution factor used to give the transformation efficiency in CFUs per microgram. In this example, a 1 to 10 dilution was used and 100 microliters of a 1 milliliter solution was plated, giving a final dilution factor of 0.01. In the undigested plasmid lane, the circular DNA may appear as two or three different bands of varying brightness. This is because the circular, uncut DNA may exist in several different conformation states, such as supercoiled, open circle, or more linear, and each of these move through the gel at different rates. Analysis of the recovered plasmid DNA digestion indicated that the plasmid used has an expected size of pUC19 DNA, 2,686 base pairs.
