Introduction
Phosphatidylethanolamine मिथाइल (PeMT) एंजाइमों monomethyl-पे, डाइमिथाइल-पीई और / या देने के लिए पीई, monomethyl पीई या डाइमिथाइल-पीई पर मिथाइल समूह दाता के रूप में एस -adenosylmethionine (एसएएम) का उपयोग कर एक या एक से अधिक मिथाइल समूहों के सहसंयोजक लगाव को उत्प्रेरित phosphatidylcholine (पीसी)। इन एंजाइमों पशु कोशिकाओं और कवक में लगभग हर जगह होते हैं। उन्होंने यह भी कुछ पौधों 1 और बैक्टीरिया का लगभग 10%, यूकेरियोट्स 2 के साथ बातचीत है कि विशेष रूप से उन लोगों में पाया जा सकता है।
PEMTs ही नहीं, बल्कि अन्य महत्वपूर्ण सेलुलर कार्यों को पूरा करने से पशु कोशिकाओं में मुख्य लिपिड वर्ग है, जो पीसी के उत्पादन में योगदान करके सेल के जीव विज्ञान के लिए प्रासंगिक हैं। स्तनधारियों में, PEMTs मुख्य रूप से वे बहुत कम घनत्व वाले लिपोप्रोटीन के सामान्य स्राव के लिए आवश्यक हैं जहां जिगर में व्यक्त कर रहे हैं और वे भी आहार प्रेरित मोटापा 3, atherosclerosis 4 में योगदान है, और इंसुलिन का विरोधमंजूरी 5। इसके अतिरिक्त, स्तनधारी PeMT भी हालांकि निचले स्तर पर, adipocytes में व्यक्त की, और वसा के जमाव 6, 7 में भाग रहे हैं। कैंसर के विकास 8, एपोप्टोसिस 9, और सेल के विकास को 10 में PeMT भूमिका भी प्रदर्शन किया गया है। बैक्टीरिया में, PeMT एंजाइमों मेजबान संयंत्र 11 के साथ सामान्य कोशिकाओं के विकास 2, डाह 2, और सहजीवन के लिए महत्वपूर्ण होने के लिए दिखाया गया है।
वर्तमान प्रोटोकॉल का लक्ष्य और तर्क एंजाइम को शुद्ध करने के लिए आवश्यकता के बिना पूरे सेल के अर्क से PeMT गतिविधि को मापने के लिए है। दो अलग प्रोटोकॉल PeMT गतिविधि को मापने के लिए विकसित किया गया है। पहला और सबसे आम एक इस लेख का विषय है जो पीई, पर रेडियोधर्मी सैम से tritiated मिथाइल समूह के हस्तांतरण के उपाय। इस प्रोटोकॉल मूल रूप से एक समझ हासिल करने के लिए खमीर 12 और स्तनधारी कोशिकाओं (यकृत) 13 से PeMT गतिविधि को मापने के लिए विकसित किया गया हैइन कोशिकाओं में पीसी जैवसंश्लेषण के Anding के साथ-साथ इन एंजाइमों की विशिष्टता निर्धारित करने के लिए। बाद में, इस तकनीक को और प्रोटोजोआ परजीवी 14 (15 यद्यपि परख के लिए एक बुनियादी पीएच मान का उपयोग) में इस तरह के बैक्टीरिया के रूप में 2 अन्य प्रकार की कोशिकाओं के लिए लागू किया गया है। इस तकनीक को पूरे सेल अर्क के रूप में अच्छी तरह से शुद्ध एंजाइम के साथ इस्तेमाल किया जा सकता है, और संभवतः किसी भी सेल निकालने प्रणाली को लागू किया जा सकता है। एक गैर रेडियोधर्मी परख भी एस -adenosylhomocysteine, सैम 16 की transmethylation उत्पाद के enzymatic मात्रा का ठहराव पर निर्भर करता है कि डिजाइन किया गया है। यह रेडियोधर्मिता को शामिल नहीं करता उत्तरार्द्ध परख और अधिक सुविधाजनक हो सकता है लेकिन यह शुद्ध एंजाइमों के लिए ही उपयुक्त है।
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. सेल निकालने की तैयारी
- 20 मिमी HEPES pH7.4, 100 यू / एमएल पेनिसिलिन, / एमएल स्ट्रेप्टोमाइसिन, 5 माइक्रोग्राम / एमएल हीम 100 माइक्रोग्राम के साथ पूरक 1x M199 के बने एक माध्यम में 26 डिग्री सेल्सियस पर हवा तंग टोपी के साथ बंद एक बाँझ प्लास्टिक की बोतल में Leishmania कोशिकाओं को विकसित मिलाते हुए बिना, 0.35 ग्राम / एल नाको 2 एच, 0.1 मिमी एडिनाइन, और 2 माइक्रोग्राम / एमएल biopterin। वे 1-2 10 x 7 / एमएल के एक सेल घनत्व तक पहुँचने जब 4 डिग्री सेल्सियस पर 5 मिनट के लिए 1,500 ग्राम पर centrifugation द्वारा कोशिकाओं फसल।
- एक विंदुक के साथ सतह पर तैरनेवाला त्यागें और ठंड फॉस्फेट बफर खारा की संस्कृति की मात्रा (137 मिमी NaCl, 2.7 मिमी KCl की छमाही में एक सीरम वैज्ञानिक pipet के साथ सेल गोली resuspending द्वारा कोशिकाओं को धोने, 10 मिमी ना 2 4 HPO, 1.8 मिमी 2 के.एच. पीओ 4, pH7.4)। BL2 सुरक्षा के दिशा निर्देशों के अनुसार सेल supernatants फेकें।
- अपकेंद्रित्र कोशिकाओं को फिर से 4 डिग्री सेल्सियस पर 5 मिनट के लिए 1,500 ग्राम पर। एक विंदुक के साथ सतह पर तैरनेवाला त्यागें। टी आगे बढ़ेंया अगले चरण ओ -80 डिग्री सेल्सियस (अप करने के लिए तीन महीने) में लंबी अवधि के भंडारण के लिए तरल नाइट्रोजन में सेल गोली फ्रीज तस्वीर।
- 2x lysis बफर (0.5 एम सुक्रोज, 0.1 एम TrisHCl, PH7.5, 2 मिमी EDTA, और 2x protease अवरोध कॉकटेल) तैयार है और बर्फ पर 4 डिग्री सेल्सियस पर रहते हैं।
- 2x lysis बफर के बराबर मात्रा में (ताजा या फ्रोजन) सेल गोली Resuspend। कांच के मोती की 1x मात्रा में जोड़ें। भंवर सख्ती 10 मिनट के लिए 4 डिग्री सेल्सियस पर।
- 1x lysis बफर के 2 संस्करणों और मिश्रण जोड़ें। 10 मिनट के लिए 4 डिग्री सेल्सियस पर 1,500 XG पर अपकेंद्रित्र सेल के अर्क अटूट कोशिकाओं और नाभिक गोली।
- एक ताजा शांत अपकेंद्रित्र ट्यूब में एक pipet के साथ सतह पर तैरनेवाला स्थानांतरण और प्रयोग के पूरा होने तक बर्फ पर सेल के अर्क रहते हैं।
2. इस तरह Bicinchoninic एसिड परख के रूप में प्रोटीन आकलन किट का उपयोग सेल निकालने के प्रोटीन एकाग्रता का निर्धारण
- Bicinchoninic एसिड तैयार (बीसीए) समाधान बीसीए और तांबे (द्वितीय) के मिश्रण से (1 मिलीग्राम / ट्यूब) एस49 के अनुपात में ulfate: 1 (वी / वी)।
- के प्रोटीन मानकों को तैयार 0, 10, 20, 30, 40, 50, और बीसीए समाधान के 1 मिलीलीटर aliquots में एक 10 मिलीग्राम / एमएल गोजातीय सीरम albumin (बीएसए) शेयर समाधान गिराए द्वारा 60 माइक्रोग्राम / एमएल।
- डुप्लिकेट में बीसीए समाधान के 1 मिलीलीटर में कोशिकाओं के अर्क के 2 μl जोड़ें। एक पूर्व गर्म 60 डिग्री सेल्सियस पानी के स्नान में 10 मिनट के लिए मानकों और प्रोटीन के नमूने सेते हैं।
- स्थानांतरण के नमूने 3 मिनट के लिए बर्फ करने के लिए। 562 एनएम के तरंग दैर्ध्य में एक स्पेक्ट्रोफोटोमीटर के साथ मानकों और प्रोटीन के नमूने के absorbance के उपाय।
- निर्माता प्रोटोकॉल में वर्णित के रूप में एक संदर्भ के रूप में बीएसए मानक का उपयोग सेल के अर्क के प्रोटीन एकाग्रता की गणना। 1x lysis बफर के साथ मिलीलीटर 10 मिलीग्राम / के एक प्रोटीन एकाग्रता के लिए सेल के अर्क पतला।
ट्यूब प्रति 200 μl में 3. एंजाइमी परख
नोट: एक रासायनिक हुड में निम्नलिखित कदम ले।
- एक 15 मिलीलीटर में दो प्रतियों में प्रत्येक नमूने के परीक्षणशंक्वाकार ट्यूब। ट्यूब प्रति 20 μl 1 एम TrisHCl पीएच 7.5 तैयार है और बर्फ पर रहते हैं। प्रत्येक ट्यूब के लिए आरटी पर;: (समाधान रोक 1 (वी / वी) 1) क्लोरोफॉर्म / मेथनॉल के 2 मिलीलीटर तैयार करें।
- Pipet बर्फ पर प्रत्येक 15 मिलीलीटर शंक्वाकार ट्यूब में 1 एम TrisHCl पीएच 7.5 से 20 μl।
- यहाँ से विकिरण सुरक्षा निर्देशों का पालन करें। सैम के 50.06 माइक्रोन की कुल के लिए ट्यूब प्रति [मिथाइल 3 एच] adenosyl- एल-मेथिओनिन और 50 माइक्रोन ठंड सैम - 0.06 माइक्रोन (0.2 μCi) एस के बराबर जोड़ें। ठंडे पानी की μl एक्स जोड़ें जहां एक्स = 200 (20 (+ सेल के अर्क के लिए 20 () + ठंड और रेडियोधर्मी सैम की मात्रा) बफर के लिए) ट्यूब प्रति।
- एक पूर्व गर्म 30 डिग्री सेल्सियस पानी के स्नान करने के लिए प्रत्येक शंक्वाकार ट्यूब स्थानांतरण। रिएक्शन शुरू करने के लिए प्रत्येक ट्यूब सेल अर्क (प्रोटीन की 200 ग्राम के बराबर) के 20 μl जोड़ें। वांछित समय (0-45 मिनट) के लिए सेते हैं।
- क्लोरोफॉर्म / मेथनॉल के 2 मिलीलीटर जोड़कर प्रतिक्रिया बंद करो (1: 1; वी / वी; रोक समाधान) प्रत्येक ट्यूब। (आरटी के लिए 20-25 शंक्वाकार ट्यूब स्थानांतरण6; सी)।
4. लिपिड निष्कर्षण
नोट: एक रासायनिक हुड में निम्नलिखित कदम ले।
- Enzymatic प्रतिक्रिया नमूना युक्त प्रत्येक ट्यूब करने के लिए पानी के 700 μl जोड़ें। सख्ती 30 सेकंड के लिए भंवर। आरटी पर 5 मिनट के लिए 1,500 XG पर अपकेंद्रित्र पानी चरण से जैविक अलग करने के लिए।
- एक विंदुक के साथ एक नया 15 मिलीलीटर शंक्वाकार ट्यूब में कम जैविक चरण स्थानांतरण। 30 सेकंड के लिए सख्ती ट्यूब और भंवर युक्त प्रत्येक "कम चरण" करने के लिए पानी की 1 मिलीलीटर जोड़ें। अपकेंद्रित्र फिर 5 मिनट के लिए 1,500 XG पर पानी चरण से जैविक अलग करने के लिए।
- एक विंदुक के साथ एक जगमगाहट ट्यूब में कम जैविक चरण स्थानांतरण। एन 2 की एक धारा के तहत सूखी नमूने हैं। गैर निगमित रेडियोधर्मी सैम और विकिरण दिशानिर्देशों के अनुसार रेडियोधर्मी शंक्वाकार ट्यूबों युक्त पानी चरणों फेकें।
- 2 मिलीग्राम / जगमगाहट तरल की ट्यूब जोड़ें। एक जगमगाहट cou के साथ शामिल रेडियोधर्मिता मापनेnter 'निर्माताओं प्रोटोकॉल और साधन के उपयोग के अनुसार।
- निम्नलिखित सामान्य समीकरण का उपयोग nmol / मिलीग्राम प्रोटीन में enzymatic गतिविधि की गणना:
सीपीएम मूल्य 10 x 3 एक्स [सैम (मिमी) कुल (रेडियोधर्मी और ठंडे) एकाग्रता] रेडियोधर्मी सैम (सीआइ / mmol) x का एक्स 5 विशिष्ट गतिविधि [रेडियोधर्मी सैम की एकाग्रता (मिमी)]
नोट: ऊपर प्रोटोकॉल निर्भर एक सैम एकाग्रता या एक प्रोटीन निर्भर PeMT परख के लिए बदला जा सकता है। सैम एकाग्रता निर्भर एंजाइमी परख के लिए, समय, जबकि प्रोटीन निर्भर PeMT परख, सैम एकाग्रता के लिए परख करने के लिए जोड़ रहे हैं निरंतर (रैखिक सीमा में है, जो 15 मिनट) और ठंड सैम के विभिन्न मात्रा में रखा जाता है (हम मिमी 0.05 चुना) और समय निरंतर (15 मिनट) रखा जाता है। इसके अलावा बफर के पीएच कुछ PeMT एंजाइमों एक अलग इष्टतम पीएच मान है, तो जरूरत के रूप में बदला जा सकता है।
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
चित्रा 1 एक सब्सट्रेट के रूप में अंतर्जात पीई का उपयोग कर एक एंजाइम स्रोत के रूप में Leishmania पूरे सेल निकालने के साथ बाहर किया गया था, जो एक समय निर्भर PeMT परख को दिखाती है। जैविक चरण में रेडियोधर्मिता की मात्रा को जगमगाहट गिनती द्वारा मात्रा था। जिसके परिणामस्वरूप संख्या पीई पर स्थानांतरित tritiated मिथाइल समूहों की राशि की गणना करने के लिए उपयोग किया गया। PeMT गतिविधि लगभग 20 मिनट के लिए रैखिक था। यह तो यह एक और 15 मिनट के लिए स्थिर रहा, जिसके बाद लगभग 30 मिनट पर एक पठार पर पहुंच गया। जैसी कि उम्मीद थी कोई सेल अर्क परख (चित्रा 2) को जोड़ा गया था, जब PeMT गतिविधि नहीं पाया गया। इसके अलावा, इस गतिविधि एल के एक अवरोध है, जो 100 माइक्रोन octadecyltrimethylammonium ब्रोमाइड की उपस्थिति में समाप्त कर दिया गया था प्रमुख पीई Methyltransferases Lmj PEM1 और Lmj PEM2 14। PeMT गतिविधि भी प्रोटीन एकाग्रता dependen थाटी, और इस गतिविधि एंजाइमी परख (चित्रा 3) के लिए आवेदन किया प्रोटीन की मात्रा को रेखीय समानुपाती था। अन्त में, एक सैम एकाग्रता निर्भर PeMT परख जिसमें सैम की बढ़ती सांद्रता (चित्रा 4) का परीक्षण किया गया, बाहर किया गया था। PeMT गतिविधि लगभग 0.5 मिमी के सैम एकाग्रता में एक पठार पर पहुंच गया। सब एक साथ, इन चार assays के PeMT गतिविधि विशिष्ट है और एंजाइम (एस) को शुद्ध करने के लिए आवश्यकता के बिना पूरे सेल के अर्क से मापा जा सकता है कि प्रदर्शित करता है।
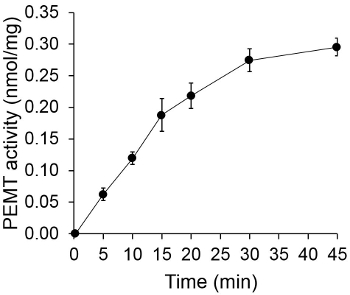
चित्रा 1. समय निर्भर PeMT परख। एंजाइमी परख समय के एक समारोह के रूप में पूरे Leishmania सेल के अर्क का 0.2 मिलीग्राम के साथ दो प्रतियों में दो बार प्रदर्शन किया गया था। PeMT गतिविधि प्रोटीन और पी के मिलीग्राम प्रति पीई पर स्थानांतरित nmol मिथाइल समूहों के रूप में प्रतिनिधित्व किया हैएर घंटा। सैम युक्त परख समाधान के लिए जोड़ा जा रहा से पहले: (मात्रा से 1, 1) समय 'ओ' के लिए, सेल के अर्क पहले क्लोरोफॉर्म / मेथनॉल से बना रोक समाधान के 2 मिलीलीटर के साथ मिलाया गया। मानक विचलन दिखाए जाते हैं।
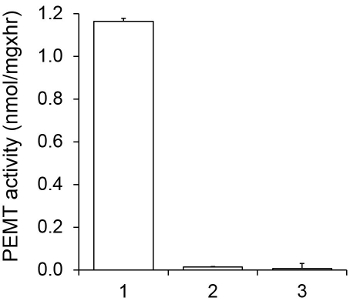
PeMT परख 0.05 मिमी सैम की उपस्थिति में 15 मिनट के लिए दो प्रतियों में दो बार बाहर किया गया था विशिष्टता PeMT गतिविधि। चित्रा 2.। 1, 0.2 मिलीग्राम प्रोटीन निकालने; 2, कोई सेल निकालने; 3, 0.2 मिलीग्राम प्रोटीन और 0.1 मिमी octadecyltrimethylammonium ब्रोमाइड। मानक विचलन दिखाए जाते हैं।
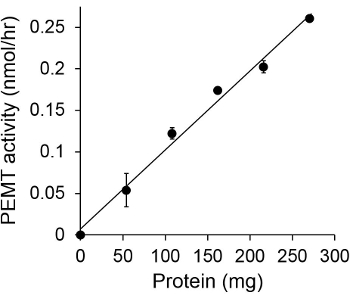
चित्रा 3. प्रोटीन निर्भर PeMT परख। एंजाइमी परख में दो बार प्रदर्शन किया गया था15 मिनट के लिए 0.05 मिमी सैम के साथ अनुपस्थिति में (बिंदु 'ओ') या Leishmania प्रोटीन के विभिन्न मात्रा की उपस्थिति नकली। मानक विचलन दिखाए जाते हैं।
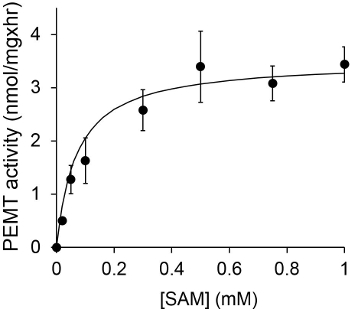
चित्रा 4. PeMT परख 15 मिनट के लिए सैम के विभिन्न सांद्रता की उपस्थिति में पूरे Leishmania सेल के अर्क का 0.2 मिलीग्राम के साथ दो प्रतियों में दो बार बाहर किया गया था। मानक विचलन दिखाए जाते हैं।
Subscription Required. Please recommend JoVE to your librarian.
Discussion
यह सरल, त्वरित PeMT परख एक प्रोटीन के स्रोत के रूप में पूरे सेल निकालने का उपयोग पीई पर सैम से रेडियोधर्मी मिथाइल समूहों के हस्तांतरण का परिणाम है कि पीई के methylated रूपों की मात्रा का ठहराव की अनुमति देता है। यह तेजी से संवेदनशील, प्रतिलिपि प्रस्तुत करने योग्य, और शुद्ध एंजाइमों 17 के लिए भी उपयुक्त है। ब्याज की मिथाइल बल्कि पीई 12,13,18,19 करने से इन substrates के लिए विशिष्ट है, तो Monomethyl- या डाइमिथाइल-पीई परख करने के लिए जोड़ा जा सकता है। शुद्ध PeMT एंजाइम का इस्तेमाल किया जाता है, तो पीई परख करने के लिए जोड़ा जा सकता है। इस प्रोटोकॉल की एक सीमा परख प्रतिक्रियाओं उत्पादों (monomethyl-पे, डाइमिथाइल-पे, या पीसी) की पहचान नहीं करता है। 20,21 में वर्णित के रूप में हालांकि, प्रतिक्रिया उत्पादों (monomethyl-पे, डाइमिथाइल-पे, पीसी) की पहचान को आगे एक आयामी पतली परत क्रोमैटोग्राफी द्वारा विश्लेषण किया जा सकता है। इसके अलावा, इस तरह के एस -adenosylhomocysteine और 5'-methylthioadenosine के रूप में सैम गिरावट उत्पादों के कुछ PeMT गतिविधि ख बाधित कर सकते हैंY प्रतिक्रिया निषेध। हालांकि, Leishmania एक एस -adenosylhomocysteine hydrolase 22 के पास जो cleaves एस -adenosylhomocysteine एडिनाइन और एस -ribosylhomocysteine, और एडिनाइन और methylthioribose-1-फॉस्फेट 23 पैदा करता है जो एक methylthioadenosine phosphorylase में। हालांकि, यह एस -adenosylhomocysteine hydrolase और methylthioadenosine phosphorylase गतिविधियों PeMT गतिविधि का कोई निषेध होता है कि इतनी कुशलता, क्रमश: एस -adenosylhomocysteine और methylthioadenosine metabolize करने के लिए पर्याप्त उच्च रहे हैं कि क्या ज्ञात नहीं है। उदाहरण में संबंधित शुद्ध, पुनः संयोजक एंजाइमों के अलावा सैम गिरावट उत्पादों 24,25 से प्रतिक्रिया निषेध राहत देने के लिए परख करने के लिए जोड़ा जा सकता है ब्याज की सेल में अनुपस्थित -adenosylhomocysteine hydrolase और / या 5'-methylthioadenosine चयापचय एंजाइम होते हैं, एस, 26।
इस प्रोटोकॉल में चार महत्वपूर्ण कदम उठाए हैं: मैं)protease अवरोध कॉकटेल पाउडर बस का उपयोग करने से पहले बफर करने के लिए (1.1 कदम) से जोड़ा जाना है; द्वितीय) के अर्क) 1.4 चरण के बाद की तैयारी (के बाद निम्नलिखित घंटे के भीतर उपयोग किया जा करने के लिए कर रहे हैं पूरे सेल; iii) लिपिड निष्कर्षण कदम (4.2 और 4.4 कदम), सावधानी अतिरिक्त tritiated सैम होता है जो अंतरावस्था या पानी चरण के किसी भी स्थानांतरण नहीं करने के लिए लागू किया जाना चाहिए, और चतुर्थ) रेडियोधर्मी एस विभाज्य दौरान - [मिथाइल 3 एच] adenosyl- एल-मेथिओनिन और फ्रीज के दोहराया चक्र के रूप में प्राप्त होने पर ठंड अभिकर्मक और की कमी के लिए खाते सकता है जो एडिनाइन और एस -pentosylmethionine 26,27,28 को हाइड्रोलिसिस द्वारा पीछा 5'-methylthioadenosine और homoserine लैक्टोन में यह नीचा पिघलना औसत दर्जे का PeMT गतिविधि। बुरा पूरे सेल अर्क भी कोई enzymatic गतिविधि के लिए जिम्मेदार हो सकता है। इस मामले में, सेल निकालने की गुणवत्ता में एक और नाम से जाना जाता enzymatic गतिविधि को मापने के द्वारा मूल्यांकन किया जा सकता है। PeMT गतिविधि के पिछले है, कमी PeMT एन पर भरोसा कर सकते हैंzyme monomethyl पीई या डाइमिथाइल-पीई बजाय पीई के लिए विशिष्ट किया जा रहा है। Monomethyl पीई और / या परख करने के लिए डाइमिथाइल-पीई substrates के अलावा PeMT गतिविधि बहाल हो सकती है।
एक वैकल्पिक, गैर रेडियोधर्मी सैम निर्भर मिथाइल परख homocysteine के एंजाइमी मात्रा का ठहराव, सैम 29 की transmethylation उत्पादों पर निर्भर करता है कि विकसित किया गया है। इस प्रोटोकॉल सफलतापूर्वक पीई विशिष्ट मिथाइल गतिविधि 16 को मापने के लिए लागू किया गया है। दुर्भाग्य से, इस परख पूरे सेल निकालने के लिए केवल शुद्ध एंजाइम के लिए उपयुक्त है और नहीं है, और या तो प्रतिक्रिया उत्पादों की पहचान का खुलासा नहीं करता।
वर्तमान प्रोटोकॉल संभवतः किसी भी प्रकार की कोशिका के लिए लागू किया जा सकता है। साथ ही, इस परख आहार प्रेरित atherosclerosis के खिलाफ जीवाणु संक्रमण या उपन्यास चिकित्सा लड़ने के लिए नए विरोधी माइक्रोबियल यौगिकों के परीक्षण के संदर्भ में ब्याज की एक PeMT के लिए विशिष्ट संभावित दवाओं का परीक्षण करने के लिए इस्तेमाल किया जा सकता है, मोटापा, और इंसुलिन प्रतिरोध।
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
कोई हितों के संघर्ष की घोषणा नहीं
Materials
| Name | Company | Catalog Number | Comments |
| S-[Methyl-3H]adenosyl-L-methionine (specific activity of 5-15 Ci/mMole) | Perkin Elmer | NET155050UC | Aliquot the reagent and freeze at -20 °C; follow radiation safety guidelines while using this reagent |
| Protease inhibitor cocktail | Roche Life Sciences | 11836170001 | dilute it fresh |
| Glass beads, acid washed, 425-600 mm | Sigma Aldrich | G8772 | |
| Bicinchoninic acid solution | Sigma Aldrich | B9643 | |
| Copper (II) sulfate | Sigma Aldrich | C2284 | |
| Scintillation counter MicroBeta2 with 1-detector | Perkin Elmer | 2450-0010 | |
| Spectrophotometer Biomate 3 | Thermo Scientific | 840208300 | |
| BSA stock solution (10 mg/ml) | New England Biolabs | B9001S | |
| Scintillation liquid | Research Product International Corp | 111198 | |
| S-(5'-Adenosyl)-L-methionine chloride (hydrochloride) | Cayman Chemicals | 13956 | dilute the reagent in 20 mM HCl and freeze aliquots at -80 °C |
References
- Keogh, M. R., Courtney, P. D., Kinney, A. J., Dewey, R. E. Functional characterization of phospholipid N-.methyltransferases from Arabidopsis and soybean. J Biol Chem. 284 (23), 15439-15447 (2009).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim Biophys Acta. 1831 (3), 503-513 (2013).
- Gao, X., et al. Decreased lipogenesis in white adipose tissue contributes to the resistance to high fat diet-induced obesity in phosphatidylethanolamine N-.methyltransferase-deficient mice. Biochim Biophys Acta. 1851 (2), 152-162 (2015).
- Zhao, Y., et al. Lack of phosphatidylethanolamine N-.methyltransferase alters plasma VLDL phospholipids and attenuates atherosclerosis in mice. Arterioscler Thromb Vasc Biol. 29 (9), 1349-1355 (2009).
- Vance, D. E. Phospholipid methylation in mammals: from biochemistry to physiological function. Biochim Biophys Acta. 1838 (6), 1477-1487 (2014).
- Nishimaki-Mogami, T., Suzuki, K., Takahashi, A. The role of phosphatidylethanolamine methylation in the secretion of very low density lipoproteins by cultured rat hepatocytes: rapid inhibition of phosphatidylethanolamine methylation by bezafibrate increases the density of apolipoprotein B48-containing lipoproteins. Biochim Biophys Acta. 1304 (1), 21-31 (1996).
- Noga, A. A., Zhao, Y., Vance, D. E. An unexpected requirement for phosphatidylethanolamine N-.methyltransferase in the secretion of very low density lipoproteins. J Biol Chem. 277 (44), 42358-42365 (2002).
- Li, D., et al. Epigenetic repression of phosphatidylethanolamine N-.methyltransferase (PEMT) in BRCA1-mutated breast cancer. Oncotarget. 5 (5), 1315-1325 (2014).
- Cui, Z., Houweling, M., Vance, D. E. Suppression of rat hepatoma cell growth by expression of phosphatidylethanolamine N-.methyltransferase-2. J Biol Chem. 269 (40), 24531-24533 (1994).
- Cui, Z., Shen, Y. J., Vance, D. E. Inverse correlation between expression of phosphatidylethanolamine.N-.methyltransferase-2 and growth rate of perinatal rat livers. Biochim Biophys Acta. 1346 (1), 10-16 (1997).
- Minder, A. C., de Rudder, K. E., Narberhaus, F., Fischer, H. M., Hennecke, H., Geiger, O. Phosphatidylcholine levels in.Bradyrhizobium japonicum. membranes are critical for an efficient symbiosis with the soybean host plant. Mol Microbiol. 39 (5), 1186-1198 (2001).
- Kodaki, T., Yamashita, S. Yeast phosphatidylethanolamine methylation pathway. Cloning and characterization of two distinct methyltransferase genes. J Biol Chem. 262 (32), 15428-15435 (1987).
- Tanaka, Y., Amano, F., Maeda, M., Nishijima, M., Akamatsu, Y. Purification and properties of phosphatidyl-N-.monomethylethanolamine N-.methyltransferase, the enzyme catalyzing the second and the third steps in the phosphatidylethanolamine N-.methyltransferase system, from mouse liver microsomes. Jpn J Med Sci Biol. 43 (3), 59-73 (1990).
- Bibis, S. S., Dahlstrom, K., Zhu, T., Zufferey, R. Characterization of Leishmania major phosphatidylethanolamine methyltransferases LmjPEM1 and LmjPEM2 and their inhibition by choline analogs. Mol Biochem Parasitol. 196 (2), 90-99 (2014).
- deRudder, K. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti. mutants deficient in phospholipid N-.methyltransferase still contain phosphatidylcholine. J Bacteriol. 179 (22), 6921-6928 (1997).
- Aktas, M., Narberhaus, F. In vitro characterization of the enzyme properties of the phospholipid N-.methyltransferase PmtA from Agrobacterium tumefaciens. J Bacteriol. 191 (7), 2033-2041 (2009).
- Ridgway, N. D., Vance, D. E. Phosphatidylethanolamine N-.methyltransferase from rat liver. Methods Enzymol. 209, 366-374 (1992).
- Gaynor, P. M., Carman, G. M. Phosphatidylethanolamine methyltransferase and phospholipid methyltransferase activities from Saccharomyces cerevisiae. Enzymological and kinetic properties. Biochim Biophys Acta. 1045 (2), 156-163 (1990).
- Arondel, V., Benning, C., Somerville, C. R. Isolation and functional expression in Escherichia coli. of a gene encoding phosphatidylethanolamine methyltransferase (EC 2.1.1.17) from Rhodobacter sphaeroides. J Biol Chem. 268 (21), 16002-16008 (1993).
- Wessel, M., Klusener, S., Godeke, J., Fritz, C., Hacker, S., Narberhaus, F. Virulence of Agrobacterium tumefaciens. requires phosphatidylcholine in the bacterial membrane. Mol Microbiol. 62 (3), 906-915 (2006).
- Klusener, S., Aktas, M., Thormann, K. M., Wessel, M., Narberhaus, F. Expression and physiological relevance of Agrobacterium tumefaciens. phosphatidylcholine biosynthesis genes. J Bacteriol. 191 (1), 365-374 (2009).
- Henderson, D. M., et al. Cloning of the gene encoding Leishmania donovani.S.-adenosylhomocysteine hydrolase, a potential target for antiparasitic chemotherapy. Mol Biochem Parasitol. 53 (1-2), 169-183 (1992).
- Koszalka, G. W., Krenitsky, T. A. 5'-Methylthioadenosine (MTA) phosphorylase from promastigote of Leishmania donovani. Purine and Pyrimidine Metabolism in Man V, Adv Exp Med Biol. Nyhan, W. L., Thompson, L. F., Watts, R. W. E. 131, Springer US. 559-563 (1986).
- Biastoff, S., Teuber, M., Zhou, Z. S., Dräger, B. Colorimetric activity measurement of a recombinant putrescine N.-methyltransferase from Datura stramonium. Planta Med. 72 (12), 1136-1141 (2006).
- Hendricks, C. L., Ross, J. R., Pichersky, E., Noel, J. P., Zhou, Z. S. An enzyme-coupled colorimetric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 326 (1), 100-105 (2004).
- Cannon, L. M., Butler, F. N., Wan, W., Zhou, Z. S. A stereospecific colorimetric assay for (S.,S.)-adenosylmethionine quantification based on thiopurine methyltransferase-catalyzed thiol methylation. Anal Biochem. 308 (2), 358-363 (2002).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S.-adenosyl-L-methionine. Biochemistry. 25 (15), 4444-4449 (1986).
- Wu, S. E., Huskey, W. P., Borchardt, R. T., Schowen, R. L. Chiral instability at sulfur of S.-adenosylmethionine. Biochemistry. 22 (12), 2828-2832 (1983).
- Dorgan, K. M., et al. An enzyme-coupled continuous spectrophotometric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 350 (2), 249-255 (2006).



